黄酒不仅是世界三大古酒之一,在我国亦有悠久的历史,且深受人们的喜爱[1]。房县黄酒作为我国黄酒的重要分支,起于周、而盛于唐,有“御酒”和“皇酒”之称。此外,作为国家地理标志保护产品,房县黄酒酿造工艺亦已入选湖北省非物质文化遗产[2]。房县黄酒以糯米为原料,经蒸煮、晾凉和接入当地制作的黄酒加工用小曲发酵而成。房县黄酒加工用小曲实质上为米酒曲,其以糯米为原料辅以辣蓼草(Polygonum lapathifolium L.var.salicifolium Sihbth)制作而成。目前,房县黄酒大多仍采用家庭式生产,而制曲工艺和制作环境的不同导致小曲的品质存在较大差异。酿酒行业自古以来就有“曲,酒之骨”的说法[3-4],这说明了小曲对于房县黄酒品质的重要性。由此可见,解析房县黄酒加工用小曲中微生物的构成和多样性对后续房县黄酒品质的提升具有积极意义。
针对米酒曲中微生物多样性的研究是一个逐渐发展的过程,宁亚丽等[5]通过纯培养技术发现吉林延边朝鲜族地区米酒曲中的优势细菌隶属于乳杆菌属(Lactobacillus)和肠杆菌属(Enterobacter);王艳萍等[6]通过分离鉴定确定了毛霉属(Mucor)是米酒发酵过程中的优势真菌属;而向凡舒等[3]通过MiSeq高通量测序技术对建始地区米酒曲中细菌和真菌多样性解析,发现建始地区米酒曲具有较高的微生物多样性,细菌以假单胞菌属(Pseudomonas)、片球菌属(Pediococcus)、魏斯氏菌属(Weissella)、芽孢杆菌属(Bacillus)、肠杆菌属(Enterobacter)、泛菌属(Pantoea)、克雷伯菌属(Klebsiella)和乳球菌属(Lactococcus)为主,而真菌以淀粉霉属(Amylomyces)、复膜孢酵母属(Saccharomycopsis)和威克汉姆酵母属(Wickerhamomyces)为主。近年来,随着测序技术的迅速发展,超高深测序技术也逐渐走入科研人员的视野。采用以MiSeq为代表的高通量测序技术对传统发酵食品微生物多样性进行解析时,其通常获得3~10万条不等的高质量序列,测序量在50 M碱基以下,而超高深度宏基因组测序技术通常获得上亿条高质量序列,测序量在20 Gb碱基以上,这也为解析环境中的微生物多样性奠定了坚实的基础。通过宏基因组分箱(Binning)技术可以对环境中微生物的基因组进行组装,摆脱了依靠纯培养技术的研究方式,可以在种水平,甚至可以在株水平上对环境中微生物多样性进行研究[7]。YANG等[8]通过超高深度宏基因组测序在株水平上解析了奶酪中微生物的多样性,发现了4株疑似新种,并认为部分疑似新种对奶酪的品质形成具有较大的影响。
本研究从湖北省十堰市房县地区采集了3份黄酒加工用小曲样品,使用超高深宏基因组测序技术对其微生物构成进行解析,并结合宏基因组Binning技术构建了主要微生物的基因组,进一步完善了黄酒加工用小曲中微生物的相关信息。
1 材料与方法
1.1 材料与试剂
房县黄酒加工用小曲样本采集自湖北省十堰市房县的农户家中,共采集3个,编号分别为FX1~FX3,所有样品均以糯米为主要原料,且添加了辣蓼草,直径为2.5~3.0 cm,无虫眼,制作时间在60 d左右。
QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒,德国QIAGEN公司;Illumina Novaseq 6000高通量测序平台配套试剂,美国Illumina公司;10×PCR buffer、FastPfu Fly DNA聚合酶和dNTPs Mix,北京全式金生物技术有限公司。
1.2 仪器与设备
R930机架式服务器,美国DELL公司;ND-2000C微量紫外分光光度计,美国Nano Drop公司;Illumina Novaseq 6000高通量测序平台,美国Illumina公司;DYCP-31DN电泳仪,北京六一科技生物有限公司;Fluor Chem FC3型化学发光凝胶成像系统,美国ProteinSimple公司;vetiri梯度基因扩增仪,美国AB公司。
1.3 实验方法
1.3.1 宏基因DNA提取和测序
房县黄酒加工用小曲样品置于灭菌研钵中碾碎,称取约0.5 g于2 mL无菌无酶的离心管中,使用QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒提取其宏基因组DNA,检验合格的DNA送至北京诺禾致源科技股份有限公司使用Illumina Novaseq 6000高通量测序平台进行宏基因组测序。
1.3.2 生物信息学分析
使用Seqprep软件对下机序列中含N碱基进行识别和删除,并对序列的3′端和5′端进行质量剪切,剔除平均质量<20 bp或剪切后长度<50 bp的序列[9]。基于FastQC软件对序列进行修剪,将其与人类基因组比对,移除人类基因组和低质量序列。
通过Metaphlan2软件将获得的高质量序列与标准数据库比对以获得房县黄酒加工用小曲样品中微生物的注释信息[10]。使用MegaHit软件将高质量序列组装成contigs[11],并挑选长度>1 500 bp的contigs构建序列集;基于MetaBAT2和Maxbin2软件构建序列集进行宏基因组Binning[12-13],并使用Das tool软件对获得的基因组重新评估和组装以初步获得宏基因组组装基因组(metagenome assembly genomes,MAGs)[14];使用CheckM软件对每一个MAG的质量进行评估[15],并依照完整度和污染率将MAGs划分为高质量、中等质量和低质量。
使用FastANI软件将MAGs与国家生物技术信息中心(National Center for Biotechnology Information,NCBI)非冗余核苷酸序列数据库(Nucleotide Sequence Database,NT)和人体肠道微生物的基因组集(Unified Human Gastrointestinal Genome,UHGG)比对以获得MAGs的分类学地位;使用Rpkm软件对MAGs的相对丰度进行评估[16]。
基于蛋白质直系同源簇(Cluster of Orthologous Group,COG)数据库对MAGs的功能进行注释,并使用Resistance Gene Identifier(RGI)对MAGs中的耐药基因(antibiotic resistance genes,ARGs)进行识别和注释。
1.3.3 多元统计学分析
使用R软件计算MAGs在不同分类水平上的相对丰度,统计ARGs的种类和数量。所有图形的可视化均使用R软件或Origin 2021完成。
2 结果与分析
2.1 房县黄酒加工用小曲中微生物的组成
移除人类基因组和低质量序列后,本研究3个样品共获得445 376 602条高质量序列,测序总量为62.21 Gb碱基,每个样本平均获得148 458 867条高质量序列,平均测序量为20.74 Gb。基于上述超高深测序数据,本研究首先对房县黄酒加工用小曲中微生物的组成进行了分析,其微生物在门和种水平上的注释结果如图1所示。
房县黄酒加工用小曲中共注释到5个菌门,由图1-a可知,其中相对含量>1.0%的菌门有3个,分别为硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)和子囊菌门(Ascomycota),平均相对含量分别为86.61%、8.96%和3.49%。由此可见,房县黄酒加工用小曲中的细菌主要隶属于Firmicutes和Proteobacteria,而真菌则主要隶属于Ascomycota。房县黄酒加工用小曲中共注释到96个菌种,由图1-b可知,其中相对含量>1.0%的菌种分别为戊糖片球菌(Pediococcus pentosaceus,24.04%)、食窦魏斯氏菌(Weissella cibaria,18.86%)、融合魏斯氏菌(Weissella confusa,16.15%)、干酪大球菌(Macrococcus caseolyticus,8.15%)、类肠膜魏斯氏菌(Weissella paramesenteroides,8.02%)、假肠膜明串珠菌(Leuconostoc pseudomesenteroides,2.96%)、植物乳杆菌(Lactobacillus plantarum,2.52%)、酿酒酵母(Saccharomyces cerevisiae,2.13%)、乳酸乳球菌(Lactococcus lactis,2.07%)、匹兹不动杆菌(Acinetobacter pittii,1.65%)、热带念珠菌(Candida tropicalis,1.36%)和菠萝泛菌(Pantoe ananatis,1.07%)。
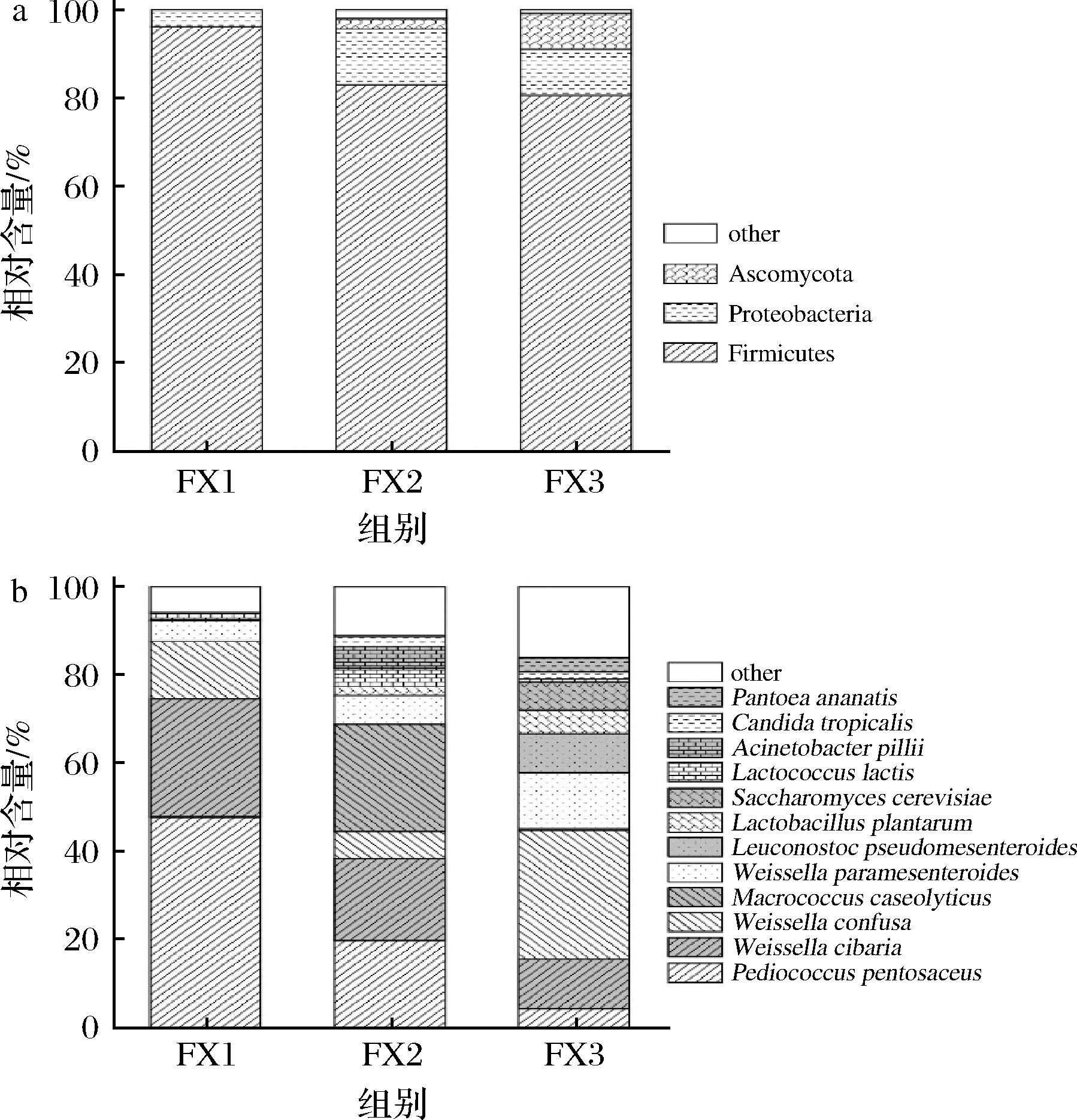
A-门;B-种
图1 房县黄酒加工用小曲中主要菌门和种的相对含量堆砌图
Fig.1 Stacked plot of the relative abundance of the main bacterial phyla and species in Huangjiu Xiaoqu of Fangxian
房县黄酒加工用小曲实质上为米酒曲,而米酒曲在以孝感米酒为代表的华中地区米酒、以大竹米酒为代表的西南地区米酒和以东兰墨米酒为代表的广西地区米酒酿造中均广泛使用。王文平等[17]发现广西南宁地区米酒曲细菌类群以Lactococcus为主,而湖北孝感地区以Weissella为主,其研究发现不同地区米酒曲的细菌类群存在一定的差异。向凡舒等[4]发现湖北恩施土家族苗族自治州宣恩县米酒曲中微生物在门水平以Firmicutes、Proteobacteria和Ascomycota为主,在属水平以Pediococcus和Pseudomonas为主。综上所述,房县黄酒加工用小曲中微生物结构与其他地区存在明显的相似性,但部分微生物类群的丰度仍然存在一定的差异,这可能与制作环境和测序方法相关[18-19]。
2.2 组装数据概述
随着测序技术和生物信息学的迅速发展,科研人员已经不满足基于Mark基因对环境样本中的微生物构成和多样性进行解析。因此,基于宏基因组的Binning技术被越来越多的应用于解析环境样本中的微生物多样性。本研究首先对3份房县黄酒加工用小曲的宏基因组序列进行Bining,其MAGs质量如图2所示。

图2 房县黄酒加工用小曲中15个MAGs的完整度和 污染率分析
Fig.2 Estimated completeness and contamination of 15 recovered MAGs in Huangjiu Xiaoqu of Fangxian
由图2可知,通过对3个房县黄酒加工用小曲的宏基因组数据进行过滤、质控、组装(contigs长度>1 500 bp)和分箱共构建了15个MAGs。通过质量分析发现,高质量MAGs(完整度>80%,污染率<10%)有8个(占比53.33%),中等质量MAGs(完整度>50%,污染率<30%)有5个(占比33.33%),而低质量MAGs(完整度<50%或污染率>30%)仅有2个(占比13.33%),后续分析均基于高质量MAGs完成。与其他环境样本(粪便、土壤和污水等)所不同[20],房县黄酒加工用小曲中的微生物复杂程度较低,与其他传统发酵食品中微生物的复杂程度类似,这可能是导致本研究所得到的MAGs数量较少的原因之一[21]。
2.3 MAGs的组成与分布
本研究首先评估了高质量MAGs与UHGG数据库和NCBI数据库中基因组的平均核苷酸相似度(average nucleotide identity,ANI),其结果如图3所示。

图3 房县黄酒加工用小曲中高质量MAGs的比对分析
Fig.3 Comparison analysis of high-quality MAGs in Huangjiu Xiaoqu of Fangxian
由图3可知,8个高质量的MAGs中有6个与数据库中的基因组具有较高的平均核苷酸相似度(ANI值≥97%),而剩余2个高质量MAGs与数据库中基因组的平均核苷酸相似度较低(ANI值<97%)。由此说明,8个高质量的MAG中有6个可以在种水平上被注释,而其他2个为疑似的新物种(FX1_3,Weissella sp.;FX3_5,Lactobacillus sp.)。高质量MAGs的相对丰度如图4所示。
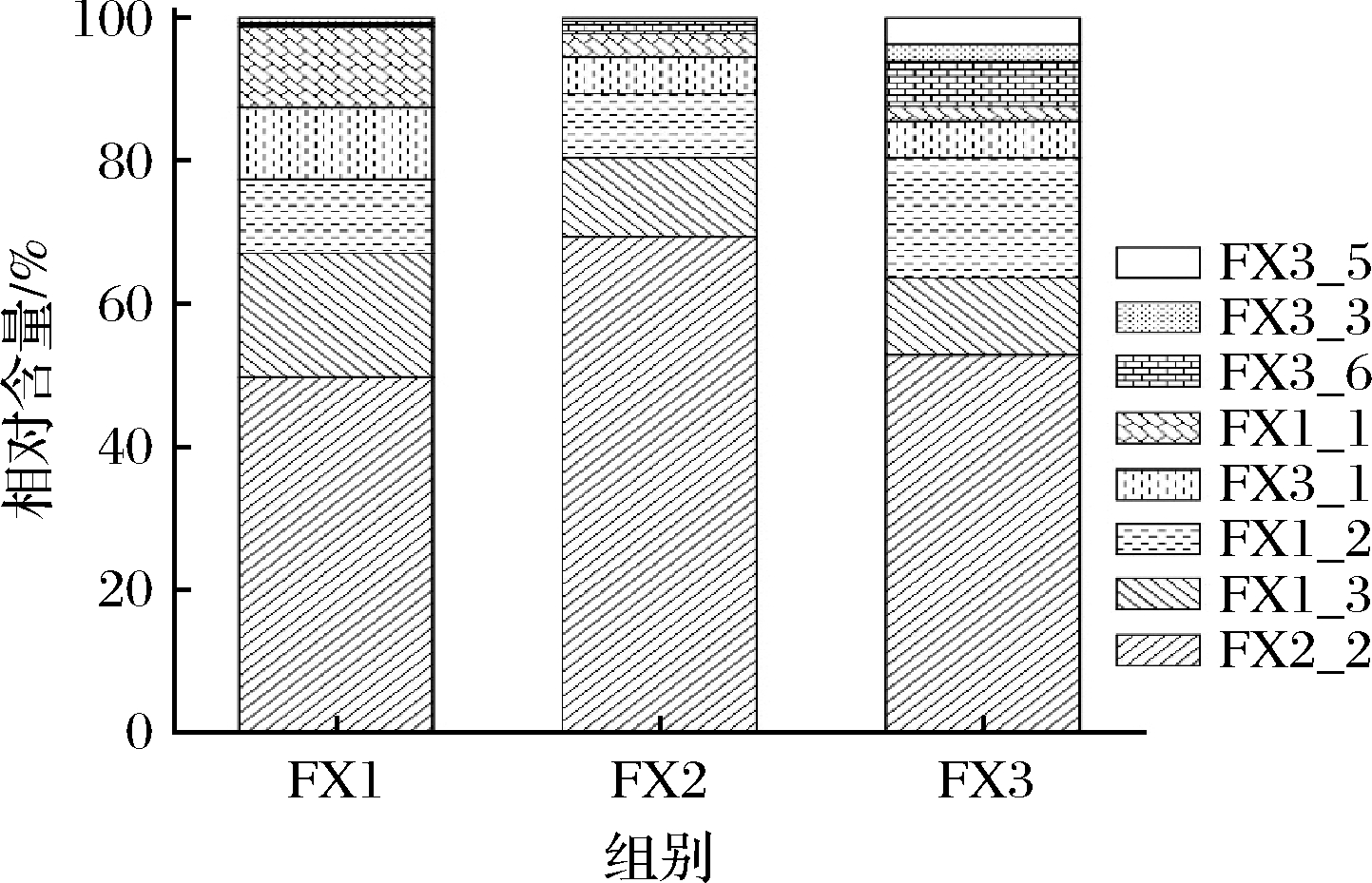
图4 房县黄酒加工用小曲中高质量MAGs的相对含量
Fig.4 Relative abundance of high-quality MAGs in Huangjiu Xiaoqu of Fangxian
由图4可知,FX2_2(Macrococcus caseolyticus)在高质量MAGs中的占据主导地位(57.35%),其次是FX1_3(Weissella cibaria)和FX1_2(Weissella paramesenteroides),相对含量分别为13.06%和12.03%。进一步研究发现,隶属于Pediococcus(FX3_1和FX1_1)、Enterobacter(FX3_3)、Lactobacillus(FX3_5和FX3_6)、球菌属(FX2_2)和Weissella(FX1_2和FX1_3)的MAGs累计相对含量分别为12.32%、1.24%、4.00%、57.35%和25.09%。
本研究通过Binning技术共组装出15个MAGs,并发现了2个疑似的新物种,其隶属于Weissella和Lactobacillus 2个细菌属。相关研究显示,Weissella和Lactobacillus是各地米酒曲中主要细菌类群,向凡舒等[3]在解析湖北省恩施土家族苗族自治州建始地区米酒曲中微生物多样性时发现,两者在米酒曲细菌中的相对含量分别为9.62%和2.18%。王玉荣等[22]使用湖北孝感和四川成都地区来源的米酒曲进行了米酒的发酵,发现不同地区来源的米酒曲对米酒产品的滋味品质具有显著的影响,并认为米酒曲中的微生物是造成品质差异的最主要因素。因此,这些上述两个疑似新物种的存在可能与房县独特的生境有关,并可能直接或间接影响着房县黄酒的滋味品质[23]。在后续的研究中可以通过基因组建模确定2株疑似新种的基本营养需求[24],结合“培养组学”手段获得微生物菌株,进而为后续探究房县黄酒品质的提升提供菌株支持。
2.4 MAGs的功能分析
本研究在解析房县黄酒加工用小曲中微生物构成的基础上进一步对微生物的功能基因进行甄别,并基于COG对功能基因进行了注释,结果如图6所示。

a-功能注释分析热图(C-能量生产和转换;D-细胞周期控制、 细胞分裂、染色体分割;E-氨基酸转运与代谢;F-核苷转运与 代谢;G-碳水化合物运输和代谢;H-辅酶转运与代谢;I-脂质 转运与代谢;J-翻译、核糖体结构与生物发生;K-转录;L-复制、 重组和修复;M-细胞壁/膜/包膜生物发生;N-细胞运动;O-翻译后 修饰,蛋白质周转,伴侣;P-无机离子运输与代谢;Q-次生代谢产 物的合成、转运和分解代谢;R-一般功能预测;S-未知功能; T-信号转导机制;U-细胞内运输,分泌和囊泡运输;V-防御机制; Y-核外结构);b-Upset图
图5 房县黄酒加工用小曲中高质量MAGs的功能注释分析
Fig.5 Functional annotation analysis of high-quality MAGs in Huangjiu Xiaoqu of Fangxian
本研究从高质量MAGs中共注释到1 913个COGs,这些COGs分别隶属于21个一级功能层。由图5-a可知,碳水化合物运输和代谢、核苷转运与代谢和翻译、核糖体结构与生物发生在高质量MAGs中,具有较高的表达水平,而细胞运动和细胞内运输,分泌和囊泡运输等表达水平较低。值得注意的是,FX3_5相较于与其他高质量MAGs,碳水化合物运输和代谢具有更高的表达水平。由图5-b可知,在所有高质量MAGs中均存在的COGs有408个,而仅在FX3_5中存在的COGs有96个,且通过对96个COGs分析发现其大多隶属于一级功能层G(碳水化合物运输和代谢)。高质量MAGs所携带的ARGs的结果如图6所示。
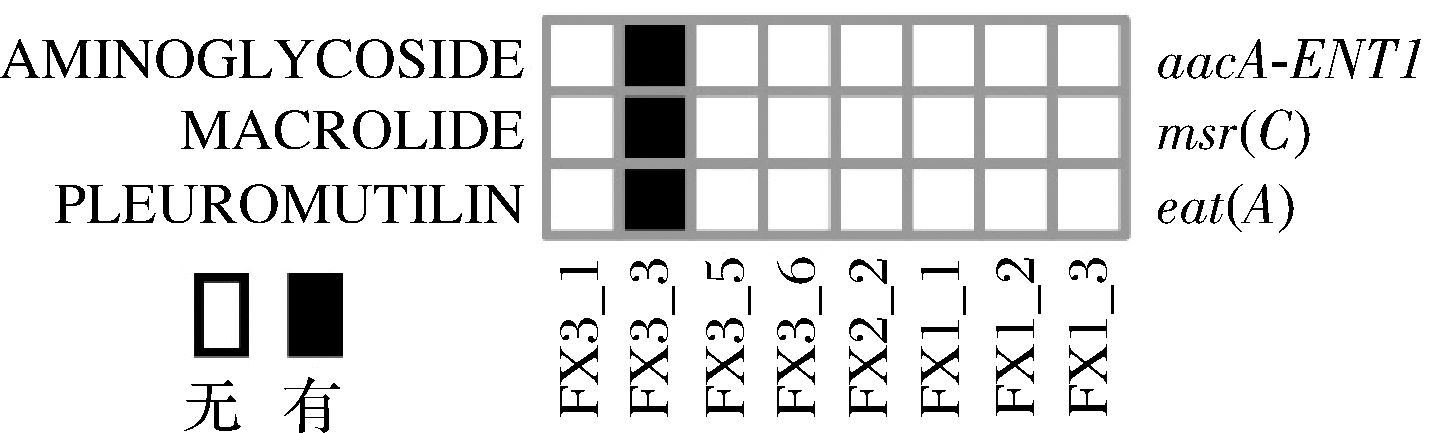
图6 ARG宿主与其携带ARGs的对应关系
Fig.6 The mapping relationship between ARG-carrying MAGs and the carried ARGs
由图6可知,在8个高质量的MAGs中鉴定出1个MAG携带ARGs,占比12.50%,且鉴定到的MAG为乳酸肠球菌(Enterococcus lactis)。FX3_3中一共鉴定到3种ARGs,分别为氨基糖苷类、大环内酯类和截短侧耳素,分别分布在aacA-ENT1、msr(C)和eat(A)等基因上。由此可见,乳酸肠球菌可能具有多重耐药性。
功能基因分析显示疑似的新物种(Lactobacillus sp.)具有较高碳水化合物运输和代谢能力,而碳水化合物运输和代谢是房县黄酒发酵过程中必不可少的。房县黄酒常以糯米或籼米为基质,接入小曲自然发酵而成,其中的微生物群系对房县黄酒品质的形成具有重要的作用。由此可见,Lactobacillus sp.可能积极参与了房县黄酒的发酵过程[25],并在房县黄酒的品质形成过程中发挥着重要的作用。与发酵性能同等重要的是发酵剂的安全性,研究结果显示乳酸肠球菌含有多个耐药基因,可能具有多重耐药性,但并不能确定其是否具有迁移潜力[26]。因此,在后续的研究中进一步扩大房县黄酒加工用小曲的采集范围和数量,在采用超高深度宏基因组测序的基础上,与高通量染色体构象捕获技术结合来解析噬菌体或质粒与ARGs宿主的关系,评估传统发酵食品的安全性是十分必要的。
3 结论
本研究发现Firmicutes、Proteobacteria和Ascomycota为房县黄酒加工用小曲中的优势菌门,而Pediococcus pentosaceus、Weissella cibaria和Weissella confusa等为优势种。通过Binning获得的8个高质量MAGs隶属于5个细菌属,其中2个为疑似的新物种(Weissella sp.和Lactobacillus sp.),且Lactobacillus sp.可能通过碳水化合物的运输和代谢积极参与房县黄酒的发酵进程。
[1] 孙宝国. 国酒[M].北京:化学工业出版社, 2019.
SUN B G.Chinese National Alcohols[M].Beijing:Chemical Industry Press, 2019.
[2] 陈程. 房县黄酒[M].武汉:长江出版社, 2019.
CHEN C.Fangxian Huangjiu[M].Wuhan:Changjiang Press, 2019.
[3] 向凡舒, 朱媛媛, 邓风, 等.建始地区米酒曲细菌和真菌多样性研究[J].食品工业科技, 2021, 42(1):126-131.
XIANG F S, ZHU Y Y, DENG F, et al.Bacterial and fungal diversity of rice wine koji in Jianshi aera[J].Science and Technology of Food Industry, 2021, 42(1):126-131.
[4] 向凡舒, 刘雪婷, 代程洋, 等.基于MiSeq高通量测序技术宣恩地区米酒微生物多样性解析[J].食品工业科技, 2020, 41(21):128-132;138.
XIANG F S, LIU X T, DAI C Y, et al.Analysis of microbial diversity of rice wine in Xuanen area based on MiSeq high-throughput sequencing technology[J].Science and Technology of Food Industry, 2020, 41(21):128-132;138.
[5] 宁亚丽, 吴跃, 何嫱, 等.基于高通量测序技术分析朝鲜族传统米酒及其酒曲中微生物群落多样性[J].食品科学, 2019, 40(16):107-114.
NING Y L, WU Y, HE Q, et al.Analysis of microbial community diversity in Chinese Korean traditional rice wine and its starter culture using high-throughput sequencing[J].Food Science, 2019, 40(16):107-114.
[6] 王艳萍, 程巧玲, 张阳, 等.米酒醪中优势微生物菌相组成的初步研究[J].中国酿造, 2008,27(5):12-14.
WANG Y P, CHENG Q L, ZHANG Y, et al.Study on predominant microflora in glutinous rice wine[J].China Brewing, 2008,27(5):12-14.
[7] YOU L J, YANG C C, JIN H, et al.Metagenomic features of traditional fermented milk products[J].LWT, 2022, 155:112945.
[8] YANG C C, YOU L J, KWOK L Y, et al.Strain-level multiomics analysis reveals significant variation in cheeses from different regions[J].LWT, 2021, 151:112043.
[9] BROWN J, PIRRUNG M, MCCUE L A.FQC Dashboard:Integrates FastQC results into a web-based, interactive, and extensible FASTQ quality control tool[J].Bioinformatics, 2017, 33(19):3 137-3 139.
[10] TRUONG D T, FRANZOSA E A, TICKLE T L, et al.MetaPhlAn2 for enhanced metagenomic taxonomic profiling[J].Nature Methods, 2015, 12(10):902-903.
[11] LI D H, LIU C M, LUO R B, et al.MEGAHIT:An ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph[J].Bioinformatics, 2015, 31(10):1 674-1 676.
[12] KANG D D, LI F, KIRTON E, et al.MetaBAT 2:An adaptive binning algorithm for robust and efficient genome reconstruction from metagenome assemblies[J].PeerJ, 2019, 7(7):e7359.
[13] WU Y W, SIMMONS B A, SINGER S W.MaxBin 2.0:An automated binning algorithm to recover genomes from multiple metagenomic datasets[J].Bioinformatics, 2016, 32(4):605-607.
[14] SIEBER C M K, PROBST A J, SHARRAR A, et al.Recovery of genomes from metagenomes via a dereplication, aggregation and scoring strategy[J].Nature Microbiology, 2018, 3(7):836-843.
[15] PARKS D H, IMELFORT M, SKENNERTON C T, et al.CheckM:Assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes[J].Genome Research, 2015, 25(7):1 043-1 055.
[16] WAGNER G P, KIN K, LYNCH V J.Measurement of mRNA abundance using RNA-seq data:RPKM measure is inconsistent among samples[J].Theory in Biosciences, 2012, 131(4):281-285.
[17] 王文平, 熊英梅, 陈赛浙, 等.不同地区米酒曲细菌多样性解析及表型预测[J].食品工业科技, 2021, 42(20):134-139.
WANG W P, XIONG Y M, CHEN S Z, et al.Analysis of bacterial diversity and prediction of phenotype in rice wine koji from different regions[J].Science and Technology of Food Industry, 2021, 42(20):134-139.
[18] YANG C C, ZHAO F Y, HOU Q C, et al.PacBio sequencing reveals bacterial community diversity in cheeses collected from different regions[J].Journal of Dairy Science, 2020, 103(2):1 238-1 249.
[19] MANDE S S, MOHAMMED M H, GHOSH T S.Classification of metagenomic sequences:Methods and challenges[J].Briefings in Bioinformatics, 2012, 13(6):669-681.
[20] 李毅, 夏呈强, 冯焱, 等.通过宏基因组分箱方法揭示南极联合冰川土壤中ARG宿主[J].生态学杂志,2021,40(9):2 832-2 841.
LI Y, XIA C Q, FENG Y, et al.Deciphering hosts of antibiotic resistance genes by metagenomic binning approaches in Union Glacier, Antarctica[J].Chinese Journal of Ecology, 2021, 40(9):2 832-2 841.
[21] WALSH A M, MACORI G, KILCAWLEY K N, et al.Meta-analysis of cheese microbiomes highlights contributions to multiple aspects of quality[J].Nature Food, 2020, 1(8):500-510.
[22] 王玉荣,张俊英, 胡欣洁, 等.湖北孝感和四川成都地区来源的酒曲对米酒滋味品质影响的评价.食品科学, 2015, 36(16): 207-210.
WANG Y R, ZHANG J Y, HU X J, et al. Evaluation of the influence of rice wine koji from Xiaogan in Hubei and Chengdu in Sichuan on the taste and quality of rice wine. Food Science, 2015, 36(16): 207-210.
[23] 王菲, 钟小丹, 戴彩霞, 等.原料米和酒曲对米酒滋味品质影响的评价[J].食品研究与开发, 2017, 38(20):80-84.
WANG F, ZHONG X D, DAI C X, et al.A comparative study on the taste profile characterization of rice wine fermented by different kinds of rice and rice wine koji[J].Food Research and Development, 2017, 38(20):80-84.
[24] SCHÖPPING M, GASPAR P, NEVES A R, et al.Identifying the essential nutritional requirements of the probiotic bacteria Bifidobacterium animalis and Bifidobacterium longum through genome-scale modeling[J].NPJ Systems Biology and Applications, 2021, 7(1):47.
[25] FANG R S, ZHOU W Y, CHEN Q H.Ethyl carbamate regulation and genomic expression of Saccharomyces cerevisiae during mixed-culture yellow rice wine fermentation with Lactobacillus sp.[J].Food Chemistry, 2019, 292(9):90-97.
[26] ZHAO R X, YU K, ZHANG J Y, et al.Deciphering the mobility and bacterial hosts of antibiotic resistance genes under antibiotic selection pressure by metagenomic assembly and binning approaches[J].Water Research, 2020, 186:116318.