炎症被认为是许多慢性病发展的关键因素,世界卫生组织发布的《2019年世界卫生统计》显示,在全球十大死因的疾病中,有八项都直接或间接来源于炎症反应[1]。炎症反应被描述为机体对外源性损伤做出的一般、复杂和有益的免疫反应,具有全身性、非特异性的特点[2]。然而,过度的炎症反应会对机体产生损害,引起各种炎症介质的过度表达,产生有利于多种慢性疾病发生的条件,最终引起如癌症、动脉粥样硬化、关节炎、心脏病和胰岛素抵抗等慢性疾病的发生[3]。目前,具有抗炎效果的天然产物受到研究者的青睐,尤其是食源性生物活性肽,因其安全无毒而逐渐成为研究热点。
菲律宾蛤仔(Ruditapes philippinarum)又名杂色蛤、花蛤等,属软体动物门双壳纲帘蛤目帘蛤科蛤仔属,广泛分布在中国南北海区,是中国四大经济养殖贝类之一[4]。蛤类具有丰富的药用价值,《本草经疏》记载“蛤蜊其性滋润而助津液,故能润五肺、止消渴,开胃也。咸能入血软坚,故主妇人血块及老癖为寒热也”。现代医学研究表明,菲律宾蛤仔富含蛋白质、矿物元素和氨基酸,是一种高蛋白、低脂肪的优良水产贝类[5]。其蛋白酶解产物具有降血压[6]、抗氧化[6]、降血糖[7]、护肝[8]、减肥减脂[9]等多种生物活性。
传统生物活性肽的制备方法通常采用酶水解蛋白后进行一系列的分离、纯化以及活性鉴定,但存在过程繁琐,历时长且获得高纯度活性肽难度大,产量低等缺点[10]。利用生物信息学和分子对接技术筛选活性肽,能够高效筛选出特定活性肽片段,缩短纯化的时间,并对多肽进行生物活性预测,获得效果优良的目标肽[11]。田文慧等[12]利用分子对接技术从啤酒中筛选出2条寡肽,能和DPP-Ⅳ结合紧密,具有降血糖潜力。LUO等[13]通过药效团模拟、分子对接和分子动力学模拟筛选出PD-L1的小分子抑制剂,具有潜在的抗癌活性。
本研究以菲律宾蛤仔作为研究对象,分析鉴别其肌肉组织主要蛋白组成,利用虚拟酶解和分子对接技术,从菲律宾蛤仔中筛选出抗炎肽,通过合成寡肽,进行体外细胞实验验证,获得具有良好抗炎活性肽,为菲律宾蛤仔生物活性肽的筛选提供理论依据。
1 材料与方法
1.1 数据库与软件
NCBI(http://www.ncbi.nlm.nih.gov);BIOPEP-UWM(https://biochemia.uwm.edu.pl/biopep-uwm/);PeptideRanker(http://bioware.ucd.ie/-ompass/biowareweb/Serverpages/peptide-ranker.php);ToxinPred(http://www.imtech.res.in/raghava/toxinpred/);INNOVAGEN(http://www.innovagen.com/proteomics-tools);RCSB PDB(https://www.rcsb.org/);Pymol 2.1;AutoDock Tools-1.5.6;Discovery Studio 2019;AutoDock Vina;Schrodinger2019.01;Chem3D。
1.2 材料与仪器
新鲜菲律宾蛤仔,广东省湛江市霞山水产品批发市场;RAW264.7小鼠巨噬细胞,中国科学院细胞库。丙烯酰胺、SDS(化学纯)、脂多糖(lipopolysaccharide,LPS),Sigma公司;PMSF、EDTA(超级纯)、过硫酸铵(化学纯),AMERCSO公司;CHAPS、DTT、IAM、胰蛋白酶,Promega公司;尿素,分析纯,GibcoBRL公司;甲酸、甲醇、乙腈,均为质谱级,Fisher Scientific公司;PBS、RPMI 1640、青霉素-链霉素双抗,Gibco公司;胎牛血清、CCK-8试剂盒,ZETA公司;一氧化氮试剂盒,上海碧云天公司。
Thermo Q-Exactive质谱仪、细胞培养箱、Varioskan Flash型全自动酶标仪,Thermo公司;垂直版型电泳仪,Bio-rad公司;离心机,eppendorf公司;超声破碎仪,宁波新芝公司;双向等电聚焦装置,GE公司。
1.3 实验方法
1.3.1 菲律宾蛤仔全蛋白的提取及鉴定
1.3.1.1 全蛋白提取
参考ZI等[14]的方法稍作改动。新鲜菲律宾蛤仔开壳取肉,取其软体部位(去除内脏团等杂质),用PBS缓冲液清洗后称取1.0 g组织,加入液氮研磨成粉末,加入含有10%三氯乙酸的预冷丙酮,-20 ℃沉淀2 h,离心后用丙酮清洗后除去丙酮,加入500 μL裂解液,混匀后超声波提取5 min,悬液在15 ℃,20 000×g,离心30 min后收集上清液,4 ℃备用。
1.3.1.2 双向凝胶电泳
根据UDONSOM等[15]的方法稍作改动。在提取的全蛋白溶液中加入终浓度50 mmol/L 0.8% IPG混合液,混匀后于10 ℃,20 000×g离心10 min,取出胶条后室温平衡10 min,在每根胶条上覆盖2 mL矿物油,逐步升高电压至4 kV进行等电聚焦,待等电聚焦后的胶条在平衡液中还原烷基化处理后,立即放置于提前配制好的SDS-PAGE上面,轻轻按压,胶条一端加入maker后,覆盖上一层0.5%琼脂糖固定住,电泳至溴酚蓝指示剂跑出胶底。
1.3.1.3 高丰度蛋白质的鉴定
根据UDONSOM等[15]的方法稍作修改。使用ImageMaster 2D软件进行胶图灰度值分析,挑选蛋白强度百分比大于2%的蛋白点,切除后用超纯水清洗2次,再加入1 mL胶内脱色液(50%乙腈,25 mmol/L NH4HCO3溶液)清洗2次。加入乙腈脱水至胶粒完全变白后真空抽干乙腈,加入10 mmol/L DTT溶液,胶粒完全吸收后放入56 ℃水浴锅内孵育1 h,孵育完毕加入55 mmol/L IAM溶液,暗室室温孵育45 min后加入25 mmol/L NH4HCO3溶液清洗2次,再使用脱色液继续清洗2次,至胶粒完全变白后抽干乙腈。向胶粒中加入胰蛋白酶储液,充分吸收后再加入25 mmol/L NH4HCO3溶液没过胶粒,37 ℃水浴酶解过夜,往酶解液中依次加入脱色液和乙腈,分别抽提肽段30 min,合并来自于同一个胶粒的肽段,真空抽干备用。每管使用10 μL终体积分数为0.1%的甲酸复溶肽段,上样5 μL,进行质谱鉴定。
质谱条件:离子源电压设置为1.8 kV,一级质谱扫描范围设置为350~2 000 Da,扫描分辨率设置为70 000,二级质谱扫描范围依赖于一级母离子m/z自动选择,分辨率设置为17 500。
1.3.2 主要蛋白的虚拟酶解及肽库的建立
虚拟酶解采用BIOPEP-UWM在线工具,选择Bioactive peptides-ENZYME(S) ACTION模块[16],选取鉴定得到的1条主要蛋白质(原肌球蛋白)的序列输入到对话框,分别选择糜蛋白酶(EC 3.4.21.1)、胰蛋白酶(EC 3.4.21.4)、胃蛋白酶(pH 1.3,EC 3.4.23.1)、木瓜蛋白酶(EC 3.4.22.2)、碱性蛋白酶(EC 3.4.21.62)、胃蛋白酶(EC 3.4.23.1)+糜蛋白酶(EC 3.4.21.1)+ 胰蛋白酶(EC 3.4.21.4)、胃蛋白酶(EC 3.4.23.1)+胰蛋白酶(EC 3.4.21.4)、木瓜蛋白酶(EC 3.4.22.2)+碱性蛋白酶(EC 3.4.21.62)对原肌球蛋白序列虚拟酶解,再将虚拟酶解后的肽段与BIOPEP数据库中的肽段进行比较,筛选出未经报道过的二肽、三肽、四肽、五肽、六肽,建立虚拟酶解肽库。
1.3.3 菲律宾蛤仔生物活性肽的筛选
1.3.3.1 生物活性、毒性和理化性质预测
使用PeptideRanker在线程序(http://bioware.ucd.ie/-compass/biowareweb/Serverpages/peptide-ranker.php)对以上虚拟酶解肽库中的寡肽进行潜在生物活性的可能性分析,选取生物活性评分≥0.3的肽段,认为其具有潜在的生物活性[17];使用ToxinPred在线程序(http://www.imtech.res.in/raghava/toxinpred/)预测寡肽的潜在毒性[18],使用INNOVAGEN在线程序(http://www.innovagen.com/proteomics-tools)预测寡肽的水溶性和理化性质。
1.3.4 分子对接
1.3.4.1 肽段、受体结构准备
通过以上方法共筛选出8条寡肽用于分子对接验证。使用Schrodinger2019.01软件构建,并通过Chem3D对所构建的多肽进行能量最小化,转化为pdb格式后导入AutoDock Tools-1.5.6软件并添加原子电荷、分配原子类型,将所有柔性键默认可旋转,保存为pdbqt文件。TLR2(PDB ID:6 NIG)、TLR4(PDB ID:2Z63)[19]的晶体结构从蛋白质数据库(https://www.rcsb.org/)中获得,采用Pymol 2.1软件删除无关小分子后导入AutoDock Tools-1.5.6软件,删除水分子、添加氢原子以及设置原子类型,保存为pdbqt文件。
1.3.4.2 分子对接
使用Discovery Studio 2019 对准备好的蛋白和肽段进行半柔性对接。将处理后的8条多肽作为小分子配体,2个蛋白靶点作为受体,通过AutoDock Vina进行批量分子对接,对分子对接结果进行分析,多肽与蛋白的结合作用可视化采用Pymol2.1软件。
1.3.5 体外抗炎效果测定
寡肽的人工合成采用固相合成法(纯度>98%),利用LPS诱导的RAW264.7细胞炎症模型验证寡肽的体外抗炎活性[20]。
1.3.5.1 寡肽对RAW264.7细胞活力的影响
根据黄平等[20]的方法稍加修改。取对数生长期的RAW264.7细胞以3×105个细胞/mL接种于96孔板中,待细胞贴壁后,弃去上清液,无菌操作下进行样品干预。对照组加入RPMI 1640培养基100 μL、实验组分别加入质量浓度为25、50、100、200、400 μg/mL的样品溶液100 μL,在含有5%CO2、37 ℃的培养箱干预24 h后,弃去上清液,避光条件下加入体积分数为10%的CCK-8溶液100 μL,于培养箱中孵育30 min,使用酶标仪在450 nm下测定各组吸光度值。细胞活力按公式(1)计算:
细胞活力![]()
(1)
式中:A1,样品组的吸光度值;A空,空白孔的吸光度值;A0,对照组的吸光度值。
1.3.5.2 寡肽对RAW264.7细胞产NO能力的影响
根据黄平等[20]的方法稍加修改,方法同1.3.5.1节。将处理后的96孔板放入含有5%CO2、37 ℃的培养箱干预2 h后,模型组和各实验组分别加入终质量浓度为1 μg/mL的LPS,于培养箱中干预24 h后,收集上清液,按照试剂盒说明书进行测定,制作标准曲线(y=0.015 7x+0.059 2,R2=0.999 5)。
1.4 数据处理
应用Pymol 2.1软件对分子对接可视化。所有试验设3组平行,数据以平均值±标准差表示,采用SPSS 26.0、Excel 2019和GraphPad 9.2.0等软件对数据进行分析处理和图表的制作,P<0.05为显著性差异。
2 结果与分析
2.1 菲律宾蛤仔软体部位中主要蛋白质的鉴定
如图1-a所示,菲律宾蛤仔软体部位(去除内脏团等杂质)全单白的2-DE图像显示出其蛋白质主要分布在图中的Ⅰ、Ⅱ、Ⅲ、Ⅳ四个区域,分子质量主要分布在35~45 kDa和低于14.4 kDa范围内。通过ImageMaster 2D软件分析后,可找到1 582个蛋白点,如图1-b所示,从中挑选出强度值最大的6个蛋白胶点(ID为205、206、311、312、313、316)(图1-b)进行质谱鉴定。结果如表1所示,菲律宾蛤仔中的主要蛋白质分别为原肌球蛋白(tropomyosin)、组蛋白H4(histone H4)等。本研究选取的原料为菲律宾蛤仔软体部位(去除内脏团等杂质),主要组成部分为足肌和闭壳肌,其是支撑贝类肌肉运动的机构蛋白质,而原肌球蛋白是肌原纤维蛋白中最稳定的蛋白质之一,能调节肌动球蛋白的收缩或松弛反应,与肌肉运动密切相关[21]。本研究最终选取原肌球蛋白进行后续分析。
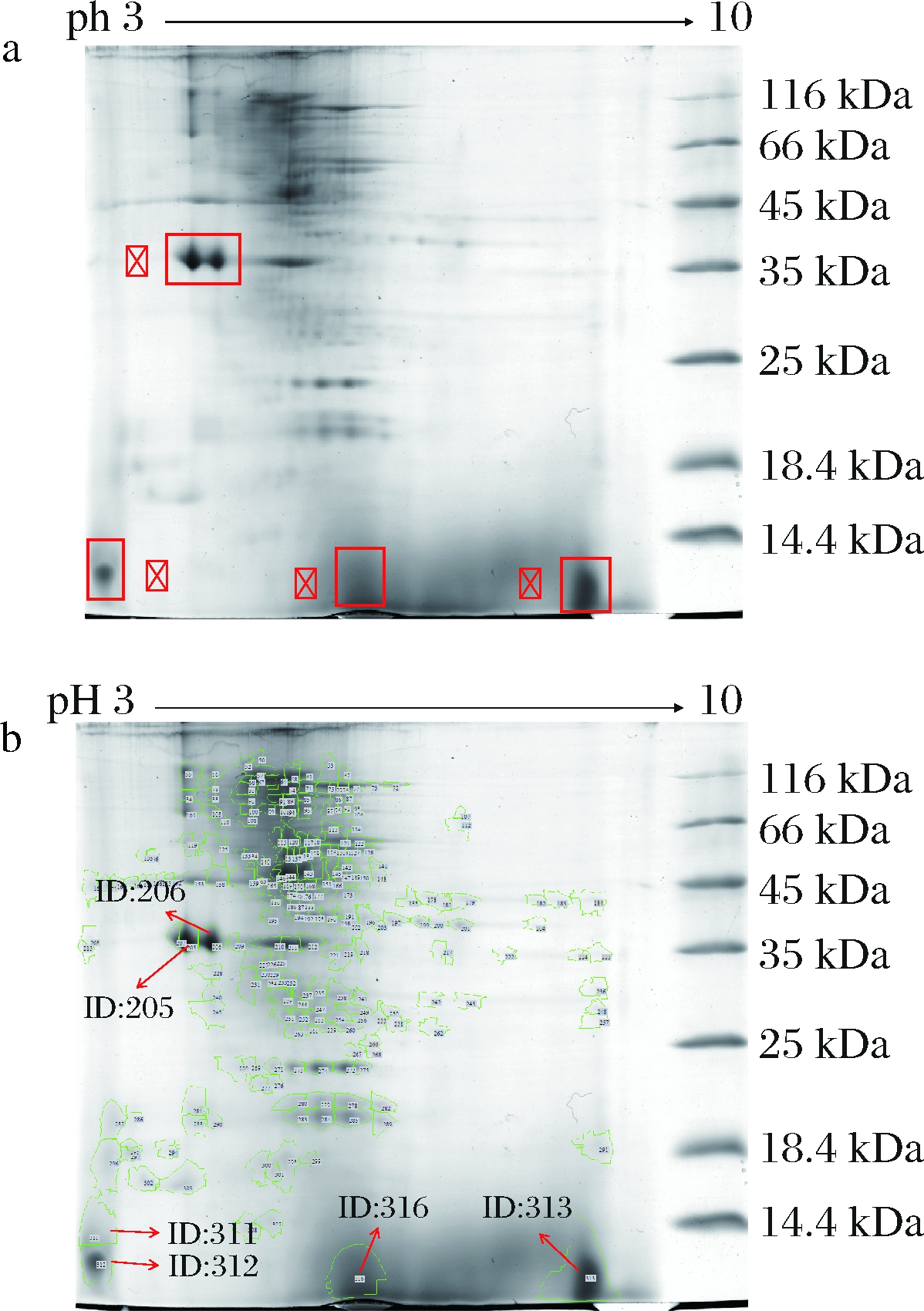
a-2-DE原图谱;b-使用ImageMaster 2D软件分析灰度后的2-DE图谱
图1 菲律宾蛤仔全单白的2-DE图谱
Fig.1 2-DE map of R.philippinarum total protein
表1 菲律宾蛤仔主要蛋白质质谱鉴定结果
Table 1 Identification of main protein mass spectrometry of R.philippinarum
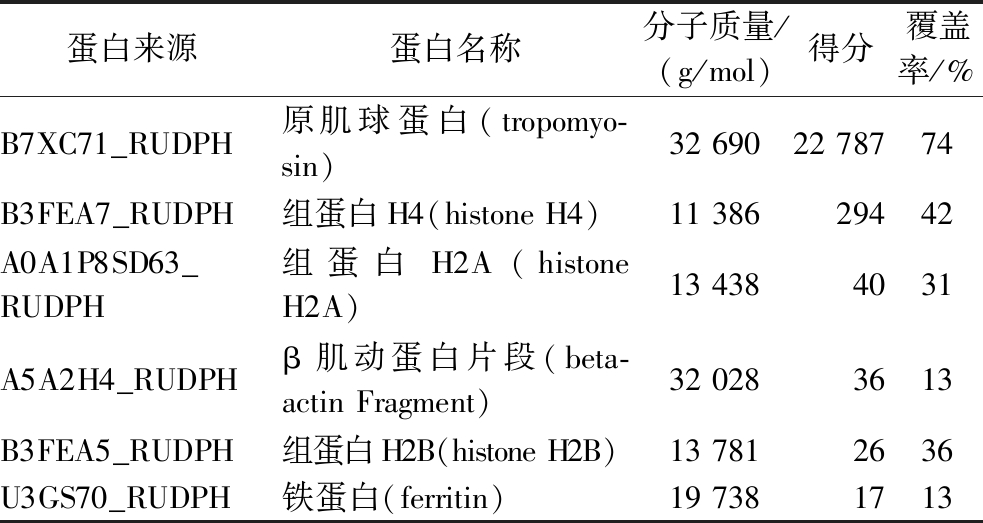
蛋白来源蛋白名称分子质量/(g/mol)得分覆盖率/%B7XC71_RUDPH原肌球蛋白(tropomyo-sin)326902278774B3FEA7_RUDPH组蛋白H4(histoneH4)1138629442A0A1P8SD63_RUDPH组蛋白H2A(histoneH2A)134384031A5A2H4_RUDPHβ肌动蛋白片段(beta-actinFragment)320283613B3FEA5_RUDPH组蛋白H2B(histoneH2B)137812636U3GS70_RUDPH铁蛋白(ferritin)197381713
2.2 虚拟酶解结果及生物活性肽的筛选
通过BIOPEP-UWM工具对原肌球蛋白进行虚拟酶解,经8种酶解方式虚拟酶解后比对BIOPEP数据库,如表2所示,共得到132条未经报道过的二~六肽。再通过PeptideRanker对这132条肽段进行评分,筛选出具有潜在活性的肽段(评分≥0.3)共10条(表3),其中二肽3条、三肽2条、四肽4条、五肽1条。水溶性和毒性通常是评价药物在体内代谢转运分布的重要指标,生物活性肽在机体内发挥生物活性的前提首先是对机体无毒害作用[22],因此通过分析 10 条寡肽的理化性质、毒性和水溶性发现,其分子质量较小,多数在500 g/mol以下,表明以上10条寡肽可能具有很好的肠道消化吸收特性,以便于更好地发挥生物活性[22];10条寡肽均为无毒性的优良肽段;最终筛选出8条水溶性寡肽,即CK、DL、MD、AMKL、CKAL、DQTF、GYTR、MQAMK。通过分析8条寡肽的氨基酸序列,发现多数水溶性寡肽的氨基酸序列中含有一些疏水性氨基酸,如苯丙氨酸(Phe)、亮氨酸(Leu)、丙氨酸(Ala)等,说明这些寡肽具有构成活性肽的前提基础[23],且一些寡肽的C末端由碱性氨基酸组成,如精氨酸(Arg)、赖氨酸(Lys),这与文献[24]报道的结果一致,说明碱性氨基酸对肽的水溶性具有一定的影响。GUHA等[24]的研究表明,多数抗炎肽的序列中富含带正电荷的疏水性氨基酸,尤其是当这些氨基酸存在于N端或C端,这些氨基酸被认为在抗炎反应中起重要作用,此外,谷氨酰胺(Gln)也被认为是参与抗炎活性的关键氨基酸[25],这与本研究筛选出的8条寡肽的氨基酸组成规律相似。
表2 菲律宾蛤仔原肌球蛋白虚拟酶解肽库
Table 2 Polypeptide library of virtual enzymatic hydrolysis from tropomyosin in R.philippinarum
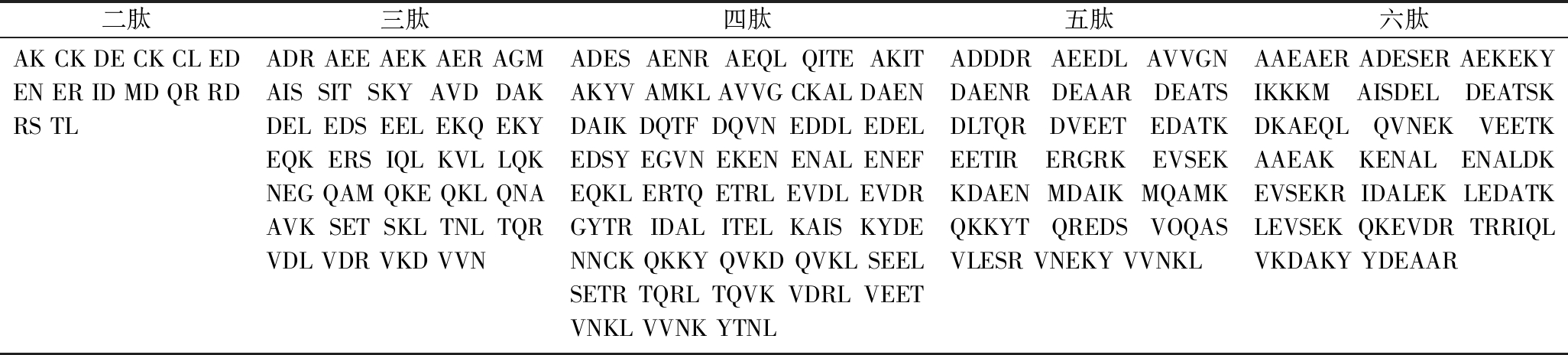
二肽三肽四肽五肽六肽AKCKDECKCLEDENERIDMDQRRDRSTLADRAEEAEKAERAGMAISSITSKYAVDDAKDELEDSEELEKQEKYEQKERSIQLKVLLQKNEGQAMQKEQKLQNAAVKSETSKLTNLTQRVDLVDRVKDVVNADESAENRAEQLQITEAKITAKYVAMKLAVVGCKALDAENDAIKDQTFDQVNEDDLEDELEDSYEGVNEKENENALENEFEQKLERTQETRLEVDLEVDRGYTRIDALITELKAISKYDENNCKQKKYQVKDQVKLSEELSETRTQRLTQVKVDRLVEETVNKLVVNKYTNLADDDRAEEDLAVVGNDAENRDEAARDEATSDLTQRDVEETEDATKEETIRERGRKEVSEKKDAENMDAIKMQAMKQKKYTQREDSVOQASVLESRVNEKYVVNKLAAEAERADESERAEKEKYIKKKMAISDELDEATSKDKAEQLQVNEKVEETKAAEAKKENALENALDKEVSEKRIDALEKLEDATKLEVSEKQKEVDRTRRIQLVKDAKYYDEAAR
表3 菲律宾蛤仔原肌球蛋白酶解肽的活性评价及理化性质分析
Table 3 Activity evaluation and physicochemical property analysis of the selected oligopeptides

肽多肽序列PeptideRanker评分分子质量/(g/mol)PIpH7时的净电荷预测水溶性预测毒性二肽CK0.46249.339.130.9优无毒性DL0.33246.260.69-1优无毒性MD0.57264.300.88-1优无毒性三肽AGM0.82277.343.770差无毒性QAM0.49277.343.770差无毒性四肽AMKL0.49461.6210.191优无毒性CKAL0.40433.579.130.9优无毒性DQTF0.52509.510.76-1优无毒性GYTR0.33495.539.831优无毒性五肽MQAMK0.39607.799.881优无毒性
2.3 分子对接
分子对接是通过计算机平台模拟分子间相互作用方式,并预测其结合模式和亲和力的常用方法[26],在多肽活性方面应用广泛,分子间结合能越低,表明肽与蛋白的亲和力越好,则理论上肽段的抗炎活性更强[27]。TLR2和TLR4是toll样受体(toll-like receptors,TLRs)家族受体参与炎症反应信号传导的常用受体,细胞膜上的TLRs可特异性识别外来刺激,从而激活炎症反应信号通路[19]。分子对接结果如表4所示,8条寡肽中有4条与TLR2蛋白结合紧密,结合能低于-5 kcal/mol,2条与TLR4蛋白结合紧密,结合能低于-5 kcal/mol,其中寡肽DQTF和GYTR与TLR2的结合能分别为-7.70和-6.70 kcal/mol,与TLR4的结合能分别为-6.20和-5.50 kcal/mol,结合紧密且匹配度高,因此选用这2条寡肽进行下一步分析。
表4 寡肽的分子对接得分
Table 4 Molecular docking scores of oligopeptides
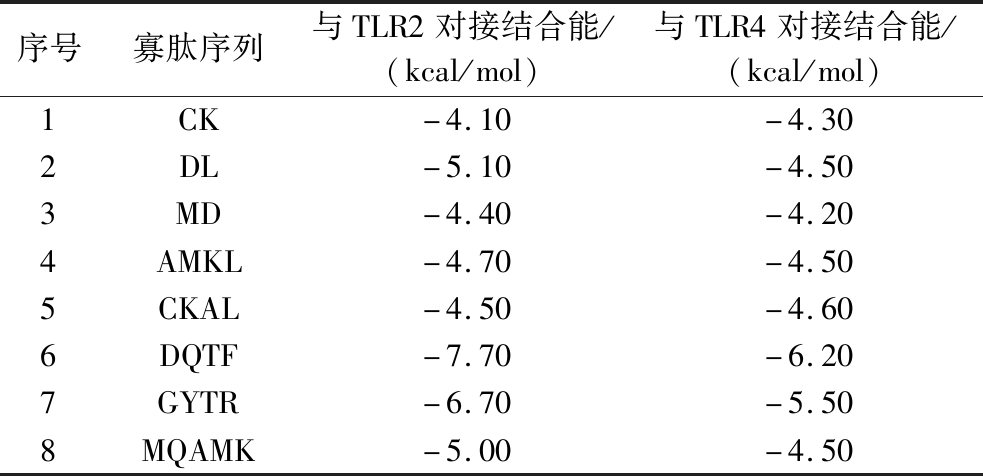
序号寡肽序列与TLR2对接结合能/(kcal/mol)与TLR4对接结合能/(kcal/mol)1CK-4.10-4.302DL-5.10-4.503MD-4.40-4.204AMKL-4.70-4.505CKAL-4.50-4.606DQTF-7.70-6.207GYTR-6.70-5.508MQAMK-5.00-4.50
2.4 DQTF和GYTR与TLR2、TLR4的分子对接作用构象分析
如图2所示,寡肽DQTF和TLR2蛋白的结合匹配较好,能够进入蛋白位点深处,与TLR2蛋白的氨基酸残基TYR-332,LEU-289,VAL-351形成氢键,与PHE-284,ILE-341,VAL-343,LEU-328,LEU-289形成疏水相互作用,使得DQTF能够牢牢地锚定在位点空腔内,使其与蛋白形成稳定的复合物。

a-复合体的三维结构;b-活性表面图;c-复合体的细节绑定方式(下同)
图2 DQTF-TLR2的分子对接图
Fig.2 Molecular docking diagram of DQTF-TLR2
如图3所示,寡肽DQTF和TLR4蛋白的GLN-430,SER-381,ASP-405,THR-357,ARG-355等氨基酸形成很强的氢键相互作用,且氢键距离短,结合能力强,可以有效促进多肽与蛋白形成稳定的复合物,另外DQTF还可以与TYR-451,HIS-426,PHE-377等氨基酸形成很好的疏水相互作用,对稳定蛋白口袋的多肽分子有着重要贡献。
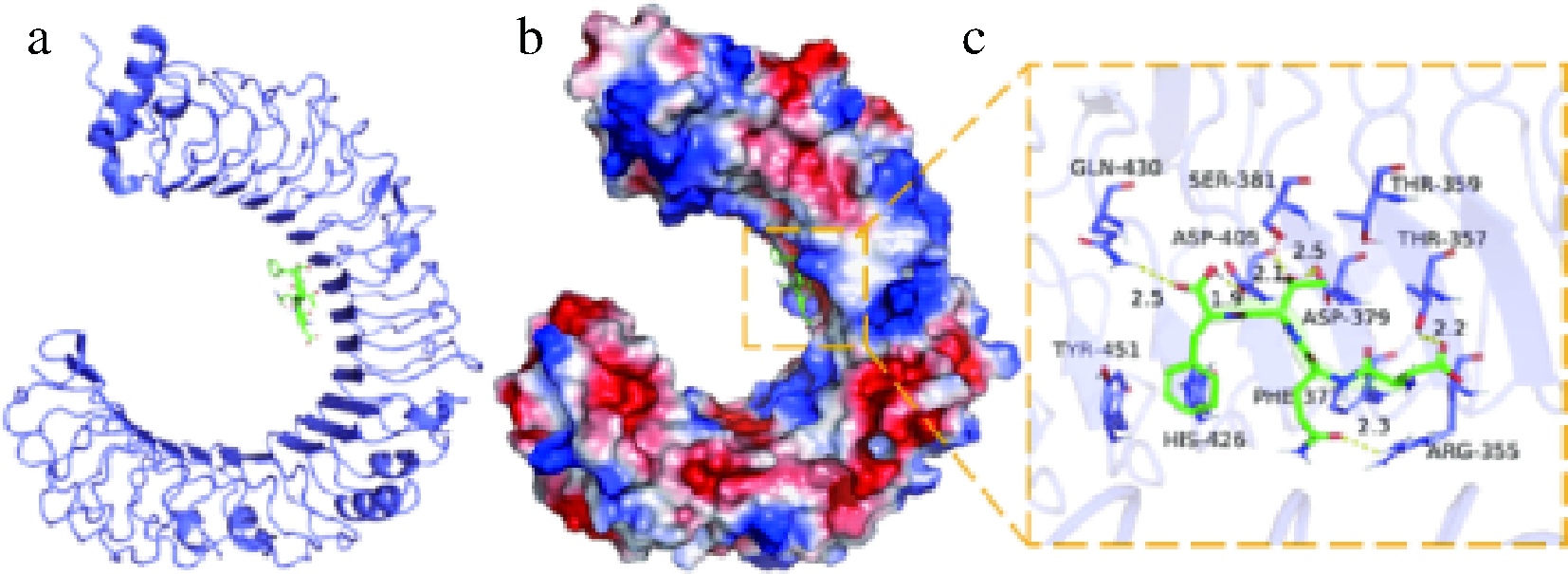
图3 DQTF-TLR4的分子对接图
Fig.3 Molecular docking diagram of DQTF-TLR4
如图4所示,寡肽GYTR与TLR2通过与PHE-284,ILE-314,VAL-343,LEU-312,VAL-348,LEU-331,LEU-289,ILE-261等氨基酸形成疏水相互作用,与TYR-326形成很强的氢键相互作用,另外,该多肽的苯环能够与TLR2蛋白的TYR-326苯环以及LEU-317形成pi共轭相互作用,这些相互作用可以有效促进多肽与蛋白形成稳定的复合物。
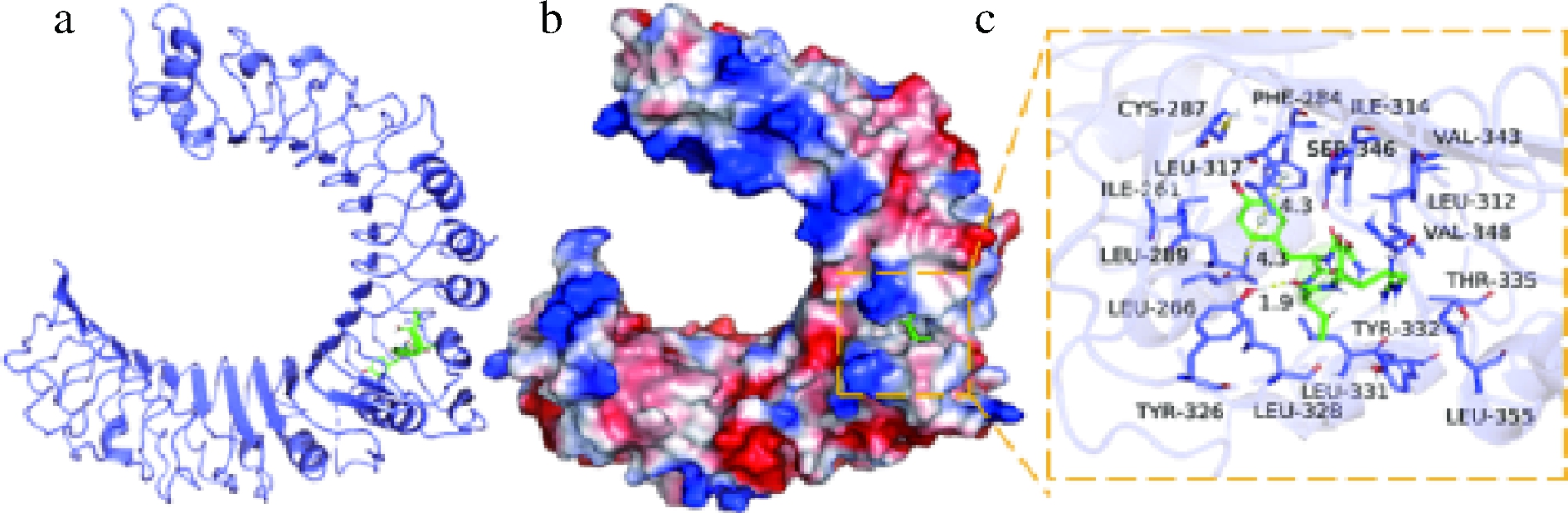
图4 GYTR-TLR2的分子对接图
Fig.4 Molecular docking diagram of GYTR-TLR2
如图5所示,GYTR与TLR4蛋白的活性位点存在相互作用的氨基酸残基有GLU-135,GLY-111,ARG-87,PHE-63,GLU-42,ASN-137,PRO-113,GLU-89,PRO-65,ASN-64等。GYTR含有络氨酸以及亮氨酸,具有一定的疏水性,能够与活性位点氨基酸PHE-63,PRO -113,PRO-65形成疏水相互作用,对稳定蛋白空腔中的小分子有着重要作用。另外,该寡肽还能够与GLU-135,GLU-42,ASN-137,GLU-89氨基酸形成多个氢键相互作用,对锚定蛋白空腔中的小分子有着重要贡献。综上,以上2条寡肽可以通过与TLR2、TLR4蛋白分子形成氢键、疏水相互作用等作用力而紧密结合,起到抑制作用。

图5 GYTR-TLR4的分子对接图
Fig.5 Molecular docking diagram of GYTR-TLR4
2.5 体外抗炎活性验证
2.5.1 DQTF和GYTR对RAW264.7细胞活力的影响
细胞活力是反应机体生命特征的一个重要指标,其变化可以体现细胞的增殖或死亡[28],是评价多肽有无毒副作用的重要指标。本研究对筛选出来的2条寡肽进行体外抗炎活性验证,通过CCK-8法探究不同浓度的DQTF和GYTR对RAW264.7细胞活力的影响,结果如图6所示。与对照组相比,25~400 μg/mL范围的DQTF和GYTR均能显著提高细胞的活力(P<0.001),表明2条寡肽对细胞无毒害作用且可显著促进细胞增殖。
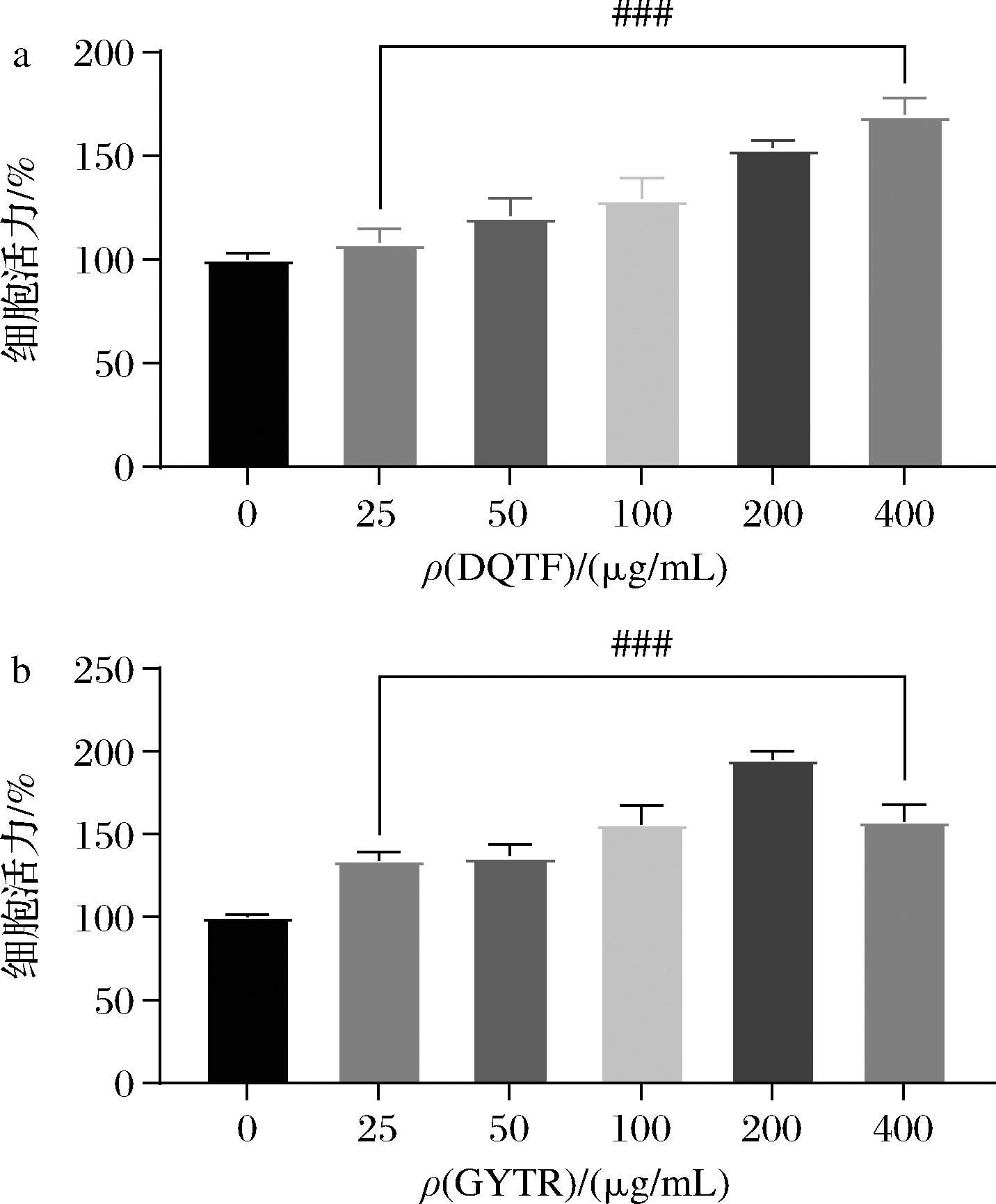
图6 DQTF和GYTR对RAW264.7细胞活力的影响
Fig.6 Effect of DQTF and GYTR on the viability of RAW264.7 cells
注:与对照组相比,###表示P<0.001。
2.5.2 DQTF和GYTR对LPS诱导的RAW264.7细胞产NO含量的影响
经LPS刺激后的巨噬细胞会产生炎症反应,释放出一系列炎症介质,其中NO作为一种气体介质也参与调节各种炎症反应,过量的NO会引起机体过度炎症[29]。本研究对筛选出来的2条寡肽进行体外抗炎活性验证,通过Greiss法检测寡肽对LPS诱导的RAW264.7细胞炎症模型产生NO含量的影响。结果如图7所示,与对照组相比,经LPS刺激后的细胞产NO含量极显著增加(P<0.001),说明造模成功;与LPS组相比,DQTF干预组在100~400 μg/mL范围内,细胞产NO含量显著下降(P<0.01),且呈浓度依赖性,GYTR在25~400 μg/mL范围内,细胞产NO含量呈浓度依赖性显著下降(P<0.001),这与LEE等[30]通过传统酶解手段分离出的菲律宾蛤仔抗炎肽的效果一致,并且本研究的NO抑制水平(P<0.001)更优于LEE等的结果(P<0.01),表明DQTF和GYTR是具有优良抗炎活性的寡肽。但2条肽段对于其他炎症介质的调控水平还未知,因此下一步的研究将集中在DQTF和GYTR对调节LPS诱导的RAW264.7细胞产生其他炎症介质水平的调控上。
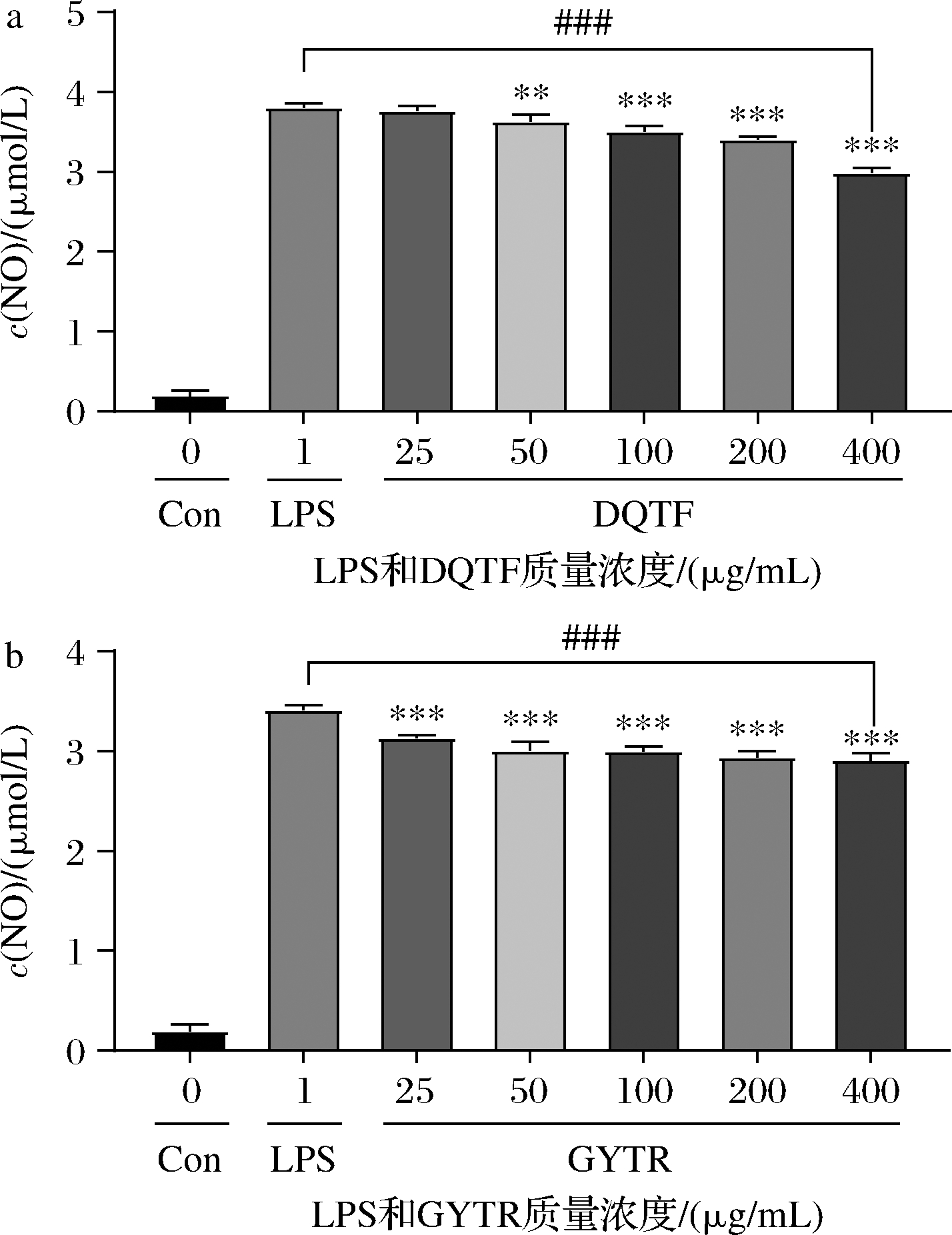
图7 DQTF和GYTR对RAW264.7细胞产NO的影响
Fig.7 Effect of DQTF and GYTR on NO productions by RAW264.7 cells
注:与 LPS 组相比,*表示P<0.05,**表示P<0.01,***表示P<0.001。
3 结论
本文建立一种简便、快速筛选生物活性肽的方法。首先使用2-DE技术从菲律宾蛤仔的全蛋白中分离其高丰度蛋白质,通过LC-MS/MS鉴定出蛋白序列后,选取其中主要蛋白(原肌球蛋白)进行分析。结合在线程序BIOPEP-UWM对其进行虚拟酶解,共得到132条寡肽,再对寡肽进行生物活性预测、理化性质分析,筛选出8条未经报道且水溶性好、对人体无毒害的寡肽,利用分子对接模拟了肽段与TLR2、TLR4蛋白的结合模式,最终筛选出2条结合紧密的肽段,对其进行作用模式分析,并利用LPS诱导的RAW264.7细胞模型验证其体外抗炎活性,结果表明2条寡肽是优良的抗炎肽。本实验通过虚拟酶解手段结合分子对接模拟小分子与大分子蛋白的结合模式来筛选生物活性肽,建立了一种快速、高效的筛选生物活性肽的方法,为菲律宾蛤仔来源的生物活性肽的筛选和开发提供了方法参考。
[1] LIU W L,CHEN X W,LI H,et al.Anti-inflammatory function of plant-derived bioactive peptides:A review[J].Foods (Basel,Switzerland),2022,11(15):2361.
[2] XU J Z,YI M Q,DING L J,et al.A review of anti-inflammatory compounds from marine fungi,2000—2018[J].Marine Drugs,2019,17(11):636.
[3] HOTAMISLIGIL G S.Inflammation,metaflammation and immunometabolic disorders[J].Nature,2017,542(7640):177-185.
[4] 饶梦微,章超桦,林海生,等.菲律宾蛤仔肉不同提取物呈味特性[J].广东海洋大学学报,2022,42(1):90-97.
RAO M W,ZHANG C H,LIN H S,et al.Sensory characteristic of different extracts from Ruditapes philippinarum[J].Journal of Guangdong Ocean University,2022,42(1):90-97.
[5] 杨金兰,李刘冬,黄珂,等.菲律宾蛤仔全脏器的营养成分分析与评价[J].中国渔业质量与标准,2014,4(2):26-31.
YANG J L,LI L D,HUANG K,et al.Analysis and evaluation on nutrients in whole viscera of Ruditapes philippinarum[J].Chinese Fishery Quality and Standards,2014,4(2):26-31.
[6] YU Y,FAN F J,WU D,et al.Antioxidant and ACE inhibitory activity of enzymatic hydrolysates from Ruditapes philippinarum[J].Molecules (Basel,Switzerland),2018,23(5):1 189.
[7] ZHANG Y,PAN D D,YANG Z C,et al.Angiotensin I-Converting enzyme (ACE) inhibitory and dipeptidyl Peptidase-4 (DPP-Ⅳ) inhibitory activity of umami peptides from Ruditapes philippinarum[J].LWT,2021,144:111265.
[8] WANG Q,LIU F J,WANG X M,et al.Preparation and hepatoprotective activities of peptides derived from mussels (Mytilus edulis) and clams (Ruditapes philippinarum)[J].Marine Drugs,2022,20(11):719.
[9] SONG Y H,CAI Q L,WANG S L,et al.The ameliorative effect and mechanisms of Ruditapes philippinarum bioactive peptides on obesity and hyperlipidemia induced by a high-fat diet in mice[J].Nutrients,2022,14(23):5066.
[10] JO C,KHAN F F,KHAN M I,et al.Marine bioactive peptides:Types,structures,and physiological functions[J].Food Reviews International,2017,33(1):44-61.
[11] 赵贵琴,李婷婷,宋敏杰,等.分子对接技术筛选鲈鱼肌球蛋白中黄嘌呤氧化酶抑制肽[J].中国食品学报,2021,21(6):81-91.
ZHAO G Q,LI T T,SONG M J,et al.Screening of xanthine oxidase inhibitory peptide from bass myosin by molecular docking[J].Journal of Chinese Institute of Food Science and Technology,2021,21(6):81-91.
[12] 田文慧,孙丽平,张翠,等.白啤中二肽基肽酶-IV抑制肽的虚拟筛选及活性分析[J].食品科学,2022,43(10):81-87.
TIAN W H,SUN L P,ZHANG C,et al.Virtual screening of activity evaluation of dipeptidyl peptidase-IV inhibitory peptides in white beer[J].Food Science,2022,43(10):81-87.
[13] LUO L X,ZHONG A,WANG Q,et al.Structure-based pharmacophore modeling,virtual screening,molecular docking,ADMET,and molecular dynamics (MD) simulation of potential inhibitors of PD-L1 from the library of marine natural products[J].Marine Drugs,2021,20(1):29.
[14] ZI J,ZHANG J Y,WANG Q H,et al.Proteomics study of rice embryogenesis:Discovery of the embryogenesis-dependent globulins[J].Electrophoresis,2012,33(7):1129-1138.
[15] UDONSOM R,REAMTONG O,ADISAKWATTANA P,et al.Immunoproteomics to identify species-specific antigens in Neospora caninum recognised by infected bovine sera[J].Parasite (Paris,France),2022,29:60.
[16] MINKIEWICZ P,DZIUBA J,IWANIAK A,et al.BIOPEP database and other programs for processing bioactive peptide sequences[J].Journal of AOAC International,2008,91(4):965-980.
[17] MOONEY C,HASLAM N J,POLLASTRI G,et al.Towards the improved discovery and design of functional peptides:Common features of diverse classes permit generalized prediction of bioactivity[J].PLoS One,2012,7(10):e45012.
[18] GUPTA S,KAPOOR P,CHAUDHARY K,et al.In silico approach for predicting toxicity of peptides and proteins[J].PLoS One,2013,8(9):e73957.
[19] JI C F,ZHANG Z Y,CHEN J R,et al.Immune-enhancing effects of a novel glucan from purple sweet potato Ipomoea batatas (L.) lam on RAW264.7 macrophage cells via TLR2-and TLR4-mediated pathways[J].Journal of Agricultural and Food Chemistry,2021,69(32):9313-9325.
[20] 黄平,洪静霞,米杰,等.羊栖菜多酚通过核转录因子-κB/丝裂原活化蛋白激酶通路缓解脂多糖诱导的RAW264.7细胞炎症反应[J].食品科学,2022,43(23):141-148.
HUANG P,HONG J X,MI J,et al.Polyphenols from Sargassum fusiforme alleviate lipopolysaccharide-induced inflammatory reaction in RAW264.7 cells through nuclear transcription factor-κB/mitogen-activated protein kinase pathway[J].Food Science,2022,43(23):141-148.
[21] 章超桦,薛长湖.水产食品学[M].北京:中国农业出版社,2010.
ZHANG C H,XUE C H.Aquatic Food Science[M].Beijing:China Agricultural Press,2010.
[22] 陈艳楠,邱智军,刘学强,等.紫贻贝(Mytilus edulis)蛋白计算机模拟消化物活性的生物信息学分析[J].食品与发酵工业,2022,48(15):185-192.
CHEN Y N,QIU Z J,LIU X Q,et al.Bioinformatics analysis of the bioactivities of in silico digest from Mytilus edulis proteins[J].Food and Fermentation Industries,2022,48(15):185-192.
[23] 孙洁,李燕,郑昌亮,等.中国毛虾二肽基肽酶-IV抑制肽的分离纯化与结构鉴定[J].食品与发酵工业,2023,49(1):160-167.
SUN J,LI Y,ZHENG C L,et al.Isolation,purification and structural identification of dipeptidyl peptidase-IV inhibitory peptide from China shrimp[J].Food and Fermentation Industries,2023,49(1):160-167.
[24] GUHA S,MAJUMDER K.Structural-features of food-derived bioactive peptides with anti-inflammatory activity:A brief review[J].Journal of Food Biochemistry,2019,43(1):e12531.
[25] GONZ LEZ-MONTOYA M,HERN
LEZ-MONTOYA M,HERN NDEZ-LEDESMA B,SILV
NDEZ-LEDESMA B,SILV N J M,et al.Peptides derived from in vitro gastrointestinal digestion of germinated soybean proteins inhibit human colon cancer cells proliferation and inflammation[J].Food Chemistry,2018,242:75-82.
N J M,et al.Peptides derived from in vitro gastrointestinal digestion of germinated soybean proteins inhibit human colon cancer cells proliferation and inflammation[J].Food Chemistry,2018,242:75-82.
[26] TAO X,HUANG Y K,WANG C,et al.Recent developments in molecular docking technology applied in food science:A review[J].International Journal of Food Science &Technology,2020,55(1):33-45.
[27] SU L J,WANG Y,WANG J M,et al.Structural basis of TLR2/TLR1 activation by the synthetic agonist diprovocim[J].Journal of Medicinal Chemistry,2019,62(6):2938-2949.
[28] 朱丹丹,江世贵,黄建华,等.斑节对虾性腺组织、细胞的原代培养条件优化[J].水产科学,2019,38(2):163-172.
ZHU D D,JIANG S G,HUANG J H,et al.Optimization of tissue and primary cell culture of tiger shrimp Penaeus monodon[J].Fisheries Science,2019,38(2):163-172.
[29] 储倩.醇溶性鲟鱼软骨多肽的制备及抗炎活性研究[D].镇江:江苏大学,2021.
CHU Q.Preparation of ethanol-soluble polypeptide from sturgeon cartilage and anti-inflammatory activity[D].Zhenjiang:Jiangsu University,2021.
[30] LEE S J,KIM E K,KIM Y S,et al.Purification and characterization of a nitric oxide inhibitory peptide from Ruditapes philippinarum[J].Food and Chemical Toxicology,2012,50(5):1660-1666.