冷鲜猪肉通常贮藏于0~4 ℃,因肉嫩味美、营养卫生等优点而深受消费者喜爱,也因营养丰富且水分含量高而极易腐败。微生物生长繁殖尤其是优势腐败菌是引起肉类腐败变质的主要原因,包括假单胞菌、热杀索丝菌和乳酸菌等[1]。其中,热杀索丝菌(Brochothrix thermosphacta)属于兼性厌氧的革兰氏阳性菌,能在4 ℃低温条件下快速生长繁殖,引起肉类品质劣变,是导致托盘包装冷鲜猪肉腐败变质的主要腐败菌[2]。而托盘结合聚乙烯保鲜膜又是超市销售冷鲜猪肉时广泛采用的一种包装形式。因此,监测PE包装冷鲜猪肉中热杀索丝菌的含量,对于保证冷鲜猪肉食用和商品价值具有重要意义。
常见的微生物检测方法包括国标规定的平板菌落计数法[3]、新兴的菌落测试片法[4]以及基于荧光定量PCR测定[5]等方法,但这些方法都存在着需要样品预处理和专业人员操作、耗时费力、具有破坏性、难以批量检测等缺点。因此,有必要建立一种快速无损的方法检测冷鲜猪肉中的微生物含量。高光成像技术(hyperspectral imaging,HSI)是一种集计算机科学、光学、传感器、信号采集及仪器分析等多种技术为一体的新型快速无损检测技术,分辨率高,可同时获取样品的图像信息和光谱信息[6],其在非包装肉类微生物检测上的应用研究备受关注[7]。TAO等[8]和庄齐斌等[9]利用高光谱成像技术建立了预测冷鲜猪肉中菌落总数的模型,预测集相关系数(correlation coefficient of prediction,Rp)均达到了0.90以上,预测准确度较高;ACHATA等[10]基于可见-近红外光谱范围内的HSI技术开发了快速预测牛背最长肌上菌落总数的模型,Rp2最高可达0.96;何鸿举等[2]利用近红外高光谱成像技术实现了冷鲜鸡肉中热杀索丝菌含量的快速评估,Rp可达到0.931。然而,迄今为止,基于高光成像技术预测微生物的研究主要集中在非包装冷鲜肉中菌落总数的预测上,对冷鲜肉中特定腐败菌的预测研究较少,且鲜少有研究关注包装冷鲜肉中微生物的检测。
鉴于高光谱成像技术在肉品微生物检测上所展现出的简单、快速和无损等优势,本研究尝试以4 ℃贮藏条件下接种热杀索丝菌的非包装和PE包装冷鲜猪大排肉为实验对象,获取其在可见-近红外(400~1 000 nm)和短波红外(1 000~2 000 nm)波段内的高光谱数据,选择不同的光谱预处理和特征波长筛选算法,建立基于全波段和特征波长的模型以实现非包装和PE包装冷鲜猪大排肉中热杀索丝菌的定量预测,为批量、快速、无损检测PE包装冷鲜猪大排肉品质提供了方法参考和数据支撑,有助于促进冷鲜肉品行业发展。
1 材料与方法
1.1 菌种、材料与试剂
热杀索丝菌(B.thermosphacta)ACCC 03872,购买于中国农业微生物菌种保藏管理中心,于南京农业大学食品科技学院实验室4 ℃条件下保藏;冷鲜猪大排肉(宰后冷却24 h),购于江苏南京北华联超市,30 min内运至实验室;营养琼脂,上海盛思生化科技有限公司;NaCl,国药集团化学试剂有限公司;无水乙醇,广东光华科技股份有限公司;STAA琼脂培养基(含STAA添加剂),海博生物技术有限公司。
1.2 主要仪器与设备
SW-CJ-2FD型洁净工作台,苏州安泰空气技术有限公司;LDZF-50KB型立式压力蒸汽灭菌锅,上海申安医疗器械厂;UV-1800型紫外分光光度计,日本岛津公司;JA2003型电子天平,上海精密科学仪器有限公司;CTHI-250B型恒温恒湿箱,美国施都凯设备公司。
ImSpector V10E型光谱成像仪、ImSpector N25E成像光谱仪,芬兰Specim公司;ICLB1620CCD相机,美国Imperx公司;IT3900ER卤素光源(150 W),美国Illumination Technologies公司;Raptor EM285CL相机,英国Raptor Photonics公司;IRCP0076-ICOMB001移动平台及高光谱分析软件等,五铃光学股份有限公司。
1.3 实验方法
1.3.1 样品处理与接种
参照顾欣哲[11]的方法,略有改动。将保存于斜面试管的热杀索丝菌接种至营养琼脂培养基,30 ℃下活化培养24 h,然后挑选典型热杀索丝菌单菌落进行划线培养,30 ℃下培养24 h。用0.85%(质量分数)无菌生理盐水冲洗培养基上的热杀索丝菌制成菌悬液,调整菌悬液浓度至103~104 CFU/mL,备用。
在无菌超净台中,将猪大排肉分割成10 cm×1 cm(直径×厚度)的圆柱体,共获得200块猪大排肉样品。用75%(体积分数)乙醇溶液擦拭表面后置于紫外灯下灭菌15 min,再将预灭菌的样品浸泡在热杀索丝菌悬液中15 s,取出后沥干多余水分。将接菌样品随机分成2组,即包装组和非包装组,每组100块样品。包装组样品表面接触包装一层无菌PE保鲜膜,非包装组样品不进行任何接触包装处理。每个样品都被单独置于培养皿中后于(4±1) ℃恒温恒湿培养箱中贮藏10天(包括第0天),每隔24 h从包装组和非包装组中各取出10块样品进行高光谱数据采集和微生物检测。
1.3.2 高光谱数据采集与校正
为探究PE保鲜膜对样品光谱反射率的影响,本文采集了非包装和PE包装冷鲜猪大排肉以及PE保鲜膜的高光谱数据。利用标准黑板能够完全吸收入射光的性质,采集完全覆盖了PE保鲜膜的标准黑板的高光谱图像,以获取PE保鲜膜的相对反射率。采集数据前,高光谱成像系统预热30 min。可见-近红外高光谱的分辨率为2.8 nm,有效波段范围为400~1 000 nm,数据采集模式为反射。依据预实验,其工作参数设置为[12]:相机镜头与样本距离28 cm,光源与样品距离31 cm(斜上方呈45°夹角),光源强度45 W,曝光时间3 ms,平台移速7.23 mm/s。短波红外高光谱的分辨率为6.2 nm,有效波段范围为1 000~2 000 nm,数据采集模式也为反射。依据预实验,其工作参数设置为:相机镜头与样本距离26 cm,光源与样品距离31 cm(斜上方呈45°夹角),光源强度255 W,曝光时间3.5 ms,平台移速13.78 mm/s。
为减小样品间厚度差异性、弱化相机暗电流效应、减小外围环境光线对高光谱图像的影响,需对采集的高光谱图像进行校正[13]。具体校正方法为:采集聚四氟乙烯白板(反射率99.9%)获得全白反射图像,盖住相机镜头采集全黑反射图像,然后通过公式(1)获得校正后图像。
(1)
式中,R,校正后图像;I0,初始反射图像;Ir,全白反射图像;Br,全黑反射图像。
1.3.3 热杀索丝菌的测定
参照GB 4789.2—2022《食品安全国家标准 食品微生物学检验 菌落总数测定》,略有改动。无菌条件下,取10 g冷鲜猪大排肉样品剪碎,置于装有90 mL体积分数0.85%无菌生理盐水的无菌均质袋,振荡30 min,梯度稀释后选取3个适当稀释度,取0.1 mL稀释液涂布于STAA琼脂培养基上,在(30±1) ℃下培养(48±1) h后进行菌落计数。
1.4 数据处理
1.4.1 光谱提取与预处理
本实验通过MATLAB R2016a软件选取高光谱图像内整块猪大排肉作为感兴趣区域(range of interests,ROI),获取并计算每个样品ROI区域内的平均光谱值,以此作为样品的光谱。为减少试验操作人员和仪器带来的误差,采用标准正态变量法(standard normal variate,SNV)、多元散射校正(multiplicative scatter correction,MSC)、正交信号校正法(orthogonal signal correction,OSC)、平滑法(smoothing)、一阶导数法(first derivative,1 st)等算法进行光谱预处理。
1.4.2 光谱特征波段筛选
高光谱数据量大,不免存在大量干扰信息,导致模型精度和运行速度降低,因此挑选特征波段有助于提高模型的鲁棒性和泛化能力,提升系统的计算速度和运行能力。本文采用连续投影算法(successive projections algorithm,SPA)和竞争性自适应重加权算法(competitive adaptive reweighted sampling,CARS)筛选光谱的特征波长。SPA是一种常见的特征波长筛选方法,能够挑选冗余信息量最少的波长,减少波长数量,在近红外光谱、高光谱等领域的定性和定量分析中应用广泛[14]。CARS可根据回归系数的绝对值进行特征波长筛选,能通过交互验证得出模型均方根误差最低的子集,从而筛选出最佳特征波长组合[15]。
1.4.3 模型的建立与评价
本研究采用偏最小二乘(partial least squares,PLS)和支持向量机(support vector machine,SVM)建立基于高光谱的热杀索丝菌的定量预测模型。通过校正集决定系数(determination coefficient of calibration,Rc2)、校正集均方根误差(root mean square error of calibration,RMSEC)、预测集决定系数(determination coefficient of prediction,Rp2)、预测集均方根误差(root mean square error of prediction,RMSEP)以及相对分析误差(relative percent deviation,RPD)[公式(2)]评价模型的准确性和预测能力。Rc2和Rp2越接近1,RMSEC和RMSEP越小,模型精度越高[16]。RPD<2.5,表明模型预测能力差;RPD值在2.5~3.0,表明模型预测能力较好;RPD>3.0,表明模型预测能力非常好[17]。
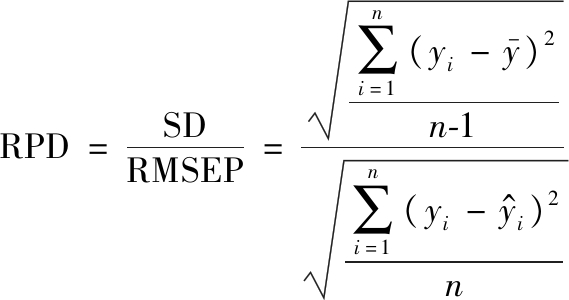
(2)
式中,SD,标准差;RMSEP,预测集均方根误差;yi,实测值;![]() 平均值;
平均值;![]() 预测值;n,样品数量。
预测值;n,样品数量。
2 结果与分析
2.1 猪大排肉中热杀索丝菌含量的测定结果
4 ℃贮藏条件下,非包装和PE包装冷鲜猪大排肉中热杀索丝菌含量的测定结果如表1所示。参照WU等[18]的方法,将非包装和PE包装冷鲜猪大排肉中热杀索丝菌含量的测量值分别从小到大排序,每4个样品中随机选取1个样品作为预测集,3个作为校正集,最终非包装组和PE包装组的微生物数据均以3∶1的比例被分为校正集(75个样本)和预测集(25个样本)。此外,表1显示校正集中热杀索丝菌含量的变化范围可以覆盖预测集中的变化范围,且校正集和预测集中热杀索丝菌含量均匀分布。
表1 校正集和预测集样品中热杀索丝菌含量的测定结果
Table 1 Results for the measurement of concentrations of B.thermosphacta in the calibration set and the prediction set
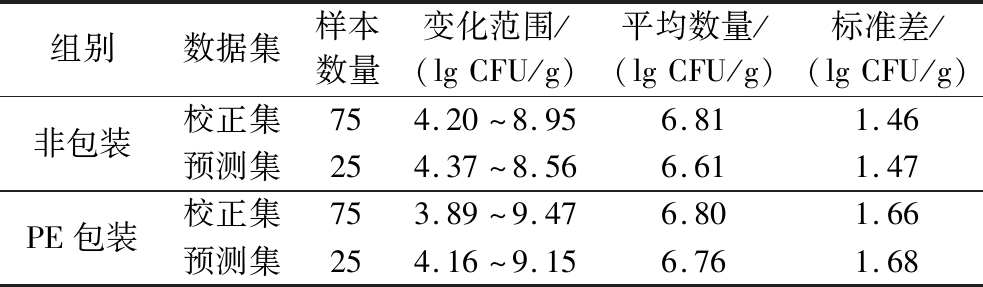
组别数据集样本数量变化范围/(lgCFU/g)平均数量/(lgCFU/g)标准差/(lgCFU/g)非包装校正集754.20~8.956.811.46预测集254.37~8.566.611.47PE包装校正集753.89~9.476.801.66预测集254.16~9.156.761.68
2.2 猪大排肉样品的光谱特征
非包装和PE包装冷鲜猪大排肉在不同波段的平均光谱如图1所示。在400~1 000 nm和1 000~2 000 nm波段范围内,非包装组和PE包装组样品的平均光谱曲线总体走势基本一致,吸收峰相似度较高,但光谱值大小存在差异。PE包装冷鲜猪大排肉的光谱反射率略低于非包装的,其原因可能是光线在穿透PE保鲜膜时发生透射、反射、散射和折射,造成发射率部分损失。
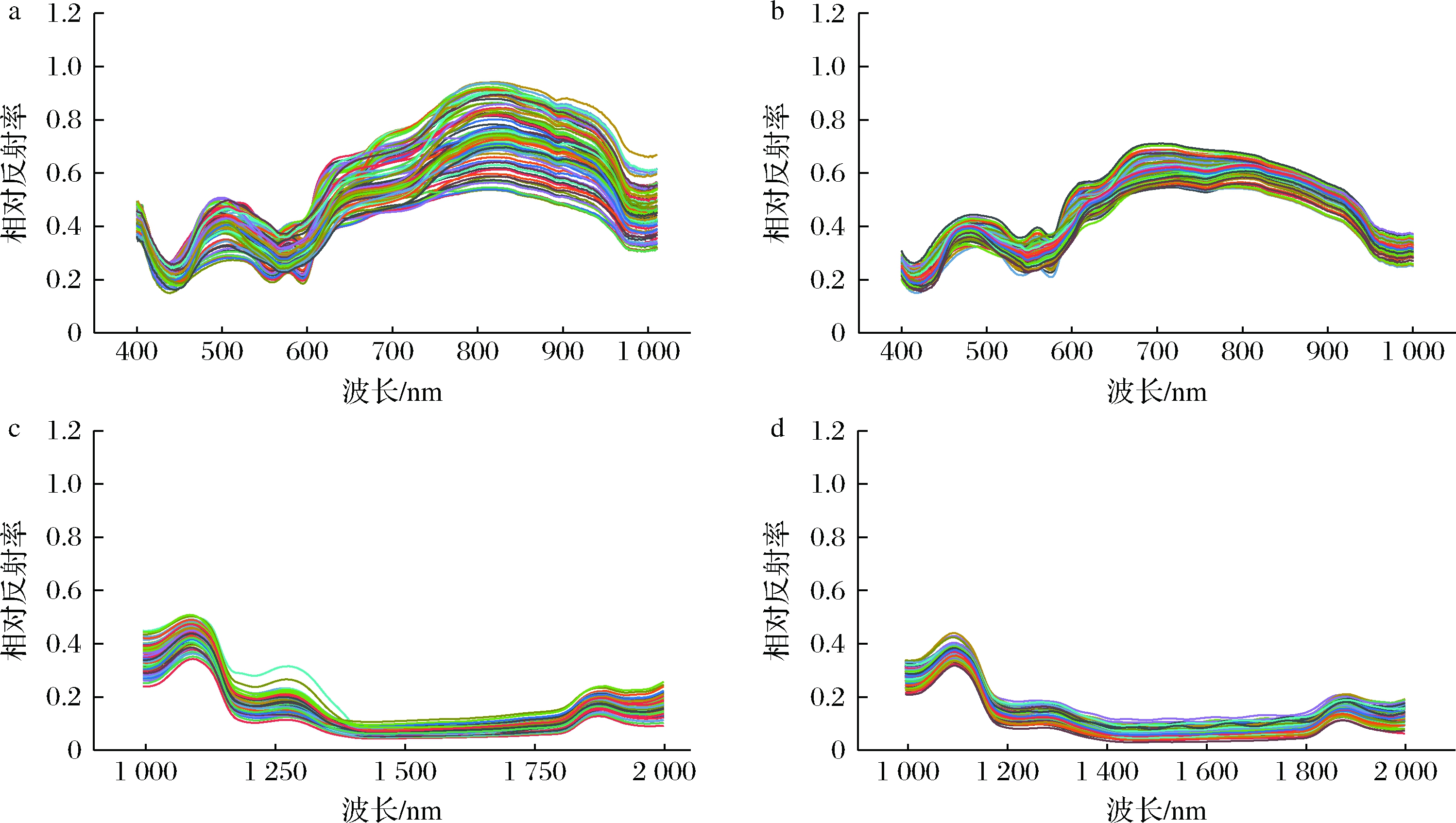
a-400~1 000 nm,非包装;b-400~1 000 nm,PE包装;c-1 000~2 000 nm,非包装;d-1 000~2 000 nm,PE包装
图1 非包装和PE包装猪大排肉在不同波段下的平均光谱
Fig.1 Average spectra of unpackaged and PE-packaged pork chops at different wavelengths
图2显示PE保鲜膜在400~1 000 nm和1 000~2 000 nm内的相对反射率均低于0.2且并无明显的特征峰,这在一定程度上说明PE保鲜膜对冷鲜猪大排肉光谱反射率的影响较小,解释了非包装和PE包装组样品反射光谱曲线相似的原因。
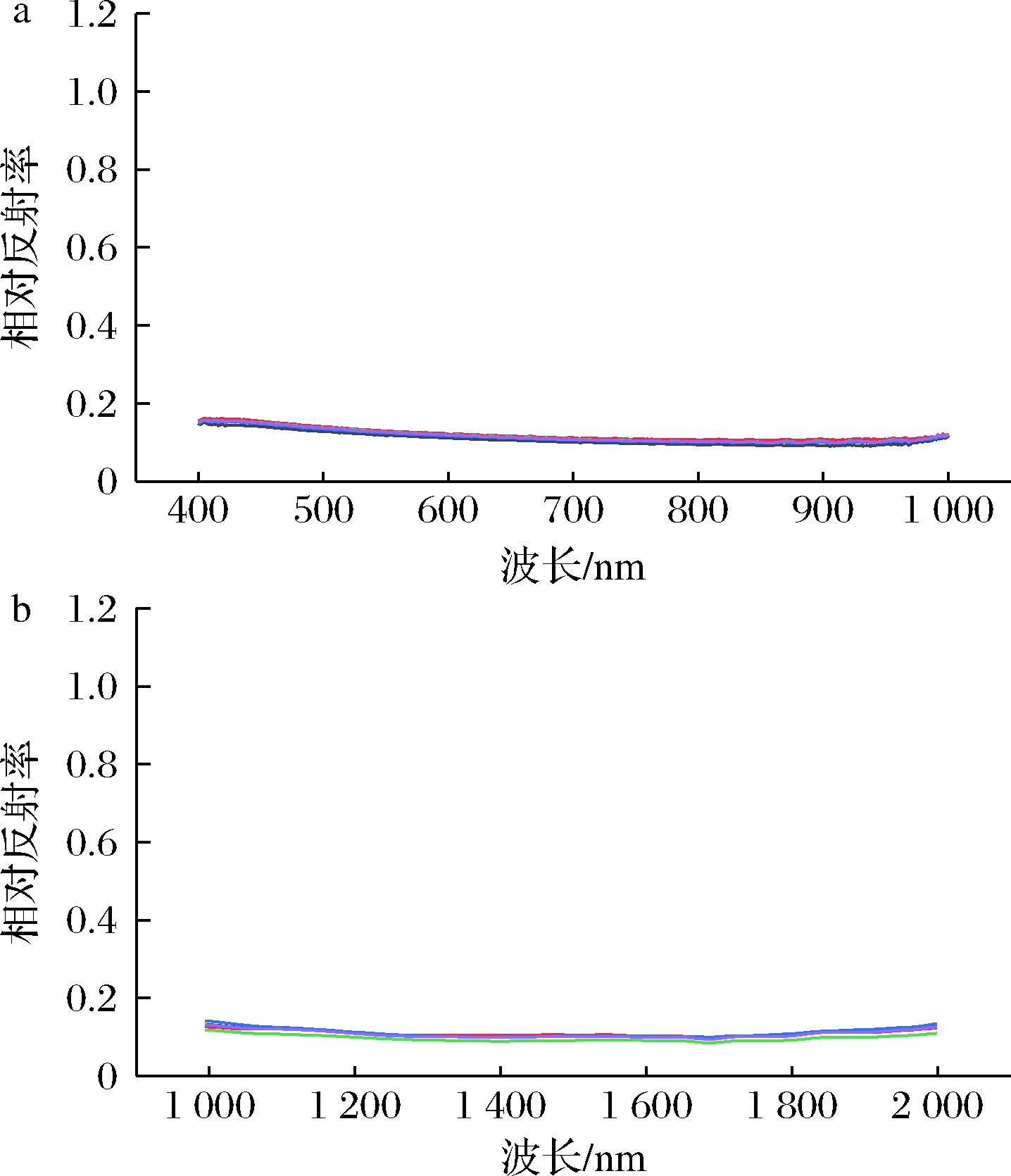
a-400~1 000 nm;b-1 000~2 000 nm
图2 PE保鲜膜在不同波段内的反射光谱图
Fig.2 Reflectance spectra of PE cling film in the different wavelengths
在400~1 000 nm内,非包装和PE包装冷鲜猪大排肉在440、560、590、970 nm处存在较为明显的吸收峰。其中,440 nm处的吸收峰和血红素的卟啉有关[19],560和590 nm处的吸收峰和肌红蛋白等色素有关[20],970 nm处的吸收峰和水分密切相关[21]。在1 000~2 000 nm内,非包装和PE包装冷鲜猪大排肉在1 040、1 220、1 400、1 820和2 000 nm处有明显吸收。其中,1 040和1 400 nm处的吸收峰归因于蛋白质分子结构官能团N—H键一级和二级倍频的伸缩振动,2 000 nm处的吸收峰源于水分子中O—H键的合频吸收[22],1 220 nm和1 820 nm的吸收峰与C—H键的二级和一级倍频吸收有关[23]。
2.3 基于全波段的热杀索丝菌定量预测模型的建立
基于4种经典的预处理算法(SNV、MSC、1 st、Smoothing和OSC)分别构建了400~1 000 nm和1 000~2 000 nm波段下非包装和PE包装冷鲜猪大排肉中热杀索丝菌的定量PLS和SVM模型,结果如表2所示。在400~1 000 nm全波段内,基于不同光谱预处理算法建立的非包装猪大排肉中热杀索丝菌的定量PLS模型中,1 st-PLS模型预测效果最优(Rp2=0.935,RPD=3.827),而在SVM模型中SNV-SVM预测精度最高(Rp2=0.897,RPD=3.095);基于不同预处理算法构建的PE包装猪大排肉中热杀索丝菌定量PLS和SVM模型中的最佳模型分别是OSC-PLS模型(Rp2=0.943,RPD=4.188)和OSC-SVM模型(Rp2=0.923,RPD=3.668)。在1 000~2 000 nm全波段内,经过不同光谱预处理得到非包装组热杀索丝菌的最佳定量PLS和SVM模型分别是OSC-PLS模型(Rp2=0.802,RPD=1.978)和SNV-SVM模型(Rp2=0.479,RPD=1.317);通过不同光谱预处理得到PE包装组热杀索丝菌的最佳定量PLS和SVM模型分别是1 st-PLS模型(Rp2=0.675,RPD=1.791)和OSC-SVM模型(Rp2=0.552,RPD=1.491)。
表2 基于400~1 000 nm和1 000~2 000 nm全波段定量预测非包装和PE包装冷鲜猪大排肉中热杀索丝菌的PLS和SVM模型
Table 2 PLS and SVM models for quantifying B.thermosphacta in unpackaged and PE-packaged chilled pork chops based on full wavebands in 400-1 000 nm and 1 000-2 000 nm

组别建模方法400~1000nm1000~2000nm预处理校正集预测集校正集预测集Rc2RMSEC/(lgCFU/g)Rp2RMSEP/(lgCFU/g)RPDRc2RMSEC/(lgCFU/g)Rp2RMSEP/(lgCFU/g)RPD非包装PLSSVMRaw0.9190.4140.9070.4593.2100.7900.6650.6730.9081.620SNV0.9610.2880.9250.3963.7210.8280.6020.7530.7871.869MSC0.9600.2890.9240.3983.7020.8270.6030.7540.7851.874OSC0.9430.3460.9200.4093.6020.8300.5980.8020.7441.978Smoothing0.9030.4500.8930.4873.0250.5041.0210.4991.0561.3931st0.9480.3290.9350.3853.8270.8060.6390.6880.8501.731Raw0.9070.4470.8450.5702.5850.3481.2020.1081.3801.066SNV0.9480.3320.8970.4763.0950.4491.0860.4791.1171.317MSC0.9460.3390.8950.4813.0630.2451.2620.2661.2631.165OSC0.9230.4050.8750.5222.8230.8520.5680.6511.2131.213Smoothing0.8980.4720.8290.5982.4640.0101.4500.1121.4431.0201st0.8770.5110.8660.5422.7180.2771.2460.3541.1821.245PE包装PLSSVMRaw0.9210.4620.9060.5103.3010.7570.8100.5871.1241.498SNV0.9260.4470.9220.4633.6360.8080.7200.6111.0601.588MSC0.9260.4480.9220.4633.6360.8080.7200.6121.0601.588OSC0.9450.3850.9430.4024.1880.8500.6380.6561.0101.667Smoothing0.9150.4790.8880.5573.0220.7480.8260.6031.1051.5241st0.9380.4080.9330.4313.9060.7740.7830.6750.9401.791Raw0.9630.3210.8670.6032.7920.3951.3570.5221.3221.273SNV0.9830.2170.8730.6112.7550.9060.5220.4471.3071.288MSC0.9730.2700.8690.6172.7290.5461.1850.4401.2701.326OSC0.9560.3510.9230.4593.6680.7810.7730.5521.1291.491Smoothing0.9620.3270.8610.6172.7290.4111.3710.3671.4021.2011st0.8860.5610.8110.7172.3480.3701.3710.3021.4411.168
注:Raw表示不采用预处理,下同。
综上,基于400~1 000 nm全波段构建的热杀索丝菌定量PLS和SVM模型中,无论是非包装组还是PE包装组,PLS模型都表现出最佳预测精度,其最优预处理方式分别为1 st和OSC,RPD均大于3.0,表明两个模型的预测能力非常好。其原因可能是PLS模型易于识别系统信息与噪声变量且能够通过选择主因子来筛除无效信息并提高模型精度[24],而SVM模型对参数和核函数的选定较复杂且对噪声变量敏感,其预测性能易受到影响[17],这在校正集和预测集数据集中分布在理想预测线上下的图3-a1和3-b1中得到了印证。同时,这也表明即使PE保鲜膜对光谱信息会产生一定影响,但建模精度仍能达到较高水平[25]。而基于1 000~2 000 nm全波段构建的PLS和SVM定量模型中有相似现象,即非包装和PE包装组热杀索丝菌的最佳预测模型均是PLS模型,不同之处在于最优预处理算法(OSC和1 st)不同,且两模型的RPD均小于2.5,表明模型预测能力较差,这也反映在了校正集及预测集中预测值和实测值均较为接近的图3-a2和3-b2中。由此可见,相比于1 000~2 000 nm全波段光谱值,基于400~1 000 nm全波段的PLS模型更适合用于定量预测非包装和PE包装组中热杀索丝菌,这与马骥[6]基于400~1 000 nm和1 000~2 000 nm的高光谱数据预测冷鲜猪肉中菌落总数的研究结论一致,即400~1 000 nm的高光谱数据更适合冷鲜猪肉中菌落总数的预测。

图3 不同波段下非包装(a1和a2)和PE包装(b1和b2)猪大排肉中热杀索丝菌最佳预测结果
(1:400~1 000 nm;2:1 000~2 000 nm)
Fig.3 Best prediction results of B.thermosphact in unpackaged (a1 and b1) and PE-packaged (a2 and b2) pork chops at different wavelengths
2.4 基于特征波长的热杀索丝菌定量预测模型的建立
采用SPA和CARS算法分别从原始光谱和预处理(SNV、MSC、1 st、Smoothing和OSC)后的光谱中筛选特征波长,结果如表3所示。在400~1 000 nm波段范围内,利用SPA算法分别从非包装和PE包装组冷鲜猪大排肉样品的高光谱数据中筛选出9和14个特征波长,波段数量减少率分别为97.9%和96.7%;而采用CARS算法分别从非包装和PE包装组的高光谱数据中筛选出20和25个特征波长,其波段数量减少率与SPA算法相当,分别为95.2%和94.0%。此外,在1 000~2 000 nm波段范围内,利用SPA算法分别从非包装和PE包装组的高光谱数据中筛选出特征波长10和14个,波段数量减少率分别为93.0%和90.2%;而采用CARS算法分别从非包装和PE包装组的高光谱数据中筛选出特征波长14和21个,波段数量减少率90.2%和85.3%,略低于SPA算法。
表3 基于SPA和CARS算法的特征波长筛选结果
Table 3 Results for the selection of feature wavelengths based on SPA and CARS algorithm

组别波段筛选方法400~1000nm1000~2000nm波段数波长/nm减少比率/%波段数波长/nm减少比率/%非包装SPA9436.41、452.78、577.20、598.60、615.80、635.93、683.67、980.25、993.3097.910994.80、1019.96、1051.99、1104.47、1124.52、1144.75、1165.15、1213.41、1894.34、1929.4393.0CARS20440.49、541.77、543.18、582.89、667.72、722.97、724.42、823.93、832.72、834.19、835.65、863.50、897.17、900.09、901.56、961.37、978.80、988.95、993.30、994.7495.214994.80、1007.33、1019.96、1124.52、1241.35、1837.47、1851.76、1858.89、1894.34、1901.39、1936.40、1950.30、1957.22、1998.3990.2PE包装SPA14422.85、432.34、437.77、496.95、557.32、575.77、584.32、600.03、634.49、653.25、692.39、765.33、815.13、999.0996.714994.80、1013.63、1039.10、1144.75、1185.73、1269.53、1290.82、1377.01、1420.62、1604.23、1736.26、1823.13、1866.00、1971.0290.2CARS25406.69、425.56、428.27、429.62、455.52、571.51、585.75、598.60、622.98、638.82、640.26、653.25、657.59、661.93、663.38、666.27、692.39、711.31、769.72、772.65、774.11、845.91、850.31、949.72、999.0994.021994.80、1007.33、1032.70、1039.10、1124.52、1144.75、1199.54、1248.37、1255.41、1312.21、1319.37、1326.54、1427.91、1604.23、1611.59、1655.72、1685.08、1758.09、1794.32、1823.13、1922.4485.3
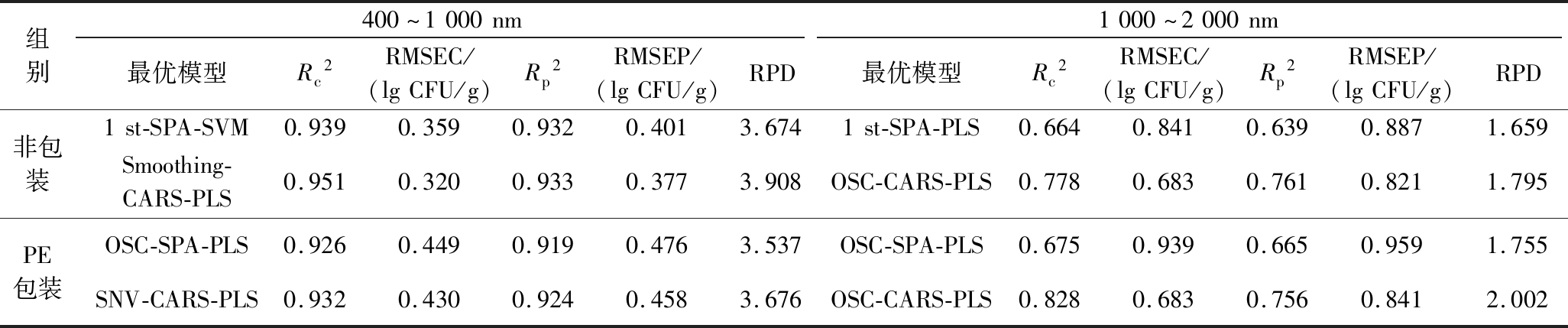
基于SPA和CARS算法筛选的特征波长构建了非包装和PE包装组的猪大排肉中热杀索丝菌的PLS和SVM定量模型,建模结果如表4所示(仅展示基于SPA和CARS算法结合不同预处理算法获得的最优预测模型)。在400~1 000 nm波段范围内,非包装组中最佳的热杀索丝菌定量预测模型分别是1 st-SPA-SVM模型(Rp2=0.932,RPD=3.674)和Smoothing-CARS-PLS模型(Rp2=0.933,RPD=3.908),RPD均大于3.0,展示出极佳的预测性能;PE包装组中最优的热杀索丝菌定量预测模型分别是OSC-SPA-PLS模型(Rp2=0.919,RPD=3.537)和SNV-CARS-PLS模型(Rp2=0.924,RPD=3.676),RPD均大于3.0,表明模型性能极佳。与全波段相比,采用SPA算法提取特征波长构建非包装和PE包装组中热杀索丝菌的预测模型时,模型预测精度均有所下降,但Rp2仅分别降低了0.003和0.024,且仅分别使用了2.1%和3.3%的波长信息;而基于CARS算法筛选的特征波长的最优热杀索丝菌定量模型精度也略微下降,但![]() 仅分别降低了0.002和0.019,且仅使用了4.8%和6.0%的波长信息。虽然基于全波段的模型精度略好于特征波长,但基于特征波长的模型预测性能良好,这无疑在一定程度上消除了数据冗余,减小了运算量,提高了运算效率。图4-a1、4-b1、4-c1和4-d1显示上述最佳建模结果,其显示无论是校正集还是预测集中预测值和实测值均较为接近,同样表明在400~1 000 nm波段内基于SPA和CARS算法的非包装和PE包装组热杀索丝菌的最佳定量模型具有较高预测精度,具有一定实际应用价值。
仅分别降低了0.002和0.019,且仅使用了4.8%和6.0%的波长信息。虽然基于全波段的模型精度略好于特征波长,但基于特征波长的模型预测性能良好,这无疑在一定程度上消除了数据冗余,减小了运算量,提高了运算效率。图4-a1、4-b1、4-c1和4-d1显示上述最佳建模结果,其显示无论是校正集还是预测集中预测值和实测值均较为接近,同样表明在400~1 000 nm波段内基于SPA和CARS算法的非包装和PE包装组热杀索丝菌的最佳定量模型具有较高预测精度,具有一定实际应用价值。

1:400~1 000 nm;2:1 000~2 000 nm;a:非包装-SPA;b:非包装-CARS;c:PE包装-SPA;d:PE包装-CARS
图4 基于SPA和CARS算法的非包装和PE包装猪大排肉中热杀索丝菌最佳预测结果
Fig.4 Best prediction results of B.thermosphacta in unpackaged and PE-packaged pork chops based on SPA and CARS algorithms
表4 基于SPA和CARS算法筛选的特征波段的非包装和PE包装组中热杀索丝菌PLS和SVM定量模型
Table 4 PLS and SVM models for quantifying B.thermosphact in unpackaged and PE-packaged groups based on feature bands from SPA and CARS algorithms
组别400~1000nm1000~2000nm最优模型Rc2RMSEC/(lgCFU/g)Rp2RMSEP/(lgCFU/g)RPD最优模型Rc2RMSEC/(lgCFU/g)Rp2RMSEP/(lgCFU/g)RPD非包装1st-SPA-SVM0.9390.3590.9320.4013.6741st-SPA-PLS0.6640.8410.6390.8871.659Smoothing-CARS-PLS0.9510.3200.9330.3773.908OSC-CARS-PLS0.7780.6830.7610.8211.795PE包装OSC-SPA-PLS0.9260.4490.9190.4763.537OSC-SPA-PLS0.6750.9390.6650.9591.755SNV-CARS-PLS0.9320.4300.9240.4583.676OSC-CARS-PLS0.8280.6830.7560.8412.002
此外,由表4可知,在1 000~2 000 nm波段范围内,非包装组中热杀索丝菌的最优定量模型分别是1 st-SPA-PLS模型(Rp2=0.639,RPD=1.659)和OSC-CARS-PLS模型(Rp2=0.761,RPD=1.795);PE包装组热杀索丝菌的最优定量模型分别是OSC-SPA-PLS模型(Rp2=0.665,RPD=1.755)和OSC-CARS-PLS模型(Rp2=0.756,RPD=2.002)。以上最优定量模型的RPD均小于2.5,说明经过SPA和CARS算法筛选特征波长后仍无法提高基于1 000~2 000 nm内的光谱数据建立的非包装和PE包装猪大排肉热杀索丝菌的定量预测模型,模型精度较差,无法用于实际。从图4-a2、4-b2、4-c2和4-d2中也能清楚观察到此结果,无论是校正集还是预测集中预测值和实测值均较为接近。由此可知,无论是采用SPA算法还是CARS算法筛选1 000~2 000 nm波段内的特征波长后建立非包装和PE包装猪大排肉中热杀索丝菌的定量模型,所得模型预测效果均不尽人意,预测精度明显低于基于400~1 000 nm波段内的光谱数据所建立的模型。
对比SPA和CARS两种算法筛选特征波长后建立的热杀索丝菌定量预测模型,发现CARS算法筛选的特征波长所建模型精度更高,其原因可能是CARS算法筛选的特征波长数量较多,特征信息更加丰富,然而这也在一定程度上包含了部分无效冗余信息,降低了模型的运算速度,这和龙家美[26]的研究结果一致。另一方面,相比之下,SPA算法筛选的特征波长数目较少但所建模型预测精度较高,考虑到检测时间、运算速度等因素,选择SPA算法筛选的特征波长或将具有更好的实际应用性。对比基于400~1 000 nm的全波段和特征波长建立的非包装和PE包装冷鲜猪大排肉中热杀索丝菌的定量模型,发现PE包装组的多个模型![]() 均能达到0.9以上且RPD大于3.0,表明PE保鲜膜对预测结果的干扰较小,所建模型仍有较高的预测性能。
均能达到0.9以上且RPD大于3.0,表明PE保鲜膜对预测结果的干扰较小,所建模型仍有较高的预测性能。
3 结论与讨论
本文利用可见-近红外(400~1 000 nm)和短波红外(1 000~2 000 nm)高光谱成像系统采集非包装和PE包装冷鲜猪大排肉的光谱信息,选择不同的光谱预处理方式结合SPA和CARS算法筛选特征波长,建立了基于全波段和特征波长的非包装和PE包装冷鲜猪大排肉中热杀索丝菌的定量PLS和SVM模型。结果表明,在400~1 000 nm和1 000~2 000 nm内,PE包装组光谱值略低于非包装组,PE保鲜膜对光谱响应的影响不明显。基于两个全波段构建的非包装和PE包装猪大排肉中热杀索丝菌的定量PLS和SVM模型,PLS模型均展现出最佳预测效果,且相比于1 000~2 000 nm,基于400~1 000 nm内全波段光谱值更适合用于定量预测非包装和PE包装组的热杀索丝菌,最优预处理方式分别为1 st和OSC,RPD均大于3.0,模型预测性能非常好。此外,无论是基于SPA算法还是CARS算法筛选的400~1 000 nm波段内的特征波长定量预测非包装和PE包装组中热杀索丝菌的效果均优于1 000~2 000 nm内的,其中,基于SPA算法筛选的特征波长建立的预测模型在最大限度减少波段的同时预测精度较高,非包装组最优模型为1 ![]() RPD=3.674),PE包装组最优模型为
RPD=3.674),PE包装组最优模型为![]() RPD=3.537)。综上,400~1 000 nm内的光谱值适合于定量预测非包装和PE包装猪大排肉中的热杀索丝菌,这为HSI技术在PE包装或者其他包装冷鲜肉中微生物的批量检测奠定了理论基础。尽管如此,1 000~2 000 nm波段下的模型预测效果差的原因仍不明朗,PE保鲜膜及其他种类的包装对光谱响应的具体影响还有待进一步深入研究。
RPD=3.537)。综上,400~1 000 nm内的光谱值适合于定量预测非包装和PE包装猪大排肉中的热杀索丝菌,这为HSI技术在PE包装或者其他包装冷鲜肉中微生物的批量检测奠定了理论基础。尽管如此,1 000~2 000 nm波段下的模型预测效果差的原因仍不明朗,PE保鲜膜及其他种类的包装对光谱响应的具体影响还有待进一步深入研究。
[1] 邓森荣,向蓉,魏琦麟,等.冷鲜肉微生物种类及微生物预测模型的研究进展[J].广东农业科学,2021,48(3):133-141.
DENG S R,XIANG R,WEI Q L,et al.Research progress in microbial species and microbial prediction model of chilled meat[J].Guangdong Agricultural Sciences,2021,48(3):133-141.
[2] 何鸿举,蒋圣启,马汉军,等.基于NIR高光谱技术快速预测冷鲜鸡肉热杀索丝菌含量[J].食品工业科技,2020,41(13):241-246;252.
HE H J,JIANG S Q,MA H J,et al.NIR hyperspectral imaging technology for rapid prediction of Brochothrix thermosphacta in fresh chilled chicken[J].Science and Technology of Food Industry,2020,41(13):241-246;252.
[3] 张婧.平板计数法与纸片法检测食品微生物菌落总数的比较分析[J].中国食品,2021(3):92.
ZHANG J.Comparative analysis of microbial colony count of food by plate counting and paper tablet method[J].China Food,2021(3):92.
[4] 张建军,唐轶君,李晶,等.基于测试片法与平板计数法对不同类型食品中菌落总数结果的比较分析[J].食品安全质量检测学报,2021,12(14):5606-5612.
ZHANG J J,TANG Y J,LI J,et al.Comparative analysis of the results of total number of colonies in different types of food based on test piece method and plate counting method[J].Journal of Food Safety &Quality,2021,12(14):5606-5612.
[5] 周慧,支竹伟,刘涵,等.多重实时荧光定量PCR检测肴肉中的特定腐败菌[J].粮食与食品工业,2014,21(3):86-89;92.
ZHOU H,ZHI Z W,LIU H,et al.Multiplex real-time quantitive PCR detection of specific spoilage organisms in Yao meat[J].Cereal &Food Industry,2014,21(3):86-89;92.
[6] 马骥.基于高光谱成像技术的加工猪肉品质快速检测方法研究[D].广州:华南理工大学,2019.
MA J.Research on rapid detection methods for the quality of processed pork based on hyperspectral imaging technology[J].Guangzhou:South China University of Technology,2019.
[7] 曲佳欢,马骥,孙大文,等.高光谱成像在食品质量评估方面的研究进展与应用(一)[J].肉类研究,2012,26(4):40-45.
QU J H,MA J,SUN D W,et al.Researches and applications of hyperspectral imaging in food industry(Ⅰ)[J].Meat Research,2012,26(4):40-45.
[8] TAO F F,PENG Y K.A nondestructive method for prediction of total viable count in pork meat by hyperspectral scattering imaging[J].Food and Bioprocess Technology,2015,8(1):17-30.
[9] 庄齐斌,郑晓春,杨德勇,等.基于高光谱反射特性的猪肉新鲜度和腐败程度的对比分析[J].食品科学,2021,42(16):254-260.
ZHUANG Q B,ZHENG X C,YANG D Y,et al.Comparative analysis of pork freshness and spoilage based on hyperspectral reflection characteristics[J].Food Science,2021,42(16):254-260.
[10] ACHATA E M,OLIVEIRA M,ESQUERRE C A,et al.Visible and NIR hyperspectral imaging and chemometrics for prediction of microbial quality of beef Longissimus dorsi muscle under simulated normal and abuse storage conditions[J].LWT,2020,128:109463.
[11] 顾欣哲.基于电子鼻响应信息的猪肉假单胞菌生长预测模型的构建[D].南京:南京农业大学,2017.
GU X Z.Constructing the predictive growth model of Pseudomonas in pork based on electronic nose[D].Nanjing:Nanjing Agricultural University,2017.
[12] ZHOU B J,FAN X,SONG J,et al.Growth simulation of Pseudomonas fluorescens in pork using hyperspectral imaging[J].Meat Science,2022,188:108767.
[13] LI J L,SUN D W,CHENG J H.Recent advances in nondestructive analytical techniques for determining the total soluble solids in fruits:A review[J].Comprehensive Reviews in Food Science and Food Safety,2016,15(5):897-911.
[14] 周孟然,余道洋,胡锋,等.基于连续投影算法的食用油激光诱导荧光光谱特征波长筛选[J].河南师范大学学报(自然科学版),2021,49(2):46-53.
ZHOU M R,YU D Y,HU F,et al.Continuous projection algorithm for screening characteristic wavelength of laser induced fluorescence spectrum of edible oil[J].Journal of Henan Normal University (Natural Science Edition),2021,49(2):46-53.
[15] 郭城,吴卫成,谌迪,等.基于ATR-FTIR结合CARS算法测定蜂王浆中水溶性蛋白质和总糖含量[J].中国食品学报,2021,21(11):183-190.
GUO C,WU W C,CHEN D,et al.Determination of water-soluble protein and total sugar in royal jelly based on attenuated total reflection mid-infrared spectroscopy and CARS algorithm[J].Journal of Chinese Institute of Food Science and Technology,2021,21(11):183-190.
[16] 杜尚广.基于近红外光谱技术快速评价芸苔属种子活力[D].南昌:南昌大学,2015.
DU S G.Rapid assessment of seed vigor of Brassica based on near on infrared spectroscopy[D].Nanchang:Nanchang University,2015.
[17] 张亚坤.基于多尺度高光谱成像的大豆养分检测方法研究[D].哈尔滨:东北农业大学,2018.
ZHANG Y K.Research on detection method of soybean nutrient based on multiscale hyperspectral imaging technology[D].Harbin:Northeast Agricultural University,2018.
[18] WU D,SUN D W.Potential of time series-hyperspectral imaging (TS-HSI) for non-invasive determination of microbial spoilage of salmon flesh[J].Talanta,2013,111:39-46.
[19] CHEN L J,YANG Z L,HAN L J.A review on the use of near-infrared spectroscopy for analyzing feed protein materials[J].Applied Spectroscopy Reviews,2013,48(7):509-522.
[20] SWATLAND H J.Internal Fresnel reflectance from meat microstructure in relation to pork paleness and pH[J].Food Research International,1997,30(8):565-570.
[21] SIVERTSEN A H,KIMIYA T,HEIA K.Automatic freshness assessment of cod (Gadus morhua) fillets by Vis/Nir spectroscopy[J].Journal of Food Engineering,2011,103(3):317-323.
[22] 程玮玮.低温保藏猪肉有机生物大分子光谱成像规律与快速检测方法研究[D].广州:华南理工大学,2018.
CHENG W W.Study on spectral imaging characteristics and rapid detection method of pork meat organic macromolecules preserved at low temperature[D].Guangzhou,South China University of Technology,2018.
[23] KAMRUZZAMAN M,ELMASRY G,SUN D W,et al.Prediction of some quality attributes of lamb meat using near-infrared hyperspectral imaging and multivariate analysis[J].Analytica Chimica Acta,2012,714:57-67.
[24] 张伏生,汪鸿,韩悌,等.基于偏最小二乘回归分析的短期负荷预测[J].电网技术,2003,27(3):36-40.
ZHANG F S,WANG H,HAN T,et al.Short-term load forecasting based on partial least-squares regression[J].Power System Technology,2003,27(3):36-40.
[25] 陈雅,姜凯译,李耀翔,等.基于近红外的PE包装蓝莓新鲜度无损检测[J].包装工程,2022,43(7):1-10.
CHEN Y,JIANG K Y,LI Y X,et al.Nondestructive detection of freshness of PE packaged blueberries based on NIR[J].Packaging Engineering,2022,43(7):1-10.
[26] 龙家美.基于高光谱和多光谱成像技术的干制红枣水分、VC和总糖含量无损检测[D].南京:南京农业大学,2021.
LONG J M.Non-destructive detection of moisture,VC and total sugar content of dried jujube based on hyperspectral and multispectral imaging technology[D].Nanjing:Nanjing Agricultural University,2021.