几丁质是以N-乙酰氨基-β-葡萄糖为单位,由β-1,4-糖苷键连接的多聚糖,根据(C8H13NO5)n分子链排列方向,可将几丁质分为α型、β型以及γ型:α型中分子链以反向平行方式排列;β型中分子链以正交方式排列;γ型中分子链以蜂巢方式排列[1-2]。几丁质是节肢动物外骨骼和丝状真菌细胞壁的主要组成成分,来自于海洋的虾、蟹壳几丁质含量高达14%~17%,是人类获得几丁质的主要来源[3]。降解几丁质的方法主要包括化学法、微生物发酵、酶水解等[4]。其中化学法主要是通过强酸、强碱等方式进行处理,伴随而来的是资源浪费以及环境污染。因此通过微生物发酵法和酶水解降解几丁质是未来必然的选择。而其中,微生物发酵法所用微生物可能具有致病性,酶解法则能够直接利用不同来源的几丁质酶降解,避免致病性微生物对人体的危害,因此,应用几丁质酶,特别是来源海洋微生物几丁质酶,能够有效降解几丁质生产N-乙酰氨基葡萄糖,尤其在海产品加工中,具有重要的经济意义[5]。
几丁质酶是一种能够将几丁质彻底降解为N-乙酰氨基葡萄糖单体或寡聚体的糖苷水解酶[6]。根据其作用方式的不同,可分为内切几丁质酶与外切几丁质酶:内切几丁质酶通过随机切割几丁质内部的β-1,4-糖苷键发挥酶解作用,生成N-乙酰氨基葡萄糖寡聚体或几丁质二糖;外切几丁质酶则是从非还原端依次切割几丁二糖单位,生成几丁二糖或N-乙酰氨基葡萄糖单体[7-9]。源于细菌的几丁质酶多属于糖苷水解酶18(glycoside hydrolase 18,GH18)家族,根据催化域氨基酸序列相似性,GH18家族几丁质酶又分为A、B、C 3个亚家族,目前对A家族外切几丁质酶研究最为广泛[7]。根据CAZy数据库统计,微生物来源的几丁质酶主要存在于细菌,其中变形菌门、厚壁菌门、放线菌门中大量菌种可产几丁质酶,变形菌门的弧菌属是细菌几丁质酶重要来源,该属微生物可以几丁质为唯一能量来源迅速生长,同时分泌GH18家族的多种几丁质酶,实现协同高效降解天然几丁质目的[10]。目前研究发现,弧菌所产生的几丁质酶在pH 5.0~10.0能够保持86%以上活性,在50 ℃下保温1 h能够剩余60%以上活性,与陆地来源几丁质酶相比,具有较好的热稳定性以及pH稳定性[11]。
本研究从天津渤海滩涂获得一株产几丁质酶弧菌,并对其进行分子鉴定,及其发酵所产几丁质酶的最适酶解条件和酶稳定性进行探索。为筛选分离高活性产几丁质酶微生物提供有效方法,并对弧菌几丁质酶及其酶学性质进行研究,为降解利用几丁质提供基础研究数据。
1 材料与方法
1.1 材料与设备
1.1.1 样品来源
滩涂沉积物:中国天津市滨海新区东疆湾海岸,北纬N:39°0′11.55″,东经E:117°49′54.71″,取样置于车载冰箱,立即带回实验室进行菌株筛选及分离纯化。
1.1.2 培养基
富集培养基(g/L):粉状几丁质4.0、酵母粉3.0、蛋白胨3.0、MgSO4·7H2O 0.5、KH2PO4 0.7、K2HPO4 0.3、FeSO4·7H2O 0.01。
筛选培养基(g/L):质量分数0.5%胶体几丁质溶液40 mL、酵母粉3.0、蛋白胨3.0、MgSO4·7H2O 0.5、KH2PO4 0.7、K2HPO4 0.3、FeSO4·7H2O 0.01、琼脂粉20.0(固体培养基加入)。
发酵培养基(g/L):质量分数0.5%胶体几丁质溶液40 mL、酵母粉5.0、蛋白胨5.0、MgSO4·7H2O 0.5、KH2PO4 0.7、K2HPO4 0.3、FeSO4·7H2O 0.01[12]。
所有培养基均121 ℃灭菌20 min。
1.1.3 仪器与设备
TDA-8002型水浴锅,天津市外环实验电炉有限公司;752紫外光栅分光光度计,上海准确科学仪器有限公司;SPX-250B-Z型生化培养箱,上海迅雷有限公司医疗设备厂;pHSJ-4A型实验pH计,上海生物仪器有限公司;P型移液器,德国艾朋朵夫公司;TCL-12台式高速冷冻离心机,中科院生物物理所技术服务公司;UV-1型三用紫外分析仪,上海化工电光仪器厂;DL102型电热鼓风干燥箱,天津市化工实验仪器厂;Dionex Ultimate 3000 HPLC系统,赛默飞世尔科技公司。
1.2 实验方法
1.2.1 胶体几丁质的制备
参考文献[13]的方法制备胶体几丁质。将5.0 g粉状几丁质加入到100 mL浓盐酸中,缓慢搅拌至无颗粒黏稠液体状态,然后将其放入4 ℃冷藏24 h。在通风橱向浓盐酸处理后的液体中加入超纯水,6 000 r/min离心20 min,多次超纯水离心洗涤至pH为中性。
1.2.2 DNS试剂的制备
参考文献[14]的方法制备DNS试剂。
1.2.3 菌株的筛选及鉴定
1.2.3.1 富集培养
取滩涂沉积物约5.0 g于50 mL富集培养基中,37 ℃、180 r/min摇瓶培养72 h。
1.2.3.2 透明圈菌落的筛选及纯化
将富集培养后的培养液稀释至10-6,取0.1 mL菌液涂布至固体筛选培养基。37 ℃培养2 d,挑取产生透明圈的单菌落,将单菌落在筛选平板上继续纯化3代。
1.2.3.3 菌种的16S rDNA基因测序以及生理生化鉴定
以纯化后的菌落基因组为模板,引物27F:5′-AGAGTTTGATCCTGGCTCAG-3′和1492R:5′-GGTTACCTTGTTACGACTT-3′进行PCR扩增。将扩增产物送至金唯智生物技术有限公司进行测序。测序结果在NCBI数据库中进行序列比对并进行序列分析。
根据伯杰氏手册以及GB 4789.7—2013《食品安全国家标准 食品微生物学检验 副溶血性弧菌检验》进行菌株生理生化鉴定。
1.2.3.4 几丁质酶基因的克隆
根据弧菌来源的几丁质酶氨基酸保守区域DGCDID和YGRGW,设计简并引物GH18A-F:5′-GAYGGNGTDGAYATHGAYTGGGA-3′和GH18A-R:5′-CCARCCNCGRCCRTACATNG-3′。以纯化后的菌株基因组为模板,克隆几丁质酶保守区域,送至金唯智生物技术有限公司进行测序。对其保守区段进行序列分析,找到所对应的几丁质酶基因全长并克隆,使用MEGA 11软件构建进化树。
1.2.4 发酵上清液的SDS-PAGE及蛋白质谱分析
发酵液4 ℃、6 000 r/min离心15 min,上清液即为粗酶液。粗酶液使用10 kDa分子截留量超滤管浓缩10倍后,SDS-PAGE检测粗酶液中蛋白表达情况,并将样品送至中国科学院微生物研究所质谱与功能组学平台进行蛋白质谱分析。
1.2.5 粗酶液酶活力的测定
粗酶液酶活力的测定采用DNS法,并配制1.0 mg/mL的N-乙酰氨基葡萄糖标准溶液绘制标准曲线[2,15]。
酶活力单位(U)的定义[16]:在55 ℃条件下水浴30 min,1 min催化底物产生1 μmol N-乙酰氨基葡萄糖所需要的酶量为一个酶活力单位U。
1.2.6 粗酶液水解产物分析
浓缩后的粗酶液与胶体几丁质在最适条件下进行反应不同时间,将水解产物进行HPLC分析。流动相:V(正辛烷磺酸钠磷酸缓冲液)∶V(乙腈)=97∶3,液体使用前经0.45 μm滤膜进行过滤除杂并超声波去除气泡;流速0.6 mL/min;色谱柱:SB-C18(4.6 mm×250 mm,5 μm);柱温30 ℃;190 nm下进行紫外检测;进样量10 μL;标准品:0.4 mg/mL N-乙酰氨基葡萄糖和1.6 mg/mL氨基葡萄糖混合标品。
1.2.7 酶学性质测定
1.2.7.1 最适底物及其底物浓度的选择
将酶液分别于质量分数为1.0%的天然几丁质、壳聚糖和胶体几丁质以1∶9(体积比)反应体系于55 ℃反应30 min,加入DNS后煮沸显色,在540 nm处测量吸光度。
确定最适底物后,以0.5%、1.0%、1.5%和2.0%质量分数的底物与酶液以9∶1(体积比)反应体系于55 ℃反应30 min,加入DNS后煮沸5 min后显色,在540 nm处测量吸光度。
1.2.7.2 最适温度及pH
将酶液与底物在1∶9(体积比)反应体系混合,分别于25、30、35、40、45、50、55、60、65、70 ℃下反应30 min,加入DNS后煮沸显色,测量540 nm处吸光度,计算相对酶活力。
配制pH为4.0、5.0、6.0、7.0、8.0、9.0、10.0缓冲液的体积分数为1.0%的底物,将酶液与底物以1∶9(体积比)反应体系于最适温度下反应30 min,加入DNS后煮沸显色,测量540 nm吸光度,计算相对酶活力。
1.2.7.3 温度及pH的稳定性
确认最适温度后,进行温度稳定性实验。将酶液置于最适pH缓冲液并分别于30、40、45、50、55、60、70 ℃保温1 h,再在最适条件下与底物反应30 min,DNS法测量540 nm处吸光度,计算相对酶活力。
将酶液10倍稀释至不同pH的缓冲溶液中,37 ℃保温1 h后,于最适温度和最适pH条件下与底物反应30 min,DNS法测量540 nm处吸光度,计算相对酶活力。
1.2.7.4 金属离子及表面活性剂对于酶活力的影响
分别配制含有Mg2+、Zn2+、Cu2+、Mn2+、Li+、Ca2+、Fe2+、Na+、K+的溶液和EDTA溶液,终浓度为10 mmol/L;配制体积分数为0.1%的SDS、吐温-20、吐温-80溶液。将各溶液和酶液充分混匀后于37 ℃保温1 h,再以与底物体积比为1∶9的反应体系于最适温度和最适pH条件下反应30 min,DNS法测量540 nm处吸光度,计算相对酶活力,对照组酶活力为100%。
2 结果与分析
2.1 产几丁质酶菌株的筛选及鉴定
经过富集培养后,从样品中共筛选出20株于几丁质酶筛选培养基中产生透明圈的菌株,通过比较透明圈与菌落直径比,挑选出比值最大的Y-8菌株作为出发菌株进行后续试验(圈径比于120 h达到最大值,圈径比值3.6)。
2.1.1 Y-8菌株菌落形态观察
如图1所示,Y-8菌株于固体筛选培养基平板上37 ℃培养24 h,菌落直径2 mm,菌落表面湿润凸起,边缘整齐。染色观察为革兰氏阴性细菌,呈短弧状。
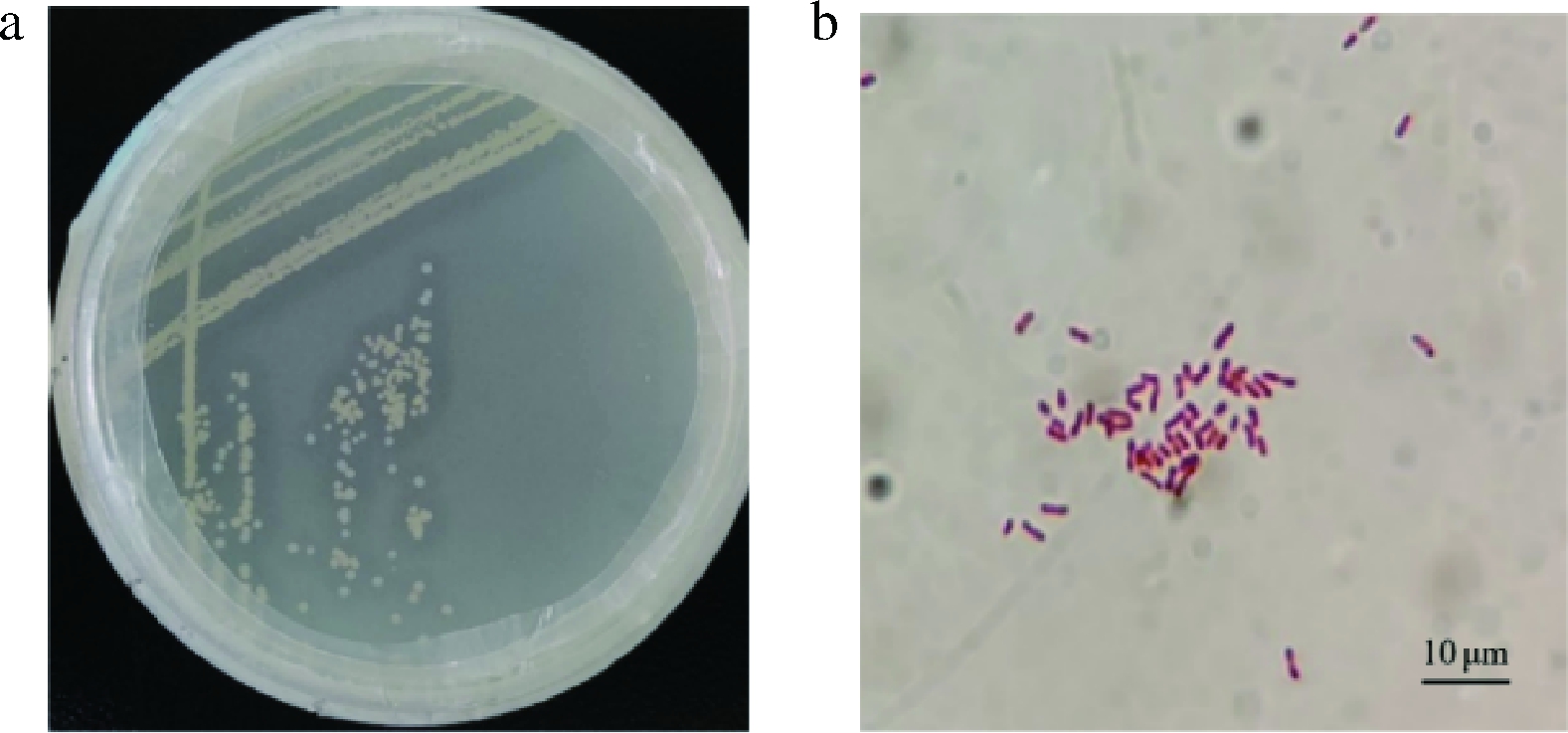
a-Y-8单菌落;b-Y-8显微镜形态
图1 菌株Y-8单菌落形态观察
Fig.1 Colony morphology of bacterial Y-8
2.1.2 16S rDNA序列扩增及其测序分析
以Y-8菌株基因组为模板,以27F和1492R为引物,经PCR扩增其部分16S rDNA,其测序结果为1 420 bp。经GenBank数据库检索鉴定,Y-8菌株属于弧菌属(Vibrio sp.)。
2.1.3 菌株生理生化鉴定
菌株生理生化鉴定结果如表1所示,Y-8菌株的生理生化特性与副溶血性弧菌完全符合,确定Y-8菌株为副溶血性弧菌(Vibrio parahaemolyticus)。
表1 菌株Y-8生理生化鉴定
Table 1 Physiological and biochemical identification of strain Y-8

生理生化检测项目实验结果标准结果革兰氏染色--氧化酶++动力++蔗糖--葡萄糖++甘露醇++分解葡萄糖产气--乳糖--H2S--赖氨酸脱羧酶++V-P--ONPG--
注:+-表示阳性;--表示阴性。
2.1.4 Y-8菌株的几丁质酶基因克隆与分析
为获得Y-8菌株几丁质酶基因序列,对5个来自弧菌属的几丁质酶氨基酸序列进行对比分析,氨基酸多序列比对结果如图2-a所示,找到几丁质酶中保守的氨基酸序列DGVDID和YGRGW。

a-多序列比对图;b-系统发育树
图2 多序列比对及基于几丁质酶氨基酸序列构建Y-8菌株NJ系统发育树
Fig.2 Multi-sequence alignment and Neighbor-joining phylogenetic tree of strain Y-8 based on the amino acid sequence of chitinase
注:多序列比对中所选取的几丁质酶氨基酸序列来自于Vibrio splendidus(RIH72604.1),Vibrio parahaemolyticus(OCP51821.1),Vibrio parahaemolyticus(OAR41713.1),Vibrio harveyi CAIM 1792(EMR37610.1),Vibrio cholerae LMA3 984-4(AEA79673.1)
这与连文浩[16]研究中细菌来源的GH18家族催化活性中心的氨基酸序列“DGXDXD”和“YXRGW”一致,利用此保守的氨基酸序列设计的简并引物,以Y-8菌株基因组为模板进行保守域核苷酸序列克隆并测序,与已知的弧菌属来源几丁质酶核苷酸序列比对,结果与几丁质酶MBE4005454.1、几丁质酶HAS3048952.1保守区相似度达99.76%和99.43%。以几丁质酶MBE4005454.1序列为参考,设计上下游引物,克隆Y-8菌株几丁质酶chi1基因全长并测序,最终与MBE4005454.1核苷酸序列全长相似度达到99.68%。最终从Y-8菌株中克隆到2条几丁质酶基因序列,其中chi1大小为2 547 bp预测编码848个氨基酸,理论分子质量90.0 kDa,chi2大小为3 165 bp预测编码1 054个氨基酸,理论分子质量111.83 kDa;二者分别与几丁质酶MBE4005454.1及HAS3048952.1相似度达99.76%和99.43%。
根据氨基酸序列构建系统发育树图2-b,进一步确认Y-8菌株是副溶血性弧菌,且具有2种几丁质酶Chi1和Chi2。
2.2 Y-8菌株几丁质酶的SDS-PAGE及质谱分析
将Y-8菌株几丁质酶发酵液上清液进行SDS-PAGE分析,如图3所示,电泳结果与克隆的几丁质酶基因chi1和chi2所编码的蛋白质理论分子质量90.0 kDa和111.83 kDa一致。
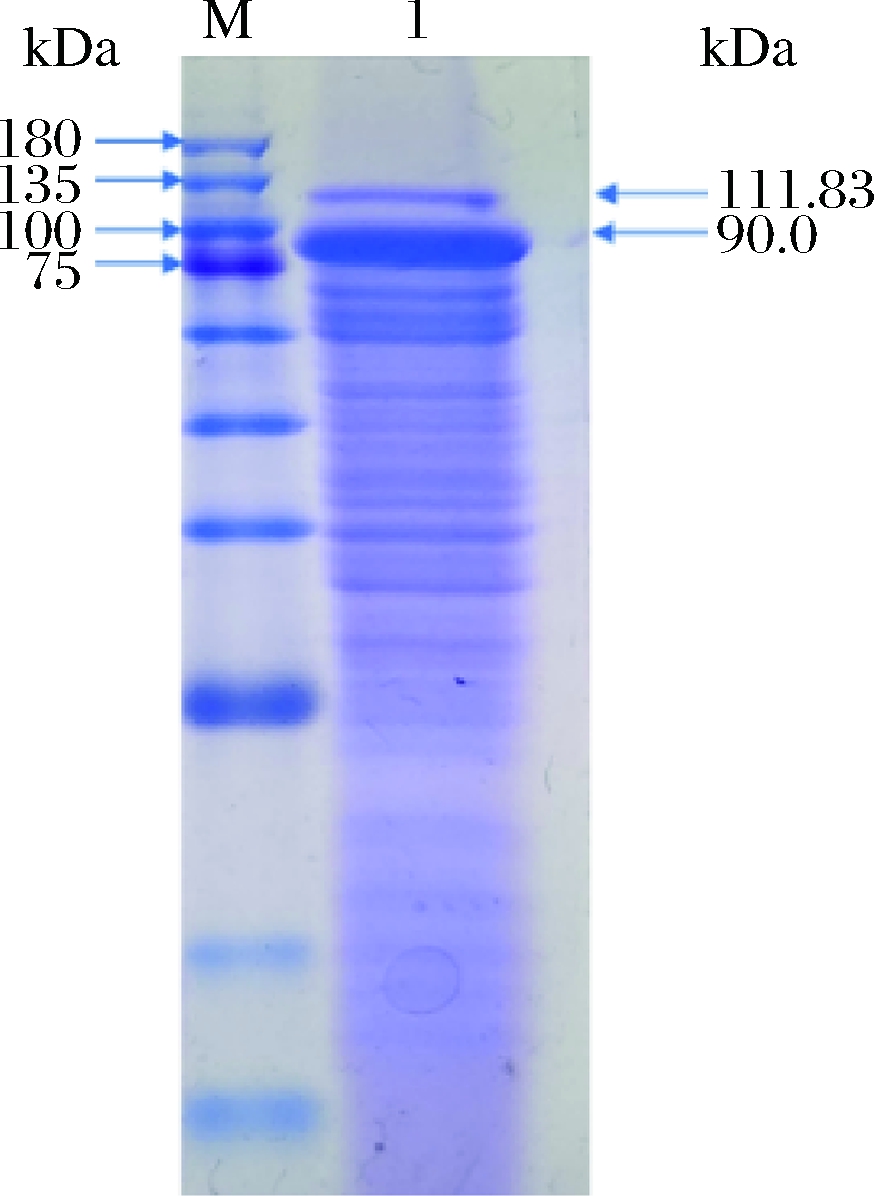
图3 发酵上清液的SDS-PAGE
Fig.3 SDS-PAGE of fermentation supernatant
如图4所示,质谱测定几丁质酶分子质量大小分别为87.6和112.9 kDa,与通过基因序列分析所得到的结果基本一致。其中Chi1在蛋白质谱分析显示具有NCBI数据库中几丁质酶特征氨基酸序列ANRDTFVASVK和AMLDELEAETGR,该肽段均位于几丁质酶Chi1的ChiA催化模块中;Chi2蛋白质谱分析显示具有几丁质酶特征氨基酸序列MGADHPR、VIGYFTSWR、DIPWEQLTHINYAFVSIGSDGK、GHFGALATYK、INIGVPYYTR和VGDQNYPINPK,前5个肽段均位于几丁质酶Chi2的ChiA催化模块,第6个肽段位于几丁质酶Chi2的ChiC催化模块。证明Y-8菌株确实能够产生2种几丁质酶Chi1以及Chi2,并且Chi1具有ChiA外切酶活性、Chi2具有ChiA外切酶和ChiC内切酶活性。

a-Chi1蛋白质谱;b-Chi2蛋白质谱
图4 蛋白质谱分析
Fig.4 Amino acid sequence analysis of protein spectrum
注:上标表示该段氨基酸序列被检测到的次数。
2.3 Y-8菌株发酵时间与产几丁质酶的关系及水解产物研究
绘制不同浓度N-乙酰氨基葡萄糖溶液OD540值标准曲线,线性拟合方程为y=3.905 4x-0.225 2,相关系数R2=0.99,线性关系很好。取不同发酵时间Y-8菌株发酵上清液,进行几丁质酶活力测定,结果如图5-a所示,从24 h至48 h,发酵液中几丁质酶迅速增加,当发酵时间为48 h时,几丁质酶活力达到最大值,此后酶活力呈现缓慢下降趋势。由此可见,Y-8菌株在此条件下培养48 h达到产酶高峰。
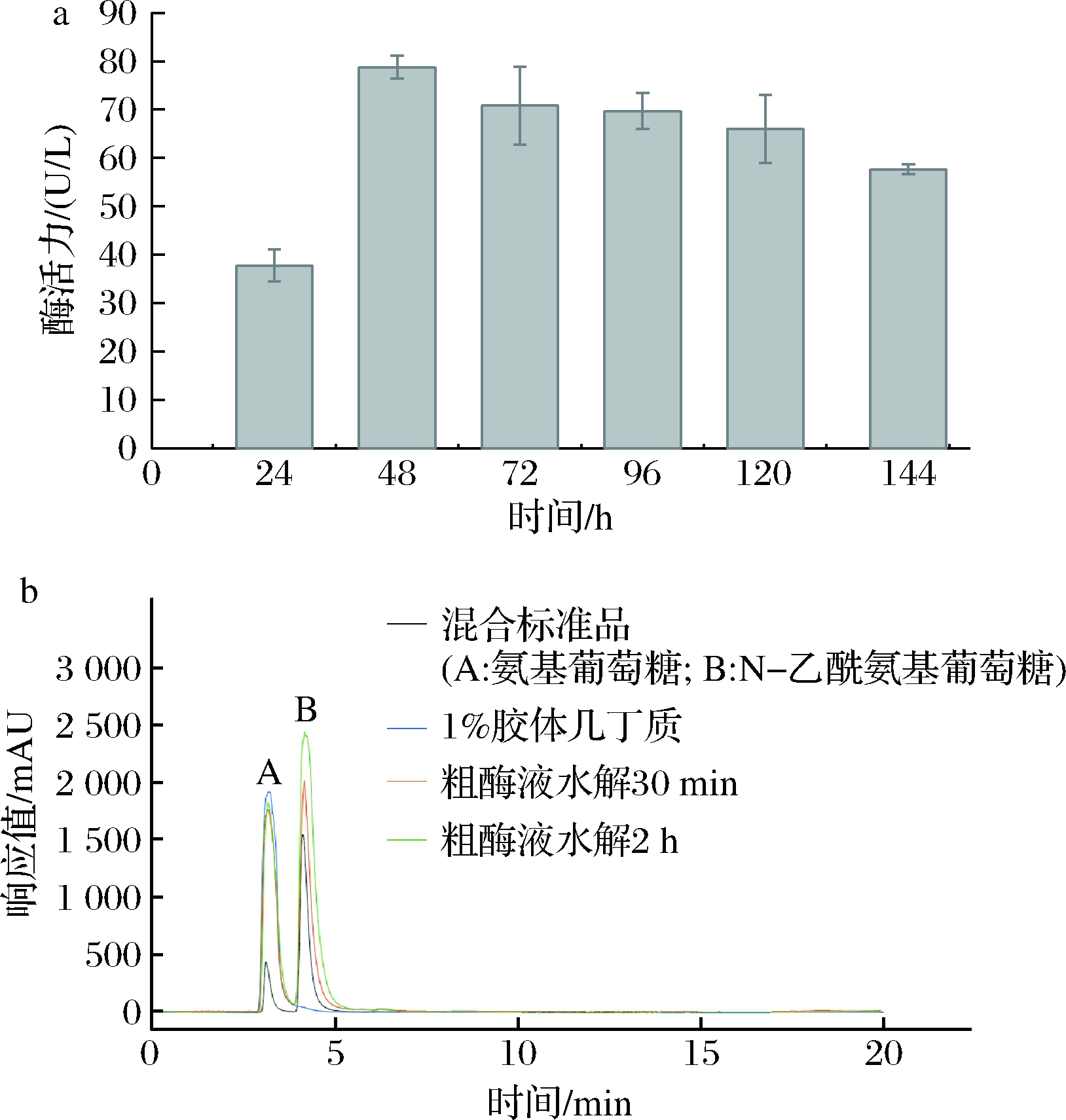
a-几丁质酶活力;b-水解产物HPLC分析
图5 不同发酵时间中几丁质酶活力和水解产物HPLC分析
Fig.5 Determination of chitinase activity at different fermentation time points and HPLC analysis of hydrolysate
注:水解产物HPLC分析中A峰为氨基葡萄糖吸收峰、B峰为N-乙酰氨基葡萄糖吸收峰
如图5-b所示,1%胶体几丁质中含有氨基葡萄糖但不含N-乙酰氨基葡萄糖,可能是因为制备胶体几丁质时几丁质被强酸水解所导致的;胶体几丁质经粗酶液水解30 min后,在4 min位置附近出现水解产物峰,经与氨基葡萄糖和N-乙酰氨基葡萄糖混合标准品比对,确认该水解产物为N-乙酰氨基葡萄糖,表明Y-8粗酶液能够水解胶体几丁质,生成单一产物N-乙酰氨基葡萄糖,并随着水解时间延长而增加,水解2 h后生成N-乙酰氨基葡萄糖1.02 mg/mL;但不同水解时间,反应体系中氨基葡萄糖含量没有变化,进一步证明酶解体系中的氨基葡萄糖来自于底物胶体几丁质,而非由几丁质酶水解产生。
2.4 Y-8菌株几丁质酶酶学性质的研究
2.4.1 Y-8菌株几丁质酶最适底物及其浓度的研究
Y-8几丁质酶水解不同底物效率差异较大,几丁质酶水解底物主要有壳聚糖、天然几丁质以及胶体几丁质,壳聚糖是将几丁质脱乙酰化的产物,本实验中所用的壳聚糖脱乙酰度在85%以上;天然几丁质是将几丁质只进行机械磨碎,不涉及结构的改变;胶体几丁质是通过强酸处理降低几丁质聚合度后的产物。
如图6-a所示,以胶体几丁质为底物时酶活性最高,以此为100%的酶活力;壳聚糖为底物时,相对酶活力达到11.28%;天然几丁质为底物时相对酶活力最低,仅为4.28%。推测其主要原因为:天然几丁质相邻长链间通过氨基与羰基形成氢键而聚合,导致几丁质酶底物结合域难以与N-乙酰氨基葡萄糖长链相结合,使得Y-8菌株产生的几丁质酶对天然几丁质的降解活性较差;通过盐酸处理后的胶体几丁质,在强酸条件下降低几丁质分子内链间聚合度,降低几丁质的致密性同时实现去乙酰化,这有助于底物与酶结合,实现更高的酶解作用。因此,Y-8能够更有效地降解胶体几丁质。
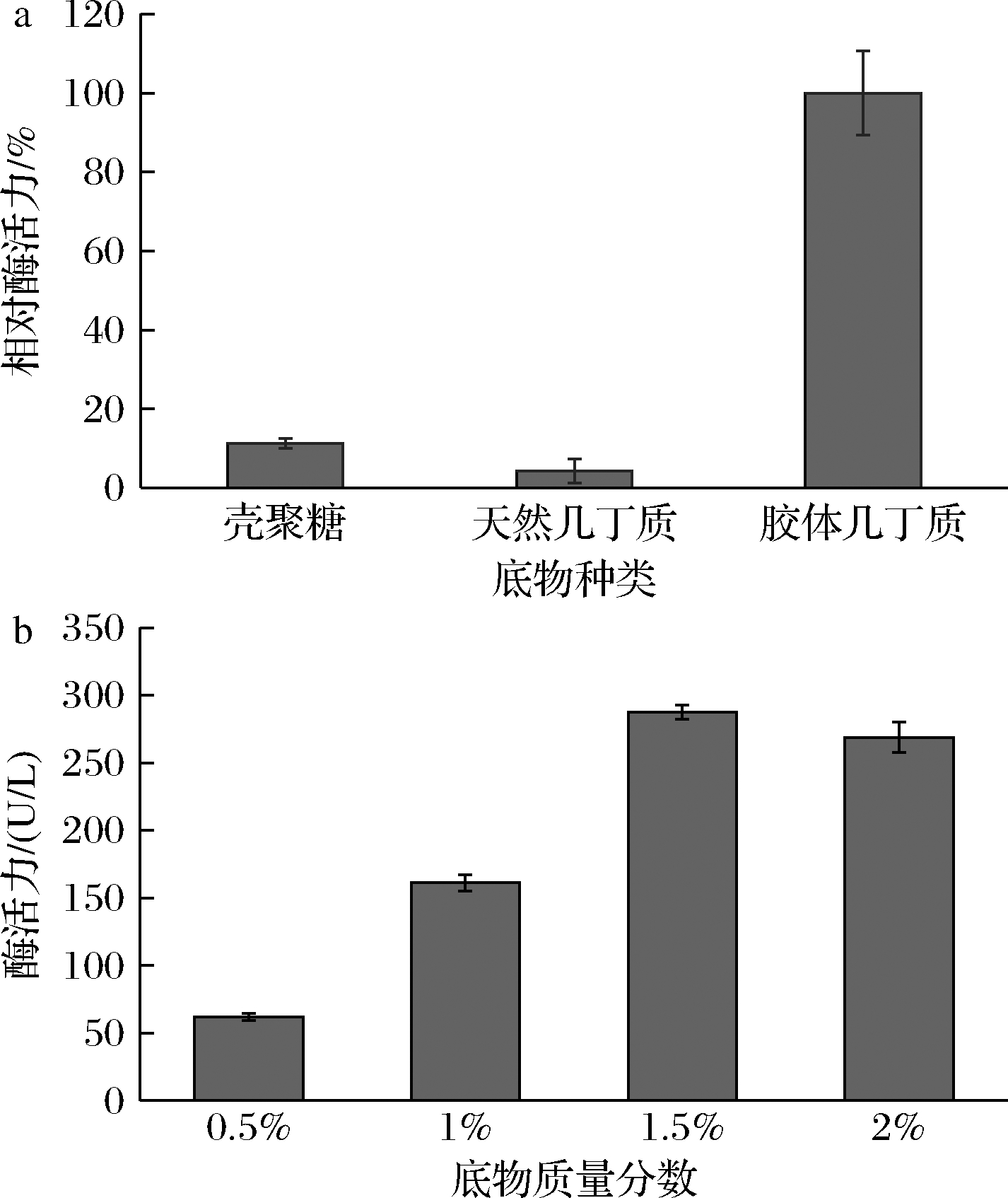
a-相对酶活力;b-底物浓度
图6 不同底物的相对酶活力及最适底物浓度
Fig.6 Relative enzyme activity on different substrates and optimal substrate concentration
如图6-b所示,随底物浓度增加,几丁质酶活力呈现上升趋势,当底物质量分数达到1.5%时几丁质酶活力最高,确定最适底物质量分数为1.5%的胶体几丁质。
2.4.2 Y-8菌株几丁质酶最适酶解温度及pH的研究
几丁质酶一般具备较广的温度适应性,如图7-a所示,随温度升高,粗酶液酶活力升高,在55 ℃时达到峰值,这表明该酶在55 ℃酶解能力最高,之后随温度升高酶解能力迅速降低,总体呈钟罩形曲线。
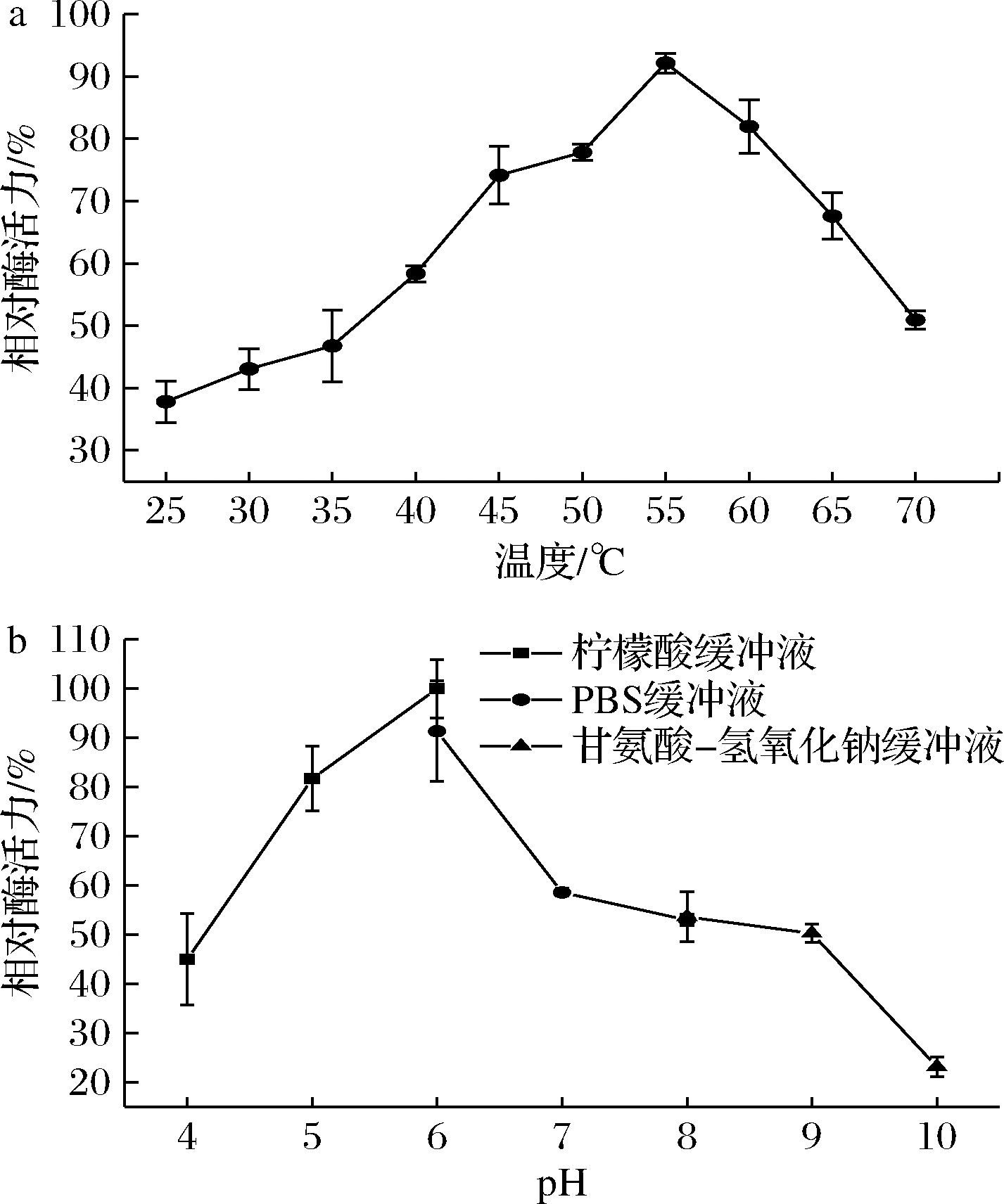
a-酶解温度;b-pH
图7 最适酶解温度及pH
Fig.7 Optimal enzymalytic temperature and pH
如图7-b所示,该几丁质酶最适pH为6.0,为酸性酶,当pH超过6.0以后,酶解效率大幅下降。实验结果符合细菌几丁质酶多为酸性酶这一特性。
2.4.3 Y-8菌株几丁质酶温度及pH稳定研究
如图8-a所示,几丁质酶在45 ℃及以下能保持70%以上的酶活力,50~60 ℃酶活力维持在35%~50%,70 ℃、1 h后仅具有30%酶活力,表明该酶高温具有一定的耐受性。如图8-b所示,pH 4.0~9.0的相对酶活性均在55%以上,在pH 10.0以上环境中酶活性急剧下降,表明该酶具有较宽泛的pH适应性。
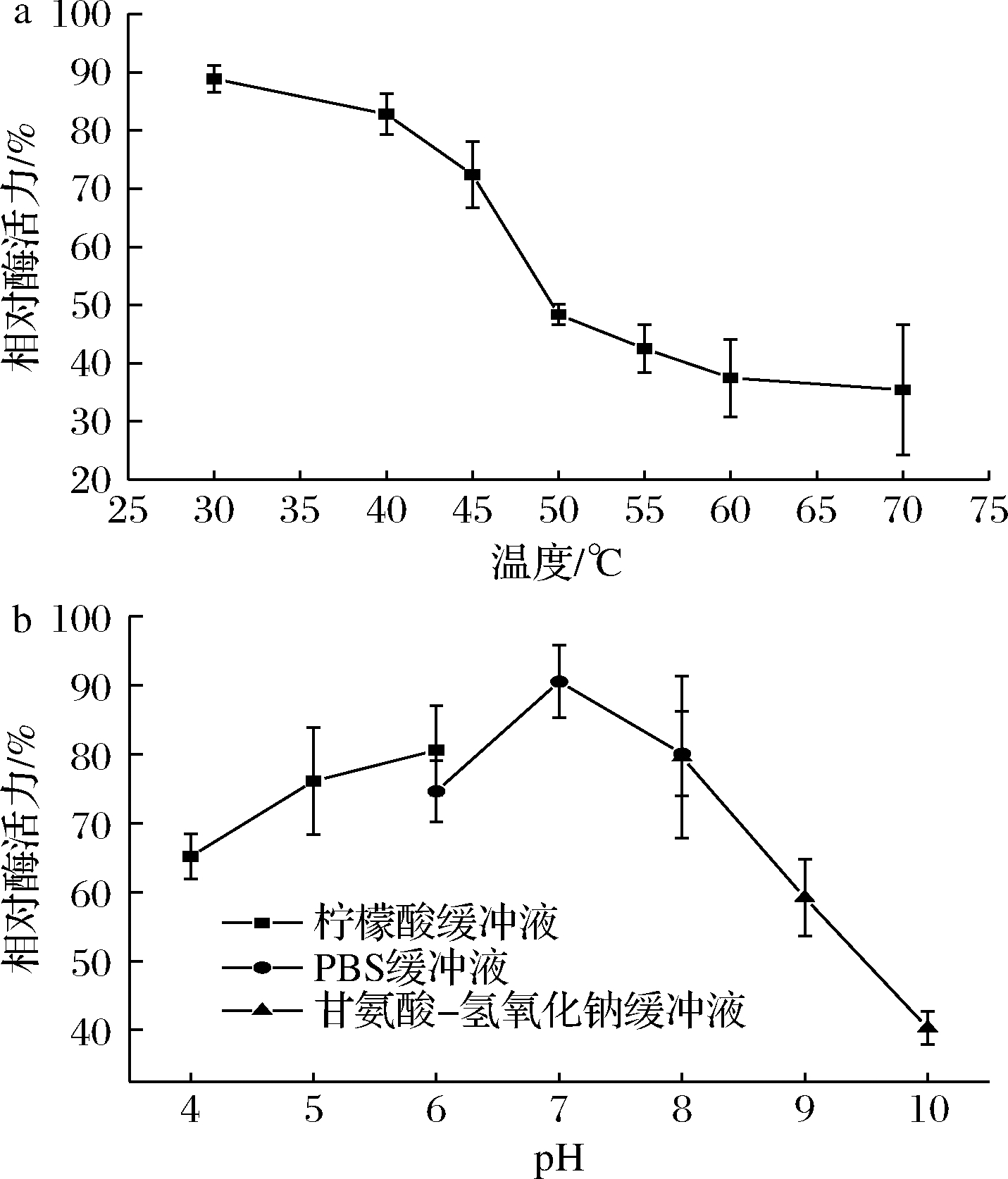
a-温度稳定性;b-pH稳定性
图8 温度及pH稳定性曲线
Fig.8 Temperature and pH stability curves
2.4.4 金属离子及化学试剂对于Y-8几丁质酶活力的影响
金属离子对于维持酶的三维结构甚至是催化过程有着十分重要的意义。因此本实验选取9种金属离子、EDTA以及3种常见表面活性剂测定其对于Y-8产生的几丁质酶活力的影响,结果如表2所示。
表2 金属离子对于酶活力的影响
Table 2 Effect of metal ions on enzyme activity
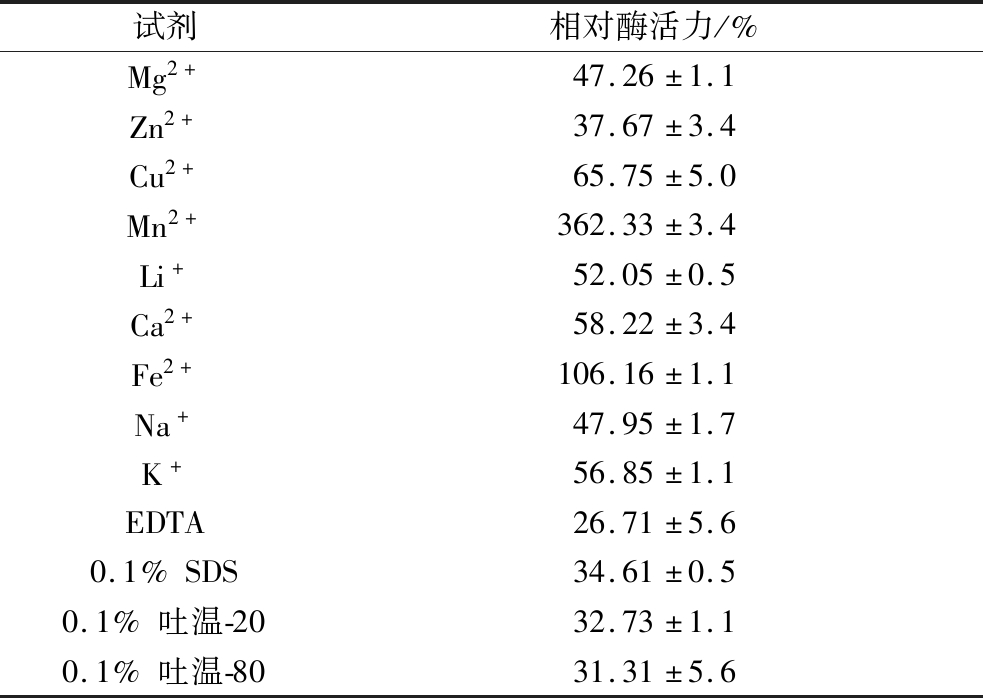
试剂相对酶活力/%Mg2+47.26±1.1Zn2+37.67±3.4Cu2+65.75±5.0Mn2+362.33±3.4Li+52.05±0.5Ca2+58.22±3.4Fe2+106.16±1.1Na+47.95±1.7K+56.85±1.1EDTA26.71±5.60.1%SDS34.61±0.50.1%吐温-2032.73±1.10.1%吐温-8031.31±5.6
Mn2+和Fe2+对于酶活力能够起到促进作用,Mn2+促进效果最为明显,能够达到对照的362%。其余金属离子均展现出一定程度的抑制作用;EDTA作为金属离子螯合剂,在10 mmol/L的浓度下出现大幅度抑制作用,证明该几丁质酶的酶活力中心依赖金属离子;3种表面活性剂(SDS、吐温-20、吐温-80)均对该酶有抑制作用,这可能与表面活性剂的添加影响酶的三维构象有关。
3 讨论
海洋中环境复杂,大多数营养物质往往以复杂的聚合物形式存在,为了使细胞更好地吸收营养,海洋中的弧菌往往产生多种胞外多糖水解酶,它们能够以几丁质为唯一能量来源并且迅速生长,这与它们能够产生不同类别几丁质酶并且存在协同降解作用相关[11]。弧菌属产生的几丁质酶均为GH18家族几丁质酶,以Chitinase A最为广泛,在pH 4~7时活性最高,pH 3~10保有活力,并且对高温高盐条件耐受性好,50~60 ℃保温6 h能剩余80%以上酶活力,并且降解几丁质能力优秀[17]。
本文从渤海滩涂中筛选出一株能够降解几丁质的副溶血性弧菌,并对其2个几丁质酶基因及氨基酸序列进行分析,其所含有的保守序列催化序列“DXXDXDXE”以及底物结合序列“SXGG”,普遍存在于细菌来源的几丁质酶中,并且能够形成(β/α)8-TIM桶状结构[18-19],这种桶状结构能够有效帮助酶与底物结合。Chi1中存在1个ChiA催化区域,属于GH18家族,具有外切酶活性,ChiA具有碳水化合物运输和代谢功能,蛋白空间构造深且窄,能够从还原端依次切割,主要产生单糖及双糖[17],如交替单胞菌株O-7[20]中,ChiA在几丁质降解过程中起主要作用并且对粉末状几丁质具备降解活性,水解产物主要为二糖。Chi2中存在ChiA和ChiC两个催化区域,ChiC具有内切酶活性能够从内部随机切割几丁质长链,产生双糖和寡聚糖[11],如粘质沙雷氏菌2170菌株[19]中,ChiC能够从胶体几丁质中水解产生(GlcNAc)6~(GlcNAc)3。
本文筛选到的副溶血性弧菌Y-8菌株几丁质酶最适反应温度为55 ℃,最适pH为6.0,郑家敏等[2]筛选到一株弧菌GR52,其几丁质酶基因在大肠杆菌表达后,发现其最适反应温度50 ℃、最适pH为5.0,与本文发现的几丁质酶的最佳作用温度和pH非常接近。同时,弧菌GR52几丁质酶在pH 5.0~10.0的条件下能够保持较好的稳定性,而本论文Y-8菌株几丁质酶在pH 4.0~9.0相对酶活力在50%以上,证明来源于弧菌属几丁质酶更适应于酸性环境[21]。此外,Mn2+、Fe2+对于Y-8的几丁质酶酶促反应有促进作用,相对酶活力分别达362%、106%,这表明Y-8几丁质酶的酶活力中心需Mn2+、Fe2+作为激活剂,同时受金属离子螯合剂抑制。与前人研究结果Zn2+、Na+、Cu2+、Mg2+等金属阳离子可以提高几丁质酶活性,而Mn2+会对几丁质酶具有显著的抑制作用或者无影响[14,22-23]有所不同。
4 结论
本文从渤海滩涂获得一株高效水解胶体几丁质的菌株,经细菌生理生化实验以及分子生物学鉴定为副溶血性弧菌(Vibrio parahaemolyticus),并命名为Y-8。通过SDS-PAGE以及蛋白质谱分析,证明Y-8菌株产生2种几丁质酶Chi1和Chi2,并具有弧菌几丁质酶典型保守氨基酸序列催化序列“DXXDXDXE”以及底物结合序列“SXGG”。该几丁质酶以胶体几丁质为底物时,最终水解产物经HPLC分析为单一N-乙酰氨基葡萄糖。由此推断,Y-8菌株几丁质酶复合酶系协同降解胶体几丁质,并将其彻底水解为单糖,但具体协同降解体系比例以及相关酶学性质仍需进一步探究。酶学性质分析表明,Y-8菌株所产几丁质酶最适温度为55 ℃,温度耐受性好,45 ℃保温1 h后能剩余70%以上酶活性;最适pH为6.0,pH 4.0~9.0、37 ℃保温1 h稳定性良好,剩余酶活力55%以上;此外,Mn2+具有显著的激活作用,使酶活力提高至362%,但受到EDTA以及表面活性剂的抑制。本文筛选Y-8菌株能够产生2种几丁质酶,降解胶体几丁质生成单一产物N-乙酰氨基葡萄糖,可用于降解几丁质生产N-乙酰氨基葡萄糖,为以海洋甲壳类外骨骼来源的几丁质,工业化生产N-乙酰氨基葡萄糖提供几丁质酶的来源。
[1] 高英豪.几丁质酶基因Cht6和Cht10在果蝇翅表皮发育中的功能研究[D].太原:山西大学,2021.
GAO Y H.Functional analysis of Cht6 and Cht10 in win cuticle development in Drosophila[D].Taiyuan:Shanxi University,2021.
[2] 郑家敏,梁燕辉,朱凡,等.几丁质酶基因的克隆表达及酶学性质[J].微生物学通报,2018,45(5):1027-1034.
ZHENG J M,LIANG Y H,ZHU F,et al.Cloning,expression and characterization of the chitinase gene from Vibrio sp.GR52[J].Microbiology China,2018,45(5):1027-1034.
[3] 肖宇,于宏伟,马爱进,等.球磨、超声和盐酸处理对几丁质的微观结构和酶促脱乙酰效率的影响[J/OL].食品科学,2022.http://kns.cnki.net/kcms/detail/11.2206.TS.20220803.1658.002.html.
XIAO Y,YU H W,MA A J,et al.Effects of ball milling,ultrosonic and hydrochloric acid treatment on the microstructure and enzymatic deacetylation efficacy of chitin[J/OL].Food Science,2022.http://kns.cnki.net/kcms/detail/11.2206.TS.20220803.1658.002.html.
[4] 杨绍青,刘学强,刘瑜,等.酶法制备几种功能性低聚糖的研究进展[J].生物产业技术,2019(4):16-25.
YANG S Q,LIU X Q,LIU Y,et al.Advance in enzymatic production of several functional oligosaccharides[J].Biotechnology &Business,2019(4):16-25.
[5] 张新月,张月琪,王凤彪,等.海洋细菌来源几丁质酶的研究进展[J].食品工业科技,2021,42(22):383-389.
ZHANG X Y,ZHANG Y Q,WANG F B,et al.Research progress of microbial chitinase from marine bacteria[J].Science and Technology of Food Industry,2021,42(22):383-389.
[6] ARAKANE Y,MUTHUKRISHNAN S.Insect chitinase and chitinase-like proteins[J].Cellular and Molecular Life Sciences,2010,67(2):201-216.
[7] TRAN T N,DOAN C T,NGUYEN M T,et al.An exochitinase with N-acetyl-β-glucosaminidase-like activity from shrimp head conversion by Streptomyces speibonae and its application in hydrolyzing β-chitin powder to produce N-acetyl-d-glucosamine[J].Polymers,2019,11(10):1600.
[8] 黎欣宇,邓旭,蒋思婧,等.C端结构域截短提高苏云金芽孢杆菌来源几丁质酶的活力[J].微生物学报,2022,62(4):1379-1389.
LI X Y,DENG X,JIANG S J,et al.C-terminal domain truncation improves the activity of chitinase derived from Bacillus thuringiensis[J].Acta Microbiologica Sinica,2022,62(4):1379-1389.
[9] 赵沙,颜子娟,张舒,等.细菌几丁质酶结构、功能及分子设计的研究进展[J].生物化学与生物物理进展,2021(7):1179-1191.
ZHAO S,YAN Z J,ZHANG S,et al.Research progress on structure,function and molecular design of bacterial chitinase[J].Progress in Biochemistry and Biophysics,2021(7):1179-1191.
[10] RATHORE A S,GUPTA R D.Chitinases from bacteria to human:Properties,applications,and future perspectives[J].Enzyme Research,2015,2015:1-8.
[11] 刘力睿,潘杰,李猛.微生物几丁质酶的研究进展,应用及展望[J].生物资源,2020,42(5):494-504.
LIU L R,PAN J,LI M.Recent research progress,application and future perspectives on microbial chitinases[J].Biotic Resources,2020,42(5):494-504.
[12] 王悦.高产几丁质酶菌株的分离鉴定与酶学性质研究[D].天津:天津科技大学,2017.
WANG Y.Isolation and identification of high chitinase-producing bacterium and the properties of its chintinase[D].Tianjin:Tianjin University of Science and Technology,2017.
[13] 郝之奎.Chitinolyticbacter meiyuanensis的筛选鉴定及其发酵产几丁质酶研究[D].无锡:江南大学,2011.
HAO Z K.Chitinolyticbacter meiyuanensis isolation,identification,and fermentation for chitinase production[D].Wuxi:Jiangnan University,2011.
[14] 郑家敏.几丁质酶高产菌株选育及酶学性质研究[D].福州:福州大学,2018.
ZHENG J M.Breeding and enzymatic properties of high yield chitinase-producing strain[D].Fuzhou:Fuzhou University,2018.
[15] KONAGAYA Y,TSUCHIYA C,SUGITA H.Purification and characterization of chitinases from Clostridium sp.E-16 isolated from the intestinal tract of the South American Sea lion (Otaria flavescens)[J].Letters in Applied Microbiology,2006,43(2):187-193.
[16] 连文浩.产几丁质酶微生物的筛选及基因的克隆表达[D].福州:福州大学,2015.
LIAN W H.Gene cloning and expression of chitinase from microorganisms isolated from various environments[D].Fuzhou:Fuzhou University,2015.
[17] 周玉玲,蒋思婧,贺妮莎,等.微生物几丁质酶研究进展及其在N-乙酰氨基葡萄糖制备中的应用[J].微生物学报,2021,61(8):2192-2204.
ZHOU Y L,JIANG S J,HE N S,et al.Research progress of microbial chitinase and its application in the preparation of N-acetylglucosamine[J].Acta Microbiology Sinica,2021,61(8):2192-2204.
[18] 龚福明,何彩梅,唐小艳,等.微生物几丁质酶的研究进展及应用现状[J].中国酿造,2017,36(8):20-25.
GONG F M,HE C M,TANG X Y,et al.Research progress and application status of microbial chitinase[J].China Brewing,2017,36(8):20-25.
[19] SUZUKI K,SUGAWARA N,SUZUKI M,et al.Chitinases A,B,and C1 of Serratia marcescens 2170 produced by recombinant Escherichia coli:Enzymatic properties and synergism on chitin degradation[J].Bioscience,Biotechnology,and Biochemistry,2002,66(5):1075-1083.
[20] ORIKOSHI H,NAKAYAMA S,MIYAMOTO K,et al.Roles of four chitinases (ChiA,ChiB,ChiC,and ChiD) in the chitin degradation system of marine bacterium Alteromonas sp.strain O-7[J].Applied and Environmental Microbiology,2005,71(4):1811-1815.
[21] 吴昊,刘嘉荔,杨静文,等.双酶协同降解胶体几丁质及其作用机制[J].食品科学,2022,43(6):74-80.
WU H,LIU J L,YANG J W,et al.Synergistic enzymatic degradation of colloidal chitin and its mechanism[J].Food Science,2022,43(6):74-80.
[22] 丁志雯,刘耀东,黄志发,等.产几丁质酶海洋细菌Dyadobacter sp.CZW019的筛选、鉴定及酶学性质研究[J].江苏海洋大学学报(自然科学版),2021,30(2):22-29.
DING Z W,LIU Y D,HUANG Z F,et al.Screening,identification and enzymatic properties of a chitinase-producing marine bacterium Dyadobacter sp.CZW019[J].Journal of Jiangsu Ocean University (Natural Science Edition),2021,30(2):22-29.
[23] 高聪,张阿磊,陈可泉,等.Chitinibacter sp.GC72的筛选鉴定及其几丁质降解产物研究[J].食品与生物技术学报,2015,34(1):15-20.
GAO C,ZHANG A L,CHEN K Q,et al.Isolation and characterization of a chitin-degrading strain Chitinibacter sp.GC72 and identification of its chitinase degradation[J].Journal of Food Science and Biotechnology,2015,34(1):15-20.