白酒是中国特有的蒸馏酒,由多种微生物通过固态混合发酵制备而成,根据生产工艺的差异逐步演变形成了公认的12种香型。其中,浓香型白酒是以粮谷为原料,采用浓香大曲为糖化发酵剂,经泥窖固态发酵,固态蒸馏、陈酿、勾调而成的,不直接或间接添加食用酒精及非自身发酵产生的呈色呈香呈味物质的白酒[1]。浓香型白酒采用独特的泥窖发酵,造就了浓香型白酒特有的“窖香”味,是形成“窖香浓郁、绵柔甘冽、香味协调”的酒体风格的关键,也是浓香型白酒与其他香型白酒的最大区别。窖池中微生物菌种的种类、丰度、群落结构以及代谢活性直接影响着白酒酿造环境的微生物平衡,促进白酒典型香气的形成,进而影响浓香型白酒的品质[2-3],因此,微生物在浓香型白酒酿造过程中处于核心地位,发挥着关键作用。尽管对浓香型白酒酿造微生物的关注越来越高,但是微生物在白酒酿造过程中的作用机理尚未完全解析,功能微生物、代谢特性与白酒风味物质形成的内在联系也未完全阐释明晰[4]。
传统的平板培养法是分析浓香型白酒酿造过程微生物群落结构的直接方法,但由于分离生境的复杂性,微生物生长的自然环境条件无法满足,微生物间共同协作的方式研究不透彻[5],培养基选择不合理等因素[6-7],导致用传统的培养技术对不同生境微生物的可培养性都很低[8-9],严重阻碍了对微生物群落多样性的深入解析。随着研究技术的进步,针对白酒酿造过程中复杂微生物群落结构的解析主要集中在分子生态学方法,单链构象多态性分析(single strand conformation polymorphism,PCR-SSCP)技术[10]、变性梯度凝胶电泳(denaturing gel gradient electrophoresis,PCR-DGGE)技术[11]和rRNA基因克隆文库技术[12]等。然而,由于PCR-DGGE和PCR-SSCP在很大程度上依赖于所设计的PCR引物的特异性,rRNA基因的克隆文库分析依赖于PCR扩增产物的连接转化效率,因此上述技术不能准确全面地反映微生物群落组成[13]。高通量测序技术具有通量高、分辨率高、成本低等优势[14],可以绕开“大部分环境微生物不可培养”的壁垒,全面客观地解析样品中微生物群落的多样性和功能组成,同时获得定性及相对定量信息[15],是分析浓香型白酒窖池等复杂样品中微生物菌群结构的首选方法,目前已经广泛应用于浓香型[16-18]、酱香型[19-20]和清香型[21-22]白酒酿造微生物的群落结构研究中[16-18]。因此,本研究拟利用高通量测序技术,全面解析浓香型白酒窖池中微生物的群落结构,在分子生物学水平上探究特征性微生物资源,为浓香型白酒窖池中特征菌种的定向筛选和微生物可培养组的研究奠定基础。
1 材料与方法
1.1 样品采集
样品采集于四川宜宾市某浓香型白酒生产企业一车间、二车间和五车间的窖池,每个车间各采集1份窖泥(N)和酒醅(J)样品,合计6份样品。详细记录样品的采集信息,包括样品名称、采样地区、采样地点、样品类型和窖池窖龄等,具体见表1。
表1 浓香型白酒窖池样品采样信息
Table 1 Sampling information of cellars samples in strong-flavor Baijiu
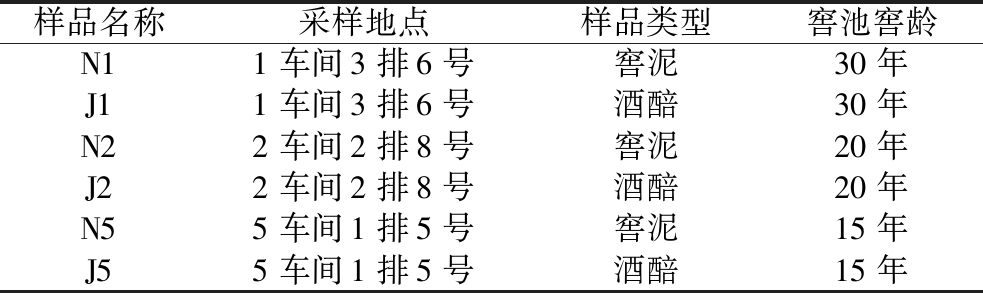
样品名称采样地点样品类型窖池窖龄N11车间3排6号窖泥30年J11车间3排6号酒醅30年N22车间2排8号窖泥20年J22车间2排8号酒醅20年N55车间1排5号窖泥15年J55车间1排5号酒醅15年
具体取样方法为:窖泥上部取样点距地面约0.3 m,中部取样点距地面约1 m,上部和中部分别从窖池四壁取样;窖池下部取样点距地面约2 m,下部分别从窖池四壁和底部取样;将上述窖泥样品混匀作为一个窖泥样品。采用五点法,从四周和中间取酒醅,混匀作为1个酒醅样品。窖泥和酒醅样品均由当天出池的窖池中采样获得,采集后的样品密封,-20 ℃保存待用。
1.2 宏基因组DNA的提取与纯化
采用CTAB法提取样品的宏基因组DNA,利用CTAB、蛋白酶K和SDS破碎细胞,酚氯仿异戊醇抽提提取窖泥和酒醅样品的宏基因组DNA,具体提取方法参考文献[23]。使用Cycle-pure Kit(D6492,Omega)对提取得到的粗DNA进行纯化,以满足后续建库的要求。
1.3 PCR扩增及测序
以质量合格的宏基因组DNA为模板,使用16S rRNA基因V3-V4区通用引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAA T-3′)进行PCR扩增[24]。使用Agencourt AMPure XP磁珠对PCR扩增产物纯化,进行文库构建,使用Agilent 2100 Bioanalyzer对文库的片段范围及浓度进行检测,选择HiSeq高通量测序平台对质控合格的文库进行测序。
1.4 数据分析
数据分析主要包括测序数据过滤、Tag拼接、OTU聚类、物种注释、物种复杂度和组间物种差异分析等流程。利用iTools Fqtools fqcheck(v.0.25)、cutadapt、readfq等软件对下机数据质控,去除接头、低质量序列和嵌合体得到Clean Data。使用软件FLASH(Fast Length Adjustment of Short reads,v1.2.11)[25],利用重叠关系将双末端测序得到的成对Reads组装成一条序列,得到高变区的Tags。利用软件USEARCH(v7.0.1090)[26]将拼接好的Tags聚类为操作分类单元(operational taxonomic unit,OTU)。通过RDP classifer(v2.2)[27]软件将OTU代表序列与RDP(Release11.5)数据库比对进行物种注释,并在界、门、纲、目、科、属和种水平上统计分析各样品中的细菌物种组成。使用mothur(v.1.31.2)[28]进行样品Alpha多样性指数分析,包括chao、ace、shannon,simpson以及coverage等指数,共同表征样品的细菌群落结构的丰富度和多样性。利用phytools软件,根据unweighted unifrac距离矩阵进行UPGMA聚类分析,并将聚类结果与各属水平上的物种相对丰度整合展示,来确定样品中细菌的系统发育关系和群落结构的差异。使用R软件(v3.4.1)构建花瓣图,展示所有样品共有OTU的数目,结合RDP数据库注释信息,确定共有OTU的分类学地位;选取丰度排名前10的细菌物种,利用R(v3.4.1)软件分析细菌物种在每个分组中的平均相对丰度以及差异显著性,探究不同分析样品的关键微生物。
2 结果与分析
2.1 窖池样品中细菌群落结构解析
利用HiSeq 2500测序平台对浓香型白酒窖池样品进行高通量测序,解析窖泥和酒醅样品中的细菌群落结构。测序结果表明,6个样品共获得337 982条Tags,每个样品的Tags数目在45 125~63 218,酒醅样品的Tags数高于窖泥样品的Tags数。6个样品共获得1 527 OTUs,物种累积曲线趋于平稳,每个样品的OTUs数目在91~381,窖泥样品的OTUs数高于酒醅样品的OTUs数。
2.1.1 窖池样品细菌多样性研究
Alpha多样性是指特定生态系统内的物种多样性,是反映物种丰富度和均匀度的综合指标,主要包括Chao、Ace、Shannon、Simpson和Coverage指数。其中,Chao和Ace指数用于估计样品中的物种总数,指数值越大,表示样品中群落丰富度越高。Shannon和Simpson指数主要反映群落的多样性,Shannon指数越大,说明样品中细菌物种多样性越高;Simpson指数越大,说明样品中细菌物种多样性越低。深度测序指数Coverage是反映测序深度的指标,其值越接近于1,说明测序深度越合理,即测序深度已经覆盖到样品中所有的物种。
使用mothur软件对浓香型白酒窖池中的细菌群落结构的Alpha多样性进行分析。结果表明,6个样品的Chao指数在91.26~408,Ace指数在94.81~417.68,物种总数最高的样品为N2,最低为J2。样品的Shannon指数在2.79~3.82;Simpson指数在0.06~0.15,物种多样性最高的样品为J1,最低的为J5。此外,6个样品的Coverage均在0.999之上,表明测序深度已经基本覆盖到样本中所有的物种,多样性指数的具体结果见表2。
表2 浓香型白酒窖池样品多样性指数
Table 2 Bacterial diversity index of cellars samples in strong-flavor Baijiu
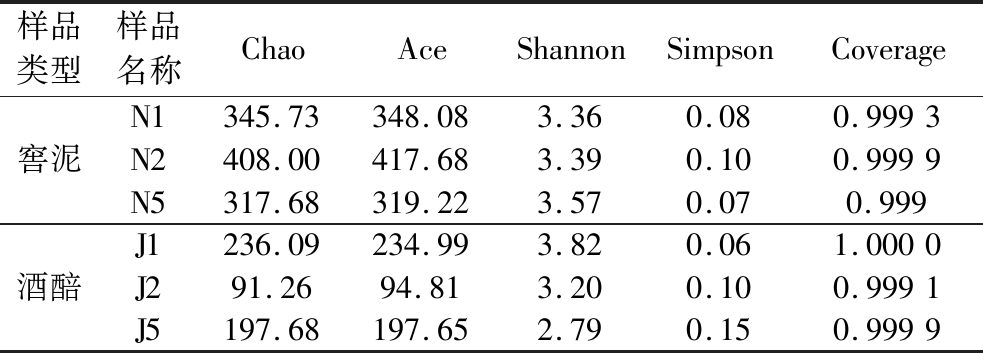
样品类型样品名称ChaoAceShannonSimpsonCoverage窖泥N1345.73348.083.360.080.9993N2408.00417.683.390.100.9999N5317.68319.223.570.070.999酒醅J1236.09234.993.820.061.0000J291.2694.813.200.100.9991J5197.68197.652.790.150.9999
从样品类型上看,窖泥样品Chao指数平均值为357.14,高于酒醅样品(平均值为175.01),酒醅样品Chao指数的变化程度高于窖泥样品(图1-a);窖泥样品的Shannon指数平均值为3.44,高于酒醅样品(平均值为3.27),但酒醅样品Shannon指数的变化程度高于窖泥样品(图1-b),这表明窖泥中的细菌物种总数和多样性均高于酒醅,推测这可能与窖泥常年反复使用,从而实现细菌的富集有关。

a-Chao指数;b-Shannon指数
图1 浓香型白酒窖泥和酒醅样品细菌多样性指数比较
Fig.1 Comparison of bacterial diversity index between pit muds and fermented grains samples in strong-flavor Baijiu
2.1.2 窖池样品细菌聚类分析
利用phytools软件,根据unweighted unifrac距离矩阵对6个样品进行UPGMA聚类分析,并将聚类结果与各属水平上的物种相对丰度整合展示,来确定样品的群落结构的差异。聚类结果表明(图2),窖泥样品和酒醅样品分别聚类在不同的分支,表明不同车间的窖泥样品之间、酒醅样品之间微生物群落结构组成较为相似;窖泥样品和酒醅样品之间的微生物群落结构组成差异较大。窖泥样品内,样品N1和N5构成了一个独立的分支,表明样品N1和N5的细菌群落结构更为相似,与样品N2的差异较大;酒醅样品也出现类似的聚类结果,样品J1和J5的细菌群落结构更为相似,与样品J2的差异较大,推测此次采样的一车间样品和五车间样品较为相似,与二车间样品差别较大。

图2 浓香型白酒窖泥和酒醅样品UPGMA聚类分析
Fig.2 UPGMA cluster analysis of pit muds and fermented grains samples in strong-flavor Baijiu
2.1.3 窖池样品细菌组成和丰度分析
窖池样品细菌群落结构分析结果表明(图3),窖泥样品中的细菌主要来自于厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),其中厚壁菌门细菌的丰度超过了60%;而酒醅样品中的细菌主要来自于厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria),其中厚壁菌门细菌的丰度最高,占比在37.7%~68.4%。16S rRNA基因V3~V4区进行扩增子测序的目的产物片段长度约400 bp,在属水平上对细菌物种注释较为准确,因此本研究主要在属水平上分析细菌物种组成和相对丰度。在相对丰度超过1%的种属中,梭菌属(包括Clostridium IV和Clostridium sensu stricto)、岩石单胞菌属(Petrimonas)、沉积物棒菌属(Sedimentibacter)、嗜蛋白菌属(Proteiniphilum)、互养单胞菌属(Syntrophomonas)和氨基酸杆状菌属(Aminobacterium)是3个窖泥样品共有的细菌菌属,而土壤芽胞杆菌(Solibacillus)是一车间窖泥的主要菌群,占比达到14.16%;乳杆菌属(Lactobacillus)是二车间窖泥的主要菌群,占比达到28.23%。乳杆菌属(Lactobacillus)、丙酸杆菌属(Propionibacterium)、梭菌属(Clostridium sensu stricto)和甲基杆菌属(Methylobacterium)是3个酒醅样品共有的细菌菌属,而土壤芽胞杆菌(Solibacillus)是一车间酒醅的主要菌群,占比达到14.56%;埃希氏菌属(Escherichia)是二车间酒醅的主要菌群,占比达到24.78%;拉梅尔芽胞杆菌(Rummeliibacillus)是五车间酒醅的主要菌群,占比达到10.48%,详细结果见图3。

图3 浓香型白酒窖池样品细菌种类和丰度
Fig.3 Bacterial species and abundance of cellar pool sample in strong-flavor Baijiu
2.1.4 窖池样品中关键细菌分析
使用R软件(v3.1.1)构建花瓣图分析了窖池样品中的共有微生物,通过RDP classifer软件将OTU代表序列与RDP数据库比对进行物种注释,确定了6个窖池样品共有的17个OTUs的准确的分类学地位,其中有7个OTUs鉴定到种水平,分别为运动氨基酸杆状菌(Aminobacterium mobile)、永达尔氏梭菌(Clostridium ljungdahlii)、产乙酸嗜蛋白质菌(Proteiniphilum acetatigenes)、嗜硫岩石单胞菌(Petrimonas sulfuriphila)、海洋原绿球菌(Prochlorococcus marinus)、森林土壤芽胞杆菌(Solibacillus silvestris)和萨勒河沉积物棒菌(Sedimentibacter saalensis)。6个OTUs鉴定到属水平,包括梭菌属(含Clostridium IV和Clostridium sensu stricto)、乳杆菌属(Lactobacillus sp.)、共养单胞菌属(Syntrophomonas sp.)和Candidatus Cloacamonas属。4个OTUs鉴定到属以上的水平,分别是瘤胃球菌科(Ruminococcaceae)、梭菌目(Clostridiales)和厚壁菌门(Firmicutes),详细结果见表3。
表3 浓香型白酒窖池样品共有OTU列表
Table 3 Common OTUs list of cellar pool samples in strong-flavor baijiu
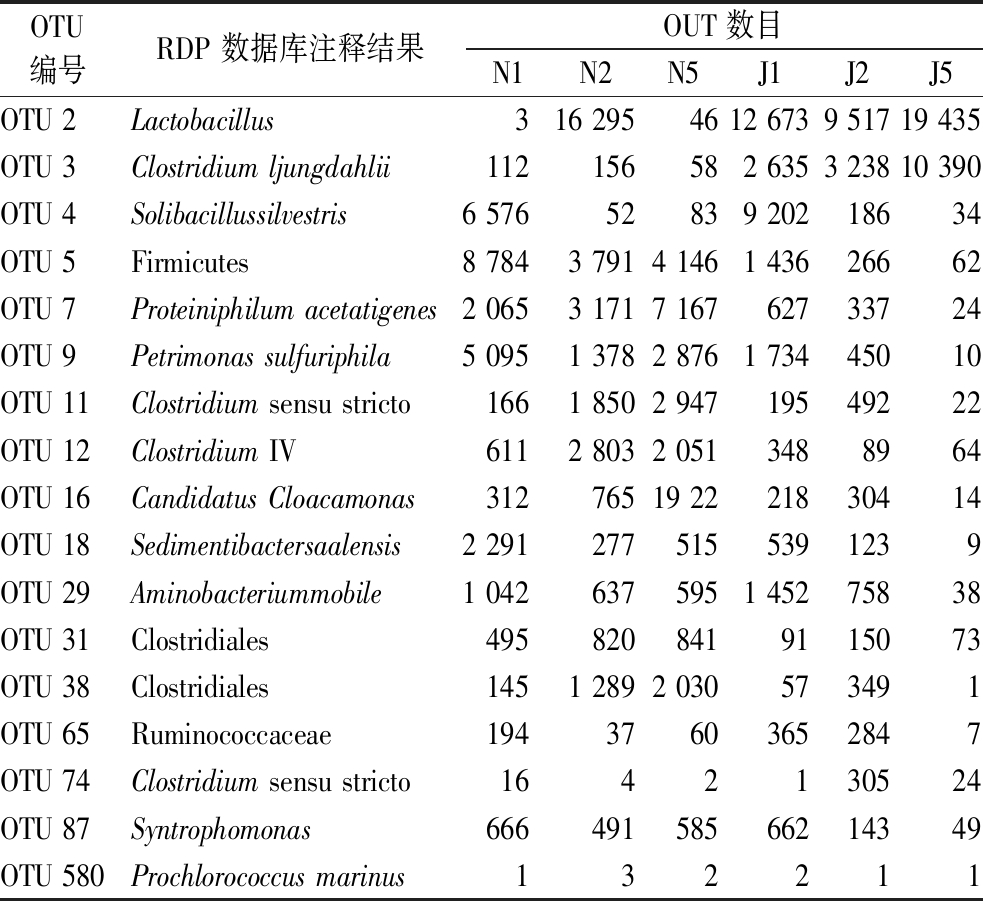
OTU编号RDP数据库注释结果OUT数目N1N2N5J1J2J5OTU2Lactobacillus 316295 4612673951719435OTU3Clostridiumljungdahlii112156582635323810390OTU4Solibacillussilvestris65765283920218634OTU5Firmicutes878437914146143626662OTU7Proteiniphilumacetatigenes20653171716762733724OTU9Petrimonassulfuriphila509513782876173445010OTU11Clostridiumsensustricto1661850294719549222OTU12ClostridiumIV611280320513488964OTU16CandidatusCloacamonas312765192221830414OTU18Sedimentibactersaalensis22912775155391239OTU29Aminobacteriummobile1042637595145275838OTU31Clostridiales4958208419115073OTU38Clostridiales14512892030573491OTU65Ruminococcaceae19437603652847OTU74Clostridiumsensustricto1642130524OTU87Syntrophomonas66649158566214349OTU580Prochlorococcusmarinus132211
9个OTUs来自梭菌纲(Clostridia),占比最多,主要包括梭菌科(Clostridiaceae)、瘤胃菌科(Ruminococcaceae)和共养单胞菌科(Syntrophomonadaceae),属于产氢产酸细菌,常与产甲烷菌共生[29]。梭菌属菌种是影响浓香型白酒风味物质形成的主要微生物,在合成己酸、己酸乙酯、丁酸等典型风味物质方面发挥着重要的作用。本研究中发现的共有微生物中的梭菌主要来自于严格意义厌氧的梭菌属(Clostridium sensu stricto),包括永达尔氏梭菌(C.ljungdahlii)、酒梭菌(Clostridium liquoris)和酪丁酸梭菌(Clostridium tyrobutyricum)等,均能在窖池的厌氧环境下生成丁酸、酪丁酸、乙醇、乙酸等风味物质[30]。瘤胃菌科细菌能够发酵纤维素类物质生成乙醇、乙酸、乳酸、H2和CO2等前体物质。岩石单胞菌属(Petrimonas)和互养单胞菌属(Syntrophomonas)菌株主要与产甲烷菌互营共生,为甲烷菌生长提供代谢底物[31]。运动氨基酸杆状菌(A. mobile)属于降解氨基酸的厌氧细菌,能够利用发酵丝氨酸、苏氨酸、甘氨酸和丙酮酸,与甲烷杆菌共培养时,能够利用丙氨酸、谷氨酸、缬氨酸、亮氨酸、异亮氨酸和天门冬氨酸[32]。萨勒河沉积物棒菌(S. saalensis)能够利用甘氨酸、精氨酸和赖氨酸产生乙酸和丁酸。产乙酸嗜蛋白质菌(P.acetatigenes)能够将丙酮酸转化成乙酸和CO2,不产生H2S[33]。森林土壤芽胞杆菌(S.silvestris)主要产生酪蛋白酶等水解酶,降解窖池中的原料产生小分子物质供细菌生长。乳杆菌属(Lactobacillus sp.)能够产生乳酸,耐受高浓度乙酸和乙醇[34],乳酸和乳酸乙酯是窖泥中重要的风味物质,但乳酸含量过多,会导致窖泥形成乳酸亚铁、乳酸钙等结晶,引起窖泥老化,二车间窖泥样品中乳酸菌的丰度明显高于一车间和五车间,乳酸菌成为优势菌群将导致窖泥微生物群落结构不稳定及窖泥质量退化,因此推测二车间窖泥的质量不如一车间和五车间。
为了进一步探究浓香型白酒窖池样品中的潜在关键微生物菌种,比较分析了窖泥和酒醅样品中关键微生物的差异。选取丰度排名前10的细菌物种,利用R(v3.4.1)软件分析细菌物种在每个分组中的平均相对丰度以及差异显著性,探究不同分析样品的关键微生物。物种差异分析结果见图4,梭菌属(Clostridium IV)、嗜蛋白质菌(Proteiniphilum)、岩石单胞菌(Petrimonas)、沉积物棒菌(Sedimentibacter)和喜热菌属(Caloramator)在窖泥样品中的相对丰度高于酒醅样品,这5个属均属于产氢产酸细菌种属,代谢产物包括丁酸、乙醇、乙酸、乳酸、H2和CO2等,常与产甲烷菌共生,这与窖泥细菌菌种的特性是一致的。
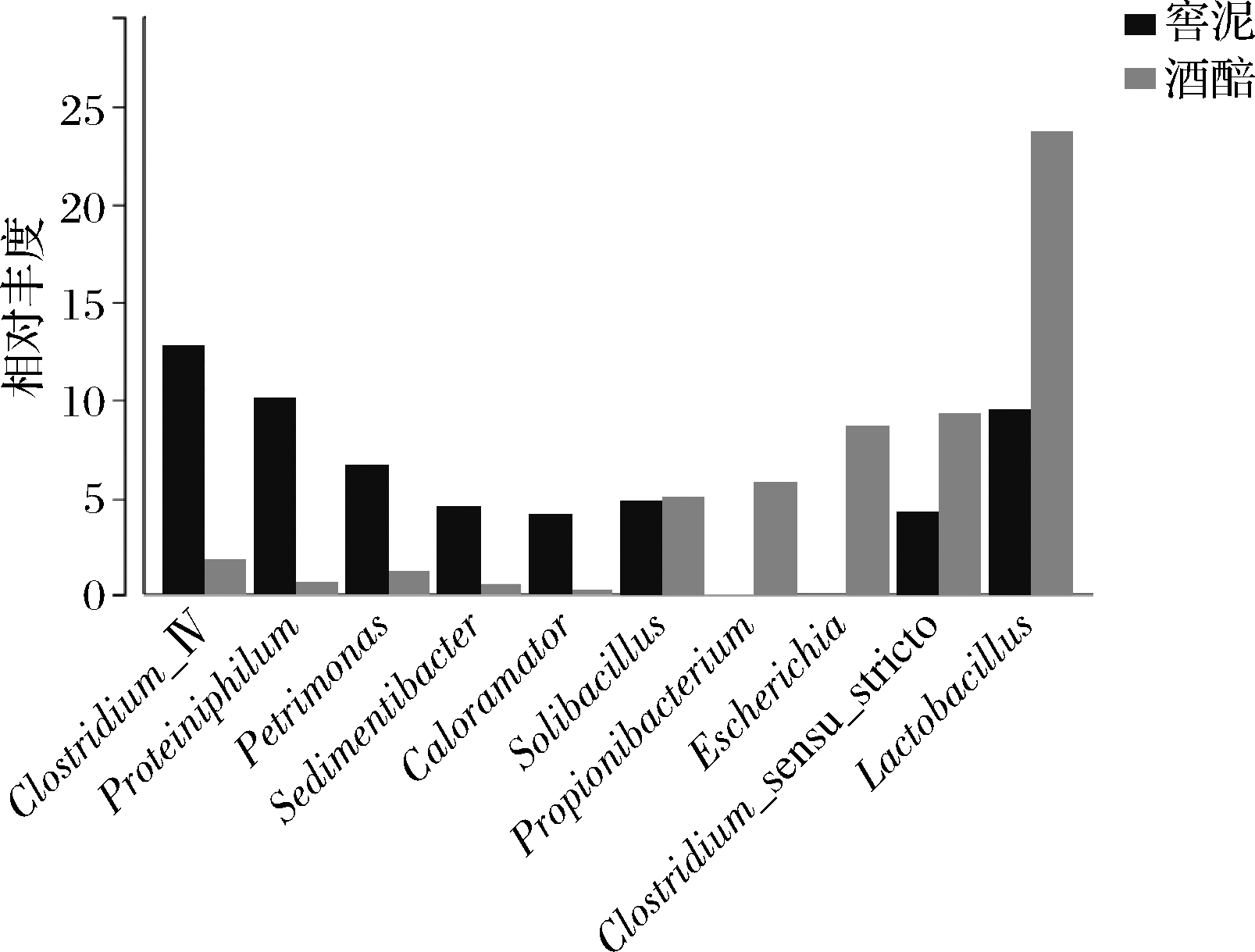
图4 浓香型白酒窖泥和酒醅样品关键微生物差异
Fig.4 Key microbial differences of pit muds and fermented grains samples in strong-flavor Baijiu
窖泥样品中的梭菌主要来自Clostridium IV簇,包括纤维素梭菌(Clostridium cellulosi)、柔嫩梭菌(Clostridium leptum)和球孢梭菌(Clostridium sporosphaeroides)[35],其中纤维素梭菌(C.cellulosi)能够降解纤维素[36],促进窖池中原料的利用,生成的小分子物质有利于窖池中其他微生物的生长。柔嫩梭菌(C.leptum)不但具备代谢生成丁酸的能力,还是目前报道的梭菌益生菌[37]。丙酸杆菌属(Propionibacterium)、埃希氏菌属(Escherichia)、严格厌氧梭菌(Clostridium sensu stricto)和乳杆菌属(Lactobacillus)在酒醅样品中的相对丰度高于窖泥样品。随着发酵的进行,体系内酸度和乙醇浓度的升高,O2含量逐渐减少,不耐受高酸度、高乙醇浓度、厌氧等不利条件的菌株逐渐衰亡,能够适应这些条件的菌株逐渐成为优势菌种。其中,丙酸杆菌属菌株能够代谢生成丙酸,严格厌氧梭菌能够代谢产生丁酸、酪丁酸等,乳杆菌属菌株在酒醅中能够产生乳酸、乙酸等风味物质,乳酸能够与其他微生物代谢产生的乙醇生成乳酸乙酯,增加酒体的醇甜感、醇厚度,是重要的呈香物质[30]。埃希氏菌属(Escherichia)多为环境条件致病菌,主要存在于二车间酒醅样品J2中,在一定程度上反映出二车间酒醅的质量不如一车间和五车间,与二车间窖泥的质量不如一车间和五车间以及聚类分析研究的结果一致。
3 结论
本研究利用高通量测序技术全面解析浓香型白酒窖泥和酒醅的细菌群落结构,探究了浓香型白酒窖池中潜在的关键细菌菌种。窖泥样品中的细菌物种总数和多样性均高于酒醅样品,窖泥样品中关键的细菌菌种主要来自梭菌属(Clostridium IV)、岩石单胞菌属(Petrimonas)、沉积物棒菌属(Sedimentibacter)、嗜蛋白菌属(Proteiniphilum)和喜热菌属(Caloramator);酒醅样品中关键的细菌菌种主要来自乳杆菌属(Lactobacillus)、丙酸杆菌属(Propionibacterium)、梭菌属(Clostridium sensu stricto)和埃希氏菌属(Escherichia)。本研究为浓香型白酒窖池中特征性菌种的定向筛选和微生物可培养组的研究奠定了基础。
[1] 国家市场监督管理总局,国家标准化管理委员会.白酒质量要求 第1部分:浓香型白酒:GB/T 10781.1—2021[S].北京:中国标准出版社,2021.
State Administration for Market Regulation,Standardization Administration.GB/T 10781.1—2021 Quality requirements for baijiu—Part 1:Nongxiangxing baijiu[S].Beijing:Standards Press of China,2021.
[2] 张志伟.浓香型白酒窖泥微生物的研究[D].济南:齐鲁工业大学,2018.
ZHANG Z W.Study on microorganisms in pit mud of Luzhou-flavor liquor[D].Jinan:Qilu University of Technology,2018.
[3] 翟磊,于学健,冯慧军,等.宜宾产区浓香型白酒酿造生境中细菌的群落结构[J].食品与发酵工业,2020,46(2):18-24.
ZHAI L,YU X J,FENG H J,et al.Study on bacterial community structure in the brewing habitats of strong flavour Chinese Baijiu from Yibin region[J].Food and Fermentation Industries,2020,46(2):18-24.
[4] 张宿义,沈才洪,许德富.浓香型白酒的技术发展回顾[J].酿酒,2009,36(1):8-10.
ZHANG S Y,SHEN C H,XU D F.Review on the technological development of Luzhou-flavor style liquor[J].Liquor-Making,2009,36(1):8-10.
[5] 周燚,刘小锦,朱晨光,等.细菌中群体感应调节系统[J].微生物学报,2004,44(1):122-126.
ZHOU Y,LIU X J,ZHU C G,et al.Quorum sensing in bacteria[J].Acta Microbiologica Sinica,2004,44(1):122-126.
[6] 周楠,姜成英,刘双江.从环境中分离培养微生物:培养基营养水平至关重要[J].微生物学通报,2016,43(5):1075-1081.
ZHOU N,JIANG C Y,LIU S J.Cultivation of microorganisms from environments:Nutrient level of the culture medium is of great importance[J].Microbiology China,2016,43(5):1075-1081.
[7] 郭斌,吴晓磊,钱易.提高微生物可培养性的方法和措施[J].微生物学报,2006,46(3):504-507.
GUO B,WU X L,QIAN Y.Approaches for increasing the culturability of microorganisms[J].Acta Microbiologica Sinica,2006,46(3):504-507.
[8] KAEBERLEIN T,LEWIS K,EPSTEIN S S.Isolating “uncultivable”microorganisms in pure culture in a simulated natural environment[J].Science,2002,296(5570):1127-1129.
[9] BLOOMFIELD S F,STEWART G S,DODD C E,et al.The viable but non-culturable phenomenon explained?[J].Microbiology,1998,144(1):1-3.
[10] 罗惠波,甄攀,黄治国.浓香型白酒窖池细菌群落[J].微生物学通报,2010,37(11):1621-1627.
LUO H B,ZHEN P,HUANG Z G.Research on bacterial community in Luzhou-flavor liquors pit[J].Microbiology China,2010,37(11):1621-1627.
[11] WANG H Y,GAO Y B,FAN Q W,et al.Characterization and comparison of microbial community of different typical Chinese liquor Daqus by PCR-DGGE[J].Letters in Applied Microbiology,2011,53(2):134-140.
[12] 王明跃,张文学,王海英,等.不同窖龄窖泥古菌的系统发育多样性分析[J].应用与环境生物学报,2012,18(6):1043-1048.
WANG M Y,ZHANG W X,WANG H Y,et al.Phylogenetic diversity analysis of archaeal in the pit mud with different cellar age[J].China Industrial Economics,2012,18(6):1043-1048.
[13] SCHIRMER M,IJAZ U Z,D’AMORE R,et al.Insight into biases and sequencing errors for amplicon sequencing with the Illumina MiSeq platform[J].Nucleic Acids Research,2015,43(6):e37.
[14] 秦楠,栗东芳,杨瑞馥.高通量测序技术及其在微生物学研究中的应用[J].微生物学报,2011,51(4):445-457.
QIN N,LI D F,YANG R F.Next-generation sequencing technologies and the application in microbiology:A review[J].Acta Microbiologica Sinica,2011,51(4):445-457.
[15] 王兴春,杨致荣,王敏,等.高通量测序技术及其应用[J].中国生物工程杂志,2012,32(1):109-114.
WANG X C,YANG Z R,WANG M,et al.High-throughput sequencing technology and its application[J].China Biotechnology,2012,32(1):109-114.
[16] 刘博,杜海,王雪山,等.基于高通量测序技术解析浓香型白酒中窖泥臭味物质4-甲基苯酚的来源[J].微生物学通报,2017,44(1):108-117.
LIU B,DU H,WANG X S,et al.Detecting source of p-cresol in strong flavor Chinese liquor by high throughput sequencing[J].Microbiology China,2017,44(1):108-117.
[17] 张应刚,许涛,郑蕾,等.窖泥群落结构及功能微生物研究进展[J].微生物学通报,2021,48(11):4327-4343.
ZHANG Y G,XU T,ZHENG L,et al.Research progress on community structure and functional microorganisms of pit mud[J].Microbiology China,2021,48(11):4327-4343.
[18] 刘延波,王琳琳,金尚萍,等.不同储藏期浓香型白酒大曲的微生物多样性分析[J].中国酿造,2022,41(4):105-110.
LIU Y B,WANG L L,JIN S P,et al.Microbial diversity analysis of strong-flavor Baijiu Daqu with different storage periods[J].China Brewing,2022,41(4):105-110.
[19] 王小平,黄永光,周文美.酱香型白酒大曲细菌菌群结构及功能研究进展[J].中国酿造,2020,39(2):19-25.
WANG X P,HUANG Y G,ZHOU W M.Research progress on structure and function of bacterial community in Moutai-flavor Baijiu Daqu[J].China Brewing,2020,39(2):19-25.
[20] 莫祯妮,邱树毅,曾祥勇,等.基于Illumina Hiseq高通量测序分析酱香型白酒大曲曲皮和曲心的细菌群落差异[J].中国酿造,2022,41(2):48-52.
MO Z N,QIU S Y,ZENG X Y,et al.Difference of bacterial community in different parts of high-temperature Daqu based on Illumina Hiseq high-throughput sequencing[J].China Brewing,2022,41(2):48-52.
[21] 张双燕,廖永红,纪南,等.基于高通量测序技术分析北京清香型大曲微生物多样性[J].中国酿造,2016,35(11):49-53.
ZHANG S Y,LIAO Y H,JI N,et al.Analysis on microbial diversity of Beijing light-flavor Daqu by high-throughput sequencing[J].China Brewing,2016,35(11):49-53.
[22] 陈申习,宿智新,张磊,等.基于高通量测序的清香型和酱香型酒曲真菌群落特征研究[J].中国酿造,2021,40(7):49-53.
CHEN S X,SU Z X,ZHANG L,et al.Study on Fungal Community Characteristics of Fen-flavor and Maotai-flavor Daqu Based on high-throughput sequencing[J].China Brewing,2021,40(7):49-53.
[23] ZHOU J,BRUNS M A,TIEDJE J M.DNA recovery from soils of diverse composition[J].Applied and Environmental Microbiology,1996,62(2):316-322.
[24] ZHANG X L,TIAN X Q,MA L Y,et al.Biodiversity of the symbiotic bacteria associated with toxic marine dinoflagellate Alexandrium tamarense[J].Journal of Biosciences and Medicines,2015,3(6):23-28.
[25] HE W,ZHAO S,LIU X,et al.ReSeqTools:An integrated toolkit for large-scale next-generation sequencing based resequencing analysis[J].Genetics and Molecular Research,2013,12(4):6275-6283.
[26] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-998.
[27] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[28] SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur:Open-source,platform-independent,community-supported software for describing and comparing microbial communities[J].Applied and Environmental Microbiology,2009,75(23):7537-7541.
[29] 岳元媛,张文学,刘霞,等.浓香型白酒窖泥中兼性厌氧细菌的分离鉴定[J].微生物学通报,2007,34(2):251-255.
YUE Y Y,ZHANG W X,LIU X,et al.Isolation and identification of facultative anaerobes in the pit mud of Chinese Luzhou-flavor liquor[J].Microbiology,2007,34(2):251-255.
[30] 田雨思,衡小成,邹伟,等.浓香型白酒窖泥中梭菌的分离、鉴定及产丁酸性能的比较研究[J].食品与发酵工业,2019,45(23):60-65.
TIAN Y S,HENG X C,ZOU W,et al.Isolation and identification of clostridia from the pit mud of strong-flavor Baijiu and comparative study on butyric acid production[J].Food and Fermentation Industries,2019,45(23):60-65.
[31] 王明跃,张文学,王海英,等.不同窖龄窖泥古菌的系统发育多样性分析[J].应用与环境生物学报,2012,18(6):1043-1048.
WANG M Y,ZHANG W X,WANG H Y,et al.Phylogenetic diversity analysis of archaeal in the pit mud with different cellar age[J].Chinese Journal of Applied and Environmental Biology,2012,18(6):1043-1048.
[32] BAENA S,FARDEAU M L,LABAT M,et al.Aminobacterium mobile sp.nov.,a new anaerobic amino-acid-degrading bacterium[J].International Journal of Systematic and Evolutionary Microbiology,2000,50(Pt 1):259-264.
[33] CHEN S Y,DONG X Z.Proteiniphilum acetatigenes Gen.nov.,sp.nov.,from a UASB reactor treating brewery wastewater[J].International Journal of Systematic and Evolutionary Microbiology,2005,55(Pt 6):2257-2261.
[34] 邢敏钰,杜海,徐岩.芝麻香型白酒发酵过程中乳酸菌多样性及其演替规律[J].微生物学通报,2018,45(1):19-28.
XING M Y,DU H,XU Y.Diversity and succession of lactic acid bacteria during sesame-flavor liquor fermentation[J].Microbiology China,2018,45(1):19-28.
[35] COLLINS M D,LAWSON P A,WILLEMS A,et al.The phylogeny of the genus Clostridium:Proposal of five new Genera and eleven new species combinations[J].International Journal of Systematic Bacteriology,1994,44(4):812-826.
[36] HE Y L,DING Y F,LONG Y Q.Two cellulolytic Clostridium species:Clostridium cellulosi sp.nov.and Clostridium cellulofermentans sp.nov[J].International Journal of Systematic Bacteriology,1991,41(2):306-309.
[37] 刘又嘉,贺璐,郭艳芳,等.肠道梭菌的研究进展[J].世界华人消化杂志,2017,25(22):2007-2014.
LIU Y J,HE L,GUO Y F,et al.Research progress of intestinal Clostridium[J].World Chinese Journal of Digestology,2017,25(22):2007-2014.