在工业生物催化过程中,辅因子的高效供应是开发氧化还原酶催化反应的关键技术之一[1]。其中,辅因子再生系统是具有潜力的研究方向,可以促进生物催化反应的过程经济性和工业可行性[2-3]。人们已经提出了酶法、电化学法和光化学法再生等辅因子再生系统[2,4]。其中酶法再生是指利用2个平行的氧化还原反应酶系统分别进行酶催化底物转化和酶催化辅因子循环再生。其拥有众多优势,例如方便制备酶制剂、反应条件温和、对生物转化反应具有良好兼容性等优点[1]。
甲酸脱氢酶(formate dehydrogenase,FDH,EC 1.2.1.2)属于D-2-羟基酸脱氢酶类[5]。由FDH组成的辅因子再生系统具有较多优势,例如价格低廉、甲酸容易透过细胞膜、副产物仅为CO2、最适pH范围较广等[5]。但大多数天然的FDH为NAD+特异性,在反应过程中催化NAD+还原为NADH,因此,较多研究将FDH应用于NADH辅因子再生。为了将FDH应用于NADPH辅因子再生,寻找天然NADP+特异性FDH或改造NAD+特异性FDH的辅因子特异性成为2个主要研究方向。
巴斯德毕赤酵母甲酸脱氢酶(FDH from Komagataella phaffii,KphFDH)是典型的NAD+特异性FDH[6]。目前,对其相关研究较少,特别是改造辅因子特异性方面。本研究以KphFDH为研究对象,基于多序列比对、文献调研的结果,确定了D195、Y196为突变位点,构建了16种突变体,并表征了野生型和突变体的比酶活力和酶促反应动力学,最终得到最优的NADP+特异性突变体为KphFDHD195Q/Y196R,并成功将该突变体与来自嗜热共生细菌的内消旋-二氨基庚二酸脱氢酶突变体(Meso-diaminopimelate dehydrogenase from Symbiobacterium thermophilum,StDAPDHH227V)应用于苯丙酮酸(phenylpyruvic acid,PPA)的还原胺化反应[7-8]。
1 材料与方法
1.1 实验材料
1.1.1 菌株及质粒
大肠杆菌(Escherichia coli)JM109用于构建克隆,E.coli BL21(DE3)用于表达不同基因型的KphFDH,上述2种菌株均由实验室保存。
研究所用质粒见表1。野生型KphFDH的表达载体pET-22b(+)-FDHWT由安升达公司合成,其余质粒为突变型KphFDH表达载体,均以pET-22b(+)-FDHWT为模板构建;突变型StDAPDHH227V表达载体pET-28a(+)-StDAPDHH227由江南大学聂尧教授赠予[8]。
表1 本研究所用质粒
Table 1 Plasmids used in this study

质粒描述来源pET-22b(+)-FDHWTpET-22b(+)表达质粒,含有KphFDH野生型基因;Ampr安升达公司合成pET-22b(+)-FDHD195ApET-22b(+)表达质粒,含有KphFDHD195A突变型基因;Ampr本研究pET-22b(+)-FDHD195NpET-22b(+)表达质粒,含有KphFDHD195N突变型基因;Ampr本研究pET-22b(+)-FDHD195QpET-22b(+)表达质粒,含有KphFDHD195Q突变型基因;Ampr本研究pET-22b(+)-FDHD195SpET-22b(+)表达质粒,含有KphFDHD195S突变型基因;Ampr本研究pET-22b(+)-FDHD195A/Y196RpET-22b(+)表达质粒,含有KphFDHD195A/Y196R突变型基因;Ampr本研究pET-22b(+)-FDHD195Q/Y196HpET-22b(+)表达质粒,含有KphFDHD195Q/Y196H突变型基因;Ampr本研究pET-22b(+)-FDHD195Q/Y196PpET-22b(+)表达质粒,含有KphFDHD195Q/Y196P突变型基因;Ampr本研究pET-22b(+)-FDHD195Q/Y196RpET-22b(+)表达质粒,含有KphFDHD195Q/Y196R突变型基因;Ampr本研究pET-22b(+)-FDHD195Q/Y196SpET-22b(+)表达质粒,含有KphFDHD195Q/Y196S突变型基因;Ampr本研究pET-22b(+)-FDHD195S/Y196ApET-22b(+)表达质粒,含有KphFDHD195S/Y196A突变型基因;Ampr本研究pET-22b(+)-FDHD195S/Y196HpET-22b(+)表达质粒,含有KphFDHD195S/Y196H突变型基因;Ampr本研究pET-22b(+)-FDHD195S/Y196LpET-22b(+)表达质粒,含有KphFDHD195S/Y196L突变型基因;Ampr本研究pET-22b(+)-FDHD195S/Y196PpET-22b(+)表达质粒,含有KphFDHD195S/Y196P突变型基因;Ampr本研究pET-22b(+)-FDHD195S/Y196SpET-22b(+)表达质粒,含有KphFDHD195S/Y196S突变型基因;Ampr本研究pET-22b(+)-FDHD195S/Q197TpET-22b(+)表达质粒,含有KphFDHD195S/Q197T突变型基因;Ampr本研究pET-22b(+)-FDHD195S/Q197VpET-22b(+)表达质粒,含有KphFDHD195S/Q197V突变型基因;Ampr本研究pET-28a(+)-StDAPDHH227VpET-28a(+)表达质粒,含有StDAPDHH227V突变型基因;Kanr江南大学聂尧教授赠予
1.1.2 主要试剂
DNA定点突变引物由安升达公司合成;同源重组酶,武汉爱博泰克生物科技有限公司;Primer star DNA聚合酶、质粒提取试剂盒、胶回收试剂盒,康为世纪公司;β-烟酰胺腺嘌呤二核苷酸(β-Nicotinamide adenine dinucleotide,NAD+)、β-烟酰胺腺嘌呤二核苷酸磷酸二钠盐(β-Nicotinamide adenine dinucleotide phosphate disodium salt,NADP+·2 Na)、甲酸钠(sodium formate,NaFA),上海麦克林生化科技有限公司;酵母提取物、胰蛋白胨等常规试剂,国药集团化学试剂有限公司。
1.1.3 培养基
Luria-Bertani(LB)培养基(g/L):酵母提取物5,胰蛋白胨10,NaCl 10,配制固体培养基时加入琼脂粉10。
1.2 仪器与设备
PCR仪,杭州朗基科学仪器有限公司;恒温摇床,上海五相仪器仪表有限公司;低温离心机,美国Thermo Fisher Scientific公司;高压匀浆破碎仪,永联生物科技(上海)有限公司;AKTA蛋白纯化仪,美国GE公司;电泳仪,北京六一生物科技有限公司;SYNERGY H1多功能酶标仪,美国伯腾仪器有限公司;恒温金属浴,杭州米欧仪器有限公司。
1.3 实验方法
1.3.1 野生型KphFDH表达质粒的获取
通过NCBI查询野生型KphFDH氨基酸序列(NCBI序列号:XP_002493171.1),递交至苏州安升达公司进行密码子优化和基因合成,并将该基因插入pET-22b(+)表达载体的Nde I和Xho I多克隆位点,即得到pET-22b(+)-FDHWT表达质粒。
1.3.2 KphFDH基因定点突变
利用双片段同源重组法对KphFDH进行定点突变。以pET-22b(+)-FDHWT表达质粒为模板,利用表2中的引物进行PCR(某一突变型的正向引物与Bone-R进行PCR得到一个片段,对应突变型的反向引物与Bone-F进行PCR得到另一个片段),PCR程序如下:98 ℃预变性3 min;随后进行35个循环(98 ℃ 15 s,54 ℃ 15 s,72 ℃ 20 s),最后72 ℃保温5 min。所得2个PCR片段经DNA纯化后,进行体外同源重组(操作步骤详见ABclonal同源重组说明书)。同源重组产物直接转化至E.coli JM109,并通过测序验证突变是否正确,将正确构建的突变体质粒转化至E.coli BL21(DE3)进行蛋白表达。
表2 本研究所用引物
Table 2 Primers used in this study

引物引物序列(5′→3′)Bone-FGCGCCTGATGCGGTATTTTCBone-RGAAAATACCGCATCAGGCGCD195A-FAAGAATTACTGTACTATGCATATCAAGGCCTGCCGAD195A-RTGATATGCATAGTACAGTAATTCTTTCGGGTTAAACGD195N-FAGAATTACTGTACTATAATTATCAAGGCCTGCCGAD195N-RATAATTATAGTACAGTAATTCTTTCGGGTTAAACGCD195Q-FGAATTACTGTACTATCAGTATCAAGGCCTGCCGAAAGD195Q-RGATACTGATAGTACAGTAATTCTTTCGGGTTAAACGD195S-FGAATTACTGTACTATAGCTATCAAGGCCTGCCGAAAGAAGCD195S-RATAGCTATAGTACAGTAATTCTTTCGGGTTAAACGD195A/Y196R-FAAGAATTACTGTACTATGCGCGCCAAGGCCTGCCGAAAGAAGCD195A/Y196R-RTGGCGCGCATAGTACAGTAATTCTTTCGGGTTAAACGCD195Q/Y196H-FGAAAGAATTACTGTACTATCAGCATCAAGGCCTGCCGAAAGAAGD195Q/Y196H-RATGCTGATAGTACAGTAATTCTTTCGGGTTAAACGD195Q/Y196P-FGAAAGAATTACTGTACTATCAGCCGCAAGGCCTGCCGAAAGAAGCD195Q/Y196P-RCGGCTGATAGTACAGTAATTCTTTCGGGTTAAACGD195Q/Y196R-FGAAAGAATTACTGTACTATCAGCGCCAAGGCCTGCCGAAAGAAGCD195Q/Y196R-RGCGCTGATAGTACAGTAATTCTTTCGGGTTAAACGD195Q/Y196S-FGAAAGAATTACTGTACTATCAGAGCCAAGGCCTGCCGAAAGAAGD195Q/Y196S-RGCTCTGATAGTACAGTAATTCTTTCGGGTTAAACGD195S/Q197T-FGAATTACTGTACTATAGCTATACCGGCCTGCCGAAAGAAGCGGAAGD195S/Q197T-RATAGCTATAGTACAGTAATTCTTTCGGGTTAAACGCD195S/Q197V-FGAATTACTGTACTATAGCTATGTTGGCCTGCCGAAAGAAGCGGAAGD195S/Q197V-RATAGCTATAGTACAGTAATTCTTTCGGGTTAAACGCD195S/Y196A-FGAATTACTGTACTATAGCGCACAAGGCCTGCCGAAAGAAGCGGD195S/Y196A-RTGCGCTATAGTACAGTAATTCTTTCGGGTTAAACGCD195S/Y196H-FGAAAGAATTACTGTACTATAGCCATCAAGGCCTGCCGAAAGAAGD195S/Y196H-RATGGCTATAGTACAGTAATTCTTTCGGGTTAAACGD195S/Y196L-FGAATTACTGTACTATAGCCTGCAAGGCCTGCCGAAAGAAGCGGD195S/Y196L-RCAGGCTATAGTACAGTAATTCTTTCGGGTTAAACGCD195S/Y196P-FGAATTACTGTACTATAGCCCGCAAGGCCTGCCGAAAGAAGCGGD195S/Y196P-RCGGGCTATAGTACAGTAATTCTTTCGGGTTAAACGCCD195S/Y196S-FGAATTACTGTACTATAGCAGCCAAGGCCTGCCGAAAGAAGCGGD195S/Y196S-RGCTGCTATAGTACAGTAATTCTTTCGGGTTAAACGC
1.3.3 重组菌发酵
种子液培养:将重组菌接种于5 mL LB培养基(KphFDH重组菌培养需添加60 μg/mL氨苄青霉素,StDAPDHH227V重组菌培养需添加50 μg/mL卡那霉素),于37 ℃、220 r/min摇床上振荡培养12 h。
摇瓶发酵:将种子液以体积分数1%的接种量接入500 mL LB培养基,于37 ℃、220 r/min培养至菌体浓度OD600=0.6~0.8,加入终浓度为0.2 mmol/L IPTG于16 ℃、160 r/min条件下诱导12 h。
1.3.4 蛋白纯化
取发酵液于6 000 r/min离心10 min收集诱导后的菌体,用平衡缓冲液A(16 mmol/L Na2HPO4,4 mmol/L NaH2PO4,500 mmol/L NaCl,pH=7.4)进行重悬,高压匀浆破碎仪破碎(600 bar,5 min)后,6 000 r/min离心10 min,收集上清液。
利用AKTA蛋白纯化仪进行纯化:将镍柱对接AKTA设备;用平衡缓冲液A冲洗镍柱(1 mL/min流速,冲洗5个柱体积)直至UV基线平稳;上清液以0.5 mL/min流速进样;进样结束后,用平衡缓冲液A继续冲洗柱子直至UV基线平稳;流速改为1 mL/min,分别以不同比例的洗脱缓冲液B(16 mmol/L Na2HPO4,4 mmol/L NaH2PO4,500 mmol/L NaCl,500 mmol/L咪唑,pH=7.4)进行梯度洗脱(KphFDH的洗杂浓度为35% B液,洗脱浓度为100% B液;StDAPDHH227V的洗杂浓度为50% B液,洗脱浓度为100% B液)。
1.3.5 SDS-PAGE分析及超滤脱盐
蛋白纯化样品处理:加入适量上样缓冲液与样品混匀,煮沸5 min变性。采用YEASEN公司免封闭PAGE凝胶快速制备试剂盒进行蛋白胶制备(分离胶和浓缩胶浓度均为12.5%)。待蛋白胶凝固后,加入处理好的样品10 mL,置于SDS-PAGE缓冲液中90 V电泳2 h,至溴酚蓝带跑出胶板底部;跑胶结束后,用考马斯亮蓝R-250染色液进行染色,考马斯亮蓝染色脱色液进行脱色;脱色后,用凝胶成像系统拍照,取具有单一目的条带的蛋白样品置于超滤管中超滤脱盐,以除去蛋白样品中的咪唑并将缓冲液置换为后续实验所需的缓冲液,超滤脱盐后的蛋白样品置于4 ℃保存。
1.3.6 蛋白浓度测定
采用YEASEN公司BCA蛋白浓度测定试剂盒,具体操作详见说明书。
1.3.7 KphFDH比酶活力测定
反应在Na2HPO4-NaH2PO4缓冲液(10 mmol/L,pH=7.0)中进行,含500 mmol/L NaFA,1 mmol/L NAD+(NADP+),适量的纯化KphFDH蛋白,总反应体系为200 mL,温度为30 ℃。利用多功能酶标仪监测反应进程,将底物混合液置于96孔酶标板,添加适量的纯化KphFDH蛋白启动反应,每隔60 s测定反应生成的NADH(NADPH)在340 nm吸光度的增量[e=6.22 L/(mmol·cm)],检测5 min。酶活力定义为:在pH=7.0,T=30 ℃条件下,每分钟生成1 μmol NADH(NADPH)所需要的酶量为1个酶活力单位U。KphFDH蛋白大小按41.1 kDa计算。
1.3.8 KphFDH酶促反应动力学分析
与比酶活力测定方法相似,固定反应体系中的NaFA浓度为500 mmol/L以及适量的纯化KphFDH蛋白,调整NAD+(NADP+)浓度为0~30 mmol/L。甲酸酶促反应动力学测定:与比酶活力测定方法相似,固定反应体系中的辅因子浓度(KphFDHWT对应的NAD+浓度为2 mmol/L,KphFDHD195Q/Y196R对应的NADP+浓度为6 mmol/L)以及适量的纯化KphFDH蛋白,调整NaFA浓度为0~500 mmol/L。用GraphPad Prism 8.0处理得到的数据,将曲线拟合至Michaelis-Menten方程,得到表观kcat和Km值。
1.3.9 KphFDHD195Q/Y196R与StDAPDHH227V的体外反应
体外反应条件:反应在Na2HPO4-NaH2PO4缓冲液(50 mmol/L,pH=7.5)中进行,含20 mmol/L PPA,100 mmol/L NH4Cl,100 mmol/L NaFA,1 mmol/L NADP+,500 mg/mL KphFDHD195Q/Y196R突变体纯化蛋白,500 mg/mL StDAPDHH227V纯化蛋白,反应体系为1 mL,于37 ℃、220 r/min摇床上振荡反应240 min。作为对照组,反应体系中不加入KphFDHD195Q/Y196R突变体纯化蛋白。每隔20 min取样,加入等体积的质量分数为10%的三氯乙酸终止反应并用于后续测定PPA浓度。
通过监测PPA浓度随时间变化的关系来监测反应进程。PPA浓度测定方法:使用FeCl3显色法[8-9],0.1 mol/L的FeCl3溶解于6 mL二甲亚砜,继续添加4 mL去离子水和200 μL乙酸,将制备的FeCl3溶液置于冰浴中,将15 mL的等分取样样品添加于1 mL FeCl3溶液中,将混合物置于室温下孵育2 min,利用酶标仪测定其640 nm的吸光度。同时,配制PPA标品浓度为20、15、10、5、0 mmol/L,并测定其640 nm的吸光度。利用不同浓度的PPA标品与对应的640 nm吸光度做出标准曲线并确定取样样品中PPA的浓度。
2 结果和分析
2.1 KphFDH突变位点、突变组合的确定以及突变体制备
先前有大量改造FDH辅因子特异性的相关研究涉及来源于Candida bodinii的FDH(CboFDH)[10-11],Saccharomyces cerevisiae的FDH(SceFDH)[12-13],Candida methylica的FDH(CmeFDH)[14-15]。将KphFDH与CboFDH、SceFDH、CmeFDH进行氨基酸序列比对,结果显示4种酵母来源的FDH均有NAD+特异性FDH的“指纹序列”,即与辅因子结合的位置有经典的Rossmann折叠[16],其特征序列为(A)GXGXXGX18D(X为任何残基)(图1-a)。其中,位于Rossmann折叠末端带负电荷的天冬氨酸是FDH识别NAD+的决定残基[17],该残基后一位的酪氨酸残基也被证明参与辅因子识别并常将其作为选定位点进行突变[10,13]。根据氨基酸序列比对结果,4种酵母FDH的195,196位点的氨基酸残基均一致(图1-a)。因此,最终确定KphFDH的D195,Y196作为突变的目标位点。

a-4种酵母来源FDH的部分多序列比对(黑色方框为“指纹序列”,绿色方框为突变位点);b-FDH纯化结果示例
(M-蛋白分子质量标准;1-粗酶;2-纯化后的酶)
图1 FDH部分多序列比对结果与SDS-PAGE分析
Fig.1 The partial results of multiple sequences alignment and SDS-PAGE analysis of FDH
所有突变体来源于先前对其他酵母FDH的辅因子特异性改造相关研究。本研究所有的突变体及参考来源见表3,其中包括4种单点突变体和12种双点突变体。
表3 KphFDH突变体及参考来源
Table 3 The variants and reference of KphFDH

突变组合参考来源KphFDHD195A/Y196R[13]KphFDHD195S/Y196H[5]KphFDHD195Q/Y196R,KphFDHD195S/Y196P[11]KphFDHD195S/Q197T,KphFDHD195S/Q197V,KphFDHD195S/Y196A,KphFDHD195S/Y196L,KphFDHD195S/Y196S[15]KphFDHD195A,KphFDHD195N,KphFDHD195Q,KphFDHD195S,KphFDHD195Q/Y196H,KphFDHD195Q/Y196P,KphFDHD195Q/Y196S[10]
利用DNA定点突变将KphFDHWT突变为设计的16种突变体,并在E.coli BL21(DE3)中进行表达。如图1-b(其中1种KphFDH的表达和纯化示例)所示,KphFDH成功表达和纯化,蛋白条带位置与蛋白亚基理论大小(41.1 kDa)相符。
2.2 KphFDHWT及其突变体比酶活力测定
如表4所示,KphFDHWT对NAD+的比酶活力约为4.061 U/mg,当以NADP+为辅因子时无法催化反应进行,说明KphFDHWT是NAD+特异性FDH。
表4 KphFDHWT及其突变体比酶活力 单位:U/mg
Table 4 The specific activities of KphFDHWT and its variants

KphFDH类型NAD+NADP+KphFDHWT4.061±0.077/KphFDHD195A0.139±0.0030.011±0.001KphFDHD195N0.049±0.0010.015±0.001KphFDHD195Q0.192±0.0020.025±0.002KphFDHD195S0.216±0.0040.016±0.001KphFDHD195A/Y196R0.072±0.0030.204±0.003KphFDHD195Q/Y196H0.123±0.0020.505±0.011KphFDHD195Q/Y196P0.715±0.0030.108±0.009KphFDHD195Q/Y196R0.063±0.0021.049±0.039KphFDHD195Q/Y196S0.079±0.0010.029±0.001KphFDHD195S/Y196A0.057±0.0020.007±0.001KphFDHD195S/Y196H0.109±0.0010.095±0.003KphFDHD195S/Y196L0.252±0.0080.049±0.001KphFDHD195S/Y196P0.847±0.0060.154±0.006KphFDHD195S/Y196S0.073±0.0010.018±0.001KphFDHD195S/Q197T0.176±0.0130.029±0.001KphFDHD195S/Q197V0.189±0.0050.011±0.001
所有突变体对NAD+和NADP+均展现出一定的比酶活力。所有单点突变体对NADP+的比酶活力小于对NAD+的比酶活力。在双点突变体中,KphFDHD195A/Y196R、KphFDHD195Q/Y196H、KphFDHD195Q/Y196R对NADP+的比酶活力大于对NAD+的比酶活力,其中,改造效果最好的为KphFDHD195Q/Y196R,其对NAD+的比酶活仅为0.063 U/mg,对NADP+的比酶活力达到了1.049 U/mg。
通过比酶活力测定表明16种突变体中只有3种双点突变体对NADP+的比酶活力高于NAD+,且其中最优的改造突变体为KphFDHD195Q/Y196R。由KphFDHD195A、KphFDHD195A/Y196R和KphFDHD195Q、KphFDHD195Q/Y196R的比酶活力结果可知,在引入D195A或D195Q突变的基础上继续引入Y196R突变,可以进一步降低突变体对NAD+的特异性并提高对NADP+的特异性。同样,根据KphFDHD195Q、KphFDHD195Q/Y196H和KphFDHD195S、KphFDHD195S/Y196H的比酶活力结果,可以发现在D195Q或D195S的基础上继续引入Y196H突变,也具有相同的效果。根据KphFDHD195Q、KphFDHD195Q/Y196H、KphFDHD195Q/Y196R的比酶活力结果,可以发现引入Y196R突变比引入Y196H突变更有利于提高突变体对NADP+的特异性。
2.3 KphFDHWT及较优改造突变体的辅因子酶促反应动力学测定
为了进一步了解KphFDHWT和较优改造突变体的酶促反应动力学性质,对KphFDHWT和上述3种双点突变体进行了辅因子酶促反应动力学测定(表5)。其中,KphFDHWT对NAD+的Km为0.088 mmol/L,kcat为2.961 s-1,kcat/Km为33.648 L/(mmol·s)。
表5 KphFDHWT及部分突变体的辅因子酶促反应动力学
Table 5 The kinetics of enzymatic reaction for cofactor of KphFDHWT and patial variants
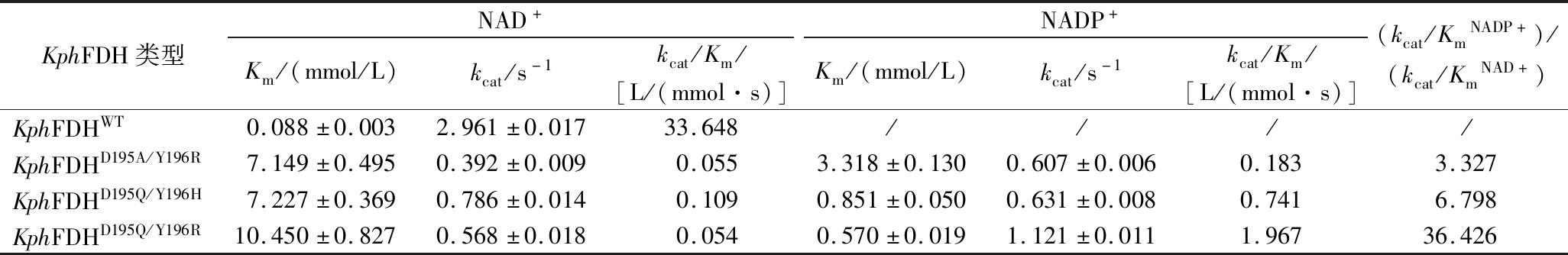
KphFDH类型NAD+NADP+Km/(mmol/L)kcat/s-1kcat/Km/[L/(mmol·s)]Km/(mmol/L)kcat/s-1kcat/Km/[L/(mmol·s)](kcat/KmNADP+)/(kcat/KmNAD+)KphFDHWT0.088±0.0032.961±0.01733.648////KphFDHD195A/Y196R7.149±0.4950.392±0.0090.0553.318±0.1300.607±0.0060.1833.327KphFDHD195Q/Y196H7.227±0.3690.786±0.0140.1090.851±0.0500.631±0.0080.7416.798KphFDHD195Q/Y196R10.450±0.8270.568±0.0180.0540.570±0.0191.121±0.0111.96736.426
在3种突变体中,KphFDHD195Q/Y196R对NAD+的亲和性最低,Km为10.450 mmol/L。对NAD+催化转化数最低的突变体为KphFDHD195A/Y196R,kcat为0.392 s-1。KphFDHD195A/Y196R和KphFDHD195Q/Y196R对NAD+的催化效率相近,均低于KphFDHD195Q/Y196H突变体,kcat/Km分别为0.055 L/(mmol·s)和0.054 L/(mmol·s)。
KphFDHD195Q/Y196R对NADP+具有最高的亲和性,Km为0.570 mmol/L;其对NADP+的催化转化数也最高,kcat为1.121 s-1。因此,该突变体对NADP+催化效率最高,kcat/Km为1.967 L/(mmol·s)。
KphFDHD195A/Y196R、KphFDHD195Q/Y196H和KphFDHD195Q/Y196R的催化特异性比率分别为3.327、6.798和36.426。所有突变体中,KphFDHD195Q/Y196R展现出最优的改造效果。
2.4 KphFDHWT和KphFDHD195Q/Y196R的甲酸酶促反应动力学测定
为了进一步表征KphFDHD195Q/Y196R的酶促反应动力学性质,本研究进一步测定了KphFDHWT以NAD+为辅因子和该突变体以NADP+为辅因子条件下的甲酸酶促反应动力学(表6)。
表6 KphFDHWT和KphFDHD195Q/Y196R的甲酸酶促反应动力学
Table 6 The kinetics of enzymatic reaction for formate of KphFDHWT and KphFDHD195Q/Y196R

KphFDH类型辅因子底物甲酸Km/(mmol/L)kcat/s-1kcat/Km/[L/(mmol·s)]KphFDHWTNAD+6.122±0.2132.898±0.0190.473KphFDHD195Q/Y196RNADP+58.36±3.7401.199±0.0230.020
KphFDHWT以NAD+为辅因子时,对甲酸的Km为6.122 mmol/L,kcat为2.898 s-1,kcat/Km为0.473 L/(mmol·s)。与KphFDHWT相比,KphFDHD195Q/Y196R以NADP+为辅因子时,对甲酸的亲和性、催化转化数和催化效率均下降。
KphFDHD195Q/Y196R以NADP+为辅因子时,对甲酸亲和性下降的现象可能归因于来源于酵母的FDH遵循有序Bi-Bi反应机制[18],具体机理还需进一步研究。
2.5 KphFDHD195Q/Y196R应用于体外NADPH辅因子再生系统
为了证明KphFDHD195Q/Y196R是否可以用于体外NADPH辅因子再生系统,本研究挑选了一个模式反应[8](图2-a),在该反应中,PPA与NH4Cl在StDAPDHH227V的催化下生成D-苯丙氨酸(D-phenylalanine,D-PA)和H2O,同时NADPH作为辅因子底物被氧化为NADP+;NADP+作为辅因子底物被KphFDHD195Q/Y196R还原为NADPH,同时消耗甲酸生成CO2。将相关反应底物和上述2种纯化后的酶添加至一锅进行反应,理论上可以驱动NADP+-NADPH辅因子循环,不断消耗PPA生成D-PA。

a-体外NADPH辅因子再生系统模式反应原理;b-StDAPDHH227V纯化结果(M-蛋白分子质量标准;1-粗酶;2-纯化后的酶);c-体外NADPH辅因子再生系统结果(对照组-添加相关底物和StDAPDHH227V纯化酶;实验组-添加相关底物、StDAPDHH227V和KphFDHD195Q/Y196R纯化酶)
图2 体外NADPH辅因子再生系统模式反应原理、StDAPDHH227V的SDS-PAGE分析以及反应结果
Fig.2 Principle of model reaction of in vitro NADPH cofactor regeneration system,SDS-PAGE analysis of StDAPDHH227V,and result of the reaction
如图2-b所示,StDAPDHH227V成功表达和纯化,蛋白条带位置与蛋白理论大小(34.2 kDa)相符。将相关底物和上述2种酶添加至一锅进行反应,并添加NADP+启动反应,通过监测底物PPA浓度随时间的变化来监测反应进程。如图2-c所示,对照组中PPA浓度在初始浓度左右波动;而添加了上述2种酶的实验组中,PPA浓度随时间下降,并在200 min后几乎完全反应。证明了KphFDHD195Q/Y196R可以应用于体外NADPH辅因子再生系统。
3 结论与讨论
目前已有大量报道将FDH应用于体外辅因子再生系统[19]。然而,天然FDH大多是NAD+特异性的,改造NAD+特异性FDH将其转变为NADP+特异性并应用于NADPH辅因子再生系统成为了一个研究方向。本研究为了改造KphFDH的辅因子特异性,根据多序列比对和文献调研的结果,确定了D195、Y196突变位点,构建了16个突变体,并表征了野生型和突变体的比酶活力和酶促反应动力学。最终得到最优的NADP+特异性突变体为KphFDHD195Q/Y196R。最后成功将该突变体应用于体外NADPH辅因子再生系统,证明其具有可应用性。本研究填补了KphFDH辅因子特异性改造方面的研究空白,并为发酵工业中体外NADPH辅因子再生系统提供了一种工具酶。但本研究仅限于对KphFDH的D195、Y196位点进行理性设计和改造,为了进一步提高其对NADP+的特异性,未来可以以该突变体出发,挖掘更多相关突变位点和突变体组合。基于目前已有体内NADPH再生筛选工具[20],未来也可以通过半理性设计的方法进一步改造KphFDH的辅因子特异性。
[1] 黄志华,刘铭,王宝光,等.甲酸脱氢酶用于辅酶NADH再生的研究进展[J].过程工程学报,2006,6(6):1011-1016.
HUANG Z H,LIU M,WANG B G,et al.Formate dehydrogenase and its application in cofactor NADH regeneration[J].The Chinese Journal of Process Engineering,2006,6(6):1011-1016.
[2] VAN DER DONK W A,ZHAO H M.Recent developments in pyridine nucleotide regeneration[J].Current Opinion in Biotechnology,2003,14(4):421-426.
[3] WICHMANN R,VASIC-RACKI D.Cofactor regeneration at the lab scale[J].Advances in Biochemical Engineering/Biotechnology,2005,92:225-260.
[4] 江金鹏,吴旭日,陈依军.解决氧化还原酶反应体系中辅酶问题的策略及其应用[J].生物工程学报,2012,28(4):410-419.
JIANG J P,WU X R,CHEN Y J.Strategy to solve cofactor issues in oxidoreductase catalyzed biocatalytic applications[J].Chinese Journal of Biotechnology,2012,28(4):410-419.
[5] TISHKOV V I,POPOV V O.Protein engineering of formate dehydrogenase[J].Biomolecular Engineering,2006,23(2-3):89-110.
[6] 蔡礼年,黄春辉,林陈水.毕赤酵母甲酸脱氢酶在大肠杆菌中的融合表达及其酶学性质[J].氨基酸和生物资源,2015,37(1):30-34.
CAI L N,HUANG C H,LIN C S.Fusion expression of formate dehydrogenase from Pichia pastoris in E.coli and its enzymatic property[J].Biotic Resources,2015,37(1):30-34.
[7] GAO X Z,HUANG F,FENG J H,et al.Engineering the meso-diaminopimelate dehydrogenase from Symbiobacterium thermophilum by site saturation mutagenesis for D-phenylalanine synthesis[J].Applied and Environmental Microbiology,2013,79(16):5078-5081.
[8] ZHANG D P,JING X R,ZHANG W L,et al.Highly selective synthesis of D-amino acids from readily available L-amino acids by a one-pot biocatalytic stereoinversion cascade[J].RSC Advances,2019,9(51):29927-29935.
[9] MOLLA G,MELIS R,POLLEGIONI L.Breaking the mirror:L-Amino acid deaminase,a novel stereoselective biocatalyst[J].Biotechnology Advances,2017,35(6):657-668.
[10] ANDREADELI A,PLATIS D,TISHKOV V,et al.Structure-guided alteration of coenzyme specificity of formate dehydrogenase by saturation mutagenesis to enable efficient utilization of NADP+[J].The FEBS Journal,2008,275(15):3859-3869.
[11] WU W H,ZHU D M,HUA L.Site-saturation mutagenesis of formate dehydrogenase from Candida bodinii creating effective NADP+-dependent FDH enzymes[J].Journal of Molecular Catalysis B:Enzymatic,2009,61(3-4):157-161.
[12] 倪敏君.酿酒酵母甲酸脱氢酶及其辅酶专一性突变体在大肠杆菌中的高表达,纯化及性质研究[D].上海:华东理工大学,2010.
NI M J.Expression,purification,and characterization of formate dehydrogenase from Saccharomyces cerevisiae and its two coenzyme-specificity mutants[D].Shanghai:East China University of Science and Technology,2010.
[13] SEROV A E,POPOVA A S,FEDORCHUK V V,et al.Engineering of coenzyme specificity of formate dehydrogenase from Saccharomyces cerevisiae[J].The Biochemical Journal,2002,367(3):841-847.
[14] GUL-KARAGULER N,SESSIONS R B,CLARKE A R,et al.A single mutation in the NAD-specific formate dehydrogenase from Candida methylica allows the enzyme to use NADP[J].Biotechnology Letters,2001,23(4):283-287.
[15] ÖZGÜN G P,ORDU E B,TÜTÜNCÜ H E,et al.Site saturation mutagenesis applications on Candida methylica formate dehydrogenase[J].Scientifica,2016,2016:4902450.
[16] GALKIN A G,KUTSENKO A S,BAJULINA N P,et al.Site-directed mutagenesis of the essential arginine of the formate dehydrogenase active centre[J].Biochimica et Biophysica Acta,2002,1594(1):136-149.
[17] LAMZIN V S,DAUTER Z,POPOV V O,et al.High resolution structures of holo and apo formate dehydrogenase[J].Journal of Molecular Biology,1994,236(3):759-785.
[18] KATO N,SAHM H,WAGNER F.Steady-state kinetics of formaldehyde dehydrogenase and formate dehydrogenase from a methanol-utilizing yeast,Candida boidinii[J].Biochimica et Biophysica Acta (BBA)-Enzymology,1979,566(1):12-20.
[19] 程峰,魏澜,王成娇,等.甲酸脱氢酶及其在手性生物制造中的应用[J].生物工程学报,2022,38(2):632-649.
CHENG F,WEI L,WANG C J,et al.Formate dehydrogenase and its application in biomanufacturing of chiral chemicals[J].Chinese Journal of Biotechnology,2022,38(2):632-649.
[20] LINDNER S N,RAMIREZ L C,KRÜSEMANN J L,et al.NADPH-auxotrophic E.coli:A sensor strain for testing in vivo regeneration of NADPH[J].ACS Synthetic Biology,2018,7(12):2742-2749.