红景天苷是传统中药红景天(Rhodiola rosea L.)的主要药理成分,具有增强免疫力、抗氧化、抗肿瘤、美白和抗衰老等多重生理功效,广泛应用于医药、食品和化妆品等领域,是一种极具应用与开发价值的高品质天然产物[1]。植物提取是目前红景天苷生产的主要方法,但红景天资源日益稀缺,并且存在生长周期长、含量低、提取工艺复杂等固有缺陷。虽然研究者后来开发了人工种植、组织培养和细胞悬浮培养等技术,在一定程度上缓解了对红景天资源的依赖,但仍未解决生长周期长和产量低的问题[2-3]。随着红景天苷药理机制和生理功效研究不断深入,市场需求与日俱增,红景天苷的生产面临极大挑战。
基于合成生物学技术构建工程菌发酵生产天然产物,是替代传统植物提取法,实现天然产物绿色、可持续生产的重要方法。目前,已有研究者通过改造模式微生物大肠杆菌和酿酒酵母,实现了红景天苷从头合成(图1)[4-5]。CHUNG等[4]和BAI等[5]通过优选合成途径关键酶[如醇脱氢酶和尿苷二磷酸(uridine diphosphate, UDP)-葡萄糖糖基转移酶]、阻断竞争途径(如pheA、feaB)、解除反馈抑制(如敲除tyrR)等策略改造大肠杆菌,可产红景天苷288、56.9 mg/L。基于相似的策略,JIANG等[6]改造酿酒酵母,摇瓶发酵,红景天苷产量达到0.73 g/L。GUO等[7]在酿酒酵母中重构中心代谢途径,5 L发酵罐分批补料发酵可产红景天苷1.82 g/L。LIU等[8]通过模块工程改造酿酒酵母,5 L发酵罐分批补料发酵,红景天苷产量达到26.55 g/L,这是目前微生物合成红景天苷的最高水平。LIU等[9]将两株改造的营养缺陷型大肠杆菌进行共培养,5 L发酵罐分批补料发酵可产红景天苷6.03 g/L。LIU等[10]在酪醇高产菌的基础上,通过增加外源基因UGT85A1拷贝数,5 L发酵罐分批补料发酵可产红景天苷9.48 g/L,这是目前大肠杆菌合成红景天苷的最高水平。

图1 大肠杆菌中重构的人工红景天苷合成途径
Fig.1 Artificial salidroside synthesis pathways constructed in E.coli
注:红色为外源基因,黑色为大肠杆菌内源基因,绿色为红景天苷合成途径
虽然目前红景天苷合成水平得到大幅度提升,但与同一代谢途径中酪氨酸(55 g/L)[11]的产量仍差距较大。同时,作者在分析文献时发现,红景天苷发酵菌体密度普遍偏低,并伴随高浓度中间产物酪醇残留,如GUO等[7]利用酿酒酵母发酵红景天苷,5 L发酵罐分批补料发酵,菌体密度仅为23.24 g DCW/L,红景天苷1.82 g/L,而酪醇却高达8.48 g/L;LIU等[9]和LIU等[10]利用大肠杆菌发酵红景天苷,5 L发酵罐分批补料发酵OD600值均不超过40,LIU等[10]还发现红景天苷工程菌OD600值较野生型大幅度下降。并且,CASADEY等[12]和本研究均发现酪醇具有良好的抑菌功能,GONZ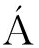 LEZ等[13]亦证实酪醇抑制细菌和酵母生长,影响细胞形态和生理状态。此外,由于大肠杆菌缺乏对外源途径的精确调控,因而无法充分发挥外源途径的生物学功能,导致产物合成受阻。因此,酪醇对菌体的生长抑制和外源途径缺乏调控是限制红景天苷产量进一步提升的瓶颈。群体感应(quorum-sensing, QS)是一种自诱导系统,可根据细胞密度自动调控基因的表达,可较好地平衡细胞生长和产物合成,目前已成功提升大肠杆菌4-羟基苯乙酸[14]、肌醇和葡糖二酸[15]产量。因此,本研究以L值-多巴高产菌大肠杆菌(Escherichia coli)DOPA 30N[16]为底盘细胞,通过筛选合成途径关键酶、优化底盘细胞和群体感应动态调控等策略,缓解中间产物酪醇的毒性作用,平衡产物合成与细胞生长,进而提高红景天苷合成水平。研究结果将有利于改变目前红景天苷生产困境,并为其他糖苷类天然产物的微生物合成提供示范和借鉴。
LEZ等[13]亦证实酪醇抑制细菌和酵母生长,影响细胞形态和生理状态。此外,由于大肠杆菌缺乏对外源途径的精确调控,因而无法充分发挥外源途径的生物学功能,导致产物合成受阻。因此,酪醇对菌体的生长抑制和外源途径缺乏调控是限制红景天苷产量进一步提升的瓶颈。群体感应(quorum-sensing, QS)是一种自诱导系统,可根据细胞密度自动调控基因的表达,可较好地平衡细胞生长和产物合成,目前已成功提升大肠杆菌4-羟基苯乙酸[14]、肌醇和葡糖二酸[15]产量。因此,本研究以L值-多巴高产菌大肠杆菌(Escherichia coli)DOPA 30N[16]为底盘细胞,通过筛选合成途径关键酶、优化底盘细胞和群体感应动态调控等策略,缓解中间产物酪醇的毒性作用,平衡产物合成与细胞生长,进而提高红景天苷合成水平。研究结果将有利于改变目前红景天苷生产困境,并为其他糖苷类天然产物的微生物合成提供示范和借鉴。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株、质粒和引物
本研究所用质粒和菌株请见附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.035417,下同),引物请见附表2(https://doi.org/10.13995/j.cnki.11-1802/ts.035417,下同)。
1.1.2 培养基
LB培养基(g/L):胰蛋白胨,10;酵母提取物,5;NaCl,10;pH 7.0~7.2,121 ℃灭菌20 min。
SOC培养基(g/L):胰蛋白胨20,酵母提取物5,葡萄糖3.6,KCl 0.186,NaCl 0.5,MgCl2 0.95,116 ℃灭菌20 min,用于电转化后复苏。
LBGT培养基[16](g/L):胰蛋白胨10,酵母提取物5,NaCl 10,葡萄糖25,微量元素10 mL,pH 7.0~7.2,116 ℃灭菌20 min。微量元素(g/L):FeSO4·7H2O 10,ZnSO4·7H2O 2.2,MnSO4·4H2O 0.58,CuSO4·5H2O 1.0,Na2B4O7·10H2O 0.2,(NH4)6Mo7O24·4H2O 0.1,35% HCl,10 mL,过滤除菌,接种前加入培养基。
补料培养基(g/L):葡萄糖500,MgSO4·7H2O 30,116 ℃灭菌20 min。
1.1.3 试剂
限制性内切酶,赛默飞世尔科技(中国)有限公司;Prime Star Max DNA聚合酶,宝日医生物技术(北京)有限公司;质粒提取和琼脂糖凝胶DNA回收试剂盒,美基生物科技(广州)有限公司;Gibson Assembly试剂盒,NEB(北京)有限公司;葡萄糖测定试剂盒,南京建成生物工程研究所;抗生素,生工生物工程(上海)股份有限公司。L-酪氨酸、L-多巴、酪醇、4-羟基苯乙酸、红景天苷和脱水四环素,百灵威科技(北京)有限公司;色谱级甲醇、三氟乙酸,安谱实验科技股份(上海)有限公司,其余均为国产分析纯试剂。
1.2 仪器与设备
UV-2450紫外-可见分光光度计、LC20-AT高效液相色谱,日本岛津公司; 2 L×4 Minibox5 Intelli-Ferm平行生物反应器,迪必尔生物工程(上海)有限公司。
1.3 实验方法
1.3.1 重组质粒的构建
采用酶切连接法和Gibson组装法构建质粒。重组质粒经菌落PCR、酶切图谱及测序正确后可使用。
酪醇合成途径质粒的构建:分别PCR扩增大肠杆菌(yqhD、yiaY、frmA、yjgB、yahk、adhP、aldB、adhE)和酿酒酵母的醇脱氢酶(alcohol dehydrogenase, ADH)基因(ADH6),插入到pBbB2K-GFP的EcoRⅠ/BamHⅠ间,获得醇脱氢酶系列表达载体。随后PCR扩增ARO10F138L/D218G并去掉终止密码子TAA,采用BglBrick组装技术[17],插入到醇脱氢酶表达载体的EcoR I/Bgl Ⅱ间,使二者通过linker(GSG)2融合表达。
红景天苷合成途径质粒构建:通过Gibson组装法构建。库叶红景天(Rhodiola sachalinensis)来源UGT73B6(GeneBanK号:AY547304)和UGT72B14(GeneBanK号:EU567325),拟兰芥(Arabidopsis thaliana)来源UGT85A1(GeneBanK号:At1 g22400),以及地衣芽孢杆菌(Bacillus licheniformis)来源UGT1(GeneBanK号:AAU40842)均经密码子优化后由金唯智(苏州)生物科技有限公司进行全基因合成。设计引物去掉yahK终止密码子,PCR扩增pBbB2K-ARO10F138L/D218G-L-yahK整个质粒以及UGT73B6、UGT72B14、UGT85A1和UGT1,琼脂糖凝胶纯化PCR产物后,按照Gibson Assembly试剂盒说明书操作,构建ARO10F138L/D218G-L-yahK分别与UGT73B6、UGT72B14、UGT85A1、UGT1融合表达的红景天苷合成途径质粒。
sgRNA表达质粒构建:采用反向PCR构建feaB敲除及pgm、galU启动子置换所需sgRNA表达质粒。
群体感应调控红景天苷合成途径质粒构建:以pBbB2k-ARO10F138L/D218G-L-yahK-L-UGT85A1和pZBK-PesaSIC-PesaRAS为模板,分别PCR扩增ARO10F138L/D218G-L-yahK-L-UGT85A1和群体感应质粒骨架,纯化PCR产物,按照Gibson Assembly试剂盒说明书,构建群体感应调控红景天苷合成途径质粒pZBK-PesaRAS-SL。
1.3.2 基因编辑
feaB基因敲除和P37置换pgm、galU天然启动子,均参照JIANG等[18]报道的方法,采用CRISPR/Cas9技术,通过同源重组的方法进行。
1.3.3 工程菌发酵
种子培养:挑取活化的单菌落接种至含20 mL LB培养基的100 mL摇瓶中,37 ℃、200 r/min培养10 h。
摇瓶发酵:测定种子液OD600值,按照初始OD600=0.1的接种量接种至装有50 mL LBGT培养基的250 mL 摇瓶中,30 ℃、200 r/min培养48 h,发酵结束后测定生物量(OD600值)、酪醇和红景天苷浓度。
发酵罐发酵:采用分批补料发酵的方式,在2 L×4平行发酵罐上执行,装液量1.2 L,以初始OD600值 0.2 左右的接种量接种,通气量1.8 L/min,发酵温度30 ℃,以氨水调节pH值恒定在7.0(死区0.1),搅拌转速400~1 200 r/min,溶氧设定为25%(死区5%)并与转速关联控制,初始葡萄糖质量浓度设置为5 g/L,采用pH-stat补料策略,每隔4 h取样测定OD600值、葡萄糖浓度、酪醇和红景天苷产量。
含有质粒的工程菌,按照要求加入相应浓度的抗生素。需要诱导表达的菌株,OD600值达到2.5时加入脱水四环素(工作质量浓度120 ng/mL)诱导。
1.3.4 酪醇和红景天苷检测
参照作者[14]前期分析4-羟基苯乙酸的方法,利用HPLC检测红景天苷和酪醇。取发酵液2 mL,10 000 r/min离心1 min,上清液0.22 μm滤膜过滤,收集滤液检测。分析条件:LC20-AT HPLC检测系统,Inertsil ODS-SP C18反相柱(GL Inertsil ODS-SP 150 mm×4.6 mm,5 μm),SPD-M20A二极管阵列检测器,流动相A为0.2%三氟乙酸,流动相B为100%甲醇,柱温30 ℃,进样体积10 μL,检测波长222 nm,采用梯度洗脱法检测。分析程序:0~20 min甲醇质量分数由14%梯度上升至45%,随后下降至14%直至30 min结束,流速0.5 mL/min。在此条件下,红景天苷、酪醇、L-酪氨酸、L-多巴和4-羟基苯乙酸可很好地分离开来(图2-a),发酵液中酪醇(图2-b)和红景天苷(图2-c)保留时间与标准品一致。

a- 标准品液相色谱图;b- 酪醇工程菌TR5发酵液液相色谱图;c- 红景天苷工程菌SL3发酵液液相色谱图
图2 HPLC分析红景天苷、酪醇标准品和工程菌发酵液的色谱图
Fig.2 HPLC analysis of standard tyrosol, standard salidroside and fermentation broth of engineered E.coli
1.3.5 酪醇对底盘细胞生长的影响
挑取活化的SLP单菌落,接种至含20 mL LB培养基的100 mL摇瓶中,37 ℃、200 r/min培养10 h,以初始OD600=0.1的接种量,分别接种至酪醇质量浓度0、2、4、6、8、10、12、14、16、18、20 g/L的5 mL LB试管中,培养12 h后测量OD600值,考察不同浓度酪醇对底盘细胞SLP生长的影响。
1.3.6 数据统计和分析
所有实验均设置3次重复,数据以“平均值±标准差”的形式呈现,采用SPSS 26.0进行方差分析和t检验,P<0.05定义为具有显著性,P<0.01定义为极显著。
2 结果与分析
2.1 红景天苷合成途径筛选
2.1.1 酪醇合成途径筛选
酪醇与UDP-葡萄糖是合成红景天苷的直接前体物,4-羟基苯乙醛则是合成酪醇的直接前体物。基于不同的4-羟基苯乙醛合成方式,大肠杆菌中构建的人工酪醇合成途径有3条,即苯丙酮酸脱羧酶(如ARO10和IpdC)-醇脱氢酶途径[19-21]、芳香醛合成酶(如AAS)-醇脱氢酶途径[4, 22]和酪氨酸脱羧酶(如TydC、AspC和HisC)-酪胺氧化酶(如TynA和TYO)-醇脱氢酶[23]途径。作者[14]前期对比了3条4-羟基苯乙醛合成途径,发现酿酒酵母来源的苯丙酮酸脱羧酶ARO10效率最高,并通过定向进化获得了催化效率大幅度提高的突变体ARO10F138L/D218G。以此为基础,将其与不同来源的醇脱氢酶融合表达,导入L-多巴高产菌DP中,以仅表达ARO10F138L/D218G的质粒为对照,发酵考察其对酪醇合成的影响,结果见图3。
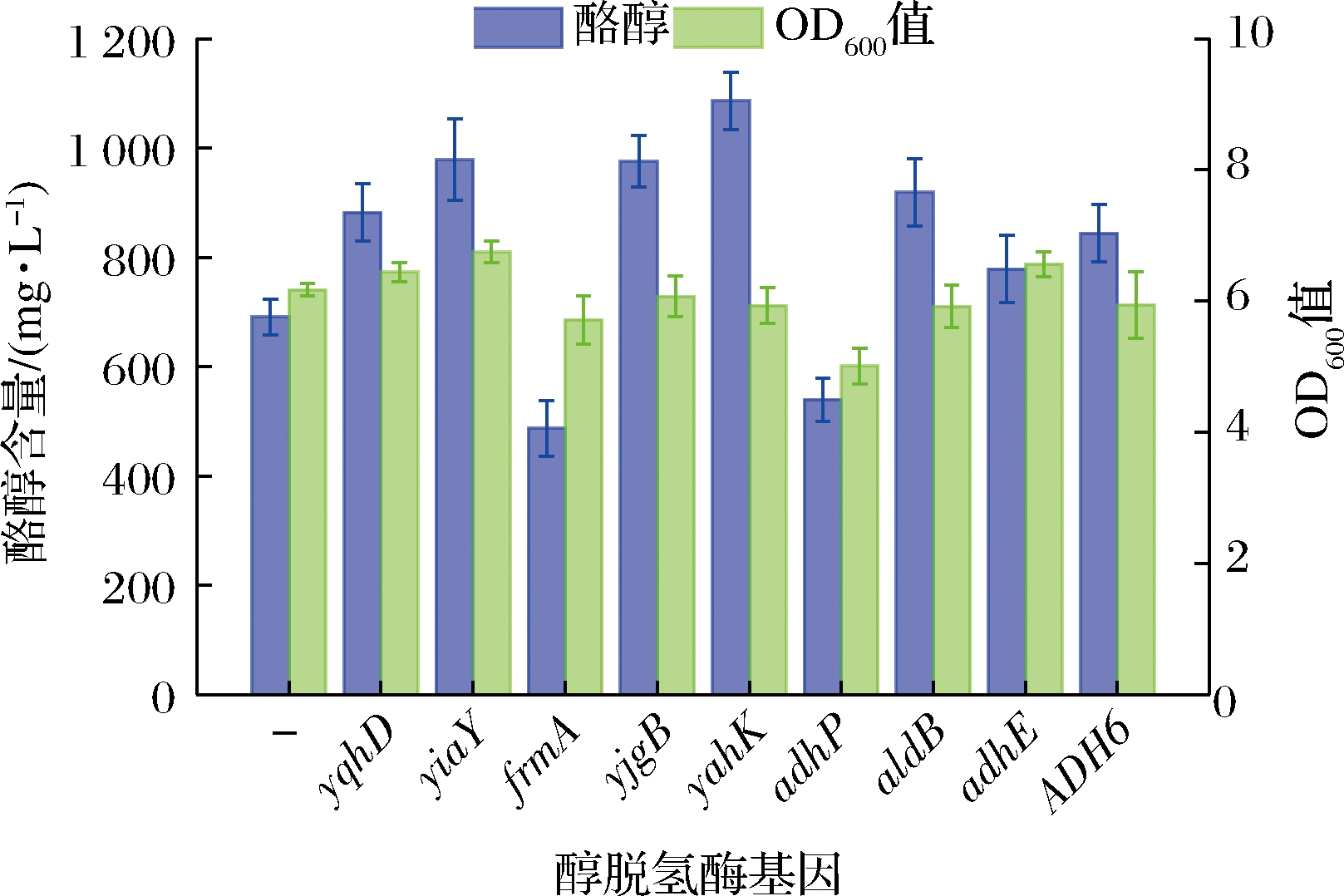
图3 过表达醇脱氢酶基因对酪醇合成的影响
Fig.3 Effects of overexpression of alcohol dehydrogenase genes on the tyrosol production
由图3可知,仅表达ARO10F138L/D218G亦可合成酪醇,说明内源醇脱氢酶可催化4-羟基苯乙醛生成酪醇,LIU等[9]、YANG等[24]和薛宇翔等[25]在酪醇合成研究中也证实了这一结论。过量表达醇脱氢酶,除frmA和adhP之外,可提高酪醇产量12.70%~57.98%,yahK效果最佳,达到(1 086.1±52.3) mg/L。但是,LIU等[9]研究结果显示,大肠杆菌内源醇脱氢酶足以饱和底物,过表达反而导致酪醇产量降低。酪醇与L-多巴均为酪氨酸衍生物,本研究出发菌株DP为L-多巴高产菌,对糖酵解、磷酸戊糖和莽草酸途径进行过多处遗传修饰和改造,并且进化后的突变体ARO10F138L/D218G催化效率大幅度提升,因而提高了代谢通量,使得内源醇脱氢酶不能充分饱和底物。LI等[20]和曾娇娇等[22]在大肠杆菌酪醇合成研究中发现,部分醇脱氢酶基因过表达后会导致酪醇产量降低,本研究中过表达frmA和adhP亦导致酪醇产量下降,其原因尚需设计实验进一步研究。
2.1.2 红景天苷合成途径筛选
尿苷二磷酸葡萄糖糖基转移酶(uridine diphosphate glucosyltransferase, UGT)是红景天苷合成的关键酶。研究显示,库叶红景天来源的UGT73B6[26]和UGT72B14[27],拟兰芥来源的UGT85A1[4,9-10]和地衣芽孢杆菌来源的UGT1[28]均可催化酪醇形成红景天苷,因此我们将上述4个UGT与最优酪醇合成途径ARO10F138L/D218G-L-yahK融合表达,考察其对红景天苷合成的影响,结果见图4。

图4 过表达UGT基因对红景天苷合成的影响
Fig.4 Effects of overexpression of UGT genes on the salidroside production
由图4可知,4个UGT均可催化酪醇生成红景天苷,UGT85A1催化效率最高,可产红景天苷(605.8±16.8) mg/L。YU等[27]通过大肠杆菌异源表达UGT72B14和UGT73B6,并进行了纯化和表征,结果显示UGT72B14催化效率较UGT73B6高出170%。FAN等[28]体外催化实验结果显示,UGT1催化效率优于UGT73B6。LIU等[9]研究发现,在大肠杆菌中UGT85A1红景天苷产量远高于UGT1。CHUNG等[4]比较了12个拟兰芥来源的UGT,发现UGT85A1红景天苷合成效率最高。本研究系统比较了4个UGT在大肠杆菌中的红景天苷合成效率,发现UGT85A1最高,因此后续实验采用UGT85A1。
2.2 优化底盘细胞提高红景天苷产量
UDP-葡萄糖和酪醇是合成红景天苷的直接前体物,二者充足和均衡的供给是红景天苷高效合成的关键。由图1分析可知,敲除feaB可阻断4-羟基苯乙酸合成途径,使代谢流转向酪醇的合成;强化表达pgm和galU有利于提高UDP-葡萄糖的供给。为此,敲除DP的feaB基因,以P37启动子置换pgm和galU天然启动子PseqA和PgalU,同时以高翻译效率UTR序列“TTTCGGAATTAAGGAGGTAATAAAT”置换其天然5′-UTR,以强化表达pgm和galU,获得菌株SLP。将最优红景天苷合成途径质粒pBbB2K-ARO10F138L/D218G-L-yahK-L-UGT85A1引入至SLP,获得菌株SLP3,发酵考察其对红景天苷合成的影响,结果见图5。
图5 优化底盘细胞对红景天苷合成的影响
Fig.5 Effect of engineering of chassis cell on the production of salidroside
注:*表示显著(P<0.05);**表示极显著(P<0.05)(下同)
由图5可知,底盘细胞优化后,红景天苷产量和酪醇产量均极显著提高(P<0.01),达到(1 156.3±44.2) mg/L,较对照SL3提高95.03%,说明敲除feaB,强化表达pgm、galU提高前体物供给的策略可有效地提高红景天苷产量。敲除feaB,可阻断4-羟基苯乙酸竞争途径,促进前体物酪醇的合成,这在CHUNG等[4]、薛宇翔等[25]和YANG等[29]的研究也证实了这一结论。同时,发酵液中酪醇比例亦极显著下降(P<0.01),由53.19%降低至42.19%,说明强化表达pgm、galU增大了UDP-葡萄糖的供给,将酪醇转化为红景天苷。但在SLP3发酵液中,仍有(842.4±22.7) mg/L酪醇残留,且OD600值亦低于正常水平,可能是酪醇对底盘细胞的毒性作用,代谢产物的积累以及质粒高表达造成的代谢负担,导致菌体生长受到抑制,进而影响红景天苷的合成。
2.3 群体感应动态调控对红景天苷合成的影响
2.3.1 酪醇对底盘细胞生长的影响
CASADEY等[12]发现酪醇具有良好的抑菌功能,GONZ LEZ等[13]发现酪醇可影响细菌和酵母的细胞形态和生理状态。此外,在大肠杆菌和酿酒酵母红景天苷合成研究中,均发现菌体发酵密度低于正常水平[6-7,9-10]。为此,考察了酪醇对底盘细胞SLP的影响,结果见图6。
LEZ等[13]发现酪醇可影响细菌和酵母的细胞形态和生理状态。此外,在大肠杆菌和酿酒酵母红景天苷合成研究中,均发现菌体发酵密度低于正常水平[6-7,9-10]。为此,考察了酪醇对底盘细胞SLP的影响,结果见图6。
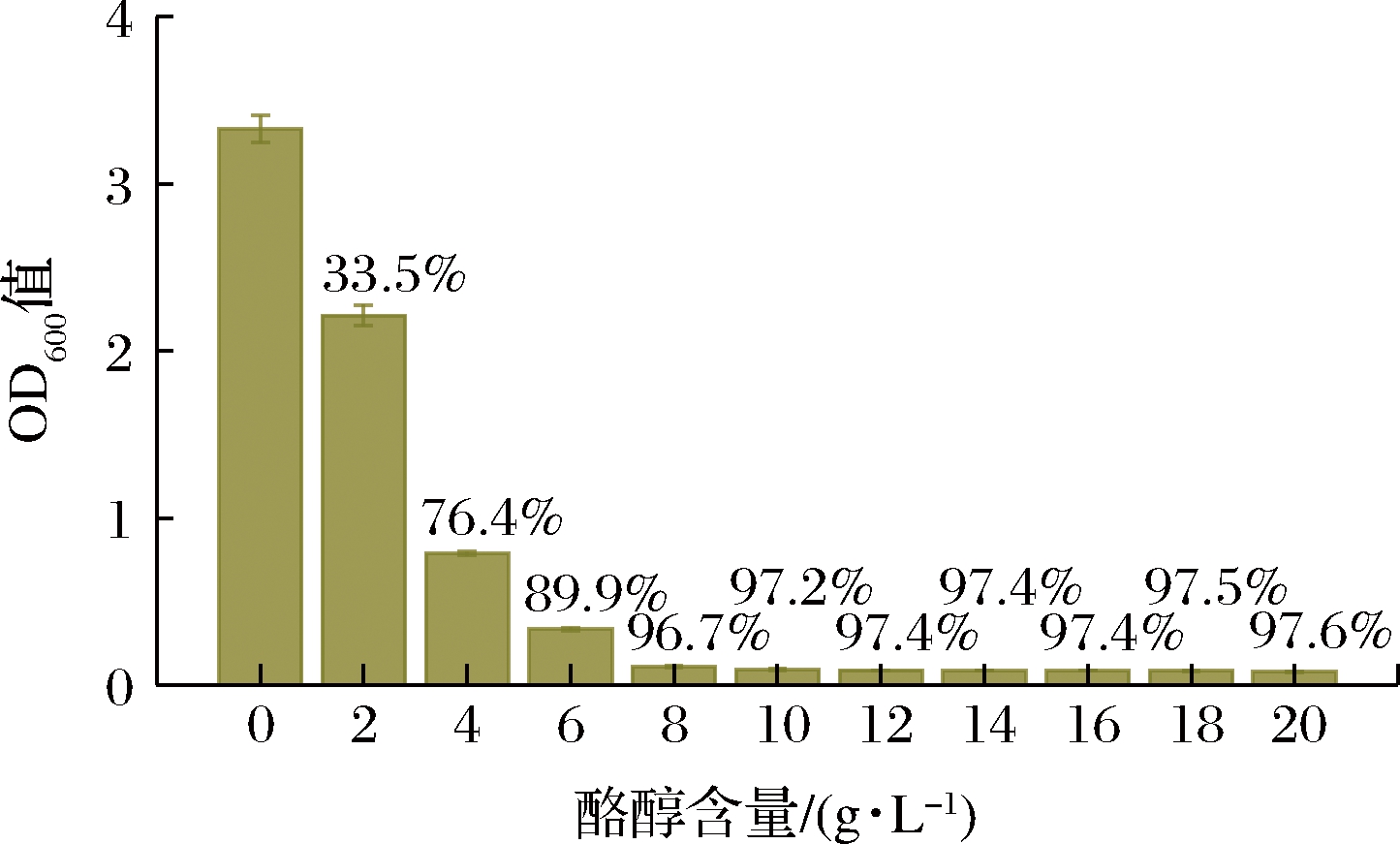
图6 酪醇对底盘细胞生长的影响
Fig.6 Effect of tyrosol on the growth of chassis cell
由图6可知,酪醇强烈抑制底盘细胞SLP生长,2 g/L 抑制率达33.5%,而8 g/L则几乎完全抑制菌体生长。此外,全细胞生物催化的酪醇产量和底物转化率均远高于从头合成[28-29],这也间接证实了酪醇对细胞的毒性作用。因此,减少中间产物酪醇积累,解除其毒性作用,是进一步提升红景天苷产量的关键。
2.3.2 群体感应动态调控对红景天苷合成的影响
群体感应是一种细胞与细胞间的通讯系统,可依据细胞密度自动调控基因表达。作者前期构建了一个兼具激活和抑制功能的群体感应质粒pZBK-PesaSIC-PesaRAS(图7-a),可以同时调控竞争途径和合成途径[14]。以群体感应激活启动子PesaR控制红景天苷合成途径ARO10F138L/D218G-L-yahK-L-UGT85A1,可动态激活合成途径的表达。早期细胞密度低时,合成途径的表达受到限制,促进细胞生长,随着细胞密度的增大,逐步增强合成途径的表达,以此实现群体感应动态调控,从而减轻中间产物酪醇对细胞的毒性作用,缓解红景天苷积累和质粒高表达造成的代谢负担,平衡细胞生长与产物合成。将红景天苷群体感应调控质粒pZBK-PesaRAS-SL(图7-b)引入底盘细胞SLP,获得工程菌SLPQ3,发酵考察其对红景天苷合成的影响。图7-c发酵结果显示,SLPQ3的红景天苷产量和OD600值较对照SLP3极显著提高(P<0.01),红景天苷产量达到(1 484.6±21.4) mg/L,OD600值达到8.18±0.23,较静态诱导表达工程菌SLP3分别提高36.3%和31.6%。此外,发酵液酪醇比例亦极显著下降(P<0.01),由44.4%下降至30.3%,说明群体感应动态在一定程度上可降低酪醇积累,减轻毒性作用,缓解生长抑制,进而提高红景天苷产量。作者在前期研究中,通过群体感应系统动态调控4-羟基苯乙酸合成途径,使其产量提高了46.4%[14]。这证实群体感应系统作为一种自诱导系统,可以减轻产物或中间产物对底盘细胞的毒性作用,较好的平衡生长和产物合成,这对高毒性化学品和其他糖苷类天然产物的生物合成具有广泛的借鉴和应用价值。

a-群体感应调控质粒;b-群体感应调控红景天苷合成途径质粒;c-群体感应调控红景天苷合成途径发酵结果
图7 群体感应动态调控对红景天苷合成的影响
Fig.7 Effect of dynamic quorum-sensing control of salidroside synthesis pathway on the production of salidroside
注:pZBK-PesaSIC-PesaRAS工作机制。在没有esaI合成的群体感应信号分子AHL存在时,esaRI70V编码的转录因子EsaRI70V可以与启动子PesaS结合而激活下游基因(MCS1位点)的转录,同样EsaRI70V可以与启动子PesaR结合抑制下游基因(MCS2位点)的表达。随着细胞生长,开始积累信号分子AHL,AHL与转录因子EsaRI70V结合,导致EsaRI70V与启动子PesaS解离,抑制其下游基因(MCS1位点)的表达,同时使EsaRI70V与启动子PesaR解离,激活其下游基因(MCS2位点)的表达。由于群体感应信号分子AHL数量与细胞密度相关,因此群体感应装置的激活和抑制均随细胞密度呈动态变化,细胞密度大,其激活和抑制越强
2.4 2 L发酵罐发酵生产红景天苷
在群体感应激活系统中,基因表达强度随细胞密度的增大而增加,为进一步验证群体感应调控工程菌SLPQ3的红景天苷生产潜力,在2 L平行生物反应器系统中同时对SLP3和SLPQ3进行发酵。图8发酵结果显示,群体感应动态调控工程菌SLPQ3最大OD600值达到46.74,较对照SLP3提高26.6%(P<0.01);红景天苷产量达到4.38 g/L,较SLP3提高48.2%(P<0.01);此外,发酵液中酪醇比例由33.67%降至17.86%(P<0.01),展现了良好的生产潜力。
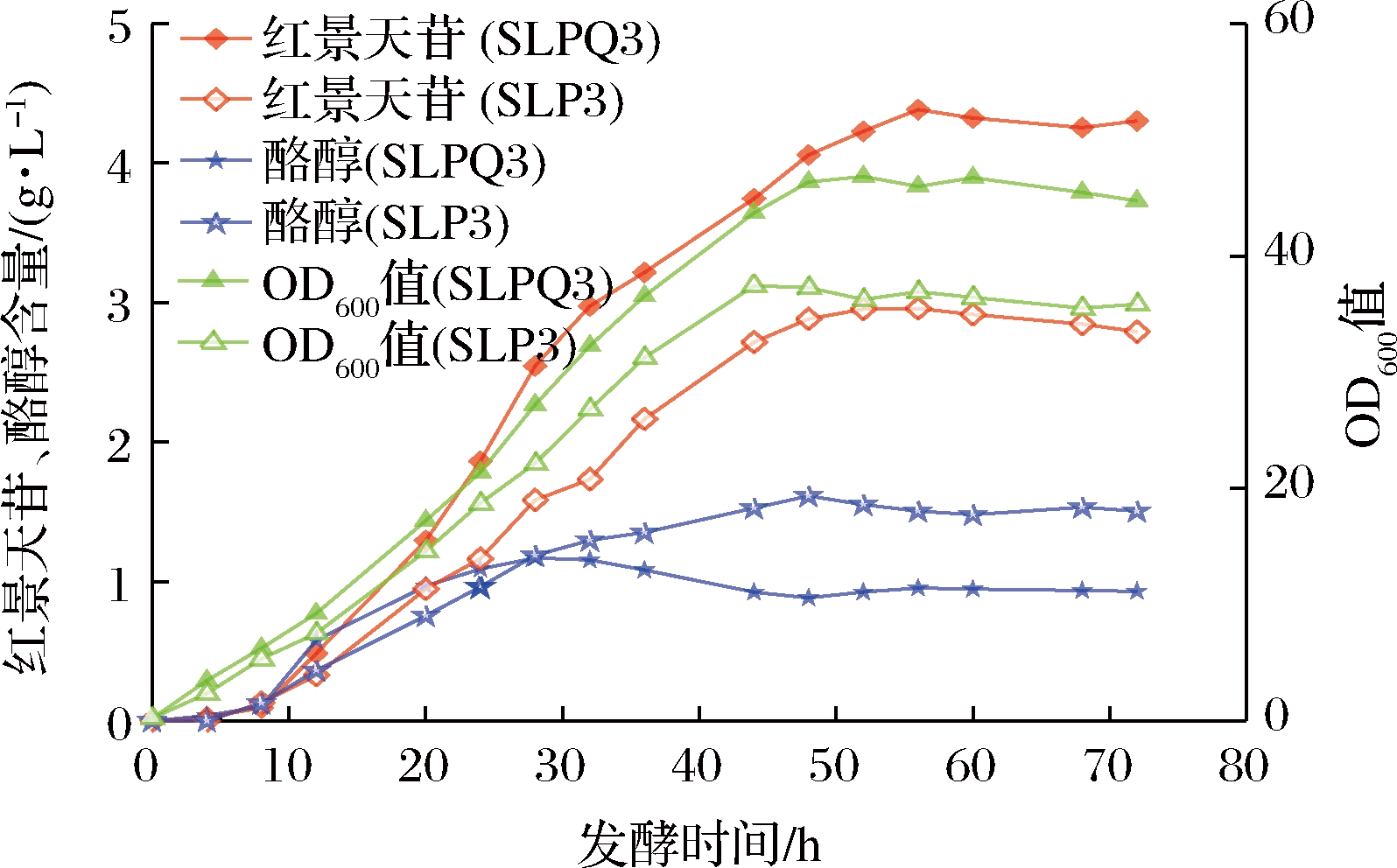
图8 群体感应调控菌株SLPQ3分批补料发酵结果
Fig.8 Fed-batch fermentation of strain SLPQ3 with dynamic quorum-sensing control of salidroside synthesis pathway
3 讨论
红景天苷是一种高品质天然产物,极具应用与开发价值。本研究在前期研究获得的苯丙酮酸脱羧酶突变体ARO10F138L/D218G基础上,通过筛选醇脱氢酶和UDP-葡萄糖糖基转移酶,红景天苷产量达到(605.8±16.8) mg/L;通过敲除竞争途径基因feaB,强化表达UDP-葡萄糖合成的关键基因pgm和galU,红景天苷产量提高95.03%,达到(1 156.3±44.2) mg/L。采用群体感应激活系统动态调控红景天苷合成途径,有效地减轻了酪醇的毒性作用,缓解了生长抑制,摇瓶发酵OD600值达到8.18±0.23,红景天苷产量达(1 484.6±21.4) mg/L,较对照SL3分别提升36.3%和32.6%。2 L发酵罐水平发酵,红景天苷产量达到4.38 g/L,OD600值达到46.74,分别较静态诱导表达提高48.2%和26.6%,展现了良好的生产能力。
本研究通过不同的策略逐步提高了红景天苷产量,但仍有较高浓度中间产物酪醇残留,且菌体发酵密度也未达到正常水平,这说明群体感应动态调控虽可减轻酪醇毒性作用,缓解生长抑制,但并不能达到完全消除的目的。适应性进化可在不需要任何遗传调控信息的条件下,快速提高细胞的耐受性[30],因此后续可通过适应性进化提高底盘细胞的酪醇耐受性。此外,还可对外源酶UGT85A1定向进化提高酶活力,将积累的酪醇转化为红景天苷,也可通过可调控基因间隔区(TIGR)、SpyCatcher-SpyTag等底物通道平衡基因表达,减少中间产物酪醇积累。红景天苷合成需要大量NADPH和ATP,目前已有大量辅因子改造提高产物合成效率的案例[31-32],因此可考虑重构NADPH合成途径或CRISPRi抑制NADPH和ATP消耗相关基因,提高NADPH和ATP供给,促进红景天苷合成。在本研究中,红景天苷摇瓶发酵水平为(1 484.6±21.4) mg/L,与LIU等[9]大肠杆菌(1 072.46 mg/L)以及LIU等[8]酿酒酵母(1 575.45 mg/L)的摇瓶发酵水平相近,但本研究发酵罐发酵水平仅为4.38 g/L,与他们(6.03 g/L)[9]和(26.55 g/L)[8]的发酵罐发酵水平存在较大差距,因此后续可通过改变发酵策略和优化发酵条件进一步释放菌株生产潜力。
[1] ZHANG X M, XIE L, LONG J Y, et al.Salidroside:A review of its recent advances in synthetic pathways and pharmacological properties[J].Chemico-Biological Interactions, 2021, 339:109268.
[2] LI Y, SHAO C H, PARK S Y, et al.Production of salidroside and polysaccharides in Rhodiola sachalinensis using airlift bioreactor systems[J].Acta Physiologiae Plantarum, 2014, 36(11):2 975-2 983.
[3] KAPOOR S, SHARMA A, BHARDWAJ P, et al.Enhanced production of phenolic compounds in compact callus aggregate suspension cultures of Rhodiola imbricata edgew[J].Applied Biochemistry and Biotechnology, 2019, 187(3):817-837.
[4] CHUNG D, KIM S Y, AHN J H.Production of three phenylethanoids, tyrosol, hydroxytyrosol, and salidroside, using plant genes expressing in Escherichia coli[J].Scientific Reports, 2017, 7:2578.
[5] BAI Y F, BI H P, ZHUANG Y B, et al.Production of salidroside in metabolically engineered Escherichia coli[J].Scientific Reports, 2014, 4:6640.
[6] JIANG J J, YIN H, WANG S, et al.Metabolic engineering of Saccharomyces cerevisiae for high-level production of salidroside from glucose[J].Journal of Agricultural and Food Chemistry, 2018, 66(17):4 431-4 438.
[7] GUO W, HUANG Q L, FENG Y H, et al.Rewiring central carbon metabolism for tyrosol and salidroside production in Saccharomyces cerevisiae[J].Biotechnology and Bioengineering, 2020, 117(8):2 410-2 419.
[8] LIU H Y, TIAN Y J, ZHOU Y, et al.Multi-modular engineering of Saccharomyces cerevisiae for high-titre production of tyrosol and salidroside[J].Microbial Biotechnology, 2020, 14(6):2 605-2 616.
[9] LIU X, LI X B, JIANG J L, et al.Convergent engineering of syntrophic Escherichia coli coculture for efficient production of glycosides[J].Metabolic Engineering, 2018, 47:243-253.
[10] LIU S, XIA Y Y, YANG H Q, et al.Rational chromosome engineering of Escherichia coli for overproduction of salidroside[J].Biochemical Engineering Journal, 2022, 184:108474.
[11] PATNAIK R, ZOLANDZ R R,GREEN D A, et al.L-tyrosine production by recombinant Escherichia coli:Fermentation optimization and recovery[J].Biotechnology and Bioengineering, 2008, 99(4):741-752.
[12] CASADEY R, CHALLIER C, ALTAMIRANO M, et al.Antioxidant and antimicrobial properties of tyrosol and derivative-compounds in the presence of vitamin B2.Assays of synergistic antioxidant effect with commercial food additives[J].Food Chemistry, 2021, 335:127576.
[13] GONZ LEZ B,V
LEZ B,V ZQUEZ J,CULLEN P J, et al.Aromatic amino acid-derived compounds induce morphological changes and modulate the cell growth of wine yeast species[J].Frontiers in Microbiology, 2018, 9:670.
ZQUEZ J,CULLEN P J, et al.Aromatic amino acid-derived compounds induce morphological changes and modulate the cell growth of wine yeast species[J].Frontiers in Microbiology, 2018, 9:670.
[14] SHEN Y P, FONG L S, YAN Z B, et al.Combining directed evolution of pathway enzymes and dynamic pathway regulation using a quorum-sensing circuit to improve the production of 4-hydroxyphenylacetic acid in Escherichia coli[J].Biotechnology for Biofuels, 2019, 12(1):94.
[15] GUPTA A, REIZMAN I M B, REISCH C R, et al.Dynamic regulation of metabolic flux in engineered bacteria using a pathway-independent quorum-sensing circuit[J].Nature Biotechnology, 2017, 35(3):273-279.
[16] WEI T, CHENG B Y, LIU J Z.Genome engineering Escherichia coli for L-DOPA overproduction from glucose[J].Scientific Reports, 2016, 6:30080.
[17] LEE T S, KRUPA R A, ZHANG F Z, et al.BglBrick vectors and datasheets:A synthetic biology platform for gene expression[J].Journal of Biological Engineering, 2011, 5:12.
[18] JIANG Y, CHEN B, DUAN C L, et al.Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system[J].Applied and Environmental Microbiology, 2015, 81(7):2 506-2 514.
[19] KOMA D, YAMANAKA H, MORIYOSHI K, et al.Production of aromatic compounds by metabolically engineered Escherichia coli with an expanded shikimate pathway[J].Applied and Environmental Microbiology, 2012, 78(17):6 203-6 216.
[20] LI X L, CHEN Z Y, WU Y F, et al.Establishing an artificial pathway for efficient biosynthesis of hydroxytyrosol[J].ACS Synthetic Biology, 2018, 7(2):647-654.
[21] XU W, YANG C, XIA Y Y, et al.High-level production of tyrosol with noninduced recombinant Escherichia coli by metabolic engineering[J].Journal of Agricultural and Food Chemistry, 2020, 68(16):4 616-4 623.
[22] 曾娇娇, 余世琴, 周景文.代谢工程改造大肠杆菌增产酪醇[J].食品与发酵工业, 2021, 47(22):8-15.
ZENG J J, YU S Q, ZHOU J W.Metabolic engineering of Escherichia coli for improving tyrosol production[J].Food and Fermentation Industries, 2021, 47(22):8-15.
[23] SATOH Y, TAJIMA K, MUNEKATA M, et al.Engineering of a tyrosol-producing pathway, utilizing simple sugar and the central metabolic tyrosine, in Escherichia coli[J].Journal of Agricultural and Food Chemistry, 2012, 60(4):979-984.
[24] YANG H Q, XUE Y X, YANG C, et al.Modular engineering of tyrosol production in Escherichia coli[J].Journal of Agricultural and Food Chemistry, 2019, 67(14):3 900-3 908.
[25] 薛宇翔, 陈献忠, 杨翠, 等.代谢工程改造大肠杆菌合成酪醇[J].食品与生物技术学报, 2019, 38(10):152-159.
XUE Y X, CHEN X Z, YANG C, et al.Metabolic engineering of Escherichia coli for tyrosol synthesis[J].Journal of Food Science and Biotechnology, 2019, 38(10):152-159.
[26] MA L Q, LIU B Y, GAO D Y, et al.Molecular cloning and overexpression of a novel UDP-glucosyltransferase elevating salidroside levels in Rhodiola sachalinensis[J].Plant Cell Reports, 2007, 26(7):989-999.
[27] YU H S, MA L Q, ZHANG J X, et al.Characterization of glycosyltransferases responsible for salidroside biosynthesis in Rhodiola sachalinensis[J].Phytochemistry, 2011, 72(9):862-870.
[28] FAN B, CHEN T Y, ZHANG S, et al.Mining of efficient microbial UDP-glycosyltransferases by motif evolution cross plant Kingdom for application in biosynthesis of salidroside[J].Scientific Reports, 2017, 7:463.
[29] YANG C, CHEN X Z, CHANG J Z, et al.Reconstruction of tyrosol synthetic pathways in Escherichia coli[J].Chinese Journal of Chemical Engineering, 2018, 26(12):2 615-2 621.
[30] LONG C P, ANTONIEWICZ M R.How adaptive evolution reshapes metabolism to improve fitness:Recent advances and future outlook[J].Current Opinion in Chemical Engineering, 2018, 22:209-215.
[31] CHEN R B, GAO J Q, YU W, et al.Engineering cofactor supply and recycling to drive phenolic acid biosynthesis in yeast[J].Nature Chemical Biology, 2022, 18(5):520-529.
[32] SHEN Y P, LIAO Y L, LU Q, et al.ATP and NADPH engineering of Escherichia coli to improve the production of 4-hydroxyphenylacetic acid using CRISPRi[J].Biotechnology for Biofuels, 2021, 14(1):100.