目前,从各种食物中提取生物活性肽已成为研究的一个热点,尤其是从食物中提取的生物活性肽由于其高组织亲和力和特异性,且无副作用,并有潜力作为药物的天然替代品而受到关注[1]。生物活性肽一般是由2~20个氨基酸分子通过肽键相互连接而成,是蛋白质水解后的片段,分子质量大多数在3 kDa以下,是对机体生命活动有益或者具有一定生物活性的肽类化合物[2]。这些生物活性肽已被证明具有抗糖尿病、降压、抗菌、抗氧化等功效[3-4]。生物活性肽常常隐藏于蛋白质一级结构序列中的一些特定区域内,必须释放它们才能发挥作用。目前,常用于获取生物活性肽的方法包括微生物发酵法、胃肠道消化酶法和蛋白酶水解法等,特别是通过体外消化释放多肽已被证实是一种有效提高食物蛋白生物活性的良好途径[5]。已有研究证实通过模拟体外消化实验获得的多肽与人体蛋白消化过程释放多肽结果更相似[6]。因此,采用该方法可更有效预测功能蛋白及其蛋白肽的生物活性。
传统的生物活性肽筛选方法主要通过分离纯化,结合体内外活性评价进行肽段的确定,整个过程费时、费力且技术难度大。随着科学技术的发展,运用计算机辅助酶法技术,通过在线数据及虚拟筛选手段研究肽的活性,能够快速高效地从中发现和预测具有较高生物活性的肽段,并能够减少工作强度,提高鉴定成功率[7]。先前已有学者通过虚拟筛选,在线预测了多肽的活性、吸收、代谢和毒性等,从鸡蛋、稻米、啤酒等中鉴定出血管紧张素转化酶(angiotensin converting enzyme,ACE)和二肽基肽酶抑制剂-Ⅳ(dipeptidyl peptidases inhibitor-Ⅳ,DPP-Ⅳ)抑制肽[8-10]。因此,本研究采用一系列生物信息学方法,从驼皮胶原蛋白模拟胃肠道消化产物中筛选生物活性肽,为驼皮胶原蛋白生物活性肽筛选提供新思路。
胶原蛋白肽是以胶原蛋白或明胶为原料,在酶的作用下得到的水解产物。与胶原蛋白相比,低分子质量的胶原蛋白肽具有更强的生物活性,如抗氧化、抑菌、降血压、抗炎等特性[11-12]。动物源性农副产品产量巨大,胶原蛋白资源丰富,但利用率不高,深加工工艺亟待优化。对于骆驼产业而言,如何合理利用驼皮得到胶原蛋白及其衍生产品,从而减少废料造成的环境污染和增加骆驼的经济附加值是目前值得关注的问题。但是,目前关于驼皮胶原蛋白方面的研究较少,本课题组前期证实低强度超声波辅助酶法可有效提高胶原蛋白的提取率[13]。因此,本研究以不同方法提取获得的驼皮胶原蛋白为研究对象,通过体外模拟胃肠消化获得更贴近人体真正消化产物的胶原蛋白多肽,并采用计算机模拟法探究其生物活性,以期为功能活性肽的开发及骆驼皮副产物的转化利用提供理论依据。
1 材料与方法
1.1 材料与试剂
5岁膳驼的皮购买于阿拉善右旗当地屠宰场。胃蛋白酶(1∶10 000),北京索莱宝科技有限公司;中性蛋白酶(1398),山东龙元生物工程有限公司;猪胃蛋白酶(P6887-250MG)、猪胰蛋白酶(T0303-1G)、NH4HCO3(生化纯),美国Sigma公司;纯水、乙腈、甲酸、三氟乙酸(tri uoroacetic acid,TFA)(均为质谱纯),美国Fisher Scientific公司。
uoroacetic acid,TFA)(均为质谱纯),美国Fisher Scientific公司。
1.2 仪器与设备
JY 88-IIN超声波细胞粉碎机,宁波新芝生物科技股份有限公司;JJ-2型组织捣碎机,常州赛普实验仪器厂;XK78-2型磁力加热搅拌器,江苏新康医疗器械有限公司;Zetasizer Nano ZS90纳米粒度电位仪,英国马尔文仪器有限公司;Q-Exactive 质谱仪,美国Thermo Fisher公司。
1.3 实验方法
1.3.1 驼皮胶原蛋白提取
根据本实验前期的驼皮胶原蛋白的提取方法[13],具体操作如下:骆驼皮经过脱毛、脱脂、去除杂蛋白等处理后,将0.5 mol/L乙酸和预处理的皮肤组织与质量浓度为40 g/L的胃蛋白酶以1∶20的比例混合,制成匀浆液。称取一定质量的上述匀浆液,采用常规酶法提取[加入胃蛋白酶于0.5 mol/L乙酸溶液,(pepsin soluble collagen,PSC)]/超声波辅助法提取[酶法基础上进行超声波,条件:200 W,20 min,(ultrasound-pepsin soluble collagen,UPSC)],在低温条件搅拌提取48 h后,以8 000 r/min离心20 min,收集上清液,得到驼皮胶原蛋白提取液。经冷冻干燥得到骆驼皮中胶原蛋白,制备胶原蛋白酶解物前在90 ℃热解2 h处理。
1.3.2 模拟体外消化
模拟胃消化道:分别取250 mg PSC和UPSC冻干样品,溶解于25 mL水中。取2 g NaCl,加7 mL浓HCl溶液,用0.2 mol/L NaOH溶液调pH值至3,用水定容至1 L。加入猪胃蛋白酶(0.008 mg/mL),使其充分溶解,得到模拟胃液(simulated gastric fluid,SGF)。取25 mL样品溶液与25 mL SGF混合,在恒温摇床振荡2 h(37 ℃,100 r/min)。每间隔30 min(0、30、60、90、120 min)取样,所取样品调pH值至7终止反应。
模拟肠消化道:取6.8 g KH2PO4于250 mL水中溶解,加入0.2 mol/L NaOH溶液77 mL,溶于500 mL水中,再用0.2 mol/L HCl将溶液pH值调至6.8。加入胆盐(0.2 mg/mL)、胰蛋白酶(0.003 mg/mL)溶于上述溶液,使其充分溶解,得到模拟肠液(simulated intestinal fluid,SIF)。将模拟胃消化2 h后的消化液与SIF等体积混合后,置于37 ℃的恒温摇床振荡继续消化2 h。每间隔30 min(30、60、90、120 min)取样,将所取样品煮沸10 min终止反应。
1.3.3 SDS-PAGE分析
利用SDS-PAGE分析法分析PSC和UPSC。采用质量分数5%浓缩胶和质量分数8%的分离胶体系进行电泳分离,用0.5 mol/L的乙酸将样品配制成0.5 mg/mL的胶原蛋白溶液,上样量为5 μL。分别采用浓缩胶电压80 V、分离胶电压100 V进行电泳。电泳结束后,使用考马斯亮蓝R-250染色胶片,并用乙醇-乙酸脱色液进行脱色,最后使用凝胶成像仪观察分析。
1.3.4 粒径分析
采用激光粒度仪测定样品的粒径大小及分布情况,测定的相关参数:物质折光率为1.59,介质折射率为1.333。
1.3.5 肽段检测
超滤脱盐:样品用Millipore 10 kDa超滤管超滤,得到的多肽混合物用Empore固相萃取小柱C18脱盐,多肽组分真空冷冻干燥后用40 μL体积分数0.1%的TFA复溶,进行LC-MS/MS分析。
色谱条件:色谱柱:Zorbax 300SB-C18 peptide traps,进样量2 μL,流速为250 nL/min。流动相:A为0.1%甲酸溶液,B为0.1%甲酸-乙腈溶液。液相梯度设置如下:0~50 min,B相线性梯度从4%~50%;50~54 min,B相线性梯度从50%~100%;54~60 min,B液维持在100%。该过程以95%的A液进行平衡。
质谱条件:采用正离子源,喷雾电压为3.2 kV;数据采集范围:300~1 800 m/z;质谱扫描模式为Full MS-ddMS2,一级质谱和二级质谱的分辨率分别为70 000(m/z 200处)和17 500(m/z 200处)。
数据库检索参数:Peptide mass tolerance:20 ppm;MS/MS tolerance:0.1 Da;Enzyme=None;Max Missed Cleavages:2;Variable modification:Oxidatio;数据库选用UniProt_camelidae_89471_20201019(数据库包含89 471条序列,下载于2020年10月19日)并结合反库;结果过滤参数:FDR<0.01。
1.3.6 驼源胶原蛋白活性肽的初筛
1.3.6.1 消化产物中生物活性肽的筛选
使用PeptideRanker程序对上述收集的多肽进行生物活性预测,并以预测得分的形式表示其生物活性的相对大小。以0.5作为活性预测的阈值,当多肽得分大于该值时,认为其具有潜在的生物活性[14]。
1.3.6.2 消化产物的性质分析
本研究使用 admetSAR(http://lmmd.ecust.edu.cn/admetsar2/)程序对所选活性肽进行在线评价, 选择人体肠道吸收(human intestinal absorption, HIA)来确定吸收特性,选择血脑屏障(blood brain barrier, BBB)穿透率和细胞色素 P450(CYP 450)和急性口服毒性做为评价指标[15]。
1.3.7 分子对接
从RCSB PDB蛋白质数据库(http://www.rcsb.org/pdb/home/home.do)检索得 ACE 靶点蛋白(PDB ID:1o86)和靶点蛋白DPP-IV(PDB ID:5 J3 J)晶体结构。在对接前去除水分子,加氢,加电荷优化的预处理,选择蛋白分子为受体。利用Chem Draw 软件(Cambridge Soft Co., Boston, MA, U.S.A)绘制多肽分子结构,在对接前进行能量最小化。使用autodock、pymol 软件(De Lano Scientific LLC, San Carlos, CA, USA)进行分子模拟对接研究[16-17]。
1.3.8 数据处理
本实验的数据统计分析使用软件 Origin Pro 8绘图。使用Proteins Plus(https://proteins.plus/)用于展示受体和配体之间的2D作用模型图。
2 结果与分析
2.1 SDS-PAGE分析
为了解PSC和UPSC经模拟胃肠消化后的水解情况,使用SDS-PAGE对不同消化时间PSC和UPSC的水解程度进行表征。根据图1观察可知,二者在模拟胃消化阶段中,消化时长在60 min内,胶原蛋白的主要条带仍然可见,当消化120 min时,β和γ链几乎被完全水解,仅在135 kDa附近的条带较明显。在消化过程中,随着时间的延长,50 kDa以下的蛋白条带逐渐变深,说明α、β和γ链均被水解为较小分子质量的肽。在开始模拟肠消化30 min后,PSC和UPSC在15%的分离胶上已无明显条带。

a-PSC;b-UPSC
图1 PSC及UPSC经模拟胃肠消化后的SDS-PAGE图谱
Fig.1 SDS-PAGE patterns of in vitro gastrointestinal digestion of PSC and UPSC
注:PSC/UPSC-0代表未消化阶段,PSC/UPSC-2代表模拟胃消化阶段,PSC/UPSC-4代表模拟肠消化阶段,(下同);T0表示未消化原始样品,T30表示消化30 min,T60表示消化60 min,T90表示消化90 min,T120表示消化120 min。
2.2 粒径分析
图2显示了在模拟胃肠消化过程中,PSC和UPSC的粒径逐渐变小。在模拟胃消化2 h后,PSC组中大颗粒组分由40~355 μm变为28~200 μm。随后的模拟肠消化阶段,大颗粒组分最终在25~140 μm。UPSC与PSC在模拟消化各阶段具有相似的变化规律,经过模拟肠消化阶段后,二者消化产物的粒径大小相似,但UPSC较PSC的粒径更小。

a-PSC;b-UPSC
图2 UPSC和PSC各消化阶段粒径分布图
Fig.2 Particle size distribution of UPSC and PSC at different digestion stages
2.3 肽段分析
由图3可知,PSC和UPSC组分别鉴定出101和138种多肽。与PSC消化产物相比,经过超声波处理后含有的多肽数量更多,说明超声波可使胶原蛋白结构更加松散,促进水解,使其在消化后可得到更多数量的多肽。由图2所示,PSC和UPSC组经过胃肠消化后多肽的分子质量主要集中在500~2 000 Da,尤其在1 000~1 500 Da数量最多,分别为47和62。此外本研究结果显示,在UPSC组鉴定出<500 Da的多肽数量更多,表明经过超声波处理后,胶原蛋白可释放出大量的短肽。

图3 UPSC和PSC不同分子质量下的肽段数量
Fig.3 The number of peptides under different molecular weights of PSC and UPSC
2.4 消化产物中生物活性肽评价
本研究通过胃蛋白酶和胰蛋白酶消化以及水解后,主要I型胶原蛋白分解释放的肽片段。通过PeptideRanker程序对消化产生的生物活性肽打分,得分大于0.5且低于15个氨基酸的的多肽有56条,其中十肽8条,十一肽11条,十三肽8条,活性肽分子质量主要集中在小于1 500 Da。由表1可知,PSC组包含31条生物活性肽,UPSC组包含47条生物活性肽。该结果表明,超声波处理对于胶原蛋白提供生物活性肽具有良好的潜力。
表1 UPSC和PSC胶原蛋白水解肽活性评价
Table 1 Activity evaluation of collagen hydrolytic peptides from UPSC and PSC
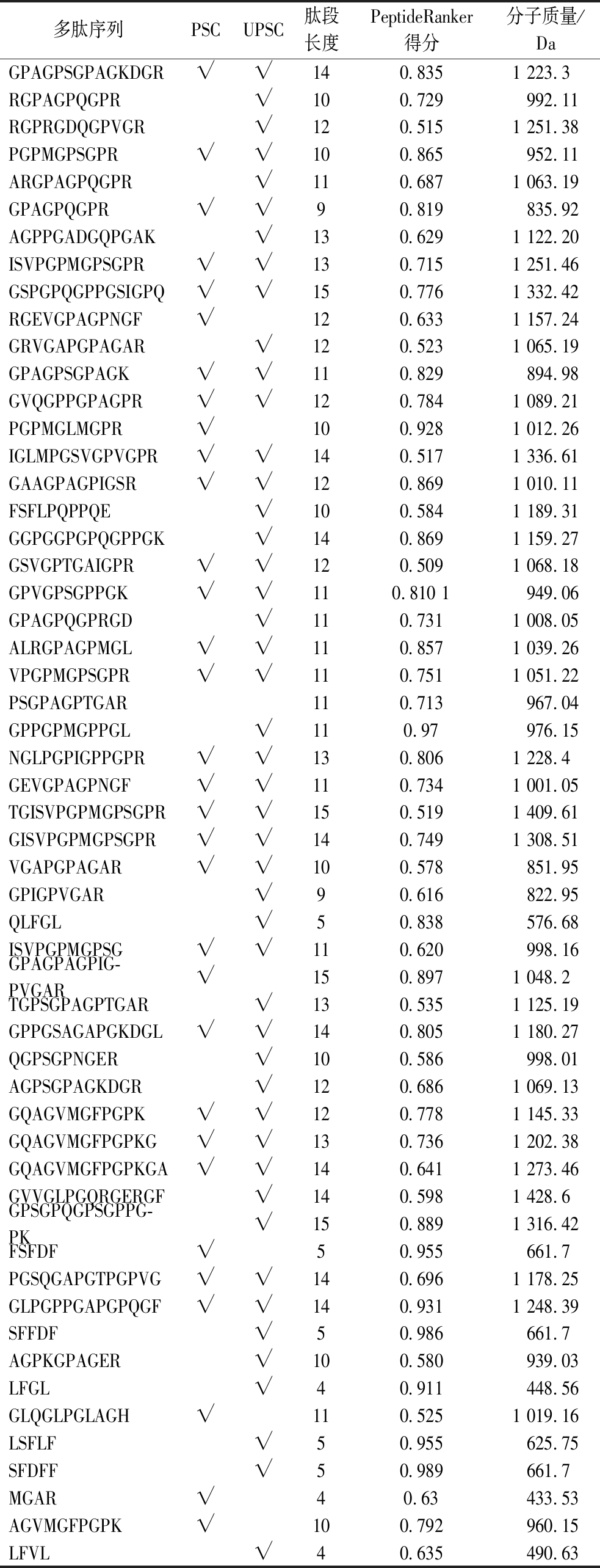
多肽序列PSCUPSC肽段长度PeptideRanker得分分子质量/DaGPAGPSGPAGKDGR√√140.8351 223.3 RGPAGPQGPR√100.729992.11RGPRGDQGPVGR√120.5151 251.38PGPMGPSGPR√√100.865952.11ARGPAGPQGPR√110.6871 063.19GPAGPQGPR√√90.819835.92AGPPGADGQPGAK√130.6291 122.20ISVPGPMGPSGPR√√130.7151 251.46GSPGPQGPPGSIGPQ√√150.7761 332.42RGEVGPAGPNGF√120.6331 157.24GRVGAPGPAGAR√120.5231 065.19GPAGPSGPAGK√√110.829894.98GVQGPPGPAGPR√√120.7841 089.21PGPMGLMGPR√100.9281 012.26IGLMPGSVGPVGPR√√140.5171 336.61GAAGPAGPIGSR√√120.8691 010.11FSFLPQPPQE√100.5841 189.31GGPGGPGPQGPPGK√140.8691 159.27GSVGPTGAIGPR√√120.5091 068.18GPVGPSGPPGK√√110.810 1949.06GPAGPQGPRGD√110.7311 008.05ALRGPAGPMGL√√110.8571 039.26VPGPMGPSGPR√√110.7511 051.22PSGPAGPTGAR110.713967.04GPPGPMGPPGL√110.97976.15NGLPGPIGPPGPR√√130.8061 228.4GEVGPAGPNGF√√110.7341 001.05TGISVPGPMGPSGPR√√150.5191 409.61GISVPGPMGPSGPR√√140.7491 308.51VGAPGPAGAR√√100.578851.95GPIGPVGAR√90.616822.95QLFGL√50.838576.68ISVPGPMGPSG√√110.620998.16GPAGPAGPIG-PVGAR√150.8971 048.2TGPSGPAGPTGAR√130.5351 125.19GPPGSAGAPGKDGL√√140.8051 180.27QGPSGPNGER√100.586998.01AGPSGPAGKDGR√120.6861 069.13GQAGVMGFPGPK√√120.7781 145.33GQAGVMGFPGPKG√√130.7361 202.38GQAGVMGFPGPKGA√√140.6411 273.46GVVGLPGQRGERGF√140.5981 428.6GPSGPQGPSGPPG-PK√150.8891 316.42FSFDF√50.955661.7PGSQGAPGTPGPVG√√140.6961 178.25GLPGPPGAPGPQGF√√140.9311 248.39SFFDF√50.986661.7AGPKGPAGER√100.580939.03LFGL√40.911448.56GLQGLPGLAGH√110.5251 019.16LSFLF√50.955625.75SFDFF√50.989661.7MGAR√40.63433.53AGVMGFPGPK√100.792960.15LFVL√40.635490.63
2.5 肽段的性质分析
本研究采用admetSAR 2.0数据库对筛选的生物活性肽段的吸收、代谢、毒性进行预测。只有通过胃肠道吸收并透过生理屏障后活性肽才能在机体内发挥功能活性从而促进人体健康[18]。据报道口服小分子活性肽主要通过胃肠吸收,活性肽的HIA性有助于预测其通过小肠的吸收率,人体的小肠吸收率≤30%时,HIA值为负数,反之为正数[19]。与其他膜屏障相比,血脑屏障是通过形成通透屏障而严格限制化合物的渗透,许多化合物和靶向药物由于无法在脑组织中达到足够浓度而不能产生期望的治疗作用[20]。如表2所示,本研究通过预测,获得5条既能通过胃肠道吸收也能通过血脑屏障的肽段,其中1条较长肽来源于传统提取法获得,其余肽均来源于超声辅助提取法所获得。
表2 胶原蛋白肽段的性质分析
Table 2 Absorption, metabolism and toxicity characteristics of peptides from
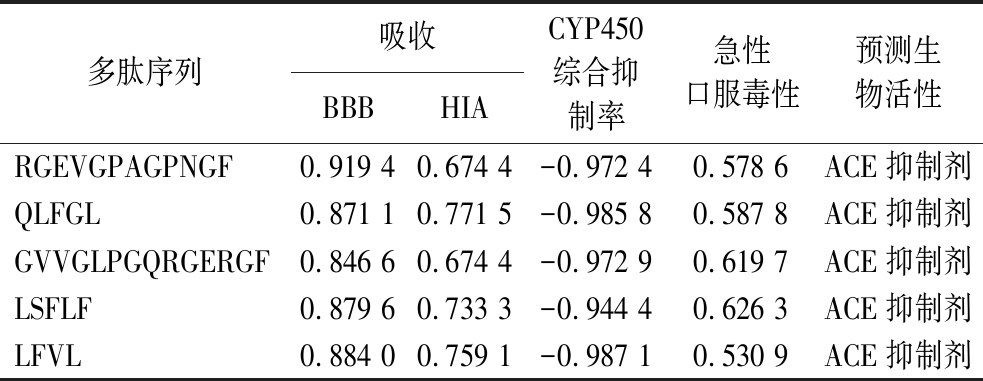
多肽序列吸收BBBHIACYP450综合抑制率急性口服毒性预测生物活性RGEVGPAGPNGF0.919 40.674 4-0.972 40.578 6ACE抑制剂QLFGL0.871 10.771 5-0.985 80.587 8ACE抑制剂GVVGLPGQRGERGF0.846 60.674 4-0.972 90.619 7ACE抑制剂LSFLF0.879 60.733 3-0.944 40.626 3ACE抑制剂LFVL0.884 00.759 1-0.987 10.530 9ACE抑制剂
CYP450是代谢过程中关键的酶,主要分布于肝脏中,少部分分布于肠、肺、肾、脑中,其主要通过参与内源性物质的生物合成和代谢及外源性物质的生物氧化和降解[21]。为了探究肽段的代谢速度,本研究选择CYP450综合抑制率作为指标。预测结果显示上述5条肽段综合抑制率均为负数,表明所选肽段能提高CYP酶系的活性,有效加快肽段在体内的代谢速度。此外,本研究选择急性口服毒性指标分析所选肽段的毒性预测指标,结果表明5条肽段急性口服毒性均为三级毒性,毒性低[22]。
2.6 ACE抑制肽的虚拟筛选
分子对接是研究分子间相互作用,并预测其结合模式和结合力的常用方法。与蛋白酶活性部位的关键氨基酸结合并产生相互作用是生物活性肽发挥蛋白酶抑制活性的前提[23]。为了进一步筛选具有较高抑制ACE抑制活性的肽段,本研究进行分子对接模拟,评价筛选的3条寡肽与ACE结合能力。据报道,自由能越低,多肽与ACE的结合作用力越强;此外,氢键相互作用在稳定对接复合体和酶催化反应中发挥重要作用,氢键的数目直接影响降压活性肽对ACE的亲和力[24]。如表3所示,3条寡肽与降压靶标ACE发生不同程度的结合,具有潜在的ACE抑制活性,其中LSFLF与ACE表现出最强的结合力,且形成的氢键数量最多。
表3 ACE抑制肽的筛选
Table 3 Screening of ACE inhibitory peptides

多肽序列预测结合能/(kcal/mol)相互作用QLFGL-7.72氢键LSFLF-9.36氢键,1π-π键LFVL-7.82氢键
ACE是一种含锌离子的二肽羧基酶,其活性中心由3个疏水腔组,腔中有锌离子。目前的研究表明三肽对ACE的抑制作用主要是由氢键相互作用及配体与活性部位锌离子的相互作用主导[25-26]。本研究虚拟筛选得到的3个寡肽均能嵌入ACE的活性口袋中,均能与ACE活性部位的关键氨基酸残基形成分子间相互作用而产生抑制作用。如图4所示,与ACE的结合中,LSFLF与Glu384A,Ala356A,Asn66A,Ser516A,Tyr62A,Arg124A形成氢键作用,同时可与Trp357形成π-π键作用,使其在ACE的活性部位空腔中更为稳定地结合。


a-3D图;b-2D图
图4 LSFLF与ACE相互作用3D图和2D图
Fig.4 3D and 2D diagram of the interaction between ACE and LSFLF
3 结论
传统酶法提取和超声辅助酶法提取技术均能使胶原蛋白发生水解,但是超声辅助酶法技术对胶原蛋白的降解程度更高。经过模拟胃肠道消化后,超声波处理后活性肽的数量增加,且肽段大部分集中于1 500 Da以下。通过ADMET生物活性预测,发现5条潜在具有ACE抑制作用的肽段。通过分子对接技术进一步虚拟筛选发现,超声辅助酶法获得的肽段LSFLF可能具有较高潜力ACE抑制作用,其主要通过与氨基酸残基形成氢键进行抑制ACE的活性。该研究结果证实超声辅助酶法提取技术能促进驼皮胶原蛋白质降解,可能释放生物活性多肽,该结果可为驼皮深度开发活性肽的高价值利用提供理论参考。
[1] SILVA DO NASCIMENTO E, ANAYA K, DE OLIVEIRA J M C, et al.Identification of bioactive peptides released from in vitro gastrointestinal digestion of yam proteins (Dioscorea cayennensis)[J].Food Research International, 2021, 143:110286.
[2] BHAT Z F, KUMAR S, BHAT H F.Bioactive peptides of animal origin:A review[J].Journal of Food Science and Technology, 2015, 52(9):5377-5392.
[3] HAJFATHALIAN M, GHELICHI S, GARC A-MORENO P J, et al.Peptides:Production, bioactivity, functionality, and applications[J].Critical Reviews in Food Science and Nutrition, 2018, 58(18):3097-3129.
A-MORENO P J, et al.Peptides:Production, bioactivity, functionality, and applications[J].Critical Reviews in Food Science and Nutrition, 2018, 58(18):3097-3129.
[4] WU R B, CHEN L L, LIU D, et al.Preparation of antioxidant peptides from salmon byproducts with bacterial extracellular proteases[J].Marine Drugs, 2017, 15(1):4.
[5] ULUG S K, JAHANDIDEH F, WU J P.Novel technologies for the production of bioactive peptides[J].Trends in Food Science &Technology, 2021, 108:27-39.
[6] LUCAS-GONZ LEZ R, VIUDA-MARTOS M, PÉREZ-ALVAREZ J A, et al.In vitro digestion models suitable for foods:Opportunities for new fields of application and challenges[J].Food Research International, 2018, 107:423-436.
LEZ R, VIUDA-MARTOS M, PÉREZ-ALVAREZ J A, et al.In vitro digestion models suitable for foods:Opportunities for new fields of application and challenges[J].Food Research International, 2018, 107:423-436.
[7] 于志鹏, 薛如阳, 赵文竹, 等.肽组学在食源性活性肽研究中应用的进展[J].食品工业科技, 2016,37(4):382-385.
YU Z P, XUE R Y, ZHAO W Z, et al.Progress on the application of peptidomics methods in food-derived bioactive peptides[J].Science and Technology of Food Industry, 2016, 37(4):382-385.
[8] 于志鹏, 樊玥, 赵文竹, 等.鸡蛋蛋白ACE抑制肽的筛选、鉴定及其作用机制[J].食品科学, 2020, 41(12):129-135.
YU Z P, FAN Y, ZHAO W Z, et al.Identification and mechanism of action of angiotensin-I converting enzyme inhibitory peptides from hen egg proteins[J].Food Science, 2020, 41(12):129-135.
[9] ZHAO W Z, ZHANG D, YU Z P, et al.Novel membrane peptidase inhibitory peptides with activity against angiotensin converting enzyme and dipeptidyl peptidase IV identified from hen eggs[J].Journal of Functional Foods, 2020, 64:103649.
[10] 石嘉怿, 梁富强, 张太, 等.基于生物信息学稻米半胱氨酸蛋白酶抑制剂来源生物活性肽的虚拟筛选及分子对接研究[J].中国粮油学报, 2021, 36(2):1-8.
SHI J Y, LIANG F Q, ZHANG T, et al.In silico identification and molecular docking study of bioactive peptides derived from rice oryzacystatins using integrated bioinformatics approaches[J].Journal of the Chinese Cereals and Oils Association, 2021, 36(2):1-8.
[11] FU Y, THERKILDSEN M, ALUKO R E, et al.Exploration of collagen recovered from animal by-products as a precursor of bioactive peptides:Successes and challenges[J].Critical Reviews in Food Science and Nutrition, 2019, 59(13):2011-2027.
[12] JIN R T, TENG X Y, SHANG J Q, et al.Identification of novel DPP-IV inhibitory peptides from Atlantic salmon (Salmo salar) skin[J].Food Research International, 2020, 133:109161.
[13] 史睿. 超声波辅助提取骆驼皮胶原蛋白及其性质研究[D].呼和浩特:内蒙古农业大学, 2021.
SHI R.Study on ultrasonic-assisted extraction and properties of collagen from camel skin[D].Hohhot:Inner Mongolia Agricultural University, 2021.
[14] TU M L, LIU H X, CHENG S Z, et al.Identification and characterization of a novel casein anticoagulant peptide derived from in vivo digestion[J].Food &function, 2019, 10(5):2552-2559.
[15] 樊玥. 基于分子对接的海水鱼ACE抑制三肽鉴定及作用机制研究[D].锦州:渤海大学, 2019.
FAN Y.Identification and action mechanism of ACE inhibitory tripeptides from marine fish based on molecular docking[D].Jinzhou:Bohai University, 2019.
[16] 林凯. 基于结构信息学定向水解曲拉酪蛋白及新型降压肽的研究[D].哈尔滨:哈尔滨工业大学, 2020.
LIN K.Screening of novel antihypertensive peptides by directed hydrolysis in qula casein based on the structural informatics[D].Harbin:Harbin Institute of Technology, 2020.
[17] 张欣格. 绵羊乳清蛋白α-glucosidase抑制肽与DPP-Ⅳ抑制肽的筛选及抑制机理研究[D].哈尔滨:哈尔滨工业大学, 2021.
ZHANG X G.Isolation and inhibition and mechanism of peptides against α-glucosidase and DPP-Ⅳ from sheep whey protein[D].Harbin:Harbin Institute of Technology, 2021.
[18] LUNDQUIST P, ARTURSSON P.Oral absorption of peptides and nanoparticles across the human intestine:Opportunities, limitations and studies in human tissues[J].Advanced Drug Delivery Reviews, 2016, 106:256-276.
[19] LIN S H, HUANG K J, WENG C F, et al.Exploration of natural product ingredients as inhibitors of human HMG-CoA reductase through structure-based virtual screening[J].Drug Design, Development and Therapy, 2015, 9:3313-3324.
[20] 陈艳楠, 邱智军, 刘学强, 等.紫贻贝(Mytilus edulis)蛋白计算机模拟消化物活性的生物信息学分析[J].食品与发酵工业, 2022, 48(15):185-192.
CHEN Y N, QIU Z J, LIU X Q, et al.Bioinformatics analysis of the bioactivities of in silico digest from Mytilus edulis proteins[J].Food and Fermentation Industries, 2022, 48(15):185-192.
[21] BANERJEE P, DUNKEL M, KEMMLER E, et al.SuperCYPsPred:A web server for the prediction of cytochrome activity[J].Nucleic Acids Research, 2020, 48(W1):W580-W585.
[22] 田文慧, 孙丽平, 张翠, 等.白啤中二肽基肽酶-IV抑制肽的虚拟筛选及活性分析[J].食品科学, 2022, 43(10):81-87.
TIAN W H, SUN L P, ZHANG C, et al.Virtual screening of activity evaluation of dipeptidyl peptidase-IV inhibitory peptides in white beer[J].Food Science, 2022, 43(10):81-87.
[23] 张颖. 牛、羊乳酪蛋白源DPP-Ⅳ抑制肽的制备、鉴定及抑制机理研究[D].北京:中国农业大学, 2016.
ZHANG Y.Enzymatic preparation, identification and inhibition mechanism of DPP-Ⅳ inhibitory peptides derived from bovine and caprine milk casein[D].Beijing:China Agricultural University, 2016.
[24] 叶贤江, 苏志琛, 林晓娟, 等.基于生物信息学与分子对接技术对坛紫菜降血压肽的筛选及活性分析[J].食品科学, 2021, 42(24):140-148.
YE X J, SU Z C, LIN X J, et al.Screening and activity evaluation of antihypertensive peptides from Porphyra haitanensis by bioinformatics and molecular docking[J].Food Science, 2021, 42(24):140-148.
[25] JIA J Q, WU Q Y, YAN H, et al.Purification and molecular docking study of a novel angiotensin-I converting enzyme (ACE) inhibitory peptide from alcalase hydrolysate of ultrasonic-pretreated silkworm pupa (Bombyx mori) protein[J].Process Biochemistry, 2015, 50(5):876-883.
[26] 吴长平. 大豆ACE抑制肽的选择性制备及其作用机理研究[D].广州:华南理工大学, 2019.
WU C P.Selective preparation and mechanism study of soybean ACE inhibitory peptides[D].Guangzhou:South China University of Technology, 2019.