水蛭素(Hirudin,H)是一种从水蛭唾液中提取的小分子多肽,具有3个分子内二硫键[1]。它能特异性结合凝血酶,是目前发现的最强凝血酶天然抑制剂[2]。水蛭素有3种主要亚型,分别为水蛭素变体(Hirudin varidant, Hv)Ⅰ、Ⅱ、Ⅲ(Hv1, Hv2, Hv3)[3],它们具有相似的N端核心结构域和对凝血酶的抑制活性。其中Hv3除了具有抗凝血作用外,在预防和治疗白内障方面也表现出巨大潜力[4]。目前,一些水蛭素变体已经在大肠杆菌(Escherichia coli)、乳酸乳球菌、丝状真菌、转基因植物、酵母菌等[4-10]多种底盘细胞中成功表达。MENDOZA-VEGA等[11]利用酿酒酵母(Saccharomyces cerevisiae) C13ABYS86表达Hv2时产量达到500 mg/L,但是其发酵周期长达60 h;KIM等[7]利用GAL1启动子在S.cerevisiae中表达水蛭素,发酵72 h产量才能达到62.1 mg/L;MATSUI等[12-13]通过构建猪腺苷酸激酶蛋白与重组水蛭素变体I(recombinant Hirudin varidant I, Rhv1)融合表达载体,实现了Rhv1在E.coli中的高水平表达,但是其以包涵体形式存在,产物没有活性。HUANG等[4]开发了一种稳定表达Rhv3的方法,并通过补料分批发酵使得Rhv3产量达到915 mg/L。然而S.cerevisiae、转基因植物、丝状真菌等发酵周期长;E.Coli具有内毒素、纯化工艺复杂,其大规模生产仍然具有不小的挑战。
谷氨酸棒杆菌(Corynebacterium glutamicum)是一种高GC含量、无内毒素的革兰氏阳性细菌,常被用来生产氨基酸等小分子物质[14-15]。由于其胞外蛋白酶活性低、具有Sec、Tat两种强大的蛋白分泌体系,被认为是一种新型重组蛋白表达系统。利用C.glutamicum表达系统进行重组蛋白生产不仅能缩短发酵周期,而且可以规避E.coli表达系统内毒素、纯化难的问题,降低重组蛋白生产成本。
本研究利用实验室前期开发的3A组装系统,在C.glutamicun中构建以组成型启动子Ptac控制表达的1~7拷贝Rhv3重组菌株,并且每一个Rhv3基因的构建过程中将信号肽进行替换,以实现Rhv3的高效分泌。同时在Rhv3基因的C端添加6个组氨酸标签,最终仅需通过简单的Ni2+纯化即可获得高纯度Rhv3蛋白。
1 材料与方法
1.1 材料与试剂
1.1.1 主要试剂
Taq DNA聚合酶、pfu DNA聚合酶、质粒提取试剂盒、胶回收试剂盒、PCR产物回收试剂盒,CWBIO;限制性内切酶、Ligation Mix,TakaRa;牛纤维蛋白原、凝血酶,索莱宝;所有试剂和化学品均为分析纯试剂。
1.1.2 菌株、质粒
不同核糖体结合位点(ribosome binding site,RBS)-目标基因融合质粒所用引物见表1。引物合成和测序由苏州安升达完成。
表1 本实验所用引物
Table 1 Primers used in this study

注:粗体为对应RBS序列。
引物名称引物序列(5′-3′)3A-RBS1-FCCGGAATTCGCGGCCGCACTAGTAAAGGAGGAGCTAGTATG3A-RBS2-FCCGGAATTCGCGGCCGCACTAGTAGAAGGAGGCTAGTATG3A-RBS3-FCCGGAATTCGCGGCCGCACTAGTAGAAAGGAGGGCTAGTATG3A-RBS4-FCCGGAATTCGCGGCCGCACTAGTGAAAGGAGGGCTAGTATG3A-RBS5-FCCGGAATTCGCGGCCGCACTAGTGAAAGGAGAGCTAGTATG3A-RBS6-FCCGGAATTCGCGGCCGCACTAGTGAAAGGCGAGCTAGTATG3A-RBS7-FCCGGAATTCGCGGCCGCACTAGTGAAAGGAGCTAGTATGp3A-RAACTGCAGCGGCCGCTTAGCTAGC
本研究中所构建菌株如表2所示。
表2 本实验所用菌株
Table 2 Strains used in this study

质粒特性来源p3A-CgR0949-TetpXMJ19 derivative, ΔlacIq, Tet, CgR0949实验室贮存p3A-PorB-TetpXMJ19 derivative, ΔlacIq, Tet, PorB实验室贮存p3A-CspA-TetpXMJ19 derivative, ΔlacIq, Tet, CspA实验室贮存p3A-CspB-TetpXMJ19 derivative, ΔlacIq, Tat, CspB实验室贮存p3A-Cg1514-TetpXMJ19 derivative, ΔlacIq,Tet, Cg1514实验室贮存p3A-Ptac-AmppXMJ19 derivative, ΔlacIq, Amp, Ptac实验室贮存p3A-Rhv3-AmppXMJ19 derivative, ΔlacIq, Amp, Rhv3实验室贮存p3A-egfp-AmppXMJ19 derivative, ΔlacIq, Amp实验室贮存p3A-KanpXMJ19 derivative, ΔlacIq, Kan实验室贮存p3A-ChlrpXMJ19 derivative, ΔlacIq, Chlr实验室贮存p3A-RBS1-egfp-Ampp3A-egfp-Amp derivative, egfp, RBS1本研究p3A-RBS2-egfp-Ampp3A-egfp-Amp derivative, egfp, RBS2本研究p3A-RBS3-egfp-Ampp3A-egfp-Amp derivative, egfp, RBS3本研究p3A-RBS4-egfp-Ampp3A-egfp-Amp derivative, egfp, RBS4本研究p3A-RBS5-egfp-Ampp3A-egfp-Amp derivative, egfp, RBS5本研究p3A-RBS6-egfp-Ampp3A-egfp-Amp derivative, egfp, RBS6本研究p3A-RBS7-egfp-Ampp3A-egfp-Amp derivative, egfp, RBS7本研究p3A-RBS1-CgR0949-Tetp3A-egfp-Tet derivative, CgR0949, RBS1本研究p3A-RBS1-CgR0949-Rhv3-Chlrp3A-egfp-Chlr derivative, RBS1,CgR0949,Rhv3,Chlr本研究p3A-RBS1-CspA-Tetp3A-egfp-Tet derivative, CspA, RBS1本研究p3A-RBS1-CspA-Rhv3-Chlrp3A-egfp-Chlr derivative, RBS1, CspA,Rhv3,Chlr本研究p3A-RBS1-PorB-Tetp3A-egfp-Tet derivative, PorB, RBS1本研究p3A-RBS1-PorB-Rhv3-Chlrp3A-egfp-Chlr derivative, RBS1, PorB,Rhv3,Chlr本研究p3A-RBS1-CspB-Tetp3A-egfp-Tet derivative, CspB, RBS1本研究p3A-RBS1-CspB-Rhv3-Chlrp3A-egfp-Chlr derivative, RBS1, CspB,Rhv3,Chlr本研究p3A-RBS1-Cg1514-Tetp3A-egfp-Tet derivative, Cg1514, RBS1本研究p3A-RBS1-Cg1514-Rhv3-Chlrp3A-egfp-Chlr derivative, RBS1, Cg1514, Rhv3,Chlr本研究p3A-M1-egfp-Kanp3A-Kan derivative, Ptac, egfp, Kan本研究p3A-M2S1-egfp-Chlrp3A-M1-egfp-Kan derivative, two copies of egfp, RBS1, Chlr本研究p3A-M2S2-egfp-Chlrp3A-M1-egfp-Kan derivative, two copies of egfp, RBS2, Chlr本研究p3A-M2S3-egfp-Chlrp3A-M1-egfp-Kan derivative, two copies of egfp, RBS3, Chlr本研究p3A-M2S4-egfp-Chlrp3A-M1-egfp-Kan derivative, two copies of egfp, RBS4, Chlr本研究p3A-M2S5-egfp-Chlrp3A-M1-egfp-Kan derivative, two copies of egfp, RBS5, Chlr本研究p3A-M2S6-egfp-Chlrp3A-M1-egfp-Kan derivative, two copies of egfp, RBS6, Chlr本研究p3A-M2S7-egfp-Chlrp3A-M1-egfp-Kan derivative, two copies of egfp, RBS7, Chlr本研究p3A-M1-CspA-Rhv3-Chlrp3A-Chlr derivative, Ptac, CspA, Rhv3, Chlr本研究p3A-M2S1-CgR0949-Rhv3-Kanp3A-M1-CspA-Rhv3-Chlr derivative, two copies of Rhv3, RBS1, CgR0949, Kan本研究p3A-M2S1-PorB-Rhv3-Kanp3A-M1-CspA-Rhv3-Chlr derivative, two copies of Rhv3, RBS1, PorB, Kan本研究p3A-M2S1-CspA-Rhv3-Kanp3A-M1-CspA-Rhv3-Chlr derivative, two copies of Rhv3, RBS1, CspA, Kan本研究p3A-M2S1-CspB-Rhv3-Kanp3A-M1-CspA-Rhv3-Chlr derivative, two copies of Rhv3, RBS1, CspB, Kan本研究p3A-M2S1-Cg1514-Rhv3-Kanp3A-M1-CspA-Rhv3-Chlr derivative, two copies of Rhv3, RBS1, Cg1514, Kan本研究p3A-M3S1-CgR0949-Rhv3-Chlrp3A-M2S1-CgR0949-Rhv3-Kan derivative, three copies of Rhv3, RBS1, CgR0949, CgR0949, Chlr本研究p3A-M3S1-PorB-Rhv3-Chlrp3A-M2S1-CgR0949-Rhv3-Kan derivative, three copies of Rhv3, RBS1, CgR0949, PorB, Chlr本研究
续表2
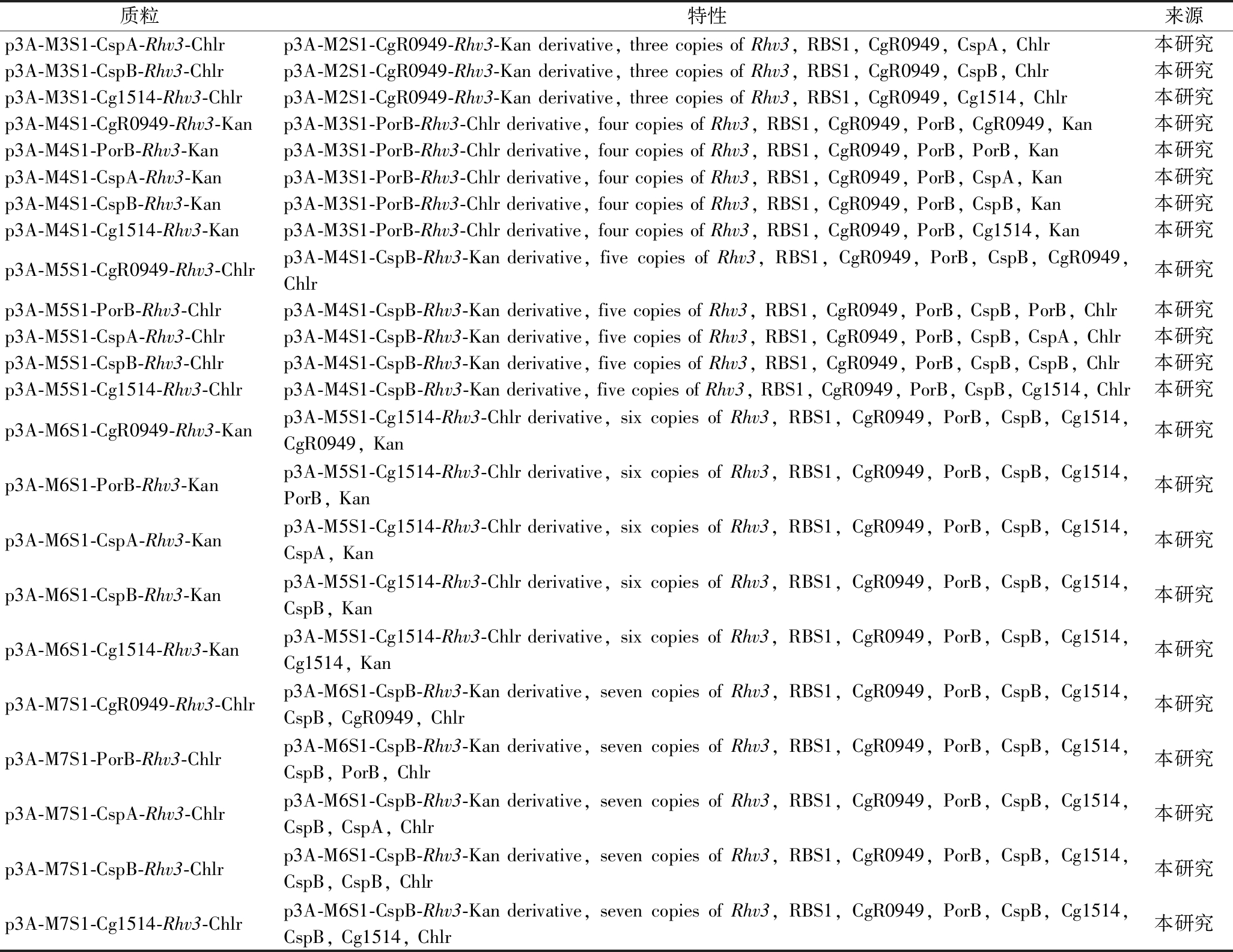
质粒特性来源p3A-M3S1-CspA-Rhv3-Chlrp3A-M2S1-CgR0949-Rhv3-Kan derivative, three copies of Rhv3, RBS1, CgR0949, CspA, Chlr本研究p3A-M3S1-CspB-Rhv3-Chlrp3A-M2S1-CgR0949-Rhv3-Kan derivative, three copies of Rhv3, RBS1, CgR0949, CspB, Chlr本研究p3A-M3S1-Cg1514-Rhv3-Chlrp3A-M2S1-CgR0949-Rhv3-Kan derivative, three copies of Rhv3, RBS1, CgR0949, Cg1514, Chlr本研究p3A-M4S1-CgR0949-Rhv3-Kanp3A-M3S1-PorB-Rhv3-Chlr derivative, four copies of Rhv3, RBS1, CgR0949, PorB, CgR0949, Kan本研究p3A-M4S1-PorB-Rhv3-Kanp3A-M3S1-PorB-Rhv3-Chlr derivative, four copies of Rhv3, RBS1, CgR0949, PorB, PorB, Kan本研究p3A-M4S1-CspA-Rhv3-Kanp3A-M3S1-PorB-Rhv3-Chlr derivative, four copies of Rhv3, RBS1, CgR0949, PorB, CspA, Kan本研究p3A-M4S1-CspB-Rhv3-Kanp3A-M3S1-PorB-Rhv3-Chlr derivative, four copies of Rhv3, RBS1, CgR0949, PorB, CspB, Kan本研究p3A-M4S1-Cg1514-Rhv3-Kanp3A-M3S1-PorB-Rhv3-Chlr derivative, four copies of Rhv3, RBS1, CgR0949, PorB, Cg1514, Kan本研究p3A-M5S1-CgR0949-Rhv3-Chlrp3A-M4S1-CspB-Rhv3-Kan derivative, five copies of Rhv3, RBS1, CgR0949, PorB, CspB, CgR0949, Chlr 本研究p3A-M5S1-PorB-Rhv3-Chlrp3A-M4S1-CspB-Rhv3-Kan derivative, five copies of Rhv3, RBS1, CgR0949, PorB, CspB, PorB, Chlr本研究p3A-M5S1-CspA-Rhv3-Chlrp3A-M4S1-CspB-Rhv3-Kan derivative, five copies of Rhv3, RBS1, CgR0949, PorB, CspB, CspA, Chlr本研究p3A-M5S1-CspB-Rhv3-Chlrp3A-M4S1-CspB-Rhv3-Kan derivative, five copies of Rhv3, RBS1, CgR0949, PorB, CspB, CspB, Chlr本研究p3A-M5S1-Cg1514-Rhv3-Chlrp3A-M4S1-CspB-Rhv3-Kan derivative, five copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, Chlr本研究p3A-M6S1-CgR0949-Rhv3-Kanp3A-M5S1-Cg1514-Rhv3-Chlr derivative, six copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CgR0949, Kan本研究p3A-M6S1-PorB-Rhv3-Kanp3A-M5S1-Cg1514-Rhv3-Chlr derivative, six copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, PorB, Kan本研究p3A-M6S1-CspA-Rhv3-Kanp3A-M5S1-Cg1514-Rhv3-Chlr derivative, six copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CspA, Kan本研究p3A-M6S1-CspB-Rhv3-Kanp3A-M5S1-Cg1514-Rhv3-Chlr derivative, six copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CspB, Kan本研究p3A-M6S1-Cg1514-Rhv3-Kanp3A-M5S1-Cg1514-Rhv3-Chlr derivative, six copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, Cg1514, Kan本研究p3A-M7S1-CgR0949-Rhv3-Chlrp3A-M6S1-CspB-Rhv3-Kan derivative, seven copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CspB, CgR0949, Chlr本研究p3A-M7S1-PorB-Rhv3-Chlrp3A-M6S1-CspB-Rhv3-Kan derivative, seven copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CspB, PorB, Chlr本研究p3A-M7S1-CspA-Rhv3-Chlrp3A-M6S1-CspB-Rhv3-Kan derivative, seven copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CspB, CspA, Chlr本研究p3A-M7S1-CspB-Rhv3-Chlrp3A-M6S1-CspB-Rhv3-Kan derivative, seven copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CspB, CspB, Chlr本研究p3A-M7S1-Cg1514-Rhv3-Chlrp3A-M6S1-CspB-Rhv3-Kan derivative, seven copies of Rhv3, RBS1, CgR0949, PorB, CspB, Cg1514, CspB, Cg1514, Chlr本研究
1.1.3 培养基
LB培养基(g/L):酵母提取物5,胰蛋白胨10,NaCl 10,用于培养E.coli DH5α。
LBB培养基(g/L):酵母提取物5,胰蛋白胨10,NaCl 10,脑心浸出液 10,用于C.glutamicum CGMCC1.15647重组菌株培养。
CGXⅡ培养基:葡萄糖 40 g/L;尿素 5 g/L;硫酸铵 20 g/L;K2HPO4 1 g/L;MgSO4·7H2O 0.25 g/L;CaCl2 10 mg/L;FeSO4·7H2O 10 mg/L;MnSO4·H2O 10 mg/L;ZnSO4·7H2O 1 mg/L;0.2 mg/L CuSO4;NiCl·6H2O 0.02 mg/L;原儿茶酸30 mg/L;3-吗啉丙磺酸(3-morpholinopropanesulfoinc acid,MOPs) 42 g/L,用于C.glutamicum CGMCC1.15647重组菌株培养。
BHI培养基:脑心浸出液 37 g/L,用于C.glutamicum CGMCC1.15647重组菌株培养。
1.1.4 仪器与设备
LongGene A 300 PCR仪,朗基科学仪器有限公司;DYCZ-24DN电泳仪,北京六一仪器厂;MicroPulser电击转化仪,Bio-Rad (USA)。
1.2 实验方法
1.2.1 单拷贝基因表达载体构建
课题组前期以BioBrick为基础,在C.glutamicum中构建了含不同抗性基因的3A组装系统[16]。克隆质粒p3A-Amp、p3A-Kan、p3A-Chlr上含有EcoR I、Nhe I、Spe I和Pst I 4个限制性内切酶位点。Nhe I、Spe I为一组同尾酶,经酶切后产生相同的黏性末端。在组装时两个黏性末端连接,形成不被其中任何限制性内切酶识别的间隔序列(Scar)。
标准化组成型启动子Ptac采用EcoR I、Nhe I酶切,标准化egfp质粒使用Spe I、Pst I酶切,标准化p3A-Kan质粒使用EcoR I、Pst I酶切。经80 ℃灭活后,按照4∶4∶1比例连接,构建以Ptac控制表达的单拷贝egfp质粒。同样地,构建以Ptac启动子、CspA信号肽控制分泌表达的单拷贝Rhv3质粒。
1.2.2 多拷贝基因表达载体构建
RBS-egfp质粒构建:以p3A-egfp-Kan为模板,使用引物3A-RBS-F和p3A-R进行PCR扩增(图1-A),将不同RBS序列引入egfp基因ATG之前。获得的片段使用EcoR I、Pst I酶切后,将目的片段连入经EcoR I、Pst I酶切的p3A-Amp中,得到含不同RBS序列的p3A-RBS-egfp-Amp质粒。
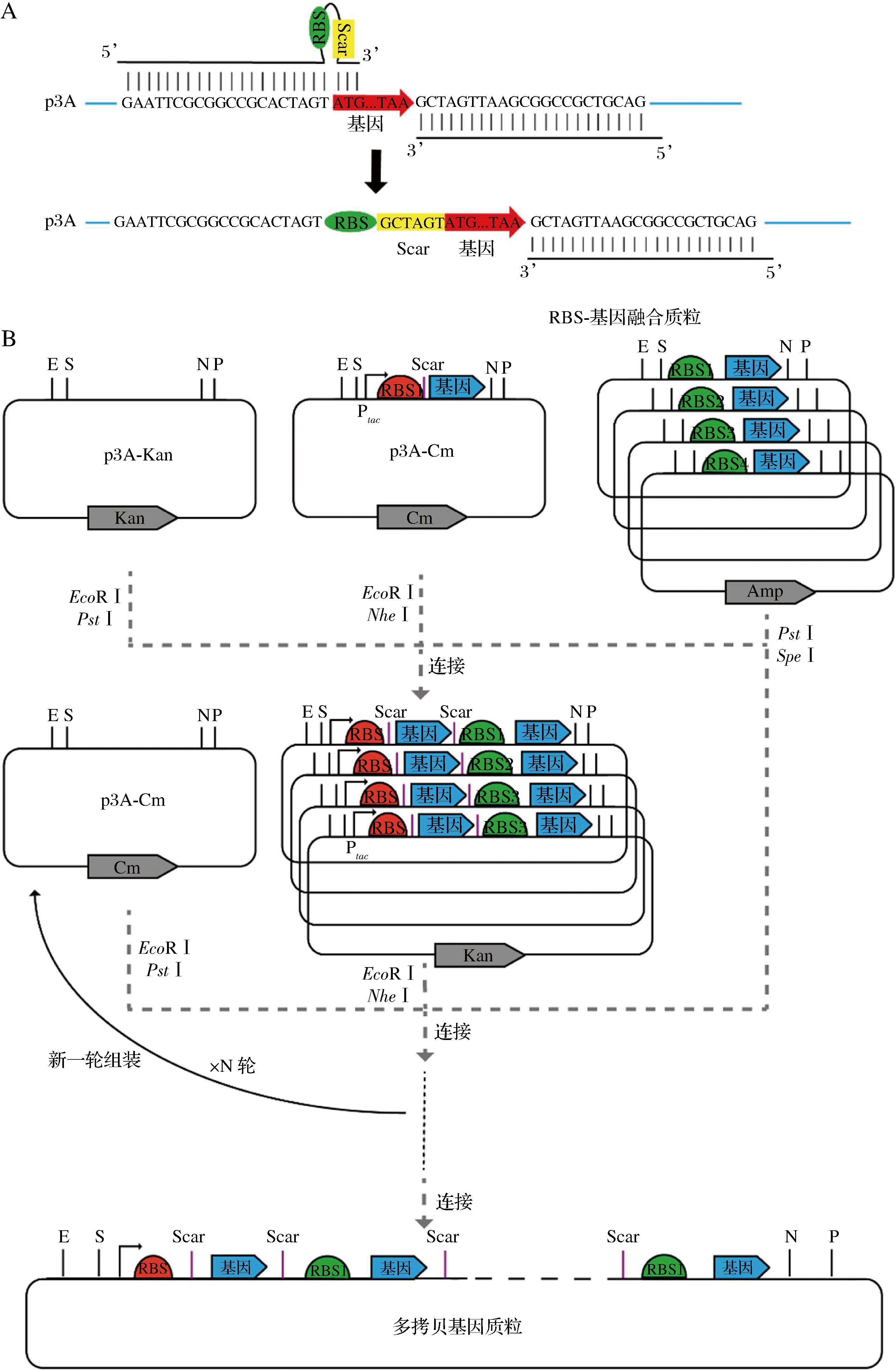
A-适用于3A组装系统的RBS通用引物示意图;B-基于3A组装的多拷贝质粒构建示意图
图1 基于3A组装的RBS通用引物开发及多拷贝质粒构建流程
Fig.1 Development of the RBS universal primer and a flow chart for the multi-copy plasmid based on 3A assembly
注:E-EcoR I酶切位点,序列为GAATTC;S-Spe I酶切位点,序列为ACTAGT;N-Nhe I酶切位点,序列为GCTAGC;P-Pst I酶切位点,序列为CTGCAG;Scar-Nhe I和Spe I连接后形成的间隔序列,序列为GCTAGT(下同)。
多拷贝egfp质粒构建:分别利用上述质粒与实验室前期构建的p3A-Ptac-egfp-Kan进行3A组装,获得含不同RBS序列的两拷贝egfp表达质粒(图1-B)。具体操作步骤参照图1-B。
RBS-CgR0949-Rhv3、RBS-PorB-Rhv3、RBS-CspA-Rhv3、RBS-CspB-Rhv3、RBS-Cg1514-Rhv3质粒构建:分别以p3A-CgR0949-Kan、p3A-PorB-Kan、p3A-CspA-Kan、p3A-CspB-Kan、p3A-Cg1514-Kan为模板,使用引物3A-SD1-F和p3A-R进行PCR扩增(图1-A),将RBS1序列引入信号肽基因CgR0949、PorB、CspA、CspB、Cg1514的ATG之前。得到的片段使用EcoR I、Pst I酶切,连接到p3A-Amp中,得到p3A-RBS1-CspA-Amp、p3A-RBS1-CgR0949-Amp。上述质粒经EcoR I、Nhe I酶切,标准化Rhv3质粒经Spe I、Pst I酶切,连接至经EcoR I、Pst I酶切的p3A-Chlr中,获得p3A-RBS1-CgR0949-Rhv3-Chlr、p3A-RBS1-PorB-Rhv3-Chlr、p3A-RBS1-CspA-Rhv3-Chlr、p3A-RBS1-CspB-Rhv3-Chlr、p3A-RBS1-Cg1514-Rhv3-Chlr(图1-B)。
多拷贝Rhv3质粒构建:分别利用上述质粒与单拷贝p3A-Ptac-CspA-Rhv3-Chlr进行3A组装,获得含不同信号肽序列的两拷贝Rhv3表达质粒。接着选择最优表达菌株对应质粒按照上述步骤进行多轮(×N rounds)构建,获得多拷贝Rhv3表达质粒。具体操作步骤参照图1-B。
1.2.3 质粒转化
上述质粒采用电转化方法引入宿主细胞C.glutamicum CGMCC1.15647中[17]。
1.2.4 egfp荧光强度分析
将含有不同RBS序列的两拷贝egfp重组菌株接种到24孔板中过夜培养。按2%接种比例转接到含新鲜LBB培养基的24孔板中发酵24 h。取待测菌液0.2 mL,使用培养基将OD600值调至0.5左右,使用荧光分光光度计测定菌体荧光值(激发波长和发射波长分别为488 nm和507 nm)。最后计算单位OD600值荧光强度以指示不同RBS翻译强度。
1.2.5 Rhv3表达、纯化分析
将重组菌接种至含相应抗生素的培养基中过夜培养,按2%接种比例转接到新鲜培养基中发酵24 h后,菌液经12 000 r/min离心5 min收集上清液进行SDS-PAGE分析。
Rhv3蛋白序列C端添加有6个His-tag,作为纯化标记。用AKTA中的HisTrap HP色谱柱纯化Rhv3-His,蛋白纯度用SDS-PAGE分析测定。
1.2.6 凝血酶滴定
蛋白浓度检测采用BSA定量法。分别取0.5 mg/L蛋白标准溶液0、1、2、4、8、12、16、20 μL加入到96孔酶标板中,用PBS溶液稀释上述蛋白标准品溶液至终体积为20 μL。同时取适量样品加入酶标板中,按需将终体积稀释至20 μL;按照说明书配制BCA标准溶液,并向上述各孔中加入200 μL,放置在37 ℃培养箱中反应30 min;结束后使用酶标仪测定A595;最后根据蛋白标准品浓度及A595得出标准曲线,并按此曲线计算样品蛋白浓度。
Rhv3活性分析采用凝血酶滴定法。试管中加入200 μL 0.5%(质量分数)牛纤维蛋白原及100 μL适量浓度发酵上清液;接着每隔1 min加入10 μL 50、25、12.5、6.25、3.125 NIH/mL凝血酶溶液,直至观察到溶液出现凝结,此时即为反应终点。按照水蛭素与凝血酶1∶1反应,加入凝血酶的量即为水蛭素抗凝血酶活性(ATU/mL),如公式(1)所示:
发酵上清液Rhv3活性![]()
(1)
2 结果与分析
2.1 Rhv3在C.glutamicum中的表达及活性分析
将构建好的p3A-M1-CspA-Rhv3-Chlr质粒(图2-A)电转化入C.glutamicum CGMCC 1.15647中进行发酵培养,取发酵上清液进行SDS-PAGE分析。结果如图2-B所示,在15 kDa处出现明显的蛋白条带。与理论值(8 kDa)相比,目标条带大小接近理论值的2倍,猜测可能是因为2个蛋白质分子间较强的疏水作用力使Rhv3以二聚体形式分泌。为进一步优化产量,将重组菌株分别在LBB、BHI和CGXⅡ 3种培养基中培养。结果显示在CGXⅡ培养基中Rhv3几乎无表达。结合重组菌株生物量及凝血酶滴定活性分析(图2-C)可知,重组菌株在CGXⅡ培养基中生长最差,其OD600值仅为3.33,活性为0.42×103 ATU/L。相反在BHI培养基中重组菌株具有最大的生物量(26.62)和最大活性(3.02×103 ATU/L)。
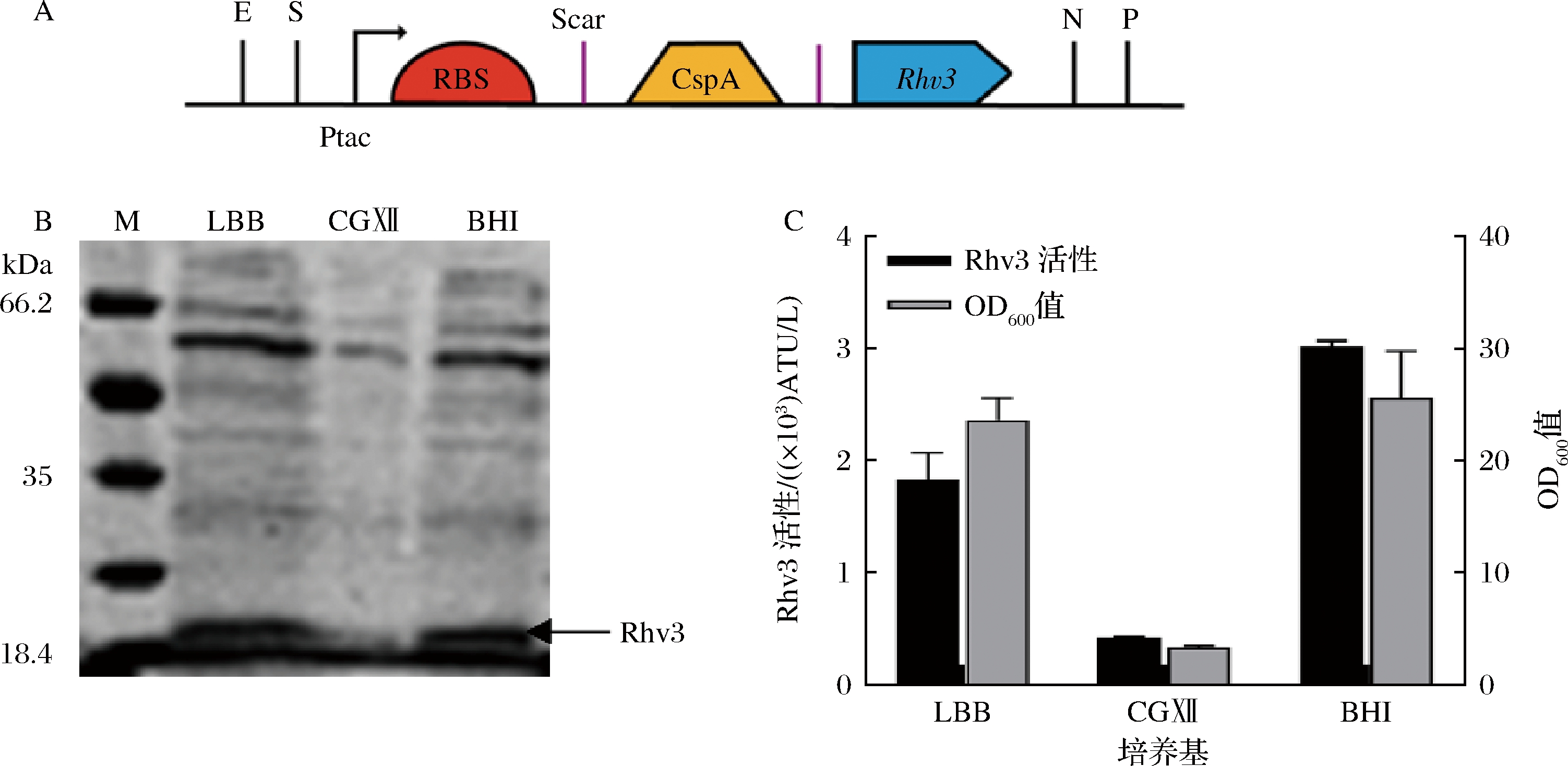
A-单拷贝Rhv3质粒结构示意图;B-不同培养基中Rhv3表达分析(M为分子质量标准);C-不同培养基中Rhv3活性分析
图2 重组菌株在不同培养基中表达情况分析
Fig.2 The expression of Rhv3 of recombinant strain under different culture conditions
2.2 RBS筛选
为了获得强RBS序列用于C.glutamicum中重组蛋白生产,本研究选取了7个不同强度的RBS序列,分别是AAAGGAGGA、AGAAGGAG、AGAAAGGAGG、GAAAGGAGG、GAAAGGA、GAAAGGCGA、GAAAGGAGA。按照图1-A方式进行PCR,构建含不同RBS序列的p3A-RBS-egfp-Amp质粒。接着得到的质粒分别与p3A-Ptac-egfp-Kan进行组装,构建两拷贝egfp质粒(图1-B)。将构建好的两拷贝egfp质粒转入C.glutamicum中,以单拷贝egfp(p3A-Ptac-egfp-Kan, POS)重组菌为对照(图3-A),通过egfp荧光强度差异比较不同RBS序列表达强度。
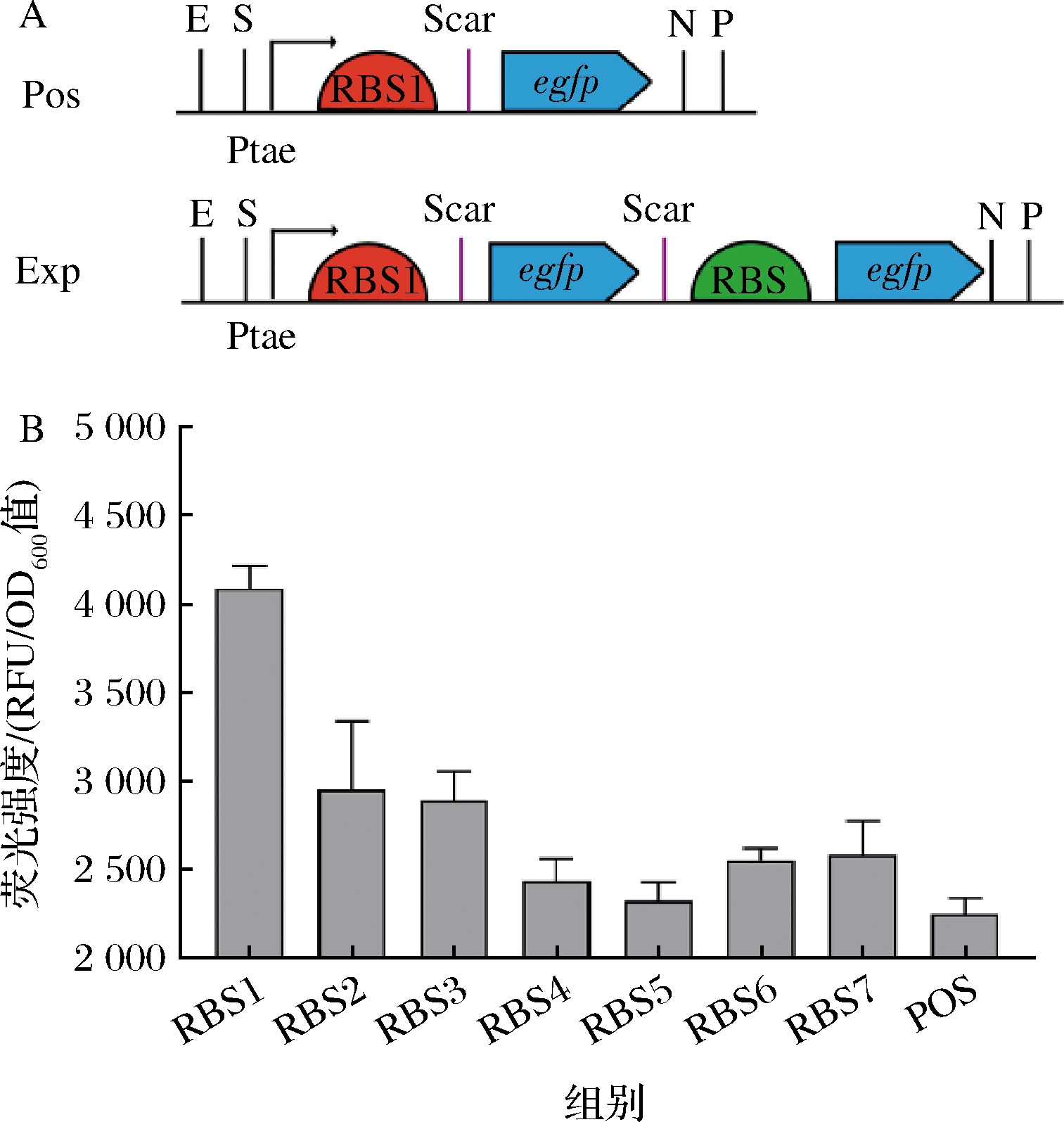
A-两拷贝egfp质粒结构示意图;B-不同RBS重组菌株单位荧光强度(RFU/OD600值)比较
图3 RBS序列筛选
Fig.3 Screening of RBS sequence
如图3-B所示,两拷贝egfp重组菌株荧光强度均高于单拷贝egfp重组菌株(POS);含不同RBS序列的两拷贝egfp重组菌株中,RBS1荧光强度最高,达到POS的1.81倍;RBS2、RBS3次之;RBS5最弱,荧光强度与POS相比几乎无增长。因此,选用RBS1序列用于C.glutamicum中重组蛋白的表达。
2.3 多拷贝Rhv3菌株构建及信号肽、拷贝数的优化表达分析
为优化Rhv3分泌水平,选择C.glutamicum内源信号肽CgR0949、PorB、CspA、CspB、Cg1514进行Rhv3多拷贝重组菌株构建(图4-A)并通过SDS-PAGE验证其表达强度。以单拷贝Rhv3重组菌做对照,通过Image J进行灰度分析发现,两拷贝Rhv3重组菌株表达量明显得到改善,并且第二拷贝Rhv3使用CgR0949、PorB、CspA、CspB和Cg1514信号肽(M2S-Cg1514)时,分泌强度分别是单拷贝菌株(M1-CspA)的1.37、1.67、1.45、2.06、2.28倍。需要注意的是,当第二拷贝Rhv3使用信号肽CspA、CspB时,SDS-PAGE上显示为两条带,猜测可能是蛋白表达过程中Rhv3蛋白出现降解造成的。

A-多拷贝Rhv3质粒结构示意图;B~G-分别为二、三、四、五、六、七拷贝Rhv3表达分析(M-蛋白分子质量标准;CK-野生型C.glutamicum)
图4 多拷贝策略优化Rhv3表达
Fig.4 Optimal expression of Rhv3 based on multi-copy strategy
注:每泳道上样15 μL(为更好的分辨Rhv3的表达,G图上样量降为7 μL),其他泳道定义取自相应菌株质粒名称中不同的部分,例如p3A-M2S-CgR0949-Rhv3-Kan,即对应泳道M2S-CgR0949。
为进一步优化Rhv3的表达,选择最优表达菌株(M2S-Cg1514)构建三拷贝Rhv3重组菌,结果如图4-C所示,Rhv3最高表达强度(M3S-CspB)是M2S-Cg1514的1.45倍。当Rhv3拷贝数到六时,Rhv3表达量达到最高(M6S-PorB),是出发菌株的4.69倍(图4-F)。然而在随后的七拷贝Rhv3表达中发现,Rhv3表达量出现下降(图4-G),猜测可能是过多重复序列积累导致其在体内发生同源重组;也可能是因为过多Rhv3合成消耗了过量核糖体,或者造成宿主代谢负担加重,导致菌株生长不良。综上,经过多轮构建及筛选,获得了一株高产Rhv3重组菌株(M6S-PorB);并且发现,在一定程度上,Rhv3表达量随着基因拷贝数的增加而升高。
2.4 Rhv3重组菌发酵培养及纯化分析
将M6S-PorB重组菌在500 mL摇瓶中进行发酵培养并检测其凝血酶活性。结果如图5-A所示,菌株在4~20 h内生物量快速增长,20 h后进入稳定期,至40 h时生物量达到最高(OD600=23.08)。SDS-PAGE显示,在4 h时上清液中能检测到明显的Rhv3蛋白条带。并且随着时间的延长,条带越来越粗(图5-B)。凝血酶滴定结果证实在40 h时,上清液中Rhv3活性达到最高,为10.91×103 ATU/L,比单拷贝Rhv3菌株提高3.61倍。经BCA蛋白定量检测分析可知,Rhv3含量达到1.89 g/L。AKTA纯化后,Rhv3纯度高于90%(图5-C)。

A-重组菌株在BHI培养基中发酵生长曲线及Rhv3活性分析;B-重组菌株Rhv3表达分析(每泳道上样3 μL,M为分子质量标准,1~12对应发酵4、8、12、16、20、24、28、32、36、40、44、48 h时Rhv3的表达情况);C-BSA标准曲线;D-经AKTA纯化后蛋白的SDS-PAGE分析(L1为目标蛋白纯化样品)
图5 重组菌株发酵培养
Fig.5 Fermentation of recombinant strain for Rhv3 production
3 讨论
为了改善重组蛋白的合成,学者们尝试过很多方法,包括替换启动子和信号肽、共表达伴侣蛋白、优化发酵工艺、提高基因拷贝数等[13,16,18-19]。对于Rhv3这种小分子多肽的生产,多数采用多拷贝策略提高生产。LIN等[20]利用2A肽构建egfp基因多拷贝串联表达菌株,egfp表达量在三拷贝时达到最高。梁伟锋等[18]利用同尾酶的不可逆转连接方式在Pichia pastoria中构建人脑源性神经营养因子(human brain derived neurotrophic factor, hBDNF)的1~6拷贝重组表达载体,使得hBDNF产量达到20 mg/L。同年,ZHONG等[21]使用常规构建方法在E.coli中构建1、2、4、8个拷贝融合人β-防御素-2(humanβ-defensin-2, hBD2)重组表达菌株,最终融合蛋白产量占总可溶性蛋白的62.2%,是目前报道的最高产量。周伟杰等[22]利用BioBrick法在P.pastoria中构建以Pgap为启动子的五拷贝血管内表皮生长因子(vascular endothelial growth factor, VEGF)重组菌,获得VEGF165产量高达0.45 g/L。
理论上讲,重组蛋白的生产与基因拷贝数呈线性关系,基因拷贝数增加会直接提高重组蛋白的生产。尽管在实际应用生产中被证实两者并没有呈现出很好的线性关系,但是这一策略仍然是提高重组蛋白生产最高效、快捷的方法。本研究通过在C.glutamicum中分泌表达Rhv3并筛选RBS、信号肽和拷贝数,使得Rhv3产量达到1.89 g/L。然而需要注意的是,RBS序列、拷贝数只是影响重组蛋白表达的因素之一,后续仍然可以通过底盘细胞改造、发酵工艺优化等手段进一步改善Rhv3的表达。
4 结论
总之,本研究在C.glutamicum表达系统中开发了3A组装的多拷贝基因表达策略。利用该策略,优化Rhv3合成过程中的RBS、信号肽以及拷贝数等,在放大培养中Rhv3分泌活性达到10.91×103 ATU/L,比之前报道的1.75×103 ATU/L[16]提高523%。与通过Linker、2A肽等进行重组蛋白表达的多拷贝融合策略相比,本研究多拷贝基因策略旨在利用一条mRNA上进行多个核糖体结合位点的翻译,实现重组蛋白表达最大化。同时3A组装的开发,缩短了载体构建周期,极大加速了重组蛋白生产效率。这一策略的开发,增强了C.glutamicum作为重组蛋白生产宿主的核心竞争力,为进一步利用C.glutamicum进行重组蛋白工业化生产奠定了基础。
[1] ZHANG J G, LAN N N.Hirudin variants production by genetic engineered microbial factory[J].Biotechnology &Genetic Engineering Reviews, 2018, 34(2):261-280.
[2] 赖宝英, 陈志峰.水蛭素对急性脑出血患者血脑屏障紧密连接蛋白表达和患者颅内压的影响[J].黑龙江中医药, 2021, 50(5):107-108.
LAI B Y, CHEN Z F.Effects of hirudin on expression of blood brain barrier tight junction protein and intracranial pressure in patients with acute cerebral hemorrhage[J].Heilongjiang Journal of Traditional Chinese Medicine, 2021, 50(5):107-108.
[3] LI T, MA J Y, XU Z H, et al.Transcriptomic analysis of the influence of methanol assimilation on the gene expression in the recombinant Pichia pastoris producing hirudin variant 3[J].Genes, 2019, 10(8):606.
[4] HUANG C C, ZHANG X R, QU J, et al.Robust preparative-scale extracellular production of hirudin in Escherichia coli and its purification and characterization[J].Journal of Industrial Microbiology and Biotechnology, 2012, 39:1487-1494.
[5 ] GIDDINGS G, ALLISON G, BROOKS D, et al.Transgenic plants as factories for biopharmaceuticals[J].Nature Biotechnology, 2000, 18:1151-1155.
[6] KIM M D, HAN K C, KANG H A, et al.Coexpression of BiP increased antithrombotic hirudin production in recombinant Saccharomyces cerevisiae[J].Journal of Biotechnology, 2003, 101(1):81-87.
[7] KIM M D, LEE T H, LIM H K, et al.Production of antithrombotic hirudin in GAL1-disrupted Saccharomyces cerevisiae[J].Applied Microbiology and Biotechnology, 2004, 65(3):259-262.
[8] LYU J, HUANG C C, ZHANG X R, et al.Extracellular secretion of anticoagulant peptide hirudin in Lactococcus lactis using SP310 mut2 signal peptide[J].Biotechnology Letters, 2012, 34(1):61-65.
[9] RADZIO R, KÜCK U.Efficient synthesis of the blood-coagulation inhibitor hirudin in the filamentous fungus Acremonium chrysogenum[J].Applied Microbiology and Biotechnology, 1997, 48(1):58-65.
[10] ROSENFELD S A, NADEAU D, TIRADO J, et al.Production and purification of recombinant hirudin expressed in the methylotrophic yeast Pichia pastoris[J].Protein Expression and Purification, 1996, 8(4):476-482.
[11] MENDOZA-VEGA O, HEBERT C, BROWN S W.Production of recombinant hirudin by high cell density fed-batch cultivations of a Saccharomyces cerevisiae strain:Physiological considerations during the bioprocess design[J].Journal of Biotechnology, 1994, 32(3):249-259.
[12] MATSUI T, SATO H, YAMAMURO H, et al.High cell density cultivation of recombinant Escherichia coli for hirudin variant 1 production[J].Journal of Biotechnology, 2008, 134(1-2):88-92.
[13] MATSUI T, SATO H, YAMAMURO H, et al.High cell density cultivation of recombinant E.coli for hirudin variant 1 production by temperature shift controlled by pUC18-based replicative origin[J].Applied Microbiology and Biotechnology, 2008, 80:779-783.
[14] KAWAGUCHI H, HASUNUMA T, OHNISHI Y, et al.Enhanced production of γ-amino acid 3-amino-4-hydroxybenzoic acid by recombinant Corynebacterium glutamicum under oxygen limitation[J].Microbial Cell Factories, 2021, 20:228.
[15] ZHANG Y C, LIU Y D, ZHANG S Y, et al.Metabolic engineering of Corynebacterium glutamicum WM001 to improve l-isoleucine production[J].Biotechnology and Applied Biochemistry, 2021, 68(3):568-584.
[16] WANG Y, GAO X, LIU X, et al.Construction of a 3A system from BioBrick parts for expression of recombinant hirudin variants Ⅲ in Corynebacterium glutamicum[J].Applied Microbiology and Biotechnology, 2020, 104:8257-8266.
[17] VAN DER REST M E, LANGE C, MOLENAAR D.A heat shock following electroporation induces highly efficient transformation of Corynebacterium glutamicum with xenogeneic plasmid DNA[J].Applied Microbiology and Biotechnology, 1999, 52:541-545.
[18] 梁伟锋, 张朝春, 杨希才.一种多拷贝毕赤酵母表达载体的构建及人脑源性神经营养因子的表达[J].微生物学报, 2005, 45(1):34-38.
LIANG W F, ZHANG C C, YANG X C.Construction of a multi-copy Pichia expression vector and expression of human brain-derived neurotrophic factor in Pichia pastoris[J].Acta Microbiologica Sinica, 2005, 45(1):34-38.
[19] SEYED HOSSEINI FIN N A, BARSHAN-TASHNIZI M, SAJJADI S M, et al.The effects of overexpression of cytoplasmic chaperones on secretory production of hirudin-PA in E.coli[J].Protein Expression and Purification, 2019, 157:42-49.
[20] LIN Y J, HUANG L H, HUANG C T.Enhancement of heterologous gene expression in Flammulina velutipes using polycistronic vectors containing a viral 2A cleavage sequence[J].PLoS One, 2013, 8(3):e59099.
[21] ZHONG Z X, XU Z N, PENG L, et al.Tandem repeat mhBD2 gene enhance the soluble fusion expression of hBD2 in Escherichia coli[J].Applied Microbiology and Biotechnology, 2006, 71(5):661-667.
[22] 周伟杰, 吴凤梅, 姚冬生, 等.通过毕赤酵母表达系统获得高纯度的重组人血管内皮生长因子(rhVEGF165)[J].生物工程学报, 2021, 37(11):4083-4094.
ZHOU W J, WU F M, YAO D S, et al.Production of high-purity recombinant human vascular endothelial growth factor (rhVEGF165) by Pichia pastoris[J].Chinese Journal of Biotechnology, 2021, 37(11):4083-4094.