酶,是一种极为高效的特殊催化剂,其化学本质是具有催化活性的蛋白质或核酸[1-2]。由于酶的基因在长达数万年的进化过程中不断地复制与突变,因此出现了诸多同源酶[3],大部分的同源酶氨基酸序列相似度较高,但催化的反应可能不同。在类胡萝卜素合成过程中,将番茄红素环化生成β-胡萝卜素的番茄红素β-环化酶(lycopene β-cyclase,LCYB)[4],和将紫黄质环化为新黄质的新黄质合酶(neoxanthin synthase,NSY)[5]序列高度相似,但是功能不尽相同。在普遍认为的原生质体祖先——蓝藻中仅含有LCYB,且这两种酶发挥活性还均结合有相同的辅酶和相似的活性中心[6-8],因此有研究认为NSY和LCYB存在进化关系,属于同源酶[4-5,9-10]。但对导致LCYB和NSY功能差异的原因,此前从未有报道。因此本研究旨在探究LCYB和NSY对番茄红素环化功能差异的原因。LCYB是生成β-胡萝卜素的关键酶之一,在代谢通路中处于上游,对LCYB的研究较多,已经鉴定识别的不同来源的LCYB超过500个。LIU等[11]通过大肠杆菌异源表达证明来自宁夏枸杞的番茄红素β-环化酶(LyLCYB)可以将番茄红素催化生成β-胡萝卜素。相较于LCYB,关于下游类胡萝卜素合成途径中将紫黄质催化生成新黄质的NSY研究较少[12],被鉴定识别的NSY远不如LCYB多。BOUVIER等[9]从番茄中克隆了大小为56 kDa的新黄质合酶(SoNSY),SoNSY和LyLCYB氨基酸序列相似性高达88.2%,且实验证明其不具备催化番茄红素的能力。
LCYB和NSY的底物均是对光、热等敏感的具有抗氧化性的类胡萝卜素[13],实现催化作用还需要有辅酶FAD和NADPH的参与[8],相较于在体内构建酶的表达系统,体外表达需要考虑底物的快速氧化以及辅酶的添加,更为困难且具有不稳定性,故构建体内类胡萝卜素代谢通路是理想的研究手段。因此本研究选择了枸杞来源的LyLCYB和番茄来源的SoNSY作为研究对象,全基因合成LyLCYB基因lyLCYB和SoNSY基因soNSY,通过构建嵌合体和点突变的方式将改造后的基因在大肠杆菌中实现异源表达,HPLC分析工程菌株发酵后的产物,以此发掘导致LyLCYB和SoNSY中番茄红素环化功能差异的关键氨基酸位点。
1 材料与方法
1.1 试剂材料
lyLCYB、soNSY、crtE、crtB、crtI由华大基因全基因合成;高保真DNA聚合酶试剂Phanta Max Super-Fidelity DNA Polymerase、DNA聚合酶试剂2×Taq Master Mix (Dye Plus)、重组克隆试剂ClonExpress Ultra One Step Cloning Kit购于诺唯赞生物公司;PCR回收试剂盒、快速质粒小提试剂盒购于天根生化科技公司;SSCS感受态细胞制备液购于上海捷瑞生物公司;番茄红素标准品、β-胡萝卜素标准品、IPTG、酵母粉、蛋白胨、琼脂粉、氨苄青霉素、卡那霉素均购于索莱宝科技公司。其他试剂未作特殊说明均为国药化学试剂分析纯。PCR引物由青岛擎科公司提供。
1.2 培养基配方
LB液体培养基:称取1.0 g胰蛋白胨,0.5 g酵母提取物,1.0 g氯化钠,溶于100.0 mL纯水中,115 ℃灭菌30 min[若需配制LB固体培养基,则在此基础上添加1.5%~2.0%(质量分数)的琼脂粉]。
氨苄青霉素溶液:称取0.1 g的氨苄青霉素粉末放置于1.5 mL的离心管中,用1.0 mL的超纯水溶解,在超净台中过孔径0.22 μm的水相滤膜,放于-20 ℃冰箱备用,且添加量为0.1%(体积分数)。
100.0 mg/mL卡那霉素溶液:方法同上[称取1.0 g 卡那霉素粉末,添加量为0.05%(体积分数)]。
1.3 生物信息学分析
利用如下生物信息学分析网站及软件分析序列,目标序列的基因查找网址:https://www.ncbi.nlm.nih.gov/;蛋白质序列转运肽预测网址[14]:http://www.cbs.dtu.dk/services/TargetP/;判定目的蛋白是否具有膜结合域网站:http://www.cbs.dtu.dk/services/TMHMM/;蛋白质结构域预测分析网站:http://smart.embl-heidelberg.de/;实验过程中用到蛋白质序列分析软件:Jalview(氨基酸序列比对)[15]、SnapGene(质粒构建)。
1.4 构建嵌合体双质粒菌株
目的片段的扩增以及质粒载体构建选用了高保真酶,并使用同源重组的方法使得各片段连接处留有15~20 bp同源序列。各种不同嵌合体构建的方式是将soNSY基因的部分片段替换为lyLCYB基因对应位置的片段。本实验室将crtE、crtB、crtI基因搭载到氨苄青霉素抗性的pTrc99a载体上构建了产番茄红素质粒[16];lyLCYB、soNSY及嵌合体基因搭载到以卡那霉素为抗性的pTac15K载体上用以探究原酶和重组酶的活性。构建质粒所需引物如表1所示,利用重组克隆试剂进行重组连接,将重组产物转入大肠杆菌(Escherichia coli)TreliefTM5α感受态中,涂布至对应抗生素的LB固体培养基上。37 ℃ 过夜培养约12 h后挑菌测序,并将测序结果正确的菌株接种至LB试管中培养6~8 h,OD600≈0.6时,用快速质粒小提试剂盒提取质粒备用。
表1 构建质粒所需引物
Table 1 Primers required constructing plasmids
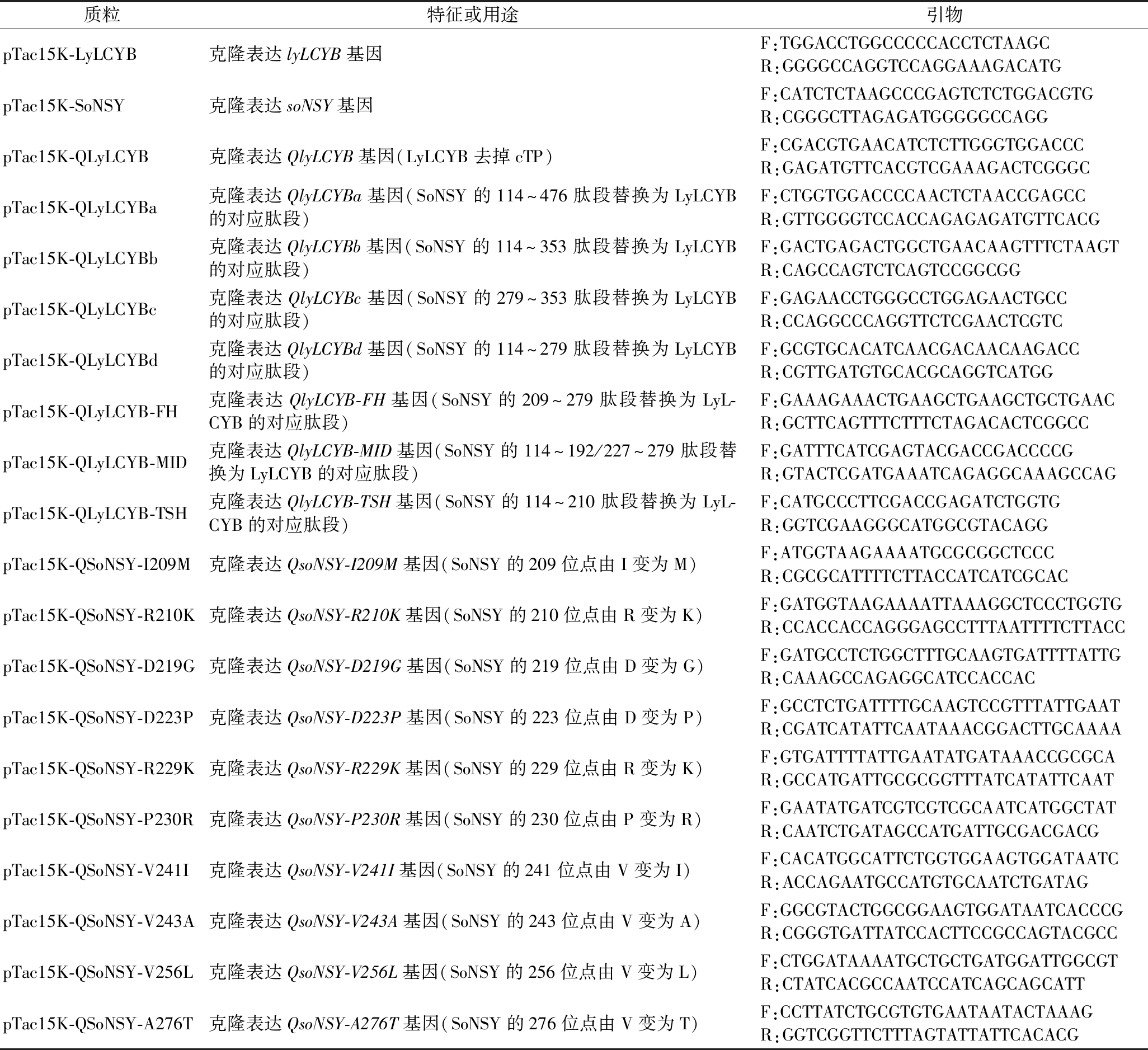
质粒特征或用途引物pTac15K-LyLCYB克隆表达lyLCYB基因F:TGGACCTGGCCCCCACCTCTAAGCR:GGGGCCAGGTCCAGGAAAGACATGpTac15K-SoNSY克隆表达soNSY基因F:CATCTCTAAGCCCGAGTCTCTGGACGTGR:CGGGCTTAGAGATGGGGGCCAGGpTac15K-QLyLCYB克隆表达QlyLCYB基因(LyLCYB去掉cTP)F:CGACGTGAACATCTCTTGGGTGGACCCR:GAGATGTTCACGTCGAAAGACTCGGGCpTac15K-QLyLCYBa克隆表达QlyLCYBa基因(SoNSY的114^476肽段替换为LyLCYB的对应肽段)F:CTGGTGGACCCCAACTCTAACCGAGCCR:GTTGGGGTCCACCAGAGAGATGTTCACGpTac15K-QLyLCYBb克隆表达QlyLCYBb基因(SoNSY的114^353肽段替换为LyLCYB的对应肽段)F:GACTGAGACTGGCTGAACAAGTTTCTAAGTR:CAGCCAGTCTCAGTCCGGCGGpTac15K-QLyLCYBc克隆表达QlyLCYBc基因(SoNSY的279^353肽段替换为LyLCYB的对应肽段)F:GAGAACCTGGGCCTGGAGAACTGCCR:CCAGGCCCAGGTTCTCGAACTCGTCpTac15K-QLyLCYBd克隆表达QlyLCYBd基因(SoNSY的114^279肽段替换为LyLCYB的对应肽段)F:GCGTGCACATCAACGACAACAAGACCR:CGTTGATGTGCACGCAGGTCATGGpTac15K-QLyLCYB-FH克隆表达QlyLCYB-FH基因(SoNSY的209^279肽段替换为LyL-CYB的对应肽段)F:GAAAGAAACTGAAGCTGAAGCTGCTGAACR:GCTTCAGTTTCTTTCTAGACACTCGGCCpTac15K-QLyLCYB-MID克隆表达QlyLCYB-MID基因(SoNSY的114^192/227^279肽段替换为LyLCYB的对应肽段)F:GATTTCATCGAGTACGACCGACCCCGR:GTACTCGATGAAATCAGAGGCAAAGCCAGpTac15K-QLyLCYB-TSH克隆表达QlyLCYB-TSH基因(SoNSY的114^210肽段替换为LyL-CYB的对应肽段)F:CATGCCCTTCGACCGAGATCTGGTGR:GGTCGAAGGGCATGGCGTACAGGpTac15K-QSoNSY-I209M克隆表达QsoNSY-I209M基因(SoNSY的209位点由I变为M)F:ATGGTAAGAAAATGCGCGGCTCCCR:CGCGCATTTTCTTACCATCATCGCACpTac15K-QSoNSY-R210K克隆表达QsoNSY-R210K基因(SoNSY的210位点由R变为K)F:GATGGTAAGAAAATTAAAGGCTCCCTGGTGR:CCACCACCAGGGAGCCTTTAATTTTCTTACCpTac15K-QSoNSY-D219G克隆表达QsoNSY-D219G基因(SoNSY的219位点由D变为G)F:GATGCCTCTGGCTTTGCAAGTGATTTTATTGR:CAAAGCCAGAGGCATCCACCACpTac15K-QSoNSY-D223P克隆表达QsoNSY-D223P基因(SoNSY的223位点由D变为P)F:GCCTCTGATTTTGCAAGTCCGTTTATTGAATR:CGATCATATTCAATAAACGGACTTGCAAAApTac15K-QSoNSY-R229K克隆表达QsoNSY-R229K基因(SoNSY的229位点由R变为K)F:GTGATTTTATTGAATATGATAAACCGCGCAR:GCCATGATTGCGCGGTTTATCATATTCAATpTac15K-QSoNSY-P230R克隆表达QsoNSY-P230R基因(SoNSY的230位点由P变为R)F:GAATATGATCGTCGTCGCAATCATGGCTATR:CAATCTGATAGCCATGATTGCGACGACGpTac15K-QSoNSY-V241I克隆表达QsoNSY-V241I基因(SoNSY的241位点由V变为I)F:CACATGGCATTCTGGTGGAAGTGGATAATCR:ACCAGAATGCCATGTGCAATCTGATAGpTac15K-QSoNSY-V243A克隆表达QsoNSY-V243A基因(SoNSY的243位点由V变为A)F:GGCGTACTGGCGGAAGTGGATAATCACCCGR:CGGGTGATTATCCACTTCCGCCAGTACGCCpTac15K-QSoNSY-V256L克隆表达QsoNSY-V256L基因(SoNSY的256位点由V变为L)F:CTGGATAAAATGCTGCTGATGGATTGGCGTR:CTATCACGCCAATCCATCAGCAGCATTpTac15K-QSoNSY-A276T克隆表达QsoNSY-A276T基因(SoNSY的276位点由V变为T)F:CCTTATCTGCGTGTGAATAATACTAAAGR:GGTCGGTTCTTTAGTATTATTCACACG
将提取的pTrc99a-crtEBI质粒利用水浴法转化到E.coli BL21(DE3)中,37 ℃过夜培养后,挑取单菌落,经过PCR验证正确后,接种到LB试管中,添加氨苄青霉素抗性过夜培养,取1.0 mL 接种到含有氨苄青霉素抗性的50.0 mL LB锥形瓶培养基中,培养至OD600值为0.5~0.6左右,利用SSCS感受态细胞制备液,制备产番茄红素的感受态细胞,随后再次利用水浴法将第二个质粒转化到自制的产番茄红素感受态细胞中,涂布于氨苄青霉素和卡那霉素双抗性的LB固体平板,过夜培养,挑取单菌落,PCR验证,最终构建好的菌株如表2所示。
表2 实验所需菌株
Table 2 Strains required for the experiment

菌株特征或用途来源E. coli DH5α质粒克隆型菌株购自擎科新业生物E. coli BL21(DE3)质粒表达型菌株购自擎科新业生物E. coli BL21 (pTrc99a-crtEBI)番茄红素工程菌本实验室构建 E. coli BL21 (pTrc99a-crtEBI+pTac15K-SoNSY)番茄红素工程菌本研究构建 E. coli BL21 (pTrc99a-crtEBI+pTac15K-LyLCYB)β-胡萝卜素工程菌本研究构建 E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYB)β-胡萝卜素工程菌本研究构建 E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYBa)β-胡萝卜素工程菌本研究构建 E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYBb)β-胡萝卜素工程菌本研究构建 E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYBc)番茄红素工程菌本研究构建 E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYBd)β-胡萝卜素工程菌本研究构建
续表2

菌株特征或用途来源E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYB-FH)β-胡萝卜素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYB-MID)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QLyLCYB-TSH)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-I209M)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-R210K)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-D219G)β-胡萝卜素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-D223P)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-R229K)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-P230R)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-V241I)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-V243A)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-V256L)番茄红素工程菌本研究构建E. coli BL21 (pTrc99a-crtEBI+pTac15K-QSoNSY-A276T)番茄红素工程菌本研究构建
1.5 发酵培养嵌合体双质粒菌株
将所需发酵菌株按照1%的添加量接种到装液量为33%(体积分数)的含有相应抗性的LB试管培养基中,在37 ℃,200 r/min摇床培养3~4 h,至菌体OD600≈0.6时,按照0.1%(体积分数)的添加量加入100 mmol/L的IPTG诱导菌株高效表达蛋白,随后37 ℃,200 r/min摇床继续培养10~12 h后终止发酵,检测产物。
1.6 HPLC分析
建立完备的检测方法有助于分析发酵产物的组成和目标产物的含量,双质粒工程菌株发酵完成后,依次经过预处理和HPLC对产物进行分析,具体操作如下:
a)制备发酵样品:取发酵完成的菌液2.0 mL至2.0 mL离心管中,12,000 r/min离心3 min,重复操作1次,在离心管中富集到4.0 mL菌液的菌体,移液枪吸取500.0 μL色谱级丙酮[17]至离心管中,用灭菌后的牙签混合均匀,并到涡旋仪上涡旋5 min,后放置于37 ℃水浴锅中,静置20 min,将样品尽可能多的萃取到溶剂丙酮中。随后12,000 r/min离心5 min,将上清液过孔径0.22 μm的尼龙滤膜后取200 μL到含有内衬管的液相小瓶中,做好标记,等待检测。
b)HPLC检测:HPLC以液体为流动相,采用高压输液系统,将具有不同极性的单一溶剂或不同比例的混合溶剂、缓冲液等流动相泵入装有固定相的色谱柱,在柱内各成分被分离后,进入检测器进行检测,从而实现对试样的分析。本实验中利用YMC Carotenoid(C-30)[17-19]类胡萝卜素分析色谱柱对样品进行分析检测,吸光度476 nm,总流速1.0 mL/min,进样量20 μL,柱温35 ℃,流动相为色谱级甲醇和甲基叔丁基醚,采用二元高压梯度洗脱25 min,具体的洗脱方法如表3所示。
表3 HPLC检测产物方法
Table 3 Methods for detection of products by HPLC

检测时间/min甲醇体积分数/%甲基叔丁基醚体积分数/%0^109010202575259010
2 结果与分析
2.1 LyLCYB和SoNSY番茄红素β-环化功能验证
全合成lyLCYB和soNSY基因后,通过基因重组技术构建了质粒pTac15K-LyLCYB和pTac15K-SoNSY,且在产番茄红素的E.coli BL21(DE3)中进行了异源表达,经过发酵,如图1所示,在含有lyLCYB基因的菌株发酵产物中检测到了β-胡萝卜素,而含有soNSY基因的菌种发酵产物中,仅发现番茄红素,没有产生β-胡萝卜素,由此判断,LyLCYB可以使番茄红素环化成β-胡萝卜素,而SoNSY不可以。
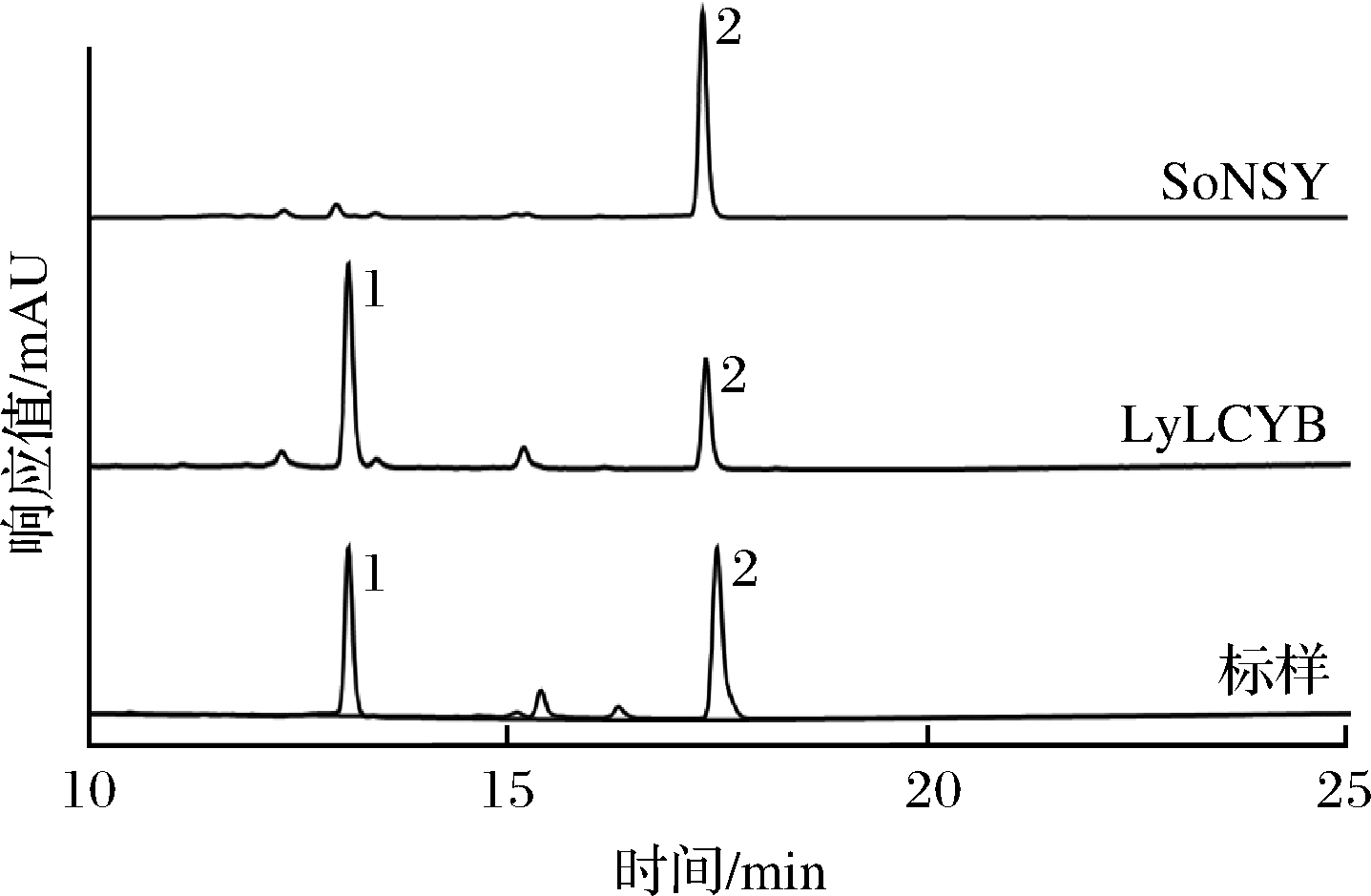
图1 LyLCYB和SoNSY番茄红素β-环化功能分析结果
Fig.1 Results of functional analysis of the LyLCYB and SoNSY lycopene β-cyclization
注:1-β-胡萝卜素;2-番茄红素(下同)。
2.2 LyLCYB和SoNSY的结构域划分
因本研究要在大肠杆菌中实现异源表达,大肠杆菌属原核生物,缺少膜状细胞器无法结合具有膜结合域的酶,因此难以使具有膜结合域的蛋白质产生酶活力[20-21]。而本研究所选的研究对象LyLCYB和SoNSY来源于具有膜状细胞器的真核高等植物,可能具有膜结合域,故首先对LyLCYB和SoNSY进行了膜结合域分析。预测的结果如图2-a所示,二者都不具备膜结合域。这对LyLCYB和SoNSY的大肠杆菌异源表达是十分有利的。
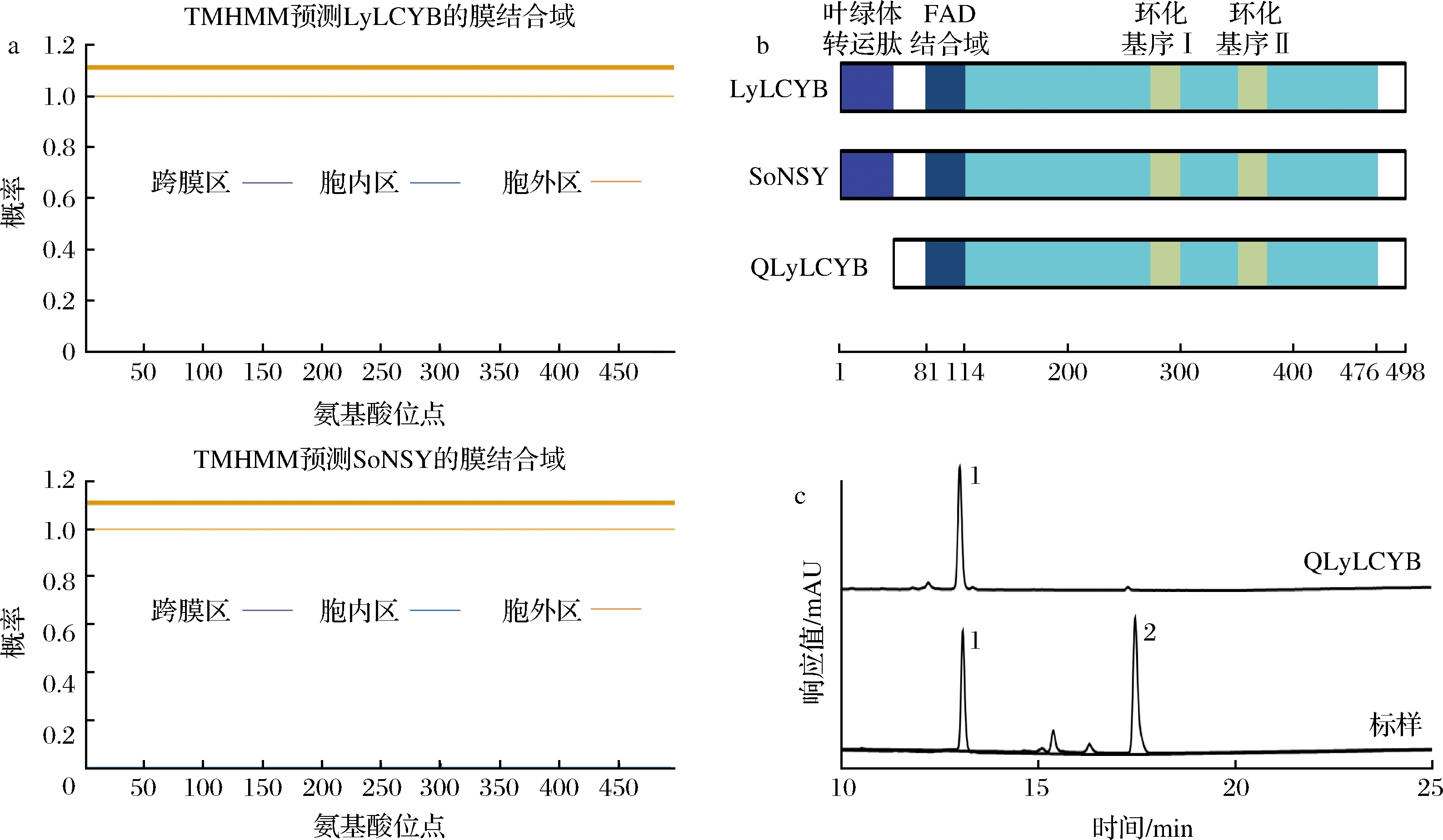
a-TMHMM-2.0预测LyLCYB和SoNSY的膜结合域;b-SMART预测LyLCYB和SoNSY结构域;c-敲除cTP的QlyLCYB基因番茄红素β-环化功能分析
图2 对LyLCYB和SoNSY的氨基酸序列分析及QLyLCYB基因番茄红素β-环化活性分析
Fig.2 Amino acid sequence analysis of LyLCYB and SoNSY and lycopene β-cyclizing activity analysis of QLyLCYB
接下来对含有498个氨基酸的LyLCYB和SoNSY序列进行了结构域划分。如图2-b所示,通过预测发现两个序列都含有自E2到C51的叶绿体转运肽(chloroplast transport peptide, cTP);且Q81-P114肽段均为两基因的FAD结合区域(FAD_binding domain);LyLCYB和SoNSY都包含两个环化保守基序(279~301Cyclase motif Ⅰ和353~367Cyclase motif Ⅱ)和蛋白N端非保守区域(477~498 NB)。考虑到cTP主要起引导作用,在真核生物中cTP完成转运职责后会被切除[22],因此真正发挥活性的天然酶中并不具备cTP,而原核生物体内不具备切除叶绿体转运肽功能的此类肽酶,因此我们敲除了lyLCYB基因中编码cTP的部分,构建了编码QLyLCYB的QlyLCYB突变基因(图2-b)。如图2-c所示,敲除cTP后,QlyLCYB仍旧具备番茄红素β-环化活性。且基于HPLC结果,含有基因QlyLCYB的菌株发酵产物中,β-胡萝卜素产物的峰面积高于含有lyLCYB基因,因此相较于原基因,敲除cTP的QlyLCYB基因环化能力提高。这可能是由于cTP在大肠杆菌中会阻碍酶发挥功能,所以去除了cTP以后,QLyLCYB对番茄红素的环化能力提高。因此本研究后续的实验均在敲除掉cTP的基因上进行。
2.3 影响番茄红素环化功能关键氨基酸的发掘
如图3-a所示,对LyLCYB和SoNSY进行序列比对,发现除cTP外,LyLCYB和SoNSY共有59个差异氨基酸位点。因此,本研究采用构建嵌合体的方式,将QSoNSY序列的部分肽段替换为对应的QLyLCYB氨基酸序列,分区域判断关键氨基酸位点所在的位置,逐步缩小范围。构建了如图3-b所示的4个嵌合体质粒,分别是pTac15K-QLyLCYBa、pTac15K-QLyLCYBb、pTac15K-QLyLCYBc、pTac15K-QLyLCYBd,将这4个嵌合体质粒导入产番茄红素的E.coli BL21(DE3)中,经过发酵分析后,在含有pTac15K-QLyLCYBa、pTac15K-QLyLCYBb、pTac15K-QLyLCYBd这3个质粒的菌株中均检测到了番茄红素和β-胡萝卜素,而含有pTac15K-QLyLCYBc质粒的菌株发酵产物中,仅发现了番茄红素(图3-c)。上述实验结果表明,导致QLyLCYB和QSoNSY对番茄红素环化差异的关键氨基酸位点在pTac15K-QLyLCYBd质粒替换的这一部分上,将这部分肽段命名为CL(N120~K277)。

a-LyLCYB和SoNSY序列比对结果;b-嵌合体构建示意图;c-含嵌合体基因菌株发酵产物HPLC结果
图3 基于LyLCYB和SoNSY序列比对构建的嵌合体功能分析
Fig.3 The analysis of chimeras’ function constructed based on sequences alignment of LyLCYB and SoNSY
在CL肽段内QLyLCYB和QSoNSY共有19个差异氨基酸位点,因此为了进一步缩小影响环化功能的关键氨基酸范围,又将CL肽段进行了分段替换,分别构建了pTac15K-QLyLCYB-FH、pTac15K-QLyLCYB-MID、pTac15K-QLyLCYB-TSH这3个质粒(图4-a),将这3个质粒进行了转化发酵分析后,如图4-b所示,含有这3个质粒的菌株中,仅含有pTac15K-QLyLCYB-FH质粒的菌株发酵产物中检测到了β-胡萝卜素,其他2个在发酵产物中没有发现β-胡萝卜素。依据上述的实验结果,将含有关键氨基酸的肽段范围缩小至FH处,即影响番茄红素环化功能的关键氨基酸在209~279。然而在FH肽段内仍旧有10个差异氨基酸位点(图4-c),因此后续采用定点突变的策略对这10个差异位点的功能进行探究。

a-嵌合体构建示意图;b-含嵌合体基因菌株发酵产物HPLC结果;c-FH片段中10个差异氨基酸位点分布
图4 影响环化功能的关键肽段FH的发掘
Fig.4 Discovery of key peptides FH affecting cyclization function
2.4 关键氨基酸位点219的发掘
为了进一步确定差异的氨基酸位点,将QSoNSY和QLyLCYB在FH区域中的10个差异氨基酸位点209、210、219、223、229、230、241、243、256和276进行突变构建点突变蛋白,即将QSoNSY上的差异位点突变QLyLCYB的对应位置的氨基酸,随后将改造后的基因转化到产番茄红素的大肠杆菌中,经过HPLC检测发酵产物,如图5所示,在含有soNSY-D219G基因的菌株中,检测到了β-胡萝卜素,这表明改造后的soNSY-D219G基因具备了番茄红素β-环化功能。而剩余9个突变位点的发酵产物均只检测到了番茄红素,并未发现β-胡萝卜素,这表明这9个突变体蛋白仍不具备环化番茄红素的能力。在此基础上,突变了LyLCYB的219位点,构建了pTac15K-LyLCYB-G219D质粒,在转化分析后发现其失去了番茄红素的环化功能,在发酵产物中仅检测到了番茄红素(图5)。上述实验结果表明,219位点是导致LyLCYB和SoNSY对番茄红素环化功能差异的关键氨基酸位点。
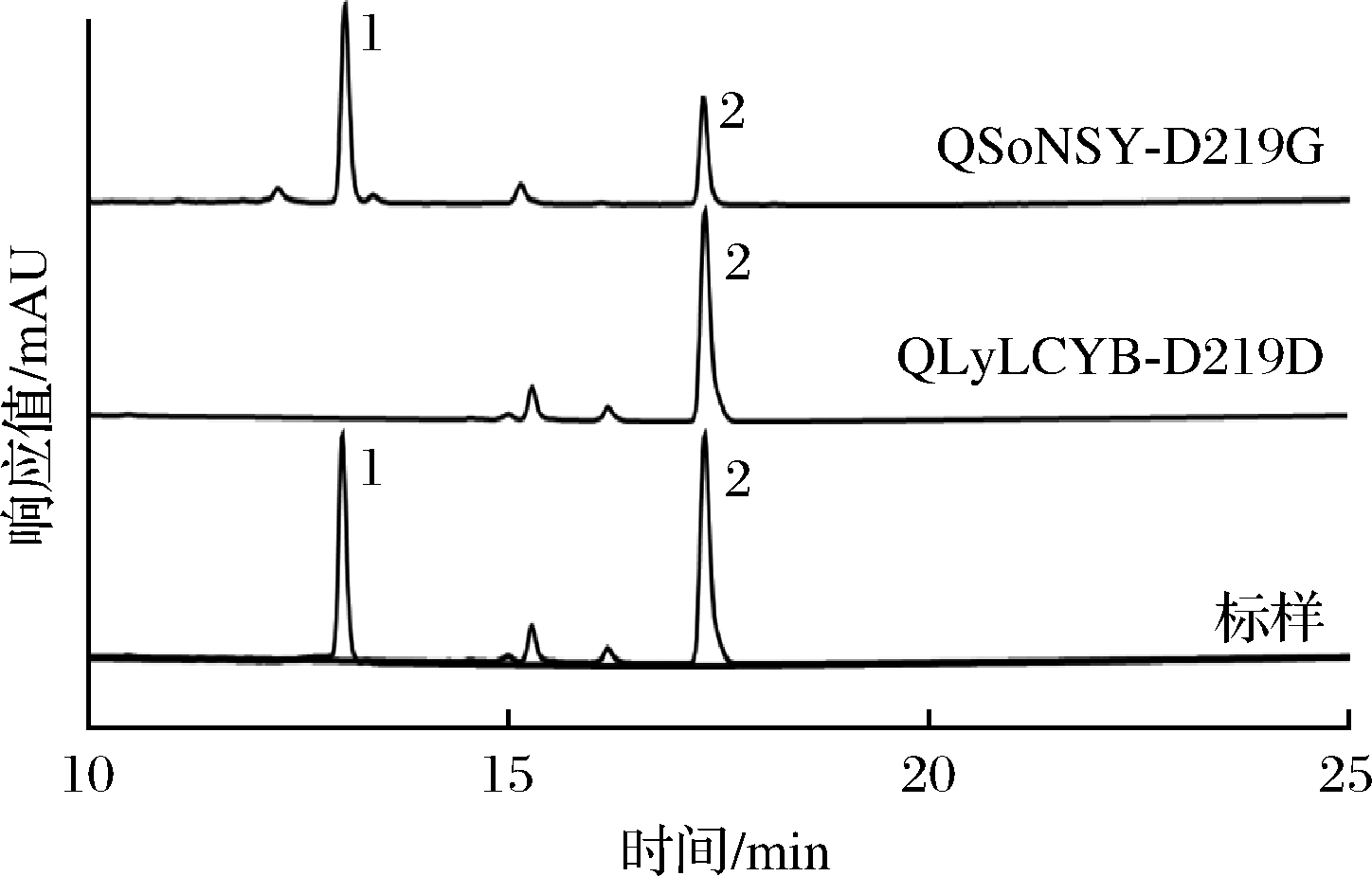
图5 基因QlyLCYB-G219D和QsoNSY-G219D番茄红素β-环化活性分析结果
Fig.5 Results of functional analysis of lycopene β-cyclization of the genes QlyLCYB-G219D and QsoNSY-G219D
3 结论
本研究通过在E.coli BL21(DE3)中构建类胡萝卜素的异源表达系统,利用嵌合体和定点突变的方式探究类胡萝卜素合成酶LyLCYB和SoNSY中影响番茄红素环化功能的氨基酸位点,并且将关键氨基酸位点的范围锁定到FH(209~279)肽段范围内,为进一步探究两个基因的差异位点将FH区域中LyLCYB和SoNSY的10个差异位点进行点突变,最终发现了含有突变体SoNSY-D219G的菌株具备了环化番茄红素的能力,而含有突变体LyLCYB-G219D的菌株环化功能消失,这表明219位点是影响LyLCYB和SoNSY番茄红素环化功能差异的关键位点。本研究为继续探究LyLCYB和SoNSY紫黄质催化功能差异提供了理论基础,同时也为LyLCYB和SoNSY酶作用机制的研究提供参考。
[1] 柳川, 弘志, 徐万祥.最初的酶是蛋白质,还是核酸?[J].世界科学, 1987(8):24-40.
LIU C, HONG Z, XU W X.Is the initial enzyme a protein, or a nucleic acid?[J].World Science, 1987(8):24-40.
[2] 许学农. 酶催化功能的本质研究与进展[J].自然杂志, 1996(6):338-340.
XU X N.Development of the study on catalytic function of enzyme[J]. Nature Magazine, 1996, 18(6):338-340.
[3] PEARSON W R.An introduction to sequence similarity (“homology”) searching[J].Current Protocols in Bioinformatics, 2013. DOI:10.1002/0471250953.bio30/s42.
[4] ZHAO Z L, LIU Z, MAO X Z.Biotechnological advances in lycopene β-cyclases[J].Journal of Agricultural and Food Chemistry, 2020, 68(43):11895-11907.
[5] AL-BABILI S, HUGUENEY P, SCHLEDZ M, et al.Identification of a novel gene coding for neoxanthin synthase from Solanum tuberosum[J].FEBS Letters, 2000, 485(2-3):168-172.
[6] YU Q J, SCHAUB P, GHISLA S, et al.The lycopene cyclase CrtY from Pantoea ananatis (formerly Erwinia uredovora) catalyzes an FADred-dependent non-redox reaction[J].The Journal of Biological Chemistry, 2010, 285(16):12109-12120.
[7] MIALOUNDAMA A S, HEINTZ D, JADID N, et al.Characterization of plant carotenoid cyclases as members of the flavoprotein family functioning with no net redox change[J].Plant Physiology, 2010, 153(3):970-979.
[8] HORNERO-MÉNDEZ D, BRITTON G.Involvement of NADPH in the cyclization reaction of carotenoid biosynthesis[J].FEBS Letters, 2002, 515(1-3):133-136.
[9] BOUVIER F, D’HARLINGUE A, BACKHAUS R A, et al.Identification of neoxanthin synthase as a carotenoid cyclase paralog[J].European Journal of Biochemistry, 2000, 267(21):6346-6352.
[10] CUNNINGHAMF X, SUN Z, CHAMOVITZ D, et al.Molecular structure and enzymatic function of lycopene cyclase from the cyanobacterium Synechococcus sp strain PCC7942[J].The Plant Cell, 1994, 6(8):1107-1121.
[11] LIU Y L, ZENG S H, SUN W, et al.Comparative analysis of carotenoid accumulation in two goji (Lycium barbarum L. and L.ruthenicum Murr.) fruits[J].BMC Plant Biology, 2014, 14:269.
[12] GIOSSI C, CARTAXANA P, CRUZ S.Photoprotective role of neoxanthin in plants and algae[J].Molecules, 2020, 25(20):4617-4633.
[13] SANDMANN G.Antioxidant protection from UV- and light-stress related to carotenoid structures[J].Antioxidants, 2019, 8(7):219.
[14] ALMAGRO ARMENTEROS J J, SALVATORE M, EMANUELSSON O, et al.Detecting sequence signals in targeting peptides using deep learning[J].Life Science Alliance, 2019, 2(5):e201900429.
[15] WATERHOUSE A M, PROCTER J B, MARTIN D M A, et al.Jalview Version 2: A multiple sequence alignment editor and analysis workbench[J].Bioinformatics, 2009, 25(9):1189-1191.
[16] 张桂林, 张亚亭, 姜宏, 等.岩藻黄质关键前体新黄质的代谢工程合成[J].南方水产科学, 2022, 18(2):57-65.
ZHANG G L, ZHANG Y T, JIANG H, et al.Metabolic engineering synthesis of neoxanthin, a key precursor of fucoxanthin[J].South China Fisheries Science, 2022, 18(2):57-65.
[17] 颜少宾, 张妤艳, 马瑞娟, 等.桃果实类胡萝卜素测定方法的研究[J].果树学报, 2012, 29(6):1127-1133.
YAN S B, ZHANG S Y, MA R J, et al.Study the measurement of carotenoids from the peach fruit[J].Journal of Fruit Science, 2012, 29(6):1127-1133.
[18] 严娟, 蔡志翔, 沈志军, 等.黄肉桃果实中类胡萝卜素提取和测定方法研究[J].果树学报, 2015, 32(6):1267-1274.
YAN J, CAI Z X, SHEN Z J, et al.Extraction and analytical methods of carotenoids in fruit of yellow flesh peach[J].Journal of Fruit Science, 2015, 32(6):1267-1274.
[19] 杜奋澄, 许杰纯, 杨雨欣, 等.辣椒果实类胡萝卜素的提取与测定方法研究[J].中国农学通报, 2019, 35(5):41-48.
DU F C, XU J C, YANG Y X, et al.Extraction and measurement methods of carotenoids from pepper fruit[J].Chinese Agricultural Science Bulletin, 2019, 35(5):41-48.
[20] 赵东旭, 冯永君, 王丰, 等.原核生物中心法则形成过程的逻辑分析[J].北京理工大学学报, 2022, 42(11):1105-1126.
ZHAO D X, FENG Y J, WANG F, et al.Logic analysis on the establishment progress of genetic central dogma in prokaryote[J].Transactions of Beijing Institute of Technology, 2022, 42(11):1105-1126.
[21] 吴名文. 原核生物与真核生物[J].生物学通报, 1981,16(6):6-7.
WU M W.Prokaryotes and eukaryotes[J].Bulletin of Biology, 1981,16(6):6-7.
[22] 姚建庄. 叶绿体转运肽进化起源的研究[D].北京:首都师范大学, 2009.
YAO J Z.The evolutionary origin of chloroplast transit peptide[D].Beijing:Capital Normal University, 2009.