乳酸分子结构相对简单,是一种极其重要的生物基化学品,在食品行业,化妆品,医药以及化工等传统行业具有广泛的应用[1],由其合成的聚乳酸在新材料行业也具有重要的应用价值[2]。目前乳酸的生产以微生物发酵为主[3],拟干酪乳杆菌是具有良好乳酸生产能力的工业乳酸菌,能够在高浓度葡萄糖条件下高效生产L-乳酸[4-5],具有广阔的应用前景。
然而,目前对于拟干酪乳杆菌的培养主要使用复合培养基或成分比较复杂的合成培养基[6-7],培养基的复杂性对拟干酪乳杆菌生理特性及其代谢调控的研究增加了困难。在全合成培养基中氨基酸类和维生素类是种类比较多的营养物质,对菌体的生长和代谢具有重要作用。氨基酸对于微生物的作用除了是蛋白质的基础组成单位外,还能在微生物生长过程中提供生长所需的能量,促进微生物能量代谢,更是很多生物体内非常重要的含氮化合物合成前体[8-10]。维生素是细胞多种酶的辅酶,参与维持和调节细胞的生理代谢[11]。因此更加合理的添加这些营养物质,对于优化菌体生长,提高乳酸产量具有重要意义。
本研究旨在基于已有的乳酸发酵全合成培养基,通过对培养基中各种氨基酸以及维生素对拟干酪乳杆菌代谢影响的研究,在保持至少不低于原全合成培养基的底物利用率以及乳酸产率的前提下,尽可能减少培养基组分,提高合成乳酸效率,为今后该菌株的代谢通量定量分析和代谢工程研究奠定基础。
1 材料与方法
1.1 菌种
拟干酪乳杆菌(Lactobacillus paracasei)由本实验室保藏。
1.2 培养基与培养条件
拟干酪乳杆菌经一级与二级斜面培养基活化[12],用50 mL超纯水重悬二级斜面菌体,将15 mL菌悬液转接至装有85 mL MRS培养基的250 mL摇瓶中作为种子瓶[13]。以20%的接种量接种于全合成培养基[14]。摇瓶培养的条件为温度37 ℃,转速130 r/min,培养48 h。2.5 L发酵罐的培养条件为发酵体积1.5 L,搅拌转速150 r/min,温度37 ℃,pH值维持在6.0左右,发酵周期为48 h。
1.3 检测方法
1.3.1 菌体光密度
在Spectrumlab 22 PC分光光度计620 nm下测定吸光值(OD值)。
1.3.2 葡萄糖浓度
采用葡萄糖氧化酶法试剂盒进行测定。
1.3.3 乳酸浓度
采用Agilent高效液相色谱检测,色谱柱为Hi-Plex H (300 mm×7.7 mm);检测波长为210 nm;流动相为0.01 mol/L硫酸;流速为0.4 mL/min;进样量为10 μL;柱温为50 ℃。
1.4 合成培养基优化方法
1.4.1 单一缺失实验
对全合成培养基中氨基酸和维生素进行单一缺失摇瓶实验,确定对菌体生长和乳酸产量有显著影响的组分。
1.4.2 单组分浓度梯度实验
对该组分的添加量设置为初始浓度的倍数,0、0.5、1、2、4倍,其余组分浓度保持不变。
1.4.3 Plackett-Burman实验
该方法在单组分浓度梯度实验的基础上,对每个因素取高(1)、低(-1)2种水平,以考察每种因素的影响水平及影响的显著性,以期从众多影响因素中筛选出显著影响乳酸生产的因素。
1.4.4 最陡爬坡实验
根据Plackett-Burman实验筛选出显著影响合成乳酸的因素,结合各因素效应的正负,设置合适的步长和爬坡方向,进而有效地确定各因素具有最大响应值的浓度范围。
1.4.5 响应面分析实验
采用Box-Behnken实验设计方法,每个因素取3个水平:-1,0,1。建立多元回归方程并计算出各因素的最佳浓度值。Plackett-Burman实验和Box-Behnken实验均通过Design Expert 8.0软件进行实验设计和结果分析。
2 结果与分析
2.1 产L-乳酸的合成培养基中氨基酸的优化
本实验在全合成培养基的基础上对20种氨基酸进行单一缺失实验,发现半胱氨酸、谷氨酸、谷氨酰胺、天冬氨酸、天冬酰胺、酪氨酸、赖氨酸、丙氨酸单一缺失后的菌体生长和乳酸产量降低最多(图1)。对这8种氨基酸进行单组分浓度梯度实验,结果显示(图2)随着氨基酸浓度的增加,其对菌体的生长和产酸存在不同程度的影响,不同氨基酸的结果也存在显著差异。半胱氨酸、谷氨酸、谷氨酰胺的浓度变化对菌体生长和产酸有明显的促进作用,但高浓度的氨基酸导致菌体生长过盛,从而使乳酸生成受到影响。天冬氨酸、天冬酰胺的浓度变化对菌体生长有一定影响,当浓度大于1倍时,菌体生物量无变化,但与乳酸产量呈正相关。酪氨酸、赖氨酸浓度变化对菌体生长影响较小,浓度在1倍以下时,对乳酸生成有促进作用。丙氨酸浓度变化对菌体生长影响较小,低浓度的丙氨酸促进产酸,高浓度的丙氨酸抑制产酸。另外,从氨基酸浓度梯度实验的数据可以看出天冬氨酸和天冬酰胺对于菌体生长和乳酸产量的影响作用相类似,考虑到微生物中广泛存在天冬酰胺酶,可将天冬酰胺转换成天冬氨酸[15],因此将天冬酰胺替换为相同浓度的天冬氨酸,其摇瓶实验的菌体生长、乳酸生成以及糖耗情况与替换前基本一致,谷氨酸与谷氨酰胺之间也同样存在转换作用[16],实验结果也相似,故选择将谷氨酰胺替换成谷氨酸。因此最终确定半胱氨酸、酪氨酸、赖氨酸、丙氨酸、天冬氨酸、谷氨酸作为合成培养基中对生长和产酸有重要作用的氨基酸。

a-单一氨基酸缺失对生长的影响;b-单一氨基酸缺失对乳酸产量的影响
图1 单一氨基酸缺失对菌体生长和乳酸产量的影响
Fig.1 Effects of single omission of amino acid on growth and lactic acid production

a-半胱氨酸;b-谷氨酸;c-谷氨酰胺;d-天冬氨酸;e-天冬酰胺;f-酪氨酸;g-赖氨酸;h-丙氨酸
图2 不同浓度梯度氨基酸对菌体生长和乳酸产量的影响
Fig.2 Effects of different concentrations of amino acid on growth and lactic acid production
2.2 产L-乳酸的合成培养基中维生素的优化
维生素的单一缺失实验同氨基酸,发现生物素、硫胺素、核黄素、烟酸、吡哆醇单一缺失后的菌体生长和乳酸产量降低最多(图3)。对这5种维生素进行单组分浓度梯度实验,结果显示(图4)不同的维生素随着浓度的增加对菌体生长和产酸存在不同的影响。其中菌体生物量与生物素和硫胺素的浓度呈正相关,生物素的效果更加明显,乳酸浓度也随着与生物素、硫胺素的浓度增加而增加,但增加到原2倍浓度之后,乳酸浓度增加趋势渐缓,生物素和硫胺素是微生物体内多种酶系统的辅酶,对糖酵解途径中多种酶的活性有促进作用,能够调节糖代谢,促进菌体生长,加快丙酮酸和乳酸的合成,在生物体的碳代谢循环中起到了至关重要的作用[17-18]。核黄素、烟酸、吡哆醇的浓度变化对菌体生长没有影响,但是对产酸合成有一定促进作用,吡哆醇的浓度在0.5倍时,产酸量达到最大,烟酸及核黄素浓度达到1倍后,乳酸增长乏力,尤其是高浓度烟酸抑制了产酸,产酸量出现了较大的下降。核黄素是细胞黄酶类辅基的组成成分,在细胞的氧化还原反应中具有重要作用[19],烟酸是NAD+和NADH的关键前体物质,NADH作为乳酸脱氢酶的辅酶参与反应[20],因此核黄素和烟酸对于乳酸的生成具有重要的调节作用[21]。吡哆醇在生物体内可转变为吡哆醛,与氨基酸的代谢密切相关[22],间接参与乳酸的代谢调控。因此最终确定生物素、硫胺素、烟酸对生长和产酸有重要作用。
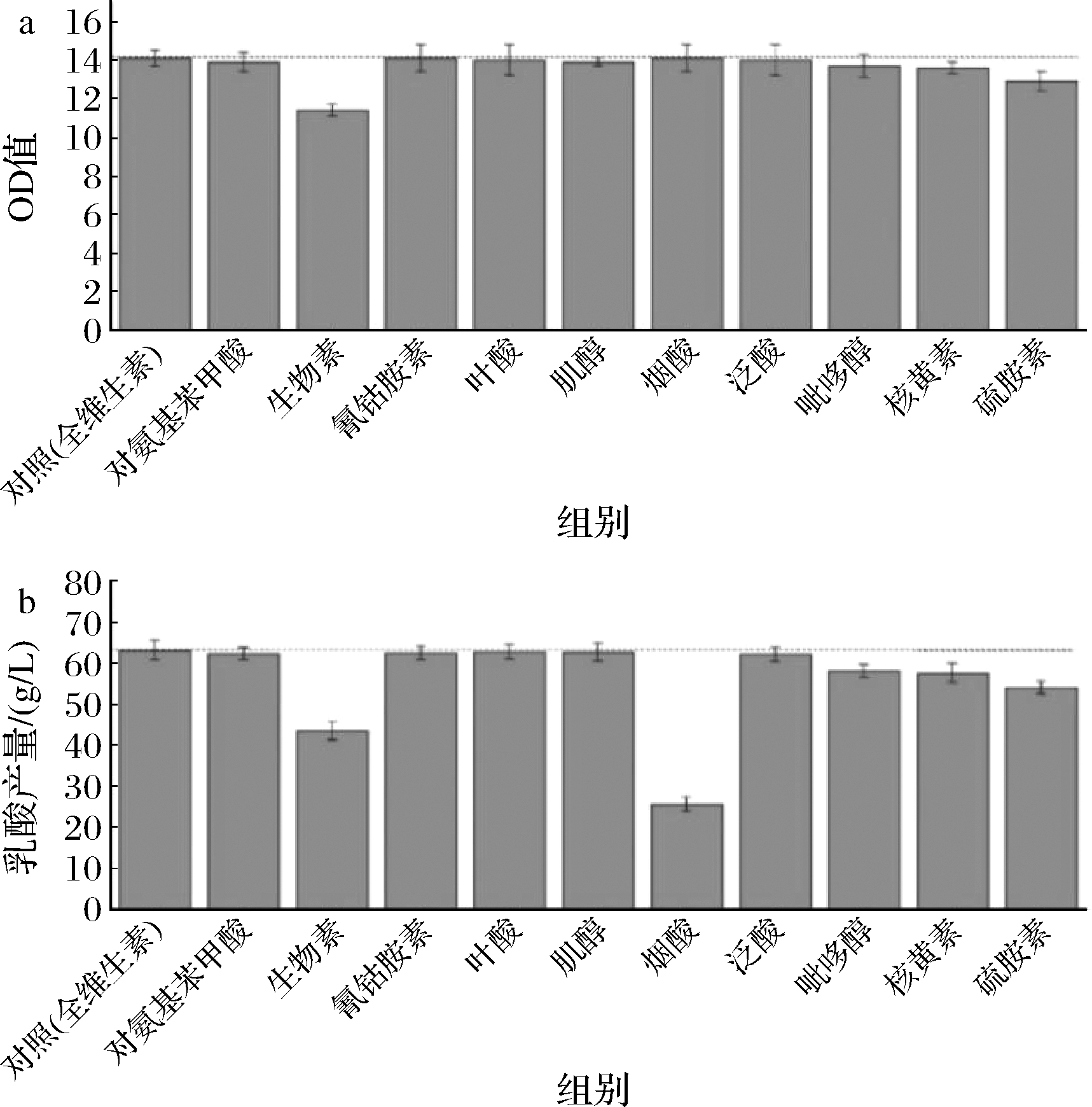
a-单一维生素缺失对生长的影响;b-单一维生素缺失对乳酸产量的影响
图3 单一维生素缺失对菌体生长和乳酸产量的影响
Fig.3 Effects of single omission of vitamin on growth and lactic acid production

a-生物素;b-硫胺素;c-核黄素;d-烟酸;e-吡哆醇
图4 不同浓度梯度维生素对菌体生长和乳酸产量的影响
Fig.4 Effects of different concentrations of vitamin on growth and lactic acid production
2.3 响应面分析优化合成培养基
在对6种氨基酸和3种维生素的单组分浓度梯度进行考察基础上,进而通过响应面分析,建立乳酸生成与影响因素间的回归关系,并最终获得产酸稳定、高效的合成培养基配方。首先通过Plackett-Burman实验分别以原浓度和原0.5倍浓度作为1和-1水平的值,实验设计因素水平及结果见表1,通过显著性分析(表2)发现,因素A(生物素)、H(谷氨酸)、J(天冬氨酸)对乳酸的生成有极显著的影响,且均为正效应,得到线性回归方程为:Y=34.69+5.38A+1.82H+1.68J,R2=0.938 1,因此确定生物素、谷氨酸、天冬氨酸作为下一步研究的影响因素。
表1 Plackett-Burman实验设计与结果
Table 1 Plackett-Burman experiment design and results

实验号A(生物素)B(硫胺素)C(烟酸)D(丙氨酸)E(酪氨酸)F(赖氨酸)G(赖氨酸)H(谷氨酸)J(天冬氨酸)乳酸预测值/(g/L)11-1111-1-1-1140.22111-1-1-11-1140.83-11-111-111137.541-111-1111-141.2511-1111-1-1-137.86-1111-1-1-11-135.97-1-1-11-111-1136.581-1-1-11-111-141.09-1-1-1-1-1-1-1-1-126.410-1-11-111-11137.811-111-1111-1-126.81211-1-1-11-11142.8
表2 偏回归系数及影响因子的显著性分析
Table 2 Significance analysis of partial regression coefficients and factors

因素影响水平平方和贡献值F值Prob(P)>F值显著性A5.3886.9484.48170.400.000 8极显著B0.891.81.54.20.023 5显著C0.080.020.020.040.641 9不显著D0.320.300.300.60.175 5不显著E0.080.020.020.050.641 9不显著F0.120.040.040.080.527 0不显著G0.220.140.140.280.293 9不显著H1.828.248.1520.080.007 1极显著J1.687.927.2816.240.007 8极显著
根据上述实验的结果设计生物素、谷氨酸、天冬氨酸的最陡爬坡路径,由于生物素、谷氨酸、天冬氨酸均为极显著正效应,所以都要沿浓度增大的方向爬坡。结果如表3所示,随着这3个因素浓度的增加,乳酸产量呈现出先升高后降低的趋势,并以第4组实验浓度值作为最陡爬坡实验的中心浓度。
表3 最陡爬坡实验设计及结果 单位:g/L
Table 3 Design and results of the steepest climbing experiment
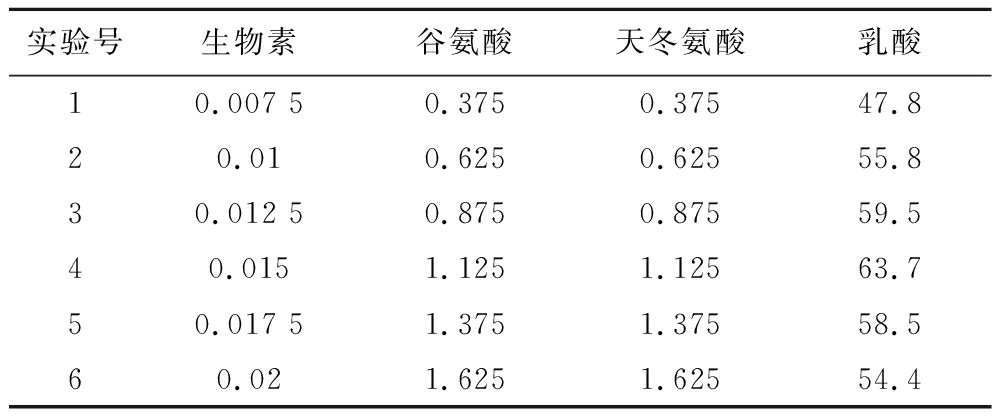
实验号生物素谷氨酸天冬氨酸乳酸10.007 50.3750.37547.820.010.6250.62555.830.012 50.8750.87559.540.0151.1251.12563.750.017 51.3751.37558.560.021.6251.62554.4
根据最陡爬坡实验确定的3个影响因素的中心浓度,对其进行3因素、3水平的响应面实验,分别以第2、4、6组的条件作为-1、0、1水平的值,实验设计与结果见表4。
表4 Box-Behnken设计表及实验结果
Table 4 Design and results of Box-Behnken experiment

实验号a(生物素)b(谷氨酸)c(天冬氨酸)乳酸/(g/L)实验值预测值111054.454.6200063.363.4301155.355.241-1059.859.6500063.863.76-11058.158.47-10157.858.580-1-157.557.59-1-1056.856.71010-158.258.31100063.663.7120-1157.857.81301-158.458.514-10-156.957.01510158.358.3
通过响应面的结果进而获得乳酸浓度对生物素、谷氨酸和天冬氨酸的多元二次回归方程为:Y=63.8+1.31a+0.77b-0.40c+0.23ab-0.025ac-0.87bc-2.74a2-3.49b2-3.19c2,R2=0.970 2,如表5所示,模型的P值小于0.05,失拟项大于0.05,说明模型显著,拟合度较好。由表6回归系数的显著性检验可知,因素a、b、a2、b2对乳酸生成的影响极显著(P<0.01),因素c2对乳酸生成的影响显著(P<0.05),其他因素对乳酸生成的影响不显著。
表5 回归模型的方差分析
Table 5 Variance analysis of regression model
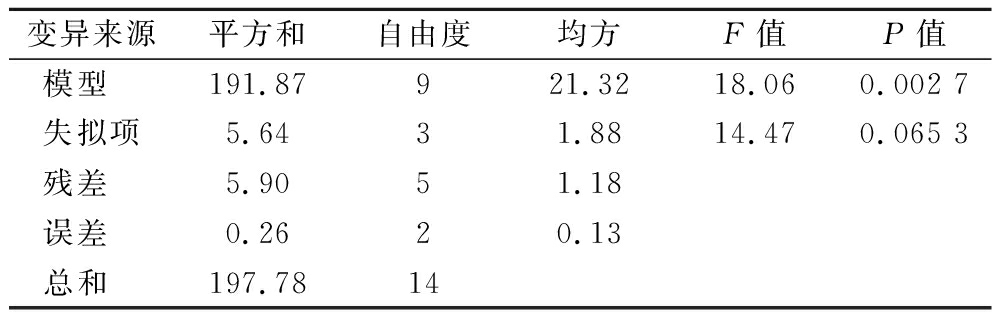
变异来源平方和自由度均方F值P值模型 191.87921.3218.060.002 7失拟项5.6431.8814.470.065 3残差 5.9051.18误差 0.2620.13总和 197.7814
表6 二次模型回归系数显著性检验
Table 6 Significance test for regression coefficients of quadratic model

因素回归系数标准误差F值Prob(P)>F值a1.310.38100.900.000 6b0.770.3811.670.006 9c-0.400.380.920.324 5ab0.230.540.300.604 6ac-0.0250.541.540.269 1bc-0.870.540.210.664 7a2-2.740.5720.940.006 0b2-3.490.5733.290.002 2c2-1.740.579.440.027 7
对二次回归方程求最值后换算可得生物素0.016 g/L,谷氨酸1.128 g/L,天冬氨酸1.120 g/L,在该浓度条件下模型预测的乳酸质量浓度为64.3 g/L,3次验证实验的平均乳酸质量浓度为64.1 g/L,与模型的预测值基本一致,说明模型对乳酸生成拟合效果较好。通过以上单组分浓度梯度实验以及响应面分析实验最终确定优化后合成培养基中的氨基酸和维生素质量浓度(g/L)分别为半胱氨酸0.375,酪氨酸0.125,赖氨酸0.3,丙氨酸0.125,天冬氨酸1.120,谷氨酸1.128,硫胺素0.007 5,烟酸0.007 5,核黄素0.007 5,吡哆醇0.007 5,生物素0.016。
2.4 合成培养基优化前后的摇瓶和2.5 L罐发酵水平比较
合成培养基优化前后摇瓶发酵结果如图5所示,在初始葡萄糖质量浓度为80 g/L的条件下,菌体生长在前期并没有太大变化,12 h之后,优化后的菌体生长速度明显加快,最终OD值为14.5,高于对照的OD值13.2。乳酸产率在前期略有增长,发酵至12 h后,乳酸产量和产率出现了较大幅度的增长,发酵至48 h时乳酸产量从54.8 g/L增加到64.2 g/L,产率从1.04 g/(L·h)增加到1.25 g/(L·h),糖酸转化率从81.9%增加到93.8%。

a-OD值随培养时间的变化;b-乳酸产量随培养时间的变化;c-葡萄糖浓度随培养时间的变化
图5 合成培养基优化前后摇瓶发酵过程特性变化
Fig.5 Profiles of fermentation process with original and optimized chemical defined medium in shake flasks
在初始葡萄糖质量浓度为200 g/L的条件下,用2.5 L发酵罐比较培养基优化前后的乳酸发酵情况(图6),与250 mL摇瓶发酵不同的是,2.5 L罐OD值在24~28 h的时候达到最大值,在这之后OD值平稳中略有下降,优化前的OD值在24 h达到最大,为11.5,优化后OD值在28 h达到最大,为12.7,优化后比优化前提高了10.4%。发酵至12 h后,乳酸生产开始明显增加,发酵至48 h时,乳酸产量从140.2 g/L 增加到165.5 g/L,增长幅度18%,产率从2.83 g/(L·h)增加到3.35 g/(L·h),糖酸转化率从82.4%增加到97.6%,较摇瓶水平进一步提高。

a-OD值随培养时间的变化;b-乳酸产量随培养时间的变化;c-葡萄糖浓度随培养时间的变化
图6 合成培养基优化前后2.5 L反应器发酵过程特性变化
Fig.6 Profiles of fermentation process with original and optimized chemical defined medium in 2.5 L bioreactor
3 结论与讨论
本文优化了拟干酪乳杆菌乳酸发酵合成培养基成分中的氨基酸和维生素组分及浓度。利用单一缺失实验确定了待优化的组分为8种氨基酸和5种维生素,而部分氨基酸和维生素的缺失并不会对菌体生长和产酸造成影响,说明这些组分并不是生长和代谢限制因素,或者本身可以合成相关组分满足菌体生长和产酸需求,因此无需从外界环境中获取。根据浓度梯度实验并通过Plackett-Burman实验确定了显著影响因素为生物素、谷氨酸、天冬氨酸,运用响应面分析方法确定了三者质量浓度分别为0.016、1.128、1.120 g/L。使用优化后的合成培养基进行拟干酪乳杆菌乳酸发酵,其最终乳酸浓度无论是在摇瓶还是在2.5 L发酵罐中都要显著高于优化前,在80 g/L葡萄糖的摇瓶中乳酸质量浓度达到64.3 g/L,在200 g/L葡萄糖的2.5 L发酵罐中乳酸质量浓度达到了165.5 g/L,糖酸转化率分别提高至93.8%和97.6%,在大幅减少氨基酸和维生素种类的情况下,取得了更好的发酵效果。
优化后的培养基中生物素、谷氨酸、天冬氨酸的添加量提高,说明这3种物质对乳酸的生成有重要作用。其中谷氨酸和天冬氨酸是三羧酸循环的重要参与者[23],二者添加量的提高使得在减少碳源流向三羧酸循环的情况下仍能保持三羧酸循环的流量,加上生物素促进糖酵解途径的作用,从而使得更多的碳源流向了乳酸生成。此外,由于代谢流实验需要对胞内外的氨基酸成分进行定量检测,而胞外氨基酸组分的减少排除了不必要氨基酸的检测干扰,也为更有效的计算代谢流提供了极大便利,因此本实验结果为后续拟干酪乳杆菌代谢流定量研究奠定了良好基础。
[1] WEE Y J, KIM J N, RYU H W.Biotechnological production of lactic acid and its recent applications[J].Food Technology and Biotechnology, 2006, 44(2):163-172.
[2] ZHAO X P, LI J C, LIU J C, et al.Recent progress of preparation of branched poly (lactic acid) and its application in the modification of polylactic acid materials[J].International Journal of Biological Macromolecules, 2021, 193:874-892.
[3] HUANG S Y, XUE Y F, YU B, et al.A review of the recent developments in the bioproduction of polylactic acid and its precursors optically pure lactic acids[J].Molecules, 2021, 26(21):6446.
[4] TIAN X W, WANG Y H, CHU J, et al.Exploring cellular fatty acid composition and intracellular metabolites of osmotic-tolerant mutant Lactobacillus paracasei NCBIO-M2 for highly efficient lactic acid production with high initial glucose concentration[J].Journal of Biotechnology, 2018, 286:27-35.
[5] TIAN X W, WANG Y H, CHU J, et al.Metabolite profiling coupled with metabolic flux analysis reveals physiological and metabolic impacts on Lactobacillus paracasei oxygen metabolism[J].Process Biochemistry, 2018, 68:1-11.
[6] 张言慧, 李晓雪, 张悦, 等.利用响应面方法优化产L-乳酸的合成培养基[J].中国酿造, 2012, 31(5):15-20.
ZHANG Y H, LI X X, ZHANG Y, et al.Optimization of a defined medium for the L-lactic acid production by reponse surface analysis method[J].China Brewing, 2012, 31(5):15-20.
[7] BRIGNONE D, RADMANN P, BEHR J, et al.Boosting the growth of the probiotic strain Lactobacillus paracasei ssp.paracasei F19[J].Archives of Microbiology, 2017, 199(6):853-862.
[8] LIN L, DU R B, WANG Y Q, et al.Regulation of auxotrophic lactobacilli growth by amino acid cross-feeding interaction[J].International Journal of Food Microbiology, 2022, 377:109769.
[9] XIAO F, GUO F F.Impacts of essential amino acids on energy balance[J].Molecular Metabolism, 2022, 57:101393.
[10] TOMITA T.Structure, function, and regulation of enzymes involved in amino acid metabolism of bacteria and archaea[J].Bioscience, Biotechnology, and Biochemistry, 2017, 81(11):2050-2061.
[11] JAEHME M, SLOTBOOM D J.Diversity of membrane transport proteins for vitamins in bacteria and archaea[J].Biochimica et Biophysica Acta (BBA)-General Subjects, 2015, 1850(3):565-576.
[12] 孙研, 王永红.不同凝结芽孢杆菌在单一及混合碳源下的发酵特性[J].食品工业科技, 2020, 41(16):74-80.
SUN Y, WANG Y H.Fermentation characteristics of different strains of Bacillus coagulans with single and mixed carbon sources[J].Science and Technology of Food Industry, 2020, 41(16):74-80.
[13] RODR GUEZ DE OLMOS A, GARRO M S.Metabolic profile of Lactobacillus paracasei subsp.paracasei CRL 207 in solid state fermentation using commercial soybean meal[J].Food Bioscience, 2020, 35:100584.
GUEZ DE OLMOS A, GARRO M S.Metabolic profile of Lactobacillus paracasei subsp.paracasei CRL 207 in solid state fermentation using commercial soybean meal[J].Food Bioscience, 2020, 35:100584.
[14] 张悦, 王泽建, 王永红, 等.拟干酪乳杆菌发酵L-乳酸合成培养基的优化[J].生物加工过程, 2011, 9(5):32-37.
ZHANG Y, WANG Z J, WANG Y H, et al.Optimization of defined culture medium for L-lactic acid fermentation by Lactobacillus paracasei[J].Chinese Journal of Bioprocess Engineering, 2011, 9(5):32-37.
[15] ZADEH HOSSEINGHOLI E, NEGHABI N, MOLAVI G, et al.In silico identification and characterization of antineoplastic asparaginase enzyme from endophytic bacteria[J].IUBMB life, 2020, 72(5):991-1000.
[16] SHIRAZIAN P, ASAD S, AMOOZEGAR M A.The potential of halophilic and halotolerant bacteria for the production of antineoplastic enzymes:L-asparaginase and L-glutaminase[J].EXCLI Journal, 2016, 15:268.
[17] SIRITHANAKORN C, CRONAN J E.Biotin, a universal and essential cofactor:Synthesis, ligation and regulation[J].FEMS Microbiology Reviews, 2021, 45(4):fuab003.
[18] WALKER C, RYU S, GIANNONE R J, et al.Understanding and eliminating the detrimental effect of thiamine deficiency on the oleaginous yeast Yarrowia lipolytica[J].Applied and Environmental Microbiology, 2020, 86(3):e02299-19.
[19] TURNER M E, HUYNH K, CARROLL R K, et al.Characterization of the Streptococcus mutans SMU.1703c-SMU.1702c operon reveals its role in riboflavin import and response to acid stress[J].Journal of Bacteriology, 2020, 203(2):e00293-20.
[20] COLOMBIÉ S, SABLAYROLLES J M.Nicotinic acid controls lactate production by K1-LDH:A Saccharomyces cerevisiae strain expressing a bacterial LDH gene[J].Journal of Industrial Microbiology and Biotechnology, 2004, 31(5):209-215.
[21] TERRADE N, MIRA DE ORDU A R.Determination of the essential nutrient requirements of wine-related bacteria from the genera Oenococcus and Lactobacillus[J].International Journal of Food Microbiology, 2009, 133(1-2):8-13.
A R.Determination of the essential nutrient requirements of wine-related bacteria from the genera Oenococcus and Lactobacillus[J].International Journal of Food Microbiology, 2009, 133(1-2):8-13.
[22] ZHANG M X, LI Y, MU Q E, et al.Effects of chlorpyrifos on the metabolic profiling of Bacillus megaterium strain RRB[J].Chemosphere, 2022, 297:134189.
[23] FERN NDEZ M,
NDEZ M, ![]() M.Amino acid catabolic pathways of lactic acid bacteria[J].Critical Reviews in Microbiology, 2006, 32(3):155-183.
M.Amino acid catabolic pathways of lactic acid bacteria[J].Critical Reviews in Microbiology, 2006, 32(3):155-183.