自然界中共生微生物的相互作用对于全球碳、氮循环不可或缺,而且这类相互作用早在几千年前就被用于食品的发酵和保藏[1]。发酵食品微生物间的相互作用可对群落成员产生有益、中性或有害的影响,其中涉及到互利共生:相互作用的微生物间彼此受益;偏害共生:相互作用时一种微生物对另一种产生不利影响且自身生长不受干扰;偏利共生:相互作用时一种微生物对另一种产生有益影响且自身生长不受干扰;竞争:发酵过程中微生物之间争夺营养素和能量;寄生:相互作用的微生物一方受益,一方受害,受害方为受益方提供营养物质而自身生长被抑制。这些相互作用是发酵食品微生物群落组装和演化的重要驱动因素,一定程度上决定了最终产品的食用品质和安全性[1-4]。但是,以上经典理论无法完全解释微生物的相互依赖性和错综复杂的网络关系[5-8],缺乏从菌种水平对微生物交叉喂养等相互作用模式的理解。
微生物群落并不是微生物个体的简单聚集,而是微生物菌种之间、与其他生物及其环境通过多种方式的相互作用(包括资源竞争、生长抑制、交叉喂养、群体感应和基因水平转移等机制)形成的复杂网络生态系统。驱动菌群相互作用的动力不仅仅局限于电子供体的交换和物质代谢通路,超过98%的微生物都是营养缺陷型,需要依赖特定的外源营养物质进行生长,包括氨基酸和维生素等营养物质的交换。微生物的营养缺陷型、合成同一物质的能量需求、对不同营养物质的吸收能力都不同,这些差异深刻影响微生物在菌群中的互作。
交叉喂养,是指一种细菌所分泌的代谢产物(包括碳源、氮源、氨基酸、维生素、核苷酸、电子供体、电子受体和其他生长因子等)被另一种细菌利用和分解代谢。这种代谢物的转移和消耗现象广泛存在于微生物群落中,可提高微生物在资源贫乏的环境中生存的能力,影响到微生物群落组成、结构、进化、毒力和抗生素敏感性等。通过从环境,从不同的微生物或宿主获得的营养可用于抵消生产某些代谢物的能量成本。在空间结构化的群落中,互利合作细菌之间的局部交换增加,从而增强了互惠性。
例如奶酪中微生物依靠真菌菌丝体分泌的化学物寻找合适的栖息地[9];并为响应其他成员分泌的低浓度抗菌物质(如H2O2),减少铁离子转运蛋白的表达[10]。借助多组学等先进技术手段,可以深入理解微生物相互作用时分子信息传递的复杂性,虽然辅助因子在微生物互作过程中具有重要作用,但本文主要关注微生物间电子供体交换、氨基酸及维生素营养缺陷的交叉喂养。
原养型微生物可在环境营养素极度缺乏的情况下合成生长必需的营养物质。但由于空间环境、微生物自身代谢造成的能量消耗及某些代谢途径缺失等因素的影响,自然界中超过98%的微生物都属于营养缺陷型[11-12]。例如除天冬氨酸和谷氨酸外,清酒乳杆菌(Lactobacillus sakei)对其他氨基酸营养缺陷[13]。在需求营养素维持生命活动的驱动下,微生物之间或微生物与环境之间通常会发生营养相互作用,以降低能量消耗的生长模式来维持微生物群落的多样性。此外,在空间结构化环境中,可增加共生微生物之间的局部营养素交换频率,增强代谢产物交叉喂养效应[11]。微生物基因组的差异导致微生物营养需求的特异性,造成了营养缺陷型微生物在选择合作伙伴时具有不同的偏好,这在一定程度上决定了微生物群落的组装和演化[12]。
微生物群落成员的代谢相互作用本质上是高度动态的,即随着发酵的进行,发酵食品营养素结构因微生物代谢反应不断发生改变[14],导致微生物交叉喂养对环境的影响也随时间发生剧烈变化。最近一项针对合成群落的研究表明,在高浓度营养素条件下,微生物相互作用强度增加不利于维持物种多样性,而在低浓度营养素条件下,相互作用强度减弱,但物种多样性增加,其中互作导致的环境pH变化是主要驱动因素[15]。此外,自然发酵香肠[16]、醋[6]、奶酪[17]等微生态的物种多样性随时间变化呈简单到复杂的趋势,这在一定程度上表明随着发酵时间的推移,微生物可利用的营养素总量在减少,需依赖更多合作伙伴提供生长必需的营养素,因此增加了交叉喂养行为,减少了营养竞争行为,并通过改变pH等创造适合合作伙伴生长的环境条件,使群落物种多样性增加[15]。另外,微生物交叉喂养时产生的一系列酶(蛋白酶、脂肪酶等)[18-19]、风味物质(乳酸、醋酸等)[2,20]、氨基酸(谷氨酸、色氨酸等)[8,21]及维生素(钴胺素、叶酸等)[21-22]等可改善发酵食品的感官品质及营养品质。
在白酒、葡萄酒等酒精发酵饮料中,酵母和乳酸菌之间的交叉喂养最为普遍。乳酸菌是酒精发酵中的优势菌属,虽然在发酵过程中对风味化合物的贡献不足,但它们可以与风味物质生产菌(酵母)交叉喂养促进风味产生。例如,在白酒发酵过程中,酿酒酵母(Saccharomyces cerevisiae)能产生3-甲硫基-1-丙醇和二甲基二硫化合物,而布氏乳杆菌(Lactobacillus buchneri)不能产生这两种风味物质。但在它们的共培养中,布氏乳杆菌显示出甲基循环高转录活性,促进了酿酒酵母生产这两种化合物以及蛋氨酸(3-甲硫基-1-丙醇和二甲基二硫化合物的前体)的合成[23]。除此之外,一些酵母菌能够产生或释放能够刺激乳酸菌生长的氨基酸和维生素。例如,在葡萄汁中观察到酿酒酵母释放的氨基酸和其他代谢产物能够刺激植物乳杆菌(Lactiplantibacillus plantarum)的生长[24]。值得注意的是,植物乳杆菌的这种代谢依赖性是单向的,并且在不同的酵母菌株之间是保守的。另一个营养来源是酵母自溶,即较弱的酵母细胞在发酵过程中死亡。这种现象的特点是在葡萄酒苹果酸乳酸发酵过程中酵母死亡释放出额外的氮源,乳酸菌可将其用作营养源[25]。在酵母自溶物的含氮组分中,含有重要的氨基酸,如精氨酸、异亮氨酸、谷氨酸和色氨酸的小组分(<1 kDa)对细菌的生长促进有益。这些交叉喂养促进了非酵母微生物在酒精饮料中的生长,对葡萄酒的品质尤其是芳香特征有巨大影响。
此外,乳品发酵过程中细菌之间也存在广泛的交叉喂养。在奶酪的发酵过程中,乳酸菌通过两种机制促进丙酸菌的生长:a)乳酸菌产生乳酸,乳酸被丙酸菌进一步代谢为丙酸、醋酸和二氧化碳[26];b)乳酸菌水解奶酪蛋白,从而为丙酸菌提供多肽和游离氨基酸。乳酸菌和丙酸菌的交叉喂养具有防腐的作用,如乳酸、丙酸和醋酸、过氧化氢、双乙酰和其他几种低分子代谢物已被证明具有抗菌的作用。在目前的模型中,保加利亚乳杆菌(Lactobacillus delbrueckii subsp.bulgaricus)为嗜热链球菌(Streptococcus thermophilus)提供肽和氨基酸,嗜热链球菌反过来产生甲酸、叶酸和其他嘌呤合成前体,促进保加利亚乳杆菌的生长之间的交叉喂养[27]。这种嗜热链球菌和保加利亚乳杆菌之间的交叉喂养极大地提升了酸奶的最终产品的质量和稳定性。
综上所述,随着营养素可用性的变化,微生物可通过营养相互作用模式塑造发酵食品微生物群落,进而影响发酵食品的食用和安全品质。
为了深入理解微生物群落的动态交互网络如何影响发酵食品群落生态系统,本文主要介绍了发酵食品中微生物电子供体交换,氨基酸、维生素交叉喂养,强调了从时空角度理解微生物动态交互网络的必要性,并探究了有效筛选关键少数菌种和理性调控微生物群落的研究策略。
1 交叉喂养类型
1.1 潜在的交叉喂养
微生物间的相互作用对于群落生态系统的组成和群落功能的发挥必不可少[3,17]。已有研究发现发酵食品中存在菌种间相互作用刺激双方生长繁殖的现象,通过调控此类相互作用可改善发酵食品的食用品质[1-2,28]。例如,奶酪发酵过程中汉逊德巴利酵母(Debaryomyces hansenii)在含盐环境下可促进解脂耶氏酵母(Yarrowia lipolytica)生长,随后酵母菌分解乳酸、蛋白质、脂肪等促进木糖葡糖球菌(Staphylococcus xylosus)和变异微球菌(Micrococcus varians)生长,这两种细菌产生的蛋白酶和脂肪酶具有改善奶酪风味和质地的功能[18-19]。酸奶发酵过程中德氏乳杆菌(Lactobacillus delbrueckii)与嗜热链球菌通过相互作用刺激二者甲酸、丙酸、叶酸等[29-31]关键生长因子的摄取与代谢,该体系的环境因子及其他营养素的可用性也可能影响二者潜在的交叉喂养效果[1]。发酵香肠加工过程中清酒乳杆菌与木糖葡萄球菌之间也存在类似的互动现象,木糖葡萄球菌的代谢产物能促进清酒乳杆菌生长[32]。由于环境和微生物代谢过程中能量消耗等因素的影响,许多微生物存在营养缺陷。因此,发酵食品中普遍存在微生物交叉喂养相互作用。通过研究微生物的代谢特征可为营养缺陷菌寻找合适的互利共生伙伴,为现代食品发酵工业食品发酵剂的筛选和组合提供了理论基础,并对发酵食品微生物交互网络有了一定的了解。但是,简单理解发酵食品中优势微生物通过摄取和产生特定的营养素并刺激其他微生物生长不足以解释微生物群落的动态交叉喂养。组学技术的发展与应用为探索发酵食品微生物间营养素交叉喂养提供了新的路径,使评估微生物动态交互网络成为可能。
1.2 电子供体交换
以往的研究只关注了微生物特定代谢产物的流动,例如酵母菌可以代谢碳水化合物产生葡萄糖和果糖供短乳杆菌(Lactobacillus brevis)和植物乳杆菌利用[33-34],并没有认识到微生物成员之间可以通过级联的方式为下一个成员提供营养素和能量(图1)[12]。因此,有人提出微生物依靠电子供体交叉喂养来的观点来描述微生物群落间碳源的动态流动与交换[35]。例如厌氧微生物降解复杂有机物,代谢生成的有机物通过群落中不同的微生物转化为短链脂肪酸、二氧化碳和甲烷[36]。发酵乳制品中同样存在微生物间电子供体交换的现象,如同型发酵的嗜酸乳杆菌(Lactobacillus acidophilus)在含葡萄糖的环境中生成最终产物乳酸,厌氧生长的薛氏丙酸杆菌(Propionibacterium shermanii)可利用葡萄糖或乳酸作为碳源和能源生成丙酸、乙酸和二氧化碳[20]。乳酸菌和丙酸菌是电子供体交换的典型例子,两者间具有碳源营养链,由乳酸菌分解代谢乳糖和葡萄糖等产生乳酸,乳酸将作为丙酸菌生长及产生丙酸和乙酸的优先碳源[2]。但是共培养中乳酸的存在无法解释其对丙酸菌所有的积极作用,乳酸菌和丙酸菌的其他代谢产物以及群落成员的影响都可能促进双方生长繁殖[20]。虽然电子供体交换是微生物生态学中公认的驱动力,但是单独的电子供体交换理论忽略了微生物间的高阶相互作用[37],不足以解释微生物网络中的动态交叉喂养[8],。因此这样的理论不能用于高效筛选组合关键微生物。最近的研究基于多组学技术和计算机模型提出“结构可达性”[38]与“结构可控性”[39-40]的概念,提供了理解微生物间营养物质需求和流动的新视角,帮助我们多尺度构建微生物动态交互网络并识别任何微生物群落中的最小驱动物种,为发酵食品菌种的筛选与组合打开了新的大门。
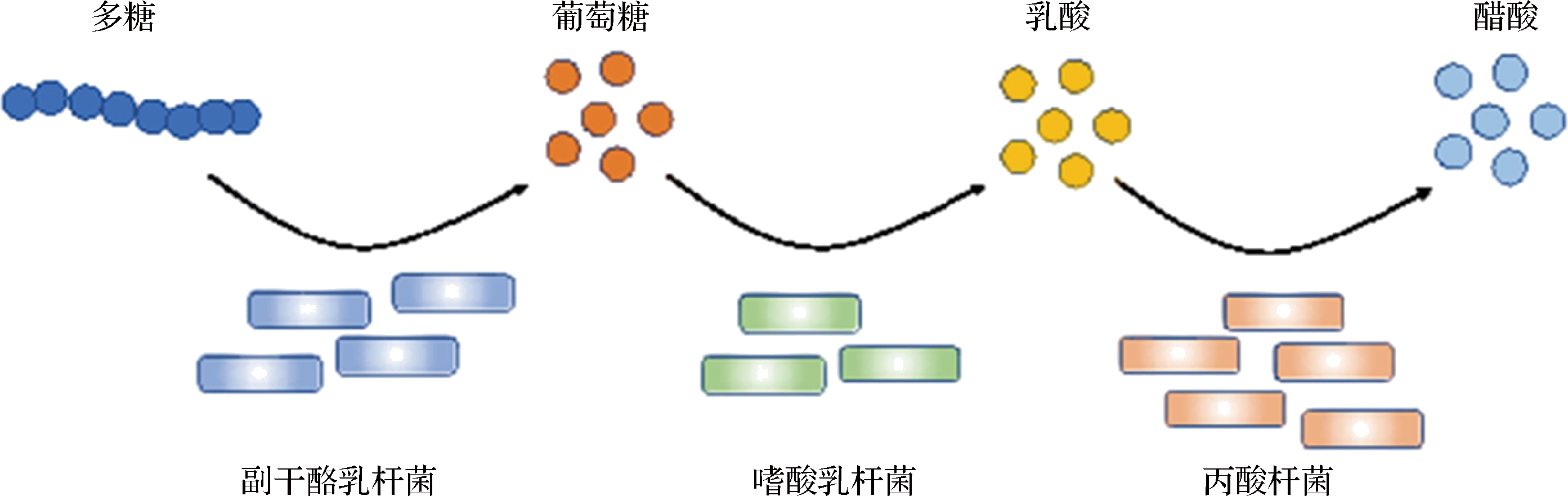
图1 发酵食品微生物电子供体交换[12,20]
Fig.1 Microbial electron donor exchange in fermented foods[12,20]
1.3 氨基酸与维生素营养缺陷
除了基于电子供体的交叉喂养外,微生物群落生态系统中氨基酸和维生素的交换同样可以促进群落组装[41]。最近一项针对949种真细菌的基因测序发现,76%的真细菌至少不能合成25种关键代谢产物中的1种[42]。另外,营养缺陷不仅存在于共生菌,还存在于自由生长的细菌。微生物的适应性优势和遗传漂变是造成营养缺陷的主要因素[11],其中包括微生物对某些代谢成本和能量需求较大的氨基酸和B族维生素营养缺陷,如色氨酸、组氨酸、酪氨酸以及维生素B12等,需从外界摄取这类营养素以获得自身生长优势[11,43]。因此,营养缺陷型细菌通常依赖群落中其他成员或周围环境提供其生长必需的营养素,或进一步与其他微生物或环境形成不同类型的共生体维持各项生命活动(图2)[44-45]。发酵食品中酵母菌能够代谢分泌氨基酸和维生素供细菌利用,而细菌同样会产生次级代谢产物供酵母菌使用[46-47]。例如在开菲尔(kefir)发酵体系中,条件培养大麦乳酸杆菌(Lactobacillus hordei)存在氨基酸和维生素营养缺陷,进一步的共培养发现酿酒酵母可为大麦乳酸杆菌提供亮氨酸、异亮氨酸、甲硫氨酸、色氨酸、缬氨酸和苯丙氨酸以及维生素B6等营养物质[21]。近期的多组学研究可从基因水平为酵母菌和细菌交叉喂养提供新见解。例如利用宏基因组联合蛋白组研究大麦乳酸杆菌与酿酒酵母之间的互动机制,通过共培养发现酿酒酵母谷氨酰胺分泌量显著增加,同时乳酸菌氨基酸透性酶基因表达及其他氨基酸生物合成途径显著上调,以摄取酵母菌分泌的氨基酸并转化利用,这种调控基因表达直接摄取氨基酸的方式可以减少自身能量消耗。另外,大麦乳酸杆菌利用酿酒酵母代谢产生核黄素,通过自身连接嘌呤代谢和戊糖磷酸途径使合成核黄素的相关酶(如核黄素合成酶)基因表达下调[22]。此外,基于转录组分析的共培养发现酸奶中嗜热链球菌和保加利亚乳杆菌之间存在氨基酸、脂肪酸、叶酸的交叉喂养,这有助于保加利亚乳杆菌嘌呤核苷酸的合成[48]。微生物可根据环境营养素的可用性调控自身基因表达缓解能量消耗产生的生存压力,随着共生体生理状态及环境营养素的变化,微生物间的交叉喂养也呈现出动态变化的规律[12]。简单理解发酵食品微生物局部时间点氨基酸、维生素等营养物质的交叉喂养及内在机制不足以全面反映微生物交叉喂养的动力学问题。因此,需在初步了解微生物营养缺陷及基因表达变化的基础上获取更多时序数据重构微生物交互网络,帮助理解微生物营养相互作用,并挖掘和操控网络节点的最小驱动物种以此来调控微生物群落的动态变化。
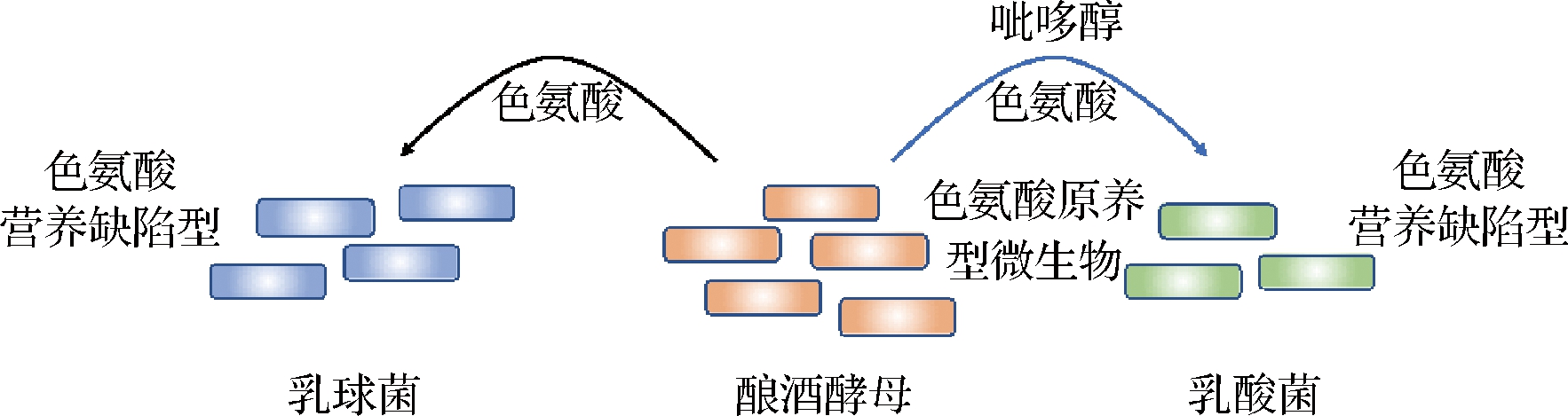
图2 发酵食品微生物氨基酸和维生素交叉喂养[8,21]
Fig.2 Microbial amino acids and vitamin cross-feeding in fermented foods[8,21]
2 动态交叉喂养
2.1 营养素的动态变化——时间角度
大量营养缺陷型细菌需要从其他微生物或环境中获取营养素,且这些营养素必须在一定时期内持续可用。微生物仅通过电子供体交换获得碳源和能量不能满足生理需求,还需依赖合作伙伴或者栖息环境提供的必要营养素。已有研究聚焦于发酵食品微生物群落特征及代谢网络结构,以寻找调控发酵食品品质的核心菌群[6,49-50],但尚不清楚核心共生菌群之间能量和营养素的定向流动是否随时间变化。例如随着发酵时间的推移,环境等因素的变化是否造成某些微生物的丰度降低或升高[51],从而直接影响其合作伙伴次级代谢产物的可用性[25,52]。虽然评估营养素动态变化对微生物交叉喂养的互动存在巨大挑战,但利用合成微生物互作模型可获得对群落组装和动态交叉喂养的新见解。最近一项研究基于代谢动力学揭示了酵母菌和乳酸菌之间的氨基酸交叉喂养:研究者首先设计了一种简单但仍保留许多复杂性的培养基(含35种成分,包括8种氨基酸,精氨酸,天冬酰胺,组氨酸,异亮氨酸,亮氨酸,蛋氨酸,缬氨酸和酪氨酸,以铵为氮源),获得酵母菌和乳酸菌(Lactiplantibacillus plantarum和Lactococcus lactis)的互作模型。随后靶向代谢组研究发现,0~18 h内酵母菌生长繁殖使胞外氨基酸(如谷氨酰胺、谷氨酸和色氨酸等)由缓慢增加至迅速上升,乳酸菌摄取利用这类氨基酸,6 h内氨基酸含量缓慢减少,当乳酸菌数量持续增加,此后30 h内胞外氨基酸浓度又降至初始值。在环境营养素可满足酵母菌-乳酸菌共生的条件下,通过氨基酸交叉喂养可以显著增加合成群落中乳酸菌的数量(酵母菌数量略增加),并在自然发酵体系(如葡萄酒)中得到很好的验证[8,53],这表明营养素的交叉喂养有助于塑造微生物群落。
最后,发酵食品中微生物交叉喂养是时间上持续变化的过程,因此如何调控并稳定微生物动态交叉喂养存在巨大挑战。有研究表明,无论是时间上连续或间歇施加控制信号,需要控制的最小驱动物种集合是一致的[38]。对于发酵食品而言,间歇控制显然比连续控制更方便,例如通过在特定的发酵时点改变发酵体系中微生物可利用的营养素比例[8]或加入某些外源微生物[54]等方式操控最小驱动物种,改变群落的交叉喂养结构,最终调控发酵食品的品质。通过时序数据建立发酵食品微生物交叉喂养动态网络并间歇操控最小驱动物种对于发酵剂的选择与发酵过程的调控具有重要作用,但要把该控制理论转化到应用还有很长一段路要走。
2.2 环境的空间结构——空间角度
微生物群落的空间结构是交叉喂养的决定因素,可以推动群落动态演变[55-56]。相比空间非结构化环境,空间结构化环境更容易使微生物接收到合作伙伴反馈的信息,进而发生交叉喂养相互作用,并可能与合作伙伴间产生“积极分类”,以维持更长久的交叉喂养关系[57-59]。为了验证这一假设,有研究测试了基因缺陷工程菌不动杆菌(Acinetobacter baylyi)和大肠埃希氏杆菌(Escherichia coli)专性组氨酸和色氨酸交叉喂养,实验发现在结构化环境中营养缺陷型菌株间优先建立交叉喂养的合作关系,从而限制了非合作菌株从中获取营养素[60]。此外,枯草芽孢杆菌(Bacillus subtilis)和印度梨形孢(Piriformospora indica)的共培养发现,二者只有在空间结构化环境中共培养才能实现连续生长[61]。然而,发酵食品中的微生物通常生长在无规律的空间非结构化环境,这会干扰微生物的分布及其对营养素的可用性,最终影响群落微生物的交叉喂养模式[62]。受水分、酸度、营养素环境因素的分布等影响,不同空间层次分布着不同的微生物群落,各个群落都存在各自的动态交叉喂养结构。例如白酒窖池中的谷物发酵,有研究者通过非培养技术(PCR-变性梯度凝胶电泳等)测定了窖池不同部位微生物差异,发现耐酸乳酸菌(Lactobacillus acetotolerans)等大量乳酸菌占据了窖池的上层空间,简单芽胞杆菌(Bacillus simplex)占据中层空间,阴沟肠杆菌(Enterobacter cloacae)占据底层空间,这一分布模式导致窖池上层产生更多乙醇,下层产生更多酸性物质[57]。尽管该研究不足以揭示窖池不同部位的微生物群落特征及代谢差异,但获得了不同空间营养素分布状况及微生物群落组装的规律。微生物和营养素的分布具有环境依赖性,为争夺“领土”菌种间将发生紧密的合作或激烈的斗争以满足自身生长,不同群落中的优势菌群往往对群落中大多数微生物的生长繁殖有一定影响[61],对其进行人为操控可引导群落内的纵向动态相互作用向有利于改善微生物生存环境的方向发展,如改善发酵食品的微生态环境优化产品品质。
3 探究微生物动态交互网络的方法
目前主要基于多组学技术绘制微生物共现网络以寻找发酵食品的核心菌群[6,17,49,63],并通过参考以往的经验选择菌群中的关键少数核心菌种以验证其对发酵食品的贡献[64]。但这种方法不能确定微生物间的动态交叉喂养关系,结果存在一定的偶然性,筛选微生物群落中关键少数菌种的效率低。因此需引入最小驱动物种这一概念,通过多组学模型和数据预测建立发酵食品微生物动态交互网络,并筛选可调控整个网络的关键节点即最小驱动物种关键少数菌种,最后在发酵食品中建立共培养实验验证关键少数菌种的营养相互作用关系及对发酵食品品质的贡献(图3)。
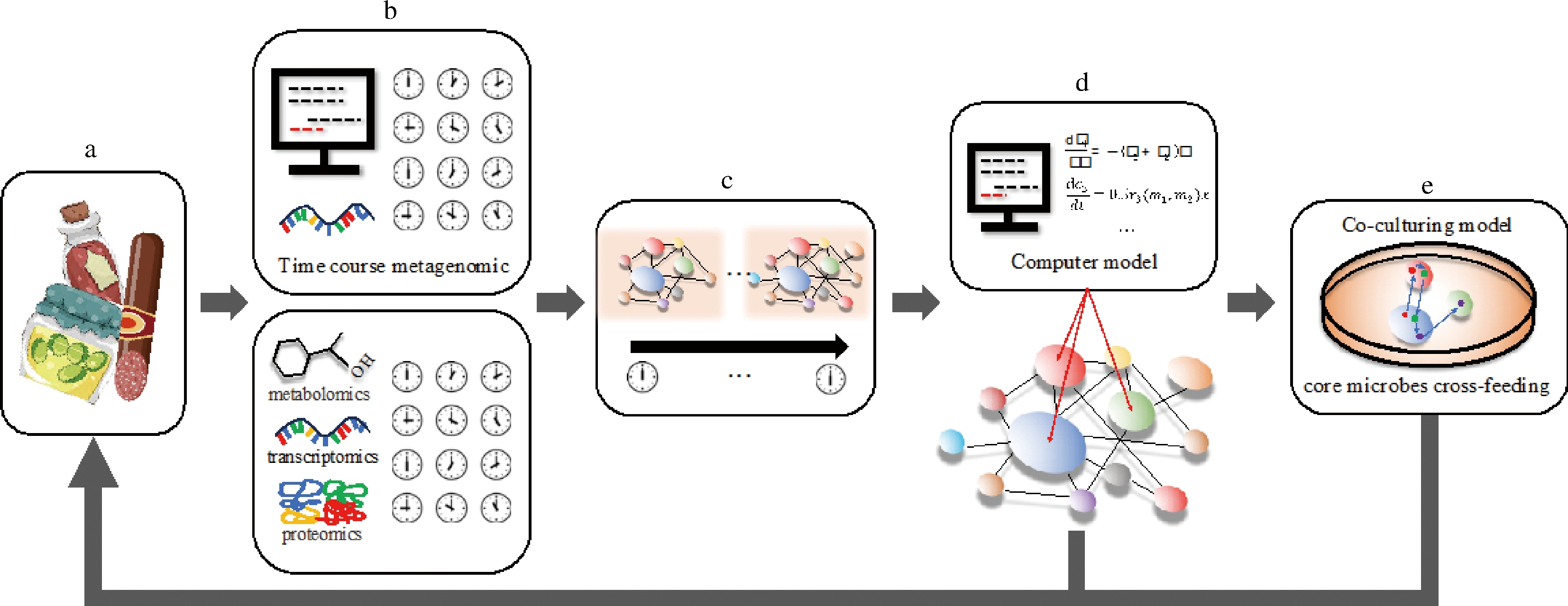
a-发酵食品;b-组学技术对发酵过程的实时监控;c-发酵过程中菌群互作网络的时序性变化;d-核心微生物群落的挖掘;e-模拟体系中核心微生物群落相互作用
图3 发酵食品中微生物动态交互网络及核心菌群筛选的研究策略
Fig.3 The strategy for microbial dynamic interactive network and screening of core community in fermented food
3.1 实验技术应用
了解发酵食品微生物动态交互网络,首先必须明确微生物群落的组成和功能特性。近年来逐渐积累的公共数据库以及宏基因测序深度和精度不断提升,有助于高分辨率纵向动态分析发酵食品微生物群落组成和功能的变化。另外,近乎完整的微生物基因组聚类分析宏基因组的分箱技术,可以从菌株水平鉴定营养缺陷[65-67]。在此基础上,联合宏转录组、宏蛋白组和宏代谢组技术,采用高通量测序、质谱方法等从mRNA、蛋白和代谢水平研究发酵食品微生物组的功能组成及代谢能力,实时监测某一条件或某一时刻微生物群落的代谢网络并整合时间序列网络图谱,帮助解析网络中的动态交叉喂养(图3-b,图3-c)[65-66]。虽然时序性多组学数据能够揭示发酵过程中微生物群落动态演化,但群落内(间)的分子相互作用容易被大量数据淹没,仅用多组学分析很难了解生态因素在整个发酵过程中的核心作用。即便提高测序深度,也很难检测到群落中的稀有物种[68]。因此,完善微生物交互网络还需借助培养组学,利用多种培养条件培养自然发酵食品中的难培养细菌,挖掘更多潜在功能微生物,分析它们的代谢特征,寻找未知功能序列等[68-69]。此外,共培养实验限定为有限种类的微生物,并根据微生物生长环境(pH变化、营养素分布等)合理设置实验室内共培养条件可用于鉴定可培养微生物和潜在功能微生物间的交叉喂养及内在机理,帮助理解微生物群落的组装和演化(图3-e)[8,70]。
3.2 建立模型
构建基于基因组序列注释和生化信息整合的基因组规模代谢网络为全面理解和理性调控微生物代谢功能提供了诸多方便,可以从代谢物和代谢流水平找到调控微生物代谢功能的关键因子,发现承担绝大多数代谢途径的关键少数菌种,反映参与微生物生理代谢的所有化合物和酶之间的相互作用,涉及到基因组、蛋白质和酶、代谢产物和代谢途径、生化反应等诸多数据。此外,通过代谢流平衡分析可以预测不同生长条件下微生物的代谢表型及代谢产物的定向流动,帮助解析微生物间的交叉喂养网络[71]。基因组规模代谢网络模型的构建需要基因组、蛋白质、代谢物、代谢途径等多种数据库联合使用,设计合理的动态模型是精准构建庞大数据之间关联性的有效方法。基于计算和数据理论的动态模型可以将复杂的时序性高通量数据以数学表达式的方式呈现,用于描述微生物代谢网络的动态并加以调控[72],建模的方法包括排序不等式[73]等。此外,基于主体的模型已被用于模拟微生物之间的相互作用,预测微生物对底物的吸收和转化反应,微生物之间的反馈机制以及外部条件对群落的影响,但该模型受限于微生物的数量,难以描述复杂微生物群落的动态[74-75];基于常微分方程(ODEs)的模型借助动力学参数常用于预测纯培养或共培养微生物的动态(涉及代谢物的消耗和生产),探究微生物对不同营养物质的偏好,帮助理解微生物如何选择合作伙伴,该模型的缺点是需要根据原位测定的数据决定模型的参数,因此仅限于研究少数微生物的相互作用[75]。由于不同模型具有特定的优缺点,因此模型的选择取决于研究目标。研究发酵食品的最终目的是从菌株水平了解群落生态系统的组装和演化,为此,基因组规模的代谢模型可作为最优选择,其他动力学模型予以辅助,并通过实验验证不断将模型优化,获取最佳预测效果。
4 总结与展望
虽然微生物营养缺陷引起的交叉喂养对塑造发酵食品微生物群落具有重要贡献,但仍缺乏菌株水平交叉喂养的纵向研究,微生物间复杂的互作动力学仅在少数共培养实验中得到证实[8,76]。宏基因组测序深度不足及不能分辨菌种的存活状况阻碍了研究者从菌株水平判断微生物交叉喂养网络[66]。由于营养素可用性及其在微生物间的流动会随时间剧烈变化[12],鉴定自然发酵食品微生物群落中的动态交互作用极具挑战,理解自然发酵食品中微生物的高阶相互作用将更加依赖于培养组学对未知菌种的挖掘[69],多组学技术解析微生物动态交互网络的纵向研究[66]以及基于高级计算工具的数据分析[60]和动力学模型的建立[72]。目前寻找自然发酵食品核心菌群仅关注其对发酵风味、安全等属性的还原程度[6,50,63-64],影响品质属性的机制大多是未知的。尽管商业发酵剂保障了发酵食品安全性和稳定性,但风味和口感不足以媲美自然发酵食品[3,77]。为了解决以上问题,未来应更多从生态学角度理解发酵食品,在建立发酵食品菌株水平动态交互网络的基础上,采用计算机建模的方式寻找并操控网络中的最小驱动物种,最终在实践中达到精确完美调控发酵食品菌群动态交互网络的目的。
[1] SIEUWERTS S, DE BOK F A M, HUGENHOLTZ J, et al.Unraveling microbial interactions in food fermentations:From classical to genomics approaches[J].Applied and Environmental Microbiology, 2008, 74(16):4997-5007.
[2] SMID E J, LACROIX C.Microbe-microbe interactions in mixed culture food fermentations[J].Current Opinion in Biotechnology, 2013, 24(2):148-154.
[3] IVEY M, MASSEL M, PHISTER T G.Microbial interactions in food fermentations[J].Annual Review of Food Science and Technology, 2013, 4(1):141-162.
[4] WINTERS M, PANAYOTIDES D, BAYRAK M, et al.Defined co-cultures of yeast and bacteria modify the aroma, crumb and sensory properties of bread[J].Journal of Applied Microbiology, 2019, 127(3):778-793.
[5] FREY-KLETT P, BURLINSON P, DEVEAU A, et al.Bacterial-fungal interactions:Hyphens between agricultural, clinical, environmental, and food microbiologists[J].Microbiology and Molecular Biology Reviews: MMBR, 2011, 75(4):583-609.
[6] WANG Z M, LU Z M, SHI J S, et al.Exploring flavour-producing core microbiota in multispecies solid-state fermentation of traditional Chinese vinegar[J].Scientific Reports, 2016, 6(1): 26818.
[7] WOLFE B E, DUTTON R J.Fermented foods as experimentally tractable microbial ecosystems[J].Cell, 2015, 161(1):49-55.
[8] PONOMAROVA O, GABRIELLI N, SÉVIN D C, et al.Yeast creates a niche for symbiotic lactic acid bacteria through nitrogen overflow[J].Cell Systems, 2017, 5(4):345-357.e6.
[9] ZHANG Y C, KASTMAN E K, GUASTO J S, et al.Fungal networks shape dynamics of bacterial dispersal and community assembly in cheese rind microbiomes[J].Nature Communications, 2018, 9(1):336.
[10] HERVE-JIMENEZ L, GUILLOUARD I, GUEDON E, et al.Postgenomic analysis of Streptococcus thermophilus cocultivated in milk with Lactobacillus delbrueckii subsp. bulgaricus:Involvement of nitrogen, purine, and iron metabolism[J].Applied and Environmental Microbiology, 2009, 75(7):2062-2073.
[11] D’SOUZA G, SHITUT S, PREUSSGER D, et al.Ecology and evolution of metabolic cross-feeding interactions in bacteria[J].Natural Product Reports, 2018, 35(5):455-488.
[12] ZENGLER K, ZARAMELA L S.The social network of microorganisms—How auxotrophies shape complex communities[J].Nature Reviews Microbiology, 2018, 16(6):383-390.
[13] CHAMPOMIER-VERG S M C, MAGUIN E, MISTOU M Y, et al.Lactic acid bacteria and proteomics:Current knowledge and perspectives[J].Journal of Chromatography B, 2002, 771(1-2):329-342.
S M C, MAGUIN E, MISTOU M Y, et al.Lactic acid bacteria and proteomics:Current knowledge and perspectives[J].Journal of Chromatography B, 2002, 771(1-2):329-342.
[14] FILANNINO P, DI CAGNO R, GOBBETTI M.Metabolic and functional paths of lactic acid bacteria in plant foods:Get out of the labyrinth[J].Current Opinion in Biotechnology, 2018, 49:64-72.
[15] RATZKE C, BARRERE J, GORE J. Strength of species interactions determines biodiversity and stability in microbial communities. Nature Ecology &Evolution, 2020, 4(3): 376-383.
[16] FERROCINO I, BELLIO A, GIORDANO M, et al. Shotgun metagenomics and volatilome profile of the microbiota of fermented sausages. Applied and Environmental Microbiology, 2018, 84(3): e02120-e02117.
[17] WOLFE B E, BUTTON J E, SANTARELLI M, et al.Cheese rind communities provide tractable systems for in situ and in vitro studies of microbial diversity[J].Cell, 2014, 158(2):422-433.
[18] ADDIS E, FLEET G H, COX J M, et al.The growth, properties and interactions of yeasts and bacteria associated with the maturation of Camembert and blue-veined cheeses[J].International Journal of Food Microbiology, 2001, 69(1-2):25-36.
[19] VAN DEN TEMPEL T, JAKOBSEN M.The technological characteristics of Debaryomyces hansenii and Yarrowia lipolytica and their potential as starter cultures for production of Danablu[J].International Dairy Journal, 2000, 10(4):263-270.
[20] LIU J A, MOON N J.Commensalistic interaction between Lactobacillus acidophilus and Propionibacterium shermanii[J].Applied and Environmental Microbiology, 1982, 44(3):715-722.
[21] STADIE J, GULITZ A, EHRMANN M A, et al.Metabolic activity and symbiotic interactions of lactic acid bacteria and yeasts isolated from water kefir[J].Food Microbiology, 2013, 35(2):92-98.
[22] BECHTNER J, XU D, BEHR J, et al.Proteomic analysis of Lactobacillus nagelii in the presence of Saccharomyces cerevisiae isolated from water kefir and comparison with Lactobacillus hordei[J].Frontiers in Microbiology, 2019, 10(325).DOI:10.3398/fmich.2019.00325.
[23] LIU J, WU Q, WANG P, et al. Synergistic effect in core microbiota associated with sulfur metabolism in spontaneous Chinese liquor fermentation. Applied and Environmental Microbiology, 2017, 83(24): e01475-e01417.
[24] PONOMAROVA O, GABRIELLI N, SÉVIN D C, et al. Yeast creates a niche for symbiotic lactic acid bacteria through nitrogen overflow. Cell Systems, 2017, 5(4):345-357.e6.
[25] JESSICA L, MARIA M, ALBERT M, et al. Saccharomyces and non-Saccharomyces competition during microvinification under different sugar and nitrogen conditions. Frontiers in Microbiology, 2016, 7: 1959.
[26] SMID E J, LACROIX C. Microbe-microbe interactions in mixed culture food fermentations. Current Opinion in Biotechnology, 2013, 24(2): 148-154.
[27] SIEUWERTS S, MOLENAAR D, VAN HIJUM S A F T, et al. Mixed-culture transcriptome analysis reveals the molecular basis of mixed-culture growth in Streptococcus thermophilus and Lactobacillus bulgaricus. Applied and Environmental Microbiology, 2010, 76(23): 7775-7784.
[28] VILJOEN B C.The interaction between yeasts and bacteria in dairy environments[J].International Journal of Food Microbiology, 2001, 69(1-2):37-44.
[29] CRITTENDEN R G, MARTINEZ N R, PLAYNE M J.Synthesis and utilisation of folate by yoghurt starter cultures and probiotic bacteria[J].International Journal of Food Microbiology, 2003, 80(3):217-222.
[30] COURTIN P, RUL F.Interactions between microorganisms in a simple ecosystem:Yogurt bacteria as a study model[J].Lait, 2004, 84:125-134.
[31] DERZELLE S, BOLOTIN A, MISTOU M Y, et al.Proteome analysis of Streptococcus thermophilus grown in milk reveals pyruvate formate-lyase as the major upregulated protein[J].Applied and Environmental Microbiology, 2005, 71(12):8597-8605.
[32] TREMONTE P, REALE A, DI RENZO T, et al.Interactions between Lactobacillus sakei and CNC (Staphylococcus xylosus and Kocuria varians) and their influence on proteolytic activity[J].Letters in Applied Microbiology, 2010, 51(5):586-594.
[33] GOBBETTI M, CORSETTI A, ROSSI J.The sourdough microflora.Interactions between lactic acid bacteria and yeasts:Metabolism of carbohydrates[J].Applied Microbiology and Biotechnology, 1994, 41(4):456-460.
[34] GOBBETTI M.The sourdough microflora:Interactions of lactic acid bacteria and yeasts[J].Trends in Food Science and Technology, 1998, 9(7):267-274.
[35] MORRIS B E L, HENNEBERGER R, HUBER H, et al.Microbial syntrophy:Interaction for the common good[J].FEMS Microbiology Reviews, 2013, 37(3):384-406.
[36] LIN L, LI Y B.Sequential batch thermophilic solid-state anaerobic digestion of lignocellulosic biomass via recirculating digestate as inoculum-Part I:Reactor performance[J].Bioresource Technology, 2017, 236:186-193.
[37] BAIREY E, KELSIC E D, KISHONY R.High-order species interactions shape ecosystem diversity[J].Nature Communications, 2016, 7(1):12285.
[38] ANGULO M T, MOOG C H, LIU Y Y.A theoretical framework for controlling complex microbial communities[J].Nature Communications, 2019, 10(1): 1045.
[39] LIU Y Y, SLOTINE J J, BARAB SI A L.Controllability of complex networks[J].Nature, 2011, 473(7346):167-173.
SI A L.Controllability of complex networks[J].Nature, 2011, 473(7346):167-173.
[40] MARAGKOUDAKIS P A, ZOUMPOPOULOU G, MIARIS C, et al.Probiotic potential of Lactobacillus strains isolated from dairy products[J].International Dairy Journal, 2006, 16(3):189-199.
[41] EMBREE M, LIU J K, AL-BASSAM M M, et al.Networks of energetic and metabolic interactions define dynamics in microbial communities[J].Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(50):15450-15455.
[42] D’SOUZA G, WASCHINA S, PANDE S, et al.Less is more:Selective advantages can explain the prevalent loss of biosynthetic genes in bacteria[J].Evolution, 2014, 68(9):2559-2570.
[43] KALETA C, SCH UBLE S, RINAS U, et al.Metabolic costs of amino acid and protein production in Escherichia coli[J].Biotechnology Journal, 2013, 8(9):1105-1114.
UBLE S, RINAS U, et al.Metabolic costs of amino acid and protein production in Escherichia coli[J].Biotechnology Journal, 2013, 8(9):1105-1114.
[44] RAINA J B, FERNANDEZ V, LAMBERT B, et al.The role of microbial motility and chemotaxis in symbiosis[J].Nature Reviews Microbiology, 2019, 17(5):284-294.
[45] ZHANG L, FENG G, DECLERCK S.Signal beyond nutrient, fructose, exuded by an arbuscular mycorrhizal fungus triggers phytate mineralization by a phosphate solubilizing bacterium[J].The ISME Journal, 2018, 12(10):2339-2351.
[46] LEROI F, PIDOUX M.Detection of interactions between yeasts and lactic acid bacteria isolated from sugary kefir grains[J].Journal of Applied Bacteriology, 1993, 74(1):48-53.
[47] VILJOEN B C. Yeast ecological interactions. Yeast′Yeast, Yeast′Bacteria, Yeast′Fungi Interactions and Yeasts as Biocontrol Agents. Heidelberg: Springer, 2006:83-110.
[48] MAYO B, RODR GUEZ J, V
GUEZ J, V ZQUEZ L, et al. Microbial interactions within the cheese ecosystem and their application to improve quality and safety. Foods, 2021, 10(3): 602-602.
ZQUEZ L, et al. Microbial interactions within the cheese ecosystem and their application to improve quality and safety. Foods, 2021, 10(3): 602-602.
[49] DE FILIPPIS F, LA STORIA A, STELLATO G, et al.A selected core microbiome drives the early stages of three popular Italian cheese manufactures[J].PLoS One, 2014, 9(2):e89680.
[50] SONG Z W, DU H, ZHANG Y, et al.Unraveling core functional microbiota in traditional solid-state fermentation by high-throughput amplicons and metatranscriptomics sequencing[J].Frontiers in Microbiology, 2017, 8:1294.
[51] MARSH A J, O’SULLIVAN O, HILL C, et al.Sequencing-based analysis of the bacterial and fungal composition of kefir grains and milks from multiple sources[J].PLoS One, 2013, 8(7):e69371.
[52] LEROI F, PIDOUX M. Characterization of interactions between Lactobacillus hilgardii and Saccharomyces florentinus isolated from sugary kefir grains. Journal of Applied Bacteriology, 1993, 74(1): 54-60.
[53] MAYO B, RODR GUEZ J, V
GUEZ J, V ZQUEZ L, et al. Microbial interactions within the cheese ecosystem and their application to improve quality and safety. Foods, 2021, 10(3): 602.
ZQUEZ L, et al. Microbial interactions within the cheese ecosystem and their application to improve quality and safety. Foods, 2021, 10(3): 602.
[54] WU Q, KONG Y, XU Y.Flavor profile of Chinese liquor is altered by interactions of intrinsic and extrinsic microbes[J].Applied and Environmental Microbiology, 2015, 82(2):422-430.
[55] JIANG X, ZERFAß C, FENG S, et al.Impact of spatial organization on a novel auxotrophic interaction among soil microbes[J].The ISME Journal, 2018, 12(6):1443-1456.
[56] HARCOMBE W R, RIEHL W J, DUKOVSKI I, et al.Metabolic resource allocation in individual microbes determines ecosystem interactions and spatial dynamics[J].Cell Reports, 2014, 7(4):1104-1115.
[57] TRIVERS R L.The evolution of reciprocal altruism[J].The Quarterly Review of Biology, 1971, 46(1):35-57.
[58] MITRI S, XAVIER J B, FOSTER K R.Social evolution in multispecies biofilms[J].Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(2):10839-10846.
[59] ESTRELA S, BROWN S P.Metabolic and demographic feedbacks shape the emergent spatial structure and function of microbial communities[J].PLoS Computational Biology, 2013, 9(12):e1003398.
[60] PANDE S, KAFTAN F, LANG S, et al.Privatization of cooperative benefits stabilizes mutualistic cross-feeding interactions in spatially structured environments[J].The ISME Journal, 2016, 10(6):1413-1423.
[61] NADELL C D, DRESCHER K, FOSTER K R.Spatial structure, cooperation and competition in biofilms[J].Nature Reviews Microbiology, 2016, 14(9):589-600.
[62] ZHENG J A, WU C D, HUANG J, et al.Spatial distribution of bacterial communities and related biochemical properties in Luzhou-flavor liquor-fermented grains[J].Journal of Food Science, 2014, 79(12):M2491-M2498.
[63] SPITAELS F, WIEME A D, JANSSENS M, et al.The microbial diversity of an industrially produced lambic beer shares members of a traditionally produced one and reveals a core microbiota for lambic beer fermentation[J].Food Microbiology, 2015, 49:23-32.
[64] WANG S L, WU Q, NIE Y, et al. Construction of synthetic microbiota for reproducible flavor compound metabolism in Chinese light-aroma-type liquor produced by solid-state fermentation. Applied and Environmental Microbiology, 2019, 85(10): e03090-e03018.
[65] ZU IGA C, ZARAMELA L, ZENGLER K.Elucidation of complexity and prediction of interactions in microbial communities[J].Microbial Biotechnology, 2017, 10(6):1500-1522.
IGA C, ZARAMELA L, ZENGLER K.Elucidation of complexity and prediction of interactions in microbial communities[J].Microbial Biotechnology, 2017, 10(6):1500-1522.
[66] FRANZOSA E A, HSU T, SIROTA-MADI A, et al.Sequencing and beyond:Integrating molecular omics for microbial community profiling[J].Nature Reviews Microbiology, 2015, 13(6):360-372.
[67] DE FILIPPIS F, PARENTE E, ERCOLINI D.Recent past, present, and future of the food microbiome[J].Annual Review of Food Science and Technology, 2018, 9(1):589-608.
[68] VILANOVA C, PORCAR M.Are multi-omics enough?[J].Nature Microbiology, 2016, 1(8):16101.
[69] LAGIER J C, DUBOURG G, MILLION M, et al.Culturing the human microbiota and culturomics[J].Nature Reviews Microbiology, 2018, 16(9):540-550.
[70] GERMERODT S, BOHL K, LÜCK A, et al. Pervasive selection for cooperative cross-feeding in bacterial communities. PLoS Computational Biology, 2016, 12(6): e1004986.
[71] BORDBAR A, MONK J M, KING Z A, et al. Constraint-based models predict metabolic and associated cellular functions. Nature Reviews Genetics, 2014, 15(2): 107-120.
[72] SONG H S, CANNON W, BELIAEV A, et al. Mathematical modeling of microbial community dynamics: A methodological review. Processes, 2015, 3(3): 699-700.
[73] XIAO Y D, ANGULO M T, FRIEDMAN J, et al. Mapping the ecological networks of microbial communities. Nature Communications, 2017, 8: 2042.
[74] AN G, MI Q, DUTTA-MOSCATO J, et al. Agent-based models in translational systems biology. WIREs Systems Biology and Medicine, 2009, 1(2): 159-171.
[75] KUMAR M, JI B, ZENGLER K, et al.Modelling approaches for studying the microbiome[J].Nature Microbiology, 2019, 4(8):1253-1267.
[76] TRISTEZZA M, DI FEO L, TUFARIELLO M, et al.Simultaneous inoculation of yeasts and lactic acid bacteria:Effects on fermentation dynamics and chemical composition of Negroamaro wine[J].LWT-Food Science and Technology, 2016, 66:406-412.
[77] PANDA S K, SHETTY P H. Innovations in Technologies for Fermented Food and Beverage Industries. Heidelberg: Springer, 2018: 25-52.