空肠弯曲菌(Campylobacter jejuni)是一种革兰氏阴性微需氧细菌,属于弯曲菌属,是一种典型的食源性人畜共患病原菌,被认为是引发细菌性急性腹泻的主要原因[1]。世界卫生组织统计,空肠弯曲菌每年在全球造成3.7万多人死亡,与空肠弯曲菌相关的食物中毒病例在持续上升[2]。除了引发肠胃炎,对于免疫力低下的人群,感染空肠弯曲菌可导致格林-巴利综合征(Guillain-Barre syndrome,GBS),容易造成呼吸肌麻痹而死亡,进一步的空肠弯曲菌感染后遗症是关节炎、结节性红斑等[3]。禽类是空肠弯曲菌的主要宿主。空肠弯曲菌在肉鸡群中的定植率为30%~90%,在3周龄以上的鸡群中定植率最高[4]。此外,空肠弯曲菌还可能污染生猪肉、牛肉、羊肉、蔬菜、水果、牛奶等。根据最新报道,空肠弯曲菌对氟喹诺酮类药物、四环素、红霉素、庆大霉素等抗生素均具有抗药性[5],因此世界卫生组织将其列为重要的耐药细菌,为了有效防治空肠弯曲菌的污染和耐药性,开发新型抗菌剂尤为迫切。
噬菌体是一种细菌病毒,它能够通过尾丝蛋白识别细菌表面的特定受体,从而特异性地识别宿主菌,并在其体内复制,最终裂解宿主菌[6]。噬菌体具有显著的特异性、增殖迅速、强大的抗菌能力,而且不会对其他正常菌群产生影响[7],因此噬菌体已成为一种广受关注的新型抗菌剂[8]。目前弯曲菌噬菌体大多数是从家禽的肠道或排泄物中分离,主要分为3组:Ⅰ组包含极少数具有大基因组的噬菌体(约320~425 kbp),Ⅱ组基因组大小介于180~190 kbp,Ⅲ组介于130~140 kbp。Ⅲ组噬菌体的裂解能力更强,在防治弯曲菌污染方面具有更大的优势。空肠弯曲菌特异性噬菌体可用于家禽养殖场,以防止或减少弯曲菌在家禽中的定植[9]。CARVALHO等[10]发现空肠弯曲菌噬菌体鸡尾酒通过口服灌胃和饲料给药时,空肠弯曲菌的数量减少约为2 lg CFU/g;LOC CARRILLO等[11]发现与未处理的对照组相比,在噬菌体给药后5 d内,鸟类盲肠内容物中空肠弯曲菌的数量下降0.55 lg CFU/g。目前只有16个弯曲菌噬菌体完成了全基因组测序,对空肠弯曲菌噬菌体进行生物学表征和基因组学分析的研究十分有限[12],关于空肠弯曲菌噬菌体用于防控家禽生产中弯曲菌污染的专利也很少,因此需要开发更丰富的空肠弯曲菌噬菌体资源,并对其进行表征和测序,是当前弯曲菌噬菌体研究的重点[13]。
本研究从青岛家禽养殖场鸡粪样品中分离一株裂解性的空肠弯曲菌噬菌体,对其进行生物学特性测定、全基因组分析及进化分析等,为空肠弯曲菌噬菌体的研究和应用提供理论基础,为防控空肠弯曲菌污染和耐药性提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
空肠弯曲菌2206(Campylobacter jejuni 2206)由青岛润达生物科技有限公司惠赠;空肠弯曲菌CICC 22936(Campylobacter jejuni CICC 22936)、沙门氏菌14028(Salmonella 14028)、铜绿假单胞菌PA01(Pseudomonas aeruginosa 01)、铜绿假单胞菌PA14(Pseudomonas aeruginosa 14)、枯草芽孢杆菌WB800(Bacillus subtilis WB800)由本实验室保存并提供。
1.1.2 样品来源
鸡粪便样品采自山东省青岛市家禽养殖场。
1.1.3 培养基和主要试剂
NZCYM琼脂培养基、NZCYM Broth,上海生工生物公司;哥伦比亚血琼脂基础培养基、脱纤维羊血、MH肉汤,青岛海博生物技术有限公司;0.22 μm无菌滤膜,美国Millipore公司。SM缓冲液:NaCl 5.8 g,MgSO4 ·7H2O 2 g,1 mol/L Tris-HCl (pH 7.5) 50 mL,加超纯水定容至1 L。1×PBS缓冲液:KH2PO4 0.2 g,Na2HPO42.17 g,NaCl 8 g,KCl 0.2 g溶解于1 L水中,调节最终pH值为7.4。
1.1.4 仪器与设备
2.5 L MGC微需氧产气包、2.5 L MGC厌氧罐,日本三菱化学株式会社;mLS-3750高温蒸汽灭菌锅,日本SANYO公司;Sigma3K15离心机,上海曦玛离心机有限公司。
1.2 空肠弯曲菌的培养及菌液制备
将Campylobacter jejuni 2206划线培养于哥伦比亚血平板,放置微需氧产气包于MGC厌氧罐42 ℃培养36~48 h。无菌接种单菌落至含有20 mL MH肉汤的小锥形瓶,42 ℃微需氧摇床培养24 h至菌液出现浑浊[14]。
1.3 噬菌体的分离纯化
取适量新鲜的鸡粪样品用SM缓冲液浸泡,放置于4 ℃ 冰箱静置过夜,使样品中的噬菌体充分溶解到缓冲液中。将混合物在室温下8 000 r/min离心15 min,通过0.22 μm无菌滤膜除菌,即为样品滤液。取500 μL样品滤液与500 μL对数生长期的空肠弯曲菌液混合,静置20 min,使噬菌体吸附到宿主菌上。将1 mL混合液和4 mL的半固体培养基NZCYM混合在一起,将混合液注入NZCYM固体平板,铺双层平板。将其放置在42 ℃的微需氧环境中培养24 h,观察噬菌斑的生长状况[15]。
挑取单个噬菌斑梯度稀释于SM缓冲液中,铺双层平板,将平板放置在42 ℃微需氧环境中培养24 h。大约纯化5~6轮,至平板上噬菌斑的形态一致。
1.4 噬菌体的电镜形态观察
将10 μL噬菌体增殖液滴加到铜网上面,静置15 min,用滤纸吸去剩余噬菌体溶液,接着滴加10 μL 2%磷钨酸染料液,继续染色10 min,最后晾干。使用透射电子显微镜,观察噬菌体的形态特征[16]。
1.5 噬菌体的生物学性质
1.5.1 噬菌体热稳定性测定
将100 μL噬菌体液与900 μL SM缓冲液混合,将其分别放入30、40、50、60、70、80 ℃水浴,分别静置30、60 min,采用双层平板法计算噬菌体在各种温度下的效价,各个温度条件采用3组平行测试[17]。
1.5.2 噬菌体pH稳定性测定
取100 μL噬菌体液与900 μL不同pH值(2~13)的PBS缓冲液混合,于42 ℃水浴加热2 h后,采用双层平板法测定噬菌体在不同pH缓冲液的效价,每个pH 做3组平行。
1.5.3 噬菌体最佳感染复数测定
感染复数(multiple of infection,MOI)是指在特定时间内可吸附的噬菌体与宿主菌的比值,是表征裂解性噬菌体侵染能力的一个重要指标。取1 mL对数期的空肠弯曲菌菌液(1.0×107 CFU/mL),分别按照0.001、0.010、0.100、1.000、10.000、100.000的感染复数比例加入1 mL稀释不同浓度的噬菌体,在42 ℃微需氧摇床,120 r/min条件培养6 h。将混合培养物以12 000 r/min离心2 min。取出100 μL的上清液,梯度稀释,然后用双层平板法测定噬菌体的效价。做3组平行实验,噬菌体效价最高的一组感染复数即为最佳感染复数[18]。
1.5.4 噬菌体一步生长曲线测定
一步生长曲线测定是衡量噬菌体裂解能力的一个重要生物学指标,能够反映噬菌体潜伏期的长短、裂解量大小等特性[19]。取1 mL对数期的空肠弯曲菌菌液,再加入1 mL效价为1.0×105 PFU/mL的噬菌体,按照最佳感染复数(MOI=0.01),将混合液放入42 ℃微需氧厌氧罐中,120 r/min孵育20 min,使噬菌体充分吸附到宿主菌上。将混合物离心2 min,弃去上清液,用MH肉汤洗涤2次,将沉淀用MH肉汤重悬加入20 mL MH肉汤中,在42 ℃的微需氧摇床中以120 r/min的速度进行培养。定时取样,直至300 min。每30 min取样1次测定效价,重复3次实验。以时间为横坐标,以噬菌体效价的对数值为纵坐标,绘制一步生长曲线[20]。
1.5.5 噬菌体对宿主菌生长的抑制作用
分别将1 mL对数期的空肠弯曲菌液(1.0×107CFU/mL)与1 mL稀释不同浓度的噬菌体液,分别按照感染复数为0.01,0.10,1.00的比例混合接种至20 mL MH肉汤。对照组为1 mL不含噬菌体的样品。在42 ℃微需氧摇床120 r/min培养24 h,每隔6 h测量混合物的OD600值。每次做3组平行。
1.5.6 噬菌体裂解谱测定
噬菌体的裂解谱测定采用点斑法。取100 μL宿主菌液,包括空肠弯曲菌2206,空肠弯曲菌CICC 22936,沙门氏菌14028,铜绿假单胞菌PA01,铜绿假单胞菌PA14,枯草芽孢杆菌WB800于5 mL半固培养基中,混匀后倒置于平板中。取5 μL噬菌体液点滴到含有不同菌液的双层平板上。过夜培养观察有无噬菌斑。
1.6 噬菌体基因组测序与分析
用病毒基因组DNA提取试剂盒提取噬菌体vB_Cj_QDYZ的DNA[21],送至上海凌恩生物公司进行测序。使用RAST软件对噬菌体进行基因预测[22],确定全基因组所有的开放阅读框(open reading frame, ORF)。根据BLASTp比对工具进行蛋白比对,推测每个ORF最相近的蛋白功能,即完成噬菌体的全基因组功能注释[23]。
使用BLASTn对噬菌体vB_Cj_QDYZ的全基因组进行比对,并下载相似性较高的空肠弯曲菌噬菌体基因组,构建全基因组水平的进化树。使用BLASTp对噬菌体vB_Cj_QDYZ的主要衣壳蛋白序列、内溶素序列进行比对,利用MEGA 7.0软件,构建系统进化树。
1.7 噬菌体在牛奶中的抑菌效果
将500 μL浓度为1.0×106 CFU/mL的空肠弯曲菌液与500 μL噬菌体增殖液(1.0×108 PFU/mL)混合于5 mL牛奶中,在42 ℃ 恒温培养箱中孵育。对照组为500 μL无菌PBS缓冲液。分别于0 h和4 h取出样品,对其进行梯度稀释,在合适梯度吸取100 μL涂布于哥伦比亚血平板上,42 ℃微需氧条件培养24~36 h,并计算哥伦比亚血平板上的空肠弯曲菌的单菌落总数。细菌数的计算如公式(1)所示:
细菌数/(CFU/mL)=菌落计数×10×稀释倍数
(1)
2 结果与分析
2.1 空肠弯曲菌噬菌体的分离和纯化
通过双层平板法分离得到一株空肠弯曲菌的裂解性噬菌体。根据宿主菌及分离样品来源,将噬菌体命名为vB_Cj_QDYZ。经过6轮纯化,噬菌斑的大小和形态比较一致,如图1-A所示,它们的直径仅为1~2 mm,边缘光滑,没有任何晕环;噬菌斑清晰透亮,说明噬菌体的裂解能力较强。

A-噬菌斑形态;B-噬菌体的透射电镜形态
图1 噬菌体 vB_Cj_QDYZ的形态特征
Fig.1 Morphological characteristics of phage vB_Cj_QDYZ
2.2 噬菌体透射电镜形态观察
如图1-B所示,噬菌体vB_Cj_QDYZ在透射电子显微镜下,有一个正二十面体的头部(直径约100.81 nm)和一条可伸缩的尾部(长度约65.90 nm)。根据ICTV第九次病毒类型研究报告,噬菌体vB_Cj_QDYZ属于有尾噬菌体目,肌尾噬菌体科[24]。目前大多数弯曲菌噬菌体都是肌尾噬菌体,具有终端泡和空头结构,有肌鞘并且可收缩。只有极少数弯曲菌噬菌体是长尾和短尾噬菌体。
2.3 噬菌体的生物学性质
2.3.1 噬菌体的热稳定性
如图2-A所示,噬菌体vB_Cj_QDYZ在30~60 ℃效价较稳定,且维持较高水平,大约为107 PFU/mL。当噬菌体在70 ℃和80 ℃条件下孵育30 min,噬菌体效价明显降低至102 PFU/mL;孵育60 min时,噬菌体完全失活。综上,噬菌体vB_Cj_QDYZ的温度耐受范围为30~60 ℃。

A-噬菌体的温度稳定性;B-噬菌体的pH稳定性;C-噬菌体的一步生长曲线;D-噬菌体对宿主的生长抑制作用
图2 噬菌体 vB_Cj_QDYZ的生物学特性
Fig.2 Biological characteristics of phage vB_Cj_QDYZ
2.3.2 噬菌体的pH稳定性
如图2-B所示,噬菌体vB_Cj_QDYZ在pH值为3~12时效价较稳定,基本维持在大约106 PFU/mL。当噬菌体在pH=2的酸性环境或者pH=13的碱性环境孵育2 h后,完全失活。综上,噬菌体vB_Cj_QDYZ的pH值耐受范围是3~12。
2.3.3 噬菌体的最佳感染复数
根据表1的数据,当感染复数为0.01时,噬菌体的效价达到最高,为2.07×108 PFU/mL,因此vB_Cj_QDYZ的最佳感染复数为0.01。
表1 噬菌体vB_Cj_QDYZ最佳感染复数
Table 1 The optimal MOI of phage vB_Cj_QDYZ

噬菌体效价/(PFU/mL)宿主菌浓度/(CFU/mL)MOI效价/(PFU/mL)1.0×1071.0×1051004.00×1061.0×1071.0×106101.40×1071.0×1071.0×10711.05×1081.0×1061.0×1070.11.21×1081.0×1051.0×1070.012.07×1081.0×1041.0×1070.0013.50×107
表2 噬菌体vB_Cj_QDYZ的裂解谱
Table 2 The host range of phage vB_Cj_QDYZ

菌株裂解性Campylobacter jejuni 2206+Campylobacter jejuni CICC 22936+Pseudomonas aeruginosa 01-Pseudomonas aeruginosa 14-Salmonella 14028-Bacillus subtilis WB800-
注:+表示能裂解宿主菌,-表示不能裂解宿主菌。
2.3.4 噬菌体的一步生长曲线
噬菌体的一步生长曲线主要由潜伏期、裂解期和平稳期组成。如图2-C所示,噬菌体的潜伏期是60 min。从第60 min以后,噬菌体的效价突然大规模增加,呈现指数式增长,因此噬菌体的裂解期是150 min。在最后的60 min, 噬菌体的效价不再出现明显改变,此阶段为平稳期。爆发量是由噬菌体的效价和宿主菌的数量决定的。在爆发末期,噬菌体的效价大约为4.5×108 PFU/mL,而感染初期宿主菌的含量为1.0×107 CFU/mL,因此爆发量为45 PFU/cell。
2.3.5 噬菌体对宿主菌的生长抑制作用
由图2-D可知,噬菌体vB_Cj_QDYZ与空肠弯曲菌2206在不同感染复数混合培养后,初始OD600值基本相同,随着培养时间的延长,在微需氧条件培养第6 h时,对照组OD600值已达到0.145, 加了不同浓度噬菌体组的OD600值是0.08。在培养第24 h,对照组OD600值为0.434,噬菌体组依然是0.08。表明噬菌体可在不同MOI下高效裂解宿主菌,噬菌体具有很强的裂解能力,可以有效抑制宿主菌的生长,具有良好的应用前景和抗菌潜力。
2.3.6 噬菌体的裂解谱
空肠弯曲菌噬菌体vB_Cj_QDYZ可以裂解实验室的两株空肠弯曲菌,点斑法可以产生清晰透亮的噬菌斑。但是对于铜绿假单胞菌、沙门氏菌、枯草芽孢杆菌的平板,则没有清晰透亮的噬菌斑产生。充分说明空肠弯曲菌噬菌体vB_Cj_QDYZ具有很强的特异性。研究表明空肠弯曲菌噬菌体的裂解谱相窄,特异性强。
空肠弯曲菌噬菌体vB_Cj_QDYZ可以裂解实验室的两株空肠弯曲菌,点斑法可以产生清晰透亮的噬菌斑。但是对于铜绿假单胞菌、沙门氏菌、枯草芽孢杆菌的平板,则没有清晰透亮的噬菌斑产生。充分说明空肠弯曲菌噬菌体vB_Cj_QDYZ具有很强的特异性。研究表明空肠弯曲菌噬菌体的裂解谱相窄,特异性强。
2.4 噬菌体vB_Cj_QDYZ全基因组分析
全基因组测序结果显示:噬菌体vB_Cj_QDYZ基因组为双链DNA,基因组全长为130 627 bp, G+C含量为26%。根据基因组大小在130~140 kbp,推断该噬菌体为Ⅲ组空肠弯曲菌噬菌体,属于CP8 virus。研究表明Ⅲ组噬菌体是目前分离最多、抑菌效果最好的空肠弯曲菌噬菌体。
在噬菌体全基因组中不含抗生素耐药基因、过敏原基因和毒力因子。进一步说明该噬菌体应用的安全性,具有良好的应用前景和抗菌潜力。噬菌体vB_Cj_QDYZ在NCBI上的序列号为OQ515481.1。
2.5 噬菌体vB_Cj_QDYZ功能注释
噬菌体 vB_Cj_QDYZ全基因组的功能注释如图3所示,全基因组一共有165个ORF,3个tRNA。其中有65个ORF编码已知功能,另外100个编码假设蛋白。对65个具有已知功能的ORF分类,主要有4种功能的蛋白:噬菌体结构蛋白、裂解蛋白、包装蛋白、与噬菌体复制和代谢有关的蛋白。具体的注释结果见附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.035791)。
附表1 噬菌体vB_Cj_QDYZ基因组功能注释
Attached Table 1 ORF analysis of Campylobacter jejuni phage vB_Cj_QDYZ genome

开放阅读框ORF开始start结束stop功能function164065receptor binding protein 32786637receptor binding protein 331 470835receptor binding protein 241 7381 580receptor binding protein 253 0921 755hypothetical protein64 7623 116recombination endonuclease75 1384 815hypothetical protein86 1905 195recombination endonuclease97 0476 178gp55 Sigma factor for T4 late transcription107 1408 102putative major capsid protein118 1108 757head completion protein1210 5208 742putative homing endonuclease1310 62911 660gp15 tail sheath stabilizer and completion protein1411 65712 112hypothetical protein1512 34512 154hypothetical protein 1612 67112 369hypothetical protein1712 82512 658hypothetical protein1813 30812 925hypothetical protein1913 83813 401hypothetical protein2014 55313 993hypothetical protein2114 89414 550hypothetical protein2214 98914 891hypothetical protein2315 31214 983hypothetical protein2415 55815 355hypothetical protein2515 79415 561hypothetical protein2616 57215 847hypothetical protein2716 86716 583hypothetical protein2817 75516 901hypothetical protein2918 06817 835hypothetical protein3018 27018 049hypothetical protein3118 71418 340hypothetical protein3219 18418 795hypothetical protein3319 79719 390hypothetical protein3420 29119 848head completion protein3520 67420 291None3620 98920 708None3721 86420 986putative RNA ligase3822 14221 867hypothetical protein3922 54422 302hypothetical protein4022 99022 541hypothetical protein4123 19322 987hypothetical protein4223 30023 196hypothetical protein4323 70623 287hypothetical protein4424 03523 763hypothetical protein 4524 26024 042hypothetical protein4624 65124 250hypothetical protein4724 99024 790hypothetical protein 4826 05425 023putative poly A polymerase4927 63026 257ribonucleotide reductase large subunit5028 75327 617ribonucleotide reductase small subunit5129 12528 766hypothetical protein 5229 48929 118hypothetical protein5329 69229 486hypothetical protein 5431 20329 728putative homing endonuclease5532 27931 236recombination and repair protein5632 93632 598hypothetical protein5733 56432 938hypothetical protein5834 53633 586sliding clamp loader subunit
续表

开放阅读框ORF开始start结束stop功能function5935 17534 594hypothetical protein 6035 42635 166hypothetical protein 6135 86235 416putative dUTP pyrophosphatase6236 24235 850hypothetical protein 6337 52236 254putative NAD(FAD)-utilizing dehydrogenase6437 97937 575hypothetical protein 6538 15937 983hypothetical protein 6638 78138 215endolysin6739 43538 800hypothetical protein 6840 96439 648topoisomerase II6942 62440 999hypothetical protein7044 48442 634topoisomerase II large subunit7145 78644 584DNA helicase7246 39645 947hypothetical protein7346 70846 445hypothetical protein 7446 76347 164hypothetical protein7547 70047 182hypothetical protein7648 37447 733hypothetical protein7748 42049 073hypothetical protein 7849 07049 732hypothetical protein7949 72552 382hypothetical protein8052 38453 340hypothetical protein 8153 35553 708hypothetical protein 8253 71654 477hypothetical protein8354 49954 960hypothetical protein8454 95056 086hypothetical protein8556 08356 769putative baseplate hub subunit and tail lysozyme8657 36857 081hypothetical protein8758 97557 356putative Hef-like homing endonuclease8859 43358 987hypothetical protein8960 06659 494hypothetical protein9060 61060 083putative tail tube protein9161 21660 638gp19 tail tube protein II9261 77861 218hypothetical protein9363 31861 762hypothetical protein9465 40863 315gp6 baseplate wedge subunit9565 75565 408gp25 baseplate wedge subunit9666 49765 787sliding clamp DNA polymerase accessory protein9767 52966 546amidinotransferase family protein9868 85167 610DNA primase-helicase subunit9969 53768 851hypothetical protein 10073 80969 709hypothetical protein; putative tail fibre protein10174 88573 860putative ribonuclease H10275 00674 872hypothetical protein10375 38975 003putative clamp loader subunit10476 35875 441putative ssDNA binding protein10578 70476 470putative homing endonuclease10679 47778 740putative DNA end protector protein10780 19679 480putative large terminase10880 70680 233hypothetical protein10981 36480 699thymidylate synthetase11081 60283 371hypothetical protein11183 36884 822hypothetical protein11286 74784 837tail sheath protein11387 25686 909hypothetical protein11488 19987 246hypothetical protein11589 54388 221DNA ligase11689 71789 589hypothetical protein 11790 01389 714hypothetical protein
续表

开放阅读框ORF开始start结束stop功能function11890 50390 003nuclease activity protein11990 89890 521hypothetical protein12091 78491 014hypothetical protein12192 37691 795hypothetical protein12292 47293 560Hef7112395 47593 547hypothetical protein12496 40495 481ribonucleoside-diphosphate reductase large subunit12597 74996 442hypothetical protein12699 24497 847major capsid protein127100 02299 276hypothetical protein128100 662100 033gp21 prohead core scaffold and protease129100 826100 659hypothetical protein130102 533100 827capsid assembly protein131102 578103 279hypothetical protein132103 282104 013neck protein133104 043105 371terminase subunit134105 400105 621hypothetical protein 135105 624106 355tail tube protein III136107 795106 350hypothetical protein137108 534107 767phosphatidylserine decarboxylase138108 841108 545co-chaperonin GroES139109 406108 897hypothetical protein 140109 906109 409hypothetical protein141110 085109 942hypothetical protein 142112 480110 117putative DEAD/DEAH box helicase143114 057112 537putative helicase144114 519114 100putative repair and recombination protein145115 246114 521hypothetical protein146116 803115 421von Willebrand factor A-like domain proteinRNA 1117 530117 448tRNA-Tyr-GTARNA 2117 620117 549tRNA-Tyr-GTARNA 3117 894117 823tRNA-Tyr-GTA147118 724118 083hypothetical protein148119 457118 708putative RecB family exonuclease149119 811119 467hypothetical protein 150120 482119 868hypothetical protein 151120 723120 580hypothetical protein 152120 960120 853hypothetical protein153123 647120 984DNA polymerase154124 723123 686putative methyltransferase155125 123124 710hypothetical protein156125 560125 120hypothetical protein157126 065125 562thymidine kinase158126 250126 062hypothetical protein 159127 194126 250DNA primase subunit160127 687127 178hypothetical protein 161128 101127 775hypothetical protein 162128 187128 642tail completion and sheath stabilizer protein163128 924128 643hypothetical protein 164129 775129 110hypothetical protein165130 515129 811receptor binding protein 4

图3 噬菌体vB_Cj_QDYZ基因组功能注释
Fig.3 Genome annotation of phage vB_Cj_QDYZ
噬菌体结构蛋白有:ORF10(主要衣壳蛋白),ORF131(衣壳组装蛋白),ORF11和ORF34(头部完成蛋白),ORF133(颈部蛋白),ORF13和ORF167(尾鞘稳定剂和完成蛋白),ORF90、ORF91和ORF136(尾管蛋白),ORF113(尾鞘蛋白),ORF100(尾丝蛋白)。其中,尾管蛋白是噬菌体编码尾部的蛋白,与吸附宿主菌密切相关。尾丝蛋白是噬菌体特异性识别宿主的物质基础,噬菌体的尾丝蛋白在特定微生物检测净化等方面具有重要意义。
噬菌体裂解相关蛋白有2个:ORF66(内溶素)和ORF85(尾部溶菌酶)。噬菌体可能是通过表达这两种蛋白来裂解宿主菌的。
噬菌体包装蛋白:ORF107(末端酶大亚基),ORF134(末端酶小亚基)。噬菌体包装蛋白主要在噬菌体包装时起重要作用。其他蛋白包括受体结合蛋白:ORF1、ORF2、ORF3、ORF4,与噬菌体结合细菌表面受体相关。
与噬菌体复制和代谢有关的蛋白有:ORF6、7(重组核酸内切酶),ORF8(转录调控因子),ORF37(RNA连接酶),ORF48(聚合酶),ORF55(重组和修复蛋白),ORF63(脱氢酶),ORF68(拓扑异构酶),ORF97(氨基转移酶家族蛋白),ORF101(核糖核酸酶),ORF158(DNA聚合酶),ORF162(胸苷激酶),ORF164(DNA引物酶亚基),ORF12、ORF54和ORF87(归巢核酸内切酶),ORF71、ORF144和ORF145(解旋酶),ORF61(dUTP焦磷酸酶),ORF153(核酸外切酶)。其中,重组和修复蛋白的功能是将噬菌体基因组整合到宿主菌上。归巢核酸内切酶参与DNA内含子的水平转移和基因表达的调控。解旋酶是一类解开氢键的酶,一般在噬菌体DNA复制过程中起到催化双链DNA 解旋的作用。焦磷酸酶,参与DNA合成。核酸外切酶,具有从分子链的末端顺次水解磷酸二酯键而生成单核苷酸作用的酶。
2.6 噬菌体vB_Cj_QDYZ比较基因组分析
噬菌体vB_Cj_QDYZ与空肠弯曲菌噬菌体CP39、CJLB-10、CP30A、CPX、CP8的同源性分别为98.30%、94.77%、94.61%、97.69%、97.82%,比对覆盖率分别为95%、91%、92%、92%、92%。弯曲菌噬菌体分为2个属Fletchervirus和Firehammervirus[25]。与vB_Cj_QDYZ相似度最高的噬菌体来自Eucampyvirinae亚科,Fletchervirus属。初步推测空肠弯曲菌噬菌体vB_Cj_QDYZ可能是来自Eucampyvirinae亚科,Fletchervirus属的新成员。
2.7 噬菌体vB_Cj_QDYZ系统进化分析
如图4-A所示,基于全基因组序列构建的进化树结果表明,噬菌体vB_Cj_QDYZ与Campylobacter phage CP39 具有最近的亲缘关系。NCBI检索发现,Campylobacter phage CP39是Eucampyvirinae亚科、Fletchervirus属的成员。研究表明CP39是肌尾噬菌体。因此推测vB_Cj_QDYZ是肌尾噬菌体(Myoviridae)科、Eucampyvirinae亚科、Fletchervirus属的成员。

A-基于全基因组构建的进化树;B-基于内溶素序列构建的进化树;C-基于主要衣壳蛋白构建的进化树
图4 噬菌体vB_Cj_QDYZ进化关系分析
Fig.4 Phylogenetic relationships of phage vB_Cj_QDYZ
如图4-B所示,噬菌体vB_Cj_QDYZ的内溶素和Campylobacter phage CPX的内溶素相似度最高;如图4-C所示, vB_Cj_QDYZ的主要衣壳蛋白与Campylobacter phage CP39的最相近。标尺代表遗传距离,遗传距离越短代表亲缘关系越近。明显看出,图4-B和图4-C的进化树标尺较长,遗传距离较大。因此推断噬菌体vB_Cj_QDYZ可能为一株新的空肠弯曲菌噬菌体。
2.8 噬菌体在牛奶中的抑菌效果
如图5所示,加入空肠弯曲菌噬菌体vB_Cj_QDYZ后,在正常环境下放置4 h后,通过平板计数法测定,哥伦比亚血平板上的空肠弯曲菌菌落总数从初始的1.0×107 CFU/mL降低至1.17×105 CFU/mL,降低了2个lg CFU/mL,充分说明噬菌体vB_Cj_QDYZ在牛奶中的杀菌率为99%。牛奶是容易被弯曲菌污染的食品,选择牛奶作为研究对象具有实际参考价值。综上所述,噬菌体vB_Cj_QDYZ具有良好的抗菌潜力。
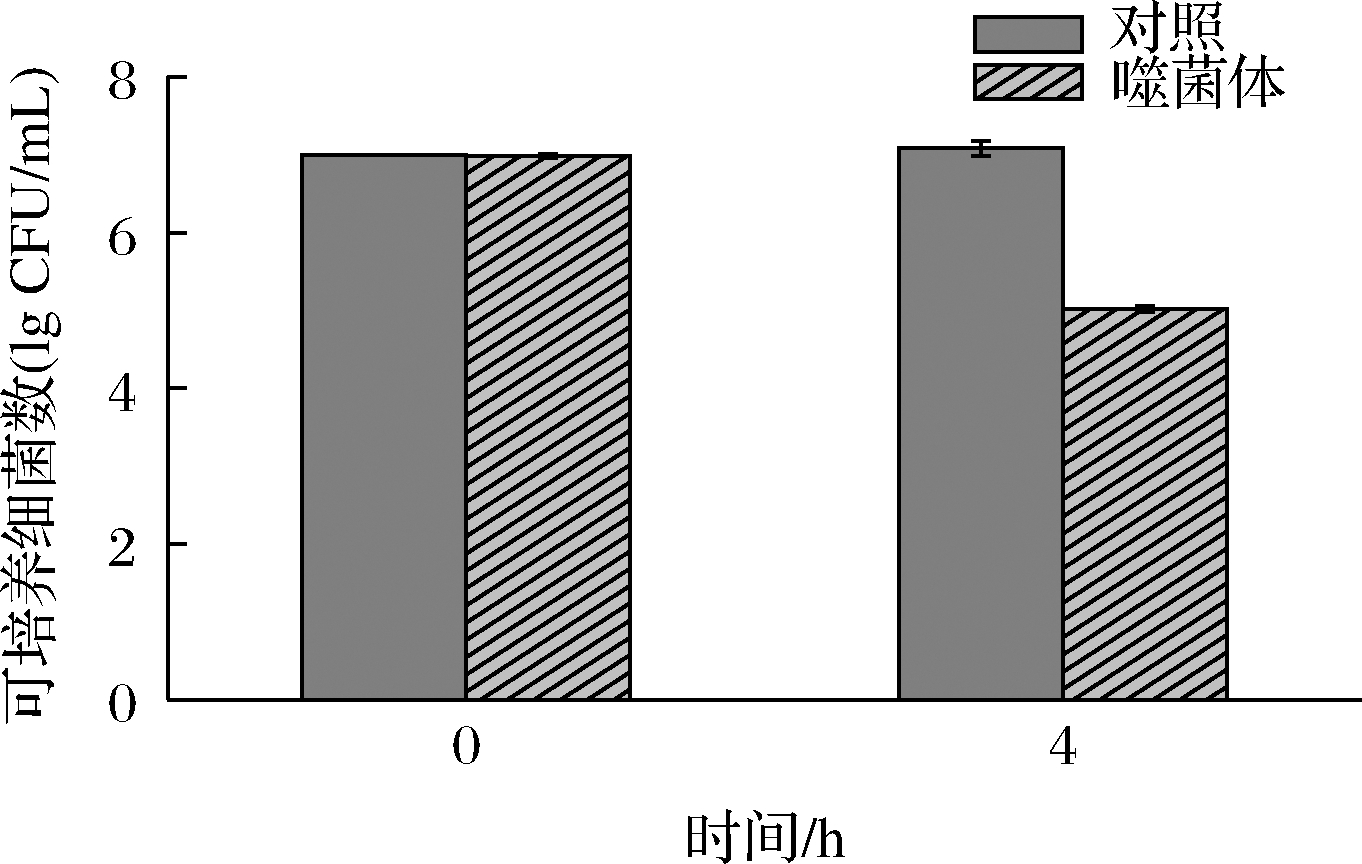
图5 噬菌体 vB_Cj_QDYZ在牛奶中的抑菌效果
Fig.5 Antibacterial effect of phage vB_Cj_QDYZ in milk samples
3 结论与讨论
本研究以空肠弯曲菌2206为宿主菌,从鸡粪中分离出一株空肠弯曲菌噬菌体,将其命名为vB_Cj_QDYZ。噬菌体在双层平板上可形成清晰透亮的噬菌斑,这说明噬菌体具有很强的裂解能力。透射电镜形态表明,噬菌体有一个正二十面体的头部(直径约100.81 nm)和一条可伸缩的尾部(长度约65.90 nm),具有弯曲菌噬菌体典型的终端泡和空头结构,属于有尾噬菌体目,肌尾噬菌体科。大多数弯曲菌噬菌体属于肌尾噬菌体。
生物学特性结果显示,噬菌体具有良好的热稳定性和pH稳定性,表现出噬菌体在某些恶劣环境下具有一定的适用性。噬菌体vB_Cj_QDYZ在30~60 ℃,pH值为3~12的环境下效价较稳定,这为噬菌体在实际家禽生产中的防控提供了理论依据。空肠弯曲菌主要寄居在鸡等家禽的肠道中,鸡的正常体温是39.6~43.6 ℃,鸡肠道的pH值主要为2~4,这说明噬菌体vB_Cj_QDYZ可以在鸡的肠道中存活且保持较高效价,具有广泛的应用范围。噬菌体vB_Cj_QDYZ的最佳感染复数为0.01,充分表明了噬菌体的侵染能力较强。一步生长曲线表明,噬菌体的潜伏期是60 min,裂解期是150 min,爆发量是45 PFU/cell。因此,空肠弯曲菌噬菌体vB_Cj_QDYZ具有较短的潜伏期和较强的裂解能力。此外,加入不同感染复数的噬菌体显著抑制了空肠弯曲菌的生长,表现出噬菌体vB_Cj_QDYZ具有很强的裂解能力。噬菌体vB_Cj_QDYZ在牛奶中的杀菌率为99%,实际应用价值较高,具有开发为新型空肠弯曲菌抗菌剂的潜力。
基因组学研究有利于从基因水平更深入了解空肠弯曲菌噬菌体。噬菌体vB_Cj_QDYZ基因组为双链DNA,基因组全长为130 627 bp, G+C含量为26%。基因组大小位于130~140 kbp,根据ICTV对弯曲菌噬菌体的最新分类,噬菌体vB_Cj_QDYZ被命名为CP8 virus,且属于Ⅲ组噬菌体,特异性受体是荚膜多糖,对空肠弯曲菌具有较强的裂解性能。Ⅲ组噬菌体是目前抑菌效果最好的空肠弯曲菌噬菌体。噬菌体一共有165个ORF,3个tRNA。其中65个ORF具有功能。此外,以全基因组序列构建的进化树显示,噬菌体vB_Cj_QDYZ与Campylobacter phage CP39具有最近的亲缘关系以主要衣壳蛋白和内溶素序列构建的进化树表明,噬菌体与其他的空肠弯曲菌噬菌体遗传距离较远,推断噬菌体vB_Cj_QDYZ属于Fletchervirus属的新成员。此外,噬菌体 vB_Cj_QDYZ不含有抗生素耐药基因、毒力基因、过敏原基因等,安全性较高,适合于实际应用生产。
本研究分离纯化一株裂解能力较强、稳定性良好、安全性较高的新型空肠弯曲菌噬菌体,能够有效抑制空肠弯曲菌的生长,且特异性较强,具有开发为新型空肠弯曲菌抗菌剂的潜力,丰富了空肠弯曲菌噬菌体的资源,为防治空肠弯曲菌污染提供理论基础和技术指导。
[1] YOUNG K T, DAVIS L M, DIRITA V J.Campylobacter jejuni:Molecular biology and pathogenesis[J].Nature Reviews Microbiology, 2007, 5(9):665-679.
[2] AL HAKEEM W G, FATHIMA S, SHANMUGASUNDARAM R, et al.Campylobacter jejuni in poultry:Pathogenesis and control strategies[J].Microorganisms, 2022, 10(11):2134.
[3] HUMPHREY T, O′BRIEN S, MADSEN M.Campylobacters as zoonotic pathogens:A food production perspective[J].International Journal of Food Microbiology, 2007, 117(3):237-257.
[4] CONNERTON P L, TIMMS A R, CONNERTON I F.Campylobacter bacteriophages and bacteriophage therapy[J].Journal of Applied Microbiology, 2011, 111(2):255-265.
[5] WAGENAAR J A, VAN BERGEN M A P, MUELLER M A, et al.Phage therapy reduces Campylobacter jejuni colonization in broilers[J].Veterinary Microbiology, 2005, 109(3-4):275-283.
[6] SHARMA S, CHATTERJEE S, DATTA S, et al.Bacteriophages and its applications:An overview[J].Folia Microbiologica, 2017, 62(1):17-55.
[7] PÉREZ PULIDO R, GRANDE BURGOS M J, G LVEZ A, et al.Application of bacteriophages in post-harvest control of human pathogenic and food spoiling bacteria[J].Critical Reviews in Biotechnology, 2016, 36(5):851-861.
LVEZ A, et al.Application of bacteriophages in post-harvest control of human pathogenic and food spoiling bacteria[J].Critical Reviews in Biotechnology, 2016, 36(5):851-861.
[8] D’ANGELANTONIO D, SCATTOLINI S, BONI A, et al.Bacteriophage therapy to reduce colonization of Campylobacter jejuni in broiler chickens before slaughter[J].Viruses, 2021, 13(8):1428.
[9] RICHARDS P J, CONNERTON P L, CONNERTON I F.Phage biocontrol of Campylobacter jejuni in chickens does not produce collateral effects on the gut microbiota[J].Frontiers in Microbiology, 2019, 10:476.
[10] CARVALHO C M, GANNON B W, HALFHIDE D E, et al.The in vivo efficacy of two administration routes of a phage cocktail to reduce numbers of Campylobacter coli and Campylobacter jejuni in chickens[J].BMC Microbiology, 2010, 10:232.
[11] LOC CARRILLO C, ATTERBURY R J, EL-SHIBINY A, et al.Bacteriophage therapy to reduce Campylobacter jejuni colonization of broiler chickens[J].Applied and Environmental Microbiology, 2005, 71(11):6554-6563.
[12] USHANOV L, LASAREISHVILI B, JANASHIA I, et al.Application of Campylobacter jejuni phages:Challenges and perspectives[J].Animals, 2020, 10(2):279.
[13] 倪娟,潘道东,孙杨赢,等.弯曲菌噬菌体在家禽生产中的防控进展[J].食品安全质量检测学报,2022,13(3):703-709.
Ni J, PAN D D, SUN Y Y, et al.Progress in the prevention and control of Campylobacter phage in poultry production[J].Journal of Food Safety &Quality,2022,13(3):703-709
[14] 英航宁,李凤鸣,李洁等.宿主菌富集空肠弯曲菌噬菌体培养条件的优化[J].微生物学通报,2021,48(9):3176-3183.
YING H N, LI F M, LI J, et al.Optimization of culture conditions for enrichment of Campylobacter jejuni phages by host bacteria[J].Microbiology China[J].2021,48(9):3176-3183.
[15] 宋召军,姜启东,范正杨,等.一株空肠弯曲菌噬菌体的分离及其生物学特性鉴定[J].中国抗生素杂志,2018,43(8):1020-1025.
SONG Z J, JIANG Q D, FAN Z Y, et al.The isolation and characterization of a lytic phage infecting Campylobacter jejuni[J].2018,43(8):1020-1025.
[16] LIU B, ZHENG T, QUAN R, et al.Biological characteristics and genomic analysis of a novel Vibrio parahaemolyticus phage phiTY18 isolated from the coastal water of Xiamen China[J].Frontiers in Cellular and Infection Microbiology, 2022:1610.
[17] WANG L K, ZHANG J Y, LIU X, et al.Biological characterization and complete genome analysis of a novel Stenotrophomonas maltophilia phage vB_SM_ytsc_ply2008005c[J].Virus Research, 2022, 318:198856.
[18] XIANG Y Y, LI W Y, SONG F, et al.Biological characteristics and whole-genome analysis of the Enterococcus faecalis phage PEf771[J].Canadian Journal of Microbiology, 2020, 66(9):505-520.
[19] SøRENSEN M C H, GENCAY Y E, BRøNDSTED L.Methods for initial characterization of Campylobacter jejuni bacteriophages[J]. Campylobacter jejuni:Methods and Protocols, 2017:91-105.
[20] XUAN G H, KONG J N, WANG Y F, et al.Characterization of the newly isolated Pseudomonas phage vB_Pae_LC3I3[J].Virus Research, 2023, 323:198978.
[21] GONZ LEZ-G
LEZ-G MEZ J P, L
MEZ J P, L PEZ-CUEVAS O, CASTRO-DEL CAMPO N, et al.Genomic and biological characterization of the novel phages vB_VpaP_AL-1 and vB_VpaS_AL-2 infecting Vibrio parahaemolyticus associated with acute hepatopancreatic necrosis disease (AHPND)[J].Virus Research, 2022, 312:198719.
PEZ-CUEVAS O, CASTRO-DEL CAMPO N, et al.Genomic and biological characterization of the novel phages vB_VpaP_AL-1 and vB_VpaS_AL-2 infecting Vibrio parahaemolyticus associated with acute hepatopancreatic necrosis disease (AHPND)[J].Virus Research, 2022, 312:198719.
[22] HAN K, ZHU Y N, LI F, et al.Genomic analysis of Acinetobacter phage BUCT629, a newly isolated member of the genus Obolenskvirus[J].Archives of Virology, 2022, 167(4):1197-1199.
[23] YUAN Y, XI H Y, DAI J X, et al.The characteristics and genome analysis of the novel Y.pestis phage JC221[J].Virus Research, 2020, 283:197982.
[24] FURUTA M, NASU T, UMEKI K, et al.Characterization and application of lytic bacteriophages against Campylobacter jejuni isolated from poultry in Japan[J].Biocontrol Science, 2017, 22(4):213-221.
[25] ZAMPARA A, AHERN S J, BRIERS Y, et al.Two distinct modes of Lysis regulation in Campylobacter Fletchervirus and Firehammervirus phages[J].Viruses, 2020, 12(11):1247.