生淀粉酶是一类可以在低于淀粉糊化温度下,直接作用于淀粉颗粒的水解酶类[1],包括α-淀粉酶、生淀粉糖化酶[2]、生淀粉普鲁兰酶等。其中生淀粉糖化酶(raw starch glucoamylase,RSGA)作为降解淀粉过程中最重要的淀粉酶之一,可以从淀粉分子非还原端依次水解α-1,4糖苷键,产物仅为葡萄糖。在低温条件下直接降解淀粉不仅能简化生产工艺,提高生产效率,而且降低了能源消耗,具有广阔的应用前景。已报道的生淀粉糖化酶主要来源于黑曲酶[1]、米曲霉[3]、青霉菌[4]等,可以降解不同种类的生淀粉颗粒。然而,目前RSGA对生淀粉降解催化效率低,酶的最适反应温度较高,大都处于65~75 ℃[2, 5],难以实现大规模应用。
N-糖基化修饰是蛋白质生物合成中常见的翻译后修饰之一[6]。通常氨基酸序列具有Asn-Xaa-Ser/Thr的排列特征,其中Xaa为除Pro以外的任意氨基酸[7]。蛋白糖基化途径关键酶为寡糖转移酶,它对Asn-Xaa-Thr的偏好高于Asn-Xaa-Ser。据文献报道,前者的糖基化频率是后者的2~3倍[8]。一系列的研究强调了Asn-Xaa-Thr对蛋白质催化效率的关键作用。例如,在56位点引入Asn-Xaa-Thr基序后,内切葡聚糖酶对CMC-Na和β-D-葡聚糖的催化效率(kcat/Km)分别提高到原来的1.20和1.28倍[9]。另外,前期对Aspergillus fumigatus来源的RSGA糖基化研究中,在113位点引入Asn-Xaa-Thr糖基化基序,突变体Q113T生玉米淀粉的降解效率提高21%[10]。因此,通过将糖基化基序中Ser突变为Thr,可能是一种提高RSGA催化效率有效的方法。
在以前研究中,我们成功在Komagataella phaffii GS115中表达A.fumigatus来源的RSGA,并发现糖基化对RSGA降解生淀粉的效率具有重要作用[5]。为提高RSGA对生玉米淀粉的催化效率,本研究基于NetNGlyc 1.0分析,将3个具有Asn-Xaa-Ser糖基化基序特征的位点替换为Asn-Xaa-Thr形式,通过对突变体酶学性质进行分析,获得催化效率提高的突变体。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
Escherichia coli JM109 (Invitrogen, Carlsbad, USA) 用于基因克隆,E.coli BL21(DE3) 为大肠杆菌表达宿主,K.phaffii GS115 (His-) (Invitrogen, Carlsbad, USA) 为酵母表达宿主。pPIC9K/α-RSGA和质粒pET20b(+)/S-RSGA 质粒为前期研究所构建并保存[5]。
1.1.2 引物
该研究中使用的引物如表1所示。
表1 本研究所用的引物
Table 1 Primers used in this study

引物名称序列(5′-3′)S123T-FCTGCAGACCGTGAGCAATCCGACCGGTGGTTTAGCS123T-RGCTCACGGTCTGCAGATAAGCTTGGCTGTTCACATACS424T-FGCTCACGCCATGAACAACGGCACCTTAAGTGAACAGTTCS424T-RGTTGTTCATGGCGTGAGCTTGCACAATGCTAACGTAGCCS612T-FGGGAAAGCGACCCGAACCGTACCTACACAGTTCCS612T-RGGTTCGGGTCGCTTTCCCACACGATGCTG
1.1.3 试剂
Primer STAR DNA聚合酶、Primer STARMAX DNA聚合酶、Sal I快切酶、Dpn I消化酶,TaKaRa公司(大连);柱回收试剂盒、胶回收试剂盒、十二烷基硫酸钠-聚丙烯酰氨凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)蛋白电泳试剂盒、标准蛋白,Invitrogen;质粒提取试剂盒、无氨基酵母氮源(不含氨基酸)、氨苄青霉素、卡纳霉素、G418及琼脂糖,生工生物工程(上海)股份有限公司;玉米淀粉,吉林中粮生化有限公司;蛋白胨和酵母粉,OXIOD公司;其他试剂均为国产分析纯。
1.1.4 培养基
LB(Luria-Bertani)培养基(g/L):酵母粉5,蛋白胨10,NaCl 10,pH 7.0。固体培养基在此基础上添加2%琼脂粉。
TB(Terrific-Broth)发酵培养基(g/L):甘油5,蛋白胨12,酵母粉24,KH2PO4 2.31,K2HPO4 16.37。
马铃薯葡萄糖肉汤培养基(potato dextrose broth,PDB)(g/L):酵母粉10,蛋白胨20,葡萄糖 20。
甘油缓冲复合培养基(buffered glycerol-complex medium, BMGY)(g/L):酵母粉10,蛋白胨20,甘油10,无氨基酵母氮源(yeast nitrogen base, YNB) 13.4,生物素 4×10-4,pH 6.0的磷酸钾缓冲液 0.1 mol/L,其中YNB、生物素采用膜过滤除菌。
诱导表达培养基(buffered methanol-complex medium, BMMY) (g/L):酵母提取物 10,蛋白胨 20,甲醇 10,YNB 13.4,生物素 4×10-4,甲醇 10,pH 6.0的磷酸钾缓冲液 0.1 mol/L。
最小糊精酶固体培养基(minimal extrase medium, MD) (g/L):葡萄糖 20,YNB 13.4,生物素 4×10-4,琼脂 20。
1.2 实验方法
1.2.1 突变体质粒的构建
基于PCR原理进行重组质粒的构建。以质粒pPIC9K/α-RSGA和pET20b(+)/S-RSGA为模板,分别使用引物对S123T-F/R、S424T-F/R和S612T-F/R进行质粒扩增,将获得的PCR产物经Dpn I消化模板后回收并转化至JM109,涂布于含100 μg/mL氨苄抗生素的LB固体平板。经菌落PCR验证后将正确的菌落进行质粒提取和序列鉴定,获得重组质粒20b-S123T、20b-S424T、20b-S612T、9K-S123T、9K-S424T和9K-S612T。质粒提取参考试剂盒说明书,测序由生工生物工程(上海)股份有限公司完成。本研究使用的引物见表1。
1.2.2 重组质粒的转化与培养
将重组质粒20b-S123T、20b-S424T和20b-S612T转化E.coli BL21(DE3) 感受态,涂布在氨苄抗性的LB固体平板并置于37 ℃培养箱9~12 h。将平板上出的单菌落接种到LB液体培养基并在37 ℃培养8~9 h,接下来以4%接种量转接到TB发酵培养基,待OD600值达到1时添加0.1 mmol/L IPTG进行诱导,并在20 ℃培养48 h,于不同时间取样并测定酶活性。
将重组质粒9K-S123T、9K-S424T和9K-S612T经Sal I线性化并回收酶切产物。在电压1 500 V、电容25 μF及电阻200 Ω条件下电击转化K.phaffii GS115,涂布于MD平板并放置于30 ℃培养箱待转化子生长。长出单菌落转接到含有4 mg/mL G418的YPD平板,最终获得3个突变体S123T、S424T和S612T。将以上突变体接种在装有40 mL BMGY培养基的250 mL摇瓶中,30 ℃培养24 h。收集发酵液并离心,用无菌生理盐水将菌体洗涤2~3次后重悬于50 mL BMMY培养基当中,30 ℃甲醇诱导蛋白表达,发酵120 h,不同时间取样测定酶活力。
1.2.3 重组蛋白的纯化
将发酵结束的样品经4 000×g离心10 min并收集上清液。使用镍亲和层析纯化柱(GE Healthcare)进行蛋白及突变体的分离纯化。目标蛋白在75 mmol/L咪唑条件下洗脱下来。样品经脱盐柱(GE Healthcare)脱盐,获得最终的纯酶。
1.2.4 最适反应温度及稳定性测定
将纯化后的纯酶分别在40、50、60、70、80 ℃条件下反应10 min,以可溶性淀粉为底物,pH 4.6的醋酸钠缓冲液中测定糖化酶活性,以最高酶活力为100%计算其他温度下的相对值[11]。将同样浓度的纯酶在60 ℃条件下保温120 min,每间隔30 min取样并测定残余酶活力,以未处理前的酶活力为100%。
1.2.5 动力学参数的测定
动力学参数的测定在含有0~2%生玉米淀粉的醋酸钠缓冲液(50 mmol/L, pH 4.6)中进行[12]。使用OriginPro 2021b进行非线性回归曲线的拟合。
1.2.6 酶活力的测定
糖化酶活力测定:底物为2%可溶性淀粉溶液,反应体系为400 μL底物,400 μL pH 4.6 的乙酸-乙酸钠缓冲液,200 μL酶液,于40 ℃反应10 min后,立即加入1 mL二硝基水杨酸(dinitrosalicylic acid,DNS)终止反应,用DNS法测定体系中产生的还原糖(以葡萄糖计),对照为灭活后酶液,依据葡萄糖标准曲线,计算酶活反应体系中产生还原糖[13]。
糖化酶酶活力定义:在pH 4.6,40 ℃反应条件下,1 min水解2%可溶性淀粉溶液产生1 μmol还原糖所需的酶量定义为1个酶活力单位(U)。
生淀粉糖化酶酶活力测定:以2%的生玉米淀粉悬浮液作为底物(用pH 4.6乙酸-乙酸钠缓冲液配制),反应体系为20 mL底物,0.5 mL酶液,于40 ℃反应60 min,立即加入0.5 mL 0.1 mol/L NaOH终止反应,对照为灭活后酶液,反应结束后反应液于8 000 r/min离心5 min,上清液用DNS法测定还原糖浓度(以葡萄糖计)。
生淀粉糖化酶酶活力定义:在pH 4.6,40 ℃反应条件下,1 h水解2%生玉米淀粉悬浮液产生1 μmol 还原糖所需的酶量定义为1个酶活力单位(U)[13]。
1.2.7 蛋白分析
使用BCA试剂盒(天根生物,北京)测定纯化蛋白浓度。采用120 g/L聚丙烯酰胺凝胶进行SDS-PAGE分析,并使用考马斯蓝R-250染色,分子凝胶成像系统分析蛋白条带。蛋白去糖基化反应体系为混合7 μL纯酶(2~10 μg蛋白)、1 μL 10×糖苷缓冲液3和2 μL endo-β-N-乙酰氨基葡萄糖苷酶H(Endo H, New England Biolabs, USA),将该体系放置在37 ℃金属浴处理12 h,样品进行SDS-PAGE分析。
1.2.8 生物信息分析
N-糖基化位点预测通过在线网站NetNGlyc 1.0 Server (http://www.cbs.dtu.dk/services/NetNGlyc/)进行分析。基于Trichoderma reesei葡萄糖淀粉酶(PDB ID:2vn4)的晶体结构,通过I-TASSER (https://zhanggroup.org/ITASSER/)生成RSGA的模型结构。
2 结果与分析
2.1 糖基化位点预测及分析
N-糖基化是毕赤酵母中常见的翻译后修饰,且不同糖基化基序产生的糖基化效果具有一定差异[8]。通过RSGA氨基酸序列分析及糖基化位点预测发现自身存在有2个糖基化位点N422(Asn422-Gly423-Ser424)和N610 (Asn610-Arg611-Ser612),均为Asn-Xaa-Ser形式的糖基化基序。为优化糖基化位点,将Ser424和Ser612分别替换为Thr,获得突变体S424T和S612T。通过NetNGlyc 1.0 Server对突变后序列进行糖基化位点预测,结果显示突变体S424T糖基化预测阈值由0.541 1提高到0.610 2;突变体S612T预测阈值由0.515 0提高到0.581 4,表明氨基酸替换为Thr后被糖基化的效率更高(表2)。另外RSGA还存在一个特殊的糖基化位点N121(Asn121-Pro122-Ser123),将其糖基化基序替换为Asn-Xaa-Thr形式,N-糖基化评估结果显示,该位点由原先的非糖基化位点转变为糖基化位点(表2)。RSGA模拟结构显示糖基化位点处于不同结构域,N121位于不规则loop区,N422位于催化结构域的α-螺旋,N610则处于淀粉结合域(图1)。

图1 RSGA潜在糖基化位点
Fig.1 The potential N-glycosylation sites presented in the modeled structure of RSGA
注:实线框为催化结构域;虚线框为淀粉结合域。
表2 RSGA糖基化位点的预测
Table 2 Prediction of N-glycosylation sites in RSGA
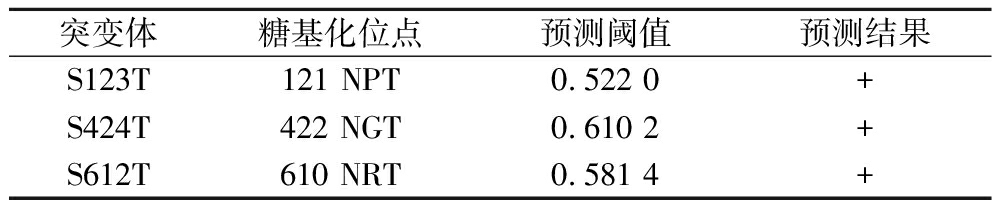
突变体糖基化位点预测阈值预测结果S123T121 NPT0.522 0+S424T422 NGT0.610 2+S612T610 NRT0.581 4+
注:“+”表示正向结果。
2.2 突变体表达纯化与糖基化分析
K.phaffii表达系统具有天然的蛋白翻译后修饰系统[6],是蛋白异源表达的优势宿主之一[14]。通过定点诱变方式构建获得重组质粒9K-S123T、9K-S424T和9K-S612T,经Sal I线性化转化至K.phaffii进行糖基化修饰,获得突变体S123T、S424T和S612T。由于K.phaffii表达的重组蛋白C端均融合有6×His标签,因此通过镍亲和层析完成重组蛋白的纯化。N-糖基化在不同酶之间存在显著差异可导致酶在SDS-PAGE上的蛋白条带变化。使用去糖基化酶EndoH对纯酶进行处理,如SDS-PAGE分析所示,RSGA以及突变体条带上方均出现不同程度的弥散现象,分为主条带和弥散条带(图2)。通常情况下在主条带上面存在弥散条带意味着发生不均一的糖基化[15]。对于RSGA,EndoH去糖基化仅去除弥散条带,不影响其主条带的位置,主要发生异质糖基化。EndoH处理前后,S424T和S612T显示出与RSGA相同的蛋白带模式。而突变体S123T糖基化效果显著,经Endo H处理后,很明显由原先的两条带变成一条带,且条带大小与主条带相近,表明该突变体发生异质糖基化[16]。
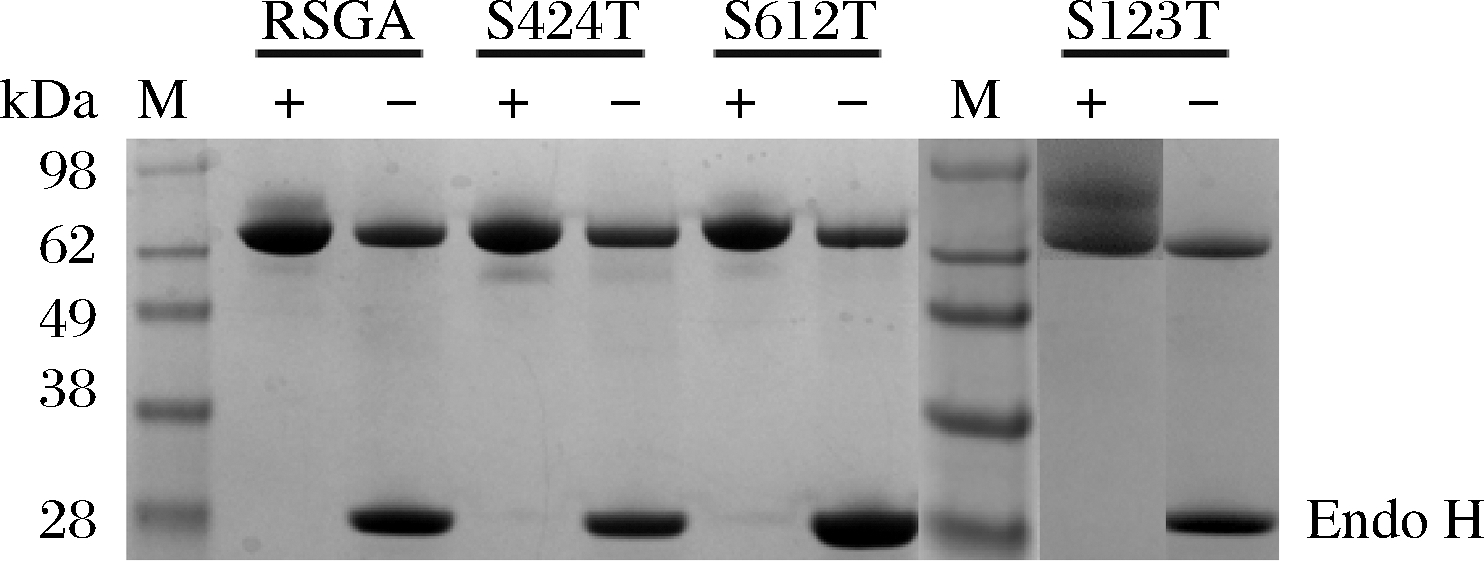
图2 糖基化突变体SDS-PAGE分析
Fig.2 SDS-PAGE analysis of RSGA and mutants
2.3 糖基化对酶的比酶活力的影响
前期研究中,通过组合突变去除RSGA存在的糖基化修饰,结果引起比酶活力的急剧下降[5],表明糖基化修饰对RSGA催化活性的重要性。将RSGA氨基酸序列中存在的Asn-Xaa-Ser糖基化基序替换为Asn-Xaa-Thr,能有效促进其比酶活力的提高。以生玉米淀粉为底物,在40 ℃,pH 4.6条件下测定的突变体活性以及蛋白浓度结果如图3-a所示。突变体S123T和S424T生淀粉酶活力分别是RSGA酶活力的1.25倍和1.02倍。此外,相同处理条件下,突变体S123T的蛋白表达量提高了9.3%。进一步对突变体的比酶活力分析发现,它们都有不同幅度的提高(图3-b)。突变体S424T和S612T比酶活力较RSGA分别提高10%和5%。另外,引入的糖基化突变体S123T对生玉米淀粉的催化活性提高17%,这可能是由于淀粉链和附着在Asn121上的额外聚糖链之间的动态相互作用,促进了酶与底物之间的降解反应。此外,将突变体在E.coli BL21(DE3)中表达,排除残基突变带来的影响。对重组突变体酶的比酶活力进行表征发现,突变体比酶活力呈现下降趋势,表明在S123T、S424T和S612T的催化反应中糖基化起积极作用,而不是残基突变(图3-b)。
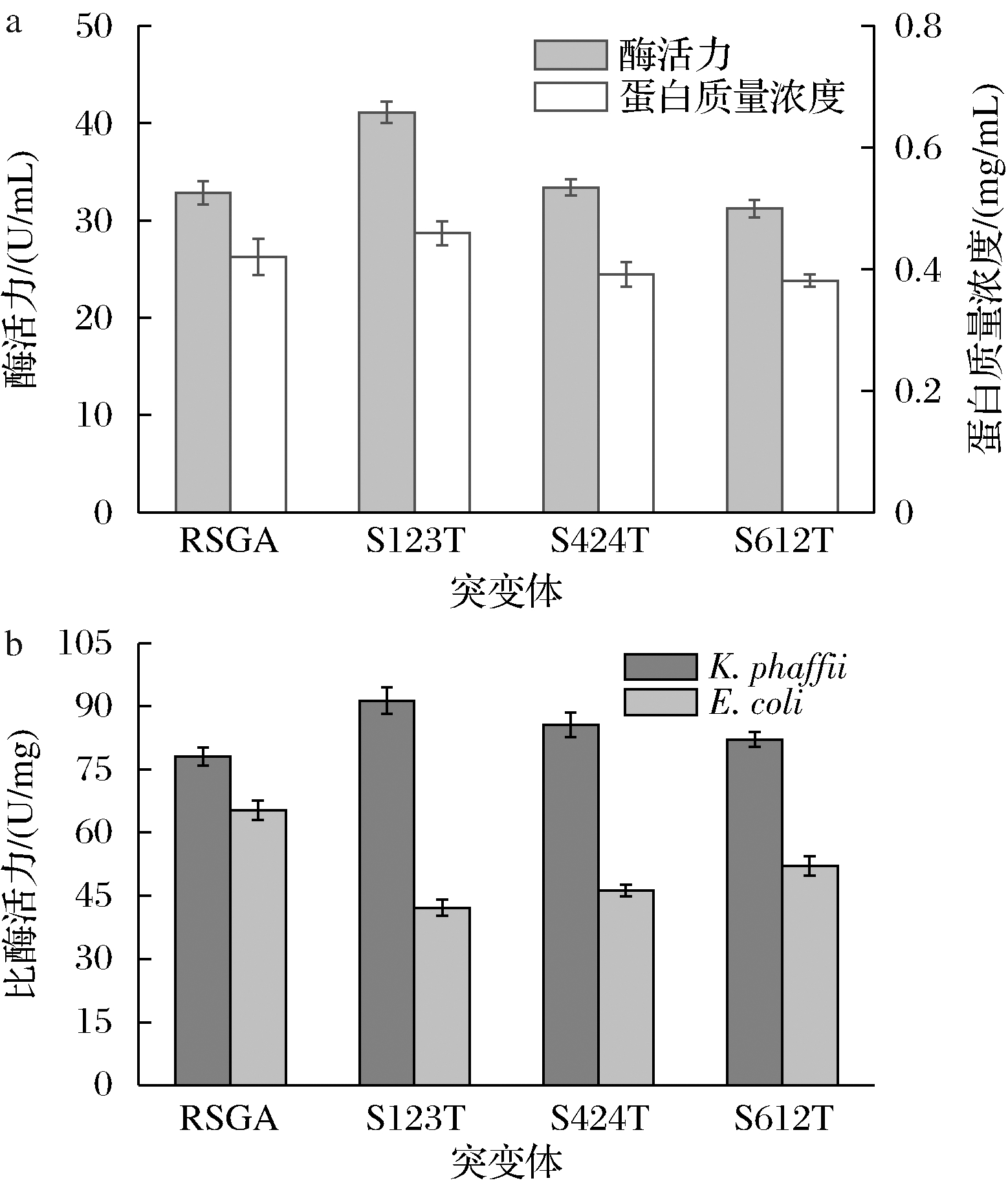
a-RSGA及突变体酶活力和蛋白质量浓度分析;b-RSGA及突变体在K.phaffii和E.coli表达后的比酶活力
图3 RSGA及突变体的蛋白表达水平及比酶活力分析
Fig.3 Protein expression levels and specific enzyme activity analysis of RSGA and mutants
2.4 糖基化对酶热稳定性的影响
已有研究表明,糖基化会影响酶的稳定性。将纤维二糖水解酶中的糖基化位点Asn384突变后,该酶在62 ℃条件下失活速度加快[17],表明糖基化修饰对稳定性具有促进作用。然而,在内切葡聚糖酶的63位点引入Asn-Xaa-Thr糖基化基序后,酶的热稳定性降低[9]。因此,为进一步明确优化后糖基化位点对RSGA酶学性质的影响,以可溶性淀粉为底物,测定RSGA和糖基化突变体的最适反应温度和在60 ℃下酶的稳定性。首先分析突变体的最适反应温度,在不同反应温度下孵育10 min后测定酶活力(40~80 ℃)。结果如图4-a所示,突变体S123T最适反应温度下降到60 ℃,低于Penicillium oxalicum来源的生淀粉糖化酶的最适反应温度65 ℃[2]。最适反应温度的降低更加有利于RSGA在低于淀粉糊化温度下降解生淀粉的应用,一定程度上降低能源消耗。进一步将RSGA与突变体纯酶在60 ℃条件下处理2 h,每间隔30 min取样并测定样品的残余酶活力。结果如图4-b所示,突变体S612T在孵育60 min后仍保留64%的残余酶活力,较RSGA的残余酶活力提高14%,酶的稳定性有所提高,这可能与糖基化的位点有关。突变体S612T是在Asn610位点处附着聚糖链,该位点处于淀粉结构域柔性区域部分[5]。通常情况下,柔性区域内的聚糖链可能会限制构象空间,从而提高其稳定性[18]。突变体S123T和S424T在孵育30 min后,酶的活性急剧下降,仅保留了35%,酶的热稳定性降低。不同位点糖基化修饰对酶产生的稳定效果具有差异,以上结果与前期引入不同位点的糖基化修饰结果是一致的[10]。

a-RSGA及突变体最适反应温度;b-RSGA及突变体温度稳定性分析
图4 RSGA及其糖基化突变体酶学性质分析
Fig.4 Stability analysis of RSGA and mutants
2.5 糖基化对酶促动力学参数的影响
酶促动力学参数是酶的基本特征常数之一,反应酶与底物之间的催化效果。糖基化使蛋白特定氨基酸上增加了多糖链,两者之间共价调节糖蛋白从而影响动力学性质[19],从而一定程度上增加了底物和蛋白之间的亲和力,促进酶与底物结合[20]。以生玉米淀粉为底物,在40 ℃,pH 4.6条件下考察了RSGA以及糖基化突变体在催化反应效率方面的变化及差异。结果表明,S123T、S424T和S612T的kcat/Km值分别是RSGA值的1.31、1.29和1.15倍(表3)。虽然突变体S123T和S424T的kcat值有所降低,但从降低的Km值可以看出,3个突变体均显著提高了对生玉米淀粉的亲和力,与催化效率提升的趋势是一致的,表明亲和力是突变体催化效率提高的主要原因(表3)。残基Asn121处于相对灵活的loop区域,这可能促进活性位点上氨基酸的功能,其附着的聚糖链不可避免与淀粉链发生相互作用,从而促进对生玉米淀粉的降解效率[10]。另外,RSGA结构模型显示,Asn422和Asn610分别位于酶的催化域和淀粉结合域(图1)。Asn422位点的糖基化可能引入了与残基的相互作用,有利于增强酶与底物之间的亲和力[5]。淀粉结合域包含2组淀粉结合位点S1和S2,对生淀粉的结合具有重要作用,而糖基化位点Asn610更加靠近淀粉结合位点,这可能使连接在Asn610上的聚糖链通过氢键相互作用促进淀粉结合位点与生玉米淀粉的结合,从而提高酶与底物之间的催化效率[5]。
表3 RSGA及突变体动力学分析
Table 3 Kinetic parameters of RSGA and its mutants against raw corn starch
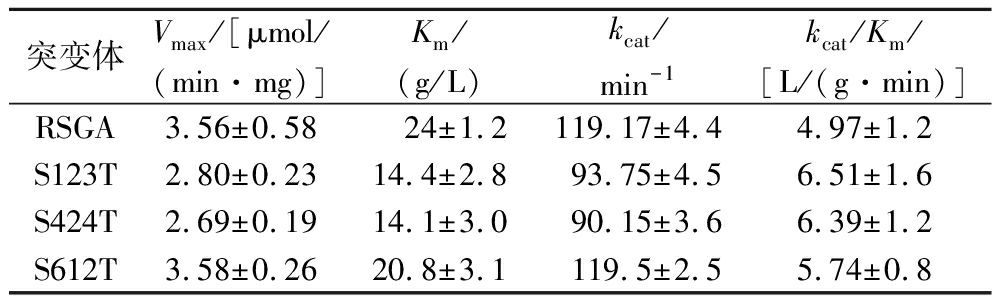
突变体Vmax/[μmol/(min·mg)]Km/(g/L)kcat/min-1kcat/Km/[L/(g·min)]RSGA3.56±0.58 24±1.2119.17±4.44.97±1.2S123T2.80±0.2314.4±2.893.75±4.56.51±1.6S424T2.69±0.1914.1±3.090.15±3.66.39±1.2S612T3.58±0.2620.8±3.1119.5±2.55.74±0.8
注:数值为3次重复的平均值±标准差。
3 结论
生淀粉糖化酶作为可以在淀粉糊化温度下降解生淀粉的水解酶,简化了淀粉糖生产工艺并降低能源消耗。本研究为提高A.fumigatus来源的RSGA对生玉米淀粉的催化效率,将RSGA氨基酸序列中3个含有Asn-Xaa-Ser糖基化基序替换为Asn-Xaa-Thr并利用NetNGlyc 1.0进行糖基化评估。突变体在K.phaffii中完成糖基化并获得突变体S123T、S424T和S612T。经去糖基化酶EndoH处理及SDS-PAGE分析确定突变体均成功糖基化。在此基础上研究了突变体S123T、S424T和S612T糖基化对催化活性、热稳定性以及酶促动力学参数的影响。通过将糖基化基序中的Ser突变为Thr,突变体S123T、S424T和S612T对生玉米淀粉催化效率分别提高到RSGA的1.31、1.29和1.15倍。此外,突变体S123T引入新的糖基化使其表达量提高了9.3%,且最适反应温度从70 ℃降低到60 ℃。本研究通过RSGA的糖基化基序改造,有效提高了酶的催化效率,是一种改善酶催化效率的新策略。
[1] SUN H Y, ZHAO P J, GE X Y, et al. Recent advances in microbial raw starch degrading enzymes[J]. Applied Biochemistry and Biotechnology, 2010, 160(4):988-1003.
[2] XU Q S, YAN Y S, FENG J X. Efficient hydrolysis of raw starch and ethanol fermentation: A novel raw starch-digesting glucoamylase from Penicillium oxalicum[J]. Biotechnology for Biofuels, 2016, 9:216.
[3] HATA Y, ISHIDA H, KOJIMA Y, et al. Comparison of two glucoamylases produced by Aspergillus oryzae in solid-state culture (koji) and in submerged culture[J]. Journal of Fermentation and Bioengineering, 1997, 84(6):532-537.
[4] ZHANG M Y, ZHAO S, NING Y N, et al. Identification of an essential regulator controlling the production of raw-starch-digesting glucoamylase in Penicillium oxalicum[J]. Biotechnology for Biofuels, 2019, 12:7.
[5] SONG W Y, TONG Y, LI Y, et al. Expression and characterization of a raw-starch glucoamylase from Aspergillus fumigatus[J]. Process Biochemistry, 2021, 111:97-104.
[6] WANG N, WANG K Y, XU F F, et al. The effect of N-glycosylation on the expression of the tetanus toxin fragment C in Pichia pastoris[J]. Protein Expression and Purification, 2020, 166:105503.
[7] WEERAPANA E, IMPERIALI B. Asparagine-linked protein glycosylation: From eukaryotic to prokaryotic systems[J]. Glycobiology, 2006, 16(6):91R-101R.
[8] KASTURI L, CHEN H, SHAKIN-ESHLEMAN S H. Regulation of N-linked core glycosylation: Use of a site-directed mutagenesis approach to identify Asn-Xaa-Ser/Thr sequons that are poor oligosaccharide acceptors[J]. The Biochemical Journal, 1997, 323(Pt 2):415-419.
[9] HAN C, WANG Q Q, SUN Y X, et al. Improvement of the catalytic activity and thermostability of a hyperthermostable endoglucanase by optimizing N-glycosylation sites[J]. Biotechnology for Biofuels, 2020, 13:30.
[10] SONG W Y, LI Y Y, TONG Y, et al. Improving the catalytic efficiency of Aspergillus fumigatus glucoamylase toward raw starch by engineering its N-glycosylation sites and saturation mutation[J]. Journal of Agricultural and Food Chemistry, 2022, 70(39):12672-12680.
[11] FANG W, XUE S S, DENG P J, et al. AmyZ1: A novel α-amylase from marine bacterium Pontibacillus sp. ZY with high activity toward raw starches[J]. Biotechnology for Biofuels, 2019, 12(1):1-15.
[12] 刘庆涛. Bacillus paralicheniformis酸性脲酶表达、分子改造及用于黄酒中氨基甲酸乙酯消减[D]. 无锡: 江南大学, 2019.
LIU Q T. Expression and molecular engineering of Bacillus paralichenifromis acid urease for reducing urethane in A model rice wine[D]. Wuxi: Jiangnan University, 2019.
[13] 韦荣霞, 张梁, 石贵阳. Aspergillus sp. RSD生淀粉糖化酶的分离纯化及酶学性质[J]. 微生物学通报, 2014, 41(1):17-25.
WEI R X, ZHANG L, SHI G Y. Purification and characterization of a raw starch-digesting glucoamylase from Aspergillus sp. RSD[J]. Microbiology China, 2014, 41(1):17-25.
[14] WANG J J, WANG X L, SHI L, et al. Methanol-independent protein expression by AOX1 promoter with trans-acting elements engineering and glucose-glycerol-shift induction in Pichia pastoris[J]. Scientific Reports, 2017, 7:41850.
[15] WEI W, CHEN L, ZOU G, et al. N-glycosylation affects the proper folding, enzymatic characteristics and production of a fungal β-glucosidase[J]. Biotechnology and Bioengineering, 2013, 110(12):3075-3084.
[16] XU G F, WU Y W, ZHANG Y L, et al. Role of N-glycosylation on the specific activity of a Coprinopsis cinerea laccase Lcc9 expressed in Pichia pastoris[J]. Journal of Bioscience and Bioengineering, 2019, 128(5):518-524.
[17] QI F F, ZHANG W X, ZHANG F J, et al. Deciphering the effect of the different N-glycosylation sites on the secretion, activity, and stability of cellobiohydrolase I from Trichoderma reesei[J]. Applied and Environmental Microbiology, 2014, 80(13):3962-3971.
[18] DOTSENKO A S, GUSAKOV A V, VOLKOV P V, et al. N-linked glycosylation of recombinant cellobiohydrolase I (Cel7A) from Penicillium verruculosum and its effect on the enzyme activity[J]. Biotechnology and Bioengineering, 2016, 113(2):283-291.
[19] MOSER J W, WILSON I B H, DRAGOSITS M. The adaptive landscape of wildtype and glycosylation-deficient populations of the industrial yeast Pichia pastoris[J]. BMC Genomics, 2017, 18(1):1-16.
[20] JEOH T, MICHENER W, HIMMEL M E, et al. Implications of cellobiohydrolase glycosylation for use in biomass conversion[J]. Biotechnology for Biofuels, 2008, 1(1):1-13.