鸡蛋过敏是一种过敏性疾病,能够引发严重的临床症状,甚至导致休克、死亡,严重影响着人类的生活质量和生命安全[1]。鸡蛋过敏多发于在婴幼儿时期,占婴幼儿和儿童食物过敏的35%;同时在成人中也存在广泛,占成年人食物过敏的12%[2-3]。在我国,鸡蛋是除牛奶外,第二大导致婴幼儿和儿童过敏的食物[4]。而在某些发达国家,鸡蛋过敏的患病率甚至超过了牛奶[5]。鸡蛋过敏原主要有6种,其中4种存在于蛋清中,包括卵类黏蛋白(ovomucoid,OVM,Gal d 1)、卵白蛋白(ovalbumin,OVA,Gal d 2)、卵转铁蛋白(ovotransferrin,OVT,Gal d 3)和溶菌酶(lysozyme,Lys,Gal d 4),另外2种存在于蛋黄中,分别为 α-卵黄蛋白(α-livetin,Gal d 5)和卵黄糖蛋白 42(yolkglycoprotein42,Gal d 6)[6]。其中,溶菌酶是存在于鸡蛋蛋清中的一种过敏原蛋白,占蛋清含量的3.5%,分子质量为14.3 kDa,它常被用于制药产品、奶酪和葡萄酒行业。但多项研究表明,鸡蛋过敏患者血液中存在溶菌酶特异性IgE抗体[7-8],这说明Gal d 4也是一类重要的蛋清过敏原。
抗原表位,即抗原决定簇,是过敏原分子中决定抗原特异性的特殊化学基团。表位按照结构特点通常分为线性表位和构象表位,前者由连续的氨基酸组成,后者则是不相连的氨基酸残基通过折叠所形成的空间结构,按照与细胞结合的类型可以分为T细胞表位和B细胞表位[9]。B细胞表位是可被B细胞表面受体或B细胞分泌的抗体特异性识别并相互结合的表位[10]。T细胞表位是通过主要相容组织复合体(major histocompatibility complex,MHC)分子提呈的,CD8+T细胞表达的T细胞抗原受体(T cell receptor,TCR)识别的表位由MHC-I类分子提呈,CD4+T细胞表达的TCR识别的表位由MHC-II类分子提呈[11]。T细胞表位属于线性表位,没有构象结构域。它们不能与细胞结合的IgE交联或激活肥大细胞和嗜碱性粒细胞,但可以引发T细胞产生增殖、分化,是决定过敏反应的关键。因此,获得B细胞表位和T细胞表位信息是充分了解食物过敏致敏机理的基础。
目前,已有大量研究对鸡蛋蛋清致敏原Gal d 1、Gal d 2 和 Gal d 3 的线性表位进行了研究。但目前关于Gal d 4的线性表位尚未被表征。目前来说,线性表位的分析方法主要有传统化学切割法、重叠合成肽库法、核磁共振分析法、质谱技术以及生物信息学预测方法[12]。重叠合成肽等方法由于其高昂的实验成本以及较大的盲目性使其不适合大规模应用[13],生物信息学方法是一种新兴的预测线性表位的方法[14],其原理是对已知的线性表位特点进行归纳后,发现亲水性残基与表面可及性较高区域可能位于分子表面,因此这些区域相比较而言形成抗原表位的可能性较大,可塑性较高的区域表明这部分具有较好的柔韧性,也容易形成利于抗体能够识别的空间结构,是表位所在区域的概率也较高[15]。生物信息学方法对食物过敏原表位的预测有利于研究潜在的抗原表位,在一定程度上可以减少分析过敏原结构与功能所消耗的时间与费用[16]。
因此,本研究拟通过使用生物信息学方法预测鸡蛋过敏原Gal d 4的线性表位,并对其线性表位进行空间定位,随后对预测所得B细胞线性表位进行鉴定,为进一步认识鸡蛋过敏原Gal d 4的结构与功能奠定一定基础。
1 材料与方法
1.1 生物信息学方法分析鸡蛋过敏原Gal d 4 的线性表位
在Uniprot数据库获取鸡蛋过敏原Gal d 4的氨基酸序列。使用SignalP-5.0 services软件对Gal d 4信号肽进行预测。使用生物信息学软件DNAStar Protean中的Hoop-woods的氨基酸亲水性方案[17]、Emini的表面可及性算法[18]、Kparlus-Schuzl的可塑性分析[19]和Jameson-wolf的抗原指数分析方案[20],对Gal d 4的一级序列性质进行分析,综合分析这些参数预测结果的重叠部分后,得出DNAStar预测所得的鸡蛋过敏原Gal d 4的B细胞线性表位序列。
分别使用Immunomedicine Group中的predicted antigenic peptides(IGPAP)、Bepipred-2.0 server和ABCpred在线工具对Gal d 4的B细胞线性表位进行预测。筛选以上3种方法预测所得的B细胞线性表位的重叠区域作为最终所得的Gal d 4的B细胞线性表位序列。
分别利用SYFPEITHI、NetMHC和NetCTL三种在线工具预测Gal d 4 的CD8+ T细胞表位。表位肽段长度设置为9 AA。MHC类型选择人白细胞抗原(human leukocyte antigen, HLA)-I类分子HLA-A*02:01(A2)和HLA-A*03:01(A3)作为限定条件。SYFPEITHI分析结果选取得分在前2%的肽段作为可能性较大的表位肽段;NetMHC分析结果亲和值<50 nm的肽段作为强结合肽段,50 nm<亲和值<500 nm的肽段为弱结合肽段;NetCTL预测所得表位鉴定的阈值为0.75。
利用NetMHCⅡpan 4.0 Serer在线工具预测Gal d 4 的CD4+ T细胞表位。表位肽段的长度设置为15 AA,选取HLA-Ⅱ类分子 (HLA-DRB1*0101、HLA-DRB1*0301、HLA-DRB1*0401、HLA-DRB1*0701、HLA-DRB1*0901、HLA-DRB1*1101、HLA-DRB1*1302、HLA-DRB1*1501)作为预测限定条件。表位肽段与HLA-Ⅱ类分子的亲和性阈值为50 nm,亲和值<50 nm的肽段为强结合肽段,50 nm<亲和值<500 nm的肽段为弱结合肽段。
1.2 生物信息学方法分析鸡蛋过敏原Gal d 4二级结构
使用DNAStar Protean载入Uniprot数据库中下载的FASTA格式的鸡蛋过敏原Gal d 4的氨基酸序列,对其二级结构使用Chou-Fasman以及Garnier-Robson方案进行预测分析。
1.3 空间定位
在PDB数据库中查找鸡蛋过敏原Gal d 4的三级结构模型,将使用生物信息学软件预测所得的Gal d 4的B细胞线性表位序列进行空间定位。
1.4 免疫活性鉴定
生物信息学软件预测所得的B细胞线性表位经Fmoc法固相合成,采用高效液相色谱分析纯度。收集的6名鸡蛋过敏患者的血清,采用间接竞争ELISA实验对合成的预测线性表位进行IgE结合活性验证。根据FU等[21]的方法略有改动,使用纯化的Gal d 4包被在96孔板上孵育过夜。3%牛血清白蛋白封闭2 h后,合成肽(B1~B5)与单个血清样品在37 ℃下孵育1.5 h,一抗为收集的鸡蛋过敏患者血清,山羊抗人IgE-辣根过氧化物酶为二抗,稀释度均为1∶50。H2SO4终止反应后,在450 nm处测定吸光度。IgE结合活性抑制率按公式(1)计算:
抑制率/%=(1-OD450/OD0)×100
(1)
式中:OD0,空白样吸光度;OD450,450 nm处吸光度。
1.5 数据处理
数据统计与图标绘制工具主要为DNAStar软件、SignalP- 5.0 services(https://services.healthtech.dtu.dk/)、Predicted Antigenic Peptides(http://imed.med.ucm.es/)、Bepipred-2.0 server(https://webs.iiitd.edu.in/raghava/abcpred/ABC_submission.html)、ABCpred(http://crdd.osdd.net/raghava/abcpred/)、SYFPEITHI(http://www.syfpeithi.de/bin/MHCServer.dll/EpitopePrediction.htm)、NetMHC(https://services.healthtech.dtu.dk/service.php?NetMHC-4.0)、NetCTL(https://services.healthtech.dtu.dk/service.php?NetCTL-1.2)。
2 结果与分析
2.1 鸡蛋过敏原Gal d 4信号肽预测
如图1所示,1MRSLLILVLCFLPLAALG18前18个氨基酸残基为Gal d 4信号肽的可能性为99.72%。信号肽是引导新和成的蛋白质向分泌通路转移的短肽链,通常疏水性较强,不适合作为线性抗原表位。
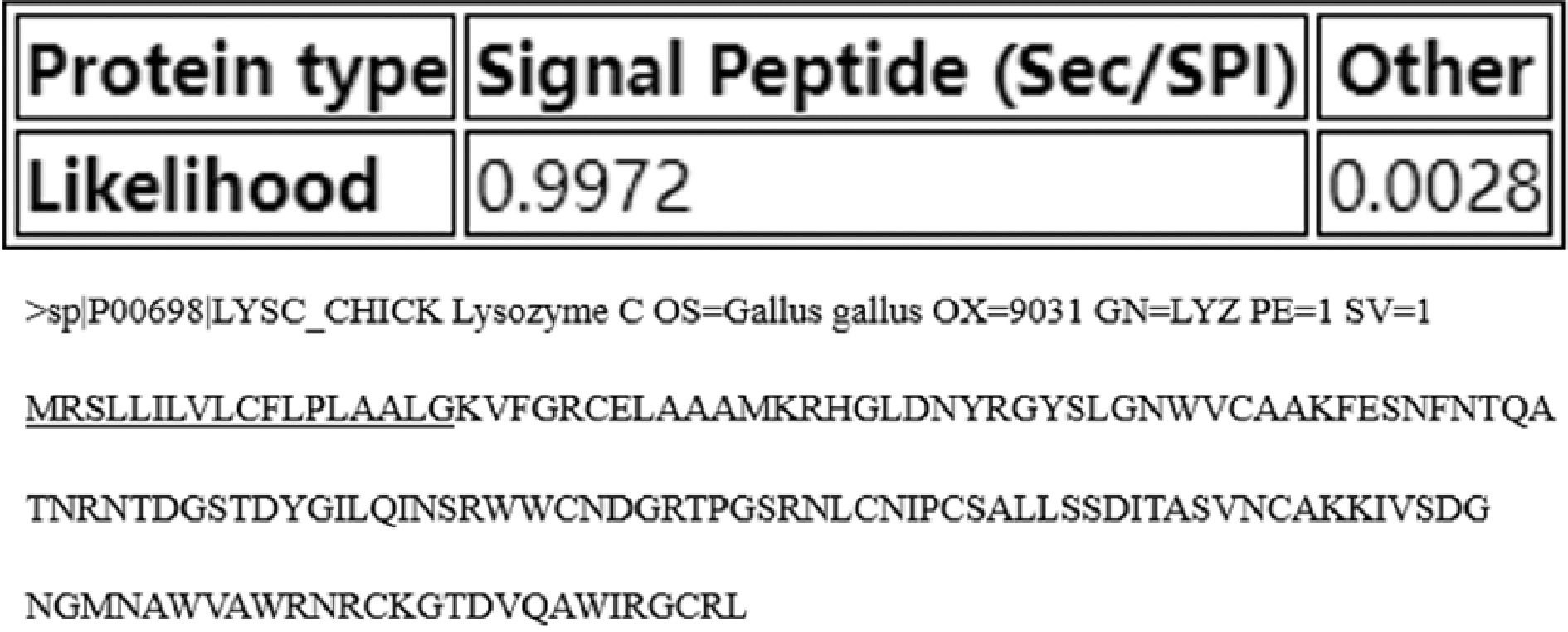
图1 SignalP- 5.0 services预测所得Gal d 4信号肽序列
Fig.1 The Gal d 4 signal peptide sequence was predicted by signalP-5.0 Services
注:图中下划线部分为预测所得信号肽区域。
2.2 DNAStar预测鸡蛋过敏原Gal d 4的B细胞线性表位
亲水性和表面可及性高的区域一般分布在蛋白质分子的表面,易产生表位与抗体结合的区域,而可塑性较强的区域由于其易发生折叠和扭曲,因此,也容易形成利于抗体能够识别的空间结构[22]。如图2所示,在氨基酸亲水性方案分析中,亲水性指数>0表明亲水性好,鸡蛋过敏原Gal d 4的亲水性区域分布较为均匀,亲水性较好的氨基酸肽段为AA 22~26、AA 27~37、AA 52~55、AA 60~69、AA 84~92、AA 111~122和AA 130~137;以表面可及性指数>1为筛选条件,表面可及性较好的氨基酸肽段为AA 29~41、AA 52~67、AA 84~91和AA 128~136;在可塑性方案分析中,AA 35~40、AA 52~70、AA 83~93、AA 102~107、AA 117~122和AA 131~137的氨基酸肽段具有一定的柔韧性,形成表位的可能性较大,容易与抗体进行嵌合[23];在抗原指数分析中,以抗原指数>0为筛选条件,结果显示抗原指数较高的氨基酸肽段为AA 19~26、AA 27~45、AA 51~72、AA 77~94、AA 102~107、AA 110~124、AA 128~137和AA 143~146,这些区域可能含有潜在的优势抗原表位。
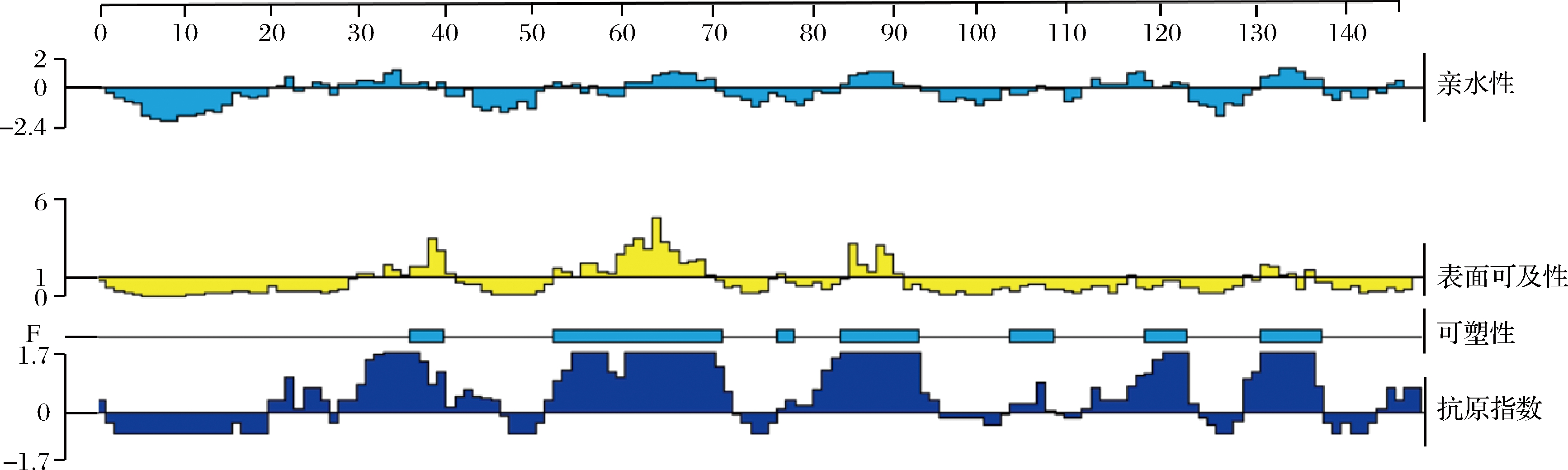
图2 DNAStar对Gal d 4的一级氨基酸序列分析
Fig.2 Sequence analysis of primary amino acid of Gal d 4 by DNAStar
综合以上参数,筛选出同时满足4个条件的氨基酸序列作为鸡蛋过敏原Gal d 4的B细胞线性表位候选氨基酸序列。结果如表1所示,DNAStar预测Gal d 4 B细胞线性表位为:19KVFGRCEL26、27AAAMKRHGLDNYRG40、52FESNFNTQATNRNTDG67、83NDGRTPGSRNL93、117VSDGNGMN124和130RNRCKGTD137。
表1 DNAStar预测Gal d 4 B细胞线性表位
Table 1 DNAStar prediction of B cell linear epitopes of Gal d 4

序号氨基酸序列起始位点1KVFGRCEL192AAAMKRHGLDNYRG273FESNFNTQATNRNTDG524NDGRTPGSRNL835VSDGNGMN1176RNRCKGTD130
2.3 IGPAP预测鸡蛋过敏原Gal d 4的B细胞线性表位
进一步采用IGPAP预测鸡蛋过敏原Gal d 4的B细胞线性表位,根据KOLASKAR等[24]的方法预测抗原表位,结果如表2所示,IGPAP预测Gal d 4的B细胞线性表位为:4LLILVLCFLPLAALGKVFGRCELAA28、43LGNWVCAA50、92NLCNIPCSALLSSDITASVNCAKKI116和134KGTDVQAWIRG144。
表2 IGPAP预测Gal d 4 B细胞线性表位
Table 2 IGPAP prediction of B cell linear epitopes of Gal d 4

序号氨基酸序列起始位点1LLILVLCFLPLAALGKVFGRCELAA 42LGNWVCAA433NLCNIPCSALLSSDITASVNCAKKI924KGTDVQAWIRG134
2.4 Bepipred预测鸡蛋过敏原Gal d 4的B细胞线性表位
同时,采用Bepipred-2.0 server在线工具使用隐马尔可夫模型和亲水性参数评分预测Gal d 4 B细胞线性表位序列[25]。结果如表3所示,Bepipred-2.0 server预测Gal d 4 B细胞线性表位为:19KVFGRCELAAAMKRHGLDNYRGYSLGN45、57NTQATNRNTDGST-DYGIL74、76INSRWWCNDGRTPGSRNLCNI96和115KIVSDGNGMNAWVAWRNRCKGTDVQAWIRG144。
表3 Bepipred预测Gal d 4 B细胞线性表位
Table 3 Bepipred prediction of B cell linear epitopes of Gal d 4

序号氨基酸序列起始位点1KVFGRCELAAAMKRHGLDNYRGYSLGN192NTQATNRNTDGSTDYGIL573INSRWWCNDGRTPGSRNLCNI764KIVSDGNGMNAWVAWRNRCKGTDVQAWIRG115
2.5 ABCpred预测鸡蛋过敏原Gal d 4 B细胞线性表位
最后,将鸡蛋过敏原Gal d 4氨基酸序列提交到ABCpred网络服务器后,利用人工神经网络模型预测出Gal d 4的B细胞线性表位序列[26]。结果如表4所示,ABCpred预测Gal d 4 B细胞线性表位为:19KVFGRCELAAAMKR32、44GNWVCAAKFESNFNTQA60、65TDGSTDYGILQ75、78SRWWCND84、87TPGSRNLCNIP97和132RCKGTDVQAWI142。
表4 ABCpred预测Gal d 4 B细胞线性表位
Table 4 ABCpred prediction of B cell linear epitopes of Gal d 4
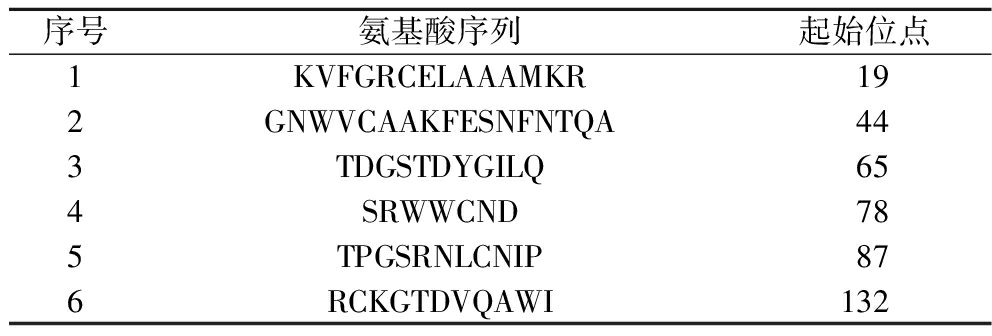
序号氨基酸序列起始位点1KVFGRCELAAAMKR192GNWVCAAKFESNFNTQA443TDGSTDYGILQ654SRWWCND785TPGSRNLCNIP876RCKGTDVQAWI132
2.6 鸡蛋过敏原Gal d 4 B细胞线性表位的综合分析
综合DNAStar、IGPAP、Bepipred-2.0 server以及ABCpred预测所得的Gal d 4 B细胞线性表位序列结果,如图3所示,4种软件预测重叠氨基酸序列为:19KVFGRCELAA28、92NL93和134KGTD137。3种软件预测重叠氨基酸序列为:29AMKR32、44GN45、57NTQA60、65TDG67、83ND84、87TPGSR91、94CNI96、132RC133、138VQAWI142。因此,综合以上结果,将3种及以上软件预测重叠氨基酸数≥3的序列作为鸡蛋过敏原Gal d 4的B细胞线性表位,这些氨基酸序列是B细胞表位的可能性较大,可作为研究重点。如表5所示,综合分析所得Gal d 4的B细胞线性表位为:19KVFGRCELAAAM-KR32、57NTQA60、65TDG67、87TP-GSRNLCNI96和132RCKGTDVQAWI142。
表5 不同软件预测Gal d 4 B细胞线性表位的氨基酸序列
Table 5 Amino acid sequences of B cells linear epitopes of Gal d 4 predicted by different software

序号氨基酸序列起始位点B1KVFGRCELAAAMKR19B2NTQA57B3TDG65B4TPGSRNLCNI87B5RCKGTDVQAWI132

图3 不同软件预测Gal d 4的B细胞线性表位的综合分析
Fig.3 Comprehensive analysis of B cells linear epitopes of Gal d 4 predicted by different software
注:圆角矩形方框内为4种软件预测重叠氨基酸序列;直角矩形方框内为3种软件预测重叠氨基酸序列。
2.7 鸡蛋过敏原Gal d 4的CD8+ T细胞表位预测
SYFPEITHI、NetMHC和NetCTL三种在线工具可切断肽链羧基的蛋白酶体并分析对肽段进行加工和转运的速率,从而计算得分。预测所得Gal d 4 的CD8+ T细胞表位结果如表6所示,与HLA-I类分子结合较好的表位肽段有42SLGNWVCAA50、100ALLSSDITA108。
表6 预测所得Gal d 4 的CD8+ T细胞表位
Table 6 Comprehensive analysis of CD8+ T cell epitopes of Gal d 4
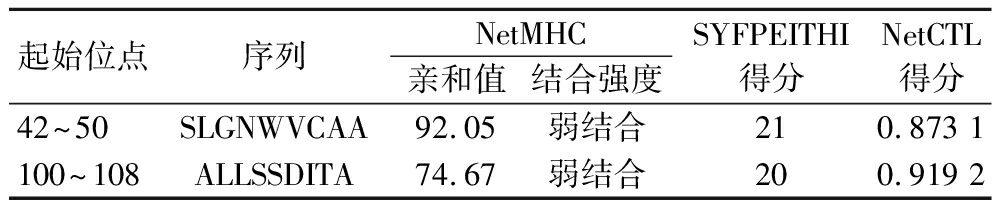
起始位点序列NetMHC亲和值结合强度SYFPEITHI得分NetCTL得分42~50SLGNWVCAA92.05弱结合210.873 1100~108ALLSSDITA74.67弱结合200.919 2
注:亲和值越小结合越强,亲和值越大结合越弱。
2.8 鸡蛋过敏原Gal d 4的CD4+ T细胞表位预测
利用NetMHCⅡpan 4.0 Serer 在线工具预测Gal d 4 的CD4+ T细胞表位结果如表7所示。每个HLA-Ⅱ类分子结合的肽段数量都有所不同,其中,HLA-DRB1*0301、HLA-DRB1*0401和HLA-DRB1*1501分别含有4、5、4个强结合肽段,大部分HLA-Ⅱ类分子都含有数目不同的弱结合肽段。
表7 预测所得Gal d 4 的CD4+ T细胞表位
Table 7 Comprehensive analysis of CD4+ T cell epitopes of Gal d 4
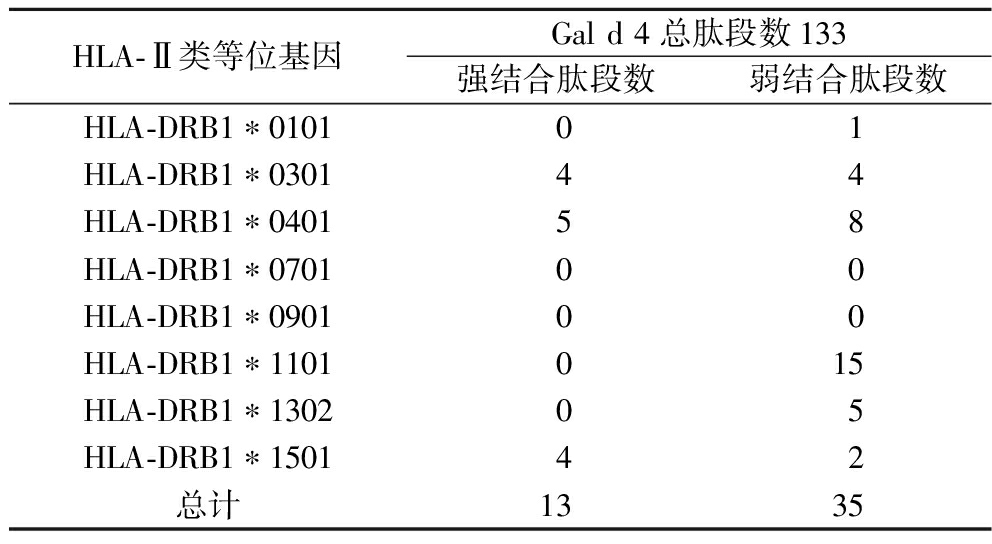
HLA-Ⅱ类等位基因Gal d 4总肽段数133强结合肽段数弱结合肽段数HLA-DRB1∗010101HLA-DRB1∗030144HLA-DRB1∗040158HLA-DRB1∗070100HLA-DRB1∗090100HLA-DRB1∗1101015HLA-DRB1∗130205HLA-DRB1∗150142总计1335
2.9 鸡蛋过敏原Gal d 4的T细胞表位综合分析
鸡蛋过敏原Gal d 4的T细胞表位综合分析如表8所示。根据CD4+ T抗原表位覆盖CD8+ T细胞抗原表位的原则,得到2条优势表位,即42SLGNWVCAA50和100ALLSSDITA108。
表8 预测所得Gal d 4 的T细胞表位综合分析
Table 8 Comprehensive analysis of T cell epitopes of Gal d 4
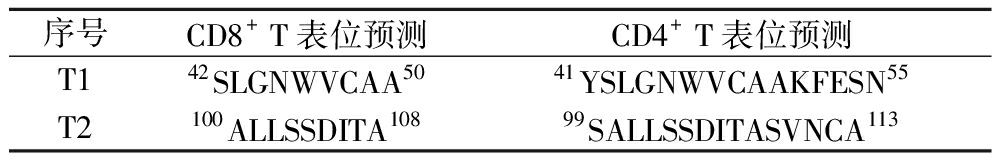
序号CD8+ T表位预测CD4+ T表位预测T142SLGNWVCAA5041YSLGNWVCAAKFESN55T2100ALLSSDITA10899SALLSSDITASVNCA113
2.10 鸡蛋过敏原Gal d 4的二级结构预测
图4为使用DNAStar软件中的Chou-Fasman和Garnier-Robson方案对Gal d 4二级结构进行预测的结果[27]。分析可知,2种方案预测该蛋白二级结构的类型中α-螺旋和β-转角结构占据了主要优势,β-折叠次之。总体来看,Gal d 4二级结构中有序的螺旋、转角和折叠占据了主要优势,无规则卷曲的比例较小,反映了该蛋白具有较为致密的结构。

图4 Gal d 4的二级结构预测
Fig.4 Prediction of secondary structure of Gal d 4
2.11 鸡蛋过敏原Gal d 4线性表位的空间定位
通过在PDB数据库下载鸡蛋过敏原Gal d 4三维空间结构,利用PyMOL软件对表5和表8中预测所得的线性表位在鸡蛋过敏原Gal d 4的空间结构中进行定位,结果如图5所示。如图5-A1所示,图中彩色区域是预测所得B细胞线性表位在Gal d 4分子飘带模型的空间结构中的定位,5个模拟表位均已在图中用数字标注。其中,19KVFGRCELAAAMKR32位于α-螺旋与无规则卷曲的连接处;57NTQA60位于β-折叠与无规则卷曲的连接处;65TDG67位于β-折叠与β-转角的连接处;87TPGSRNLCNI96和132RCKGTDVQAWI142位于无规则卷曲区域。α-螺旋与β-折叠的结构特征能够较好的保持稳定,β-转角与无规则卷曲具有较好的可塑性以及表面可及性,有利于抗原表位的形成,这表明5个模拟表位成为B细胞线性表位的潜力较大。图5-A2~图5-A3分别为Gal d 4表面分子模型正视图与翻转180°后的背视图。如图所示,2个视图中的5个B细胞模拟表位基本完全暴露在外,具有较好的表面可及性以及较大的接触面积,利于在溶液的环境中与抗体互相作用。值得注意的是,在图5-A2和图5-A3中,19KVFGRCELAAAMKR32、57NTQA60和132RCKGTDVQAWI142、65TDG67和87TPGSRNLCNI96在氨基酸序列上不连续,却在空间结构上邻近,使其具有成为构象表位的潜力。

A-预测所得Gal d 4 B细胞线性表位;B-预测所得Gal d 4 T细胞表位;A1-预测所得B细胞线性表位在Gal d 4 分子飘带模型中的定位;A2~A3预测所得B细胞线性表位在Gal d 4表面分子模型暴露情况;B1-预测所得T细胞表位在Gal d 4分子飘带模型中的定位;B2~B3预测所得T细胞表位在Gal d 4表面分子模型暴露情况
图5 Gal d 4的B细胞线性表位空间定位
Fig.5 Spatial localization of linear epitopes of B cells of Gal d 4
图5-B为预测所得T细胞表位在Gal d 4空间结构的定位。42SLGNWVCAA50和100ALLSSDITA108均位于α-螺旋与无规则卷曲的连接处,能较好的与MHC分子结合形成抗原肽从而被T细胞识别诱发免疫反应。
2.12 鸡蛋过敏原Gal d 4的B细胞线性表位的空间定位
预测所得的B细胞线性表位IgE结合活性验证结果如图6所示。
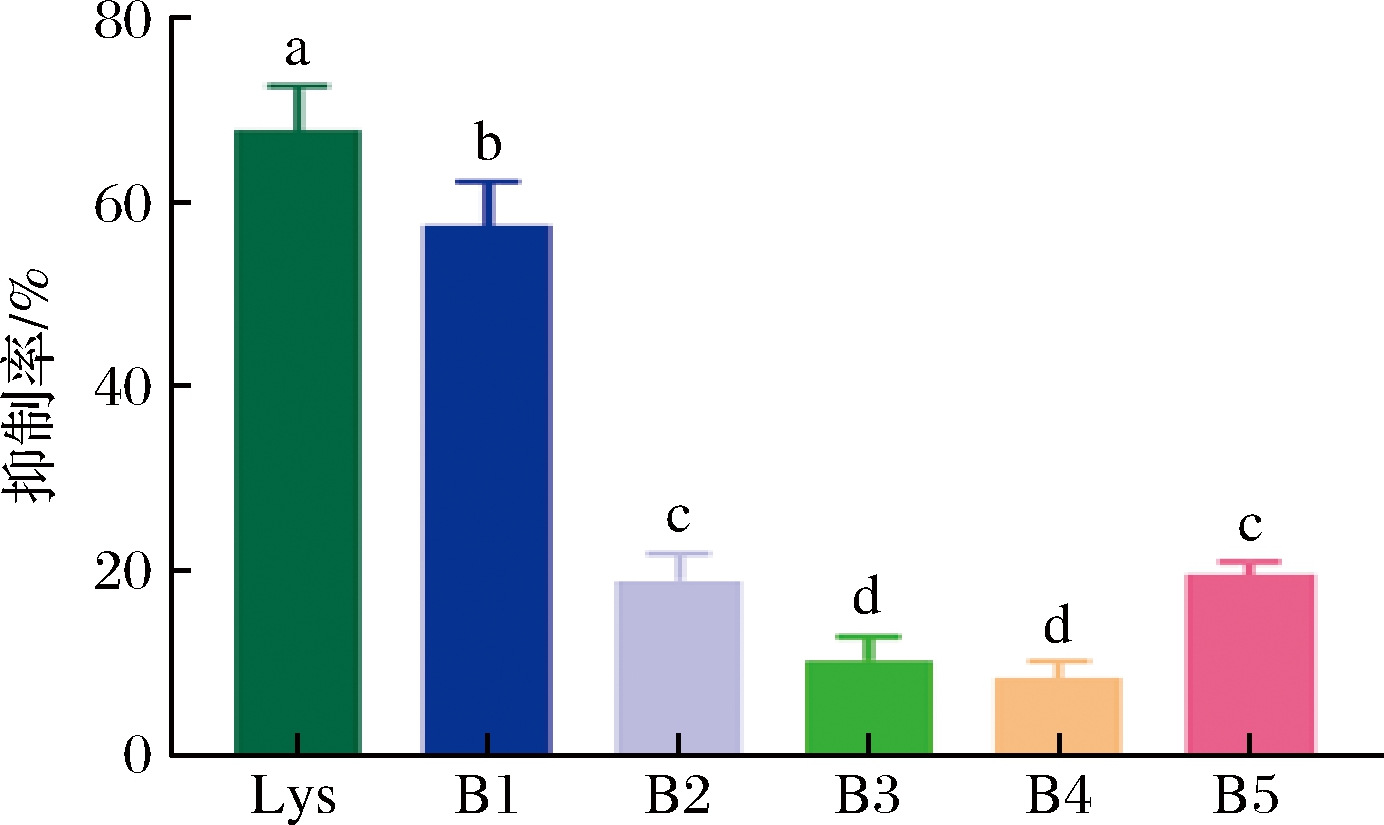
图6 Gal d 4的B细胞线性表位鉴定
Fig.6 Identifcation of linear epitopes of B cells of Gal d 4
注:Lys为鸡蛋溶菌酶纯品,不同字母标记表示为具有统计学上的差异性。
B1对鸡蛋过敏病人血清与致敏蛋白的特异性结合具有显著的抑制作用,这表明其免疫活性较强,具有一定的致敏性。B2~B5肽段则未显示出明显的抑制作用,表明其致敏潜力较小。因此,我们推断B1:19KVFGRCELAAAMKR32为Gal d 4 的B 细胞线性表位。
3 结论与讨论
本研究通过生物信息学方法对鸡蛋过敏原Gal d 4的线性表位进行预测分析,最终筛选出5个B细胞线性表位,分别为:19KVFGRCELAA AMKR32、57NTQA60、65TDG67、87TPGSRNLCNI96和132RCKGTDVQAWI142;2个T细胞抗原表位,分别为:42SLGNWVCAA50和100ALLSSDITA108。B细胞线性表位大部分位于亲水性、表面可及性以及可塑性较强的区域。蛋白质的疏水性氨基酸被包被于蛋白内部,亲水性氨基酸一般位于蛋白表面,因而,亲水性较强的区域易于抗体结合产生相互作用;表面可及性主要指溶剂分子接触抗原氨基酸的可能性,间接反映了与抗体的结合能力;可塑性强的区域的氨基酸易发生折叠和扭曲,有利于抗原与抗体的嵌合[28]。LI等[29]预测所得水牛乳β-乳球蛋白表位也位于亲水性、表面可及性以及可塑性较强的区域。结合鸡蛋过敏原Gal d 4的二级结构以及三维空间结构可知,Gal d 4的空间结构是由3个α-螺旋、1个β-折叠以及部分β-转角和无规则卷曲组成的。由二硫键形成α-螺旋在维持蛋白质空间结构稳定性方面起到非常重要的作用[30];无规则卷曲多暴露在蛋白分子表面,有利于其与抗体嵌合;同时α-螺旋和β-转角具有较高的氢键能,可以保持蛋白质的结构,并与抗体产生强烈的相互作用[31],这些使Gal d 4蛋白形成致密的球状分子,为其致敏性提供了稳定的结构基础。在本研究中预测所得的5个B细胞线性表位位于无规则卷曲与α-螺旋、β-转角的连接处或无规则卷曲处,马秀丽等[32]通过生物信息学方法预测的芝麻过敏原蛋白Ses i 3的B细胞线性表位大多分布在β-转角和无规则卷曲区域。而李雪娇等[33]通过生物信息学方法预测小麦过敏原B细胞线性表位则位于无规卷曲处和α-螺旋与无规卷曲的连接处,这与本研究预测结果相似。将5个B细胞线性表位定位在分子表面发现,5个表位大部分暴露在分子表面,具有较好的表面可及性以及较大的接触面积,利于在溶液的环境中与抗体互相作用。值得注意的是,在本研究中发现,非连续的B细胞模拟线性表位片段19KVFGRCELAAAMKR32、57NTQA60和132RCKGTDVQAWI142、65TDG67和87TPGSRNLCNI96在空间结构上邻近,这使其具有了形成构象表位的潜力。这表明,连续的氨基酸序列在成为线性表位的同时,具有一定的潜力与空间上邻近的其他具有较大可能成为抗原表位的氨基酸序列形成构象表位。GUO等[34]在对核桃过敏原Jug r 1的研究中也证实了表位既可以是氨基酸的线性序列(线性表位),又可以是三维结构的基序(构象表位),这与本研究结果一致。
T细胞表位是能被MHC分子递呈和T细胞识别的抗原表位多肽,在细胞免疫中起着重要作用。目前T细胞表位生物信息学技术比较成熟,尤其是与MHC-I类分子结合的CD8+ T细胞表位,预测的精确率已达80%以上,虽然与MHC-II类分子结合的CD4+ T细胞表位预测稍逊于前者,但随着生物信息学的发展也日趋成熟[35]。PASCAL等[36]使用NetMHCⅡpan-2.0算法预测Ara h 2肽段与HLA-II类分子结合力强的区域,最终鉴定了Ara h 2中的4个T细胞抗原表位。本研究预测所得T细胞表位经空间定位后发现,42SLGNWVCAA50大部分被包藏于蛋白分子内部,可能于消化后暴露增强其致敏性。
但从理论和实际效果看,针对线性表位相关预测方法虽然取得了一定的进展,但同时也存在着一定的缺陷,缺乏统一的标准进行评估和比较[37]。也有研究表明,仅仅依靠氨基酸序列展现的性质所预测得到的模拟表位的方法并不牢靠,需要与非表位特性的特征结合起来以提高预测模拟表位的准确性[38]。对绝大多数食物过敏而言,过敏原初次接触机体,通过一系列反应,刺激B细胞产生IgE抗体,过敏原再次接触,其B细胞表位与IgE抗体结合,诱发过敏反应,因此B细胞的线性表位十分重要。本研究通过固相肽合成方法[39]结合间接竞争ELISA对预测所得模拟表位进行鉴定,以此提高表征B细胞线性表位的精确性。结果表明,只有B1肽段具有较好的IgE结合活性,说明其成为B细胞线性表位的潜力巨大。剩余4个预测所得模拟表位展现较弱的抑制作用,可能是因为其氨基酸序列中包含了鸡蛋溶菌酶的部分B细胞线性表位但并不完全,也有可能是因为鸡蛋过敏患者的差异性,导致其特异性结合效果并不理想。
综上,本研究首次对鸡蛋过敏原Gal d 4的B细胞线性表位和T细胞表位同时进行表征,获得了5个模拟B细胞线性表位:19KVFGRCELAAAMKR32、57NTQA60、65TDG67、87TPGSRNLCNI96和132RCKGTD-VQAWI142以及2个模拟T细胞抗原表位:42SLGNWVCAA50和100ALLSSDITA108,对其在Gal d 4的三维结构模型上进行空间定位,最后通过固相肽合成结合间接竞争ELISA对预测所得模拟表位进行鉴定,得到一个优势B细胞线性表位:19KVFGRCELAAAMKR32。这为进一步明确溶菌酶Gal d 4的表位提供了依据,同时也为鸡蛋过敏原的识别与检测提供了参考。
[1] MURARO A, LEMANSKE R F Jr, CASTELLS M, et al.Precision medicine in allergic disease-food allergy, drug allergy, and anaphylaxis-PRACTALL document of the European Academy of Allergy and Clinical Immunology and the American Academy of Allergy, Asthma and Immunology[J].Allergy, 2017, 72(7):1006-1021.
[2] DONA D W, SUPHIOGLU C.Egg allergy:Diagnosis and immunotherapy[J].International Journal of Molecular Sciences, 2020, 21(14):5010.
[3] LOH W, TANG M L K.The epidemiology of food allergy in the global context[J].International Journal of Environmental Research and Public Health, 2018, 15(9):2043.
[4] 陈寅. 牛奶、鸡蛋过敏原的鉴定、纯化及应用研究[D].天津:天津医科大学, 2011.
CHEN Y.Identification, purification and application research of allergens from milk and hen’s egg[D].Tianjin:Tianjin Medical University, 2011.
[5] MARTORELL A, ALONSO E, BONÉ J, et al.Position document:IgE-mediated allergy to egg protein[J].Allergologia et Immunopathologia, 2013, 41(5):320-336.
[6] MARTOS G, L PEZ-FANDI
PEZ-FANDI O R, MOLINA E.Immunoreactivity of hen egg allergens:Influence on in vitro gastrointestinal digestion of the presence of other egg white proteins and of egg yolk[J].Food Chemistry, 2013, 136(2):775-781.
O R, MOLINA E.Immunoreactivity of hen egg allergens:Influence on in vitro gastrointestinal digestion of the presence of other egg white proteins and of egg yolk[J].Food Chemistry, 2013, 136(2):775-781.
[7] INFANTE S, L PEZ-MATAS M
PEZ-MATAS M  , CARNÉS J, et al.Allergy reaction mediated by Gal d 4 (lysozyme) after the induction of tolerance with egg[J].Annals of Allergy, Asthma &Immunology, 2014, 113(4):491-492.
, CARNÉS J, et al.Allergy reaction mediated by Gal d 4 (lysozyme) after the induction of tolerance with egg[J].Annals of Allergy, Asthma &Immunology, 2014, 113(4):491-492.
[8] ELBANY C, DE BOISSIEU D, KARILA C, et al.Unusual Lysozyme-induced anaphylaxis in an egg-allergic child[J].Authorea Preprints, 2021.DOI:10.22541/au.163664664.49008784/v1
[9] LIU C Q, SATHE S K.Food allergen epitope mapping[J].Journal of Agricultural and Food Chemistry, 2018, 66(28):7238-7248.
[10] PALOMARES O, AKDIS M, MART N-FONTECHA M, et al.Mechanisms of immune regulation in allergic diseases:The role of regulatory T and B cells[J].Immunological Reviews, 2017, 278(1):219-236.
N-FONTECHA M, et al.Mechanisms of immune regulation in allergic diseases:The role of regulatory T and B cells[J].Immunological Reviews, 2017, 278(1):219-236.
[11] 李欣, 马鑫, 谭宏凯, 等.T细胞及其表位在食物过敏中的研究进展[J].食品与生物技术学报, 2020, 39(10):18-25.
LI X, MA X, TAN H K, et al.Research progress in T cells and their epitopes in food allergy[J].Journal of Food Science and Biotechnology, 2020, 39(10):18-25.
[12] 刘阳星月, 张迪, 姚亚亚, 等.大豆过敏原11S球蛋白G2中A2链结合表位的预测[J].食品科学, 2018, 39(18):152-158.
LIU Y, ZHANG D, YAO Y Y, et al.Mapping of ig G-binding epitopes on the A2 chain of the major soybean allergen 11S globulin G2[J].Food Science, 2018, 39(18):152-158.
[13] EL-MANZALAWY Y, HONAVAR V.Recent advances in B-cell epitope prediction methods[J].Immunome Research, 2010, 6(2):S2.
[14] SUN P P, JU H X, LIU Z B, et al.Bioinformatics resources and tools for conformational B-cell epitope prediction[J].Computational and Mathematical Methods in Medicine, 2013, 2013:943636.
[15] RUBINSTEIN N D, MAYROSE I, HALPERIN D, et al.Computational characterization of B-cell epitopes[J].Molecular Immunology, 2008, 45(12):3477-3489.
[16] RAHIMPOUR E, SAMAD-SOLTANI T.Immunoinformatics:A basic bibliometric analysis[J].ImmunoAnalysis, 2021, 1(1):2.
[17] HOPP T P, WOODS K R.Prediction of protein antigenic determinants from amino acid sequences[J].Proceedings of the National Academy of Sciences of the United States of America, 1981, 78(6):3824-3828.
[18] EMINI E A, HUGHES J V, PERLOW D S, et al.Induction of hepatitis A virus-neutralizing antibody by a virus-specific synthetic peptide[J].Journal of Virology, 1985, 55(3):836-839.
[19] KARPLUS P A, SCHULZ G E.Prediction of chain flexibility in proteins[J].Naturwissenschaften, 1985, 72(4):212-213.
[20] JAMESON B A, WOLF H.The antigenic index:A novel algorithm for predicting antigenic determinants[J].Bioinformatics, 1988, 4(1):181-186.
[21] FU L L, WANG J B, NI S Q, et al.Identification of allergenic epitopes and critical amino acids of major allergens in Chinese shrimp (Penaeus chinensis) by immunoinformatics coupled with competitive-binding strategy[J].Journal of Agricultural and Food Chemistry, 2018, 66(11):2944-2953.
[22] POSCH A, WEISS W, WHEELER C, et al.Sequence analysis of wheat grain allergens separated by two-dimensional electrophoresis with immobilized pH gradients[J].Electrophoresis, 1995, 16(1):1115-1119.
[23] LI J Y, BAI X J, LIANG Y, et al.Prediction of epitopes of Rv1410c Mycobacterium tuberculosis protein using DNAStar software[J].Chinese Journal of Cellular and Molecular Immunology, 2015, 31(4):474-477.
[24] KOLASKAR A S, TONGAONKAR P C.A semi-empirical method for prediction of antigenic determinants on protein antigens[J].FEBS Letters, 1990, 276(1-2):172-174.
[25] JESPERSEN M C, PETERS B, NIELSEN M, et al.BepiPred-2.0:Improving sequence-based B-cell epitope prediction using conformational epitopes[J].Nucleic Acids Research, 2017, 45(W1):W24-W29.
[26] ISEA R.Quantitative prediction of linear B-cell epitopes[EB/OL].2017:arXiv:1703.02453.https://arxiv.org/abs/1703.02453
[27] GORGAN L, PETRESCU-MAG V.The small heat shock proteins structure in Carassius gibelio and Oncorhynchus mykiss[J].Journal of Experimental and Molecular Biology, 2009, 10(4):19-26.
[28] 龙伟, 李欣, 高金燕, 等.生物信息学技术在食物过敏原表位预测中的应用[J].食品科学, 2014, 35(3):259-263.
LONG W, LI X, GAO J Y, et al.Application of bioinformatics technology in prediction of food allergen epitopes[J].Food Science, 2014, 35(3):259-263.
[29] LI X, CHEN H B, TONG P, et al.Epitope mapping of buffalo beta-lactoglobulin against rabbit polyclonal antibody following phage display technique[J].Journal of Food Biochemistry, 2012, 36(1):56-65.
[30] KARIMI M, IGNASIAK M T, CHAN B, et al.Reactivity of disulfide bonds is markedly affected by structure and environment:Implications for protein modification and stability[J].Scientific Reports, 2016, 6:38572.
[31] SHADDEL M, EBRAHIMI M, TABANDEH M R.Bioinformatics analysis of single and multi-hybrid epitopes of GRA-1, GRA-4, GRA-6 and GRA-7 proteins to improve DNA vaccine design against Toxoplasma gondii[J].Journal of Parasitic Diseases, 2018, 42(2):269-276.
[32] 马秀丽, 张九凯, 孙劲旅, 等.芝麻过敏原Ses i 3同源建模及B细胞线性抗原表位预测[J].中国食品学报, 2021, 21(4):9-18.
MA X L, ZHANG J K, SUN J L, et al.Homology modeling and prediction on B cell liner antigenic epitopes of Ses i 3 from Sesamum indicum[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(4):9-18.
[33] 李雪娇, 李欣芮, 范卓妍, 等.小麦主要过敏原CM16线性B细胞表位的预测及初步鉴定[J].食品科学, 2019, 40(24):136-141.
LI X J, LI X R, FAN Z Y, et al.Predicting and identifying linear B-cell epitopes of wheat allergen CM16[J].Food Science, 2019, 40(24):136-141.
[34] GUO X Y, JIANG S S, LI X R, et al.Sequence analysis of digestion-resistant peptides may be an efficient strategy for studying the linear epitopes of Jug r 1, the major walnut allergen[J].Food Chemistry, 2020, 322:126711.
[35] CREECH A L, TING Y S, GOULDING S P, et al.The role of mass spectrometry and proteogenomics in the advancement of HLA epitope prediction[J].PROTEOMICS, 2018, 18(12):1700259.
[36] PASCAL M, KONSTANTINOU G N, MASILAMANI M, et al.In silico prediction of Ara h 2 T cell epitopes in peanut-allergic children[J].Clinical &Experimental Allergy, 2013, 43(1):116-127.
[37] 张春华. 基于信息融合和计算智能的构象性B细胞表位预测方法研究[D].长春:东北师范大学, 2016.
ZHANG C H.The research of conformational B-cell epitope prediction method based on information fusion and computational intelligence[D].Changchun:Northeast Normal University, 2016.
[38] GOEDHALS D, PAWESKA J T, BURT F J.Identification of human linear B-cell epitope sites on the envelope glycoproteins of Crimean-Congo haemorrhagic fever virus[J].Epidemiology and Infection, 2015, 143(7):1451-1456.
[39] GEYSEN H M, RODDA S J, MASON T J, et al.Strategies for epitope analysis using peptide synthesis[J].Journal of Immunological Methods, 1987, 102(2):259-274.