果胶酶是指能分解果胶物质的复合酶类,一般包括原果胶酶、多聚半乳糖醛酸酶、果胶酯酶及果胶裂解酶等[1-3]。果胶裂解酶(EC 1.10.3.2)能够以β-反式消去作用断裂植物细胞中果胶的α-1,4糖苷键,形成不饱和寡聚半乳糖醛酸[2]。其中,酸性果胶裂解酶是指能在低pH条件下发挥作用的裂解酶,该酶在果汁、酿酒等工业中具有广泛应用[4]。酸性果胶裂解酶主要来自于黑曲霉(Aspergillus niger)[5]、大豆曲霉[6]、黄曲霉[7]、假丝酵母[8]和酿酒酵母[9]等。然而,天然果胶裂解酶的耐酸性还有待提高[10-14]。构建高效、易操作的表达平台是酸性果胶裂解酶分子改造的重要前提。
目前,酵母为酸性果胶裂解酶的主要表达宿主[15]。然而,酵母等真菌的基因操作周期长、菌体生长缓慢、效率低,不是酶分子改造的理想平台[16-17]。与酵母等真菌表达体系相比,大肠杆菌Escherichia coli具有发酵周期短、操作简单的优良特性,是较为理想的重组蛋白生产改造的宿主细胞。此外,一些真菌(Penicillium occitanis[18]和A.niger[19-20]等)的酸性果胶裂解酶虽已在E.coli中表达,但无活性的包涵体是其主要存在形式。研究通过体外复性,可使40%的重组P.occitanis酸性果胶裂解酶包涵体重新折叠为可溶性活性蛋白[21]。然而,体外复性显著增加了操作步骤,降低了酶蛋白制备效率[22]。
在E.coli中,蛋白标签的融合可以提高蛋白质的溶解性[23]。常用来提高溶解性的蛋白标签有碳水化合物结合结构域(carbohydrate-binding structural domain, CBM)、麦芽糖结合蛋白(maltose binding protein, MBP)、大肠杆菌硫氧还原蛋白(thioredoxins, TrxA)、谷胱甘肽巯基转移酶(glutathione S-transferase, GST)[24]。例如,有研究通过融合TrxA蛋白标签策略优化了HIV-1蛋白酶(一种逆转录病毒天冬酰胺蛋白酶)在E.coli中的异源表达,表达量从0.83 mg/L提升到2.5 mg/L,有效提高了蛋白的溶解性[25-26]。
在本研究中,通过融合蛋白标签CBM、MBP、TrxA、GST优化,提高来源于A.niger AG11的酸性果胶裂解酶pelA(GenBank No.ANI_1_624124)在E.coli BL21(DE3)中的表达水平,构建pelA在E.coli中的分子改造平台,并对其酶学性质进行分析。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
A.niger AG11、E.coli JM109、BL21(DE3)感受态细胞、质粒pET-28a(+)、蛋白标签CBM、MBP、TrxA、GST均由实验室保藏。
1.1.2 主要试剂
柱回收试剂盒、蛋白质marker,Thermo公司;大肠杆菌感受态制备试剂盒、限制性内切酶、Primer STAR Max DNA聚合酶,大连TaKaRa公司;SanPrep柱式PCR产物纯化试剂盒、质粒提取试剂盒、真菌基因组DNA快速提取试剂盒、一步克隆试剂盒、卡纳青霉素、重组HRV 3C蛋白酶、异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG),上海生工公司;蛋白胨和酵母粉,Oxoid公司;底物柑橘果胶,美国Sigma公司;Bradford蛋白浓度检测试剂盒,碧云天公司;蛋白电泳loading buffer和Bis-Tris预制凝胶,Invitrogen公司。其他常规试剂及药品为国产或进口分装。
1.1.3 培养基
LB(Luria-Bertani)液体培养基(g/L):蛋白胨10,酵母粉5,NaCl 10,自然pH。
TB(terrific broth)培养基(g/L):蛋白胨12,酵母粉24,KH2PO4·3H2O 16.4,KH2PO4 2.31,甘油5,自然pH。
马铃薯葡萄糖琼脂培养基(potato dextrose agar,PDA)培养基(g/L):葡萄糖20,土豆200,琼脂15,自然pH。
上述培养基均121 ℃灭菌15 min。
1.2 实验方法
1.2.1 A.niger基因组的提取
将A.niger孢子悬液涂布于PDA培养基,在30 ℃下静置培养4~7 d。加入无菌水刮取固体培养基表面上的孢子,经滤膜过滤后接种于PDB培养基中,30 ℃、220 r/min培养24 h。通过离心及纱布过滤收集菌丝体,置于灭菌的研钵中(加液氮)研磨至细小粉末,用离心管收集待用。A.niger基因组使用真菌基因组DNA快速提取试剂盒提取。
1.2.2 PelA基因的克隆与表达载体的构建
以A.niger AG11基因组为模板,通过NCBI中的BLAST对序列进行分析,编码pelA的基因具有1 357 bp(包括4个内含子)。使用SignalP 5.0进行信号肽预测,pelA基因5′端有信号肽,可能会影响其在E.coli中的表达,故使用表1中的引物AF和AR进行PCR扩增,得到切除信号肽,但带有内含子的pelA基因。扩增程序为:98 ℃预变性3 min;98 ℃变性10 s;55 ℃退火15 s;72 ℃延伸50 s;循环32次;最后72 ℃延伸5 min。PCR扩增产物使用琼脂糖凝胶电泳检测,对产物进行纯化并测序。使用NcoⅠ和XhoⅠ对pET-28a(+)载体和PCR产物进行双酶切。使用Solution Ⅰ分别将PCR产物连接至pET-28a(+)载体的双酶切片段,并转化至E.coli JM109。按照表1所示引物,使用Aex2F、Aex2R、Aex3F、Aex3R、Aex4F、Aex4R、Aex5F、Aex5R对重组载体进行PCR以切除内含子。用磷酸化酶处理所得PCR产物,并使用Solution Ⅰ连接酶连接,得到pelA表达载体pET-28a(+)-pelA。将其转化至E.coli JM109,挑取4~6个转化子送公司进行测序验证。将验证正确的菌株重新活化培养12 h后,参考质粒提取说明书提取质粒,得到重组质粒pET-28a(+)-pelA。
表1 本研究使用的引物
Table 1 Primers used in this study

引物名称引物序列AF5′-AGAAGGAGAATGGTCGGCGTGTCCGGCTCTG-3′AR5′-TAGTGGTGGTGGTGGTGGTGTTACAGGTTGCCCTGACCGG-3′Aex2F5′-ACAACACTCTCCTCCACTGTGTCAACAACTACTTCTACGACATCTCC-3′Aex2R5′-ACAGTGGAGGAGAGTGTTGTCC-3′Aex3F5′-TCTGGATCGACCATGTTACTACCGCCCGCATCGGTCGCC-3′Aex3R5′-AGTAACATGGTCGATCCAGACCAGGTCGCAGTCATCAAG-3′Aex4F5′-GCTGAGAACATCATCATCCAGAACATCGCGGTTACCGACATC-3′Aex4R5′-TGGATGATGATGTTCTCAGCACCG-3′Aex5F5′-CCCTCTATCAGCGTTGAATACTACAACGCTGGTGTCCTCGG-3′Aex5R5′-TATTCAACGCTGATAGAGGGAGCAT-3′CBMF5′-AAGAAGGAGATATACCATGGGCAGCATGGTCGCTACTGCCAAGTACG-3′CBMR5′-TGGACCCTGAAACAGAACTTCGAGCTTGATGAGCCTGAGGTTACCGAACTTTGAA-3′MBPF5′-GGAGATATACCATGGGCAGCATGAAAATCGAAGAAGGTAAACTGGTAATCTGG-3′MBPR5′-TGGACCCTGAAACAGAACTTCGAGAGTCTGCGCGTCTTTCAGGGCTT-3′
续表1
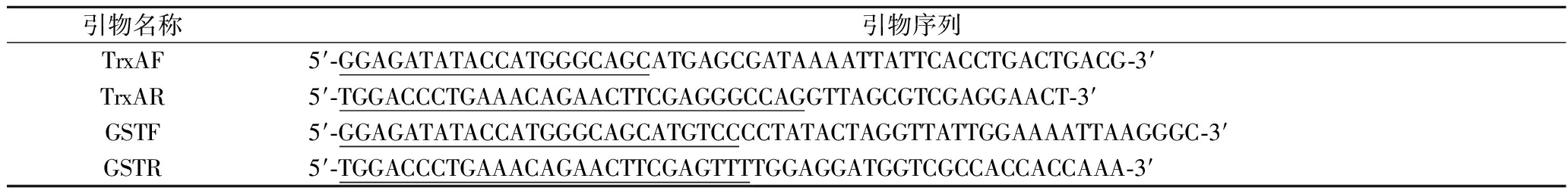
引物名称引物序列TrxAF5′-GGAGATATACCATGGGCAGCATGAGCGATAAAATTATTCACCTGACTGACG-3′TrxAR5′-TGGACCCTGAAACAGAACTTCGAGGGCCAGGTTAGCGTCGAGGAACT-3′GSTF5′-GGAGATATACCATGGGCAGCATGTCCCCTATACTAGGTTATTGGAAAATTAAGGGC-3′GSTR5′-TGGACCCTGAAACAGAACTTCGAGTTTTGGAGGATGGTCGCCACCACCAAA-3′
注:下划线表示一步克隆对应的同源臂序列。
1.2.3 重组pelA的表达
将构建的pET-28a(+)-pelA表达载体转化至E.coli BL21(DE3),得到重组菌pET-28a(+)-pelA/BL21(DE3)。将重组菌接种于LB液体培养基(含50 g/L卡那霉素)中活化,37 ℃,220 r/min培养10 h。按照3%(体积分数)接种量转接至TB发酵培养基(含50 g/L卡那霉素)中,在37 ℃,220 r/min摇床中培养至OD600约为1.0时,加入IPTG(终浓度为2 mmol/L)诱导表达,20 ℃、220 r/min培养24 h。
将重组菌发酵液离心并收集菌体,加入与发酵液等体积Tris-HCl缓冲液(50 mmol/L,pH 7.0)重悬,置于冰水浴中超声破碎。将超声破碎仪的变幅杆末端插入样品,在距离液面10~15 mm的中心位置固定,以间隙2 s、工作2 s为周期超声处理4 min。整个超声过程中样品一直处于冰浴状态。4 ℃、10 000 r/min离心20 min,分别测定菌裂解上清液和菌裂解液沉淀的酶活力,并对其进行SDS-PAGE检测。
1.2.4 蛋白标签优化提高胞内蛋白表达水平
为筛选蛋白标签CBM、MBP、TrxA、GST,采用一步克隆法分别将蛋白标签的基因序列融合到目的基因的5′端,使用表1所示引物进行PCR。基因操作参考1.2.2节的方法。挑取4~6个转化子送公司进行验证。将验证正确的菌株重新活化培养12 h后,参考质粒提取说明书提取质粒,获得重组质粒pET-28a(+)-pelA+CBM、pET-28a(+)-pelA+MBP、pET-28a(+)-pelA+TrxA、pET-28a(+)-pelA+GST。转化进感受态细胞E.coli BL21(DE3)中,构成重组菌株pET-28a(+)-pelA+CBM/BL21(DE3)、pET-28a(+)-pelA+MBP/BL21(DE3)、pET-28a(+)-pelA+TrxA/BL21(DE3)、pET-28a(+)-pelA+GST/BL21(DE3)。培养方式参考1.2.3节。取菌裂解上清液测酶活力,比较不同蛋白标签对pelA胞内表达的影响。
1.2.5 重组酶的分离纯化
使用镍亲和层析柱(HisTrapTM FF)对重组酶pelA+CBM进行分离纯化。缓冲液A:50 mmol/L Tris-HCl,pH 7.5;缓冲液B:50 mmol/L Tris-HCl,pH 7.5,500 mmol/L咪唑。取50 mL菌裂解上清液于0.22 μm 水系膜过滤后,上样至用缓冲液A平衡的HisTrapTM FF。用5%的缓冲液B冲洗纯化柱后,40%缓冲液B(含200 mmol/L咪唑)洗脱获得重组酶pelA+CBM,用脱盐柱Sephadex G25脱盐,使用SDS-PAGE检测其纯度。
1.2.6 重组酶的酶学性质测定
对脱盐后的重组pelA酶进行酶学性质分析。取出部分重组pelA+CBM酶,使用重组HRV 3C蛋白酶在4 ℃下处理16 h,以切除蛋白标签CBM。再经过镍亲和层析柱对pelA进行分离纯化,获得纯pelA。比较蛋白标签切除前后的酶学性质的差异。
最适反应温度:在pH 5.2条件下,测定不同反应温度(30~80 ℃)下的酶活力,以最大的酶活力为100%。
温度稳定性:将酶液置于不同温度(30~80 ℃)处理2 h后,在标准条件下测定残余酶活力,以未处理组的酶活力为100%。
最适反应pH:在最适反应温度下,测定不同pH值(3.0~7.0)Na2HPO4-柠檬酸盐缓冲液条件下的酶活力,使用相应pH值的缓冲液配制底物,以最大的酶活力为100%。
pH稳定性测定:将酶液置于不同pH值的(3.0~7.0)Na2HPO4-柠檬酸盐缓冲液中,40 ℃条件下处理2 h,测定残余酶活力,以未处理组的酶活力为100%。
1.2.7 重组酶的酶反应动力学参数测定
在酶的最适条件下测定质量浓度为0~5 g/L的柑橘果胶的初始酶活力。根据上述酶反应的数据,绘制Lineweaver-Burk双倒数图,计算酸性果胶裂解酶的Km和kcat。
1.2.8 重组酶活力和蛋白含量的测定
采用HCl法测定酸性果胶裂解酶活力。将5 g/L柑橘果胶溶解于0.08 mol/L琥珀酸-琥珀酸钠(pH 5.2)缓冲液中,用作底物。取2.9 mL底物,在30 ℃下预热3 min后与100 μL适当稀释的粗酶液混合,30 ℃反应30 min,加入1 mL的2 mol/L HCl终止液终止反应,在235 nm下测定其吸光度。1个标准酶活力单位定义为:在测定条件下,每分钟生成1 μmol双键所需的酶量定义为一个酶活力单位(U)。采用Bradford法测定酶蛋白浓度,蛋白标准品为牛血清蛋白。
1.2.9 统计学分析
本研究采用Origin 8.5软件进行单因素方差分析(One-way ANOVA)和Tukey’s检验(P<0.05和P<0.01),实验结果以平均值±方差表示。
2 结果与分析
2.1 产酸性果胶裂解酶重组菌株的构建及表达
以A.niger AG11基因组为模板,获得切除信号肽和内含子的pelA基因序列。电泳分析显示,PCR产物的大小约为1 077 bp,符合理论值(图1-a)。将PCR产物克隆至pET-28a(+)的Nco I-Xho I位点,得到编码pelA的质粒pET-28a(+)-pelA’。基于一步克隆法切除pET-28a(+)-pelA’中pelA的内含子序列,进一步构建得到pelA表达质粒pET-28a(+)-pelA(图1-b)。上述表达质粒序列均经测序验证。

a-pelA PCR产物的琼脂糖凝胶电泳分析;b-pET-28a/pelA载体图谱
图1 pelA基因的克隆与载体构建
Fig.1 Cloning and vector construction of pelA
SDS-PAGE分析显示,pET-28a(+)-pelA/BL21(DE3)菌裂解上清液与沉淀部分均有pelA条带(理论量38.8 kDa),但空白对照菌裂解液沉淀部分不存在该条带,菌裂解上清液隐约出现疑似pelA条带,应为背景蛋白。初步判断pelA在E.coli BL21(DE3)中成功表达,且大部分以包涵体形式存在(图2-a)。对pET-28a(+)-pelA/BL21(DE3)和空白对照菌裂解上清液与沉淀分别测定酶活力,在空白对照的菌裂解上清液与沉淀中都未检测到pelA活性,而在pET-28a(+)-pelA/BL21(DE)组的菌裂解上清液中检测到0.79 U/mL的酶活力,其菌裂解液沉淀也不具有活性(图2-b)。上述结果表明,pelA在E.coli成功表达,但主要以包涵体形式存在。大量研究表明,E.coli在表达外源蛋白时会抑制其在体内的过量表达,导致表达量降低、产生包涵体等,而将蛋白标签与目的蛋白进行融合表达,可以明显提高蛋白质产物的溶解性[21, 25-27]。因此,本研究采用融合蛋白标签的方式来提高pelA的溶解性,便于对其性质进行研究及后期改造。
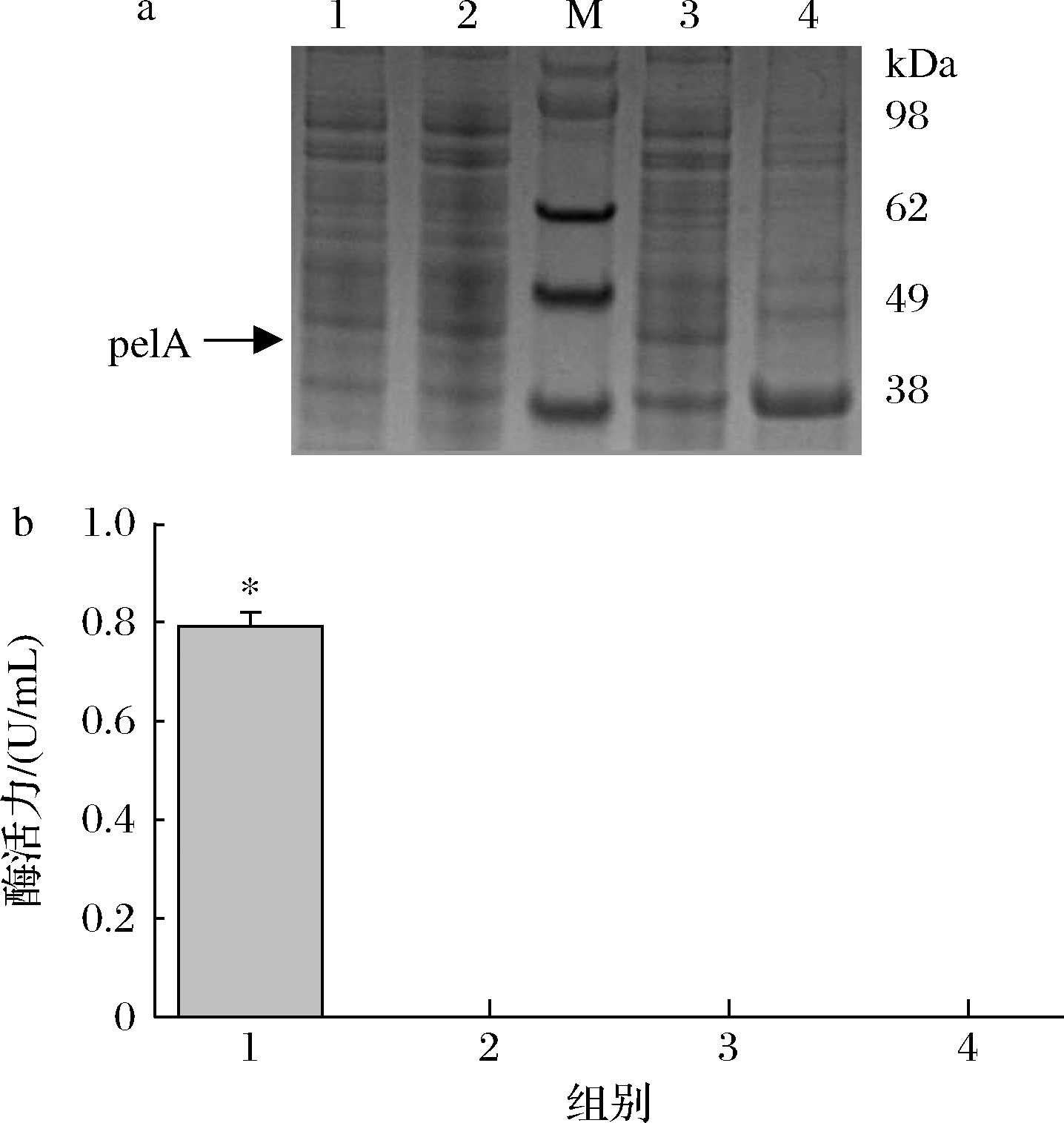
M:蛋白标准分子质量;1:pET-28a/pelA菌裂解液上清液;2:pET-28a/pelA菌裂解液沉淀;3:pET-28a(+)菌裂解液上清空白对照;4:pET-28a(+)菌裂解液沉淀空白对照
a-重组菌表达pelA的SDS-PAGE分析;b-重组菌菌裂解液上清液和菌裂解液沉淀酶活柱状图
图2 重组酶pelA的表达分析
Fig.2 Analysis of the expression of recombinant pelA
注:与pET-28a(+)菌裂解液上清液相比,*P<0.05表示差异显著。
2.2 融合标签优化提高pelA表达水平
将蛋白标签MBP、TrxA、CBM、GST的基因序列分别融合到pelA基因的5′端,构建pET-28a(+)-pelA+MBP、pET-28a(+)-pelA+TrxA、pET-28a(+)-pelA+CBM和pET-28a(+)-pelA+ GST 表达载体(图3-a)。将它们分别转化至BL21(DE3)中,并在20 ℃,220 r/min下发酵24 h。分别测定菌裂解上清液中pelA的酶活力,结果显示,蛋白标签的融合均有提高pelA活性的作用。与重组菌pET-28a(+)-pelA/BL21(DE3)产pelA的胞内活性相比,重组菌pET-28a(+)-pelA+MBP/BL21(DE3)、pET-28a(+)-pelA+TrxA /BL21(DE3)、pET-28a(+)-pelA+GST/BL21(DE3)、pET-28a(+)-pelA+CBM/BL21(DE3)的胞内活性分别提高了2.47、2.79、2.85、4.12倍。其中,重组菌pET-28a(+)-pelA+CBM/BL21(DE3)菌裂解上清液中酶活力最高,达到3.25 U/mL(图3-b)。通过SDS-PAGE结果发现,与其他3种融合蛋白标签(MBP、TrxA、GST)的蛋白条带相比,融合CBM的菌裂解液沉淀蛋白条带较细,而菌裂解上清液中的蛋白条带较粗(图3-c)。其中融合蛋白标签MBP的菌裂解上清液蛋白条带虽然很粗,但其活力不高,这可能是因为MBP标签较大,对蛋白结构功能有影响。以上结果说明,蛋白标签CBM能提高胞内表达量,有效减少包涵体的形成。而采用包涵体体外复性法,虽然使重组P.occitanis酸性果胶裂解酶的酶活力提高3.9倍[21],但蛋白制备过程繁琐,且提高效果不如融合蛋白标签CBM的方法。因此选取重组菌pET-28a(+)-pelA+CBM/BL21(DE3)对pelA酶进行表达。

M:蛋白标准分子质量;1:pelA+MBP菌裂解液上清液;2:pelA+MBP菌裂解液沉淀;3:pelA+TrxA菌裂解液上清液;4:pelA+TrxA菌裂解液沉淀;5:pelA+CBM菌裂解液上清液;6:pelA+CBM菌裂解液沉淀;7:pelA+GST菌裂解液上清液;8:pelA+GST菌裂解液沉淀(不同蛋白标签重组酶用箭头指出)
a-重组蛋白标签优化菌株表达载体构建;b-重组蛋白标签优化菌株菌裂解液上清酶活力;c-蛋白标签优化表达pelA的SDS-PAGE分析
图3 pelA融合蛋白标签的表达
Fig.3 Expression of pelA fusion protein tag
2.3 重组pelA的酶学性质
2.3.1 重组pelA的纯化和动力学参数测定
用0.22 μm的滤膜过滤菌裂解上清液,用40%的缓冲液B洗脱pelA+CBM,获得目的蛋白条带,蛋白条带与理论值相符。取出一部分洗脱蛋白,用重组HRV 3C蛋白酶在4 ℃下处理16 h,切除蛋白标签CBM,获得目的蛋白条带pelA,蛋白条带符合理论值(图4)。脱盐后测定蛋白浓度和酶活力。

M:蛋白标准分子质量;1~2:30%B液洗脱pelA+CBM;3~5:40%缓冲液B洗脱pelA+CBM;6:酶切标签后40%缓冲液B洗脱pelA
图4 镍柱亲和纯化SDS-PAGE分析
Fig.4 SDS-PAGE analysis of nickel column affinity purification
为了研究CBM对pelA催化性能的影响,测定了最适条件下pelA+CBM和pelA以柑橘果胶为底物时的动力学参数,其结果列于表2。结果显示,pelA的Km是pelA+CBM的1.66倍,表明pelA+CBM对柑橘果胶底物的亲和力比pelA的强。pelA的kcat/Km比pelA+CBM低48.81%,这表明切除CBM后pelA的催化效率降低。这些结果证明CBM可以提高pelA的催化效率,正如一些研究所证明的,CBM具有通过与底物特异性结合来增强底物与催化模块长期接触的邻近效应作用,从而提高酶的催化效率[28-29]。
表2 以柑橘果胶为底物时的重组pelA的动力学参数
Table 2 Kinetic parameters of recombinant pelA with citrus pectin as substrate

酸性果胶裂解酶Km/(g/L)kcat/(s-1)kcat/Km/[L/(g·s)]比酶活力/(U/mg)pelA+CBM2.6837.0713.8315.82pelA4.4431.447.0812.30
2.3.2 重组pelA的酶学性质测定
在30~40 ℃时,pelA+CBM 和pelA的相对酶活力均呈现上升趋势。当温度>40 ℃时,pelA的相对酶活力呈下降趋势,而pelA+CBM相对酶活力在温度>50 ℃时开始下降;反应温度>70 ℃时,相对酶活力<40%,此时pelA+CBM的相对酶活力骤降,当温度达到80 ℃时,相对酶活力低至40%以下(图5-a)。以上结果说明40 ℃为pelA的最适反应温度,而pelA+CBM的最适温度为50 ℃。
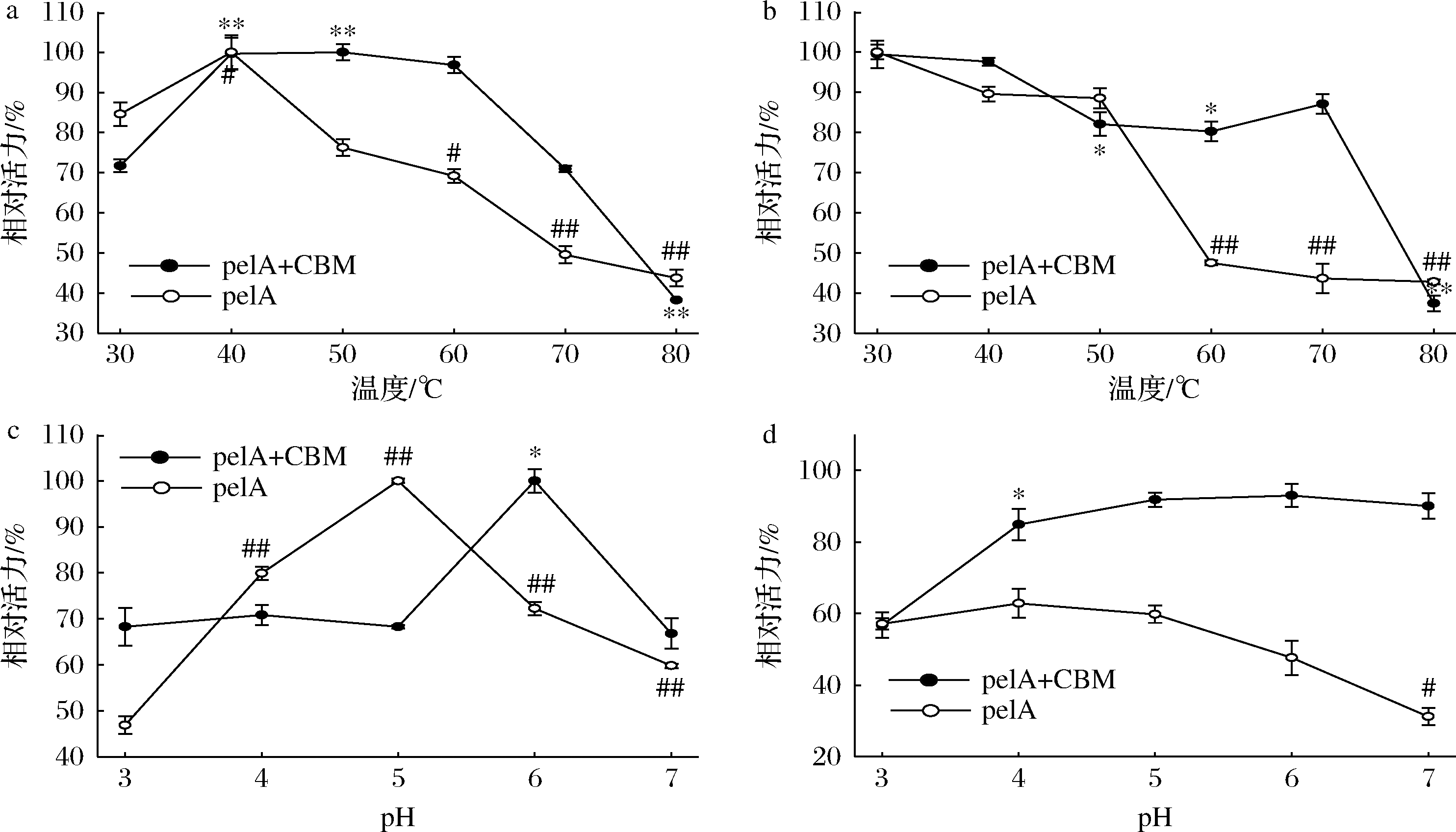
a-不同温度下酶的催化活性;b-不同温度处理2 h的剩余酶活力;c-不同pH下酶的催化活性;d-不同pH处理2 h的剩余酶活力
图5 温度和pH对蛋白标签切除前后重组pelA活性的影响
Fig.5 Effects of temperature and pH on pelA activity before and after resection of the melt-promoting tag
注:*和**分别表示pelA+CBM组中与30 ℃或pH 3.0相比,差异显著(P<0.05)和极显著(P<0.01);#和##分别表示pelA组中与30 ℃或pH 3.0相比,差异显著(P<0.05)和极显著(P<0.01)。
将pelA+CBM和pelA分别置于不同温度(30~80 ℃)孵育2 h后测定残余酶活力。当在温度为30~50 ℃时孵育2 h,pelA的相对酶活力保持在80%以上,说明该重组酶在30~50 ℃热稳定性较好。但当该重组酶在温度60 ℃时孵育2 h后,相对酶活力下降至40%以下。pelA+CBM在孵育温度为30~70 ℃时均呈现较好的稳定性,但当温度上升至80 ℃时,相对酶活力骤降,低于40%(图5-b)。上述结果表明,CBM对pelA的热稳定性有积极影响,这与研究表明的CBM具有提高酶稳定性的能力相契合[30]。
当反应体系在pH 4.0~6.0,pelA的相对酶活力均高于70%。在pH 5.0时测得最高酶活力,而当pH>6.0,酶活力下降至60%以下。pelA+CBM在pH 3.0~7.0均有60%以上的相对酶活力,且在pH 6.0时达到最高(图5-c)。由此可知,重组酶pelA和pelA+CBM在酸性条件下均具有较好的酶活力。然而,当融合CBM时,pelA的最适pH从5.0升至6.0,推测与CBM具有提高酶的稳定性能力有关。
为测定pelA和pelA+CBM在不同pH条件下的耐受性,将其分别置于不同pH(3.0~7.0)的缓冲液中,40 ℃下孵育2 h后测定残余酶活力。当pH 4.0~5.0时,pelA孵育2 h后相对酶活力保持在60%以上(图5-d),该重组酶在酸性条件下具有较好的稳定性;而当pH>5.0时,相对酶活力开始下降,在pH为7.0时相对酶活力<35%。相反,pelA+CBM在pH为4.0~7.0时均有80%以上的相对酶活力,这个结果说明,CBM对pelA在中性或碱性环境下的稳定性有影响[30]。
3 结论
酸性果胶裂解酶作为一种能在酸性环境下裂解高酯化果胶的果胶酶,在果蔬汁食品产业、医药与天然产物的提取等方面有着重要的应用。E.coli作为常用的原核表达系统,拥有生长周期短、成本低、操作简便等优势,常被用来异源表达蛋白酶。然而酸性果胶裂解酶在E.coli中多以包涵体形式存在,虽然可以通过包涵体复性的方式提高其溶解度,但操作繁琐,酶蛋白制作效率低,使E.coli无法作为酸性果胶裂解酶的分子改造平台。对此,有研究者将酸性果胶裂解酶的分子改造在真菌或酵母中进行。但存在宿主生长缓慢,实验操作步骤繁琐以及改造周期长等问题。本研究成功构建了重组pelA在E.coli中的表达系统,通过N端融合蛋白标签减少pelA包涵体的形成。该方法有效提高了蛋白溶解性,使pelA的表达量从0.79 U/mL提升至3.25 U/mL,便于对pelA进行纯化及性质测定,同时降低酶蛋白制备成本,提高分子改造效率。此外,对E.coli产pelA的酶学性质进行测定,结果表示该酶在pH 4.0~6.0、30~50 ℃均有较好的稳定性,其最适反应温度和最适反应pH分别为40 ℃和5.0。为了满足实际生产时对耐酸性和热稳定性的要求,可通过定向进化或理性设计对酶分子进行改造。本研究构建的酸性果胶裂解酶表达菌株成功使A.niger酸性果胶裂解酶在E.coli中表达,这为后续重组pelA的分子改造提供了重要的数据基础。
[1] 许均华, 李高阳, 李志坚.高产果胶酶菌株的选育及其发酵生产的研究进展[J].食品与机械, 2011, 27(1):146-150.
XU J H, LI G Y, LI Z J.Research progress in pectinase high-yield strains and the fermentation of pectinase[J].Food &Machinery, 2011, 27(1):146-150.
[2] FU Y J, LIU W, ZU Y G, et al.Enzyme assisted extraction of luteolin and apigenin from pigeonpea[Cajanus cajan (L.) Mill sp.]leaves[J].Food Chemistry, 2008, 111(2):508-512.
[3] 张海燕, 吴天祥.微生物果胶酶的研究进展[J].酿酒科技, 2006(9):82-85.
ZHANG H Y, WU T X.Research progress in pectinase[J].Liquor-Making Science &Technology, 2006(9):82-85.
[4] KASHYAP D R, VOHRA P K, CHOPRA S, et al.Applications of pectinases in the commercial sector:A review[J].Bioresource Technology, 2001, 77(3):215-227.
[5] HE Y L, PAN L, WANG B.Efficient over-expression and application of high-performance pectin lyase by screening Aspergillus niger pectin lyase gene family[J].Biotechnology and Bioprocess Engineering, 2018, 23(6):662-669.
[6] Y LD
LD Z S, MATA-G
Z S, MATA-G MEZ M A, TAR
MEZ M A, TAR C, et al.Partial purification of a polygalacturonase from a new Aspergillus sojae mutant and its application in grape mash maceration[J].International Journal of Food Science &Technology, 2017, 52(3):834-842.
C, et al.Partial purification of a polygalacturonase from a new Aspergillus sojae mutant and its application in grape mash maceration[J].International Journal of Food Science &Technology, 2017, 52(3):834-842.
[7] ANAND G, YADAV S, YADAV D.Purification and biochemical characterization of an exo-polygalacturonase from Aspergillus flavus MTCC 7589[J].Biocatalysis and Agricultural Biotechnology, 2017, 10:264-269.
[8] ATANASOVA L, DUBEY M, GRUJI M, et al.Evolution and functional characterization of pectate lyase PEL12, a member of a highly expanded Clonostachys rosea polysaccharide lyase 1 family[J].BMC Microbiology, 2018, 18(1):178.
M, et al.Evolution and functional characterization of pectate lyase PEL12, a member of a highly expanded Clonostachys rosea polysaccharide lyase 1 family[J].BMC Microbiology, 2018, 18(1):178.
[9] JOHN J, SURENDRANATHAN KAIMAL K K, SMITH M L, et al.Advances in upstream and downstream strategies of pectinase bioprocessing:A review[J].International Journal of Biological Macromolecules, 2020, 162:1086-1099.
[10] AHMED I, ZIA M A, HUSSAIN M A, et al.Bioprocessing of citrus waste peel for induced pectinase production by Aspergillus niger;its purification and characterization[J].Journal of Radiation Research and Applied Sciences, 2016, 9(2):148-154.
[11] SIDDIQUI M A, PANDE V, ARIF M.Production, purification, and characterization of polygalacturonase from Rhizomucor pusillus isolated from decomposting orange peels[J].Enzyme Research, 2012, 2012:138634.
[12] 吴倩, 周峻沛, 唐湘华, 等.Penicillium sp.JF09酸性果胶酶的发酵条件、酶学性质及其在苹果汁澄清中的应用潜力研究[J].食品科技, 2013, 38(8):42-47.
WU Q, ZHOU J P, TANG X H, et al.The acid pectinase from Penicillium sp.JF09:Fermentation optimization, enzymatic characterization and potential for application in apple juice clarification[J].Food Science and Technology, 2013, 38(8):42-47.
[13] EL ENSHASY H A, ELSAYED E A, SUHAIMI N, et al.Bioprocess optimization for pectinase production using Aspergillus niger in a submerged cultivation system[J].BMC Biotechnology, 2018, 18(1):71.
[14] AMIN F, BHATTI H N, BILAL M.Recent advances in the production strategies of microbial pectinases:A review[J].International Journal of Biological Macromolecules, 2019, 122:1017-1026.
[15] PÉREZ-FUENTES C, CRISTINA RAVANAL M, EYZAGUIRRE J.Heterologous expression of a Penicillium purpurogenum pectin lyase in Pichia pastoris and its characterization[J].Fungal Biology, 2014, 118(5-6):507-515.
[16] LI C, ZHOU J W, DU G C, et al.Developing Aspergillus niger as a cell factory for food enzyme production[J].Biotechnology Advances, 2020, 44:107630.
[17] 彭小波. 果胶裂解酶在毕赤酵母中高效表达及酶学性质分析[D].武汉:武汉轻工大学, 2018.
PENG X B.High-level expression pectate lyase in Pichia pastoris and enzymatic determination[D].Wuhan:Wuhan Polytechnic University, 2018.
[18] DAMAK N, ABDELJALIL S, KOUBAA A, et al.Cloning and heterologous expression of a thermostable pectate lyase from Penicillium occitanis in Escherichia coli[J].International Journal of Biological Macromolecules, 2013, 62:549-556.
[19] 谢茂芳, 薛保国, 吴坤.黑曲霉果胶裂解酶基因的克隆与原核表达[J].河南农业科学, 2013, 42(1):82-85.
XIE M F, XUE B G, WU K.Cloning and prokaryotic expression of pectin lyase gene from Aspergillus niger[J].Journal of Henan Agricultural Sciences, 2013, 42(1):82-85.
[20] 杨欣伟. 黑曲霉EIM-6果胶裂解酶基因的克隆与表达[D].福州:福建师范大学, 2009.
YANG X W.Cloning and expression of pectin lyase gene from Aspergillus niger EIM-6[D].Fuzhou:Fujian Normal University, 2009.
[21] HADJ SASSI A, TRIGUI-LAHIANI H, ABDELJALIL S, et al.Enhancement of solubility, purification and inclusion-bodies-refolding of an active pectin lyase from Penicillium occitanis expressed in Escherichia coli[J].International Journal of Biological Macromolecules, 2017, 95:256-262.
[22] 王颖, 董晓燕, 孙彦.蛋白质复性技术研究进展[J].生物工程进展, 2002, 22(2):61-65.
WANG Y, DONG X Y, SUN Y.Research advances in protein renaturation technology[J].Progress in Biotechnology, 2002, 22(2):61-65.
[23] KAY T.Overview of bacterial expression systems for heterologous protein production:From molecular and biochemical fundamentals to commercial systems[J].Applied Microbiology and Biotechnology, 2006, 72(2):211-222.
[24] 周俊杰. 融合标签酰基载体蛋白对外源蛋白热稳定性的影响研究[D].广州:华南农业大学, 2019.
ZHOU J J.Effect of fusion label acyl carrier protein on thermal stability of exogenous proteins[D].Guangzhou:South China Agricultural University, 2019.
[25] ECHE S, GORDON M L.Recombinant expression of HIV-1 protease using soluble fusion tags in Escherichia coli:A vital tool for functional characterization of HIV-1 protease[J].Virus Research, 2021, 295:198289.
[26] ZONDAGH J, WILLIAMS A, ACHILONU I, et al.Overexpression, purification and functional characterisation of wild-type HIV-1 subtype C protease and two variants using a thioredoxin and his-tag protein fusion system[J].The Protein Journal, 2018, 37(4):369-379.
[27] MAHALIK S, SHARMA A K, MUKHERJEE K J.Genome engineering for improved recombinantprotein expression in Escherichia coli[J].Microbial Cell Factories, 2014, 13(1):177.
[28] HOFFMAM Z B, ZANPHORLIN L M, COTA J, et al.Xylan-specific carbohydrate-binding module belonging to family 6 enhances the catalytic performance of a GH11 endo-xylanase[J].New Biotechnology, 2016, 33(4):467-472.
[29] MALGAS S, PLETSCHKE B I.The effect of an oligosaccharide reducing-end xylanase, BhRex8A, on the synergistic degradation of xylan backbones by an optimised xylanolytic enzyme cocktail[J].Enzyme and Microbial Technology, 2019, 122:74-81.
[30] TEO S C, LIEW K J, SHAMSIR M S, et al.Characterizing a halo-tolerant GH10 xylanase from Roseithermus sacchariphilus strain RA and its CBM-truncated variant[J].International Journal of Molecular Sciences, 2019, 20(9):2284.