2-苯乙醇(2-phenylethanol,2-PE)是一种有玫瑰香味和良好理化性质的高级芳香醇,天然存在于玫瑰、茉莉等植物的叶、花及提取的精油中[1],不仅如此,2-PE还可作为食品添加剂对葡萄酒、黄酒、面包等发酵食品中风味物质起增效作用[2],其作为杀菌剂、植物保鲜剂以及作为底物合成高附加值药物,在日化用品、医药卫生领域也有广泛应用[3]。目前,2-PE的生产方法主要有植物提取、化学合成和生物合成3种。植物提取法和化学合成法因原料和副产物问题造成成本高及产品质量差[4]。与之相比,生物合成法以生产周期短,反应条件温和、环保,原料廉价易得等优点逐渐成为研究热点[5]。
目前国内外发酵生产2-PE所用菌种主要有酿酒酵母(Saccharomyces cerevisiae)[6]、克鲁维酵母(Kluyveromyces)[7]、毕赤酵母(Pichia kudriavzevii)[8]等,但其生产率低,不适合工业化。基因工程改造的菌株,如WANG等[9]构建的重组酿酒酵母YS58(G1-A8-A10-A2)GDH在最佳反应条件下,5 L发酵罐2-PE产量高达6.3 g/L。YANG等[10]构建的重组大肠杆菌上调艾氏途径中苯乙醛合成酶PAAS的表达,2-PE产量为0.39 g/L。ZHANG等[11]通过过量表达苯丙酮酸途径中磷酸烯醇式丙酮酸合酶ppsA和转酮酶tktA,构建的重组大肠杆菌2-PE产量为0.32 g/L。虽然基因工程菌都有较高的2-PE产量,但高浓度的2-PE对细胞的毒性已成为微生物发酵法生产2-PE的瓶颈问题[7]。酶法合成则不会受到产物对微生物的抑制[12]。然而,大多数天然酶的活性、稳定性和选择性不符合非天然过程要求,不适合工业应用[13]。因此,可以获得稳定性好、酶活性高的蛋白质工程技术受到广泛关注。蛋白质工程通过理性设计和半理性设计提高酶的催化性能[14-18],但由于理性设计模型的限制,往往会导致改造效果不佳,因而半理性设计成为一种新的分子改造手段。近年来,利用蛋白质结构和功能的信息以及预测算法来预选合适的靶点[19],已被用于增加近平滑假丝酵母羰基还原酶的活性与稳定性[14-15],马氏克鲁维酵母醛酮还原酶的活性与立体选择性[16-17],腈水合酶的热稳定性[19]。半理性设计的方法已成为研究者改进生物催化剂有力且有效的新策略[19]。
本文拟通过易错PCR技术构建羰基还原酶CpCR突变文库,利用2,4-二硝基苯肼(2,4 dinitrophenylhydrazine,DNPH)高通量筛选阳性突变株,通过测序技术确定氨基酸突变位点,再利用半理性设计进行虚拟饱和突变,发现更好的突变位点,然后采用定点突变技术进行构建和评价,筛选得到2-PE产率高的突变酶,并研究催化时间、温度、pH、底物浓度等因素对羰基还原酶CpCR催化转化2-PE的影响,为酶法合成2-PE提供科学基础。
1 材料与方法
1.1 材料与仪器
菌株pACYC Duet-1 Escherichia coli BL21 (DE3)由本实验室构建及保存;即用型易错PCR试剂盒,北京天恩泽基因科技有限公司;限制性酶切酶Sal I和Pst I,New England Biolabs;质粒载体pACYCDuet-1,上海生工生物工程有限公司;DNPH、苯乙醛(phenylacetaldehyde, PAAH),2-苯乙醇,1-(4-氯苯基)乙醇,进口或分析纯;葡萄糖脱氢酶,安琪酵母股份有限公司;Mut Express II Fast Mutagenesis Kit V2试剂盒,Vazyme/诺唯赞生物科技有限公司。
SX-700蒸汽灭菌器、MX-307落地高速冷冻离心机,日本Tomy公司;SWCJ-2FD超净工作台,苏州净化设备有限公司;LRH-250A生化培养箱,泰宏医疗器械有限公司;UV1800紫外可见分光光度计,岛津企业管理有限公司;ZQLY-180S恒温培养摇床,上海知楚仪器有限公司,JY92-IIDN超声破碎仪、FL-GC9720气相色谱仪、C1000 Touch PCR仪、Doc XR+ Gel凝胶成像仪,Bio-Rad公司。
1.2 实验方法
1.2.1 突变文库的构建及高通量筛选
采用易错PCR的方法,易错PCR引物如下所示。
ep-F:5-ATTGCCTGCAG(Pst I)ATGACTAAAGCAGTACCAGACA-3;ep-R:5-ACTTGTCGAC(Sal I)TAAGCTTTGAATGCTTTGTCG-3。
反应体系为:3 μL Mix,20 ng pACYCDuet-1-cpcr模板,3 μL dNTP,3 μL MnCl2,1 μL ep-F(10 μmol/L),1 μL ep-R(10 μmol/L),0.5 μL Taq DNA聚合酶(5 U/μL),17.5 μL dd H2O。PCR扩增程序:95 ℃预变性30 s;95 ℃变性10 s,64 ℃退火20 s,72 ℃延伸45 s,循环30次,72 ℃继续延伸5 min。
使用限制性内切酶将PCR产物插入表达载体pACYC Duet-1 的Pst I和Sal I位点之间,再转化至E.coil BL21(DE3)感受态细胞,培养1 h,将重组细胞涂布至含有25 μg/mL氯霉素的LB平板上,37 ℃下避光过夜培养构建随机文库[20]。
利用DNPH能与醛或酮发生缩合反应,生成红色或黄色的二硝基苯腙沉淀,且所生成的硝基苯腙沉淀可被碱性溶液溶解而生成红棕色的溶液这一高灵敏度特征反应对易错PCR突变库进行96深孔板(2.2 mL)高通量筛选[21](图1)。控制酶反应体系体积为100 μL:粗酶液20 μL,10 μL 0.1 mol/L的葡萄糖溶液,10 μL的GDH,60 μL 10 mmol/L的PAAH溶液。30 ℃,200 r/min催化40 min后加入100 μL 4 g/L的DNPH后继续反应30 min。加入1 mL的100 g/L KOH溶液显色,混匀静置10 min后取上清液进行全波长扫描。确定反应体系中不同含羰基组分生成的苯腙衍生物最大吸收波长。

图1 DNPH法筛选流程
Fig.1 Screening process for DNPH method
1.2.2 半理性设计改造及验证
1.2.2.1 半理性设计改造
利用Discovery Studio 2017软件将羰基还原酶CpCR(PDB号:4OAQ)与底物PAAH分子进行半柔性CDOCKER对接,据文献报道,酶与底物结合口袋以及辅酶结合部位附近的氨基酸残基对酶活性有重要影响[22-23],因此,在易错PCR筛选出的氨基酸残基位点的基础上,结合酶与底物结合口袋及辅酶结合部位附近的对接结果,对其进行定点饱和突变。利用Mut Express II Fast Mutagenesis Kit V2试剂盒进行定点突变,构建突变文库,经测序验证后培养并保藏菌种待用。
1.2.2.2 酶活力及酶动力学参数测定
以PAAH为底物,突变体酶与wtCpCR的酶活力测定及计算方法参照文献[21]。选用0.5、1.0、1.5、2.0、3.0、4.0、5.0、6.0 mmol/L等不同浓度,测定wtCpCR和突变体酶的动力学参数Km和Vmax值。计算催化常数kcat及催化特异常数kcat/Km。比较野生型酶CpCR和突变体酶对PAAH的亲和能力和催化效率。
1.2.2.3 羰基还原酶及其突变体的热稳定性研究
取适量酶溶液,置于50 r/min、30 ℃的水浴锅中进行温育。定时取样,测定剩余酶活力,确定各种酶在不同体系中的半衰期。取适量酶溶液置于4~50 ℃条件下保温2 h,测定不同温度下剩余酶活力,计算该酶的T50(酶活力降低至初始酶活力的50%时所对应的温度)值,考察该酶温度稳定性。
1.2.3 2-PE的转化合成和检测方法
1.2.3.1 2-PE的转化合成
筛选得到的突变株经诱导表达培养后,湿菌体加入适当体积的磷酸缓冲液(0.1 mol/L,pH 7.5)重悬,超声破碎(工作4 s,停6 s,总时间25 min)后,10 000 r/min,4 ℃离心10 min,取上清液即为粗酶液。酶催化体系为:总体系10 mL,包含1 mL粗酶液(比酶活力约为3 U/mg),0.1 g葡萄糖,0.2 mL GDH,质量浓度为800 mg/L的PAAH溶液。30 ℃,200 r/min反应12 h。
1.2.3.2 2-PE的检测方法
取0.5 mL转化后的样品,加入0.1 mL 0.966 mg/mL的内标物[1-(4-氯苯基)乙醇],用3倍体积的正己烷萃取反应溶液,10 000 r/min离心2 min取上相。用0.22 μm有机滤膜过滤。色谱条件:DB-FFAP色谱柱(30 m×0.250 mm×30.25 μm);柱温采用程序升温:初温80 ℃,保持5 min后以10 ℃/min速率升至200 ℃;进样口温度260 ℃,载气N2,流速0.7 mL/min分流进样,分流比10∶1。FID检测器260 ℃,H2 40 mL/min;空气 400 mL/min;尾吹气 30 mL/min。进样量1 μL。
1.2.4 影响酶催化合成2-PE的因素实验
1.2.4.1 催化时间
取适量粗酶液,按照1.2.2节的方法转化合成2-PE,分别在0、2、4、6、8、10、12 h取样,气相色谱检测PAAH和2-PE含量,计算转化率和产率,考察酶完成催化反应所需时间。
1.2.4.2 温度
取适量粗酶液,按照1.2.2节的方法在28~38 ℃条件下转化合成2-PE,气相色谱检测PAAH和2-PE含量,通过产率考察酶最适催化温度。
1.2.4.3 pH
分别配制pH 5.5~8.5的0.1 mol/L的磷酸缓冲液,按照1.2.2节的方法分别破碎得到粗酶液,用不同pH的磷酸缓冲液配制800 mg/L的PAAH溶液,以1.2.4.2节中最适温度条件下转化合成2-PE,检测PAAH和2-PE含量,计算产率,考察酶最适催化pH。
1.2.4.4 底物浓度
取适量粗酶液,在不同质量浓度底物PAAH溶液(400~1 600 mg/L)条件下催化合成2-PE,按照1.2.2节的方法,在1.2.4.2和1.2.4.3中最适温度和pH条件下转化合成2-PE,以产率高低确定最适底物浓度。
2 结果与分析
2.1 高产2-PE突变株的筛选
在DNPH法筛选的酶反应体系中,除了底物PAAH外,其他成分包括辅酶NADPH、葡萄糖、GDH、羰基还原酶CpCR都可能与DNPH反应,从而影响DNPH检测法的准确性。图2为PAAH和反应体系中含有羰基的其他组分与DNPH反应后的全波长扫描对比图,结果显示碱性条件下PAAH与DNPH生成的对应的苯腙在510 nm波长处具有特征吸收峰,而其他干扰组分的特征吸收峰均在350~430 nm,说明反应体系中除底物以外的其他组分对DNPH检测法干扰很小,这一结论与周婕妤[24]的结论基本一致。
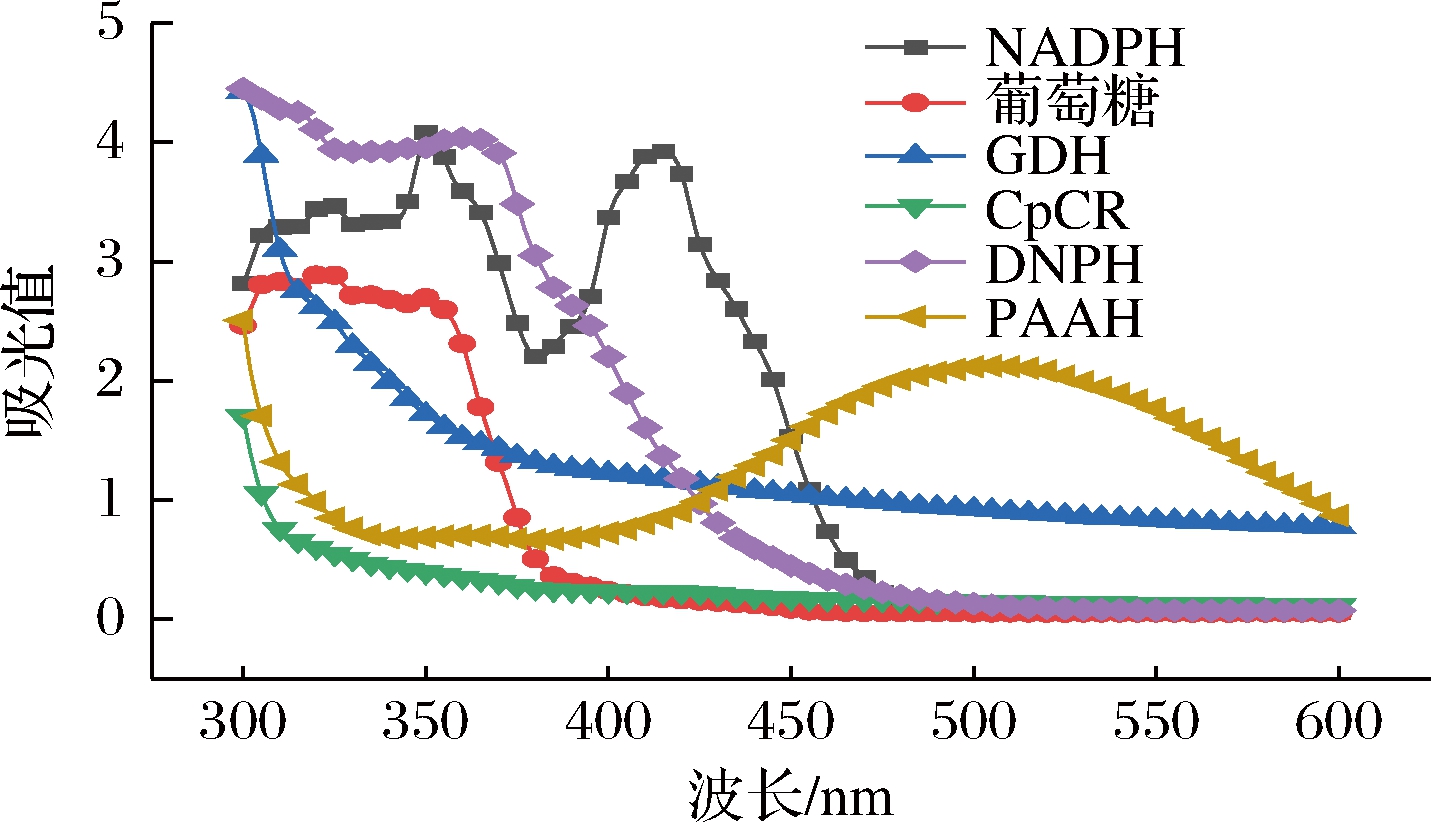
图2 NADPH、葡萄糖、GDH、CpCR、PAAH与DNPH反应的全波长扫描图
Fig.2 Absorbance spectrums of NADPH, glucose, GDH, CpCR and PAAH with DNPH
根据易错PCR反应体系构建了随机突变文库,突变后大部分突变菌株失活,仅有约10%的突变体保持对PAAH的还原活性,序列比对后根据突变氨基酸残基序列将突变体CpCR分为40~120、121~200、201~280、281~360四部分,以底物PAAH的转化率和OD510值为筛选标准的易错PCR结果如图3所示,OD510值越小、转化率越高的突变体为正突变体,选择正突变体的氨基酸残基位点进行进一步的半理性设计和酶的改造。
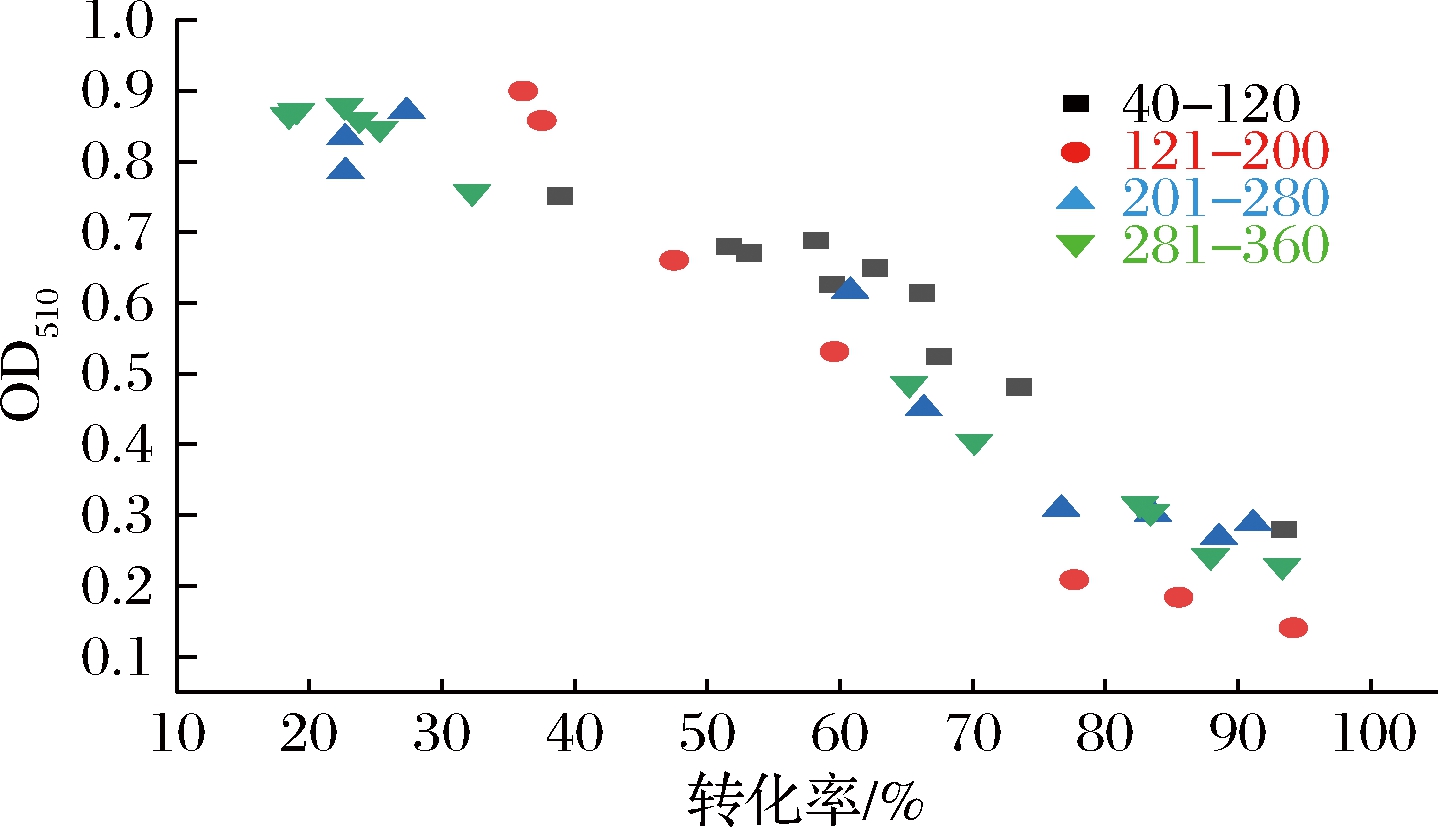
图3 易错PCR筛选结果
Fig.3 Screening of mutants from Error-prone PCR
2.2 半理性设计改造及验证
2.2.1 半理性设计改造
易错PCR筛选出的正突变体经测序、序列比对后得到Ile52、Thr54、Ala121、Leu165、Thr171、Pro284、Pro285、Lys289等氨基酸残基突变位点。CpCR(PDB号:4OAQ)与PAAH分子对接如图4-a所示,图4-b展示了酶与底物结合口袋及辅酶结合部位的氨基酸残基位点,图4-c表示酶与底物对接的氢键相互作用强弱。
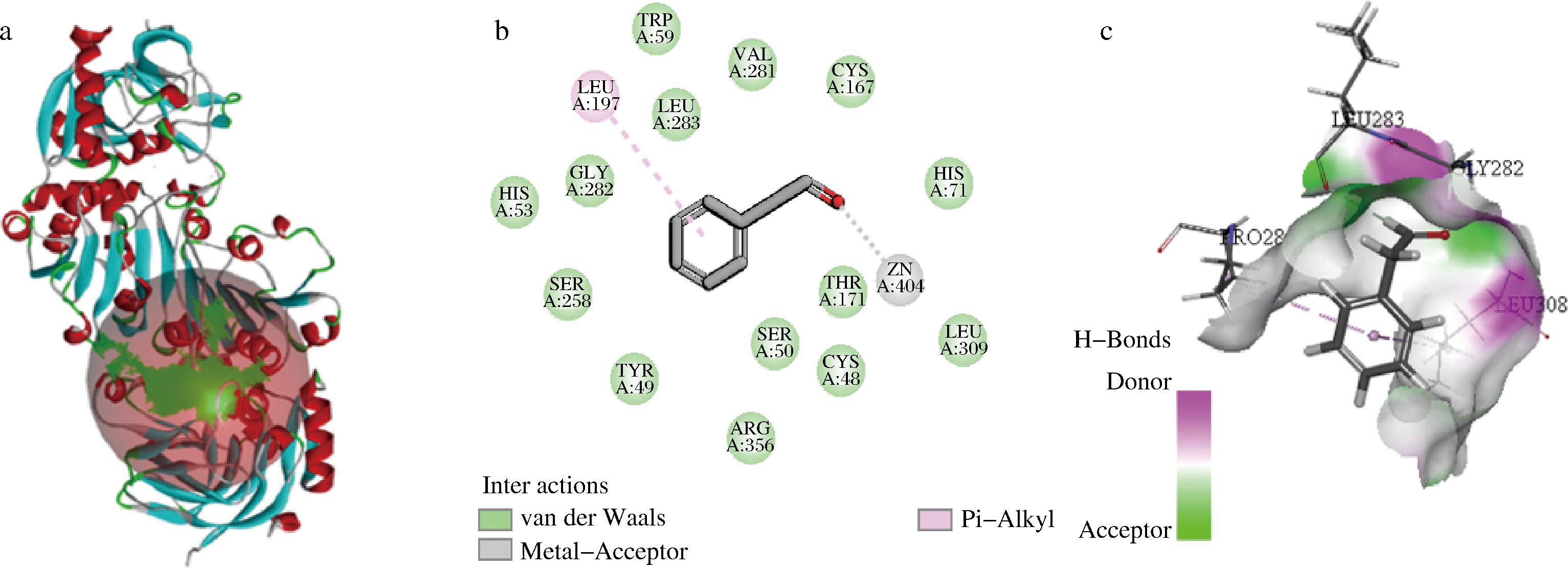
a-CpCR与PAAH对接构象图;b-CpCR与PAAH对接2D构象图;c-CpCR与PAAH对接氢键相互作用图
图4 CpCR与PAAH对接构象图及氢键相互作用图
Fig.4 Docking conformation and hydrogen bonding interaction diagrams of CpCR and PAAH
结合易错PCR筛选结果分别对其进行定点饱和突变。计算其结合突变能,结果如图5所示,根据突变自由能比较[ΔΔGmut=ΔΔGfold(mutant)-ΔΔGfold(wild type)],选择突变能最低的氨基酸进行定点突变,得到S50N、Q60W、A97K、L165D、T171F、P284E和L309W等7个突变体。
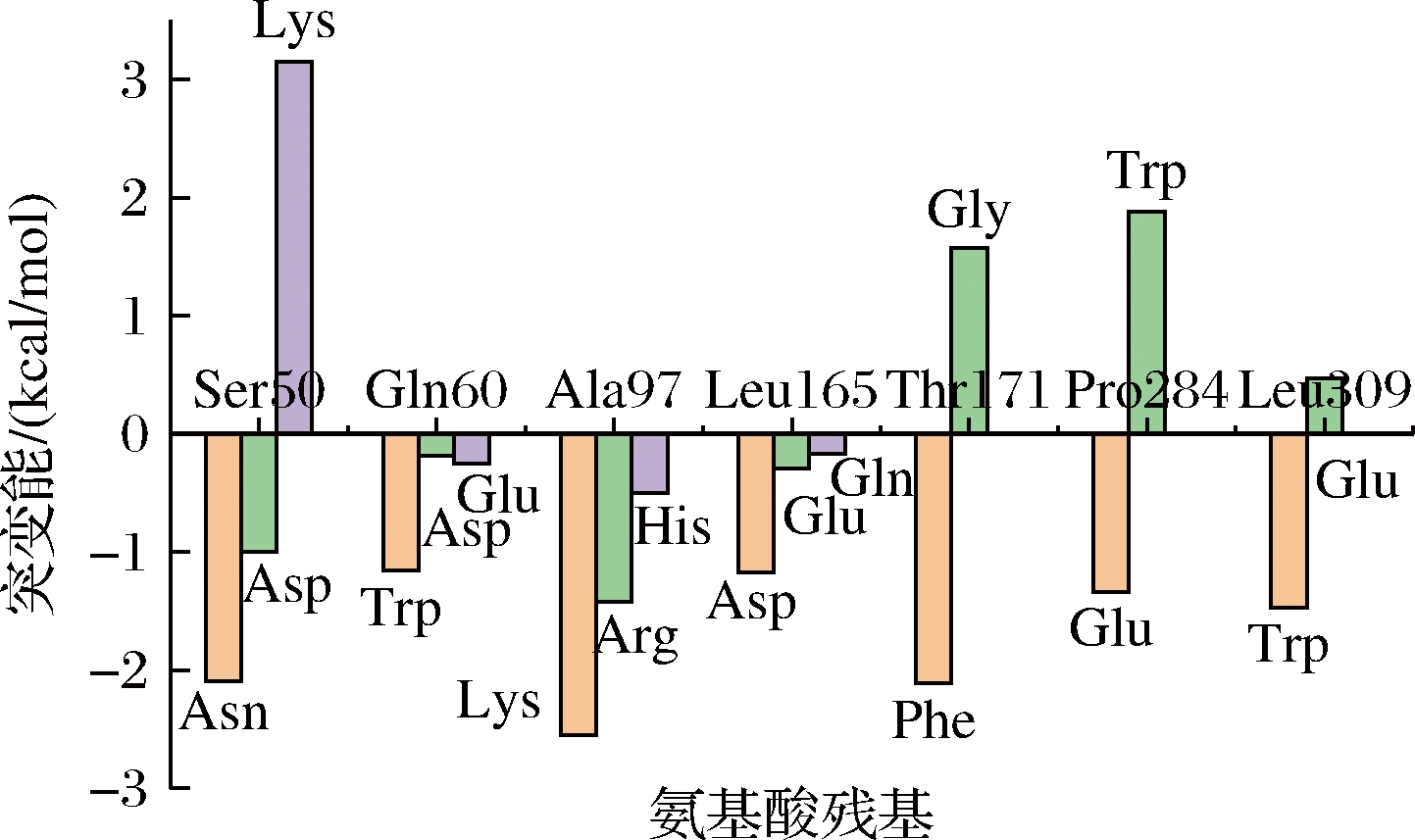
图5 不同氨基酸残基饱和突变能
Fig.5 Saturation mutation energy of different amino acid residues
2.2.2 酶活力及酶动力学参数测定
以wtCpCR作为参照,不同突变体酶的相对酶活力如图6-a所示,实验表明,T171F和L165D酶活力分别为11.46和11.96 U/mg,相比wtCpCR分别提高了10.14%和12.86%,说明171位和165位的氨基酸饱和突变增强了酶的催化能力。其余突变体酶的相对酶活力均有不同程度的减小。L309W的酶活力降至wtCpCR的10.43%,说明309位的亮氨酸是酶催化活性的关键位点。如图6-b所示,T171F和L165D催化合成2-PE的产率分别为91.31%和84.92%,相比wtCpCR分别提高了1.36倍和1.20倍。L309W产率降至2%以下,Q60W产率降低至29.9%,其余突变体酶的产率均有不同程度的提升。
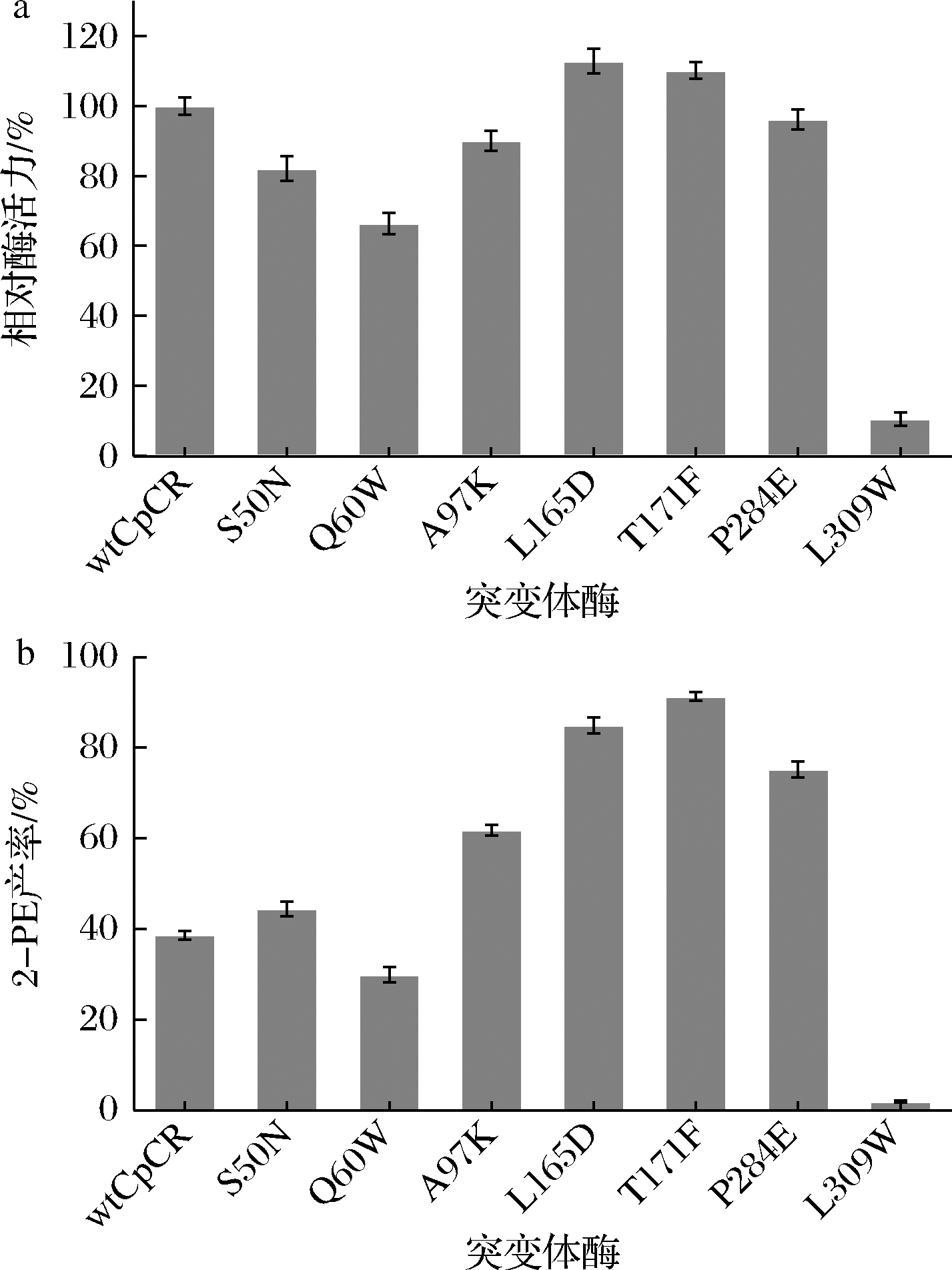
a-相对酶活力;b-催化合成2-PE产率
图6 wtCpCR和7种突变体酶的相对酶活力和转化合成2-PE的产率
Fig.6 Relative activity and 2-PE yield of wtCpCR and seven mutants
酶动力学参数和催化特性参数反映了酶结合底物能力和催化反应速率,以wtCpCR和7种突变体酶为对象测定酶对PAAH的动力学参数和催化特性参数,结果如表1所示,羰基还原酶wtCpCR及其突变体对PAAH的亲和力均较高,其中T171F和L165D的最大反应速率分别为wtCpCR的2.59倍和2.42倍。每个T171F分子和L165D分子1 s分别可以转化16个和14个左右的PAAH分子。虽然较高的Km值意味着对底物的亲和力低,但较高的kcat值更可取,因为生产应用中高底物负荷可以完全弥补低亲和力的不足,催化效率的衡量标准是高kcat值而不是高kcat/Km[25]。此外,酶与底物亲和力较低,其底物抑制可能更弱[26]。本文中T171F的2-PE产率和kcat值均高于L165D,而其kcat/Km值却低于后者,与SU等[25]的研究结论一致。
表1 不同突变株对PAAH的酶动力学参数及催化特性参数
Table 1 Enzyme kinetic parameters and catalytic characteristic parameters of different mutants for PAAH
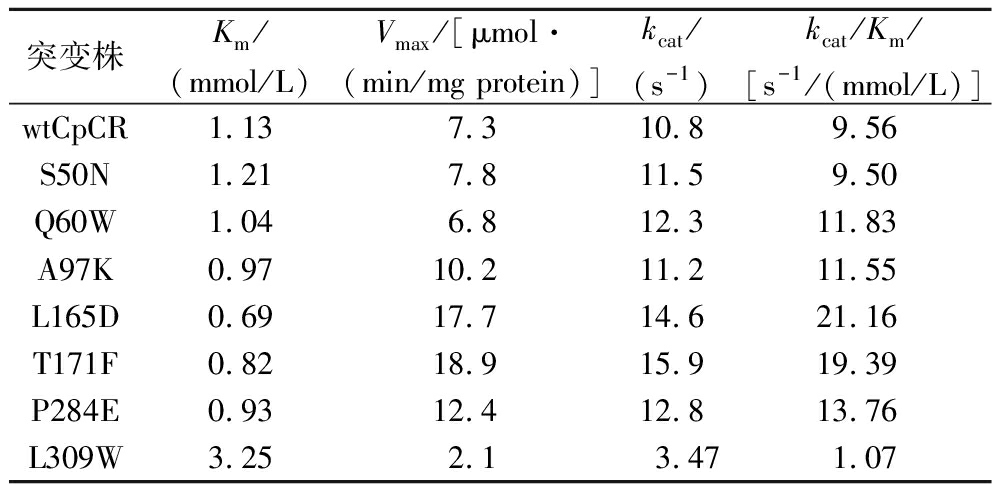
突变株Km/(mmol/L)Vmax/[μmol·(min/mg protein)]kcat/(s-1)kcat/Km /[s-1/(mmol/L)]wtCpCR1.137.310.89.56S50N1.217.811.59.50Q60W1.046.812.311.83A97K0.9710.211.211.55L165D0.6917.714.621.16T171F0.8218.915.919.39P284E0.9312.412.813.76L309W3.252.13.471.07
2.2.3 羰基还原酶及其突变体的热稳定性研究
从图7-a中可以看出,在PB缓冲液中,T171F、S50N、A97K的半衰期相比wtCpCR有所提升,其中T171F的半衰期提升至28 h。其余突变体酶半衰期均下降。由图7-b可知,突变酶的热稳定性有一定的变化,其中T171F的T50值为37 ℃,比wtCpCR提高1 ℃。L309W、Q60W和L165D的T50值为30~32 ℃,下降较大,酶的耐温性降低。
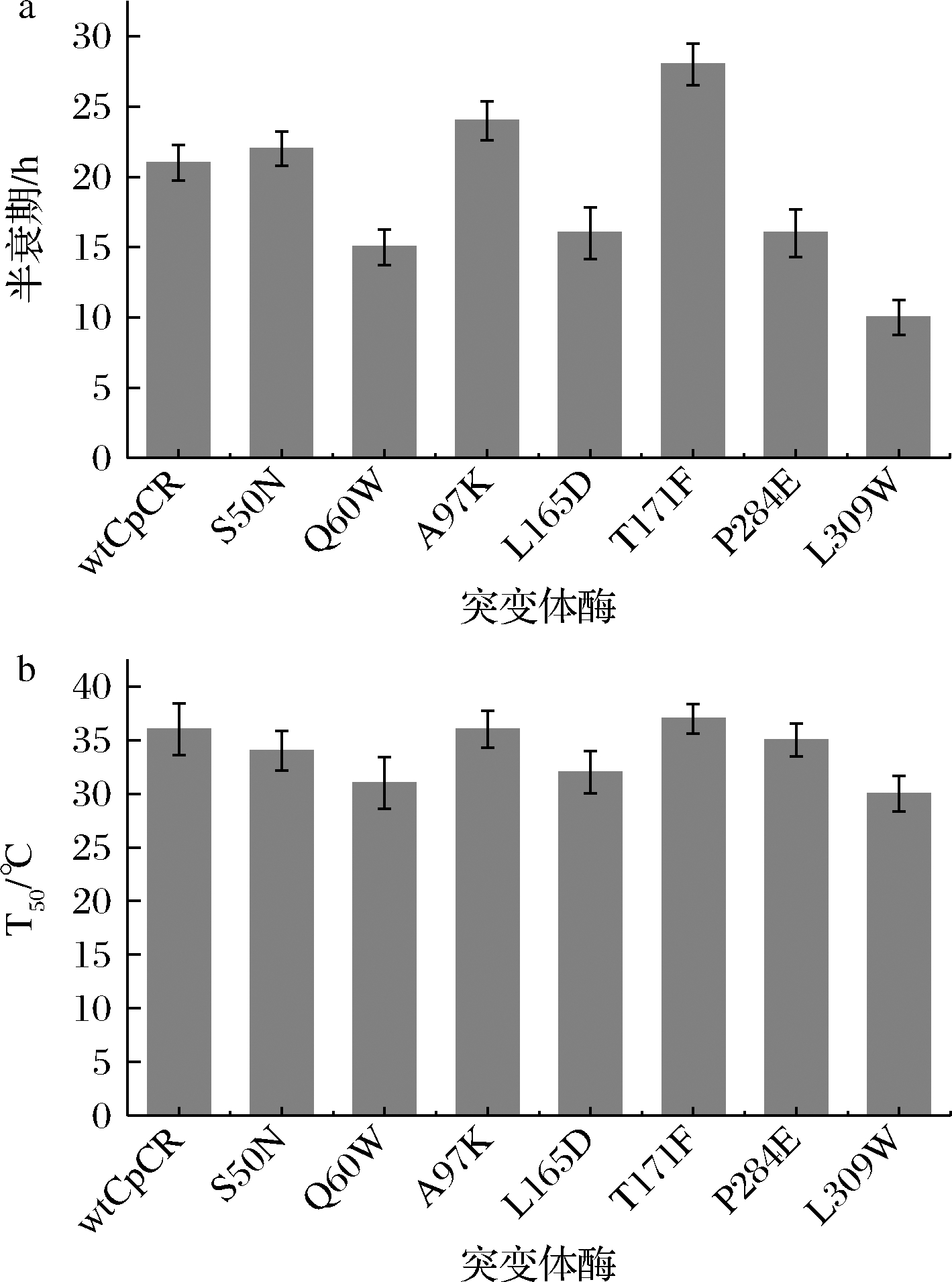
a-半衰期;b-T50值
图7 wtCpCR和6种突变体酶的半衰期和T50值变化
Fig.7 Changes of half-life and T50 of wtCpCR and six mutants
结合突变酶的相对比活力、催化合成2-PE产率、催化特性参数kcat值、以及半衰期和T50值,突变体T171F具有更强的催化能力和热稳定性,具有更好应用前景。因此,选择突变体T171F与wtCpCR进行催化合成2-PE条件研究。
2.3 突变体酶催化合成2-PE条件研究
以2.2节中筛选得到突变体酶T171F和wtCpCR催化合成2-PE的条件相对比,考察了催化时间、温度、pH、底物添加量等关键因素对酶催化合成2-PE的影响。
2.3.1 催化时间对合成2-PE的影响
酶催化相比于整细胞催化具有用时短,效率高的特点,按照1.2.4.1节中的方法,考察了突变体酶T171F和wtCpCR在12 h内2-PE的转化合成情况。结果如图8所示,wtCpCR可在10 h内基本完成转化反应,产率为42.61%,而突变体酶可在4 h内完成转化反应且产率可达90%以上,说明突变体酶T171F的催化效率相比wtCpCR有着较大的提升。
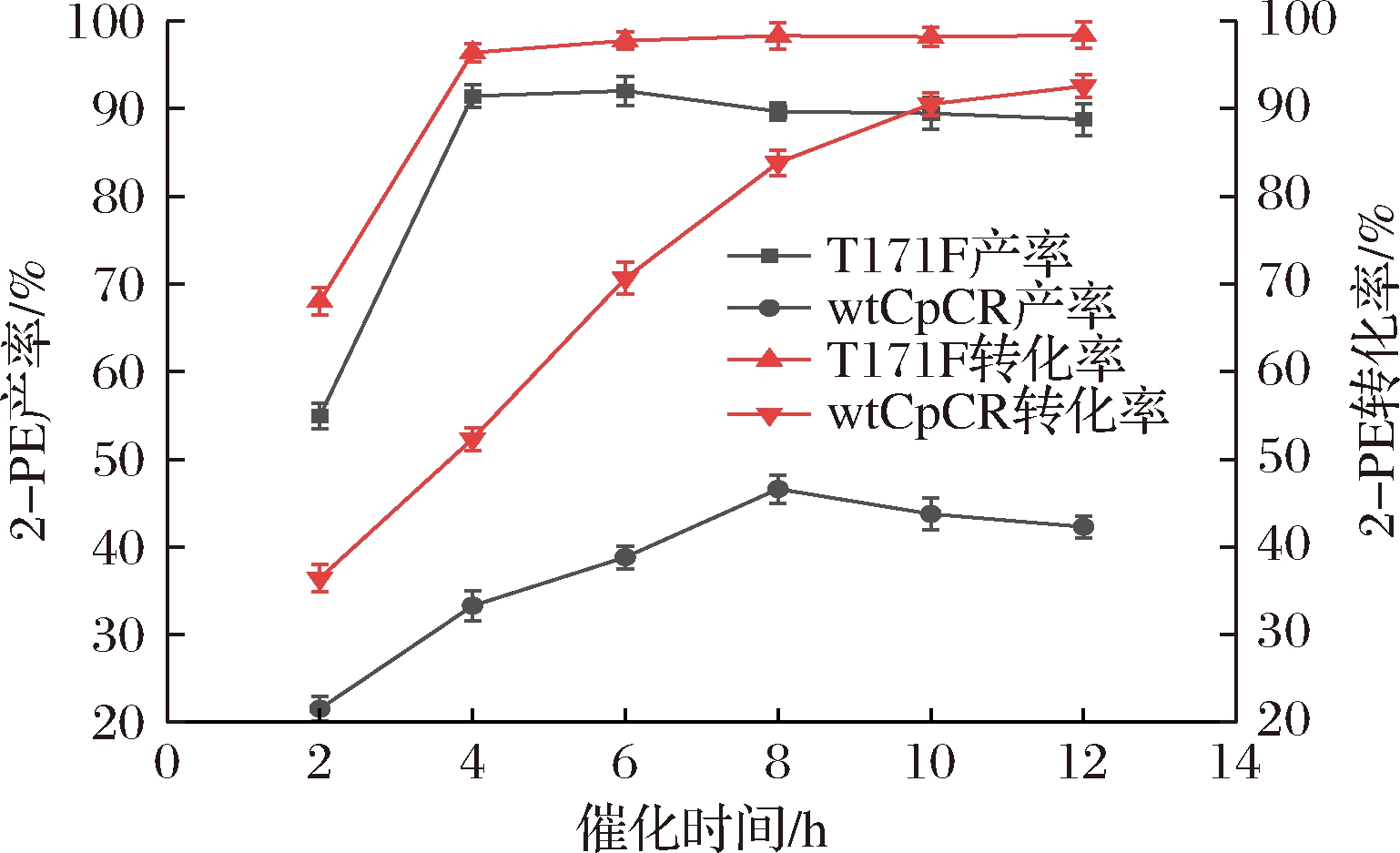
图8 酶催化时间对合成2-PE的影响
Fig.8 Effect of reaction time on enzyme synthesis of 2-PE
2.3.2 最适催化温度
温度可通过影响酶活性来影响产物的合成。参考wtCpCR温度稳定性以及最适温度条件[24],按照1.2.3.2节中的方法考察了28~38 ℃下的2-PE的合成情况。结果如图9所示,突变体酶T171F的最适催化温度为30 ℃,与wtCpCR一致。且其在28~32 ℃之间产率都能达到80%以上。
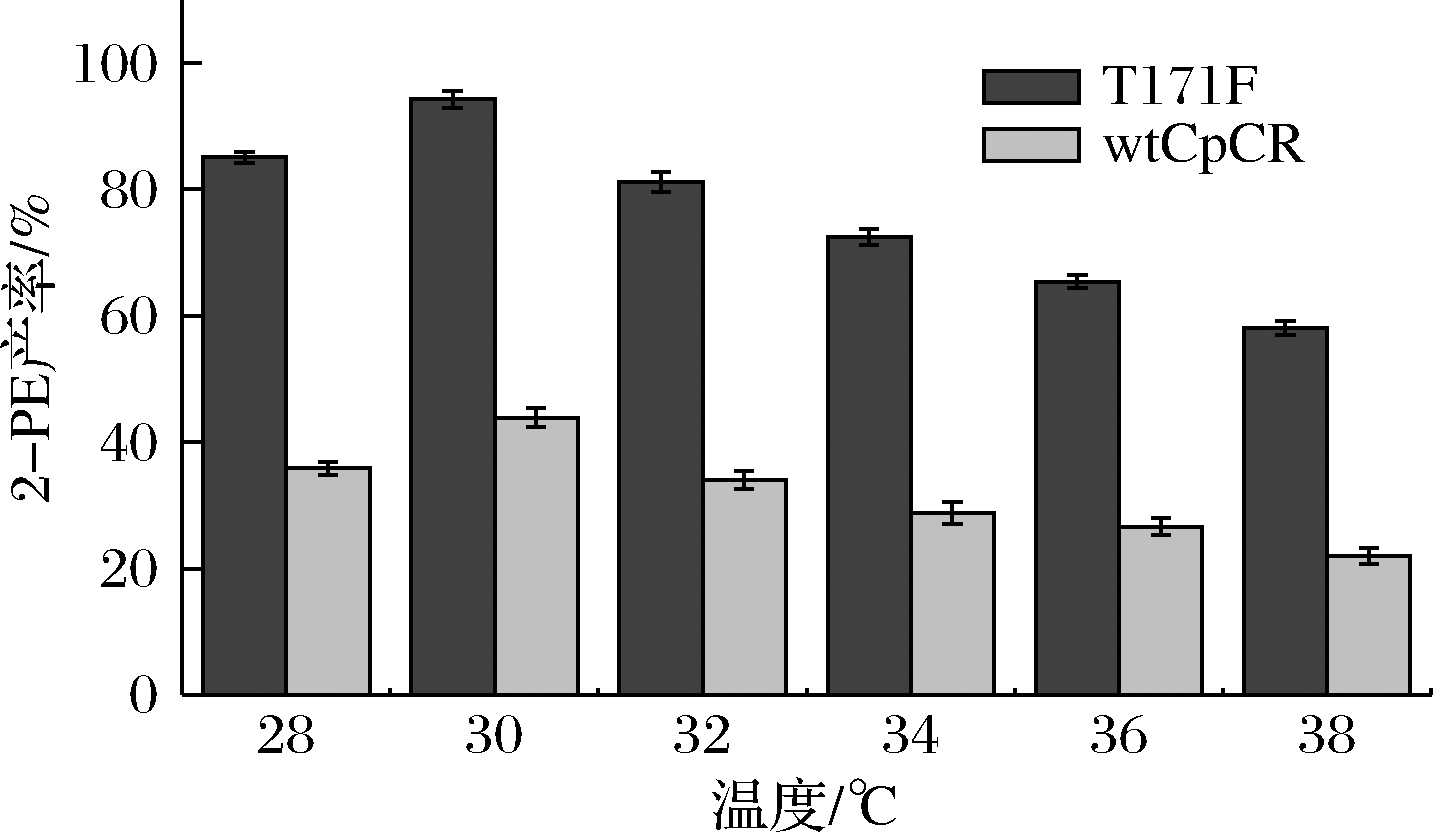
图9 温度对酶催化合成2-PE的影响
Fig.9 Effect of temperature on enzymatic synthesis of 2-PE
2.3.3 最适催化pH
pH值可以改变酶活性部位上有关基团的解离状态,从而影响酶与底物的结合,按照1.2.4.3节中的方法,考察了突变体酶T171F和wtCpCR在pH 5.5~8.5条件下2-PE的转化合成情况,结果如图10所示,wtCpCR最适催化pH值为7.0,产率可达54.7%,相比初始条件pH 7.5提高了近25%,且其在偏碱性范围内产率更高。T171F最适催化pH值为6.5,产率可达95%,但其在偏酸性范围内的产率更高,达到80%以上,且当pH值超过7.5时产率迅速降低至60%左右。说明171位的氨基酸突变改变了酶对PAAH的pH条件要求,从偏碱性改变为偏酸性。
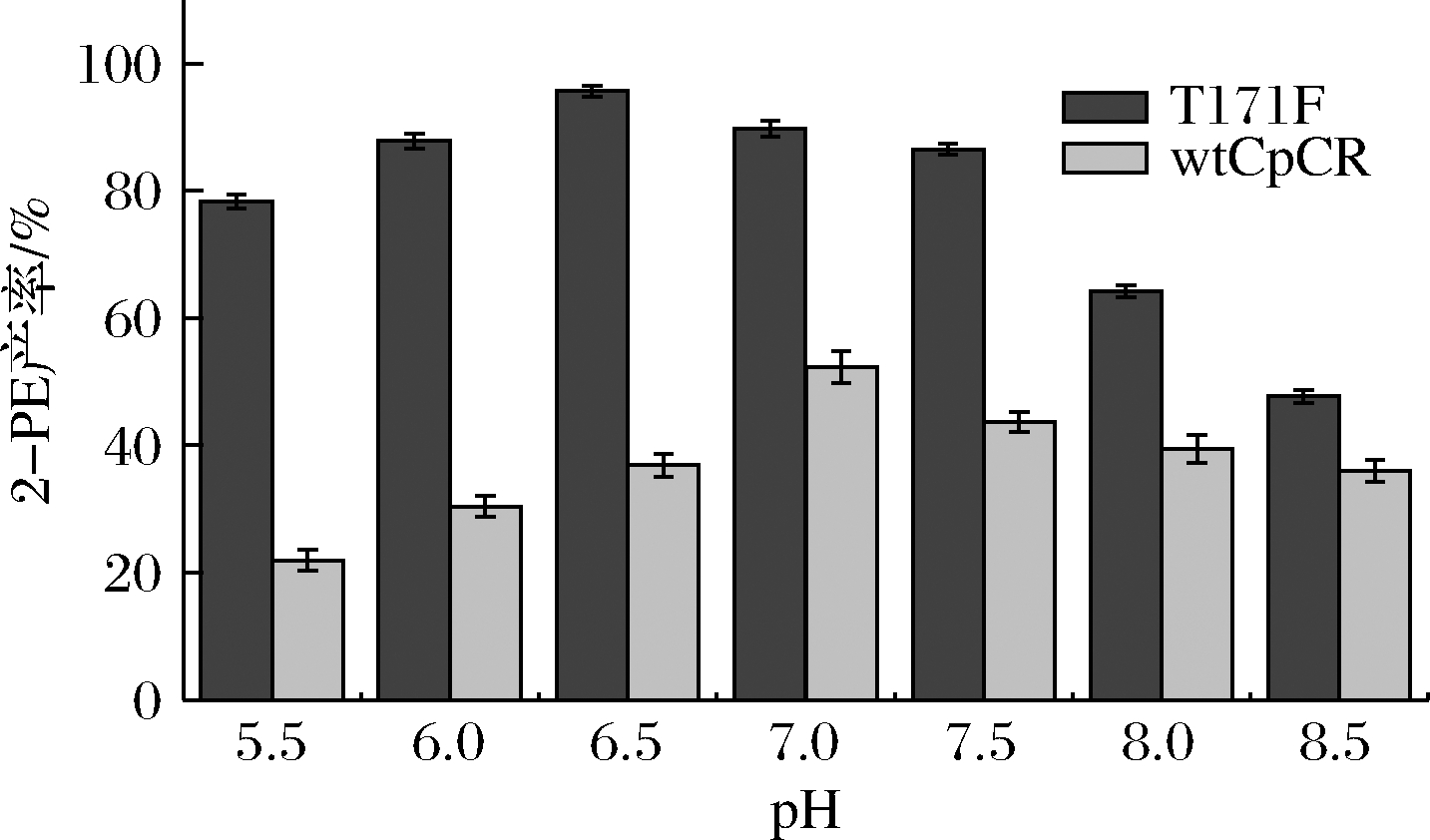
图10 pH对酶催化合成2-PE的影响
Fig.10 Effect of pH on enzymatic synthesis of 2-PE
2.3.4 底物浓度对酶催化合成2-PE的影响
底物浓度是酶催化效率的重要指标,在一定浓度的底物范围内,反应速率随底物浓度的增大而增大,但底物浓度过高时反而会出现抱团现象,导致酶底物结合口袋不适合容纳大基团底物,影响酶的催化效果[26],按照1.2.4.4节中的转化方法,考察了突变体酶T171F和wtCpCR在不同底物质量浓度(400~1 600 mg/L)下2-PE的转化合成情况。由图11可知,wtCpCR在底物质量浓度为400 mg/L时产率达到87%,但产率却随着底物浓度的增大而迅速降低,当底物质量浓度为800 mg/L时产率降至43%。而T171F的产率在底物质量浓度≤1 000 mg/L时均可达90%以上,当底物质量浓度>1 000 mg/L时,产率迅速降低。因此,突变体酶T171F能够耐受更高的底物浓度,具有更好的应用前景。
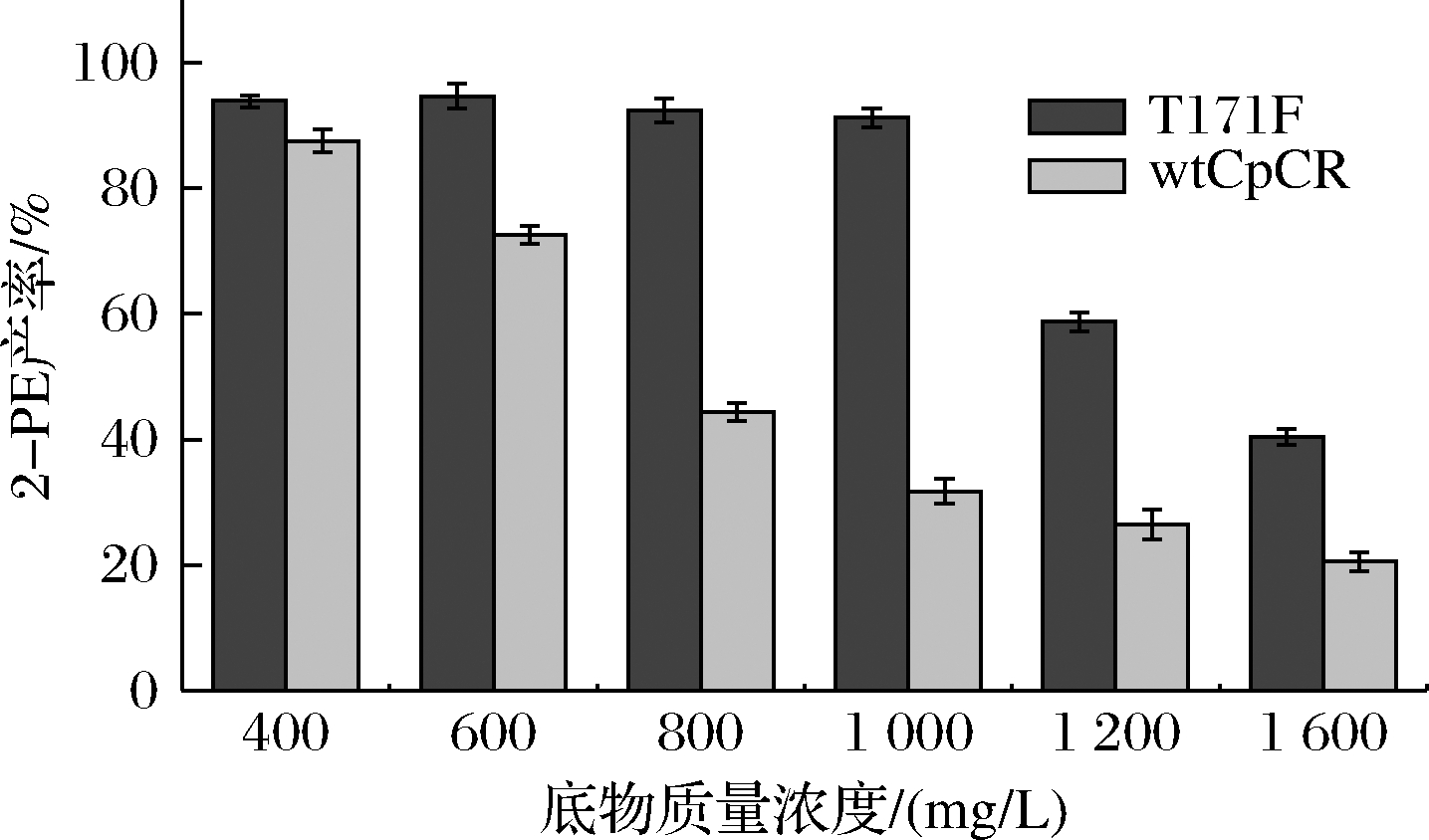
图11 底物浓度对酶催化合成2-PE的影响
Fig.11 Effect of substrate concentration on enzymatic synthesis of 2-PE
3 结论
本实验室通过易错PCR技术构建PAAH还原为2-PE的醛基还原酶突变文库,利用DNPH与醛基形成苯腙的特点高通量筛选获得正突变体,结合基因测序找出氨基酸突变位点,再通过Discovery Studio软件进行半理性设计和定点饱和突变技术构建7个突变体酶S50N、Q60W、A97K、L165D、T171F、P284E、L309W。结合相对酶活力、酶反应动力学参数、酶的热稳定性及催化合成2-PE产率分析,突变体T171F具有更强的催化能力和热稳定性。进一步研究了突变株T171F与wtCpCR进行酶催化转化2-PE工艺条件研究。结果表明,酶最适催化合成2-PE的温度为30 ℃;T171F最适pH为6.5,wtCpCR最适pH为7.0;PAAH质量浓度为1 000 mg/L时,T171F产率可达90%以上,wtCpCR则只有31.7%。而且突变体T171F相比wtCpCR催化时间由10 h降低到4 h,催化效率得到很大的提高。本研究为羰基还原酶CpCR催化合成2-PE提供重要的科学依据。
[1] QIAN X J, YAN W, ZHANG W M, et al.Current status and perspectives of 2-phenylethanol production through biological processes[J].Critical Reviews in Biotechnology, 2019, 39(2):235-248.
[2] MART NEZ-AVILA O, S
NEZ-AVILA O, S NCHEZ A, FONT X, et al.Bioprocesses for 2-phenylethanol and 2-phenylethyl acetate production:Current state and perspectives[J].Applied Microbiology and Biotechnology, 2018, 102(23):9991-10004.
NCHEZ A, FONT X, et al.Bioprocesses for 2-phenylethanol and 2-phenylethyl acetate production:Current state and perspectives[J].Applied Microbiology and Biotechnology, 2018, 102(23):9991-10004.
[3] WANG Y Q, ZHANG H, LU X Y, et al.Advances in 2-phenylethanol production from engineered microorganisms[J].Biotechnology Advances, 2019, 37(3):403-409.
[4] LUKITO B R, WU S K, SAW H J J, et al.One-pot production of natural 2-phenylethanol from L-phenylalanine via cascade biotransformations[J].ChemCatChem, 2019, 11(2):831-840.
[5] DAI J, XIA H L, YANG C L, et al.Sensing, uptake and catabolism of L-phenylalanine during 2-phenylethanol biosynthesis via the Ehrlich pathway in Saccharomyces cerevisiae[J].Frontiers in Microbiology, 2021, 12:601963.
[6] JIN D F, GU B T, XIONG D W, et al.A transcriptomic analysis of Saccharomyces cerevisiae under the stress of 2-phenylethanol[J].Current Microbiology, 2018, 75(8):1068-1076.
[7] 牛明福, 李亚恒, 陈金帅, 等.马克斯克鲁维酵母生物转化2-苯乙醇工艺优化及耐高温特性分析[J].食品与发酵工业, 2018, 44(2):15-20.
NIU M F, LI Y H, CHEN J S, et al.Optimization and characterization of 2-phenylethanol bioconversion by thermotolerant yeast Kluyveromyces marxianus[J].Food and Fermentation Industries, 2018, 44(2):15-20.
[8] MART NEZ-AVILA O, MU
NEZ-AVILA O, MU OZ-TORRERO P, S
OZ-TORRERO P, S NCHEZ A, et al.Valorization of agro-industrial wastes by producing 2-phenylethanol via solid-state fermentation:Influence of substrate selection on the process[J].Waste Management, 2021, 121:403-411.
NCHEZ A, et al.Valorization of agro-industrial wastes by producing 2-phenylethanol via solid-state fermentation:Influence of substrate selection on the process[J].Waste Management, 2021, 121:403-411.
[9] WANG Z Y, JIANG M Y, GUO X N, et al.Reconstruction of metabolic module with improved promoter strength increases the productivity of 2-phenylethanol in Saccharomyces cerevisiae[J].Microbial Cell Factories, 2018, 17(1):60.
[10] YANG C, LIU Y S, LIU W Q, et al.Designing modular cell-free systems for tunable biotransformation of L-phenylalanine to aromatic compounds[J].Frontiers in Bioengineering and Biotechnology, 2021, 9:730663.
[11] ZHANG H B, CAO M L, JIANG X L, et al.De-novo synthesis of 2-phenylethanol by Enterobacter sp.CGMCC 5087[J].BMC Biotechnology, 2014, 14:30.
[12] 龚大春, 万里, 张书银, 等.一种多酶级联制备2-PE的方法:中国, CN113355366A[P].2021-09-07.
GONG D C, WAN L, ZHANG S Y, et al.A method for preparing 2-phenylethanol by multi enzyme cascade:China, CN113355366A[P].2021-09-07.
[13] CHOI J M, HAN S S, KIM H S.Industrial applications of enzyme biocatalysis:Current status and future aspects[J].Biotechnology Advances, 2015, 33(7):1443-1454.
[14] JAKOBLINNERT A, VAN DEN WITTENBOER A, SHIVANGE A V, et al.Design of an activity and stability improved carbonyl reductase from Candida parapsilosis[J].Journal of Biotechnology, 2013, 165(1):52-62.
[15] GROSCH J H, LODERER C, JESTEL T, et al.Carbonyl reductase of Candida parapsilosis - Stability analysis and stabilization strategy[J].Journal of Molecular Catalysis B:Enzymatic, 2015, 112:45-53.
[16] SHEN W, CHEN Y, QIU S, et al.Semi-rational engineering of a Kluyveromyces lactis aldo-keto reductase KlAKR for improved catalytic efficiency towards t-butyl 6-cyano-(3R, 5R)-dihydroxyhexanoate[J].Enzyme and Microbial Technology, 2020, 132:109413.
[17] WANG Y J, YING B B, SHEN W, et al.Rational design of Kluyveromyces marxianus ZJB14056 aldo-keto reductase KmAKR to enhance diastereoselectivity and activity[J].Enzyme and Microbial Technology, 2017, 107:32-40.
[18] XIA Y Y, CUI W J, CHENG Z Y, et al.Improving the thermostability and catalytic efficiency of the subunit-fused nitrile hydratase by semi-rational engineering[J].ChemCatChem, 2018, 10(6):1370-1375.
[19] LUTZ S.Beyond directed evolution—Semi-rational protein engineering and design[J].Current Opinion in Biotechnology, 2010, 21(6):734-743.
[20] 宋婷, 王帅静, 汪沉, 等.近平滑假丝酵母ATCC 7330羰基还原酶CpCR的表达及酶学性质研究[J].食品与发酵工业, 2021, 47(3):18-24.
SONG T, WANG S J, WANG C, et al.Expression and characterization of carbonyl reductase CpCR from Candida parapsilosis ATCC 7330[J].Food and Fermentation Industries, 2021, 47(3):18-24.
[21] 盛骏桢. Anti-prelog规则羰基还原酶产生菌筛选及其在他汀侧链合成中的应用[D].杭州:浙江工业大学, 2013.
SHENG J Z.Microbial screening for carbonyl reductase following the anti-prelog’s rule and its application in the synthesis of statin side chain[D].Hangzhou:Zhejiang University of Technology, 2013.
[22] LUO X, WANG Y J, SHEN W, et al.Activity improvement of a Kluyveromyces lactis aldo-keto reductase KlAKR via rational design[J].Journal of Biotechnology, 2016, 224:20-26.
[23] ZHAO J, FRAUENKRON-MACHEDJOU V J, KARDASHLIEV T, et al.Amino acid substitutions in random mutagenesis libraries:Lessons from analyzing 3000 mutations[J].Applied Microbiology and Biotechnology, 2017, 101(8):3177-3187.
[24] 周婕妤. 双芳基醇脱氢酶立体选择性的改造及机制研究[D].无锡:江南大学, 2018.
ZHOU J Y.Modulating stereoselectivity of diaryl alcohol dehydrogenase:Engineering and mechanism studies[D].Wuxi:Jiangnan University, 2018.
[25] SU B M, SHAO Z H, LI A P, et al.Rational design of dehydrogenase/reductases based on comparative structural analysis of prereaction-state and free-state simulations for efficient asymmetric reduction of bulky aryl ketones[J].ACS Catalysis, 2020, 10(1):864-876.
[26] HAMNEVIK E, MAURER D, ENUGALA T R, et al.Directed evolution of alcohol dehydrogenase for improved stereoselective redox transformations of 1-phenylethane-1, 2-diol and its corresponding acyloin[J].Biochemistry, 2018, 57(7):1059-1062.