食品生产加工、储藏等过程中,部分食源性致病菌可在不锈钢、塑料、玻璃等食品接触表面生成胞外聚合物(extracellular polymeric substances,EPS),形成呈三维膜状结构特征的生物被膜[1-2]。在由多糖、蛋白质、核酸、脂质等生物大分子组成的EPS保护下,生物被膜状态的菌群更难以被清除,且会通过释放菌体,加剧食源性致病菌的传播[3]。因此,生物被膜被认为是食品安全的重大威胁之一,需要在食品生产供应链中持续监测[4]。
生物被膜的检测具有挑战。传统定性检测主要采用结晶紫、刚果红、银染等染色方法,但样品处理过程复杂,可重复性差,误判率高[5-6];定量检测则常用微生物培养计数法,但培养周期长,无法快速获得检测结果[7-9]。随着光学技术、电化学传感技术等发展迅速,其也被初步应用于生物被膜的快速可视化检测。例如,光学、力学、电子显微镜技术可用于观测生物被膜的三维结构和分布,但难以定量生物被膜及其组成[10];核磁共振方法可获取生物被膜基质中物质结构信息,但对样本制备和数据分析要求高[11];光谱方法理论上可实现对生物被膜的实时、无损、原位定量分析,但常见的红外光谱方法会因—OH抑制生物被膜组分的特征带,水信号过强,信噪比较低[12]。近年来,拉曼光谱技术逐渐被应用于生物被膜的检测中,其通过光的非弹性散射,无探针原位分析识别化学特征,避免了上述技术的缺点。光谱中的每个拉曼峰都是特定分子键的特征,可作为每个生化成分特定的“指纹”,易于对包含各种分子和分子基团(如蛋白质、碳水化合物、脂质和核酸)的复杂结构进行详细分析,且拉曼峰的偏移不受水的影响[13-14]。
基于此,本文从拉曼光谱的技术原理出发,介绍了拉曼光谱采集生物被膜分子信息的技术和数据分析方法,归纳了拉曼光谱可识别的生物被膜特征物质及对应关系,并总结了该技术在检测细菌黏附、生物被膜形成、信号分子调控等过程中的应用研究进展,最后讨论了通过结合其他技术,进一步提升拉曼光谱获取生物被膜特征能力的可行性。
1 拉曼光谱原理与主要技术
1.1 拉曼光谱原理
拉曼光谱是一种基于拉曼散射效应的光谱分析技术[15]。当样品暴露在可见单色光下时,样品会吸收光线,大部分光会透过样本,小部分光会被样品散射到各个方向。当特定频率的入射光与其散射光频率一致时,则被称为瑞利散射。由于该过程没有发生能量交换,也被称为弹射散射。总散射光中大约有1%,其频率和入射光不同,这部分称为拉曼散射或非弹性散射。拉曼散射又分为斯托克斯散射和反斯托克斯散射,二者均匀的分布于瑞利散射的两侧。当分子跳回到电子振动基态的较高能级时,入射光的频率大于散射光,光子发生红移,称为斯托克斯散射;当分子跳回到基态能级时,入射光频率小于散射光频率,激光光子发生蓝移,则称为反斯托克斯散射。瑞利散射和拉曼散射的能级图如图1所示。由于散射光子的频率取决于分子键的类型,因此,每个分子的拉曼光谱是唯一的,可以作为该分子的指纹谱。
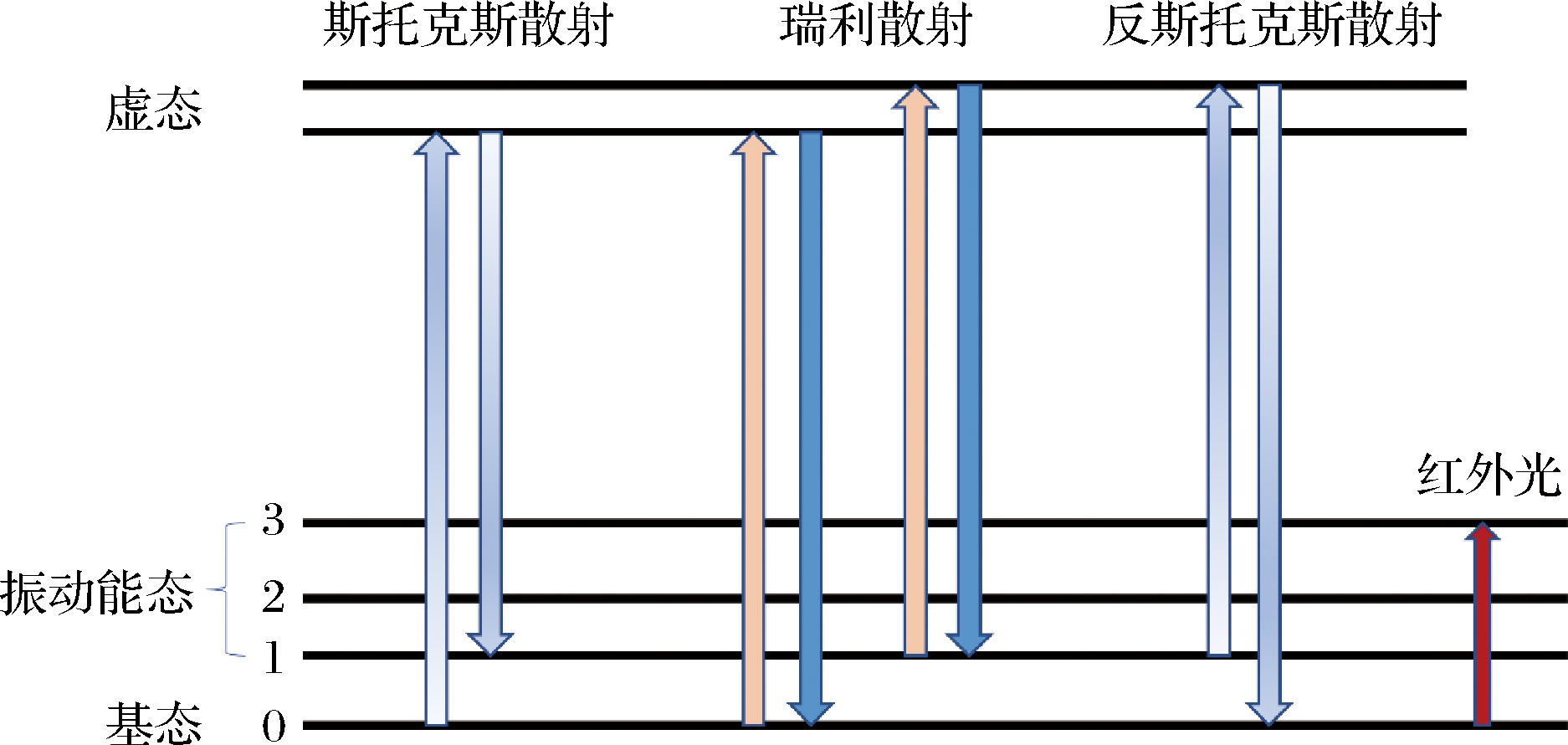
图1 瑞利散射与拉曼散射的能级图
Fig.1 Energy level diagram of Rayleigh scattering and Raman scattering
1.2 获取生物被膜拉曼光谱的主要技术
1.2.1 显微共聚焦拉曼光谱
由于生物被膜的尺度在微米至纳米水平,因此传统拉曼光谱的检测分辨率无法满足相关检测的要求。如图2所示,显微共聚焦拉曼光谱(confocal Raman microscopy,CRM)将拉曼光谱与激光扫描共聚焦显微镜(confocal laser scanning microscopy,CLSM)集成,可为微生物研究提供纳米级的空间分辨力[16]。拉曼显微光谱最初常用于微生物培养物的单细胞分析[17],VIRDIS等[18]则首次实验证明CRM可以用于监测生物被膜的形成过程,且不会影响其结构或代谢活动。对不同种类生物被膜的研究表明,CRM可以将生物被膜内的各种结构外观与其化学成分的变化相关联,并推断生物被膜基质中复杂组成物质的化学信息。
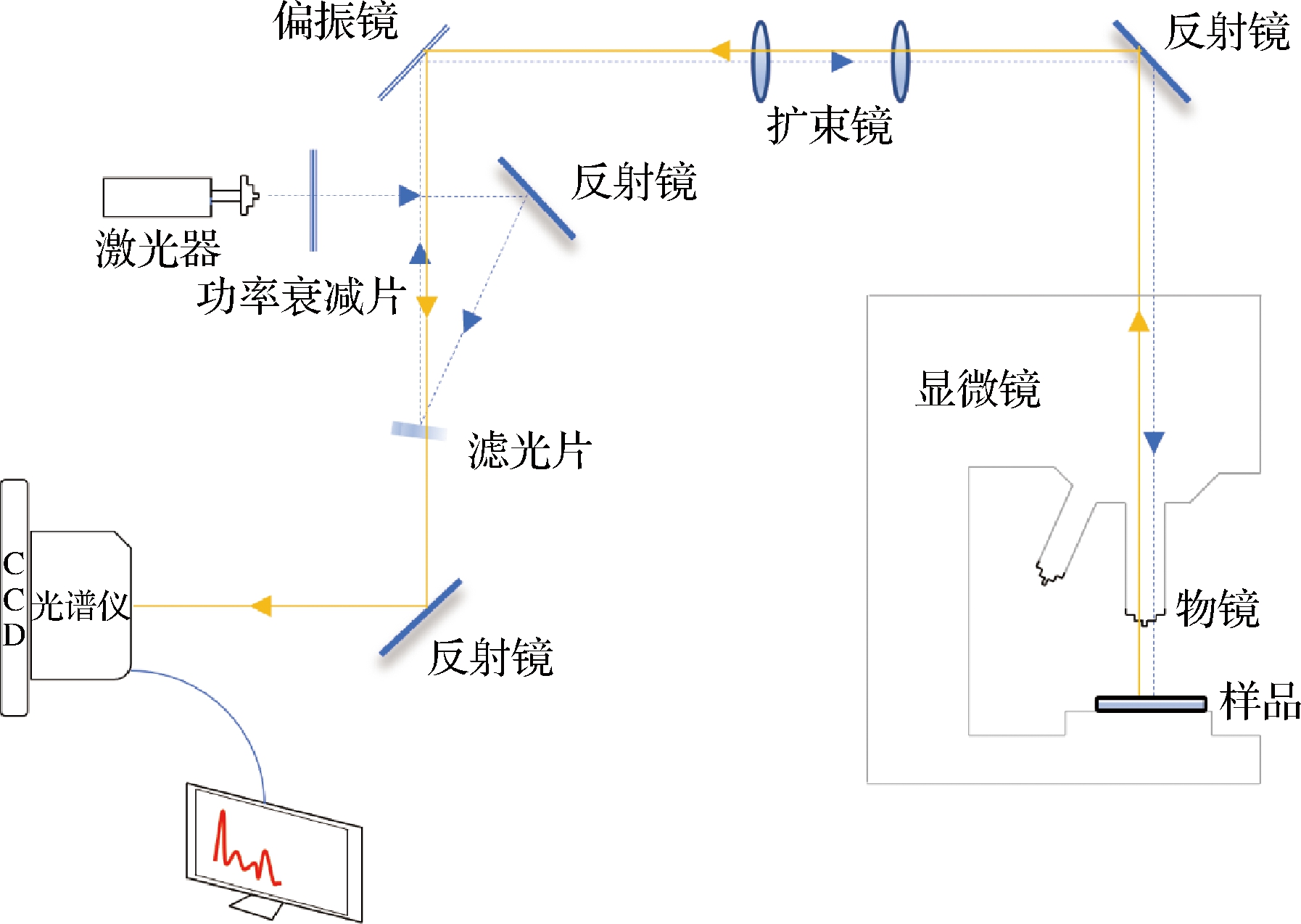
图2 显微共聚焦拉曼光谱结构图
Fig.2 Structure diagram of confocal Raman microscopy
1.2.2 表面增强拉曼散射
在利用拉曼光谱技术开展生物被膜检测过程中,常发生信号强度过低导致检测失败的情况,表面增强拉曼散射(surface-enhanced Raman scattering,SERS)技术的开发则很好地解决了这一问题。SERS是一种依靠纳米结构金属在光激发下产生的大电场来增强金属附近分子对光子的非弹性散射的技术。研究发现,使用贵金属纳米结构可以大大增强微弱的拉曼散射信号,并可通过电磁增强和化学增强机制进行解释[19]。IVLEVA等[20]率先研发了应用SERS检测生物被膜的方法,其使用盐酸羟胺还原银纳米颗粒(nano particles,NPs)作为SERS基底,将生物被膜包裹的载玻片浸入银胶体悬浮液后,进行SERS测量。随着技术优化,目前主要有2种方法制备SERS实验用生物被膜样品[21-22]:一是将生物被膜与金、银为主的金属胶体或离子溶液简单混合,然后将溶液滴注到固体基底上待测,但该方法的局限性在于拉曼信号增强仅限于胶体附近的生物被膜表面,结果的可重复性和稳定性均较差;二是将浮游菌体附着于NPs修饰过的表面形成生物被膜,该方法得到的光谱图重复性更高,数据结果更稳定,因此也是目前的主流方法[13]。
SERS为检测生物分子信号开辟了新的可能性,但目前SERS基底制备缺少标准化操作,SERS基底的化学性差异直接影响不同物质的准确鉴定。同时,SERS光谱采集中,一些金属(尤其是银)对微生物具有一定抑制作用,长时间的实验和高浓度的NPs,可能导致活菌数量显著减少,影响检测结果[23]。因此,应根据细菌种类和环境条件,进一步优化设置基底制备步骤和细菌-NPs浓度比,降低检测误差,提高SERS检测生物被膜的鲁棒性。
1.3 分析生物被膜拉曼光谱数据的方法
由于微生物样品中蛋白质、碳水化合物、核酸、脂质等化学键的分子振动所获得的光谱通常非常相似,这使得拉曼光谱数据分析成了一个难点。为了降低冗余数据的影响,一般先对光谱图进行基线扣除、平滑降噪、消除宇宙射线、归一化等预处理[24]。预处理后的数据,进一步采用化学计量学方法实现生物被膜化学组成的分析。
在微生物化学计量分析中,通常分为有监督和无监督2种基本方法。根据定性或定量的任务需求,监督方法又分为分类和回归两类模型。监督方法需要样本的先验知识,例如偏最小二乘回归(partial least-squares regression, PLSR)、支持向量机(support vector machine, SVM)判别分析、线性判别分析(linear discriminant analysis, LDA)等。SHEN等[25]建立了一种基于纤维探针拉曼光谱的细菌生物被膜分类方法,利用大肠杆菌等5种细菌建立主成分分析(principal component analysis,PCA)-LDA模型,对未知细菌生物被膜的识别率最高可达97.5%。KUSI 等[26]利用拉曼显微光谱结合SVM,对自来水中作为浮游细胞和生物被膜生长的9种细菌(如铜绿假单胞菌等)进行鉴别测定。EBERT等[27]将47例不同疾病背景患者的临床金黄色葡萄球菌的拉曼光谱图与其生物被膜形成特征相关联。对生物被膜形成的拉曼光谱进行PLSR分析,可以获得拉曼光谱与生物被膜吸光度值的线性相关性。PLSR负载系数突出了高生物被膜量和低生物被膜量之间代表核酸、碳水化合物和蛋白质的拉曼谱带的光谱差异;无监督分类方法不需要样本的先验知识,如PCA和层次聚类分析(hierarchical cluster analysis, HCA)。由于PCA具有去除冗余重叠光谱信息的能力,因此是用于区分不同生物被膜EPS成分差异的最常用的有力工具。TAHIR等[14]用拉曼光谱结合PCA表征和比较9种细菌生物被膜分泌出的2种不同种类的胞外多糖:葡聚糖和果聚糖。DU等[28]采集了不同耐干燥水平的阪崎克罗诺杆菌菌株形成的生物被膜的拉曼光谱图,PCA结果发现不同耐干燥水平的菌株主要成分存在差异,证明了多糖、蛋白质和类胡萝卜素的水平可能在菌体耐干燥环境中起重要作用。CHOO-SMITH等[29]对不同位置、深度的大肠杆菌和金黄色葡萄球菌生物被膜光谱进行HCA,发现菌落中存在不同的层,进一步的研究表明,从菌落表面获得的光谱显示出比菌落深层更高的糖原水平,来自较深层的光谱具有比表面层更高的RNA水平。
等[26]利用拉曼显微光谱结合SVM,对自来水中作为浮游细胞和生物被膜生长的9种细菌(如铜绿假单胞菌等)进行鉴别测定。EBERT等[27]将47例不同疾病背景患者的临床金黄色葡萄球菌的拉曼光谱图与其生物被膜形成特征相关联。对生物被膜形成的拉曼光谱进行PLSR分析,可以获得拉曼光谱与生物被膜吸光度值的线性相关性。PLSR负载系数突出了高生物被膜量和低生物被膜量之间代表核酸、碳水化合物和蛋白质的拉曼谱带的光谱差异;无监督分类方法不需要样本的先验知识,如PCA和层次聚类分析(hierarchical cluster analysis, HCA)。由于PCA具有去除冗余重叠光谱信息的能力,因此是用于区分不同生物被膜EPS成分差异的最常用的有力工具。TAHIR等[14]用拉曼光谱结合PCA表征和比较9种细菌生物被膜分泌出的2种不同种类的胞外多糖:葡聚糖和果聚糖。DU等[28]采集了不同耐干燥水平的阪崎克罗诺杆菌菌株形成的生物被膜的拉曼光谱图,PCA结果发现不同耐干燥水平的菌株主要成分存在差异,证明了多糖、蛋白质和类胡萝卜素的水平可能在菌体耐干燥环境中起重要作用。CHOO-SMITH等[29]对不同位置、深度的大肠杆菌和金黄色葡萄球菌生物被膜光谱进行HCA,发现菌落中存在不同的层,进一步的研究表明,从菌落表面获得的光谱显示出比菌落深层更高的糖原水平,来自较深层的光谱具有比表面层更高的RNA水平。
化学计量学模型建立后还需对模型的准确率进行验证,验证的主要方法可分为交叉验证(又称内部验证)和独立验证(又称外部验证)。交叉验证使用建立模型的光谱数据测试模型预测能力,例如K-fold交叉验证(K-fold Cross-validation)方法将数据集随机划分为K个子集,训练模型时随机选择K-n份作为训练集,剩下的n份作为测试集,是目前最常用的交叉验证技术之一[30]。交叉验证常用于检验模型的可重复性,可防止模型过拟合,还可用于插补数据缺失值;独立验证根据未用于构建模型的数据评估模型的性能,主要用于考察模型的可迁移性和泛化能力,其结果对于将该模型推向实际应用至关重要。
2 生物被膜研究中的拉曼光谱应用
在已实现的拉曼光谱生物被膜研究与应用中,主要涉及生物被膜基质组成的快速、无损检测,浮游菌体与生物被膜的差异比较,生物被膜形成和代谢过程的物质变化监测,以及生物被膜的抗逆机理探究等方面。
2.1 生物被膜基质组成的检测
食品中常见的沙门氏菌、金黄色葡萄球菌、单核细胞增生李斯特菌、副溶血性弧菌、铜绿假单胞菌、阪崎克罗诺杆菌等食源性致病菌均能在一定环境条件下形成生物被膜。与浮游菌体相比,生物被膜的拉曼谱带特征峰存在差异,通过关联不同特征峰对应的官能团,如糖类、蛋白质、核酸、脂质等,即可用于识别细菌的生物被膜。表1概括了常见食源性致病菌生物被膜拉曼特征峰与其基质内主要生物大分子的对应关系。需要注意的是拉曼光谱和SERS光谱特征反映了相似的生化性质,但SERS的增强效应可能导致谱带的偏移,两者并非完全一致。
表1 食源性致病菌生物被膜基质的代表性拉曼特征峰
Table 1 Representative Raman characteristic peaks of biofilm substrates of foodborne pathogens

食源性致病菌光谱类型特征峰/(cm-1)物质结构(官能团)峰属参考文献沙门氏菌拉曼光谱1 520CC 拉伸糖类1 330酰胺III蛋白质1 030-糖类875C—C拉伸、C—O—C、1,4糖苷键糖类[16,31]铜绿假单胞杆菌SERS光谱1 600酰胺I蛋白质1 567-核酸1 562酰胺II蛋白质1 485腺嘌呤核酸1 405COO-糖类1 355,1 085,1 181绿脓素绿脓素[32]
续表1
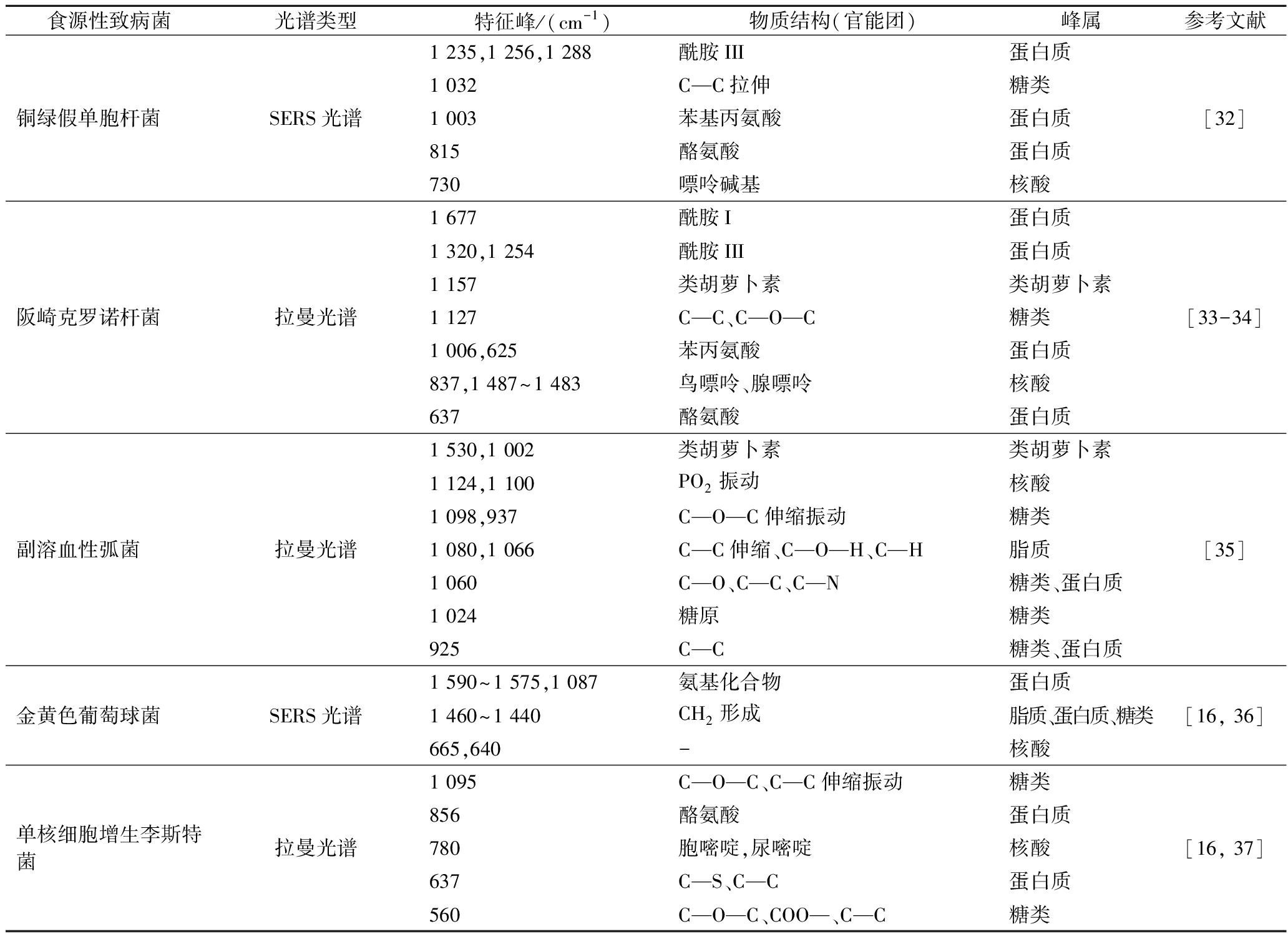
食源性致病菌光谱类型特征峰/(cm-1)物质结构(官能团)峰属参考文献铜绿假单胞杆菌SERS光谱1 235,1 256,1 288酰胺III蛋白质1 032C—C拉伸糖类1 003苯基丙氨酸蛋白质815酪氨酸蛋白质730嘌呤碱基核酸[32]阪崎克罗诺杆菌拉曼光谱1 677酰胺I蛋白质1 320,1 254酰胺III蛋白质1 157类胡萝卜素类胡萝卜素1 127C—C、C—O—C糖类1 006,625苯丙氨酸蛋白质837,1 487~1 483鸟嘌呤、腺嘌呤核酸637酪氨酸蛋白质[33-34]副溶血性弧菌拉曼光谱1 530,1 002类胡萝卜素类胡萝卜素1 124,1 100PO2振动核酸1 098,937C—O—C伸缩振动糖类1 080,1 066C—C伸缩、C—O—H、C—H脂质1 060C—O、C—C、C—N糖类、蛋白质1 024糖原糖类925C—C糖类、蛋白质[35]金黄色葡萄球菌SERS光谱1 590~1 575,1 087氨基化合物蛋白质1 460~1 440CH2形成脂质、蛋白质、糖类665,640-核酸[16, 36]单核细胞增生李斯特菌拉曼光谱1 095C—O—C、C—C伸缩振动糖类856酪氨酸蛋白质780胞嘧啶,尿嘧啶核酸637C—S、C—C蛋白质560C—O—C、COO—、C—C糖类[16, 37]
注:-为未知物质结构(官能团)。
2.2 生物被膜形成过程的检测
目前,一般认为细菌生物被膜的形成有5个阶段的动态过程:菌体附着于介质表面;细菌分泌EPS;微菌落形成;微菌落发展成成熟的生物被膜;生物被膜中浮游态细菌分散到周围环境中[38-39]。由于拉曼光谱特征可用于识别某一时间点的生物被膜基质中生物分子及含量,因此通过分析EPS拉曼特征的动态变化,理论上可以对应确认细菌生物被膜的代谢活动及其形成过程阶段,表2总结了部分食源性致病菌生物被膜形成的各阶段拉曼特征峰的变化。
表2 食源性致病菌生物被膜形成各阶段拉曼特征峰变化
Table 2 Variation of Raman characteristic peaks in each stage of biofilm formation of foodborne pathogens

食源性致病菌光谱类型特征峰/(cm-1)物质结构(官能团)备注参考文献沙门氏菌拉曼光谱531~542,1 096C—O—C、S—Sa-b↑ b-d↓863~899C—C、C—O—C、1,4糖苷键a-c↑ c-d↓1 020~1 085C—C、C—O—Ca-c↑ c-d↓1 314~1 345酰胺Ⅲa-c↑ c-d↓1 500~1 519CCa-c↑ c-d↓1 677,1 688,1 700CC、CO、酰胺Ⅰa-c↑ c-d↓[31]铜绿假单胞菌SERS光谱1 003绿脓素b-c↓1 032C—Cb-c↓1 355绿脓素a-c↑1 405COO-a-c↑1 485腺嘌呤a-c↑1 562酰胺Ⅱa-c↑1 600,1 618酰胺Ⅰa-c↑[40]
续表2

食源性致病菌光谱类型特征峰/(cm-1)物质结构(官能团)备注参考文献副溶血性弧菌拉曼光谱925C—Ca-d ↑937,1 098C—O—Ca-c↑ c-d↓1 024糖原a-c↑ c-d↓1 060C—O—C、C—C、C—Na-c↑ c-d↓1 080C—C、C—O—H、C—Ha-c↑ c-d↓1 100,1 124PO2a-c↑ c-d↓[35]金黄色葡萄球菌SERS光谱665,640核酸a-c↑1 460~1 440C—Ca-c↑1 590~1 575,1 087—NH2a-c↑[36]
注:a为生物被膜黏附期;b为微菌落形成期;c为成熟生物被膜期;d为生物被膜分散期;↑为波峰升高;↓为波峰下降。
TAN等[41]利用拉曼光谱监测了副溶血性弧菌生物被膜的形成,发现培养48 h后,生物被膜形成致密结构,拉曼谱图对应EPS中的碳水化合物和核酸含量显著上升,这一EPS拉曼谱图特征的变化同样被发现于金黄色葡萄球菌和大肠杆菌生物被膜的形成过程[26]。CHEN等[37]对食源性致病菌生物被膜中EPS利用超声探针法进行提取,研究了副溶血性弧菌和单增李斯特菌形成的单一和混合物种生物被膜的特征的动态变化,发现混合物种生物被膜中EPS的碳水化合物、蛋白质、核酸所对应的拉曼峰均有所下降。再通过苯酚-硫酸法[42]和Lowry法[43]对碳水化合物和蛋白质的含量进行量化,证实混合物种生物被膜与表面的黏附会降低,胞外多糖和蛋白质的浓度降低。通过微生物分析与拉曼光谱谱图特征相结合,为拉曼光谱对EPS的定量分析提供了理论依据。该团队还研究了副溶血性弧菌在玻璃材料表面形成的生物被膜EPS的化学成分,首次发现在副溶血性弧菌生物被膜的形成过程中类胡萝卜素起协助作用[35]。
生物被膜形成过程中,包括碳水化合物、脂质、蛋白质和核酸等在内的代谢产物和生物大分子含量通常是逐渐增加的。因此,在生物被膜初期,大多数细菌和EPS化合物的拉曼信号比较微弱,对方法的检测限要求较高。LIU等[36]报道了一种快速且新颖的SERS检测方法,用于研究金黄色葡萄球菌生物被膜的形成。通过使用带有金纳米粒子的便携式拉曼光谱仪收集结晶紫染色生物被膜的拉曼光谱,发现测量的拉曼峰强度与生物被膜的数量呈良好的线性关系。NGUYEN等[44]自制等离子体纳米间隙增强了拉曼散射信号,绿脓素检测限达到了10 ng/mL,能够检测铜绿假单胞菌早期生物被膜的形成过程。
在对细胞间群体感应(quorum sensing,QS)过程的信号分子检测方面,SERS由于具备识别金属阳离子的高灵敏度特性,也体现出了良好的效率和性能[45-46]。QS是一种细胞间通信现象,细菌通过分泌和感应特定的信号分子来监测和响应自身所处的环境和种群密度,从而在细菌种群范围内同步调节基因表达[47]。目前,传统检测生物被膜QS的手段主要是针对代谢物的同位素标记法和荧光法,但两者都需繁琐的预处理步骤。SERS由于具备识别金属阳离子的高灵敏度特性,体现出了良好的效率和性能。GUO等[48]在2片六角形氮化硼层之间封装金纳米星,开发了一种具有三明治结构的柔性黏滞便笺,该黏滞便笺可以黏贴在细菌生物被膜上,通过高灵敏度的SERS成像可实时监测分泌的信号分子。绿脓素是铜绿假单胞杆菌的一种QS信号分子,在生物被膜形成过程中起着重要作用。BODEL N等[49]以聚N-异丙基丙烯酰胺修饰的金纳米粒子(Au@pNIPAM)、二氧化钛修饰的金纳米粒子(Au@TiO2)、二氧化硅修饰的金纳米粒子(Au@SiO2)作为基底,得到绿脓素在550~900 nm段有明显特征的SERS谱图,3种基底分别对应检测限为10-10、10-9、10-14 mol/L。酰化高丝氨酸内酯类(acyl-homoserine lactones,AHLs)为革兰氏阴性细菌QS的一类重要信号分子,研究发现AHLs的SERS谱图主要由酰氨分子谱峰组成,其中酰胺I、酰胺II、酰胺III和酰胺IV的峰分别位于1 648、1 570、1 308、771 cm-1处[50]。
N等[49]以聚N-异丙基丙烯酰胺修饰的金纳米粒子(Au@pNIPAM)、二氧化钛修饰的金纳米粒子(Au@TiO2)、二氧化硅修饰的金纳米粒子(Au@SiO2)作为基底,得到绿脓素在550~900 nm段有明显特征的SERS谱图,3种基底分别对应检测限为10-10、10-9、10-14 mol/L。酰化高丝氨酸内酯类(acyl-homoserine lactones,AHLs)为革兰氏阴性细菌QS的一类重要信号分子,研究发现AHLs的SERS谱图主要由酰氨分子谱峰组成,其中酰胺I、酰胺II、酰胺III和酰胺IV的峰分别位于1 648、1 570、1 308、771 cm-1处[50]。
此外,也有研究表明SERS可以实现革兰氏阳性细菌和革兰氏阴性细菌EPS的定量检测[51-52]。然而,生物被膜样品的拉曼光谱易受到荧光强干扰的影响,所以需要对激发光波长、采集时间、物镜放大倍数、聚焦模式等测量参数不断优化。未来可从拉曼峰强度与面积的角度,进一步完善基于拉曼光谱的生物被膜的定量分析。
2.3 生物被膜逆环境过程的检测
生物被膜作为食品、医疗等领域的重大威胁之一,通常需要通过探究生物被膜的抗逆机理,针对性地开发有效抑制消杀措施。通常,抑制消杀措施会以降解DNA、蛋白质或改变官能团构象等方式导致生物被膜EPS化学成分被破坏,进而彻底清除生物被膜。因此,可通过建立处理条件与生物被膜损伤引起的拉曼光谱变化间的函数关系,实现基于拉曼光谱方法的生物被膜抗逆过程与机理分析。
在评价抗菌剂有效性方面,拉曼光谱可通过检测生物被膜中与DNA相对应的拉曼谱带,追踪抑菌与杀菌作用的机制[53-54]。HAN等[55]评估了酸性电解水去除食源性病原体单增李斯特菌和副溶血性弧菌生物被膜的能力,并通过拉曼光谱中碳水化合物C—O—C键以及酪氨酸和苯丙氨酸中芳香环的变形证明酸性电解水可以引发EPS被破坏。CHEN等[56]建立了一种高效的蓝光LED光动力灭活技术(photodynamic inactivation,PDI),用光敏剂姜黄素介导PDI根除副溶血性弧菌及其生物被膜,并通过拉曼光谱观测出PDI处理后显著降低了生物被膜EPS中蛋白质、总碳水化合物和eDNA的含量,该结果同PRABHAWATHI等[57]对功能化聚己内酰胺作用大肠杆菌生物被膜的抗生物被膜活性和生物被膜形成的研究相符。
在评价抗生素有效性方面,传统的细菌耐药性检测方法通常需要在与不同抗生素孵育期间监测细菌的生长情况,待确定目标病原体后,还需要额外18~24 h来进行药敏试验(antimicrobial susceptibility testing,AST)[58]。LU等[59]利用拉曼光谱对比分析环丙沙星、红霉素等抗生素抑制空肠弯曲菌生物被膜后波峰的变化,缩短了检测时间,并通过交叉验证评价建立了活细胞存活数的PLSR预测模型。JUNG等[60]应用拉曼光谱的基于判别模型的PCA和SVM能够以100%的准确率特异性分离抗生素治疗生物被膜后产生的光谱。对于敏感菌株,由于药物作用,细菌代谢过程受到干扰,细菌停止生长;对于耐药菌株,与未治疗的对照组相比,添加药物可能没有效果,或者由于诱导的抗生素耐药性,可能导致细菌生化特征变化。通过多元统计数据分析,可以从拉曼光谱中识别药物引起的生化变化,快速准确获得AST结果,此过程进行2~3 h即可。此外,通过分类模型或计算光谱标记带的比率对区可快速分敏感性菌株和耐药性菌株。例如,NAKAR等[61]发现大肠杆菌耐药菌株具有较高的核酸与蛋白质比率,结合机器学习模型准确区分耐药菌株与敏感菌株。
3 拉曼光谱结合其他技术检测生物被膜
为了更准确、稳定地获得生物被膜组成、结构和功能信息,还常采用拉曼光谱结合2种或多种不同的样品制备、检测、分析技术,对食源性致病菌生物被膜进行探究。
3.1 结合光谱技术
红外光谱和拉曼光谱作为2种互补的分子振动技术,结合后可共同描述生物被膜的固有化学信息[10, 62]。红外光谱可检测到改变分子永久偶极矩的振动,拉曼光谱则可在光谱中看到改变极化率的振动,红外光谱与拉曼光谱所涉及的分子振动分别通过光的吸收或非弹性散射激发。WANG等[31]通过将傅里叶变换衰减全反射红外光谱(attenuated total reflectance Fourier transform infrared spectroscopy,ATR-FTIR)和拉曼光谱结合对不锈钢表面培养的沙门氏菌生物被膜进行分析,揭示了沙门氏菌生物被膜和相应的浮游细胞之间完全不同的化学成分,以及沙门氏菌在不同营养条件下生物被膜形成之间的一些重要差异。SHARMA等[33]从FTIR技术和拉曼光谱技术获得了阪崎克罗诺杆菌全细胞成分光谱,比较了阪崎克罗诺杆菌的生物被膜细胞与浮游细胞的成分差异。
3.2 结合显微镜技术
原子力显微镜(atomic force microscope,AFM)和拉曼光谱相结合,不获得了超高分辨率,还避免了弱拉曼光谱信号所带来的困扰。AFM配备导电金属(如镀金或镀银纳米颗粒)的针尖,使其具有SERS活性。PRADHAN等[63]使用拉曼光谱揭示了微生物细胞和EPS的不同化学成分,而AFM图像清晰显示了EPS的分泌和包裹在EPS基质内的细胞,两者相结合提供了生物被膜的较为完整的成分结构信息。NIELSEN等[64]研究精油异丁香酚涂层是否可避免细菌生物被膜通过表面转移的问题,并通过拉曼光谱、AFM相结合,可视化了金黄色葡萄球菌、单增李斯特菌在不锈钢和聚乙烯表面的定殖。由于拉曼光谱检测设备稳定可靠,标准和模块化部件数量有限,因此拉曼光谱和AFM的结合相对较易。
同时,CLSM可通过荧光染色、图像分析等研究手段量化细菌生物被膜的结构特征,如:生物被膜厚度、粗糙度、孔隙度、体积覆盖率、分形维数等。但有研究表面EPS中某些分子的荧光染色特异性较低,未能被CLSM灵敏检测[65-66]。结合拉曼光谱的分子信息后,可互补实现对生物被膜基质的全面了解。例如,WAGNER等[67]将拉曼光谱技术与CLSM技术结合,同时获得了生物被膜基质内不同物质的化学信息、空间结构和深度分布信息。
3.3 结合筛选标记技术
SERS与显微镜、稳定同位素标记、微流控分析等其他工具的灵活集成,也已取得了一定进展。利用由同位素标记样品与未标记样品的拉曼光谱的差谱分析研究标记位置的分子结构和相互作用的方法,它可以补充其他分子生物学技术,用于在单细胞水平上原位研究细菌在生物被膜中的代谢过程[68]。例如,SERS与15N稳定同位素探针相结合,可以在单细胞水平上区分微生物聚集体中的氮转化,这为探索生物脱氮过程中的硝化、反硝化和厌氧氨氧化提供了一种方法[69]。MUHAMADALI等[70]首次将拉曼光谱和FTIR作为代谢指纹分析方法与稳定同位素相结合,对大肠杆菌细胞进行定量和区分,不同同位素掺入后的特定光谱位移产生了独特的聚类模式,这与介质中同位素标记含量的比率直接相关。
此外,在传统生物被膜形成过程研究中,通常局限于低剪切力和静态培养的环境条件,无法满足模拟真实动态环境的需要。对此,研究者还可结合高可重复性和高通量的微流控技术,精确控制营养和流动条件,实现研究生物被膜在不同剪切力与动态营养作用下的结构和细胞信号变化,且具有的特点。FENG等[71]通过在微流控平台上培养铜绿假单胞菌生物被膜,并使用CRM以无损和连续的方式对不同发育阶段的铜绿假单胞菌生物被膜原位表征。同时,微流控平台产生离散或连续的药物梯度,再与拉曼光谱技术相结合,也可大大缩短药敏检测时间。HUANG等[72]结合微流控技术、SERS构建了AST模型,分别对敏感、耐药的大肠杆菌和金黄色葡萄球菌进行测试,得到的AST结果与用肉汤稀释法培养的实验一致,整个实验过程仅需2 h。
4 总结与展望
拉曼光谱是一种快速、高效的检测技术,其应用于食源性致病菌生物被膜的研究表明:拉曼光谱的特征峰可用于表征生物被膜基质中的代表性化学物质,其特征峰的变化可用于识别生物被膜的形成阶段。同时,EPS、QS信号分子等拉曼谱带的改变,还可反映生物被膜在严苛环境中的物质变化与生存状态,并用于推断生物被膜的抗逆机理。在拉曼光谱技术的基础上,还可通过融入互补技术,获得更准确、稳定、全面的生物被膜信息。随着拉曼仪器的小型化、自动化、高通量化,及机器学习分析技术的成熟,将进一步促进该技术在细菌生物被膜中的应用,但目前仍存在以下突出问题:
(1)现有研究多基于实验室理想条件,尚缺少针对真实复杂食品场景的标准化样品取样与拉曼光谱预处理方法,以避免非目标待测物质的干扰;
(2)在多种微生物共存的生物被膜中,其基质的化学组成和结构也具有高度异质性,可能造成拉曼光谱特征峰差异;
(3)部分拉曼特征峰与对应的物质结构(官能团)关系尚不明确,且缺少可供参考的标准拉曼峰谱比对数据库;
(4)尽管拉曼光谱方法已初步实现了生物被膜的快速检测,但仍未有合适模型可用于定量预测评估其群体动态变化和可能造成的健康风险。
综上,未来可从以下方面开展深入探究:
其一,优化拉曼光谱待测样品的获取和预处理,通过结合微流控技术,实现待测生物被膜的高通量分选与收集,降低杂质干扰;
其二,结合生物信息学和分类模型,对不同亚型的生物被膜基质光谱差异进行比较探究,实现对高异质生物被膜群体的识别与分析;
其三,通过大量积累拉曼光谱、SERS检测未知物质的特征谱图,完善食源性致病菌的拉曼光谱数据库,构建标准峰谱比对库;
其四,引入预测微生物学方法,建立基于拉曼光谱的生物被膜动力学预测模型,为进一步考虑生物被膜的微生物定量风险评估提供基础。
[1] 程传松, 闫晓彤, 李长城, 等.食品接触表面食源性致病菌生物膜形成及控制研究进展[J].包装工程, 2021, 42(13):1-8.
CHENG C S, YAN X T, LI C C, et al.Research progress in biofilm formation and control of foodborne pathogens on food contact surfaces[J].Packaging Engineering, 2021, 42(13):1-8.
[2] 胡丽丽, 董庆利, 夏阳, 等.单增李斯特菌生物膜形成及其调控机制研究进展[J].食品与发酵工业, 2021, 47(8):276-282.
HU L L, DONG Q L, XIA Y, et al.Advances on the formation and regulation mechanism of Listeria monocytogenes biofilm[J].Food and Fermentation Industries, 2021, 47(8):276-282.
[3] VU B, CHEN M, CRAWFORD R J, et al.Bacterial extracellular polysaccharides involved in biofilm formation[J].Molecules, 2009, 14(7):2535-2554.
[4] ABDALLAH M, BENOLIEL C, DRIDER D, et al.Biofilm formation and persistence on abiotic surfaces in the context of food and medical environments[J].Archives of Microbiology, 2014, 196(7):453-472.
[5] 张君怡, 王静怡, 巴巨伟, 等.细菌生物被膜检测与清除方法研究进展[J].食品研究与开发, 2020, 41(20):218-224.
ZHANG J Y, WANG J Y, BA J W, et al.Development on detection and removal of bacterial biofilm[J].Food Research and Development, 2020, 41(20):218-224.
[6] 陈春艳, 李水红, 朱翠明.细菌生物被膜检测方法的研究进展[J].微生物学免疫学进展, 2017, 45(3):51-56.
CHEN C Y, LI S H, ZHU C M.Advances of detective methods for bacterial biofilm[J].Progress in Microbiology and Immunology, 2017, 45(3):51-56.
[7] ACHINAS S, YSKA S K, CHARALAMPOGIANNIS N, et al.A technological understanding of biofilm detection techniques:A review[J].Materials, 2020, 13(14):3147.
[8] 刘宝宝, 汪洋, 易力, 等.细菌生物被膜检测与分析方法[J].微生物学通报, 2018, 45(10):2263-2270.
LIU B B, WANG Y, YI L, et al.Detection and analysis methods of bacterial biofilm[J].Microbiology China, 2018, 45(10):2263-2270.
[9] MERINO L, PROCURA F, TREJO F M, et al.Biofilm formation by Salmonella sp.in the poultry industry:Detection, control and eradication strategies[J].Food Research International, 2019, 119:530-540.
[10] PU H, XU Y, SUN D W, et al.Optical nanosensors for biofilm detection in the food industry:Principles, applications and challenges[J].Critical Reviews in Food Science and Nutrition, 2021, 61(13):2107-2124.
[11] FYSUN O, ANZMANN T, KLEESATTEL A, et al.Detection of P.polymyxa biofilm, dairy biofouling and CIP-cleaning agents using low-field NMR[J].European Food Research and Technology, 2019, 245(8):1719-1731.
[12] STENCLOVA P, FREISINGER S, BARTH H, et al.Cyclic changes in the amide bands within Escherichia coli biofilms monitored using real-time infrared attenuated total reflection spectroscopy (IR-ATR)[J].Applied Spectroscopy, 2019, 73(4):424-432.
[13] KELE TEMUR S, AVCI E, ÇULHA M.Raman and surface-enhanced Raman scattering for biofilm characterization[J].Chemosensors, 2018, 6(1):5.
TEMUR S, AVCI E, ÇULHA M.Raman and surface-enhanced Raman scattering for biofilm characterization[J].Chemosensors, 2018, 6(1):5.
[14] TAHIR M, MAJEED M I, NAWAZ H, et al.Raman spectroscopy for the analysis of different exo-polysaccharides produced by bacteria[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2020, 237:118408.
[15] PETERSEN M, YU Z L, LU X N.Application of Raman spectroscopic methods in food safety:A review[J].Biosensors, 2021, 11(6):187.
[16] IVLEVA N P, WAGNER M, HORN H, et al.Towards a nondestructive chemical characterization of biofilm matrix by Raman microscopy[J].Analytical and Bioanalytical Chemistry, 2009, 393(1):197-206.
[17] SCHUSTER K C, URLAUB E, GAPES J R.Single-cell analysis of bacteria by Raman microscopy:Spectral information on the chemical composition of cells and on the heterogeneity in a culture[J].Journal of Microbiological Methods, 2000, 42(1):29-38.
[18] VIRDIS B, HARNISCH F, BATSTONE D J, et al.Non-invasive characterization of electrochemically active microbial biofilms using confocal Raman microscopy[J].Energy &Environmental Science, 2012, 5(5):7017-7024.
[19] ZHENG J K, HE L L.Surface-enhanced Raman spectroscopy for the chemical analysis of food[J].Comprehensive Reviews in Food Science and Food Safety, 2014, 13(3):317-328.
[20] IVLEVA N P, WAGNER M, HORN H, et al.Raman microscopy and surface-enhanced Raman scattering (SERS) for in situ analysis of biofilms[J].Journal of Biophotonics, 2010, 3(8-9):548-556.
[21] CUI L, ZHANG D D, YANG K, et al.Perspective on surface-enhanced Raman spectroscopic investigation of microbial world[J].Analytical Chemistry, 2019, 91(24):15345-15354.
[22] ZHOU H B, YANG D T, IVLEVA N P, et al.SERS detection of bacteria in water by in situ coating with Ag nanoparticles[J].Analytical Chemistry, 2014, 86(3):1525-1533.
[23] CUI L, CHEN S D, ZHANG K S.Effect of toxicity of Ag nanoparticles on SERS spectral variance of bacteria[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2015, 137:1061-1066.
[24] LUSSIER F, THIBAULT V, CHARRON B, et al.Deep learning and artificial intelligence methods for Raman and surface-enhanced Raman scattering[J].TrAC Trends in Analytical Chemistry, 2020, 124:115796.
[25] SHEN H D, RÖSCH P, PLETZ M W, et al.In vitro fiber-probe-based identification of pathogens in biofilms by Raman spectroscopy[J].Analytical Chemistry, 2022, 94(13):5375-5381.
[26] KUSI D, KAMPE B, RAMOJI A, et al.Raman spectroscopic differentiation of planktonic bacteria and biofilms[J].Analytical and Bioanalytical Chemistry, 2015, 407(22):6803-6813.
D, KAMPE B, RAMOJI A, et al.Raman spectroscopic differentiation of planktonic bacteria and biofilms[J].Analytical and Bioanalytical Chemistry, 2015, 407(22):6803-6813.
[27] EBERT C, TUCHSCHERR L, UNGER N, et al.Correlation of crystal violet biofilm test results of Staphylococcus aureus clinical isolates with Raman spectroscopic read-out[J].Journal of Raman Spectroscopy, 2021, 52(12):2660-2670.
[28] DU X J, WANG X Y, DONG X, et al.Characterization of the desiccation tolerance of Cronobacter sakazakii strains[J].Frontiers in Microbiology, 2018, 9:2867.
[29] CHOO-SMITH L P, MAQUELIN K, VAN VREESWIJK T, et al.Investigating microbial (micro)colony heterogeneity by vibrational spectroscopy[J].Applied and Environmental Microbiology, 2001, 67(4):1461-1469.
[30] WIECZOREK J, GUERIN C, MCMAHON T.K-fold cross-validation for complex sample surveys[J].Stat, 2022, 11(1).
[31] WANG H H, DING S J, WANG G Y, et al.In situ characterization and analysis of Salmonella biofilm formation under meat processing environments using a combined microscopic and spectroscopic approach[J].International Journal of Food Microbiology, 2013, 167(3):293-302.
[32] LI Y Y, HU Y, CHEN T, et al.Advanced detection and sensing strategies of Pseudomonas aeruginosa and quorum sensing biomarkers:A review[J].Talanta, 2022, 240:123210.
[33] SHARMA G, PRAKASH A.Combined use of Fourier transform infrared and Raman spectroscopy to study planktonic and biofilm cells of Cronobacter sakazakii[J].Journal of Microbiology, Biotechnology and Food Sciences, 2014, 3(4):310-314.
[34] DU X J, WANG F, LU X N, et al.Biochemical and genetic characteristics of Cronobacter sakazakii biofilm formation[J].Research in Microbiology, 2012, 163(6-7):448-456.
[35] 赵爱静. 副溶血性弧菌生物被膜形成特性的初步研究[D].上海:上海海洋大学, 2016.
ZHAO A J.Preliminary study on biofilm formation characteristics of Vibrio parahaemolyticus[D].Shanghai:Shanghai Ocean University, 2016.
[36] LIU Y, YU H, CHENG Y, et al.Non-destructive monitoring of Staphylococcus aureus biofilm by surface-enhanced Raman scattering spectroscopy[J].Food Analytical Methods, 2020, 13(9):1710-1716.
[37] CHEN P, WANG J J, HONG B, et al.Characterization of mixed-species biofilm formed by Vibrio parahaemolyticus and Listeria monocytogenes[J].Frontiers in Microbiology, 2019, 10:2543.
[38] RATHER M A, GUPTA K, BARDHAN P, et al.Microbial biofilm:A matter of grave concern for human health and food industry[J].Journal of Basic Microbiology, 2021, 61(5):380-395.
[39] HUANG Y T, CHAKRABORTY S, LIANG H.Methods to probe the formation of biofilms:Applications in foods and related surfaces[J].Analytical Methods, 2020, 12(4):416-432.
[40] KELE TEMUR S, ÇOBANDEDE Z, ÇULHA M.Biofilm formation of clinically important microorganisms on 2D and 3D poly (methyl methacrylate) substrates:A surface-enhanced Raman scattering study[J].Colloids and Surfaces B:Biointerfaces, 2020, 188:110765.
TEMUR S, ÇOBANDEDE Z, ÇULHA M.Biofilm formation of clinically important microorganisms on 2D and 3D poly (methyl methacrylate) substrates:A surface-enhanced Raman scattering study[J].Colloids and Surfaces B:Biointerfaces, 2020, 188:110765.
[41] TAN L, ZHAO F, HAN Q A, et al.High correlation between structure development and chemical variation during biofilm formation by Vibrio parahaemolyticus[J].Frontiers in Microbiology, 2018, 9:1881.
[42] NAKAMURA H, TAKAKURA K I, SONE Y, et al.Biofilm formation and resistance to benzalkonium chloride in Listeria monocytogenes isolated from a fish processing plant[J].Journal of Food Protection, 2013, 76(7):1179-1186.
[43] JANISSEN R, MURILLO D M, NIZA B, et al.Spatiotemporal distribution of different extracellular polymeric substances and filamentation mediate Xylella fastidiosa adhesion and biofilm formation[J].Scientific Reports, 2015, 5:9856.
[44] NGUYEN C Q, THRIFT W J, BHATTACHARJEE A, et al.Longitudinal monitoring of biofilm formation via robust surface-enhanced Raman scattering quantification of Pseudomonas aeruginosa-produced metabolites[J].ACS Applied Materials &Interfaces, 2018, 10(15):12364-12373.
[45] WANG X D, ZHANG Q, CHU L Q.Fabrication of polymer brush/silver nanoparticle nanocomposite as surface enhanced Raman scattering substrate for detection of quorum sensing signal molecule[J].Chinese Journal of Analytical Chemistry, 2018, 46(11):1763-1769.
[46] BODEL N G, MONTES-GARC
N G, MONTES-GARC A V, PÉREZ-JUSTE J, et al.Surface-enhanced Raman scattering spectroscopy for label-free analysis of P.aeruginosa quorum sensing[J].Frontiers in Cellular and Infection Microbiology, 2018, 8:143.
A V, PÉREZ-JUSTE J, et al.Surface-enhanced Raman scattering spectroscopy for label-free analysis of P.aeruginosa quorum sensing[J].Frontiers in Cellular and Infection Microbiology, 2018, 8:143.
[47] PRAZDNOVA E V, GOROVTSOV A V, VASILCHENKO N G, et al.Quorum-sensing inhibition by gram-positive bacteria[J].Microorganisms, 2022, 10(2):350.
[48] GUO J X, LIU Y, CHEN Y L, et al.A multifunctional SERS sticky note for real-time quorum sensing tracing and inactivation of bacterial biofilms[J].Chemical Science, 2018, 9(27):5906-5911.
[49] BODEL N G, MONTES-GARC
N G, MONTES-GARC A V, L
A V, L PEZ-PUENTE V, et al.Detection and imaging of quorum sensing in Pseudomonas aeruginosa biofilm communities by surface-enhanced resonance Raman scattering[J].Nature Materials, 2016, 15(11):1203-1211.
PEZ-PUENTE V, et al.Detection and imaging of quorum sensing in Pseudomonas aeruginosa biofilm communities by surface-enhanced resonance Raman scattering[J].Nature Materials, 2016, 15(11):1203-1211.
[50] PEARMAN W F, LAWRENCE-SNYDER M, ANGEL S M, et al.Surface-enhanced Raman spectroscopy for in situ measurements of signaling molecules (autoinducers) relevant to bacteria quorum sensing[J].Applied Spectroscopy, 2007, 61(12):1295-1300.
[51] YAN S, QIU J, GUO L, et al.Development overview of Raman-activated cell sorting devoted to bacterial detection at single-cell level[J].Applied Microbiology and Biotechnology, 2021, 105(4):1315-1331.
[52] COLNI![]()
 A, DINA N, LEOPOLD N, et al.Characterization and discrimination of gram-positive bacteria using Raman spectroscopy with the aid of principal component analysis[J].Nanomaterials, 2017, 7(9):248.
A, DINA N, LEOPOLD N, et al.Characterization and discrimination of gram-positive bacteria using Raman spectroscopy with the aid of principal component analysis[J].Nanomaterials, 2017, 7(9):248.
[53] BERNATOV S, SAMEK O, PIL
S, SAMEK O, PIL T Z, et al.Following the mechanisms of bacteriostatic versus bactericidal action using Raman spectroscopy[J].Molecules, 2013, 18(11):13188-13199.
T Z, et al.Following the mechanisms of bacteriostatic versus bactericidal action using Raman spectroscopy[J].Molecules, 2013, 18(11):13188-13199.
[54] SAMEK O, ZEMANEK P, BERNATOVA S, et al.Monitoring the influence of antibiotic exposure using Raman spectroscopy[J].Proceedings of SPIE-The International Society for Optical Engineering, 2014, 8939(1):393-398.
[55] HAN Q A, SONG X Y, ZHANG Z H, et al.Removal of foodborne pathogen biofilms by acidic electrolyzed water[J].Frontiers in Microbiology, 2017, 8:988.
[56] CHEN B W, HUANG J M, LI H H, et al.Eradication of planktonic Vibrio parahaemolyticus and its sessile biofilm by curcumin-mediated photodynamic inactivation[J].Food Control, 2020, 113:107181.
[57] PRABHAWATHI V, BOOBALAN T, SIVAKUMAR P M, et al.Functionalized polycaprolactam as an active food package for antibiofilm activity and extended shelf life[J].Colloids and Surfaces B:Biointerfaces, 2014, 123:461-468.
[58] TANNERT A, GROHS R, POPP J, et al.Phenotypic antibiotic susceptibility testing of pathogenic bacteria using photonic readout methods:Recent achievements and impact[J].Applied Microbiology and Biotechnology, 2019, 103(2):549-566.
[59] LU X N, SAMUELSON D R, RASCO B A, et al.Antimicrobial effect of diallyl sulphide on Campylobacter jejuni biofilms[J].The Journal of Antimicrobial Chemotherapy, 2012, 67(8):1915-1926.
[60] JUNG G B, NAM S W, CHOI S, et al.Evaluation of antibiotic effects on Pseudomonas aeruginosa biofilm using Raman spectroscopy and multivariate analysis[J].Biomedical Optics Express, 2014, 5(9):3238-3251.
[61] NAKAR A, PISTIKI A, RYABCHYKOV O, et al.Detection of multi-resistant clinical strains of E.coli with Raman spectroscopy[J].Analytical and Bioanalytical Chemistry, 2022, 414(4):1481-1492.
[62] SPORTELLI M C, KRANZ C, MIZAIKOFF B, et al.Recent advances on the spectroscopic characterization of microbial biofilms:A critical review[J].Analytica Chimica Acta, 2022, 1195:339433.
[63] PRADHAN N, PRADHAN S K, NAYAK B B, et al.Micro-Raman analysis and AFM imaging of Acidithiobacillus ferrooxidans biofilm grown on uranium ore[J].Research in Microbiology, 2008, 159(7-8):557-561.
[64] NIELSEN C K, SUBBIAHDOSS G, ZENG G, et al.Antibacterial isoeugenol coating on stainless steel and polyethylene surfaces prevents biofilm growth[J].Journal of Applied Microbiology, 2018, 124(1):179-187.
[65] JOHNSEN A R, HAUSNER M, SCHNELL A, et al.Evaluation of fluorescently labeled lectins for noninvasive localization of extracellular polymeric substances in Sphingomonas biofilms[J].Applied and Environmental Microbiology, 2000, 66(8):3487-3491.
[66] NEU T R, SWERHONE G D W, LAWRENCE J R.Assessment of lectin-binding analysis for in situ detection of glycoconjugates in biofilm systems[J].Microbiology, 2001, 147(2):299-313.
[67] WAGNER M, IVLEVA N P, HAISCH C, et al.Combined use of confocal laser scanning microscopy (CLSM) and Raman microscopy (RM):Investigations on EPS - Matrix[J].Water Research, 2009, 43(1):63-76.
[68] IVLEVA N P, KUBRYK P, NIESSNER R.Raman microspectroscopy, surface-enhanced Raman scattering microspectroscopy, and stable-isotope Raman microspectroscopy for biofilm characterization[J].Analytical and Bioanalytical Chemistry, 2017, 409(18):4353-4375.
[69] CUI L, YANG K, ZHOU G W, et al.Surface-enhanced Raman spectroscopy combined with stable isotope probing to monitor nitrogen assimilation at both bulk and single-cell level[J].Analytical Chemistry, 2017, 89(11):5793-5800.
[70] MUHAMADALI H, CHISANGA M, SUBAIHI A, et al.Combining Raman and FT-IR spectroscopy with quantitative isotopic labeling for differentiation of E.coli cells at community and single cell levels[J].Analytical Chemistry, 2015, 87(8):4578-4586.
[71] FENG J S, DE LA FUENTE-N
 EZ C, TRIMBLE M J, et al.An in situ Raman spectroscopy-based microfluidic “lab-on-a-chip” platform for non-destructive and continuous characterization of Pseudomonas aeruginosa biofilms[J].Chemical Communications, 2015, 51(43):8966-8969.
EZ C, TRIMBLE M J, et al.An in situ Raman spectroscopy-based microfluidic “lab-on-a-chip” platform for non-destructive and continuous characterization of Pseudomonas aeruginosa biofilms[J].Chemical Communications, 2015, 51(43):8966-8969.
[72] HUANG H K, CHENG H W, LIAO C C, et al.Bacteria encapsulation and rapid antibiotic susceptibility test using a microfluidic microwell device integrating surface-enhanced Raman scattering[J].Lab on a Chip, 2020, 20(14):2520-2528.