毛油中磷脂成分是影响植物成品油气味、口感、外观及保质期的重要因素之一,因此油脂精炼过程脱胶效果直接影响油脂精炼的效率和最终产品质量[1-4]。传统脱胶工艺存在污水排放量大、中性油脂消耗大、残磷量高等问题,因此会增加后续油脂精炼的工艺步骤[5-6]。酶法脱胶是一种植物油脂脱胶的绿色新工艺,与传统脱胶工艺相比,酶法脱胶具有脱胶完全、用水量和废水排放少、耗能低、精炼得率高等优点,因此备受世界各国油脂加工工业的关注[7-9]。磷脂酶是应用于植物油脂酶法脱胶的重要工业酶,根据作用磷脂位点不同,可以分为磷脂酶A1、磷脂酶A2、磷脂酶B和磷脂酶C,其中磷脂酶A1和磷脂酶A2是目前酶法脱胶中应用最多的磷脂酶[10-13]。
磷脂酶C(phospholipse C, PLC)是一种水解甘油磷脂Sn-3位点甘油磷酸酯键的脂类水解酶,水解产物为甘油二酯及有机磷酸酯(磷酸胆碱、磷酸乙醇胺或磷酸肌醇等)[14-15]。PLC广泛存在于原核生物和真核生物中,相关基因已在动物、植物、酵母、细菌等多种生物中鉴定与分析[16]。PLC作用于磷脂分子的酯键,生成易溶于水的磷酸酯等而被脱除,同时生成甘油二酯进而增加中性油的得率,因此PLC是油脂精炼得率最高的酶法脱胶方式。目前已报道PLC存在催化活性低、稳定性不高、微生物来源酶不易分离纯化和重组酶多以包涵体表达等问题,因此亟需寻找适用于油脂酶法脱胶的新型酶源[17-20]。本研究利用分子克隆方法从吡咯伯克霍尔德氏菌(Burkholderia puraquae)克隆表达新型PLC基因plc_bp,利用大肠杆菌表达系统实现可溶性表达,通过热处理、镍柱亲和层析和分子筛层析获得纯化重组酶,详细研究重组酶酶学性质、结构与功能以及油脂脱胶效果,为植物油脂酶法脱胶提供新的酶源,为酶法脱胶的工业化应用提供理论和应用基础。
1 材料与方法
1.1 实验材料
1.1.1 主要菌株与质粒
吡咯伯克霍尔德氏菌B.puraquae、大肠杆菌Escherichia coli DH5α、E.coli BL21-CodonPlus(DE3)-RIL和质粒pET28a,本实验室保存。
1.1.2 主要试剂与原料
PfuDNA聚合酶、限制性内切酶Nde Ⅰ和Sal Ⅰ、DNA连接试剂盒,宝生物工程(大连)有限公司;磷脂酰胆碱(phosphatidyl cholines, PC)、磷脂酰乙醇胺(phosphatidyl ethanolamine, PE)、磷脂酰甘油(phosphatidylglycerol, PG),美国Avanti公司;对硝基苯基磷酰胆碱(p-nitrophenylphosphorylcholine, NPPC)、PCR引物、卡那霉素、异丙基-那霉素、异丙基硫代半乳糖苷(isopropyl β-D-thiogalactoside, IPTG)等,生工生物工程(上海)股份有限公司;大豆油、花生油毛油,金太阳粮油股份有限公司;葵花籽油、粗菜籽油毛油,河南省淇花食用油有限公司。
1.2 实验方法
1.2.1 重组酶PLC_BP生物信息学分析
利用NCBI网站BLASTp和Clustal X 2.0软件,对B.puraquae重组磷脂酶PLC_BP及其同源蛋白氨基酸序列进行比对分析。利用同源建模软件SWISS-MODEL和Pymol对PLC_BP三维结构进行预测和分析。
1.2.2 PLC_BP基因克隆
根据B.puraquae重组磷脂酶PLC_BP基因核苷酸序列,设计并合成引物。上游引物:5′-CAGCCATATGGGCCAGAAAACGTCTGCGACC-3′;下游引物:5′-TCGCGGATCCTCAGGCAGTGGCCTTGGCC-3′。在上下游引物5′端分别设计Nde Ⅰ与BamH Ⅰ酶切位点(划线部分表示),由上海生工公司合成。100 μL PCR反应体系:模板DNA 1.0 μL(20 ng),dNTP 1.0 μL(25 mmol/L),引物(100 μmol/L)各1.0 μL,10×缓冲液10 μL,Pfu DNA聚合酶(5 U/μL)1.0 μL,超纯水43.0 μL。PCR反应条件:94 ℃ 5 min、94 ℃ 45 s、58 ℃ 60 s、72 ℃ 1.5 min,30个循环。PCR产物经双酶切后连接到pET28a上,转化大肠杆菌E.coli DH5α并筛选重组质粒,经Nde Ⅰ与BamH Ⅰ双酶切及上海生工测序验证后将其命名为pET28a-PLC_BP。
1.2.3 重组蛋白的表达与纯化
挑取平板上生长的菌落于含有50 μg/mL卡那霉素的30 mL LB液体培养基中,37 ℃、200 r/min过夜培养。按照体积分数2%接种量进行扩大培养,转接于含有50 μg/mL卡那霉素的600 mL LB液体培养基,培养至OD600 nm为0.6~0.8,加入终浓度为0.2 mmol/L的IPTG于30 ℃继续培养8 h,4 ℃、8 000×g离心20 min。向所得菌体中添加破壁buffer(50 mmol/L Tris-HCl,pH 7.9,50 mmol/L NaCl),悬浮菌体后于冰上进行超声波破碎10 min。按照破壁buffer的0.1%体积分数添加Triton X-100,于4 ℃、75 r/min振荡2.5 h,取1 mL留样为全蛋白。超声波破碎菌体后60 ℃热处理30 min,4 ℃、8 000×g离心20 min,上清液即为粗酶液。采用镍柱亲和层析方法进行目的蛋白的纯化,以20~500 mmol/L的线性咪唑(流速1 mL/min)梯度洗脱重组酶。利用Superdex 200(16/60)凝胶过滤柱进一步纯化蛋白,以缓冲液(50 mmol/L Tris-HCl,pH 7.4,200 mmol/L NaCl)洗脱目的蛋白。SDS-PAGE来检测纯化效果,蛋白质浓度用考马斯亮蓝比色Bradford方法测定。
1.2.4 重组酶PLC_BP活性的检测
在96孔板中测定PLC_BP活性,具体酶活性反应体系:1 mmol/L NPPC底物、1 μmol/L纯化酶蛋白、60%(体积分数)山梨醇、1 mmol/L MnCl2和反应缓冲液50 mmol/L Tris-HCl(pH 8.5),60 ℃反应30 min,并在405 nm下测定吸光度。酶活力单位定义:每分钟生成1 mmol对硝基苯酚所需的酶量为1个酶活力单位。
1.2.5 重组酶PLC_BP酶学性质研究
1.2.5.1 底物特异性
分别以PC、PE和PG为反应底物,于标准反应体系中测定酶活力,测定重组酶PLC_BP的底物特异性。
1.2.5.2 最适反应温度与热稳定性
最适反应温度:以1 mmol/L NPPC作为反应底物,缓冲体系为50 mmol/L的Tris-HCl(pH 8.5),反应时间30 min,在25~80 ℃,每隔5 ℃测酶活力。酶的热稳定性:取相同浓度的酶置于不同温度(50、60、70 ℃)下,分别保温0~8 h后,在标准体系下检测酶活力。
1.2.5.3 最适反应pH
在不同pH(6.0~11.0)的缓冲液下测酶活力。所用缓冲液为50 mmol/L的磷酸钠缓冲液(pH 6.0~7.5)、Tris-HCl缓冲液(pH 7.5~9.5)和3-环己氨基丙磺酸缓冲液(pH 9.5~11.0),反应温度60 ℃,底物为NPPC,对照不加酶,以相应pH的缓冲液代替。
1.2.5.4 金属离子对酶活性的影响
研究不同金属离子对PLC_BP活力的影响。在标准反应体系中分别加入不同的金属离子溶液(Zn2+、Ca2+、Mg2+、Mn2+、Fe2+、Ni2+和Cu2+)至终浓度为5 mmol/L,室温放置60 min,以NPPC为底物,以不加任何金属离子的酶促反应物为对照,60 ℃反应测酶活力。
1.2.6 酶法脱胶
参照YANG等[21]的方法稍作改进,用PLC_BP对油菜籽油、大豆油、花生油及向日葵籽油进行酶法脱胶工艺研究。100 g毛油加入0.1 mL柠檬酸溶液(450 g/L),置于可以密封的锥形瓶,10 000 r/min均质1 min,于75 ℃、500 r/min孵育20 min。将混合液10 000×g离心10 min。向上清混合液中加入一定量柠檬酸溶液(0.1 mmol/L)调节pH。加入2 mg纯化后的PLC_BP(250 U/mg),并加入2 mL去离子水,5 000 r/min均质1 min进行乳化,酶脱胶过程在65 ℃、500 r/min条件下进行。反应结束后,进行磷含量和脂肪酸分析。磷含量分析方法:油乳样品10 mL,80 ℃水浴加热10 min,10 000×g离心10 min,取5 g上清油,使用GB/T 5537—2008《粮油检验 磷脂含量的测定》中的钼蓝比色法进行磷含量分析。游离脂肪酸含量根据AOCS Ca 5a-40《Free Fatty Acids》中的方法进行测定。
2 结果与讨论
2.1 PLC_BP氨基酸序列分析
NCBI网站中BLAST分析PLC_BP的同源序列,发现在Burkholderia lata和Burkholderia cepacia等中存在同源蛋白,相似性在95%以上;PLC193蛋白来自海洋细菌Phaeobacter sp.strain MED 193,是课题组前期已经报道的具有水解NPPC和磷脂的PLC;来自Haemophilus influenza 的HiLpxH(UDP-2,3-二酰基葡萄糖胺水解酶,PDB:5K8K)是PLC_BP同源建模的模板蛋白。利用软件Clustal X 2.0比对PLC_BP的同源序列,结果如图1所示,PLC_BP及其同源蛋白具有5个保守结构域[11,15],分别为SDIHL、GDI、GNHD、HGD和GHIH,根据文献报道预测D93和H169与金属离子Mn2+存在相互作用关系。

图1 PLC_BC与同源蛋白氨基酸序列比对
Fig.1 PLC_BC alignment with amino acid sequences of congeners
注:右边序号代表氨基酸的位置;阴影部分为保守序列;“*”和“:”分别代表保守的和半保守氨基酸位点;5个保守结构域(SDIHL、GDI、GNHD、HGD和GHIH)已经表示标识;PLC_BP:B.puraquae(WP085042045.1);PLC_BL:B.lata(ABB09191.1);PLC_BC:B.cepacia(WP059531150.1);PLC193:Phaeobacter sp.strain MED 193(EAQ46983.1);HiLpxH:H.influenza(BBF08221.1)。
2.2 重组酶PLC_BP表达及纯化
重组质粒pET28a-PLC_BP在E.coli BL21中诱导表达,经热处理、镍柱亲和层析及分子筛Superdex 200层析纯化,得到纯化重组酶PLC_BP,分子质量是38.0 kDa。点突变体D93A和H169A表达纯化步骤与PLC_BP野生型酶相同,结果如图2所示。
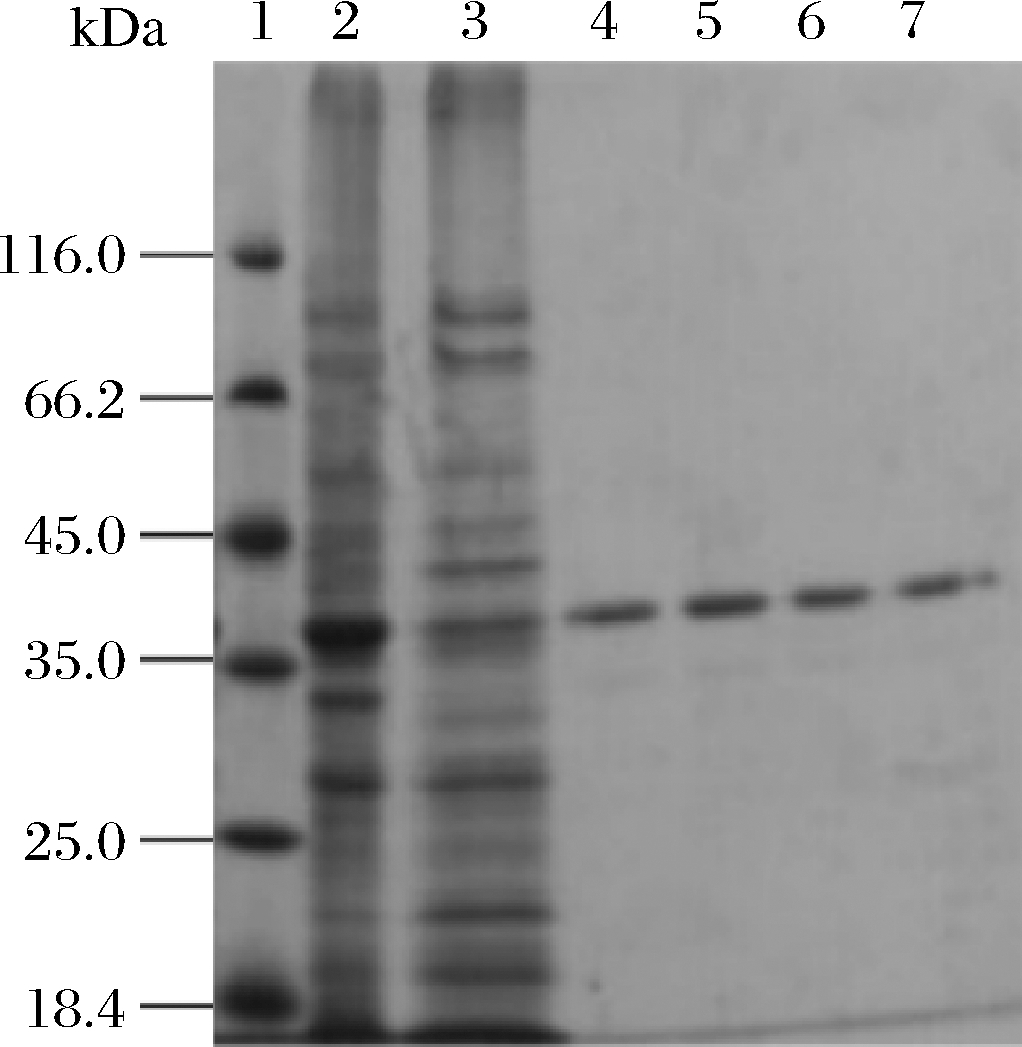
1-Marker;2-全蛋白;3-粗蛋白;4-镍柱亲核层析纯化的酶;5-分子筛Superdex 200层析纯化的酶;6-纯化后突变体D93A;7-纯化后突变体H169A
图2 纯化重组酶PLC_BP的SDS-PAGE结果
Fig.2 SDS-PAGE of purified recombinant PLC_BP
2.3 最适反应温度、热稳定性与反应pH
重组酶PLC_BP的最适温度为60 ℃,在45~65 ℃时酶活力能保持最大活力的一半以上(图3-a)。热稳定性实验表明,60 ℃处理8 h,重组酶仍然保持50%左右的活力(图3-b),结果表明重组酶PLC_BP是具有较好的热稳定性。重组酶最适pH为8.5,pH 7.5~9.5时酶活力能保持最大活力的50%以上(图3-c)。
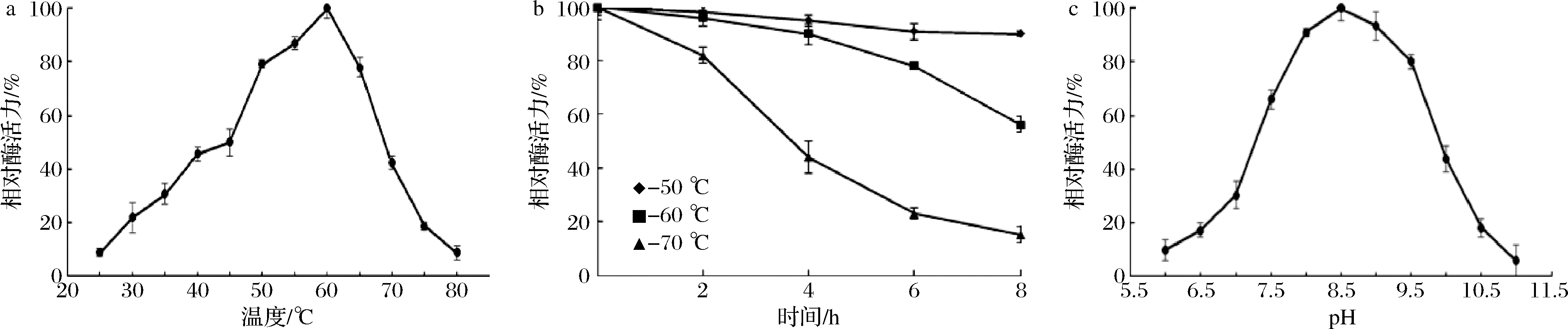
a-最适反应温度;b-热稳定性;c-最适反应pH
图3 温度和pH对酶活力的影响
Fig.3 Effects of temperature and pH on enzyme activity
2.4 金属离子对PLC_BP活力的影响
如图4所示,Mn2+具有明显的激活作用,这与文献报道部分PLC需要Mn2+作为辅助因子是一致的[10-11,15]。分别以Zn2+、Ca2+、Mg2+、Fe2+、Ni2+和Cu2+作为酶反应辅助因子,PLC_BP的活性分别下降到33.1%、15.3%、23.1%、8.3%、11.6%和3.5%,因此PLC_BP是Mn2+依赖性PLC,与氨基酸序列同源性和蛋白同源建模分析结果相一致。
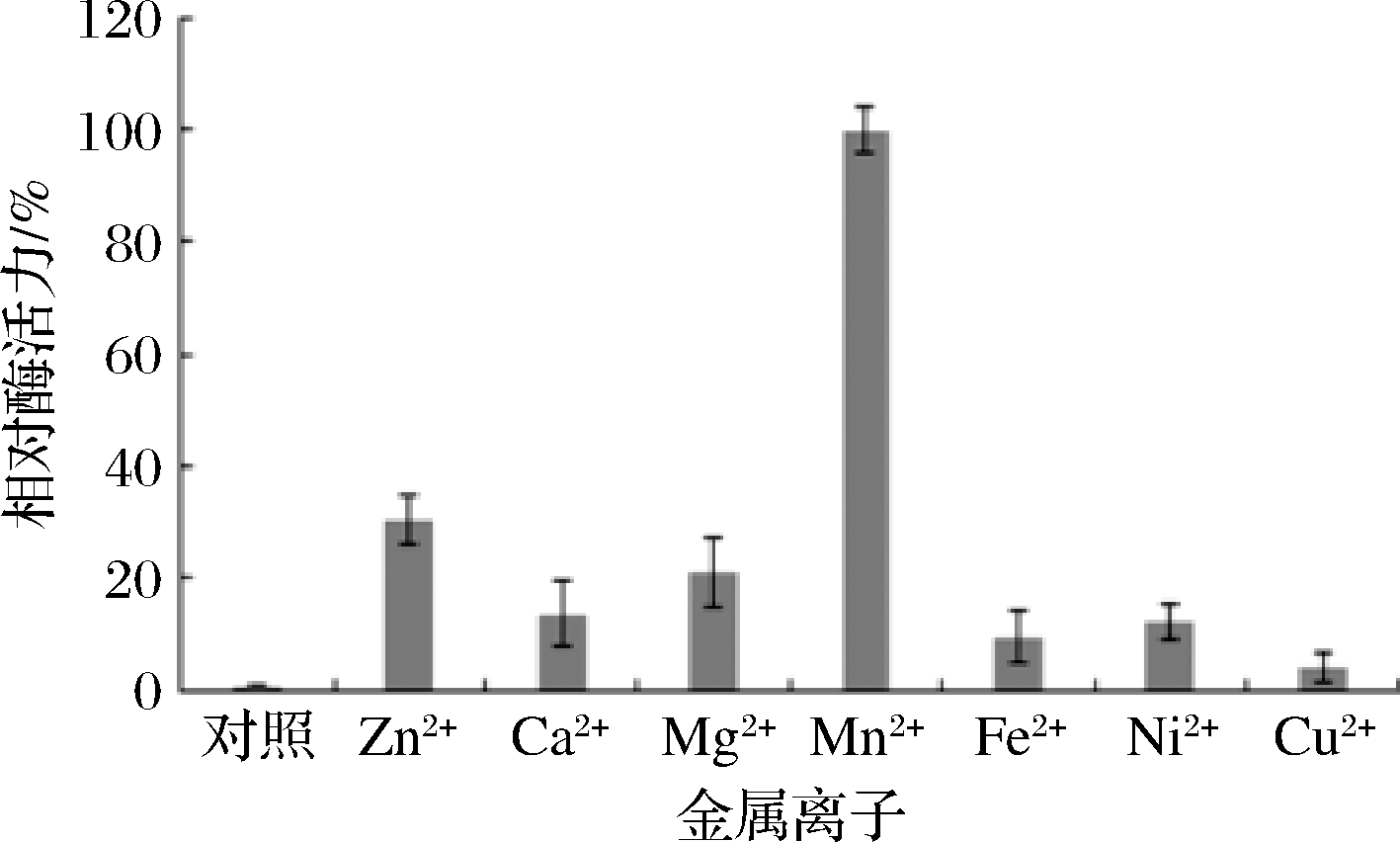
图4 金属离子对酶活力的影响
Fig.4 Effects of metal ions on enzyme activity
2.5 PLC_BP对天然磷脂的水解特异性
由表1可知, PLC_BP对不同磷脂的kcat/Km值依次为PC[7.3 s-1·(mmol/L)-1]>PE[5.7 s-1·(mmol/L)-1]>PG[1.5 s-1·(mmol/L)-1],表明PC是PLC的最佳底物,PC和PE在植物油中是含量最高的2种磷脂, PLC对PC、PE水解效果好,也为后续油脂脱胶打下了较好基础。
表1 PLC_BP对不同天然磷脂底物的动力学参数
Table 1 Kinetic parameters of PLC_BP on natural phospholipid

底物kcat/s-1Km/(mmol/L)kcat/Km/[s-1·(mmol/L)-1]NPPC54.3±4.2214.2±3.3253.5±4.7PC18.2±2.12.5±1.27.3±0.6PE20.4±1.43.6±1.75.7±1.0PG7.9±0.75.4±0.91.5±1.2
2.6 PLC结构的预测
以来自H.influenza的HiLpxH为模板(PDB:5K8K,相似性16%)建立PLC_BP的蛋白结构,如图5所示。
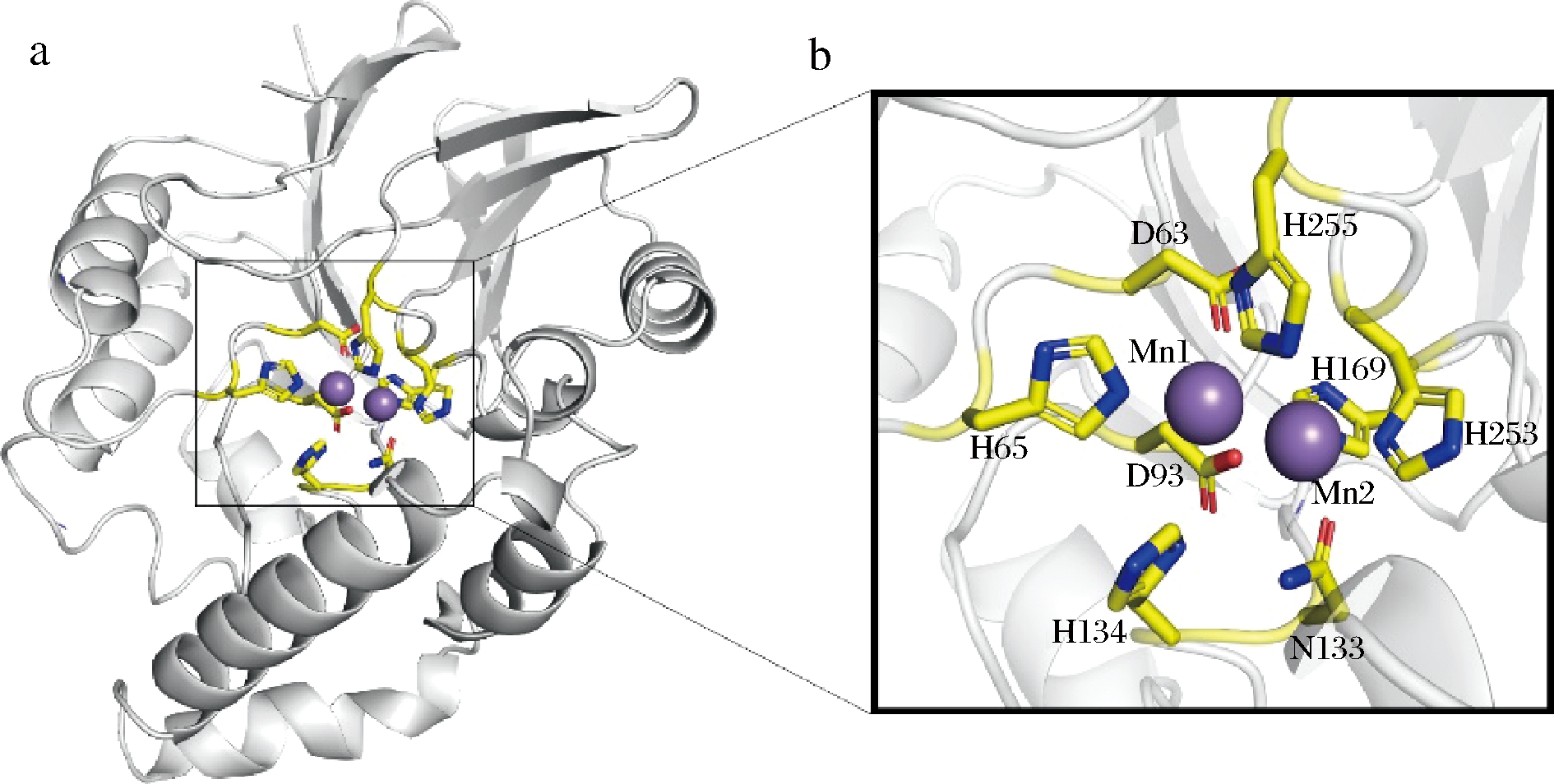
a-PLC_BP预测三维结构的双核锰离子结合位点;b-PLC_BP催化关键位点
图5 PLC_BP金属结构域结构预测
Fig.5 Structure prediction of PLC_BP metal domain
预测PLC_BP蛋白结构具有双核锰离子结合位点(图5-a),其中处于保守结构域(SDIHL、GDI、GNHD、HGD和GHIH)的残基D63、H65、D93、N133、H134、H169、H253、H255与酶催化活性关键位点,其中D93和H169与金属离子Mn2+结合存在相互作用(图5-b)[10-11,15]。通过定点突变构建Mn2+结合关键位点D93A和H169A,对比分析野生型和突变体动力学(表2),发现D93A和H169A的Km为468.4和382.6 mmol/L,大于野生型酶的Km值(214.2 mmol/L),突变体酶与底物NPPC的结合能力低于野生型酶;对比分析催化效率,突变体酶D93A和H169A的kcat/Km为8.1和6.5 s-1·(mmol/L)-1,远低于野生型酶的kcat/Km值(253.5 mmol/L),突变体酶催化效率也低于野生型酶,以上结果表明氨基酸位点D93和H169与金属离子Mn2+结合有关。
表2 PLC_BP与突变体对NPPC底物的动力学参数
Table 2 Kinetic parameters of wild-type and site-directed mutants of PLC_BP and on NPCC substrate
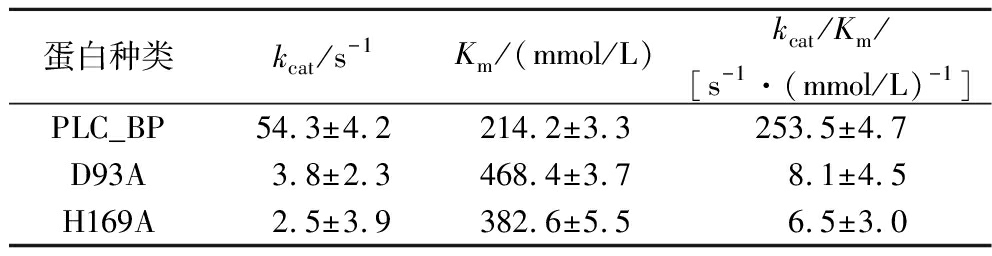
蛋白种类kcat/s-1Km/(mmol/L)kcat/Km/[s-1·(mmol/L)-1]PLC_BP54.3±4.2214.2±3.3253.5±4.7D93A3.8±2.3468.4±3.78.1±4.5H169A2.5±3.9382.6±5.56.5±3.0
2.7 PLC脱胶效果
脱胶油中磷含量降低(<10 mg/kg)满足了油脂工业精炼的要求。向柠檬酸处理后的油样中加入PLC_BP,反应5 h,菜籽油、大豆油和葵花籽油的残磷量均降至10 mg/kg内(图6)。酶脱胶反应6 h后,菜籽油、大豆油、花生油和葵花籽油的残磷量分别为3.5、4.2、2.8和3.4 mg/kg。
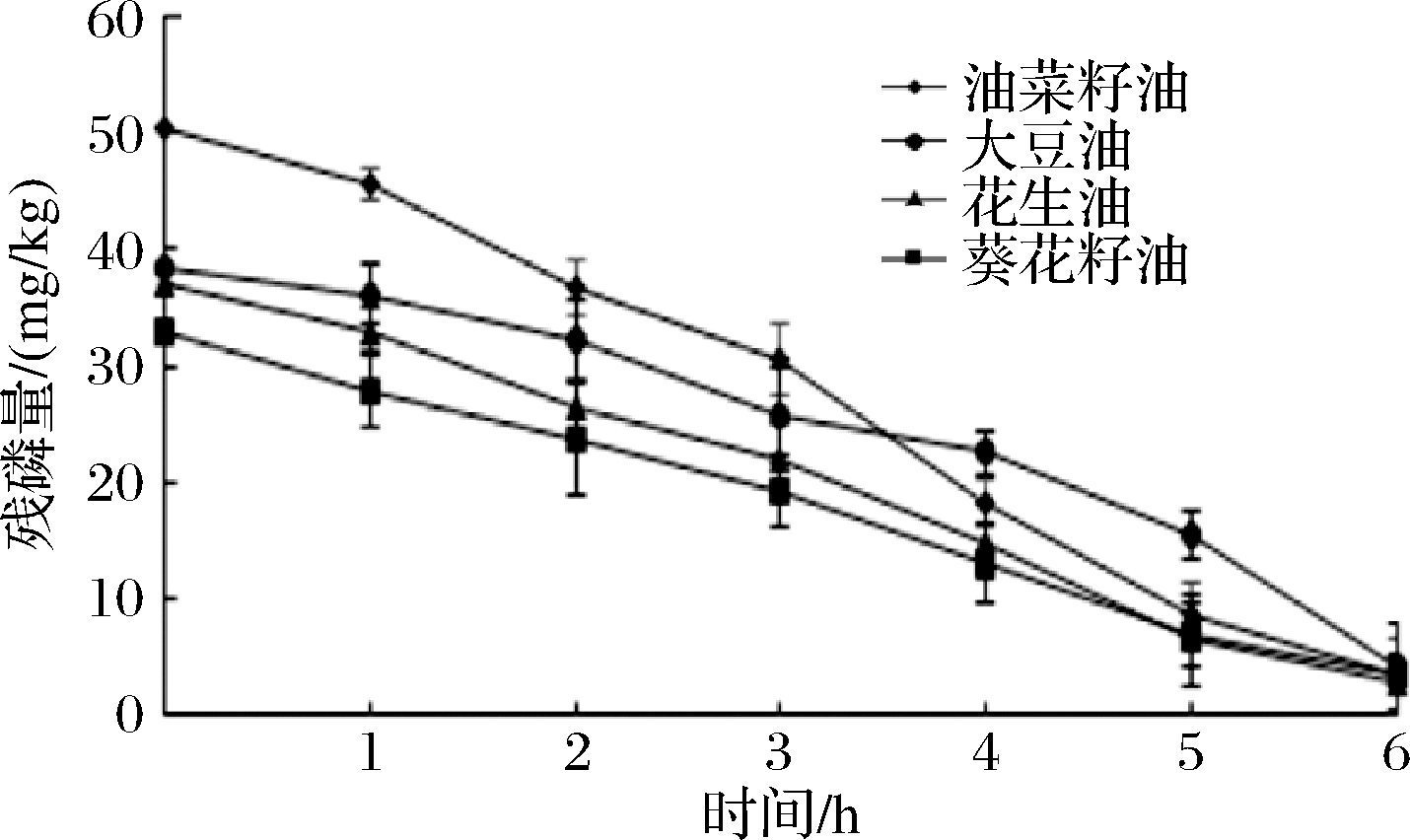
图6 反应时间对PLC_BP脱胶效果的影响
Fig.6 Effect of reaction time on degumming of PLC_BP
用PLC_BP进行毛油脱胶会使游离脂肪酸的含量略有增加。如表3所示,PLC_BP对菜籽油、大豆籽油、花生籽油和葵花籽油进行酶法脱胶后(6 h),游离脂肪酸质量分数增加了0.04%~0.08%。据报道,在酶脱胶反应过程中,残磷含量降低至100 mg/kg时,游离脂肪酸质量分数将增加约0.2%[22]。因此,本实验中发现游离脂肪酸含量的增加与磷含量的下降(29.5~46.7 mg/kg)基本一致,说明游离脂肪酸浓度增加是磷脂被酶水解的结果。以上结果表明,PLC_BP具有较好的植物油脱胶活性,是一种具有油脂工业应用前景的植物油酶法脱胶的工具酶。
表3 PLC_BP脱胶不同植物油的脂肪酸含量 单位:%
Table 3 Fatty acid content of different vegetable oils degummed by PLC_BP

反应时间/h油菜籽油大豆油花生油葵花籽油02.85±0.021.50±0.042.28±0.011.83±0.0622.87±0.031.51±0.012.30±0.041.84±0.0242.90±0.011.53±0.032.31±0.021.85±0.0362.93±0.031.55±0.062.34±0.031.87±0.05
3 结论
本文成功构建吡咯伯克霍尔德氏菌B.puraquae新型PLC原核表达载体pET28a-PLC_BP,将重组质粒转入大肠杆菌实现可溶性表达。经60 ℃热处理、镍柱亲和层析和分子筛层析纯化,得到纯化的重组磷脂酶PLC_BP,SDS-PAGE检测该酶分子质量是38.0 kDa。酶学性质研究表明,该酶的最适温度与pH分别为60 ℃与pH 8.5,在最适反应温度60 ℃处理8 h,酶活力稳定在50%以上。通过氨基酸序列同源性和蛋白质同源建模分析,发现PLC_BP具有5个保守结构域(SDIHL、GDI、GNHD、HGD和GHIH)和2个Mn2+结合关键位点(D93A和H169A)。PLC_BP可有效降低不同来源毛油中的磷脂,经过脱胶后菜籽油、大豆油、花生油和葵花籽油的残磷量分别为3.5、4.2、2.8和3.4 mg/kg,满足后续油脂精炼要求,为植物油脂酶法脱胶提供新的酶源,为酶法脱胶的工业化应用提供理论和应用基础。
[1] 冀楠, 熊昌武, 肖俊川, 等.植物油酶法脱胶通用条件的研究[J].中国油脂, 2016, 41(2):9-12.
JI N, XIONG C W, XIAO J C, et al.General condition of enzymatic degumming process for vegetable oil[J].China Oils and Fats, 2016, 41(2):9-12.
[2] 杨亚济. 大豆油酶法脱胶应用实践[J].中国油脂, 2016, 41(8):107-109.
YANG Y J.Application practice of enzymatic degumming of soybean oil[J].China Oils and Fats, 2016, 41(8):107-109.
[3] 尚建疆, 郭伟, 肖建华, 等.超声辅助菜籽毛油脱胶工艺的研究[J].现代盐化工, 2022, 49(2):43-45;53.
SHANG J J, GUO W, XIAO J H, et al.Study on ultrasonic assisted degumming process of rapeseed crude oil[J].Modern Salt and Chemical Industry, 2022, 49(2):43-45;53.
[4] 刘黔霞, 吴雪君, 杨坤, 等.植物油中磷脂含量的测定[J].云南化工, 2020, 47(4):134-135.
LIU Q X, WU X J, YANG K, et al.Comparison of methods for determination of phospholipid content in vegetable oil[J].Yunnan Chemical Technology, 2020, 47(4):134-135.
[5] 刘昌盛, 杨湄, 黄凤洪.食用植物油脱胶技术研究进展[J].农产品加工(创新版), 2010(7):47-49;56.
LIU C S, YANG M, HUANG F H.Review on recent development in vegetable degumming[J].Innovational Edition of Farm Products Processing, 2010(7):47-49;56.
[6] NIKOLAEVA T, RIETKERK T, SEIN A, et al.Impact of water degumming and enzymatic degumming on gum mesostructure formation in crude soybean oil[J].Food Chemistry, 2020, 311:126017.
[7] 许皎姣, 孙乐, 王强, 等.植物油酶法脱胶技术的研究进展[J].粮食与食品工业, 2021, 28(4):14-17.
XU J J, SUN L, WANG Q, et al.Research progress of enzymatic degumming of vegetable oil[J].Cereal &Food Industry, 2021, 28(4):14-17.
[8] 徐赢华, 王国敬, 李春, 等.酶法脱胶在植物油脂精炼中的应用进展[J].农业工程学报, 2015, 31(23):269-276.
XU Y H, WANG G J, LI C, et al.Application of enzymatic degumming on vegetable oils refining[J].Transactions of the Chinese Society of Agricultural Engineering, 2015, 31(23):269-276.
[9] 蒋晓菲, 夏袁, 金青哲, 等.菜籽毛油酸法脱胶工艺条件的优化[J].中国油脂, 2013, 38(6):4-8.
JIANG X F, XIA Y, JIN Q Z, et al.Optimization of acid-assisted degumming of crude rapeseed oil[J].China Oils and Fats, 2013, 38(6):4-8.
[10] LYU Y, YE L D, XU J, et al.Recent research progress with phospholipase C from Bacillus cereus[J].Biotechnology Letters, 2016, 38(1):23-31.
[11] BORRELLI G M, TRONO D.Recombinant lipases and phospholipases and their use as biocatalysts for industrial applications[J].International Journal of Molecular Sciences, 2015, 16(9):20774-20840.
[12] DURRANI R, KHAN F I, ALI S, et al.A thermolabile phospholipase B from Talaromyces marneffei GD-0079:Biochemical characterization and structure dynamics study[J].Biomolecules, 2020, 10(2):231.
[13] YANG P Z, JIANG S W, WU Y, et al.Recombinant expression of Serratia marcescens outer membrane phospholipase A (A1) in Pichia pastoris and immobilization with graphene oxide-based Fe3O4 nanoparticles for rapeseed oil degumming[J].Frontiers in Microbiology, 2019, 10:334.
[14] WEI T, QUARESHY M, ZHANG Y Z, et al.Manganese is essential for PlcP metallophosphoesterase activity involved in lipid remodeling in abundant marine heterotrophic bacteria[J].Applied and Environmental Microbiology, 2018, 84(15):e01109-e01118.
[15] CERMINATI S, PAOLETTI L, AGUIRRE A, et al.Industrial uses of phospholipases:Current state and future applications[J].Applied Microbiology and Biotechnology, 2019, 103(6):2571-2582.
[16] CERMINATI S, EBERHARDT F, ELENA C E, et al.Development of a highly efficient oil degumming process using a novel phosphatidylinositol-specific phospholipase C enzyme[J].Applied Microbiology and Biotechnology, 2017, 101(11):4471-4479.
[17] 邹权, 陈圣, 严明.磷脂酰胆碱特异性和磷脂酰肌醇特异性磷脂酶C酶学性质及其在油脂脱胶中的应用[J].生物加工过程, 2019, 17(2):144-151.
ZOU Q, CHEN S, YAN M.Characterization of phosphatidylcholine- and phosphatidylinositol-specific phospholipase C and its application in oil degumming[J].Chinese Journal of Bioprocess Engineering, 2019, 17(2):144-151.
[18] MARCHISIO F, DI NARDO L, VAL D S, et al.Characterization of a novel thermostable phospholipase C from T.kodakarensis suitable for oil degumming[J].Applied Microbiology and Biotechnology, 2022, 106(13-16):5081-5091.
[19] 宋玉卿, 王彤, 李丹, 等.诱变蜡状芽孢杆菌产磷脂酶C的研究[J].中国食品学报, 2018, 18(3):141-149.
SONG Y Q, WANG T, LI D, et al.Studies on mutagenesis Bacillus cereus producing phospholipase C[J].Journal of Chinese Institute of Food Science and Technology, 2018, 18(3):141-149.
[20] 汪艳红, 张梁, 顾正华, 等.乙酸钙不动杆菌磷脂酶C在大肠杆菌中重组表达、纯化及酶学性质分析[J].微生物学报, 2014, 54(10):1221-1227.
WANG Y H, ZHANG L, GU Z H, et al.Overexpression, purification and characterization of phospholipase C from Acinetobacter calcoaceticus[J].Acta Microbiologica Sinica, 2014, 54(10):1221-1227.
[21] YANG J G, WANG Y H, YANG B, et al.Degumming of vegetable oil by a new microbial lipase[J].Food Technology and Biotechnology, 2014, 44(1):101-104.
[22] JIANG F Y, HUANG S, IMADAD K, et al.Cloning and expression of a gene with phospholipase B activity from Pseudomonas fluorescens in Escherichia coli[J].Bioresource Technology, 2012, 104:518-522.