嘌呤核苷磷酸化酶(EC 2.4.2.1,purine nucleoside phosphorylase,PNP)是嘌呤补救合成途径中的关键酶,可逆的催化嘌呤核苷的磷酸化反应,生成嘌呤碱基和核糖-1-磷酸[1-2]。PNP属于N-水解和转移酶家族,普遍存在于细菌、真菌和哺乳动物中,根据PNP的寡聚状态、分子质量和底物特异性将其分成两类[3],第一类为低分子质量同源三聚体,特异性的以6-氧代嘌呤为底物,分子质量约为32 kDa,广泛存在于真核生物中,如人红细胞、牛脾脏细胞、酿酒酵母等[4-6];第二类为高分子质量同源六聚体,具有广泛的底物特异性,能够催化6-氧代嘌呤和6-氨基嘌呤等多种类型的核苷酸分解,分子质量约为26 kDa,普遍存在于细菌中,如大肠杆菌、蜡样芽胞杆菌、耐盐芽胞杆菌、嗜热栖热菌等[3,7-8]。研究发现,PNP在很多方面都起到重要作用,可作为前体物质合成利巴韦林,用作病毒性肺炎和支气管炎等病症的治疗[9],还广泛应用于低嘌呤食品的开发中。2016年,MAHOR等[10]将乳酸克鲁维酵母来源的PNP在大肠杆菌中成功重组表达,并对重组菌降低啤酒嘌呤方面的应用进行了初步的探索。在国内,李玉淼等[11]将大肠杆菌PNP基因在大肠杆菌BL21(DE3)中进行表达,酶活力较对照菌提高16倍,将酶添加到糖化醪液中,提高了嘌呤物质的利用率,能够降低发酵液中29%的游离嘌呤。
枯草芽胞杆菌(Bacillus subtilis)同大肠杆菌、毕赤酵母一样,是常见的工程菌株之一,普遍应用于外源蛋白的表达。相较于其他系统,枯草芽胞杆菌表达系统有一套高效分泌蛋白的启动子和信号肽,在蛋白表达过程中不易形成包涵体,能够稳定表达外源蛋白,易于分离纯化目的蛋白[12-13]。枯草芽胞杆菌表达存在一定的密码子偏好性,当外源基因含有非枯草芽胞杆菌偏好密码子时,将阻碍外源基因的表达。因此,根据表达宿主选择目的基因最优密码子,能有效提高目的蛋白异源表达效率,如陈灵艳等[14]通过密码子优化增加了目的基因在枯草芽胞杆菌中的表达量;KUMAR等[15]将密码子优化后的人干扰素基因转入枯草芽胞杆菌中表达,产物生成量比优化前提高30%。
本研究以NCBI中登录号CP042457.1的乳酸克鲁维酵母(Kluyveromyces lactis,Klac)PNP基因序列为基础,在不改变重组蛋白氨基酸序列的前提下,根据枯草芽胞杆菌的密码子偏好性对KlacPNP基因进行密码子优化。连接至枯草芽胞杆菌分泌表达载体,转入枯草芽胞杆菌WB600中,得到了高产PNP的重组菌株。通过摇瓶发酵及发酵条件单因素和响应面分析,初步优化了其发酵工艺。实现PNP在枯草芽胞杆菌表达系统中异源表达的同时,避免外源基因表达产生的弊端,提高PNP的表达水平,为PNP在枯草芽胞杆菌中生产奠定了基础,以期实现重组PNP的工业化生产,从而拓宽其在降低食品嘌呤中的广泛应用。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
枯草芽胞杆菌(Bacillus subtills)WB600、表达载体pBSA43为本实验室保藏,大肠杆菌DH5α感受态购自TaKaRa生物工程有限公司。
1.1.2 主要试剂、仪器
PCR引物由睿博兴科公司合成;胶回收试剂盒、PCR产物纯化回收试剂盒、质粒提取试剂盒,OMEGA公司;限制性内切酶、T4 DNA连接酶,NEB生物科技有限公司;其他化学试剂均为国产分析纯。
PCR仪、电泳仪、凝胶成像系统,Bio-Rad公司;高效液相色谱仪,美国安捷伦公司。
1.1.3 培养基
LB(Luria-Bertani)液体培养基、LB固体培养基按文献[16]的方法配制。
发酵产酶培养基(g/L):蛋白胨 9.5, 葡萄糖12,K2HPO4 8.4,KH2PO4 2.8,(NH4)2SO4 1,CaCl2 0.2,MgSO4 0.5。
以上培养基均121 ℃灭菌20 min。
1.2 实验方法
1.2.1 PNP基因的合成及基因优化
根据KlacPNP的基因序列(GenBank登录号:CP042457.1)使用软件Signal P 3.0预测序列信号肽;在基因的5′端和3′端分别引入Hind III和BamH I限制性酶切位点,交由苏州金唯智生物科技有限公司进行基因合成(PNP-YP)和密码子优化(PNP-YO),并设计鉴定引物(表1)。
表1 基因扩增引物
Table 1 Gene amplification primers
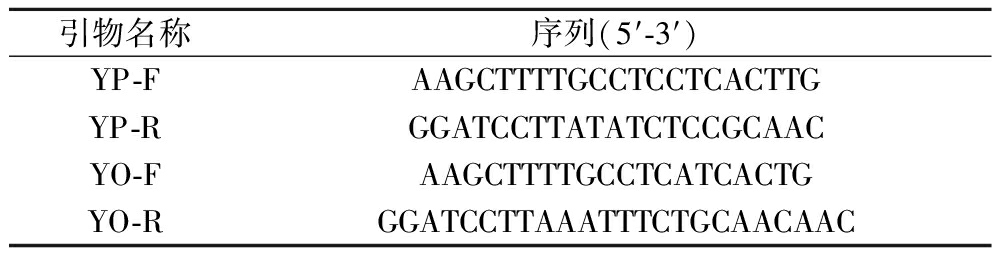
引物名称序列(5′-3′)YP-FAAGCTTTTGCCTCCTCACTTGYP-RGGATCCTTATATCTCCGCAACYO-FAAGCTTTTGCCTCATCACTGYO-RGGATCCTTAAATTTCTGCAACAAC
1.2.2 重组质粒的构建
合成的基因和载体pBSA43分别使用Hind III和BamH I进行双酶切,采用琼脂糖凝胶回收试剂盒回收目的片段。回收的目的片段按质量体积比1∶3(g∶mL)的用T4 DNA连接酶进行连接。连接产物转入到大肠杆菌DH5α感受态,加入LB培养基孵育1 h后取100 μL菌液,涂布于含100 μg/mL氨苄青霉素的LB平板上培养,12 h后挑取重组转化子,筛选鉴定,测序验证。
1.2.3 重组质粒在枯草芽胞杆菌中的表达
重组质粒在枯草芽胞杆菌WB600中的转化方法参照文献[17]。用表1中的引物进行质粒PCR鉴定,Hind Ⅲ和BamH I进行双酶切验证,获得枯草芽胞杆菌重组菌株pBSA43-YP-WB600、pBSA43-YO-WB600。
1.2.4 重组蛋白的表达和SDS-PAGE检测
分别挑取重组菌株pBSA43-YP-WB600、pBSA43-YO-WB600单菌落,接种于5 mL LB液体培养基中,培养至OD600值为0.6~0.8,按照10%接种量接至发酵产酶培养基,培养60 h后4 ℃、8 000 r/min离心10 min收集上清液,检测PNP的活力,同时进行十二烷基硫酸钠-聚丙烯酰氨凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)检测。所有试验均设置3个重复。
1.2.5 PNP活力的测定
取发酵液1 mL,8 000 r/min离心10 min,上清液即为粗酶液,酶活力参照文献[18]的方法测定。
重组PNP活力定义为:在上述反应条件下,每分钟产生1 μmol次黄嘌呤所需的酶量定义为1个酶活力单位(U/mL)。
1.2.6 PNP表达单因素试验
发酵温度37 ℃、培养基初始pH为7.0、发酵时间60 h作为初始发酵条件,分别探究发酵温度(27、32、37、42、47 ℃)、发酵时间(24、36、48、60、72 h)、初始pH值(5.0、6.0、7.0、8.0、9.0)对菌株产PNP活力的影响。
1.2.7 Box-Behnken 响应面试验设计与分析
在单因素试验的基础上,对重组菌产酶能力采用3因素3水平N=17的Box-Behnken响应面实验设计和研究,以发酵温度、培养基初始pH、发酵时间为响应面的自变量设计因素水平表(表2)。
表2 响应面因素水平表
Table 2 Response surface factors and levels

水平因素A(发酵温度)/℃B(初始pH)C(发酵时间)/h-127650032760137870
2 结果与分析
2.1 PNP基因的密码子优化及人工合成
密码子优化方式参照文献[19],在保持氨基酸序列不变的基础上,合成优化的基因序列中共替换了227个碱基,优化后的密码子序列全部是枯草芽胞杆菌的偏爱密码子,为目的基因在枯草芽胞杆菌中成功表达提供了保证。基因序列如图1所示。

图1 基因优化前后PNP基因的核苷酸序列
Fig.1 Alignment of nucleotide of the optimized and original PNP genes
注:阴影部分为相同的核苷酸。
2.2 重组质粒的构建
将合成的包含目的基因的载体和pBSA43分别用Hind III和BamH I双酶切,并利用琼脂糖凝胶回收试剂盒回收目的片段和表达载体片段,然后用T4 DNA连接酶将目的片段连接到表达载体上,并转化到大肠杆菌DH5α中,提取重组质粒PNP-YP-pBSA43、PNP-YO-pBSA43,经质粒PCR(图2),Hind Ⅲ和BamH I双酶切(图3)鉴定,说明目的基因PNP-YP、PNP-YO成功连接到表达载体pBSA43上。
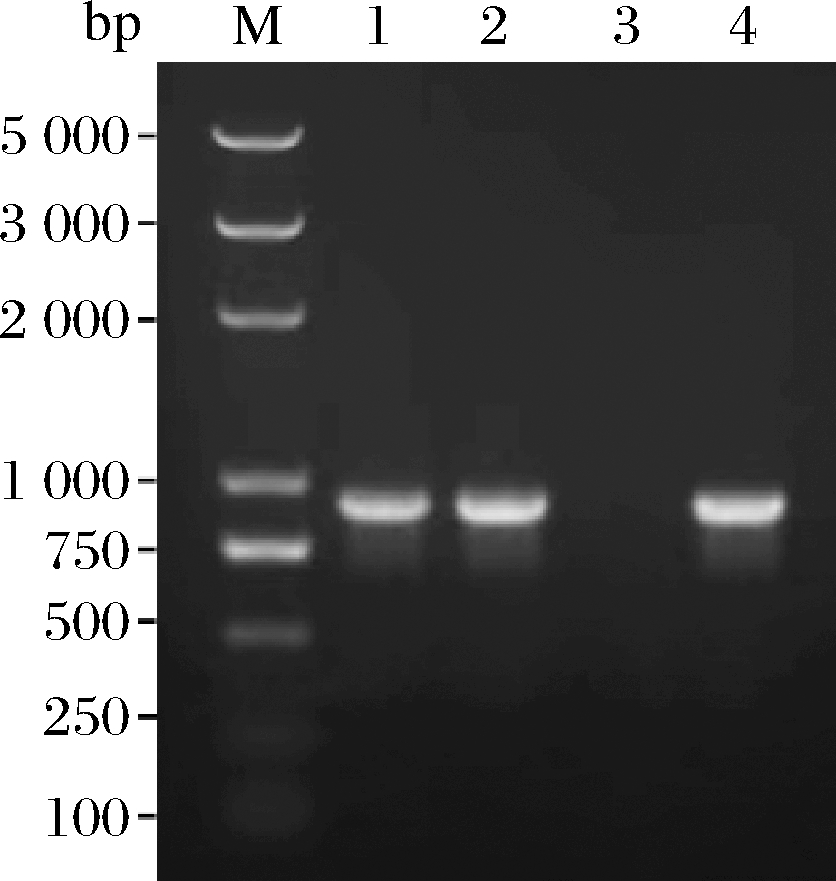
M-DNA 分子质量标准;1-PNP-YP-pBSA43 PCR 扩增产物;2-PNP-YO-pBSA43 PCR扩增产物;3-阴性对照;4-合成基因扩增产物(阳性对照)
图2 PNP基因的琼脂糖电泳检测
Fig.2 Agarose gel electrophoresis of PNP genes
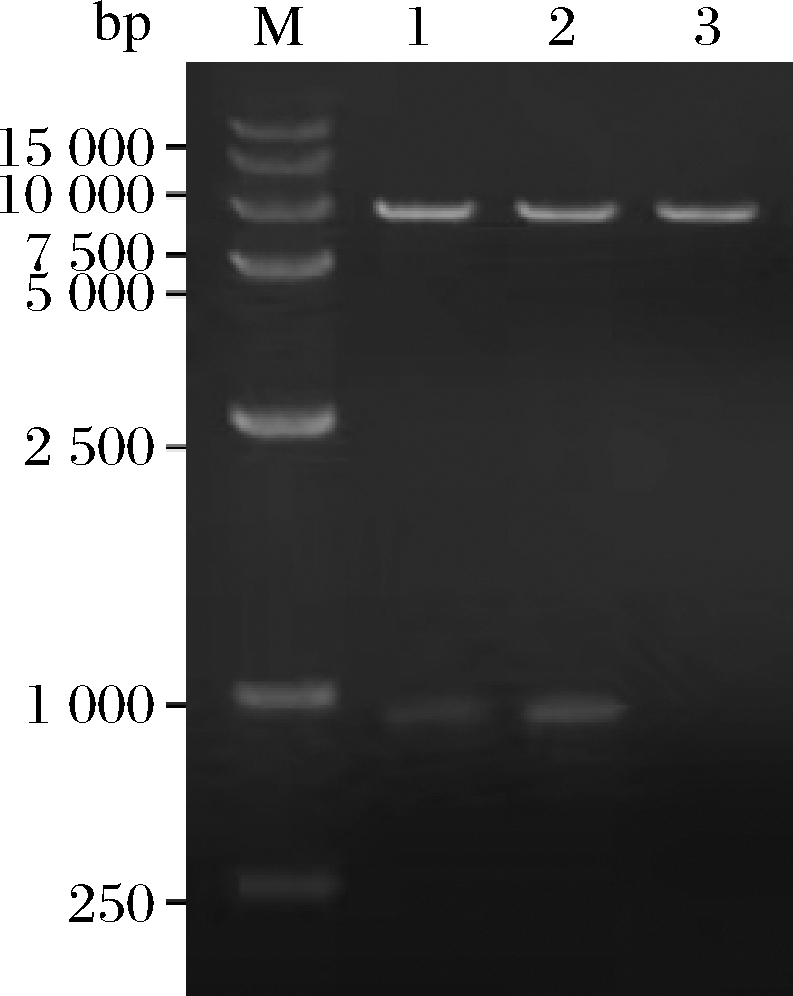
M-DNA分子质量标准;1-PNP-YP-pBSA43/Hind Ⅲ+BamH Ⅰ;2-PNP-YO-pBSA43/Hind Ⅲ+BamH Ⅰ;3-pBSA43/Hind Ⅲ+BamH Ⅰ(阴性对照)
图3 重组质粒的双酶切验证
Fig.3 Double digestion validation of recombinant plasmids
2.3 重组表达菌株的构建
鉴定正确获得的PNP-YP-pBSA43、PNP-YO-pBSA43经化学转化法转化入枯草芽胞杆菌WB600中,通过卡那霉素抗性筛选,挑取单菌落鉴定后送睿博兴科公司测序,序列正确,说明重组菌株PNP-YP-pBSA43-WB600、PNP-YO-pBSA43-WB600构建成功。
2.4 重组菌株的表达目的蛋白
2.4.1 重组PNP-YP-pBSA43-WB600、PNP-YO-pBSA43-WB600的表达
收集重组菌株PNP-YP-pBSA43-WB600和PNP-YO-pBSA43-WB600发酵60 h的上清液,以原始菌株pBSA43-WB600作为对照,进行SDS-PAGE检测,如图4所示,PNP相对分子质量约为32 kDa(泳道2、3),而对照菌株未见相应条带(泳道1)。其中,重组菌株PNP-YO-pBSA43-WB600条带比PNP-YP-pBSA43-WB600条带更粗。表明PNP-YP-pBSA43-WB600、PNP-YO-pBSA43-WB600成功在枯草芽胞杆菌中分泌表达,且密码子优化后的重组菌株PNP-YO-pBSA43-WB600比PNP-YP-pBSA43-WB600表达量更高。
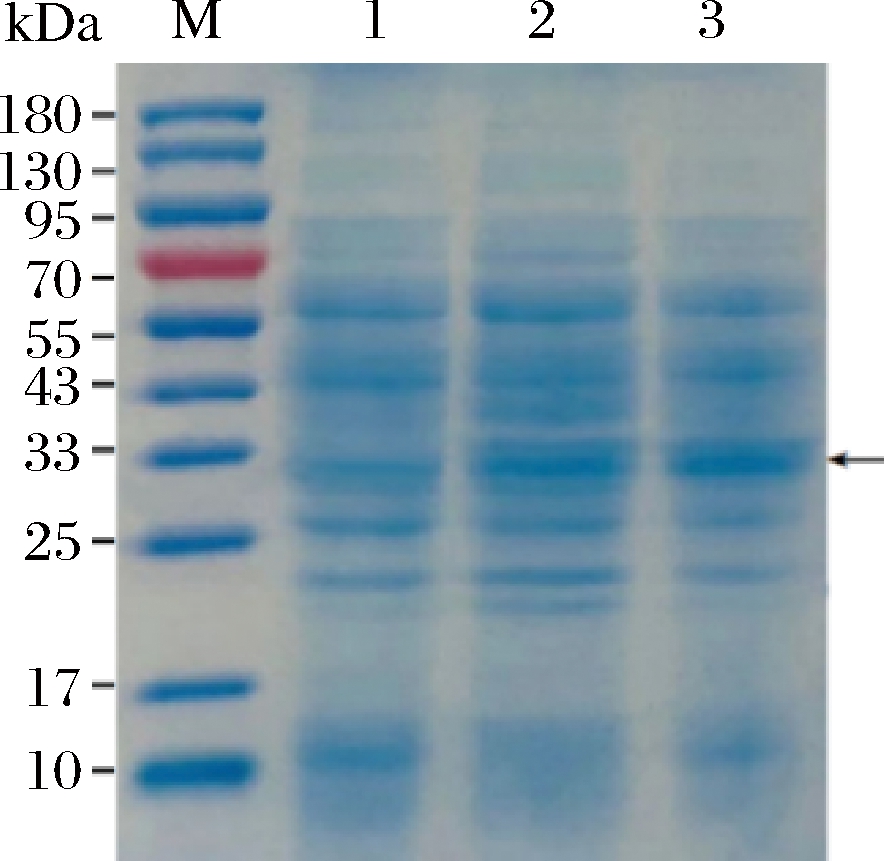
M-蛋白分子质量标准;1-pBSA43-WB600发酵液上清液(阴性对照);2-PNP-YP-pBSA43- WB600发酵液上清液;3-PNP-YO-pBSA43- WB600发酵液上清液
图4 重组菌株SDS-PAGE分析
Fig.4 SDS-PAGE analysis of recombinant strains
2.4.2 重组菌株最佳发酵时间的确定
微生物发酵过程中,利用培养基中的营养成分满足自身的生长,进而分泌蛋白,发酵时间的延长有利于胞外产物的累积。不同发酵时间的重组菌株PNP-YP-pBSA43-WB600和PNP-YO-pBSA43-WB600的PNP活力如图5所示,在24~60 h,随着发酵时间的延长,重组PNP活力逐渐增加,且密码子优化后的PNP-YO-pBSA43-WB600酶活力明显高于PNP-YP-pBSA43-WB600;发酵时间为60 h时,酶活力达到最高,此时PNP-YP-pBSA43-WB600酶活力为333.69 U/mL,PNP-YO-pBSA43-WB600酶活力为351.61 U/mL。当超过60 h后,重组PNP活力均有所下降,密码子优化后的PNP-YO-pBSA43-WB600酶活力仍然高于PNP-YP-pBSA43-WB600。因此,选择将重组菌株发酵至60 h,以实现对重组PNP的高效表达。
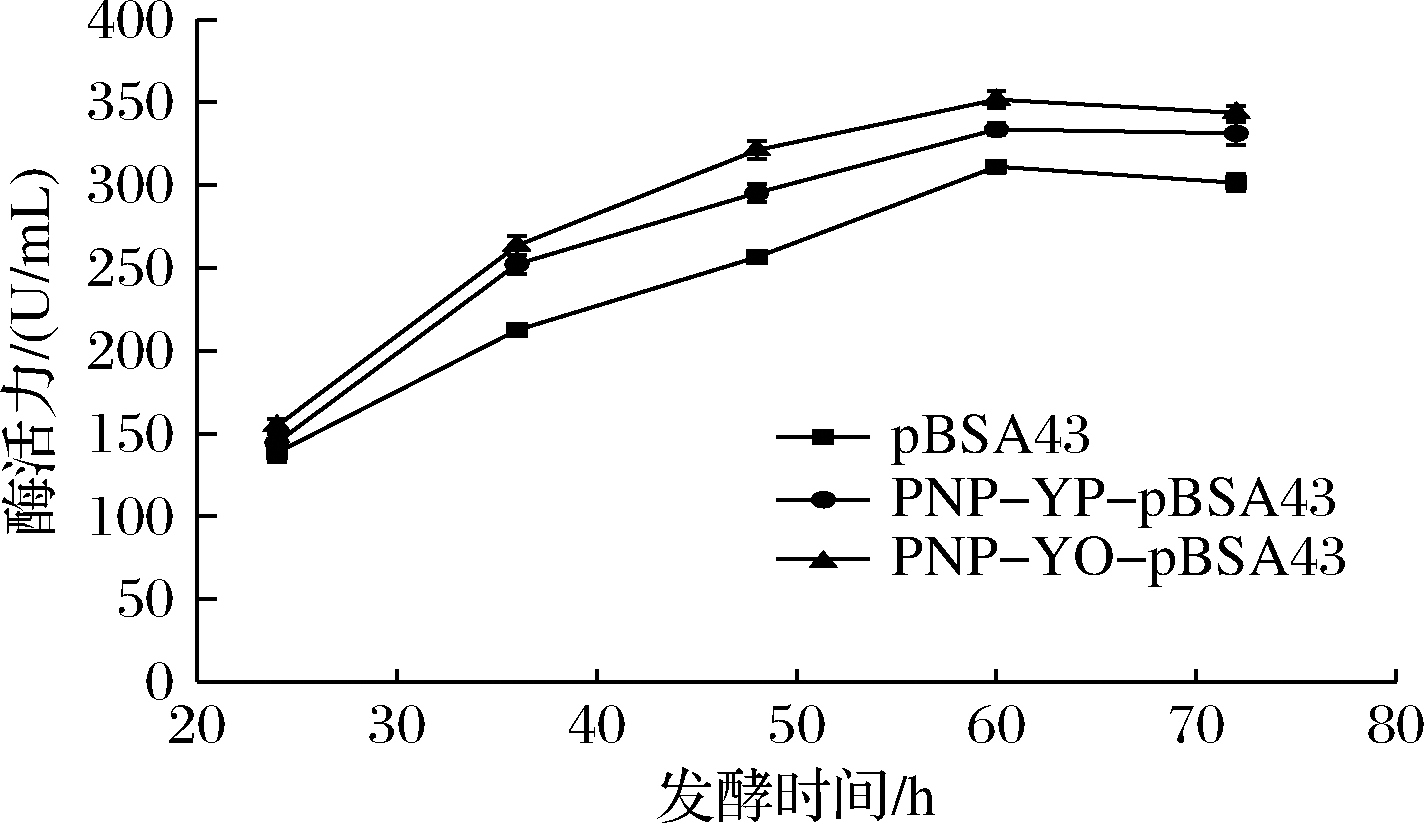
图5 发酵时间对重组PNP活力的影响
Fig.5 Effect of fermentation time on activity of recombinant PNP
2.4.3 发酵温度对重组菌株酶活力的影响
培养温度是微生物发酵中的重要条件,温度过高或过低都会影响菌体生长代谢和产酶能力。由图6可知,随着温度的升高,重组酶活力呈先升高后下降的趋势,且密码子优化后的PNP-YO-pBSA43-WB600酶活力始终高于PNP-YP-pBSA43-WB600,分析原因可能是温度低于32 ℃时,菌体生长减缓,产生酶蛋白含量少,总体酶活性不高。当发酵温度为32 ℃时,酶活力最高,PNP-YP-pBSA43-WB600为323.50 U/mL,PNP-YO-pBSA43-WB600为333.79 U/mL,由此确定重组菌株的最佳发酵温度为32 ℃。随着温度的升高,菌体生长旺盛,不利于产物的积累。当温度>37 ℃时,菌株的生长环境遭到破坏,其产酶能力下降。

图6 发酵温度对重组PNP活力的影响
Fig.6 Effect of fermentation temperature on the activity of recombinant PNP
2.4.4 初始pH对重组菌株产酶活力的影响
pH是影响细胞膜通透性和表面电荷性质的重要因素,影响着微生物对于营养物质的摄取及代谢产物的分泌,从而影响菌株生长状态和产酶能力。由图7可知,当初始pH<7时,随着pH值的升高重组菌株酶活力呈上升趋势;当初始pH=7时,酶活力达到最大值,PNP-YP-pBSA43-WB600酶活力为333.02 U/mL,PNP-YO-pBSA43-WB600为341.27 U/mL;当初始pH>7时,随着pH值的升高酶活力呈递减趋势,说明培养基初始pH升高或降低都会降低重组菌株的产酶能力,但密码子优化后的PNP-YO-pBSA43-WB600酶活力明显高于PNP-YP-pBSA43-WB600。
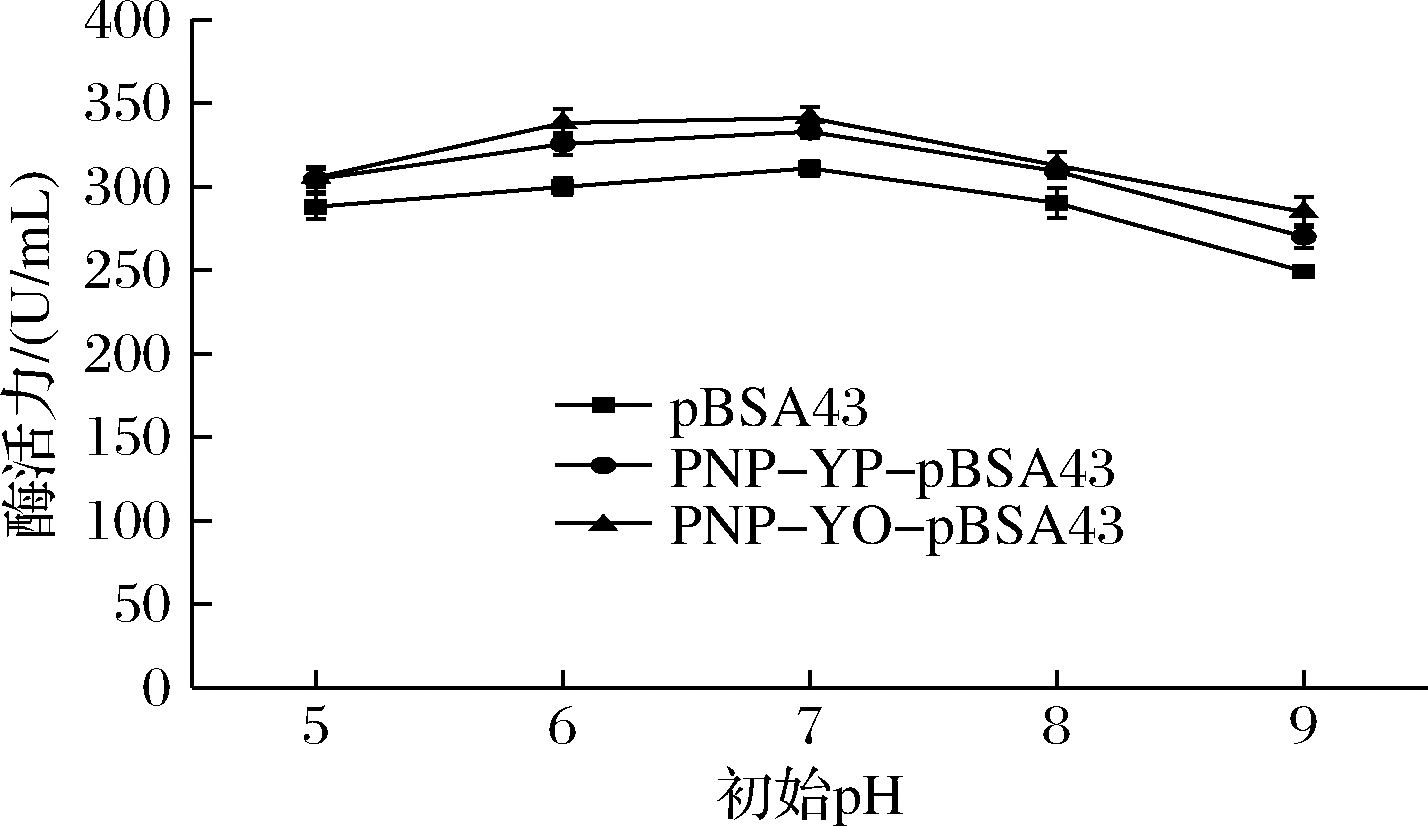
图7 培养基初始pH重组PNP活力的影响
Fig.7 Effect of initial pH of the medium on the activity of recombinant PNP
2.5 重组菌响应面实验优化
在单因素的基础上,设计了17个试验的响应分析试验,以培养基初始pH值(A)、发酵时间(B)、发酵温度(C)3个因素为自变量,发酵液酶活力为因变量,响应面实验设计与结果如表3所示。
表3 响应面实验计划表
Table 3 Response surface experiment schedule
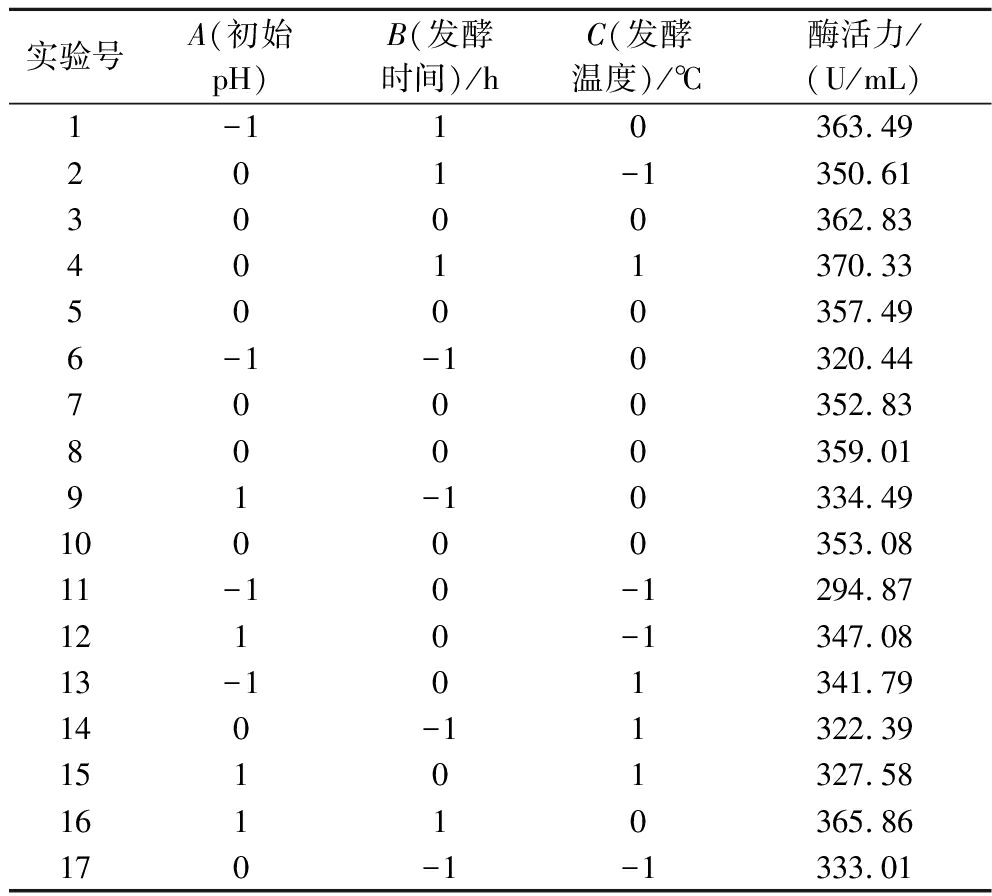
实验号A(初始pH)B(发酵时间)/hC(发酵温度)/℃酶活力/(U/mL)1-110363.49201-1350.613000362.834011370.335000357.496-1-10320.447000352.838000359.0191-10334.4910000353.0811-10-1294.871210-1347.0813-101341.79140-11322.3915101327.5816110365.86170-1-1333.01
利用Design Expert 13软件对响应面结果进行统计学分析,通过方差分析(表4)可知,在误差允许范围内,3个因素的P>F值均小于0.01,表明该模型显著且具有统计学意义。通过最小二乘法回归方程得到的重组菌PNP-YO-pBSA43-WB600产酶活力对于发酵温度、发酵初始pH和发酵时间的二次多项回归模型:酶活力(Y)=357.05+6.80A+17.49B+4.57C-2.92AB-16.6AC+7.58BC-13.62A2+2.64B2-15.60C2。当变量为1时,线性拟合系数R2=0.935 8,说明回归方程拟合度良好。
表4 回归模型方差分析结果
Table 4 Regression model ANOVA results
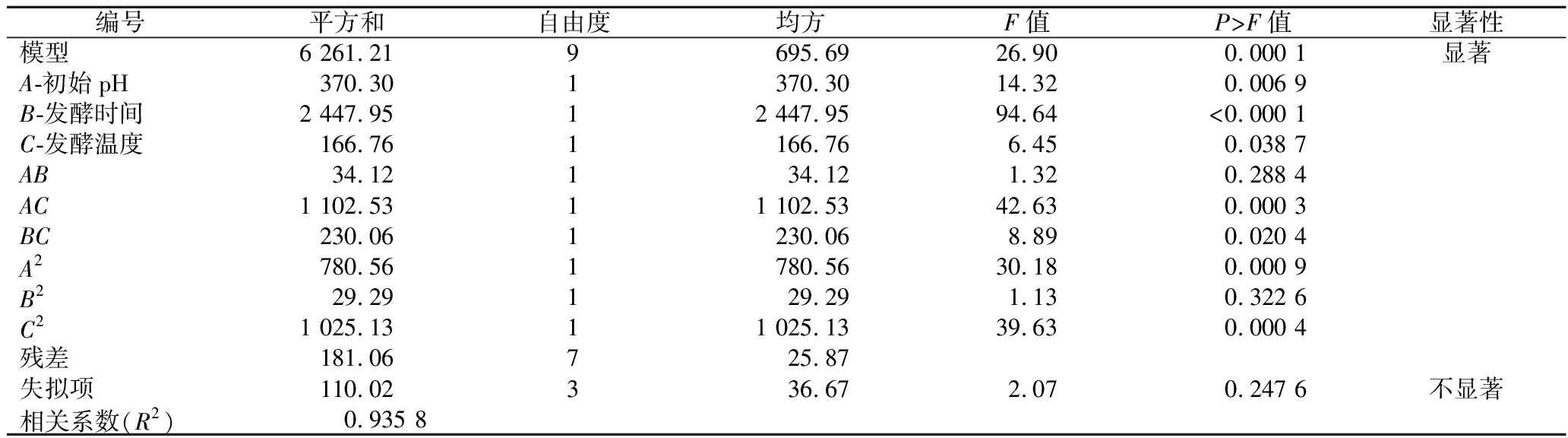
编号平方和自由度均方F值P>F值显著性模型6 261.219 695.6926.90 0.000 1显著A-初始pH370.301370.3014.320.006 9B-发酵时间2 447.9512 447.9594.64<0.000 1C-发酵温度166.761166.766.450.038 7AB34.12134.121.320.288 4AC1 102.5311 102.5342.630.000 3BC230.061230.068.890.020 4A2780.561780.5630.180.000 9B229.29129.291.130.322 6C21 025.1311 025.1339.630.000 4残差181.06725.87失拟项110.02336.672.070.247 6不显著相关系数(R2)0.935 8
响应面优化分析结果如图8所示,3个因素在不同水平对酶活力产生不同的影响,最大值处于三维模型的中心。通过回归方程求得3个因素最佳发酵条件为初始pH=6.67、发酵时间55 h、发酵温度28.7 ℃,理论预测酶活力为333.97 U/mL,在优化后的条件下摇瓶发酵酶活力达到372.79 U/mL,为预测值的111.62%。

a-培养基初始pH值和发酵时间;b-培养基初始pH值和发酵温度;c-发酵时间和发酵温度
图8 各因素交互作用响应面分析图
Fig.8 Response surface analysis of the interaction of various factors
3 结论与讨论
本研究以NCBI上登录号为CP042457.1的乳酸克鲁维酵母的PNP基因为基础,根据枯草芽胞杆菌的密码子偏好性,对KlacPNP进行密码子优化,在不改变蛋白质序列、增强目的基因在枯草芽胞杆菌中的适应性的情况下高效表达,得到了高产PNP的重组菌株。密码子优化前摇瓶发酵的酶活力为333.69 U/mL,优化后摇瓶发酵的酶活力达到351.61 U/mL。基于单因素和响应面优化确定了最佳发酵条件为:初始pH 6.67、发酵时间55 h、发酵温度28.7 ℃。最后在优化后的条件下进行发酵试验,摇瓶发酵酶活力达到372.79 U/mL,为预测值的111.62%。优化后的产量是优化前的1.12倍,得到了理想的优化结果。
前期报道中,MAHOR等[10]将klacPNP转入大肠杆菌成功表达,证明了klacPNP能够有效降低啤酒中的嘌呤含量,在klacPNP的基础上,通过点突变构建了对6-氧代嘌呤和6-氨基嘌呤具有较强底物特异性的突变体klacPNP 256D和对肌苷具有高度特异性的klacPNP 256E,二者对于啤酒中嘌呤含量都有较好的效果。TIMOFEEV等[20]将嗜热菌HB27的PNP在大肠杆菌中表达,TthPNP I高度作用于肌苷底物,每分钟降解肌苷能力330 μmol/mg,TthPNP II高度作用于腺苷底物,酶活力达到830 μmol/mg。嗜极菌嗜色还原卤单胞菌AGD 8-3[21]转化入大肠杆菌,酶活力达到28.6 U/mg。已报道人源PNP的酶活力为80 U/mg[22]。但以上重组酶表达宿主均为大肠杆菌,本试验利用枯草芽胞杆菌作为宿主细胞,通过密码子优化提高表达量,其表达乳酸克鲁维酵母来源的PNP活性有一定的提高,且对于PNP的安全生产具有重要意义。
[1] ERION M D, TAKABAYASHI K, SMITH H B, et al.Purine nucleoside phosphorylase.1.structure-function studies[J].Biochemistry, 1997, 36(39):11725-11734.
[2] TIMOFEEV V I, ZHUKHLISTOVA N E, ABRAMCHIK Y A, et al.Crystal structure of Escherichia coli purine nucleoside phosphorylase in complex with 7-deazahypoxanthine[J].Acta Crystallographica Section F Structural Biology Communications, 2018, 74(6):355-362.
[3] DESSANTI P, ZHANG Y, ALLEGRINI S, et al.Structural basis of the substrate specificity of Bacillus cereus adenosine phosphorylase[J].Acta Crystallographica Section D Biological Crystallography, 2012, 68(3):239-248.
[4] STOECKLER J D, POIROT A F, SMITH R M, et al.Purine nucleoside phosphorylase.3.reversal of purine base specificity by site-directed mutagenesis[J].Biochemistry, 1997, 36(39):11749-11756.
[5] KOELLNER G, LUI M, SHUGAR D, et al.Crystal structure of calf spleen purine nucleoside phosphorylase in a complex with hypoxanthine at 2.15 Å resolution 1[J].Journal of Molecular Biology, 1997, 265(2):202-216.
M, SHUGAR D, et al.Crystal structure of calf spleen purine nucleoside phosphorylase in a complex with hypoxanthine at 2.15 Å resolution 1[J].Journal of Molecular Biology, 1997, 265(2):202-216.
[6] LECOQ K, BELLOC I, DESGRANGES C, et al.YLR209c encodes Saccharomyces cerevisiae purine nucleoside phosphorylase[J].Journal of Bacteriology, 2001, 183(16):4910-4913.
[7] TAHIROV T H, INAGAKI E, OHSHIMA N, et al.Crystal structure of purine nucleoside phosphorylase from Thermus thermophilus[J].Journal of Molecular Biology, 2004, 337(5):1149-1160.
[8] VISSER D F, HENNESSY F, RASHAMUSE K, et al.Cloning, purification and characterisation of a recombinant purine nucleoside phosphorylase from Bacillus halodurans Alk36[J].Extremophiles, 2010, 14(2):185-192.
[9] 戴芳萍. 嘌呤核苷磷酸化酶合成利巴韦林的应用研究[D].上海:华东理工大学, 2019.
DAI F P.Study on the synthesis of ribavirin by purine nucleoside phosphorylase[D].Shanghai:East China University of Science and Technology, 2019.
[10] MAHOR D, PRIYANKA A, PRASAD G S, et al.Functional and structural characterization of purine nucleoside phosphorylase from Kluyveromyces lactis and its potential applications in reducing purine content in food[J].PLoS One, 2016, 11(10):e0164279.
[11] 李玉淼, 朱德伟, 朱洪康, 等.嘌呤核苷磷酸化酶的表达及其在啤酒中的应用[J].食品与生物技术学报, 2017, 36(12):1264-1268.
LI Y M, ZHU D W, ZHU H K, et al.Expression of purine nucleoside phosphorylase and its application in beer[J].Journal of Food Science and Biotechnology, 2017, 36(12):1264-1268.
[12] OUYANG X Y, LIU Y J, QU R D, et al.Optimizing protein-glutaminase expression in Bacillus subtilis[J].Current Microbiology, 2021, 78(5):1752-1762.
[13] HE Q, FU A Y, LI T J.Expression and one-step purification of the antimicrobial peptide cathelicidin-BF using the intein system in Bacillus subtilis[J].Journal of Industrial Microbiology &Biotechnology, 2015, 42(4):647-653.
[14] 陈灵艳, 陈先进, 蒋烨, 等.猪圆环病毒衣壳蛋白在枯草芽孢杆菌中的表达研究[J].生物技术通报, 2016, 32(5):140-145.
CHEN L Y, CHEN X J, JIANG Y, et al.Research on the expression of porcine circovirus capsid protein in the Bacillus subtilis[J].Biotechnology Bulletin, 2016, 32(5):140-145.
[15] KUMAR N, PANDEY R, PRABHU A A, et al.Genetic and substrate-level modulation of Bacillus subtilis physiology for enhanced extracellular human interferon gamma production[J].Preparative Biochemistry &Biotechnology, 2018, 48(5):391-401.
[16] 王光路, 夏俊刚, 谢希贤, 等.假交替单胞菌XM2107嘌呤核苷磷酸化酶基因克隆表达、重组蛋白纯化及酶学性质[J].微生物学报, 2010, 50(2):222-227.
WANG G L, XIA J G, XIE X X, et al.Molecular cloning, gene expression and characterization of purine nucleoside phosphorylase from Pseudoalteromonas sp.XM2107[J].Acta Microbiologica Sinica, 2010, 50(2):222-227.
[17] ZHANG X Z, ZHANG Y.Simple, fast and high-efficiency transformation system for directed evolution of cellulase in Bacillus subtilis[J].Microbial Biotechnology, 2011, 4(1):98-105.
[18] 张卫兰, 陶国庆, 刘刚, 等.氨基酸对大肠杆菌BL21产嘌呤核苷磷酸化酶的影响[J].化学与生物工程, 2010, 27(2):53-56.
ZHANG W L, TAO G Q, LIU G, et al.Influence of amino acids on the production of purine nucleoside phosphorylase by E.Coli BL21[J].Chemistry &Bioengineering, 2010, 27(2):53-56.
[19] 杨云彭, 马晓焉, 霍毅欣.密码子优化策略在异源蛋白表达中的应用[J].生物工程学报, 2019, 35(12):2227-2237.
YANG Y P, MA X Y, HUO Y X.Application of codon optimization strategy in heterologous protein expression[J].Chinese Journal of Biotechnology, 2019, 35(12):2227-2237.
[20] TIMOFEEV V I, FATEEV I V, KOSTROMINA M A, et al.The comparative analysis of the properties and structures of purine nucleoside phosphorylases from thermophilic bacterium Thermus thermophilus HB27[J].Journal of Biomolecular Structure and Dynamics, 2022, 40(8):3626-3641.
[21] ANTIPOV A N, MORDKOVICH N N, KHIJNIAK T V, et al.Cloning of nucleoside phosphorylase genes from the extremophilic bacterium Halomonas chromatireducens AGD 8-3 with the construction of recombinant producer strains of these proteins and the study of their enzymatic properties[J].Applied Biochemistry and Microbiology, 2020, 56(1):37-43.
[22] SILVA R G, CARVALHO L P S, OLIVEIRA J S, et al.Cloning, overexpression, and purification of functional human purine nucleoside phosphorylase[J].Protein Expression and Purification, 2003, 27(1):158-164.