长双歧杆菌(Bifidobacterium longum)作为人体肠道中常见的定植细菌,也是婴幼儿肠道菌群中最丰富的物种之一,对人类健康发挥着重要作用[1]。随着分类学技术的发展,B.longum的分类学地位变迁为4个亚种,分别为B.longum subsp. longum,B.longum subsp. infantis,B.longum subsp.suis和B.longum subsp.suillum[2],其中B.longum subsp.infantis与婴幼儿肠道健康密切相关,通过加速免疫系统成熟、平衡免疫系统、抑制炎症、改善肠道屏障功能、增加乙酸生成等方式对宿主有益[3]。目前B.longum subsp. infantis已列入我国《可用于食品菌种名单》和欧盟安全资格认定(qualified presumption of safety,QPS)名单中,已有菌株列入《可用于婴幼儿食品的菌种名单》[4]。YLGB-1496是1株分离自母乳的B.longum subsp. infantis,该菌株具有耐酸耐胆盐特性,能有效定植于Caco-2细胞,具有较强的调节免疫能力、抗炎[5]及抗氧化效果[6],在功能性食品、婴幼儿营养补充剂领域具有应用潜力。
研究表明益生菌的功能性和安全性特征在菌株水平具有特异性,在株水平间具有较大差异[7-8]。国内外政府监管机构及标准组织也对益生菌在菌株水平的鉴定和特征分析提出更高要求[9-10]。随着微生物鉴定技术和系统分类学的发展,菌株水平鉴定技术也持续动态更新,益生菌“种水平”分类学地位是“株水平”鉴定的必要前提[10]。菌种分类学地位的确认通常可通过形态学、生理生化特征、基质辅助激光解析电离飞行时间质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry,MALDI-TOF MS)等表型鉴定技术,并结合分子生物学16S rDNA和持家基因鉴定、平均核苷酸一致性(average nucleotide identity,ANI)分析等技术实现[11]。益生菌“株水平”精准鉴定可采用多种表型与基因型分型技术,实现益生菌鉴定溯源分析,例如基于全基因组测序(whole genome sequencing,WGS)的单核苷酸位点多态性(single nucleotide polymorphism,SNP)、多基因序列位点分析(multilocus sequence analysis, MLST)、脉冲场凝胶电泳技术(pulsed field gel electrophoresis, PFGE)、随机扩增多态性DNA(randomly amplified polymorphic DNA,RAPD)标记等技术。DURANTI等[12]对18株B.adolescentis进行cgMLST分析,通过对共有基因进行差异分析和系统发育分析,18株菌株得到良好区分。LIU等[13]采用16S rRNA、MLST、PFGE和SNP基因分型方法明确市售产品中B.animalis subsp. lactis菌株的声明准确性。基于WGS的SNP和cgMLST分析技术凭借其全面的遗传信息和较高的分辨率成为菌株鉴定分型的可靠方法[14-15]。
本研究针对B.longum subsp. infantis YLGB-1496,通过形态学、生理生化、MALDI-TOF MS等表型鉴定技术,结合全基因组测序、MLST、SNP等分子生物学技术,在菌株水平对目标菌株的分类鉴定方法开展研究,为建立适用于YLGB-1496的菌株鉴定流程奠定技术基础。通过菌株鉴定实现益生菌的精准溯源,为产品质量控制提供可靠的技术支撑,建立完善的菌株鉴定技术体系对益生菌行业的健康快速发展具有重要意义。
1 材料与方法
1.1 实验材料
1.1.1 实验菌株信息
菌株YLGB-1496-1~5均由内蒙古伊利实业集团股份有限公司提供,模式菌株B.longum subsp. infantis CICC 6069T和B.longum subsp. longum CICC 6186T由中国工业微生物菌种保藏管理中心提供。其他参比菌株分离自有明确菌株声称的益生菌菌剂、冲剂、滴剂、菌片、胶囊等产品。菌株具体信息见表1。
表1 菌株信息
Table 1 Information of strains
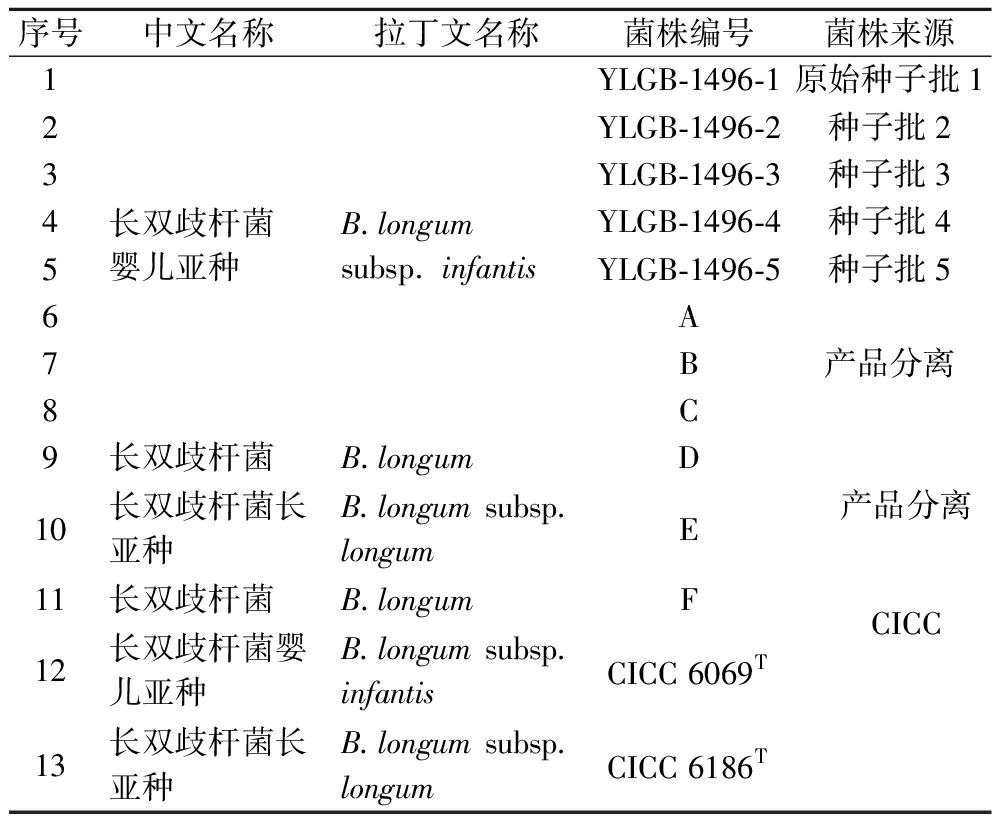
序号中文名称拉丁文名称菌株编号菌株来源12345678长双歧杆菌婴儿亚种B.longum subsp. infantisYLGB-1496-1YLGB-1496-2YLGB-1496-3YLGB-1496-4YLGB-1496-5ABC原始种子批1种子批2种子批3种子批4种子批5产品分离9长双歧杆菌B.longumD10长双歧杆菌长亚种B.longum subsp. longumE11长双歧杆菌B.longumF12长双歧杆菌婴儿亚种B.longum subsp. infantisCICC 6069T13长双歧杆菌长亚种B.longum subsp. longumCICC 6186T产品分离CICC
1.1.2 实验仪器
光学显微镜,尼康仪器有限公司;扫描电镜,日立高新技术国际贸易有限公司;MALDI-TOF MS,德国布鲁克公司;PCR仪,德国Biometra;电泳仪,美国BIO-RAD;BGISEQ-500高通量基因测序仪、BGIDL-50全自动样本加载系统,深圳华大智造科技有限公司;微量可调移液器、冷冻离心机,德国Eppendorf公司;Q800R2超声打断仪,美国Sonicator公司;Qubit 3.0核酸荧光定量仪,赛默飞世尔科技有限公司。
1.1.3 实验试剂
MRS琼脂培养基、强化梭菌培养基,美国BD公司;API 20 A培养基与试验条,法国生物梅里埃公司;BIOLOG BUA培养基、BIOLOG AN-IF接种液、BIOLOG AN MicroPlateTM,BIOLOG公司;革兰氏染色液,北京陆桥技术股份有限公司;高通量测序试剂套装BGISEQ-500RS、通用DNA文库制备试剂套装,深圳华大智造科技有限公司;Qubit®dsDNA HS Assay Kit、Qubit®ssDNA Assay Kit,赛默飞世尔科技有限公司;细菌基因组提取试剂盒E.Z.N.A.® Bacterial DNA Kit,Omega Bio-tek公司。
1.2 实验方法
1.2.1 参比菌株分离纯化
采用梯度稀释法从益生菌菌剂等样品中分离纯化目标菌株。依据产品中菌株含量信息将匀液稀释为不同稀释梯度,选择适宜稀释梯度的菌悬液涂布于MRS或强化梭菌平板上。36 ℃厌氧培养48~72 h后,选取单菌落进行MALDI-TOF MS鉴定,将鉴定为长双歧杆菌的目标菌落进行划线纯化,得到纯菌株。
1.2.2 形态学观察
菌株YLGB-1496-1接种于MRS培养基平板上,于36 ℃厌氧条件下培养48 h后,观察菌落形态,并进行革兰氏染色观察菌体形态。新鲜培养物经无菌生理盐水漂洗后用体积分数2.5%戊二醛固定过夜。离心去除戊二醛,磷酸盐缓冲液漂洗菌体,乙醇梯度脱水,CO2临界点干燥后,将样品喷金后使用扫描电镜进行菌体形态观察[16]。
1.2.3 API鉴定
菌株YLGB-1496-1于MRS培养基上36 ℃厌氧培养24 h,挑取足量菌落加入API20A培养基中制得混浊度相当于3 McFarland的菌悬液,接种API20A实验条,于36 ℃厌氧培养24~48 h后,加入附加试剂BCP、XYL、HER、H2O2判读结果,使用数据库API 20A V3.0进行鉴定。
1.2.4 BIOLOG鉴定
将菌株纯培养物转移至BUA+B培养基上,用第二代的新鲜培养物和AN-IF接种液制备菌悬液,菌悬液浊度调至65%T,然后接种BIOLOG AN MicroPlateTM鉴定板,在空气中暴露10~15 min,移至厌氧环境中36 ℃培养24 h[17]。使用BIOLOG Microstation,ML3 DC软件读取结果。
1.2.5 MALDI-TOF MS鉴定
挑取新鲜培养的实验菌株菌体加入300 μL超纯水,再加入900 μL无水乙醇涡旋振荡1 min,离心收集菌体,加入50 μL 70%(体积分数)甲酸重悬菌体细胞,加入50 μL乙腈混匀;离心取上清液加到靶板上,按照仪器说明进行鉴定分析。
1.2.6 DNA提取与WGS
采用细菌基因组提取试剂盒提取菌株的基因组DNA,使用MGIEasy通用DNA文库制备试剂套装进行建库,文库经质控合格后完成上机测序,对原始下机数据进行质控过滤:去除质量值连续≤20的碱基数达到40%的reads、去除含N碱基比例≥10%的reads、去除接头和重复污染。使用SPAdes 3.11.0软件进行拼接组装获得基因组序列。
1.2.7 基于WGS的ANI/数字DDH分析
以模式菌株B.longum subsp. longum JCM 1217T(GCA_000196555.1)、B.longum subsp. infantis ATCC 15697T (GCA_000020425.1)、B.longum subsp. suis DSM 20211T (GCA_900103055.1)、B.longum subsp. suillum Su 851T (GCA_016882605.1)为参考菌株,利用fastaANI(v 1.3)软件对测序菌株进行ANI分析。通过GGDC在线分析数据库(https://www.dsmz.de/services/online-tools/genome-to-genome-distance-calculator-ggdc)计算实验菌株和参考菌株的数字DDH值。
1.2.8 基于WGS的多基因序列位点分析(cgMLST)分析
利用Prodigal(v2.6.3)软件进行基因预测,对菌株的蛋白基因进行聚类分析CD-HIT(v 4.6.6),提取样品中共有基因,基于共有基因矩阵构建系统进化树。用TreeBeST(v 1.9.2)的PHYML(最大似然法)算法构建系统进化树,bootstraps参数设置为1 000。
1.2.9 基于WGS的SNP分析
利用MUMmer将实验菌株序列与参考序列进行比对,得到潜在SNP位点;提取参考序列SNP位点两边各100 bp序列,然后用BLAT将提取的序列和组装结果进行比对,验证SNP位点。采用BLAST、TRF、Repeatmask软件预测重复序列位点,过滤去除重复区SNP,最后得到可靠的SNP。
2 结果与分析
2.1 形态学观察
菌株YLGB-1496-1在MRS培养基中,36 ℃厌氧培养48 h,菌落白色、圆形、表面湿润、不透明、边缘整齐(图1-a)。光学显微镜下观察,菌体呈杆状,(0.5~0.6) μm×(1.2~3.8) μm,单个或成对排列,革兰氏阳性(图1-b)。扫描电镜观察,菌体呈不规则杆状,(0.4~0.6) μm×(1.3~4.7) μm,单个或成对排列(图1-c和图1-d)。菌株YLGB-1496-1的菌落和菌体形态符合长双歧杆菌典型特征[18]。

a-菌落形态;b-菌体显微形态;c、d-菌体扫描电镜形态
图1 菌株YLGB-1496-1菌落和菌体形态
Fig.1 Colony and morphology of strain YLGB-1496-1
2.2 生理生化特征
经API20A试剂条和BIOLOG鉴定系统对菌株YLGB-1496-1的酶活性、利用碳源产酸等生理生化特征进行检测。如表2所示,菌株YLGB-1496-1能水解明胶和七叶灵,过氧化酶反应阴性,能利用葡萄糖、乳糖、蔗糖、甘露醇、麦芽糖等,经API20A系统鉴定为双歧杆菌属(Bifidobacterium sp.)。
表2 菌株YLGB-1496-1的API分析结果
Table 2 API analysis results of strain YLGB-1496-1

结果吲哚产生-脲酶-葡萄糖+甘露醇+乳糖+蔗糖+麦芽糖+水杨苷-木糖+阿拉伯糖-明胶水解-七叶灵水解-甘油-纤维二糖-甘露糖+松三糖-棉子糖+山梨醇-鼠李糖-海藻糖-接触酶-芽胞/孢子-革兰氏染色+
注:“+”,阳性;“-”,阴性。
BIOLOG主要根据菌株对糖、醇、酸、酯、胺和大分子聚合物等95种碳源的利用情况进行鉴定。如表3所示,经BIOLOG AN鉴定板鉴定为B.longum subsp. infantis。目标菌株鉴定至亚种水平,与鉴定系统中生理生化反应及数据库中菌株收集的数量有关。
表3 菌株YLGB-1496-1的BIOLOG分析结果
Table 3 BIOLOG analysis results of strain YLGB-1496-1

结果N-乙酰-D-半乳糖胺-N-乙酰-D-葡糖胺-N-乙酰-β-D-甘露糖胺+w核糖醇-苦杏仁苷-D-阿拉伯糖醇-熊果苷-D-纤维二糖-α-环糊精-β-环糊精-糊精+甜醇-i-赤藓糖醇-D-果糖+L-岩藻糖+D-半乳糖+D-半乳糖醛酸+wD-龙胆二糖-D-葡萄糖酸-D-氨基葡萄糖酸-α-D-葡萄糖+葡萄糖-1-磷酸盐-葡萄糖-6-磷酸盐+w甘油-D,L-α-甘油磷酸盐-m-肌醇-α-D-乳糖+w乳果糖+麦芽糖+麦芽三糖+D-甘露醇-D-甘露糖+D-松三糖-D-蜜二糖+3-甲基-D-葡萄糖-α-甲基-D-半乳糖苷+β-甲基-D-半乳糖苷+α-甲基-D-葡萄糖苷+β-甲基-D-葡萄糖苷-异麦芽酮糖+D-棉子糖+L-鼠李糖-水杨苷-D-山梨醇-水苏糖+蔗糖+wD-海藻糖-松二糖+乙酸-甲酸-反丁烯二酸-乙醛酸-α-羟基丁酸-β-羟基丁酸-衣康酸-α-酮丁酸-α-酮戊酸-D,L-乳酸-L-乳酸-D-乳酸甲酯-D-苹果酸-L-苹果酸-丙酸-丙酮酸-丙酮酸甲酯-D-糖质酸-琥珀酰胺酸-α-琥珀酸-琥珀酸单甲酯-m-酒石酸-尿刊酸-L-丙氨酰胺-L-丙氨酸-L-丙氨酰-L-谷氨酰胺-L-丙氨酰-L-组氨酸-L-丙氨酰-L-苏氨酸-L-天冬酰胺-L-谷氨酸-L-谷氨酰胺-甘氨酰-L-天冬氨酸-甘氨酰-L-谷氨酰胺-甘氨酰-L-甲硫氨酸-甘氨酰-L-脯氨酸-L-甲硫氨酸-L-苯丙氨酸-L-丝氨酸-L-苏氨酸-L-缬氨酸-L-缬氨酸+L-天冬氨酸-2′-脱氧腺苷-肌苷+w胸苷-尿苷+w5′-单磷酸腺苷-5′-单磷酸尿苷-
注:“+”,阳性;“-”,阴性;“+w”,弱阳性。
2.3 菌株YLGB-1496及参比菌株MALDI-TOF MS鉴定结果
MALDI-TOF MS是近年来新兴的微生物快速鉴定系统,通过检测微生物核糖体等高丰度稳定表达的特征蛋白指纹图谱,与已知菌种蛋白指纹图谱比对分析,进行快速菌种鉴定。该方法已进入GB/T 33682—2017《基质辅助激光解析电离飞行时间 质谱鉴别微生物方法通则》,并且在益生菌菌种鉴定方面有较好的应用,已有研究利用此技术对动物双歧杆菌的2个亚种实现区分[19]。MALDI-TOF MS数据库中包含25种双歧杆菌蛋白指纹图谱,数据库中菌株的种类和数量越丰富,指纹图谱越完善,得到的鉴定结果也越可靠[20]。如表4所示,MALDI-TOF MS可明确YLGB-1496菌株及参比菌株的种水平,均鉴定为B.longum,分值均达到2.0以上,鉴定结果具有较高的可信度。
表4 MALDI-TOF MS鉴定结果
Table 4 Identification results of MALDI-TOF MS
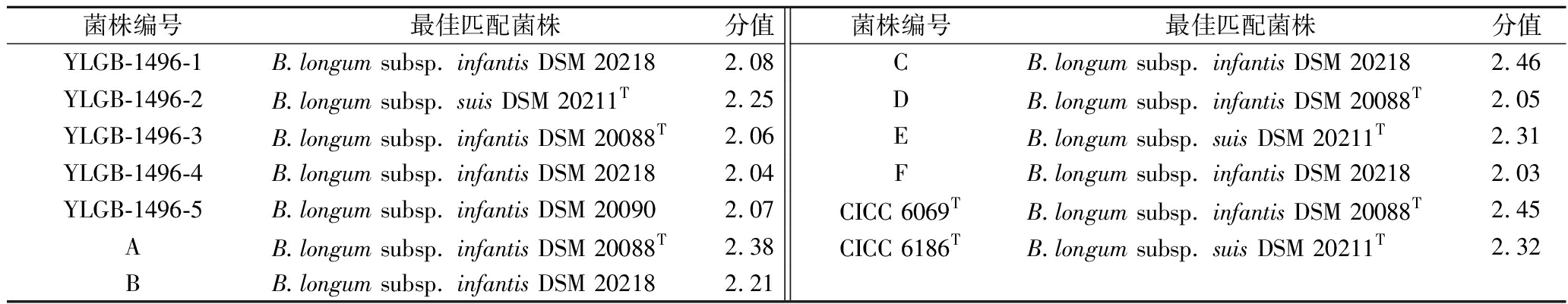
菌株编号最佳匹配菌株分值菌株编号最佳匹配菌株分值YLGB-1496-1B.longum subsp. infantis DSM 202182.08CB.longum subsp. infantis DSM 202182.46YLGB-1496-2B.longum subsp. suis DSM 20211T2.25DB.longum subsp. infantis DSM 20088T2.05YLGB-1496-3B.longum subsp. infantis DSM 20088T2.06EB.longum subsp. suis DSM 20211T2.31YLGB-1496-4B.longum subsp. infantis DSM 202182.04FB.longum subsp. infantis DSM 202182.03YLGB-1496-5B.longum subsp. infantis DSM 200902.07CICC 6069TB.longum subsp. infantis DSM 20088T2.45AB.longum subsp. infantis DSM 20088T2.38CICC 6186TB.longum subsp. suis DSM 20211T2.32BB.longum subsp. infantis DSM 202182.21
注:分值在2.00~3.00时表明鉴定结果为高可信度鉴定。
2.4 ANI分析结果
ANI于2007年由GORIS等[21]提出,指2个基因组序列之间的整体相似性,将ANI值95%~96%作为细菌菌种鉴定的有效判定指标。模式菌株B.longum subsp.infantis ATCC 15697T(ref1)、B.longum subsp.longum JCM 1217T(ref2)、B.longum subsp. suis DSM 20211T(ref3)和B.longum subsp.suillum Su 851T(ref4)的全基因组序列作为参考序列。如表5所示,实验菌株均与3个及以上的参考模式菌株全基因组的ANI比值在95%以上,可确认其种水平分类学地位,所有菌株均鉴定为B.longum。5株YLGB-1496菌株与B.longum subsp.infantis ATCC 15697T的ANI值均大于98%,具有更高的相似性,亲缘关系更近。
表5 ANI分析结果
Table 5 Results of ANI analysis
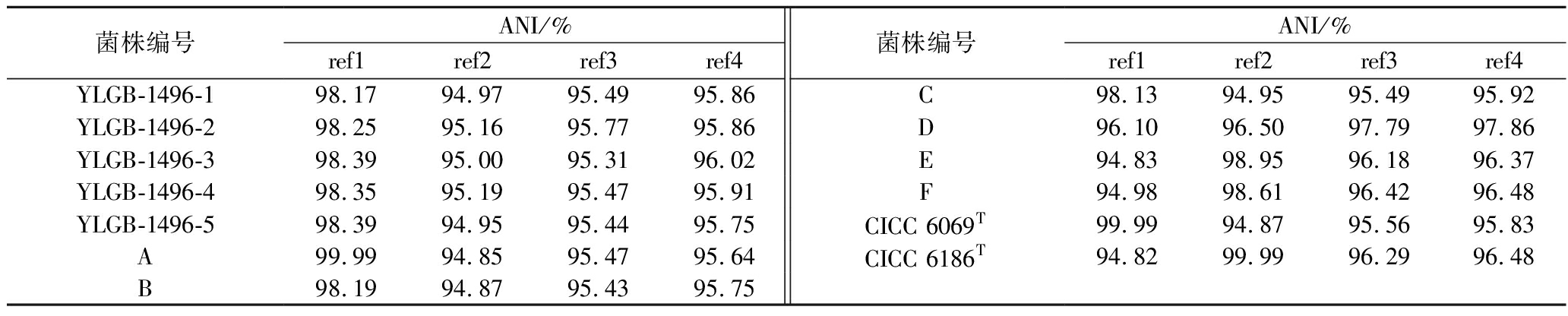
菌株编号ANI/%ref1ref2ref3ref4菌株编号ANI/%ref1ref2ref3ref4YLGB-1496-198.1794.9795.4995.86C98.1394.9595.4995.92YLGB-1496-298.2595.1695.7795.86D96.1096.5097.7997.86YLGB-1496-398.39 95.0095.3196.02E94.8398.9596.1896.37YLGB-1496-498.35 95.1995.47 95.91F94.9898.6196.4296.48YLGB-1496-598.39 94.95 95.44 95.75CICC 6069T99.9994.8795.5695.83A99.9994.8595.4795.64CICC 6186T94.8299.9996.2996.48B98.1994.8795.4395.75
2.5 数字DNA-DNA杂交(digital DNA-DNA hybridization,dDDH)分析结果
dDDH分析可作为对ANI分析结果进一步补充,选取相同的模式菌株的全基因组序列作为参考序列进行分析,结果如表6所示。研究表明通过dDDH技术可有效将目标菌株鉴定至亚种水平,其判定阈值为79%~80%,DNA-DNA杂交值>79%为同一亚种[22]。5株菌株YLGB-1496与B.longum subsp. infantis ATCC 15697T的dDDH值均>85%,与其他参考模式菌株的dDDH值均<70%,鉴定为B.longum subsp. infantis。
表6 dDDH分析结果
Table 6 Results of dDDH analysis
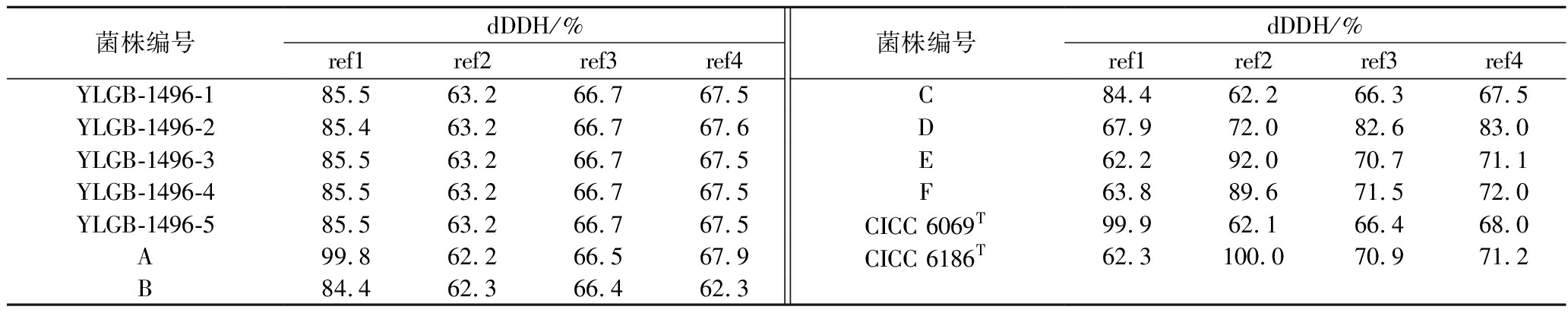
菌株编号dDDH/%ref1ref2ref3ref4菌株编号dDDH/%ref1ref2ref3ref4YLGB-1496-185.563.266.767.5C84.462.266.367.5YLGB-1496-285.463.266.767.6D67.972.082.683.0YLGB-1496-385.563.266.767.5E62.292.070.771.1YLGB-1496-485.563.266.767.5F63.889.671.572.0YLGB-1496-585.563.266.767.5CICC 6069T99.962.166.468.0A99.862.266.567.9CICC 6186T62.3100.070.971.2B84.462.366.462.3
在菌株株水平鉴定之前,基于最新方法在种或亚种水平上准确分类必不可少。结合形态学、生理生化、MALDI-TOF MS表型方法以及WGS-ANI、dDDH对菌株YLGB-1496进行了鉴定,可明确目标菌株的亚种水平分类学地位。目标菌株YLGB-1496-1~5鉴定为B.longum subsp. infantis。表型鉴定方法也可表征菌株相关的代谢能力,对菌株发酵工艺过程的优化具有指导性,但细菌处于的生长期、数据库的完善程度都会影响鉴定结果。全基因组测序目前被认为是菌种鉴定最有效和准确的方法,但其需要更高的成本和生物信息学知识来分析数据。
2.6 cgMLST分析结果
MLST是基于多位点序列的分型方法,通过串联多个基因内部片段序列,对菌株的等位基因进行多样性比较,通过不同的序列型分析菌株间的关系,在流行病学监测和进化研究方面有较广泛的应用[23]。cgMLST主要通过对菌株间的所有共有基因进行分型,可以得到较好的菌株区分效果。为进一步分析亚种内不同菌株间差异,选取EZBioCloud上28株B.longum subsp. infantis全基因组序列以扩充样本量。将所有菌株进行序列比对得到共有核心基因1 059个(图2)。以共有基因为基础,系统发育分析结果表明(图3),原始种子批菌株YLGB-1496-1与不同种子批来源菌株YLGB-1496-2~5聚类在一起,处于同一分支。5株YLGB-1496目标菌株与其他不同株和不同亚种的参比菌株可以有效区分,因此通过cgMLST可以实现目标菌株的株水平分型鉴定。
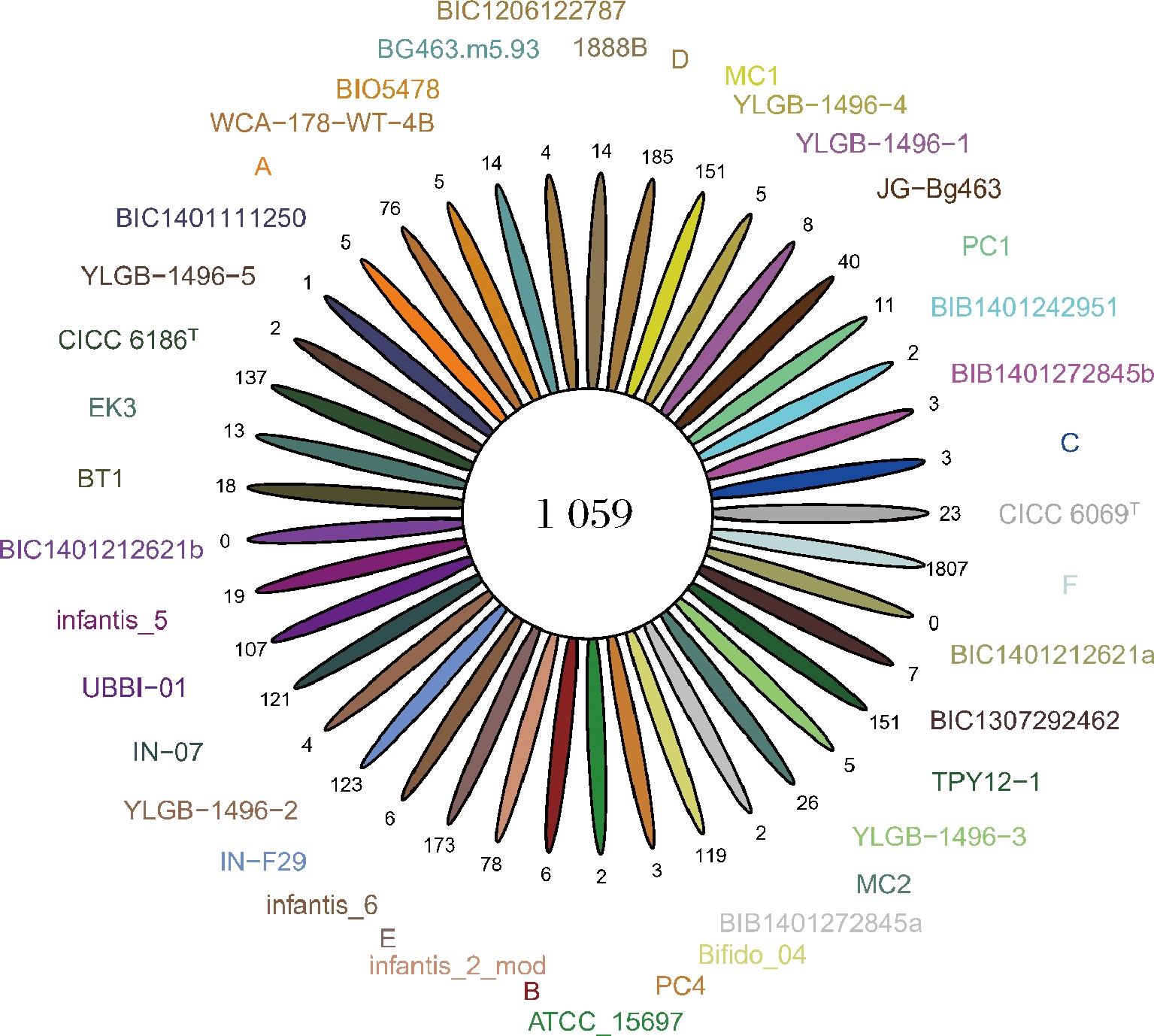
图2 菌株的共有基因和特有基因分布
Fig.2 Distribution of common and unique genes in strains
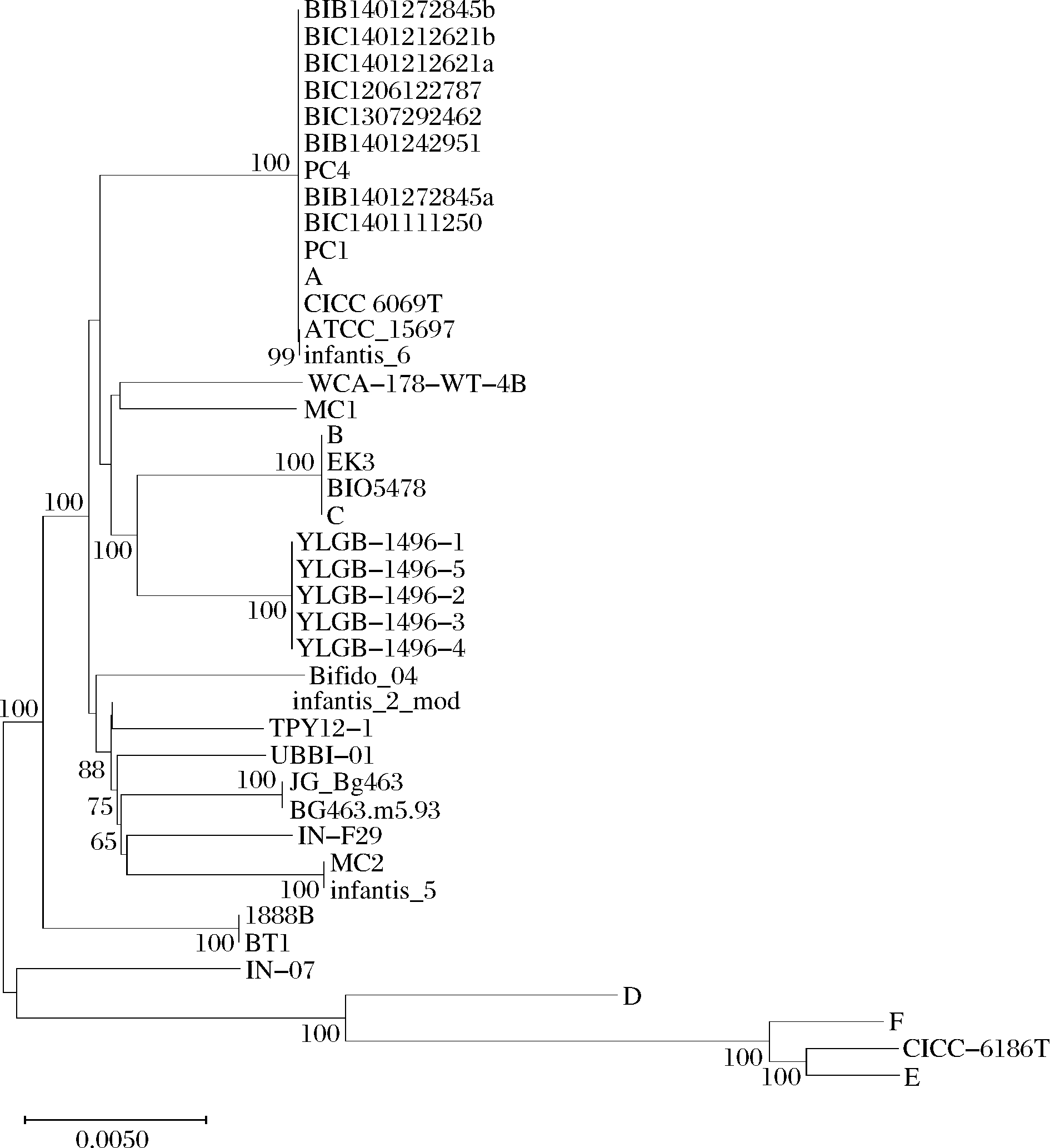
图3 菌株的cgMLST系统发育分析
Fig.3 Phylogenetic analysis of strains based on cgMLST
2.7 SNP分析结果
以YLGB-1496-1为参考,将菌株reads序列进行比对分析,SNP分析结果如表7所示。YLGB-1496-2的SNP差异为0,YLGB-1496-3的SNP差异为15,YLGB-1496-4的SNP差异为30,YLGB-1496-5的SNP差异为5,与参考序列的SNP差异较小,其他参比菌株的SNP差异均大于18 000。结合EZBioCloud上28株B.longum subsp.infantis全基因组序列分析,结果表明所有菌株与YLGB-1496-1的SNP差异均大于20 000。基于SNP差异的系统发育分析结果与cgMLST分析一致,如图4所示,5株YLGB-1496的基因序列聚集在同一分支,与其他菌株有明显区分。
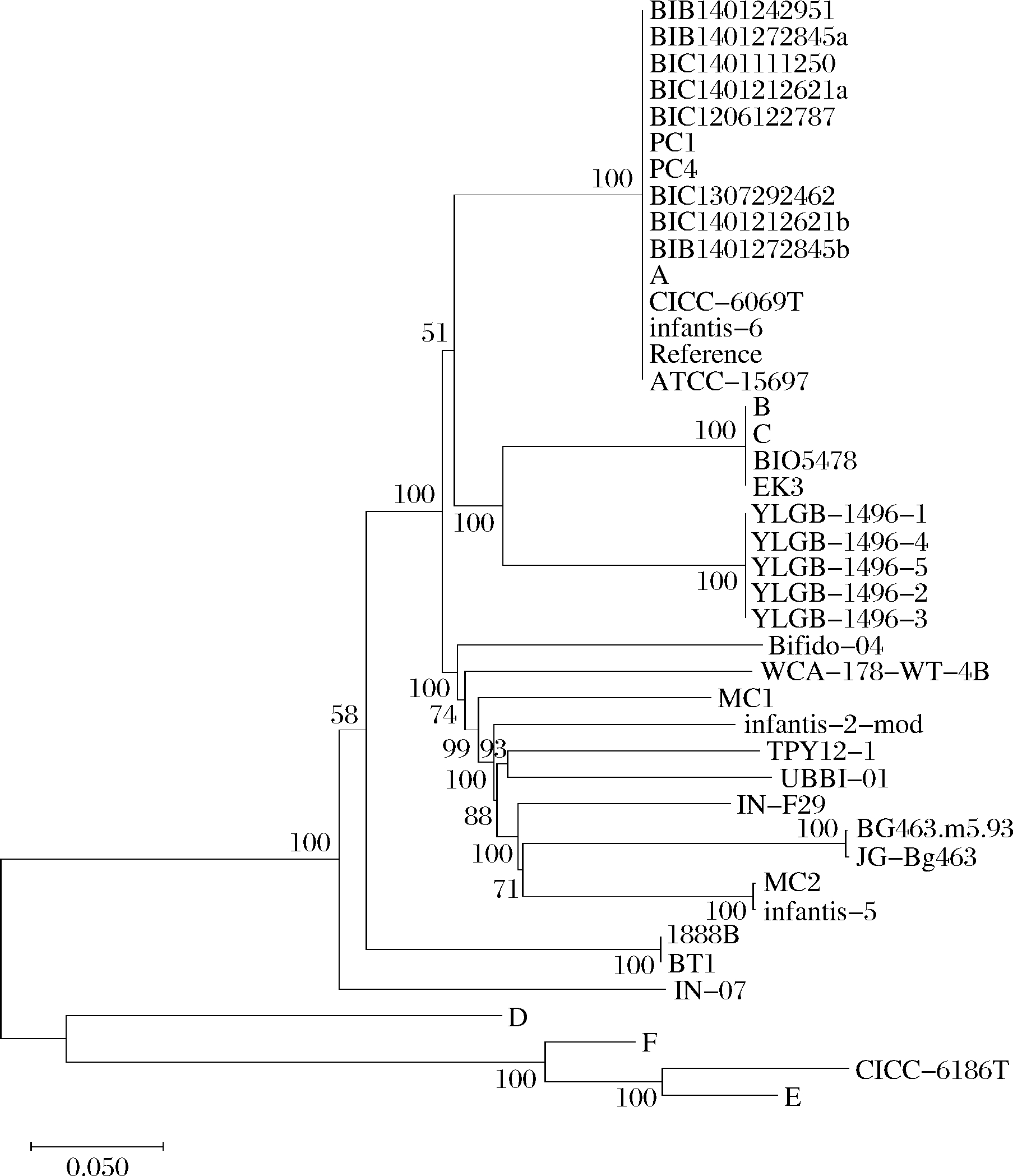
图4 菌株的SNP系统发育分析
Fig.4 Phylogenetic analysis of strains based on SNP
表7 SNP分析结果
Table 7 Results of SNP analysis
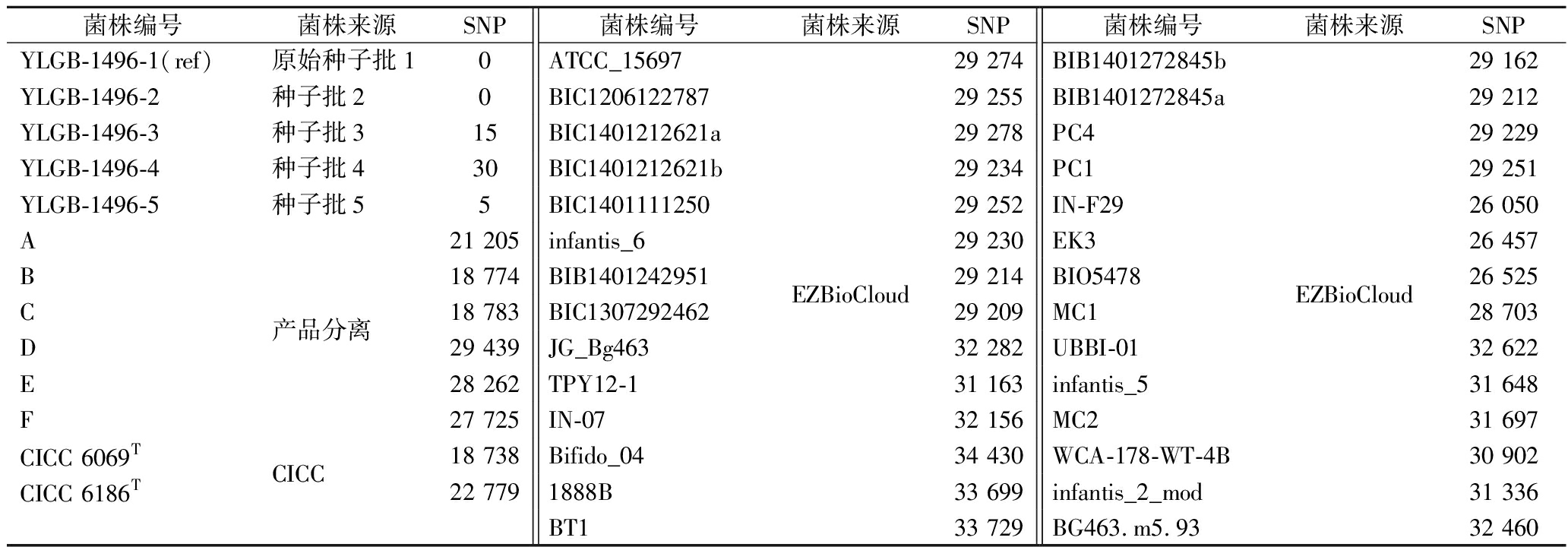
菌株编号菌株来源SNP菌株编号菌株来源SNP菌株编号菌株来源SNPYLGB-1496-1(ref)YLGB-1496-2YLGB-1496-3YLGB-1496-4YLGB-1496-5ABCDEFCICC 6069TCICC 6186T原始种子批1种子批2种子批3种子批4种子批5产品分离CICC0ATCC_156970BIC120612278715BIC1401212621a30BIC1401212621b5BIC140111125021 205infantis_618 774BIB140124295118 783BIC130729246229 439JG_Bg46328 262TPY12-127 725IN-0718 738Bifido_0422 7791888BBT1EZBioCloud29 274BIB1401272845b29 255BIB1401272845a29 278PC429 234PC129 252IN-F2929 230EK329 214BIO547829 209MC132 282UBBI-0131 163infantis_532 156MC234 430WCA-178-WT-4B33 699infantis_2_mod33 729BG463.m5.93EZBioCloud29 16229 21229 22929 25126 05026 45726 52528 70332 62231 64831 69730 90231 33632 460
目前SNP分析在益生菌菌株鉴定领域还缺少统一公认的评判标准,该方法的判定标准与目标菌株及参考菌株的种类与数量密切相关。研究表明通过对1 143株L.monocytogenes的SNP分析,21个SNP差异以内可认为菌株具有同源性[15]。本研究5株目标菌株YLGB-1496中SNP差异最大为30,注释到YLGB-1496-1与YLGB-1496-4具有28个差异基因位点。统计株内(5株)、株间(32株)和亚种内(4株)在差异位点的单核苷酸位点性态,计算4种核苷酸的占比结果如图5所示,其中13个位点(3~6,11,13,16,18,19,22,23,25,27)是该种内的多态性位点,在菌株间的多态性变化属于种内正常现象。研究表明乳酸菌菌株经过连续多次传代后会引起同株内的SNP差异,但其表型特征基本稳定[24-25]。因此基于本研究的菌株数量及数据结果分析,目标菌株YLGB-1496具有30个以内SNP差异,可与其他菌株良好区分,SNP分析可实现YLGB-1496的菌株分型鉴定。

图5 YLGB-1496-1与YLGB-1496-4 的SNP差异位点统计
Fig.5 Analysis of SNP difference sites between YLGB-1496-1 and YLGB-1496-4
3 结论与讨论
本研究建立了长双歧杆菌婴儿亚种YLGB-1496的“株”水平鉴定方法体系:通过形态学、生理生化、MALDI-TOF、基于WGS的ANI和数字DDH分析实现目标菌株YLGB-1496的亚种水平鉴定;进一步通过基于WGS的SNP和cgMLST分析有效区分目标菌株YLGB-1496与其他不同株、不同亚种的参比菌株,以及有效对不同批次来源菌株实现分型溯源,从而实现目标菌株YLGB-1496的株水平鉴定。本研究构建的目标菌株YLGB-1496鉴定技术流程,为益生菌菌株精确鉴定技术体系的构建提供了有效数据支撑和实践参考。在株水平上鉴定益生菌方法的选择,应根据菌株自身特点、不同用途或应用场景选择适当的方法。SNP和cgMLST作为分辨率较高的益生菌菌株鉴定技术,广泛应用于菌株溯源及新菌株的区分,其适用范围、判定标准与研究涵盖的目标菌株、参考菌株数量密切相关。随着试验菌株样本量的不断扩充,建立的益生菌菌株鉴定流程和评价标准将更具代表性、覆盖度和统计学意义。未来基于cgMLST和SNP分析结果,可进一步分析目标菌株YLGB-1496特异性核苷酸位点,设计株水平特异性引物,实现YLGB-1496菌株的快速精准鉴定和定量检测分析。
[1] 吴诗寅, 蔡美琴.长双歧杆菌与婴幼儿肠道健康的研究进展[J].临床儿科杂志, 2022, 40(9):715-720.
WU S Y, CAI M Q.Advances in Bifidobacterium longum and intestinal health during infancy[J].Journal of Clinical Pediatrics, 2022, 40(9):715-720.
[2] YANOKURA E, OKI K, MAKINO H, et al.Subspeciation of Bifidobacterium longum by multilocus approaches and amplified fragment length polymorphism:Description of B.longum subsp.suillum subsp.nov., isolated from the faeces of piglets[J].Systematic and Applied Microbiology, 2015, 38(5):305-314.
[3] UNDERWOOD M A, GERMAN J B, LEBRILLA C B, et al.Bifidobacterium longum subspecies infantis:Champion colonizer of the infant gut[J].Pediatric Research, 2015, 77(1):229-235.
[4] 中国卫生部, 可用于食品的菌种名单和可用于婴幼儿食品的菌种名单[Z]. 2010, 65号.
Ministry of Health,P.R.China, List of strains that can be used for food and List of strains that can be used in food for infants and young children[Z].2010, No.65.
[5] CHIU Y H, TSAI J J, LIN S L, et al.Characterisation of bifidobacteria with immunomodulatory properties isolated from human breast milk[J].Journal of Functional Foods, 2014, 7:700-708.
[6] 谢佩珊, 郭仲伟, 蔡宜钧, 等.含抗氧化的乳酸菌发酵物的食品组合物以及医药组合物:中国, CN112167345A[P].2021-01-05.
XIE P S, GUO Z W, CAI Y J, et al.Food compositions and pharmaceutical compositions containing Lactic acid bacteria fermentation with antioxidant properties:China, CN112167345A[P].2021-01-05.
[7] FAO/WHO.Joint FAO/WHO Working Group Report on Drafting Guidelines for the Evaluation of Probiotics in Food[R].London, Ontario, Canada, April 30 and May 1, 2002.
[8] HILL C, GUARNER F, REID G, et al.The International Scientific Association for Probiotics and Prebiotics consensus statement on the scope and appropriate use of the term probiotic[J].Nature Reviews Gastroenterology &Hepatology, 2014, 11(8):506-514.
[9] T/CIFST009, 食品用益生菌通则[S].中国食品科学技术学会, 2022-06-16.
T/CIFST009, General standard of probiotics for food use[S].Chinese Institute of Food Science and Technology, 2022-06-16.
[10] YAO S,BOURDICHON F,KOWALCZYK M, et al.IDF Bulletin of the IDF No.513/2021, Identification of Probiotics at the strain level-Guidance document.2021.
[11] 曹艳花, 程坤, 刘艺茹, 等.直接饲喂微生物和发酵制品生产菌株鉴定概述[J].食品与发酵工业, 2023,49(13):328-333.
CAO Y H, CHENG K, LIU Y R, et al.Overview of identification of direct feed microorganisms and fermentation production strains[J].Food and Fermentation Industries, 2023,49(13):328-333.
[12] DURANTI S, MILANI C, LUGLI G A, et al.Evaluation of genetic diversity among strains of the human gut commensal Bifidobacterium adolescentis[J].Scientific Reports, 2016, 6:23971.
[13] LIU Y, CHEN W W, YU Y, et al.Identification and characterisation of bifidobacteria in infant formula milk powder obtained from the Chinese market[J].International Dairy Journal, 2018, 80:8-16.
[14] MILANI C, DURANTI S, LUGLI G A, et al.Comparative genomics of Bifidobacterium animalis subsp.lactis reveals a strict monophyletic bifidobacterial taxon[J].Applied and Environmental Microbiology, 2013, 79(14):4304-4315.
[15] PIGHTLING A W, PETTENGILL J B, LUO Y, et al.Interpreting whole-genome sequence analyses of foodborne bacteria for regulatory applications and outbreak investigations[J].Frontiers in Microbiology, 2018, 9:1482.
[16] 翟磊, 谢九艳, 姚粟, 等.菌株CICC 6294生物学特性及其在辣椒发酵中的应用[J].食品与发酵工业, 2018, 44(7):116-121.
ZHAI L, XIE J Y, YAO S, et al.Biological properties and application in fermented chili of strain CICC 6294[J].Food and Fermentation Industries, 2018, 44(7):116-121.
[17] 程池, 杨梅, 李金霞, 等.Biolog微生物自动分析系统:细菌鉴定操作规程的研究[J].食品与发酵工业, 2006, 32(5):50-54.
CHENG C, YANG M, LI J X, et al.Biolog microbial identification system:Study on the operating regulation of bacteria identification[J].Food and Fermentation Industries, 2006, 32(5):50-54.
[18] GOODFELLOW M, KAMPFER P, BUSSE H J, et al.Bergey’s Manual of Systematic Bacteriology, Second Edition[M].New York:Springer, 2012:504.
[19] RUIZ-MOYANO S, TAO N N, UNDERWOOD M A, et al.Rapid discrimination of Bifidobacterium animalis subspecies by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J].Food Microbiology, 2012, 30(2):432-437.
[20] 吴丽娜, 王伟, 王萍, 等.基质辅助激光解吸电离飞行时间质谱法对环境中芽孢杆菌的鉴定及溯源[J].食品安全质量检测学报, 2022, 13(9):2848-2854.
WU L N, WANG W, WANG P, et al.Identification and traceability of Bacillus in the environment by matrix-assisted laser desorption lionization-time of flight mass spectrometry[J].Journal of Food Safety &Quality, 2022, 13(9):2848-2854.
[21] GORIS J, KONSTANTINIDIS K T, KLAPPENBACH J A, et al.DNA-DNA hybridization values and their relationship to whole-genome sequence similarities[J].International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 1):81-91.
[22] MEIER-KOLTHOFF J P, HAHNKE R L, PETERSEN J, et al.Complete genome sequence of DSM 30083(T), the type strain (U5/41(T)) of Escherichia coli, and a proposal for delineating subspecies in microbial taxonomy[J].Standards in Genomic Sciences, 2014, 9:2.
[23] CHAN M S, MAIDEN M, SPRATT B, et al.Database-driven multi locus sequence typing (MLST) of bacterial pathogens[J].Bioinformatics, 2001, 17(11):1077-1083.
[24] 乌云, 赵亚荣, 孔亚楠, 等.益生菌Lactobacillus plantarum P-8连续传代1000—2000代期间表型特征稳定性研究[J].中国食品学报, 2015, 15(5):20-26.
WU Y, ZHAO Y R, KONG Y N, et al.Studies on the stability of the probiotic Lactobacillus plantarum P-8 during long-term continuous subculturing from 1000 generations to 2000 generations by assessing its phenotypic characteristics[J].Journal of Chinese Institute of Food Science and Technology, 2015, 15(5):20-26.
[25] 刘天祎, 吴振, 潘道东, 等.抗氧化活性乳酸菌发酵剂的遗传稳定性研究[J].中国食品学报, 2017, 17(10):194-201.
LIU T Y, WU Z, PAN D D, et al.The genetic stability of lactic acid bacteria starter with antioxidant activity[J].Journal of Chinese Institute of Food Science and Technology, 2017, 17(10):194-201.