尿苷二磷酸糖基转移酶(uridine diphosphate glycosyltransferase,UGT)能够催化糖基配体从活化的核苷酸糖基供体转移到特定的受体分子上,从而调节受体的特性(如生物活性/溶解度和转运等)[1-2]。大部分UGT来源于植物或者动物,野生株产量不能满足工业化要求,因此需要通过微生物异源表达提高UGT的产量。目前限制UGT工业生产的主要因素为异源表达可溶性差,表达量低以及需要昂贵的尿苷二磷酸葡萄糖(uridine diphosphate glucose,UDPG)作为供体[3]。通过与蔗糖合酶(sucrose synthase,SUSy)的偶联可实现UDPG的循环再生[4-5],但是可溶性表达差的问题至今仍未有效解决。拟南芥来源的糖基转移酶UGT73C5可以催化肉桂醇,油菜素类固醇,人参二醇和脱氧雪腐镰刀菌烯醇等的糖基化,广泛应用于医药和保健食品中[6-9]。但是UGT73C5在大肠杆菌中异源表达大多以无活性的包涵体形式存在,严重限制了其工业应用。
一般来说,包涵体形成一般是由于蛋白翻译过程中表达过快或者缺失一定的后修饰从而导致不正确或不充分的折叠[10]。通过优化诱导条件[如初始细胞密度、异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)浓度、诱导时间和诱导温度]可在一定程度上改善蛋白可溶性表达,但通常效果甚微。分子伴侣共表达与助溶蛋白标签融合表达是目前研究最多的蛋白可溶性改善方法[11-12]。LASKEY等[13]在非洲爪蟾卵的浸出液中发现了分子伴侣蛋白,如GroES、GroEL、DnaK、DnaJ、GrpE和触发因子(trigger factor,TF)等,可参与协助生物大分子的折叠、组装、转运及降解等过程,从而极大程度提高目的蛋白的可溶性表达。这些分子伴侣对体外模型蛋白的正确折叠、聚集和组装具有显著影响[14-16]。例如,伴侣质粒pG-KJE8、pGro7和pG-TF2可以显著提高TtSPase的可溶性表达[17],通过与pGro7共表达,4-α-葡糖基转移酶产量提高了24.6倍[18]。另外,一些高度可溶的蛋白与目的蛋白融合表达时对目的蛋白具有助溶效果,这些蛋白往往被设计为助溶标签[19]。硫氧还蛋白Trx-tag,是一种分子质量为12 kDa的氧化还原蛋白,可催化还原蛋白质二硫键,促进目的蛋白二硫键的形成[20];谷胱甘肽巯基转移酶标签蛋白GST-tag是一个大小为26 kDa左右的转移酶,本身在大肠杆菌中可大量可溶性表达,同时也可以提高共表达的蛋白可溶性。在大多数情况下,该标签的存在还有助于保护重组蛋白免受胞外蛋白酶的降解并提高其稳定性[21]。研究发现融合Trx标签的人胰岛素可溶性表达明显提高[22],融合GST后人干扰素蛋白可实现70%的可溶表达[23]。
本研究将UGT73C5在Escherichia coli BL21(DE3)中进行异源表达,通过与分子伴侣共表达以及与助溶蛋白标签融合表达等方法提高其可溶性表达,并对不同方法表达效果进行了比较。最后,对融合酶GST-UGT73C5催化2 mmol/L肉桂醇糖基化合成络塞的反应进行了研究。
1 材料与方法
1.1 材料与试剂
本研究中用到的质粒和菌株如表1所示。来自拟南芥的UDP糖基转移酶UGT73C5 (GenBank:NP_181218.1)基因,上海亦欣生物技术有限公司优化合成。载体质粒pET-28a(+)、pET-32b(+)和pET-42b(+),本实验室收藏,E.coli DH5α和E.coli BL21(DE3)菌株分别用作质粒构建和目的基因表达的宿主。4种含有分子伴侣基因的质粒pG-KJE8、pGro7、pKJE7和pG-TF2,宝生物工程(大连)有限公司。肉桂醇及络塞,上海源叶生物有限公司。UDPG,广东昂飞生物有限公司。
表1 本研究中使用的质粒和菌株
Table 1 Strains and plasmids used in this study
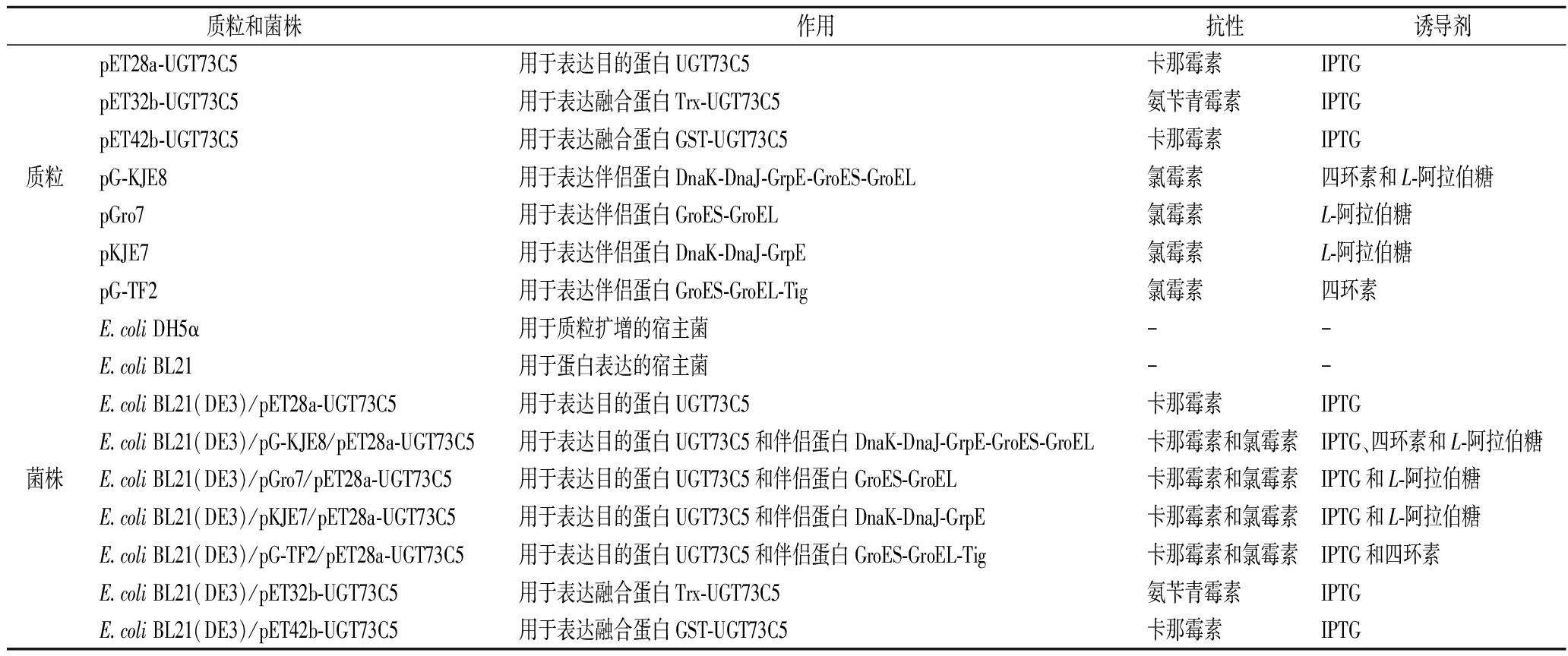
质粒和菌株作用抗性诱导剂pET28a-UGT73C5用于表达目的蛋白UGT73C5卡那霉素IPTGpET32b-UGT73C5用于表达融合蛋白Trx-UGT73C5氨苄青霉素IPTGpET42b-UGT73C5用于表达融合蛋白GST-UGT73C5卡那霉素IPTG质粒pG-KJE8用于表达伴侣蛋白DnaK-DnaJ-GrpE-GroES-GroEL氯霉素四环素和L-阿拉伯糖pGro7用于表达伴侣蛋白GroES-GroEL氯霉素L-阿拉伯糖pKJE7用于表达伴侣蛋白DnaK-DnaJ-GrpE氯霉素L-阿拉伯糖pG-TF2用于表达伴侣蛋白GroES-GroEL-Tig氯霉素四环素E.coli DH5α用于质粒扩增的宿主菌--E.coli BL21用于蛋白表达的宿主菌--E.coli BL21(DE3)/pET28a-UGT73C5用于表达目的蛋白UGT73C5卡那霉素IPTGE.coli BL21(DE3)/pG-KJE8/pET28a-UGT73C5用于表达目的蛋白UGT73C5和伴侣蛋白DnaK-DnaJ-GrpE-GroES-GroEL卡那霉素和氯霉素IPTG、四环素和L-阿拉伯糖菌株E.coli BL21(DE3)/pGro7/pET28a-UGT73C5用于表达目的蛋白UGT73C5和伴侣蛋白GroES-GroEL卡那霉素和氯霉素IPTG和L-阿拉伯糖E.coli BL21(DE3)/pKJE7/pET28a-UGT73C5用于表达目的蛋白UGT73C5和伴侣蛋白DnaK-DnaJ-GrpE卡那霉素和氯霉素IPTG和L-阿拉伯糖E.coli BL21(DE3)/pG-TF2/pET28a-UGT73C5用于表达目的蛋白UGT73C5和伴侣蛋白GroES-GroEL-Tig卡那霉素和氯霉素IPTG和四环素E.coli BL21(DE3)/pET32b-UGT73C5用于表达融合蛋白Trx-UGT73C5氨苄青霉素IPTGE.coli BL21(DE3)/pET42b-UGT73C5用于表达融合蛋白GST-UGT73C5卡那霉素IPTG
1.2 实验方法
1.2.1 重组UGT73C5菌株的构建
将合成的UGT73C5基因克隆至载体pET-28a(+)中的限制性位点BamH I和Xho I之间构建重组质粒pET28a-UGT73C5,并转化至E.coli BL21(DE3)中构建UGT73C5重组表达菌株E.coli BL21(DE3)/pET28a-UGT73C5(简称BL21-UGT73C5)。
1.2.2 UGT73C5与分子伴侣共表达菌株的构建
将pET28a-UGT73C5质粒分别和pG-KJE8、pGro7、pKJE7和pG-TF2这4种伴侣质粒同时转化E.coli BL21(DE3)感受态细胞并涂布至含有50 μg/mL卡那霉素和20 μg/mL氯霉素双抗生素的平板中构建分子伴侣共表达菌株,分别命名为E.coli BL21(DE3)/pG-KJE8/pET28a-UGT73C5(简称BL21-KJE8/UGT73C5)、E.coli BL21(DE3)/pGro7/pET28a-UGT73C5(简称BL21-RO7/UGT73C5)、E.coli BL21(DE3)/pKJE7/pET28a-UGT73C5(简称BL21-KJE7/UGT73C5)和E.coli BL21(DE3)/pG-TF2/pET28a-UGT73C5(简称BL21-TF2/UGT73C5)。
1.2.3 Trx-UGT73C5和GST-UGT73C5融合表达菌株的构建
本研究中所用到的引物见表2。质粒载体pET-32b(+)、pET-42b(+)分别带有助溶蛋白标签:硫氧还蛋白标签(thioredoxin tag, Trx-tag)和谷胱甘肽巯基转移蛋白标签(glutathione mercapto transfer protein tag,GST-tag)。通过PCR扩增pET28a-UGT73C5中的目的基因(UGT73C5),并通过ClonExpress Ⅱ一步克隆试剂盒克隆至载体pET-32b(+)与pET-42b(+)的双酶切位点BamH I和Xho I之间,构建重组质粒pET32b-UGT73C5和pET42b-UGT73C5。将这些重组质粒转化至大肠杆菌E.coli BL21(DE3)中,并分别涂布至带有100 μg/mL氨苄青霉素和50 μg/mL卡那霉素的LB平板中以构建Trx-UGT73C5和GST-UGT73C5融合酶表达菌株E.coli BL21(DE3)/pET32b-UGT73C5(简称BL21-Trx-UGT73C5)和E.coli BL21(DE3)/pET42b-UGT73C5(简称BL21-GST-UGT73C5)。
表2 本研究中使用的引物
Table 2 Primers used in this study

引物名称碱基序列32b-UGT73C5-FggccatggcgatatcggatccCATGGTGTCTGAGACA32b-UGT73C5-RgtggtggtggtggtgctcgagATTATTAGGTTCG42b-UGT73C5-FgtccatggatatcggggatccCATGGTGTCTGAGACA42b-UGT73C5-RgtggtggtggtggtgctcgagATTATTAGGTTCG
1.2.4 重组酶的表达与纯化
将不同的重组大肠杆菌表达菌株在含有相应抗生素的40 mL LB培养基中于37 ℃、200 r/min培养至OD600=0.8~0.9时,加入0.2 mmol/L IPTG(分子伴侣共表达菌株需加入0.5~4 mg/mL L-阿拉伯糖和/或1~10 ng/mL四环素),于16 ℃、200 r/min摇床中诱导20 h。通过离心(8 000 r/min、5 min)收集细胞,将细胞重悬于8 mL PBS缓冲液(pH 7.5)中,并在冰水浴中超声处理10 min。离心分离上清液与沉淀,并经SDS-PAGE进一步分析。重组酶UGT73C5、Trx-UGT73C5和GST-UGT73C5通过Ni-NTA亲和层析纯化。
1.2.5 大肠杆菌细胞光密度与细胞湿重、干重的关系
细胞湿重:收集不同体积的大肠杆菌培养液(10、20、30、40、50、60、70 mL),12 000 r/min离心10 min,弃上清液,将离心管倒置于清洁的吸水纸上约数分钟,擦净管壁上水渍,随即称量,细胞湿重等于称量的质量减去已预先称重的离心管质量。将已称量的湿细胞重悬于20 mL PBS缓冲液中以配制不同质量浓度的细胞溶液,充分混匀,吸取1 mL重悬的溶液稀释10倍后测定其在600 nm下的吸光度,并绘制细胞湿重与细胞光密度之间的标准曲线。
细胞干重:收集不同体积的大肠杆菌培养液(10、20、30、40、50、60、70 mL),12 000 r/min离心10 min,弃上清液,用去离子水洗涤沉淀两次,冻干备用。称取不同质量的冻干细胞重悬于20 mL PBS缓冲液中,以配制不同质量浓度的细胞溶液并测定600 nm下的吸光度,并绘制细胞干重与细胞光密度之间的标准曲线。
细胞光密度(OD600)与细胞干重、湿重呈良好线性关系,如图1所示。
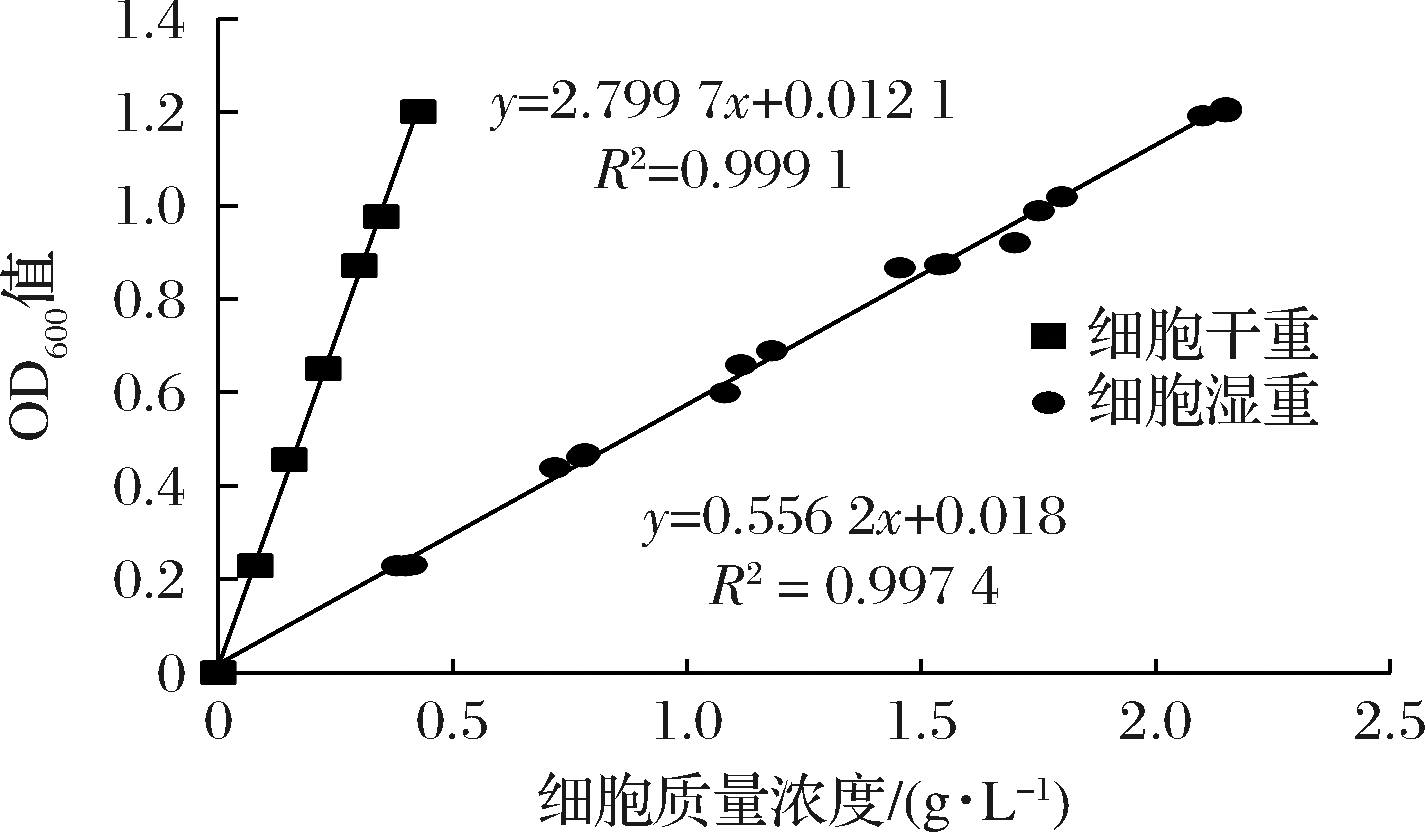
图1 大肠杆菌细胞光密度(OD600)与细胞干重、 湿重的标准曲线
Fig.1 Standard curve between E.coli cell optical density (OD600) and dry/wet weight cells
1.2.6 酶活力分析
高速离心收集40 mL LB培养基中诱导后的菌体细胞并弃尽上清液,重悬于8 mL PBS (100 mmol/L,pH 7.5)缓冲液中,稀释25倍测定其在600 nm下的吸光度并换算成其细胞干重。超声破碎后离心获取上清液,将含有100 mmol/L PBS (pH 7.5)、10 mmol/L肉桂醇、5 mmol/L UDPG和30 μL上清酶液的反应混合液(总体系300 μL)于40 ℃反应10 min,反应结束后加入600 μL甲醇以淬灭反应。反应液经12 000 r/min离心5 min并通过0.22 μm滤膜过滤后,通过HPLC分析反应产物。酶活力单位(U)定义为每分钟产生1 μmol络塞所需的酶量,酶活力(U/g细胞)定义为单位质量细胞干重所具有的酶活力。
酶的比活力通过含有100 mmol/L PBS (pH 7.5)、10 mmol/L肉桂醇、5 mmol/L UDPG和0.02 mg/mL纯酶的反应混合液(总体系300 μL)在40 ℃反应10 min测定。蛋白浓度采用Bradford法测定,比活力(U/mg 蛋白)指的是单位质量的酶所对应的活力。
1.2.7 融合酶在络塞合成中的应用
取相同质量的BL21-UGT73C5和BL21-GST-UGT73C5细胞,分别重悬于8 mL PBS (100 mmol/L,pH 7.5)缓冲液中,配制成细胞浓度为15 g/L的悬浮液,破碎离心后获取上清液。将含有100 mmol/L PBS (pH 7.5)、2 mmol/L肉桂醇、10 mmol/L UDPG和2.5 mL上清液酶液(总体系为5 mL)的混合液于40 ℃反应,在0.5、1、2、4、6、8、10、12 h分别取样并加入甲醇以淬灭反应。反应液经12 000 r/min离心5 min,并通过0.22 μm滤膜过滤后,通过HPLC分析反应产物。转化率计算如公式(1)所示:
转化率![]()
(1)
式中:Mp表示生成络塞的物质的量浓度,mmol/L;Ms表示剩余底物肉桂醇的物质的量浓度,mmol/L。
1.2.8 HPLC检测方法
使用HPLC 1260系统(Agilent Technologies,Santa Clara,CA,USA)和Diamonsil C18色谱柱(250 mm×4.6 mm×5 μm)对底物肉桂醇和糖基化产物络塞进行定量测定。肉桂醇和络塞的洗脱条件如下:50%溶剂A(H2O);50%溶剂B(乙腈);流速1 mL/min;吸光度254 nm;柱温30 ℃。
2 结果与分析
2.1 UGT73C5在大肠杆菌中的表达及活力表征
BL21-UGT73C5菌株的表达情况如图2所示,在破碎沉淀中,大约在56 kDa有一条明显条带,与理论UGT73C5的分子质量相近,而上清液中在该位置无明显条带。这说明大多数UGT73C5蛋白以不溶性包涵体形式存在。因此,增加UGT73C5可溶性表达是非常必要的。本研究中,通过HPLC检测UGT73C5对肉桂醇的糖基化活力,并以上清液酶活力表征可溶性蛋白表达量。结果表明,反应产物与络塞标准品具有相同的保留时间(大约2.5 min),而在对照样品中[E.coli BL21(DE3)/pET-28a (+)空载诱导表达]未检测到新产物的生成(图3)。通过产物络塞的标准曲线定量分析,UGT73C5在大肠杆菌中表达的酶活力为18.56 U/g细胞。
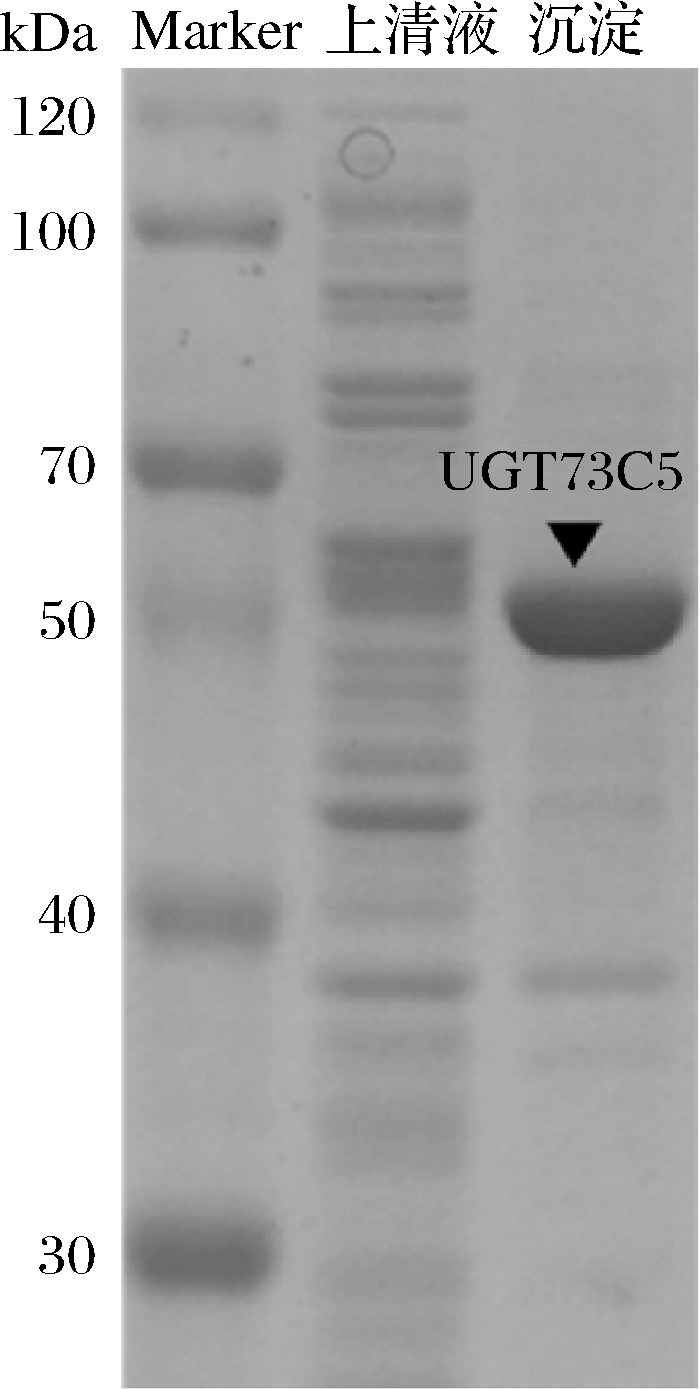
图2 UGT73C5在E.coli BL21(DE3)中异源表达的 SDS-PAGE
Fig.2 Analysis of SDS-PAGE of UGT73C5 in E.coli BL21(DE3)
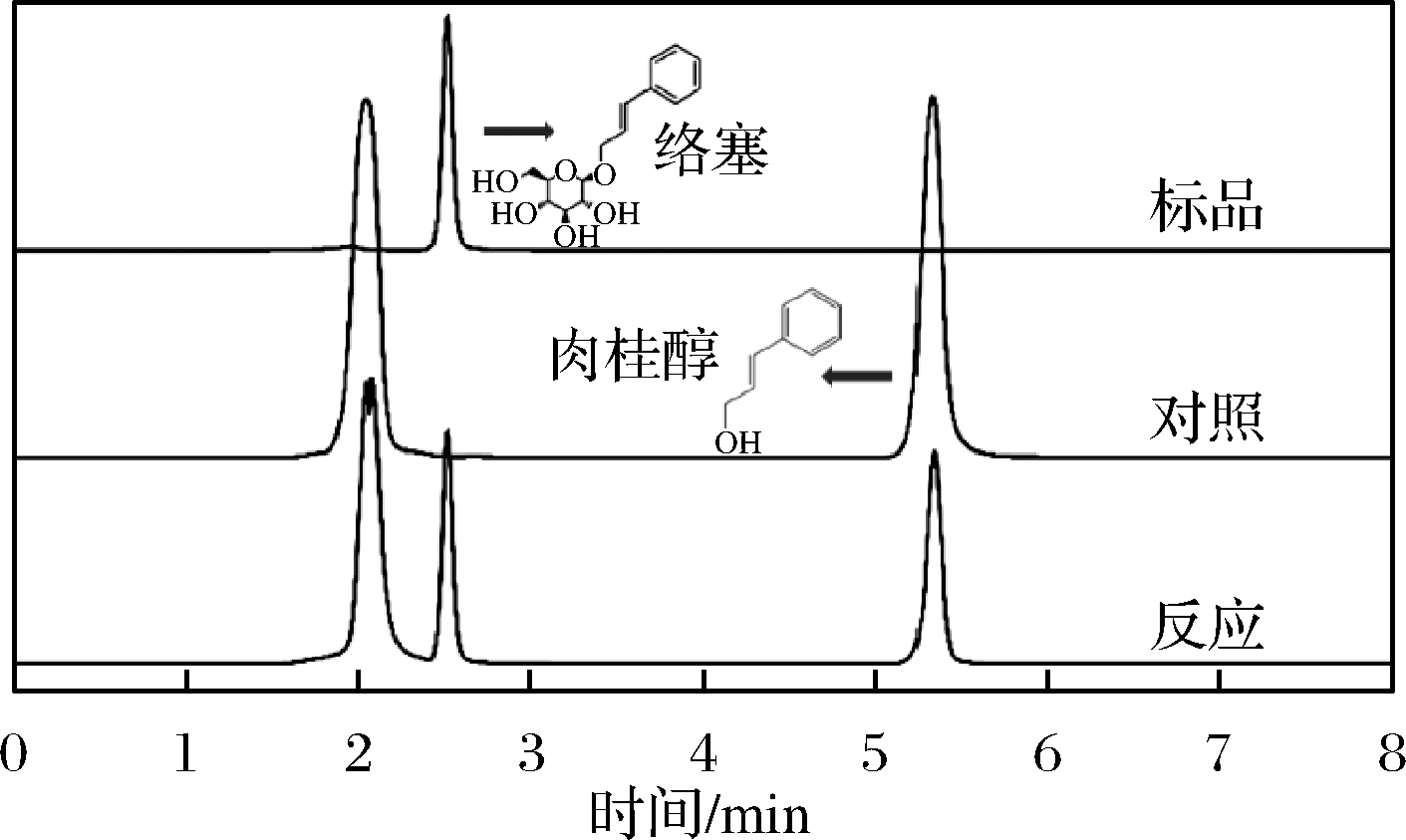
图3 UGT73C5催化肉桂醇糖基化反应的HPLC图谱
Fig.3 HPLC chromatogram of cinnamyl alcohol glycosylation by UGT73C5
2.2 分子伴侣共表达提高UGT73C5可溶性表达
为了改善UGT73C5的可溶性表达,研究了UGT73C5与4个伴侣蛋白质粒(pG-KJE8、pGro7、pKJE7和pG-TF2)的共表达情况。如图4-a所示,SDS-PAGE结果表明4种分子伴侣质粒与UGT73C5共表达成功。如图4-b所示,UGT73C5的原始酶活力为18.56 U/g细胞,添加分子伴侣蛋白质粒pG-KJE8、pGro7、pKJE7和pG-TF2后的酶活力分别为原始酶活力的1.27、1.18、0.91、1.37倍。其中质粒pG-KJE8、pGro7和pG-TF2中均含有分子伴侣GroES-GroEL、GroEL的内腔可以与变性多肽结合,在辅因子GroES调节的ATP依赖性反应中释放。GroES和GroEL作为折叠酶与多肽相互作用并帮助正确重新折叠[24],GroES-GroEL可以作为mRNA稳定剂来增加蛋白质的产生[25]。与pGro7相比,pG-KJE8与UGT73C5共表达后可溶性提高更为明显(图4-b)。原因可能是在GroES-GroEL和DnaK-DnaJ-GrpE伴侣蛋白共同辅助下,UGT73C5折叠方面表现更为良好。DnaK还可以稳定蛋白质,以供GroEL进行后续折叠[26]。GroES-GroEL和DnaK-DnaJ-GrpE在大多数蛋白质的折叠和组装中具有互补功能[27]。与pG-TF2共表达导致更高的可溶性UGT73C5表达,可能是因为触发因子TF可以识别肽底物中的芳香族和碱性氨基酸残基,然后与肽结合[28],进一步避免错误折叠,保护新生链不被蛋白酶消化,并与其他伴侣合作协助折叠。因此,伴随TF (pG-TF2)的GroES-GroEL在体内对UGT73C5产生积极影响。
2.3 重组Trx-UGT73C5和GST-UGT73C5融合蛋白的表达
虽然与分子伴侣共表达后的UGT73C5的可溶性表达有一定程度的改善,但效果并不明显。本研究进一步通过UGT73C5与Trx-tag (12 kDa)和GST-tag (26 kDa)分别融合表达,结果如图5-a所示。融合酶Trx-UGT73C5的包涵体含量仍然较高,但上清液中表达量较原始UGT73C5表达情况有明显提高。融合酶GST-UGT73C5在16 ℃下表达,几乎没有包涵体存在,而在上清液中观察到更多的蛋白表达。为了进一步提高其表达效果,提高诱导表达的温度至25 ℃,GST-UGT73C5蛋白含量显著提高,虽然有少部分包涵体出现,但大部分仍以可溶形式存在。如图5-b所示,融合酶Trx-UGT73C5酶活力为26.95 U/g细胞,是原始酶活力的1.45倍;在25 ℃诱导表达的GST-UGT73C5酶活力达到47.17 U/g细胞,是原始酶活力的2.54倍。为了研究融合标签蛋白对UGT73C5比酶活力的影响,测定了纯化后的GST-UGT73C5和Trx-UGT73C5以及原始酶UGT73C5的比活力。结果如表3所示,Trx-UGT73C5和GST-UGT73C5比活力分别为0.68、 0.72 U/mg蛋白,是原始酶比活力(0.79 U/mg蛋白)的0.86、0.91倍。以上结果表明助溶蛋白标签对糖基转移酶UGT73C5的N端修饰能够明显提高其可溶性表达,并且助溶蛋白(尤其是GST)对UGT73C5自身的催化活力没有显著影响,无需切割分离。
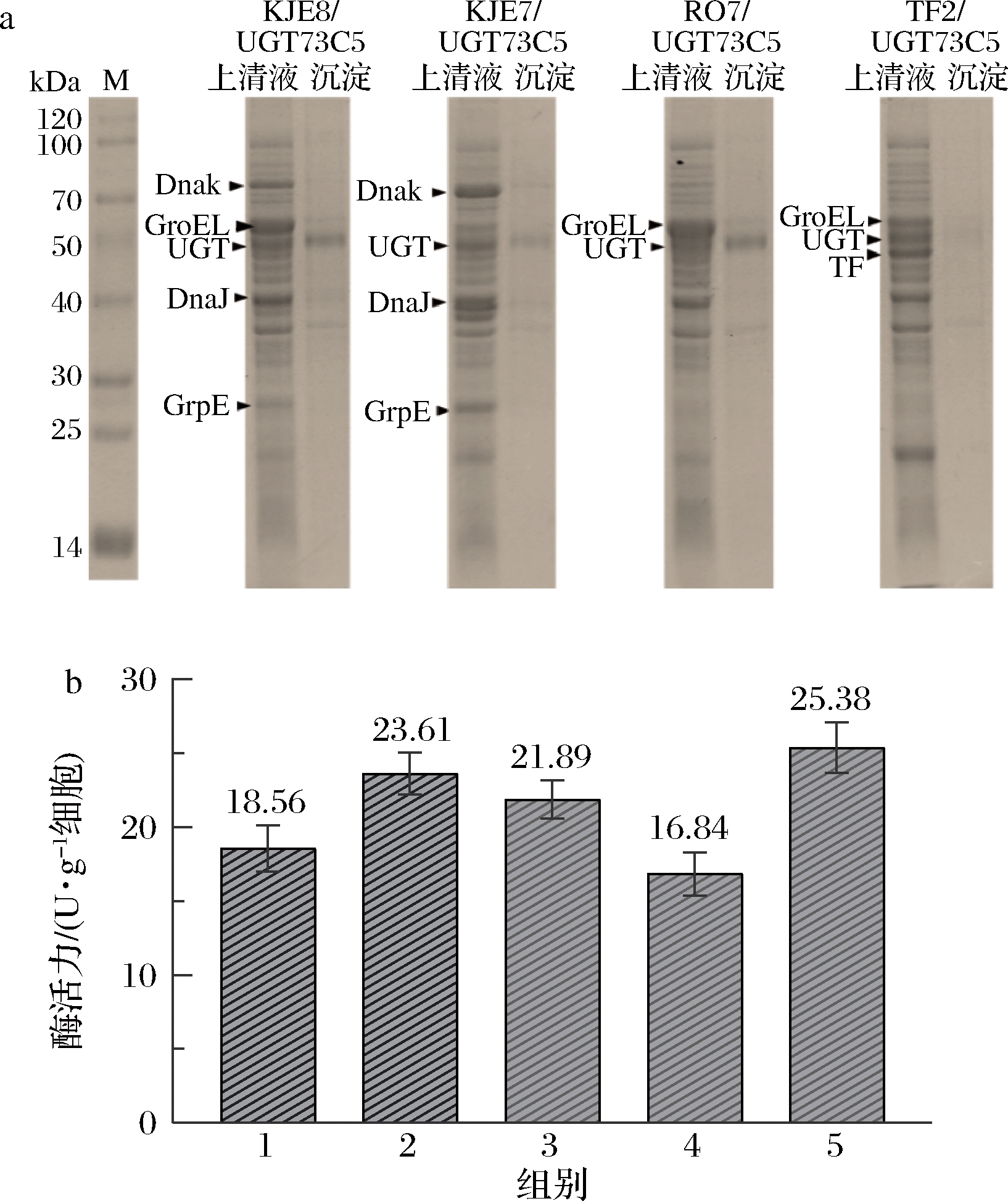
a-分子伴侣共表达SDS-PAGE分析;b-分子伴侣共表达酶活力分析 [1-BL21-UGT73C5 (16 ℃);2-BL21-KJE8/UGT73C5 (16 ℃); 3-BL21-RO7/UGT73C5 (16 ℃);4-BL21-KJE7/UGT73C5 (16 ℃); 5-BL21-TF2/UGT73C5 (16 ℃)]
图4 UGT73C5与不同分子伴侣共表达后蛋白表达及酶活力分析
Fig.4 Analysis of protein expression and activities of UGT73C5 co-expressed with different molecular chaperones

a-融合酶SDS-PAGE;b-融合酶活力分析 [1-BL21-UGT73C5 (16 ℃);2-BL21-Trx-UGT73C5 (16 ℃); 3-BL21-GST-UGT73C5 (16 ℃);4-BL21-GST-UGT73C5 (25 ℃)]
图5 UGT73C5与Trx和GST融合后蛋白表达及酶活力分析
Fig.5 Analysis of protein expression and activities of UGT73C5 fused with Trx and GST
表3 融合酶的纯酶比活力
Table 3 Specific activities of purified fusion enzymes

蛋白比活力/(U·mg-1蛋白)UGT73C50.79±0.02 Trx-UGT73C50.68±0.05 GST-UGT73C50.72±0.03
2.4 融合酶GST-UGT73C5在络塞合成中的应用
分别将融合酶GST-UGT73C5和原始酶应用于2 mmol/L肉桂醇糖基化合成络塞的反应。如图6-a所示,纯化后GST-UGT73C5(0.2 mg/mL)在6 h后能够转化96.3%的肉桂醇为络塞,与纯化后原始酶(0.2 mg/mL)转化率一致,说明增加融合标签GST后,对催化肉桂醇糖基化没有影响。对相同浓度细胞破碎后的GST-UGT73C5和原始酶粗酶催化反应比较(图6-b)发现,GST-UGT73C5在4 h内转化率就达到90.7%,6~12 h维持在93.6%,而原始酶在12 h转化率才达到88.5%。结果表明单位质量菌体融合酶GST-UGT73C5较原始酶具有更高的表达量,从而导致更高的转化率。因此,融合酶GST-UGT73C5在酶法合成络塞的应用中具有更大的潜力。
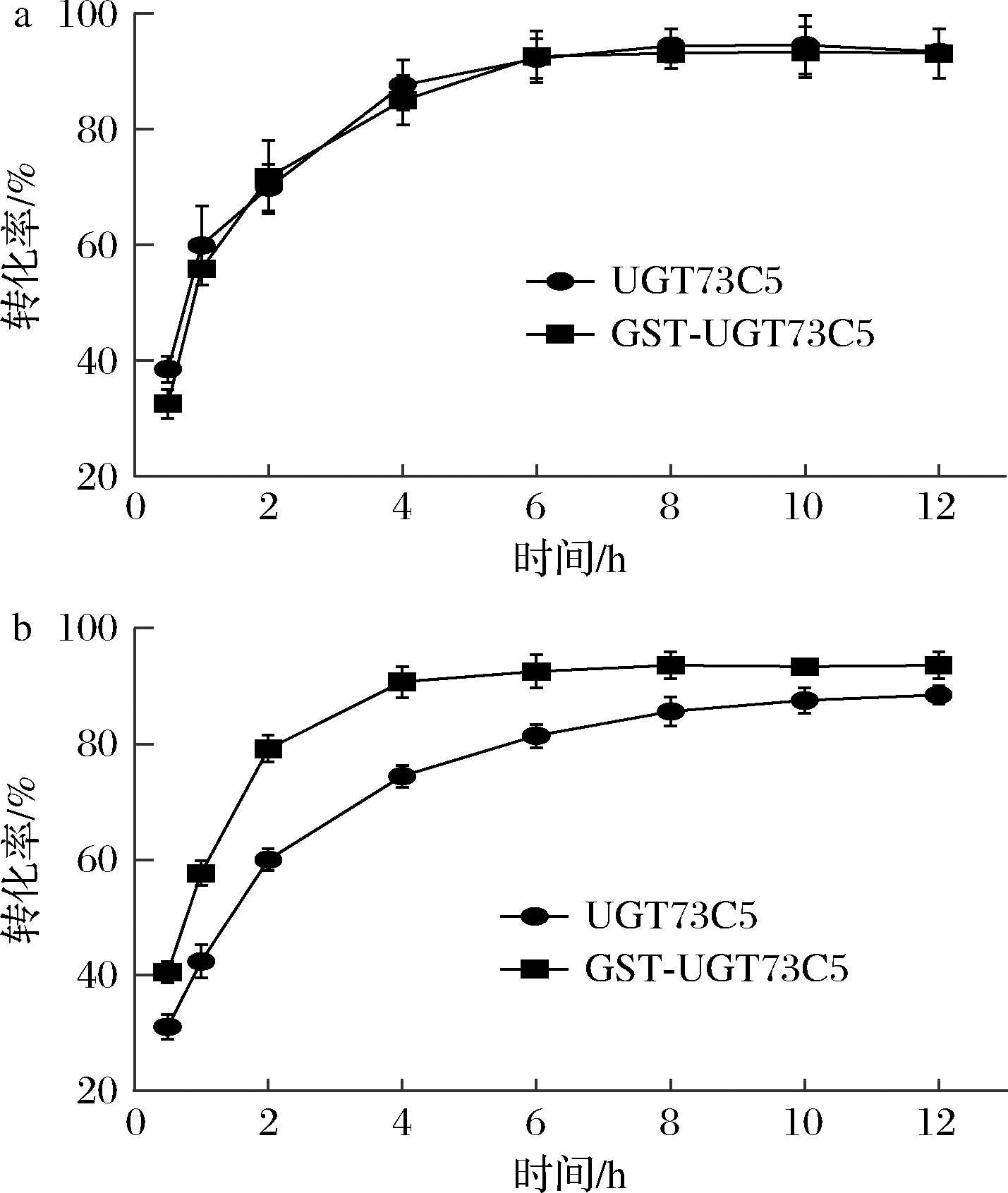
a-GST-UGT73C5与UGT73C5的纯酶反应比较;b-同等浓度 BL21-GST-UGT73C5与BL21-UGT73C5细胞破碎上清液反应比较
图6 GST-UGT73C5与UGT73C5催化肉桂醇糖基化的反应分析
Fig.6 Analysis of cinnamyl alcohol glycosylation reaction catalyzed by GST-UGT73C5 and UGT73C5
3 结论
糖基转移酶UGT73C5可以催化肉桂醇的糖基化合成络塞,然而异源表达可溶性差限制了其工业应用价值,为提高UGT73C5在大肠杆菌中的可溶性表达,本研究通过分子伴侣共表达和助溶蛋白标签融合表达的方式改善了UGT73C5可溶性差的问题。3种商业化的分子伴侣蛋白质粒pG-KJE8、pGro7和pG-TF2对UGT73C5的可溶性表达均有一定程度促进作用,其中pG-TF2表达的伴侣蛋白组合GroES-GroEL-Tig效果最好,表达量为原始酶可溶性表达量的1.37倍;融合表达了两种助溶蛋白标签硫氧还蛋白Trx-tag和谷胱甘肽巯基转移蛋白GST-Tag,其中融合蛋白GST-UGT73C5可溶性表达量最高,在25 ℃诱导条件下,为原酶初始表达量的2.54倍。纯化后GST-UGT73C5比活力与原酶相比没有明显下降,表明该N端修饰对其自身的催化活力没有显著影响,无需切割分离。在2 mmol/L肉桂醇催化反应中,同等细胞浓度的GST-UGT73C5粗酶液比原始酶UGT73C5粗酶液具有更高的转化效率。GST-UGT73C5粗酶催化反应在4 h内催化肉桂醇转化率达到90%以上,最高可达93.6%,而原始酶在12 h才达到88.5%。因此,融合酶GST-UGT73C5可能成为络塞酶法合成中更具有应用潜力的酶。
[1] NA L, LI R, CHEN X.Recent progress in synthesis of carbohydrates with sugar nucleotide-dependent glycosyltransferases[J].Current Opinion in Chemical Biology, 2021, 61:81-95.
[2] TIWARI P, SANGWAN R S, SANGWAN N S.Plant secondary metabolism linked glycosyltransferases:An update on expanding knowledge and scopes[J].Biotechnology Advances, 2016, 34(5):714-739.
[3] DE BRUYN F, MAERTENS J, BEAUPREZ J, et al.Biotechnological advances in UDP-sugar based glycosylation of small molecules[J].Biotechnology Advances, 2015, 33(2):288-302.
[4] LIU S K, LYU Y B, YU S Q, et al.Efficient production of orientin and vitexin from luteolin and apigenin using coupled catalysis of glycosyltransferase and sucrose synthase[J].Journal of Agricultural and Food Chemistry, 2021, 69(23):6 578-6 587.
[5] CHEN T, CHEN Z, WANG N, et al.Highly regioselective and efficient biosynthesis of polydatin by an engineered UGTBL 1-At SuSy cascade reaction[J].Journal of Agricultural and Food Chemistry, 2021.69(31):8 695-8 702.
[6] POPPENBERGER B, FUJIOKA S, SOENO K, et al.The UGT73C5 of Arabidopsis thaliana glucosylates brassinosteroids[J].Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(42):15 253-15 258.
[7] ZHOU W, BI H P, ZHUANG Y B, et al.Production of cinnamyl alcohol glucoside from glucose in Escherichia coli[J].Journal of Agricultural and Food Chemistry, 2017, 65(10):2 129-2 135.
[8] HU Y M, XUE J, MIU J, et al.Biocatalytic synthesis of ginsenoside Rh2 using Arabidopsis thaliana glucosyltransferase-catalyzed coupled reactions[J].Journal of Biotechnology, 2020, 309:107-112.
[9] POPPENBERGER B, BERTHILLER F, LUCYSHYN D, et al.Detoxification of the Fusarium mycotoxin deoxynivalenol by a UDP-glucosyltransferase from Arabidopsis thaliana[J].Journal of Biological Chemistry, 2003, 278(48):47905.
[10] 黄庶识, 卢明倩, 李冰,等.重组大肠杆菌表达可溶性蛋白和包涵体过程的拉曼光谱实时分析[J].中国激光, 2014, 41(12):257-265.
HUANG S S, LU M Q, LI B, et al.Real time detection on the expression of soluble protein and inclusion body in the recombinant Escherichia coli with laser tweezers Raman spectroscopy[J].Chinese Journal of Lasers, 2014, 41(12):257-265.
[11] 张磊, 唐永凯, 李红霞, 等.促进原核表达蛋白可溶性的研究进展[J].中国生物工程杂志, 2021, 41(S1):138-149.
ZHANG L, TANG Y K, LI H X, et al.Advances in promoting solubility of prokaryotic expressed proteins[J].China Biotechnology, 2021, 41(S1):138-149.
[12] 朱红裕, 李强.外源蛋白在大肠杆菌中的可溶性表达策略[J].过程工程学报, 2006, 6(1):150-155.
ZHU H Y, LI Q.Strategies for expression of soluble heterologous proteins in Escherichia coli[J]. The Chinese Journal of Process Engineering, 2006, 6(1):150-155.
[13] LASKEY R A, HONDA B M, MILLS A D, et al.Nucleosomes are assembled by an acidic protein which binds histones and transfers them to DNA[J].Nature, 1978, 275(5 679):416-420.
[14] ROBIN S, TOGASHI D M, RYDER A G, et al.Trigger factor from the Psychrophilic bacterium Psychrobacter frigidicola is a monomeric chaperone[J].Journal of Bacteriology, 2009, 191(4):1 162-1 168.
[15] DE MARCO A, DEUERLING E, MOGK A, et al.Chaperone-based procedure to increase yields of soluble recombinant proteins produced in E.coli[J].BMC Biotechnology, 2007, 7(1):32.
[16] BUKAU B, DEUERLING E, PFUND C, et al.Getting newly synthesized proteins into shape[J].Cell, 2000, 101(2):119-122.
[17] YAO D, FAN J, HAN R, et al.Enhancing soluble expression of sucrose phosphorylase in Escherichia coli by molecular chaperones[J].Protein Expression and Purification, 2020, 169:105571.
[18] DUAN X G, ZHANG X Y, SHEN Z Y, et al.Efficient production of aggregation prone 4-α-glucanotransferase by combined use of molecular chaperones and chemical chaperones in Escherichia coli[J].Journal of Biotechnology, 2019, 292:68-75.
[19] YOUNG C L, BRITTON Z T, ROBINSON A S.Recombinant protein expression and purification:A comprehensive review of affinity tags and microbial applications[J].Biotechnology Journal, 2012, 7(5):620-634.
[20] SMITH D B, JOHNSON K S.Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase[J].Gene, 1988, 67(1):31-40.
[21] ![]() MMLER A, LAWRENCE A M DE, MARCO A.Simplified screening for the detection of soluble fusion constructs expressed in E.coli using a modular set of vectors[J].Microbial Cell Factories, 2005, 4(1):34-34.
MMLER A, LAWRENCE A M DE, MARCO A.Simplified screening for the detection of soluble fusion constructs expressed in E.coli using a modular set of vectors[J].Microbial Cell Factories, 2005, 4(1):34-34.
[22] 沈加斌, 徐攀, 李鑫雨, 等.人胰岛素原基因在大肠杆菌中的可溶性表达及分离纯化研究[J].食品与生物技术学报, 2016, 35(7):721-727.
SHEN J B, XU P, LI X Y, et al.Soluble expression and purification of human proinsulin in Escherichia coli[J].Journal of Food Science and Biotechnology, 2016, 35(7):721-727.
[23] RABHI-ESSAFI I,SADOK A,KHALAF N,et al.A strategy for high-level expression of soluble and functional human interferon alpha as a GST-fusion protein in E.coli[J].Protein Engineering Design and Selection Peds, 2007, 20(5):201-209.
[24] LANGER T, PFEIFER G, MARTIN J, et al.Chaperonin-mediated protein folding:GroES binds to one end of the GroEL cylinder, which accommodates the protein substrate within its central cavity[J].The EMBO Journal, 1992, 11(13):4 757-4 765.
[25] YOON H, HONG J, RYU S.Effects of chaperones on mRNA stability and gene expression in Escherichia coli[J].Journal of Microbiology and Biotechnology, 2008, 18(2):228-233.
[26] CALLONI G, CHEN T, SCHERMANN S, et al.DnaK functions as a central hub in the E.coli chaperone network[J].Cell Reports, 2012, 1(3):251-264.
[27] GRAGEROV A, NUDLER E, KOMISSAROVA N, et al.Cooperation of GroEL/GroES and DnaK/DnaJ heat shock proteins in preventing protein misfolding in Escherichia coli[J].Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(21):10 341-10 344.
[28] PATZELT H, ![]() S, BREHMER D, et al.Binding specificity of Escherichia coli trigger factor[J].Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(25):14 244-14 249.
S, BREHMER D, et al.Binding specificity of Escherichia coli trigger factor[J].Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(25):14 244-14 249.