在海洋生物中,贝类的主要食物为藻类、原生动物等一些浮游生物,在原甲藻和鳍藻等藻类中易产生海洋生物毒素——腹泻性贝类毒素(diarrhetic shellfish poisons,DSP) [1-2]。贝类属于非选择滤食性动物,在生长过程中,难免滤食有毒藻类食物,由此引起DSP在其体内长期融积富集,当人们误食染毒的贝类后就会对身体健康产生危害,引起中毒现象,甚至会增加患结肠癌等肿瘤的风险[3-5]。由于DSP中毒现象分布广泛,人体损伤性大,应对食用贝类加强毒素检测力度,确保贝类食用安全,因此研究高效高质的贝类毒素检测方法是十分必要的。
目前腹泻性贝类毒素检测方法主要有小鼠生物测定法、酶联免疫吸附法、高效液相色谱法等[6-8]。小鼠生物测定法是应用最多的贝类毒素检测方法,但该方法受小鼠个体影响,检测结果灵敏度不高,偏差较大。酶联免疫吸附法以抗原与抗体特异性反应为基础,交叉反应时产生较大偏差。高效液相色谱法具有灵敏度和准确度高等优势,但该方法所用仪器昂贵,且前处理过程比较复杂,需要经过专业培训的人员,比较适用于实验室和较高精度需求的样本检测。这些方法均有各自的优势,但都存在费时费力,对样品具有破坏性等问题,无法实现快速无损分析。因此,开发高效、快速、无损的贝类毒素检测技术具有重要的现实意义。
近红外光谱属于分子振动光谱,主要是对含氢基团(C—H、N—H、O—H、S—H等)振动的倍频和合频进行吸收。由于不同的物质含有不同的氢基团,这些基团对近红外光吸收波长均有所不同,通过采集样品的近红外光谱,可以得到样品中含氢基团的特征信息,从而得出样品间的差异特征。近红外光谱分析技术正是根据不同的光谱特征信息及传统化学分析方法测定的样品性质或数据,通过化学计量学方法,将光谱与数据进行关联,从而实现对未知样品进行定性和定量分析[9-10]。近红外光谱技术因具有快速无损、绿色环保等优点,近年来在食品的真伪鉴定[11]、产地鉴别[12]、等级判别[13]以及微生物检测[14]等领域得到了广泛应用。在贝类检测方面,黄冠明等[15]对牡蛎(Crassostrea angulata)样本的蛋白质、水分、脂肪进行分析,LIU等[16]提出基于高光谱技术对菲律宾蛤仔(Ruditapes philippinarum)重金属污染进行检测,刘忠艳等[17]提出基于近红外光谱和多层感知机对腹泻性贝毒的检测方法。这些研究成果为贝类毒素高效、快速、无损检测提供了技术可行性。
由于光谱采集过程中存在环境、样本、操作人员等的影响,所以光谱往往存在谱峰重叠、基线漂移等干扰因素。为了消除这些干扰,需要结合化学计量学方法对光谱数据进行预处理,建立贝类毒素的准确鉴别模型。同时近红外光谱波段数量较大,波段间的相关性较高,一定程度上存在数据冗余等问题,因此数据降维一直是近红外光谱数据分析的流程之一。本文采用间隔影响分析(margin influence analysis, MIA)与连续投影算法(successive projections algorithm,SPA)相结合的方法对近红外光谱数据进行降维,然后应用偏最小二乘线性判别分析算法(partial least squares linear discriminant analysis,PLS-LDA)对腹泻性贝类毒素样本和健康样本的混合数据集进行分类,建立快速鉴别模型,以期为贝类毒素的快速无损鉴别提供一种新方法,为海产品污染物识别提供参考。
1 材料与方法
1.1 试验样品制备
样品购买自广东省湛江市东风海鲜市场,品种为翡翠贻贝。首先选取大小相似的贻贝,将其放置于塑料容器中进行驯化,以适应实验环境。驯化3 d后,选择生命力强的贻贝进行后续实验。预先准备好2个大小为119 cm×108 cm×32 cm塑料养殖箱,分别饲养健康样本(对照组)和毒素污染样本(实验组)。每个养殖箱中承装海水80 L,盐度为3%,温度为26 ℃。实验组养殖箱中加入浓度为7.3×109 细胞/L的利马原甲藻来模拟受DSP污染的海洋环境。对照组养殖箱中每天用0.5 L光合细菌喂养贻贝样本。
在实验过程中,为了保持贻贝良好的生理状态,利用氧气泵对养殖箱中的海水不断充气,海水每24 h更换1次,以保持贻贝的生活环境清洁。将贻贝样本在养殖箱中连续喂养6 d,使得DSP在贻贝样本中积累。剔除死的贻贝,共收集240个样本(每组120个样本)进行光谱采集。
1.2 光谱采集与预处理
贻贝从海水中取出后,利用近红外光谱系统(图1)获得贻贝的光谱信息,该系统由近红外光谱仪、卤素光源、光纤、计算机和可调位移平台组成。近红外光谱仪的型号为SW2520-050-NIRA(中国台湾OTO光电子有限公司)。采集贻贝的反射光谱范围在950 nm到1 700 nm之间,包括114个波段。光谱采集前,预先进行黑白校正以降低噪声[18]。

图1 近红外光谱系统
Fig.1 Near-infrared spectroscopy system
光谱采集后,将实验组DSP污染贻贝的样本内壳肉取出并冷冻,用于检测DSP含量,研究中采用GB 5009.212—2016《食品安全国家标准 贝类中腹泻性贝类毒素的测定》中的LC-MS/MS法进行检测。
采集到的原始光谱数据使用Savitzky-Golay卷积平滑求导结合标准正态变量变换用以消除光谱数据的噪声以及基线平移。对于表征样本是否为健康样本矩阵采用平均中值法预处理,去除变量中没有信息含量的均值部分。
1.3 MIA数据降维方法
MIA是一种基于模型集群分析思想,并以支持向量机(Support vector machines, SVM)的内在工作机制为基础的波段选择算法[19]。MIA方法源于间隔是反映SVM模型预测能力的一个重要指标,SVM模型的间隔越大,其结构风险就越小,其模型的泛化性能越好。也就是说,能够增加SVM模型间隔的变量是有信息变量,反之则是无信息变量甚至是干扰变量。MIA算法原理如图2所示。

图2 MIA方法的主要原理
Fig.2 Main principle of MIA method
假设有光谱训练样本集T={xi,yi|i=1,2,…,n }∈(X×Y)n,其中,n是样本数量;xi是训练样本集X内的第i个样本(p维向量), xi∈X=Rn;yi是训练样本集Y内的第i个样本的分类标签, yi∈Y={-1,1}。
(1)基于变量空间的蒙特卡洛抽样。图2中行为采样次数用N表示(一般较大),列为变量空间大小用p表示。采样过程如下:预先定义待采样变量的数量,用Q表示;每次采用无放回地从p个变量中随机选取Q个变量,得到n×Q的子数据集。重复N次,得到N个子数据集。图2每一行表示一次抽样即在变量空间随机选择Q个变量(黑色方块)。
(2)建立SVM子模型。给定一个惩罚因子C,对于(1)中每个随机抽样的子数据集,建立SVM分类模型。本研究中采用交叉验证来选择C。由此计算出其对应的N个间隔值,记为Mi,i=1,2,…,N。由图2可知每个子模型建模所用变量及其间隔。
(3)对SVM间隔进行统计分析。对于波长变量i,将N个子模型分为A类(包含波长变量i的模型)和B类(不包含波长变量i的模型)两类。基于这两类间隔的数据,可以计算得到波长变量i对应的2个分布,两类的间隔均值分别记为Mi,A和Mi,B,则波长变量i间隔分布均值的差可以表示为公式:
Dmeani=Mi,A-Mi,B
若Dmeani>0,表明SVM模型中加入波长变量i可以提高模型性能,反之亦然。对各波长变量的间隔分布,进行非参数Mann-Whitney U检验[20],计算差异显著性的P值。选择Dmeani >0且P<0.05的波长变量作为影响波段。MIA的最终目标是选择能够显著增加间隔的波长变量。
为了降低模型的复杂性,提高模型的性能,采用SPA进一步减少最终模型中包含的波段数量。
1.4 参数调优
MIA方法中有3个调优参数,分别是N、C和Q。为了考察这3个参数对模型的影响,在一系列取值下进行研究,并对结果进行了分析,以期得到一组最优化的参数。对于蒙特卡洛抽样次数,已有研究表明,N越大,得到的结果越好,但计算成本较高。考虑到计算成本和再现性,本工作中选择N=10 000。SVM的惩罚因子C用于控制对分类错误的惩罚力度,取值越大表示越不能容忍错分现象,但取值过大,容易出现过拟合。因此,选择合适的惩罚因子C是至关重要的。Q是每次采样划分子数据集的变量个数,本研究通过检验MIA识别的信息变量的再现性和相应的预测误差来确定Q的最佳取值。本研究利用交叉验证进行C和Q的优化。
为了选择合适的C和Q,本研究将贻贝的近红外数据集随机划分为训练集(96个健康样本和96个DSP样本)和测试集(24个健康样本和24个DSP样本)20次。样本的近红外光谱数据在训练集和测试集之间没有重复。本研究中,惩罚因子C在1~12取值,步长为1;子数据集变量个数Q的取值范围为10~50,步长为10。图3给出了对DSP贻贝和健康贻贝样本混合数据集执行MIA算法后得到的预测误差随参数C与Q的变化趋势。由图3可知,随着C值的增加,预测误差的变化会因Q的取值不同而不同,说明C和Q的取值均会影响MIA算法的结果,应选择预测误差最小结果对应的参数作为最优参数值。由图3-a亦可知,在采样划分子数据集的变量个数Q为30时,对于不同的C值,模型均取得较低的预测误差(图3-a中红色实线表示)。当Q=30且C=6时,算法取得了最小预测分类误差0.044 5(图3中五角星表示)。优化参数时,当对应C和Q的步长分别取值更小时,参数优化效果不明显,对模型结果影响不大。故此研究中,所选最优参数N为10 000,C为6,Q为30。
1.5 分类模型及评价标准
采用PLS-LDA,SVM和随机森林(random forest,RF)3种方法分别建立分类模型,比较不同模型对DSP贻贝、健康贻贝的识别准确率。同时,为了减少因样本集划分而导致的结果差异,提高模型稳定性,采用20次5折交叉验证法进行训练集、测试集的划分和模型训练,并将其平均值作为最终分类结果。
PLS-LDA是基于PLS算法改编的用来建立自变量与响应变量之间映射关系的一种监督分类方法。SVM是一种利用超平面对样本进行分类的方法,本研究中SVM使用的RBF作为核函数。RF是一种基于装袋和随机子空间的决策树集成方法,其输出的类别是由个别决策树输出类别的众树来决定的,可以用来解决高维数据分类等问题。
分类准确率可以直观评价模型好坏,但当使用不平衡数据集时,准确率并不能反映全面情况。本研究中引入受试者工作特征曲线(receiver operating characteristic,ROC)来衡量模型性能,它能够准确反映模型特异性和敏感性的关系,是实验准确性的综合代表,一般以ROC曲线下面积(area under the curve,AUC)作为模型评价指标,其值越大代表模型的鉴别效果越好,其值最大为1,分类器的性能与AUC成正比[21]。
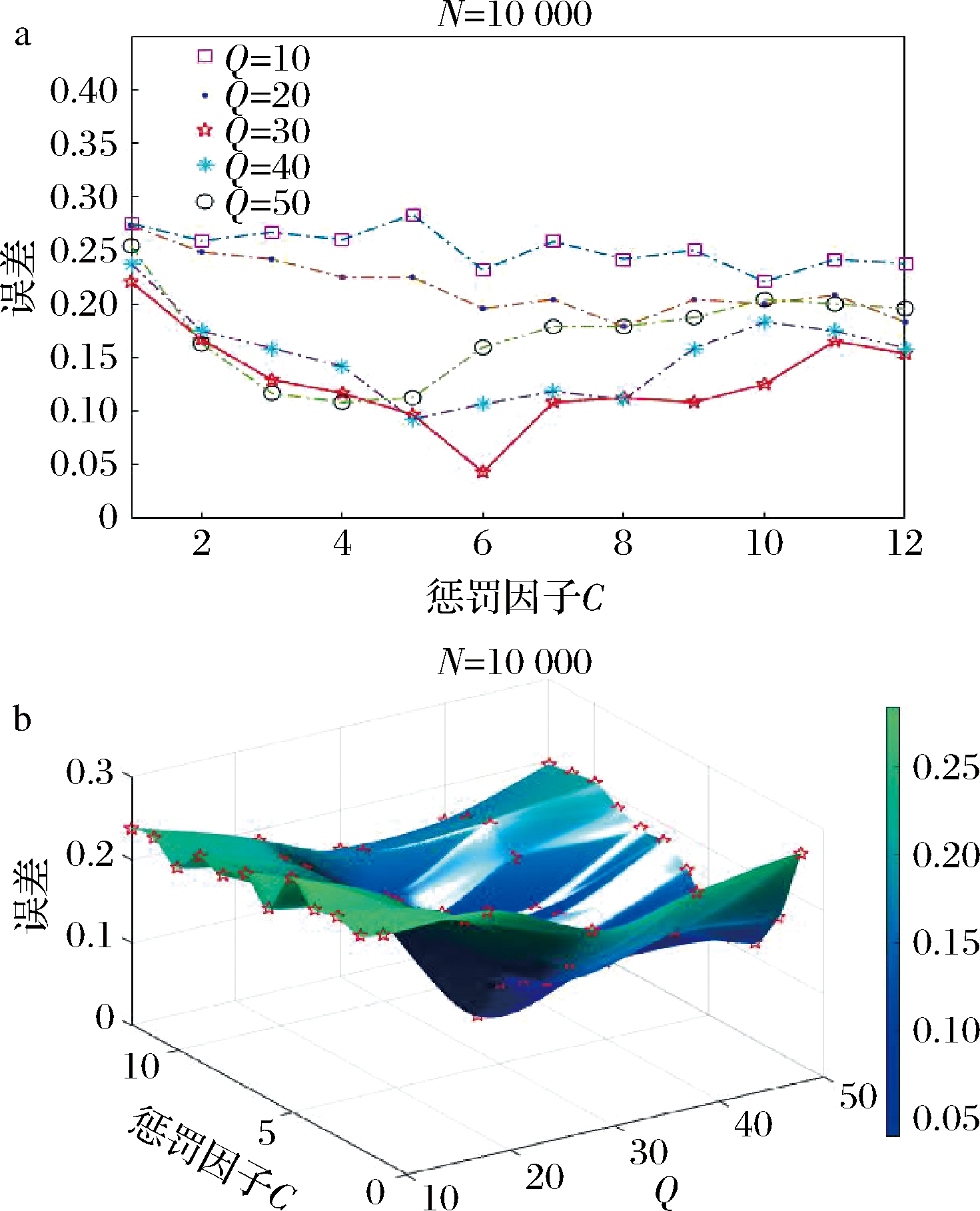
a-不同Q值下预测误差随C变化二维图; b-不同Q值下预测误差随C变化三维图
图3 惩罚因子C与采样变量数Q对预测误差的影响
Fig.3 Effect of penalty factor C and sampling variable number Q on prediction error
2 结果与分析
2.1 健康样本和DSP污染样本的原始光谱分析
混合样品集(120个DSP样品和120个健康样品)的原始近红外光谱如图4-a所示。图中光谱曲线走向趋势相似,说明二者的内部含有的化学成分大致相似。在1 050 nm附近有明显的波峰,为N—H的三级倍频[22],可能与贻贝中蛋白质的近红外吸收有关。图4-b显示了混合样本集120个DSP贻贝和健康贻贝样品平均光谱曲线,二者表现出一定的差异,在950 nm至1 080 nm波长范围内,健康样品的光谱反射率值高于DSP污染样品的光谱反射率值;在1 150~1 700 nm,DSP污染贻贝反射强度高于健康样本,在其他波长范围内,2种样品的平均光谱曲线几乎重叠。光谱采集后,对实验组样本采用LC-MS/MS法检测DSP含量为35 μg/kg。
贝类样品基质复杂,内源性化合物的存在可能会改变酶的自然环境,影响其活性。当贻贝受到海洋毒素污染时,会产生蛋白质、酶和脂质等组织成分的变化,这些变化反映在光谱曲线上。这些光谱的差异为区分DSP污染样品和健康样品提供了可行性。

a-原始光谱图;b-平均光谱图
图4 样本近红外光谱
Fig.4 Near-infrared spectra of samples
由于波长和重要背景的重叠,很难明确识别特定化学成分的波长。为了探索DSP污染和健康样本的分布,采用主成分分析(principal component analysis,PCA)提取新变量,以发现样品之间可能存在的聚类现象。第一个主成分(PC1)、第二个主成分(PC2)和第三个主成分(PC3)分别解释了总方差的95.60%、2.08%和1.32%,累计方差贡献度在99%以上。图5所示为DSP污染贻贝和健康贻贝的PCA得分图和载荷图。可以注意到,图5-a中,健康样品比DSP污染样品更紧密地聚集在一起。然而,DSP污染样品和健康样品之间仍然存在重叠。明显的重叠意味着仅仅用简单的分类方法不能够区分它们。因此,需要化学计量学方法和模式识别方法对DSP污染和健康样品进行分类。如图5-b所示,对PC1贡献最大的波段为1 030 nm,对PC2贡献最大的波段为1 470 nm和1 050 nm,对PC3贡献最大的波段为1 690 nm和1 010 nm。根据相关文献,波段1 470 nm主要对应的是与蛋白质相关的谱带,1 690 nm归属于脂质信号[23]。
在获取近红外光谱数据时,由于近红外光谱系统的工作条件和样品结构特性的变化可能会导致光谱中的基线漂移、随机噪声和多元散射效应。为了改进近红外光谱数据,通常采用光谱预处理方法来减少这些影响[24]。本研究采用Savitzky-Golay卷积平滑求导(多项式阶数为2,滑动窗口宽度为13,求导阶数为1)结合标准正态变量变换对原始光谱进行预处理,以消除光谱数据的噪声及基线漂移,可以增强鉴别模型的鲁棒性。
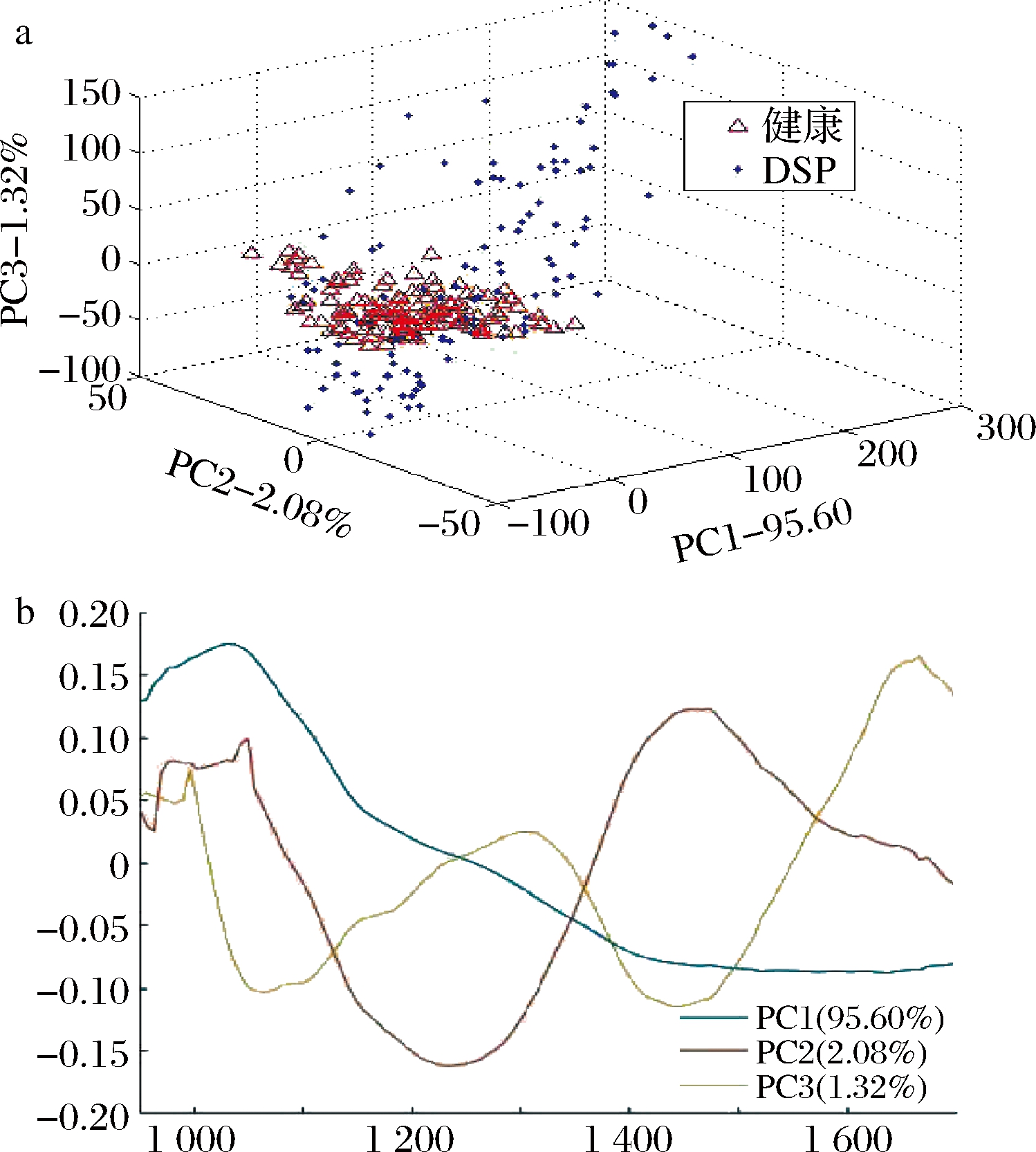
a-得分图;b-载荷图
图5 DSP样本与健康样本数据集PCA得分散点图及载荷图
Fig.5 PCA score scatter diagram and load diagram of data set for DSP sample and health samples
2.2 选取特征波长
采用MIA方法进行波长变量筛选,对于预处理后的DSP贻贝与健康贻贝混合集光谱数据,分别选出一个有信息的波长与一个无信息的波长,其对应的间隔的分布如图6所示。图6-a和图6-b所对应的分布分别为强信息变量(波段1 473.77 nm,P=6.733 2×10-12)和无信息变量(波段989.78 nm,P=1.730 2)的分布。其中,峰“1”表示含有相应波长变量的SVM模型的间隔分布,峰“0”表示不包含此波长变量的SVM模型的间隔分布。很明显,图6-a中包含波长1 473.77 nm与不包含波长1 473.77 nm变量建立模型的间隔分布存在明显差异。包含波长1 473.77 nm 变量建立的模型间隔分布发生了右移,其间隔平均值大于不包含该波长变量的,意味着该变量蕴藏着对DSP贻贝和健康贻贝进行判别的有用信息。这意味着将波长1 473.77 nm变量加入SVM模型中,可以提高模型的泛化性能。相比之下,图6-b中,包含波长989.78 nm与不包含波长989.78 nm变量所建立的SVM模型的间隔分布没有明显差异,且包含波长989.78 nm变量的SVM模型的间隔分布位于左侧,说明此变量对DSP贻贝和健康贻贝鉴别是无用的信息变量,将其加入到SVM模型中,不但不会增加,反而会减小模型的间隔,降低模型的预测能力,需要从模型中剔除。
为了建立DSP贻贝和健康贻贝混合样本数据集预测的分类模型,应该确定一个波段子集。经过MIA方法分析后,对于950~1 700 nm波段范围内的114个波长变量,各波长变量Dmean值的分布如图7所示。根据算法原理,Dmean<0为无信息变量,由图7可知,位于0线以下的均是此类变量,有25个变量被剔除。利用非参数Mann-Whitney U和Holm-Bonferroni方法进行统计检验,对剩余的89个变量按计算获得的每个波长变量P值进行排序(Dmean>0),然后按顺序选出一定数量的波长变量建模,并采用交叉验证对预测性能进行评价。由于MIA中使用了蒙特卡洛策略,所建立的SVM分类器的预测误差不能准确地再现,因此,通过运行20次MIA程序来研究分类误差随变量数量的变化。光谱数据集的平均5折交叉验证误差及标准差如图8所示。由图8可知,当模型输入变量少于35个时,平均5折交叉验证误差随变量数的增加都是先逐渐减小,然后达到最小。超过35个变量时,误差随变量数的增加逐渐增大。最终确定35个特征变量能够显著增加SVM间隔,即对DSP贻贝和健康贻贝鉴别有用的变量。
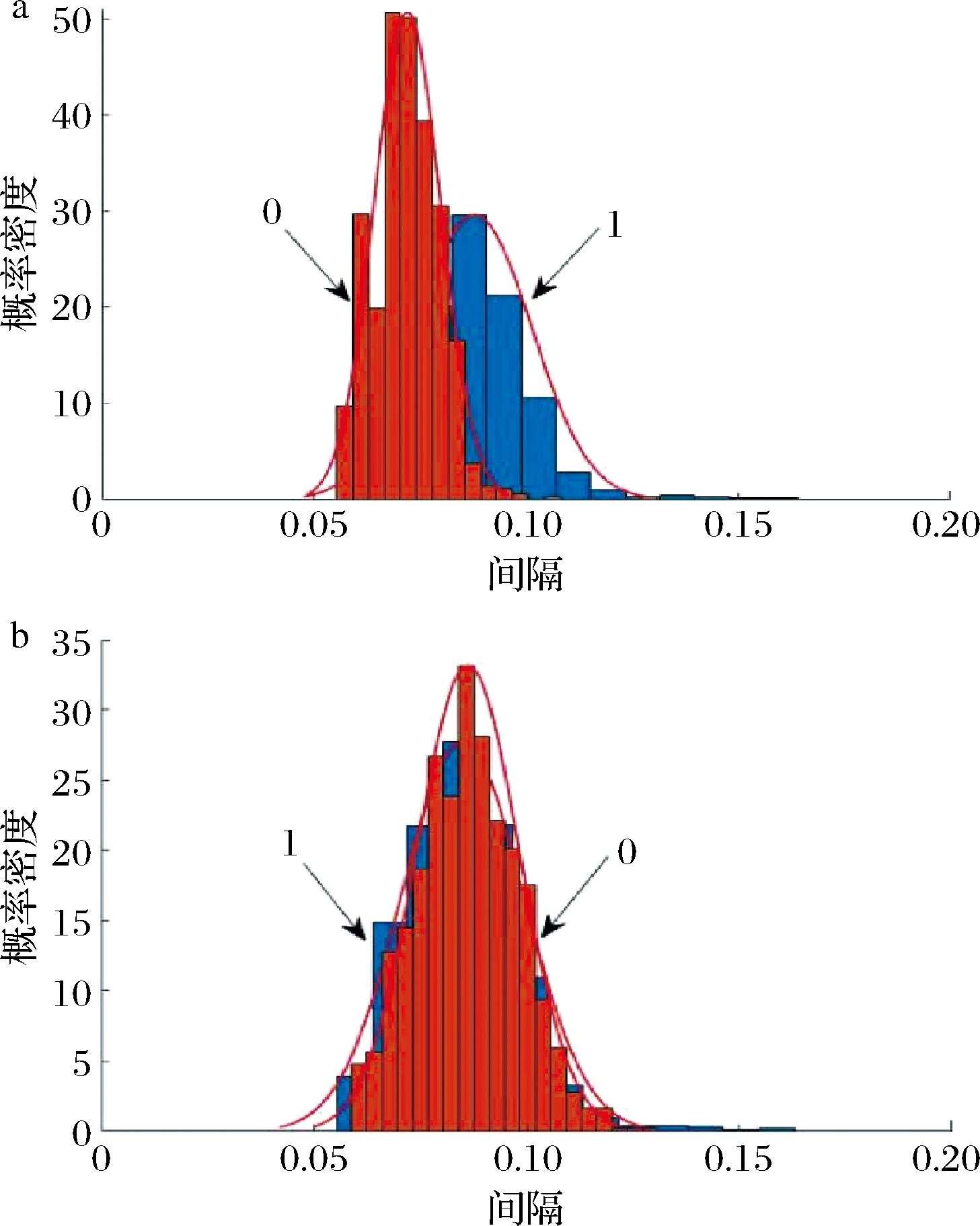
a-有信息波长1 473.77nm;b-无信息波长989.78nm
图6 两类信息变量SVM模型的间隔分布
Fig.6 Margin distribution of SVM model with two types of information variables
为了进一步减少波段数量,简化最终模型,对MIA选择的35个特征变量采用SPA获取具有最小冗余信息波段,并依据模型的交叉验证均方根误差的最小值来确定特征波长变量个数,最终筛选出来的特征波长共有7个,分别为1 029.56、1 473.77、1 480.40、1 520.18、1 526.81、1 672.67、1 692.56 nm。最后选择的特征波段如图9所示。
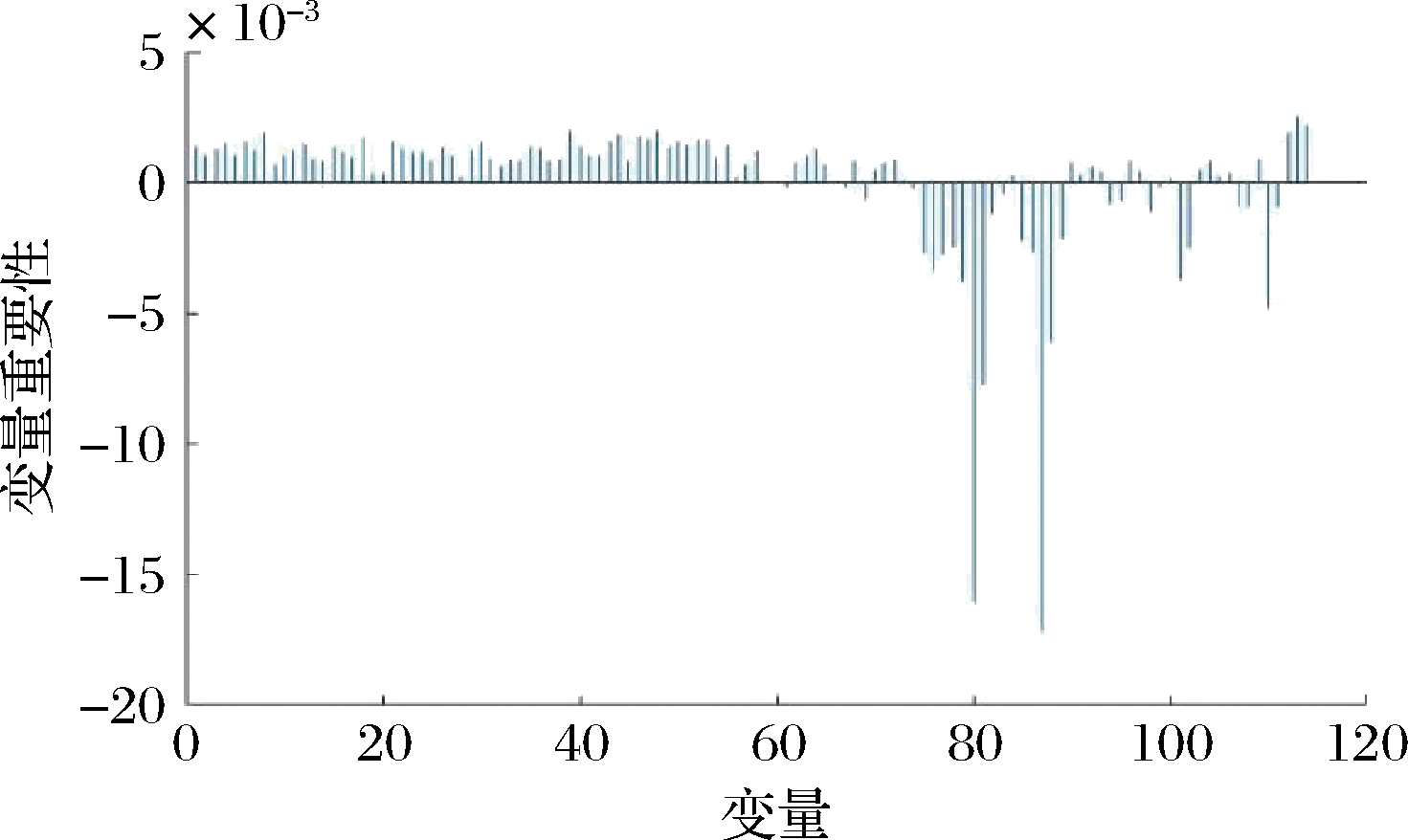
图7 运行MIA方法后每个波长变量的Dmean值
Fig.7 Dmean value of each wavelength variable after running the MIA method
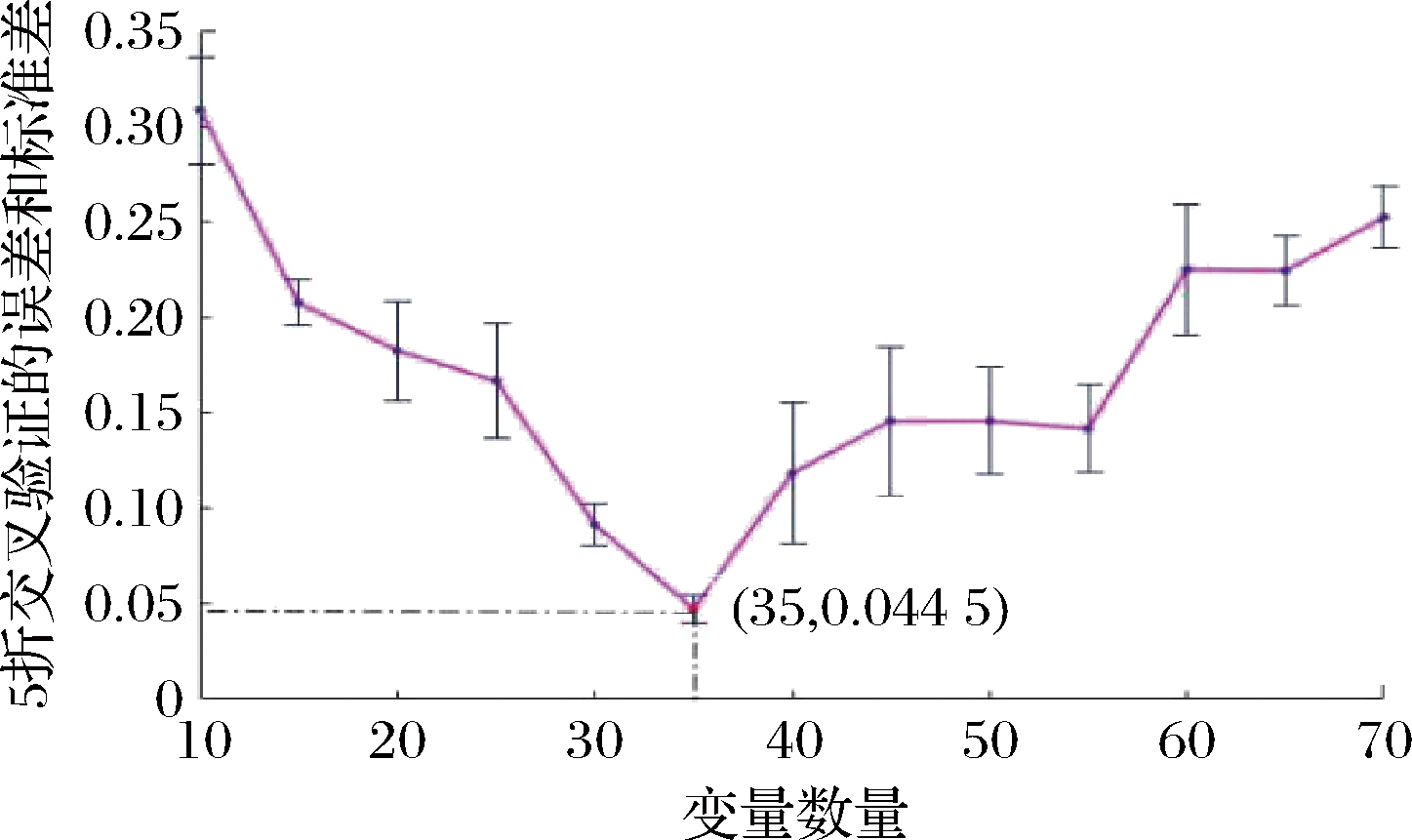
图8 选择不同变量数目时的5折交叉验证误差及标准差
Fig.8 Error and standard deviation of 5-fold cross-validation when choosing different numbers of variables
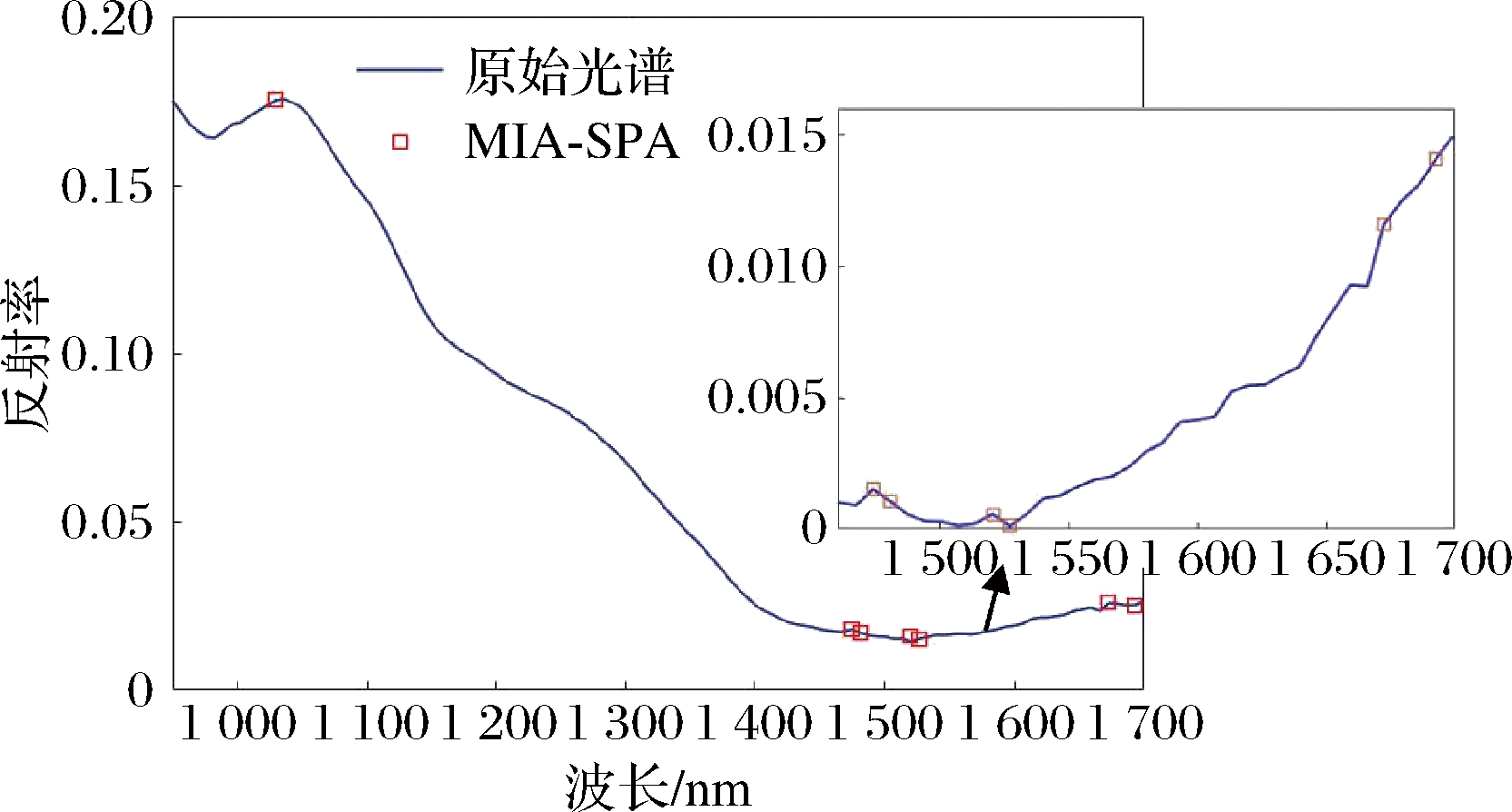
图9 MIA-SPA方法选择的波长变量的分布
Fig.9 Distribution of wavelength variables selected by the MIA-SPA method
2.3 判别模型及评价
为了构造最优鉴别模型,本项目对MIA-SPA特征波长提取方法得到的特征变量,分别应用PLS-LDA、SVM和RF建立DSP样本和健康样本的无损鉴别模型,并与MIA、SPA和全波段光谱建立的模型进行对比分析。不同降维方法下各模型的鉴别结果如表1所示。由表1可知,PLS-LDA对DSP样本和健康样本均具有很高的鉴别能力,其中DSP样本的分类准确率达到了100%;RF对DSP样本和健康样本的分类准确率偏低,不能达到分类要求;而SVM对DSP样本的分类准确率较高,达到了96.67%,但是对健康样本的分类准确率在85%以下,低于PLS-LDA的分类性能。由表1可知采用全波段建立的PLS-LDA、SVM和RF模型的分类准确率较采用降维后的变量建立的模型低,这是由于采集的样本光谱数据中包含了对表征样本特征相关性较差的波段,这些波段会降低模型的分类效果,而数据降维可筛选出更能表征样本的特征波段并将无关的波段予以删除。与MIA、SPA相比,采用MIA-SPA方法筛选的特征波长建立的模型具有较好的分类准确率,特别是MIA-SPA-PLS-LDA模型的分类效果最优,对DSP污染样本鉴别准确率达到100%。
表1 不同降维方法下各模型的分类效果
Table 1 The classification effect of each model under different dimensionality reduction methods
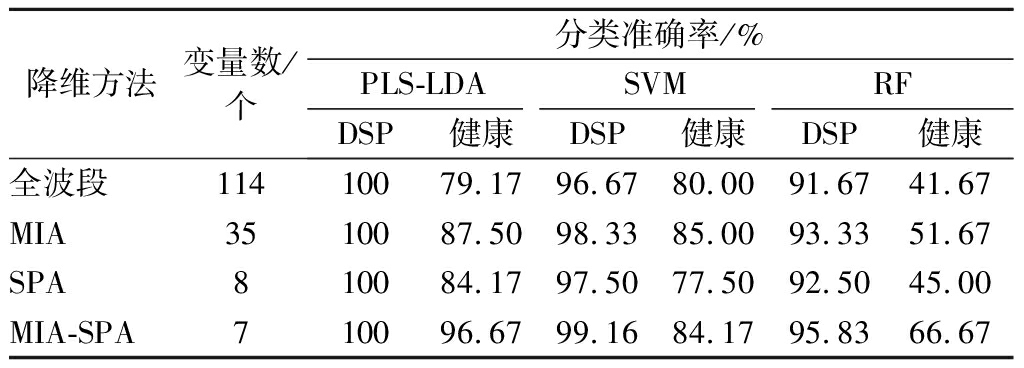
降维方法变量数/个分类准确率/%PLS-LDASVMRFDSP健康DSP健康DSP健康全波段11410079.1796.6780.0091.6741.67MIA3510087.5098.3385.0093.3351.67SPA810084.1797.5077.5092.5045.00MIA-SPA710096.6799.1684.1795.8366.67
为了进一步评价各种分类模型的性能,分别作出了上述分类模型ROC曲线。图10所示为不同降维方法下的3种鉴别模型的ROC曲线对比结果图。由图10可知,相同的降维方法得到的输入变量所建立的3类模型中,PLS-LDA模型的AUC相对SVM与RF模型的要大,说明本研究中PLS-LDA模型具有更好的性能;由图10-a可知,采用MIA-SPA降维后建立的PLS-LDA模型的AUC为0.989,均高于其他模型,说明MIA-SPA-PLS-LDA模型对DSP样本和健康样本均具有很高的鉴别能力。
2.4 讨论
本研究采用MIA-SPA-PLS-LDA结合近红外光谱技术对腹泻性贝类毒素进行检测。贻贝被DSP污染后体内发生极其复杂的化学转化和酶转化机制,将导致受污染贻贝和健康贻贝的化学成分存在差异。已有研究表明,这些差异可以被近红外光谱捕获[17,25],因此,本文采用近红外光谱检测DSP污染是有依据支撑的。本文进一步验证了近红外光谱结合波段选择方法以及判别模型,无损检测DSP污染的贻贝是可行的。
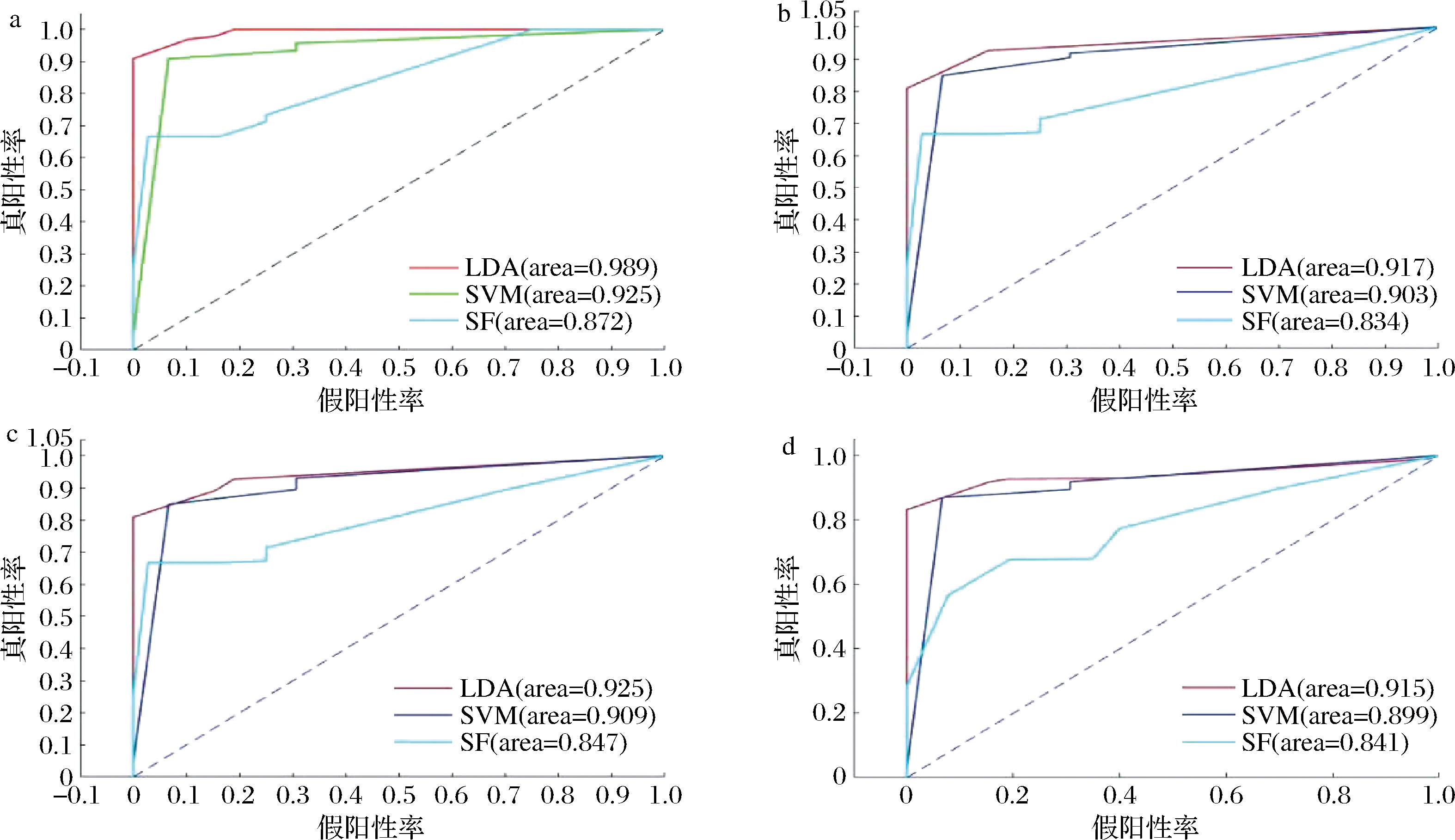
a-MIA-SPA;b-全波段;c-MIA;d-SPA
图10 不同降维方法下各分类模型的ROC曲线
Fig.10 ROC curves of each classification model under different dimensionality reduction methods
贻贝因形态及非光滑外壳可能会导致光谱变异性和光散射,因此,本文采用Savitzky-Golay卷积平滑求导结合标准正态变量变换预处理,可有效消除光谱数据的噪声及基线漂移。特征选择可以筛选出与DSP检测最相关的波段信息,同时提高分类模型的性能,本文提出采用MIA-SPA方法获取最优特征波段,据报道,在1 470 nm处发现了与蛋白质相关的谱带,在1 690 nm发现了脂类信号,说明MIA-SPA算法选择的波段可以反映光谱差异[23]。结果表明,从光谱中提取重要信息,可以实现对含DSP贻贝的快速检测。
3 结论
本文采用近红外光谱技术和化学计量学方法,对DSP污染和健康贻贝的混合样本集进行无损鉴别研究。采用MIA-SPA方法能够有效地对光谱数据进行降维(降维后变量为7个),同时结合PLS-LDA方法能够较好地对样本进行无损鉴别(DSP分类准确率为100%),通过ROC曲线分析,MIA-SPA-PLS-LDA方法的AUC最大,其模型鉴别效果最优。结果表明,MIA-SPA-PLS-LDA模型用于DSP污染快速无损鉴别是可行的,对DSP污染贻贝的检测准确率达到100%,DSP含量检出限为35 μg/kg。该研究结果可为后续各种海洋贝类毒素鉴别分析提供理论依据。
[1] YANG L, SINGH A, LANKFORD S K, et al.A rapid method for the detection of diarrhetic shellfish toxins and azaspiracid shellfish toxins in washington state shellfish by liquid chromatography tandem mass spectrometry[J].Journal of AOAC International,2020,103(3):792-799.
[2] WANG X Y, BOUZEMBRAK Y, HANS J P M, et al.Bayesian networks modeling of diarrhetic shellfish poisoning in Mytilus edulis harvested in Bantry Bay,Ireland[J].Harmful Algae,2022,112:102171.
[3] 宿志伟,赵峰,刘远平,等.固相吸附毒素跟踪技术监测牡蛎养殖区中腹泻性贝毒[J].渔业科学进展, 2016,37(6):144-150.
SU Z W, ZHAO F, LIU Y P, et al.Monitoring of diarhettic shellfish poisons in aquatic environment by solid phase adsorbent toxin tracking technology[J].Progress in Fishery Sciences,2016,37(6):144-150.
[4] CORRIERE M, SOLI O L, COSTA P R.Effects of the marine biotoxins okadaic acid and dinophysistoxins on fish[J].Journal of Marine Science and Engineering,2021,9(3):293.
O L, COSTA P R.Effects of the marine biotoxins okadaic acid and dinophysistoxins on fish[J].Journal of Marine Science and Engineering,2021,9(3):293.
[5] JIMÉNEZ-C RCAMO D, GARC
RCAMO D, GARC A C, CONTRERAS H R.Toxins of okadaic acid-group increase malignant properties in cells of colon cancer[J].Toxins,2020,12(3):179.
A C, CONTRERAS H R.Toxins of okadaic acid-group increase malignant properties in cells of colon cancer[J].Toxins,2020,12(3):179.
[6] 彭志兰, 罗海军, 王维洁, 等. 舟山海域麻痹性贝类毒素污染情况及其2种检测方法比较[J].食品安全质量检测学报, 2017, 8(4):1 436-1440.
PENG Z L, LUO H J, WANG W J, et al.Investigation of paralytic shellfish poison in waters of Zhoushan and comparison of 2 detection methods[J].Journal of Food Safety & Quality, 2017, 8(4):1 436-1 440.
[7] 胡雪莲,俞漪,曲勤凤,等.腹泻性贝类毒素(DSP)的两种快速检测方法比较[J].食品研究与开发,2012,33(12):127-130.
HU X L, YU Y, QU Q F, et al.The comparison of two screening detecting method of diarrhetic shellfish poisoning(DSP)[J].Food Research and Development,2012,33(12):127-130.
[8] 宿志伟, 赵峰, 姜雪, 等.桑沟湾养殖牡蛎中贝类毒素监测及预警[J].食品科学, 2017, 38(6):303-309.
SU Z W, ZHAO F, JIANG X, et al.Monitoring and early warning of shellfish toxins in oyster cultured in sanggou bay[J].Food Science,2017,38(6):303-309.
[9] 孟庆龙, 尚静, 黄人帅, 等.苹果可溶性固形物的可见/近红外无损检测[J].食品与发酵工业, 2020, 46(19):205-209.
MENG Q L, SHANG J,HUANG R S, et al. Nondestructive detection of soluble solids content in apple by visible-near infrared spectroscopy[J].Food and Fermentation Industries, 2020, 46(19):205-209.
[10] 褚小立,史云颖,陈瀑,等.近五年我国近红外光谱分析技术研究与应用进展[J].分析测试学报.2019,38(5):603-611.
CHU X L, SHI Y Y, CHEN P, et al.Research and application progresses of near infrared spectroscopy analytical technique in china in past five years[J].Journal of Instrumental Analysis,2019,38(5):603-611.
[11] 陈嘉, 高丽, 叶发银,等.基于近红外光谱与支持向量机的甘薯粉丝掺假快速检测[J].食品与发酵工业,2019,45(11):211-218.
CHEN J, GAO L, YE F Y, et al.Rapid detection of adulterated sweet potato starch noodle by near-infrared spectroscopy and support vector machine[J].Food and Fermentation Industries,2019,45(11):211-218.
[12] 张欣欣,李尚科,李跑,等.近红外光谱的不同产地柑橘无损鉴别方法[J].光谱学与光谱分析,2021,41(12):3 695-3 700.
ZHANG X X, LI S K, LI P, et al.A nondestructive identification method of producing regions of citrus based on near infrared spectroscopy[J].Spectroscopy and Spectral Analysis,2021,41(12):3 695-3 700.
[13] 袁瑞瑞,刘贵珊,何建国,等.可见近红外高光谱成像对灵武长枣定量损伤等级判别[J].光谱学与光谱分析,2021,41(4):1 182-1 187.
YUNA R R, LIU G S, HE J G, et al.Quantitative damage identification of lingwu long jujube based on visible near-infrared hyperspectral imaging[J].Spectroscopy and Spectral Analysis,2021,41(4):1 182-1 187.
[14] 张强,刘成海,孙井坤,等.基于支持向量机的稻谷黄曲霉毒素B1近红外无损检测[J].东北农业大学学报.2015,46(5):84-88.
ZHANG Q, LIU C H, SUN J K, et al.Near-infrared spectroscopy nondestructive determination of aflatoxin B1 in paddy rice based on support vector machine regression[J].Journal of Northeast Agricultural University,2015,46(5):84-88.
[15] 黄冠明,郭香,祁剑飞,等.葡萄牙牡蛎(Crassostrea angulata)六种化学成分近红外定量模型的建立[J].光谱学与光谱分析,2020,40(5):1 509-1 513.
HUANG G M, GUO X, QI J F, et al.Establishment of quantitative model for six chemical compositions in Crassostrea angulata by near infrared spectroscopy[J].Spectroscopy and Spectral Analysis, 2020,40(5):1 509-1 513.
[16] LIU Y, XU L L, ZENG S G, et al.Rapid detection of mussels contaminated by heavy metals using near-infrared reflectance spectroscopy and a constrained difference extreme learning machine[J].Spectrochinica Acta Part A:Molecular and Biomolecular Spectroscopy,2022,269:120776.
[17] 刘忠艳, 刘瑶, 乔付, 等.基于近红外光谱和多层感知机的贻贝中腹泻性贝毒快速无损检测[J/OL].食品与发酵工业,2022.https://doi.org/10.13995/j.cnki.11-1802/ts.032253.
LIU Z Y, LIU Y, QIAO F, et al.Rapid non-destructive detection of diarrheal shellfish poison in mussels based on near-infrared spectroscopy and multi-layer perceptron[J/OL].Food and Fermentation Industries,2022.https://doi.org/10.13995/j.cnki.11-1802/ts.032253.
[18] 何鸿举, 王魏, 王洋洋, 等. 基于近红外高光谱技术快速检测冷鲜猪肉酸价[J].食品与发酵工业, 2020, 46(10):264-270.
HE H J, WANG W, WANG Y Y, et al.NIR hyperspectral imaging technology for rapid detection of acid value in fresh chilled pork[J].Food and Fermentation Industries, 2020, 46(10):264-270.
[19] LI H D,LIANG Y Z,XU Q S,et al.Recipe for uncovering predictive genes using support vector machines based on model population analysis[J].IEEE/ACM Transactions on Computational Biology and Bioinformatics,2011,8(6):1 633-1 641.
[20] MANN H.B,WHITNEY D R.On a test of whether one of two random variables is stochastically larger than the other[J].The Annals of Mathematical Statistics, 1947, 18(1):50-60.
[21] 楚秉泉,赵艳茹,何勇.基于近红外高光谱技术和特征波谱分析方法的竹类判别研究[J].光谱学与光谱分析,2017,37(6):1 718-1 722.
CHU B Q, ZHAO Y R, HE Y.Discriminant analysis of bamboo leaf types with nir coupled with characteristic wavelengths[J].Spectroscopy and Spectral Analysis,2017,37(6):1 718-1 722.
[22] 白宗秀,朱荣光,王世昌,等.高光谱图像结合特征变量筛选定量检测羊肉中狐狸肉掺假[J].农业工程学报,2021,37(17):276-284.
BAI Z X, ZHU R G, WANG S C, et al.Quantitative detection of fox meat adulteration in mutton by hyper spectral imaging combined with characteristic variables screening[J].Transactions of the Chinese Society of Agricultural Engineering(Transactions of the CSAE), 2021, 37(17):276-284.
[23] MAMANI-LINARES L W, GALLO C, ALOMAR D.Identification of cattle, llama and horse meat by near infrared reflectance or transflectance spectroscopy[J].Meat Science,2012,90(2):378-385.
[24] TUNCER F D, DOGU D, AKDENIZ E.Efficiency of preprocessing methods for discrimination of anatomically similar pine species by NIR spectroscopy[J].Wood Material Science & Engineering,2021:1-10.
[25] LIU Y, QIAO F, XU L L, et al.Fast detection of diarrhetic shellfish poisoning toxins in mussels using nir spectroscopy and improved twin support vector machines[J].Frontiers in Marine Science,2022,9:907378.