脂肪酶(lipase, EC3.1.1.3),是一类在亲水亲脂界面分解和合成甘油三酯的酶,底物一般为长碳链(C8以上)的酰基甘油。这些酶在结构上都有α/β水解酶的折叠形式和保守的GXSXG五肽模体,属于丝氨酸水解酶类[1]。但在脂肪酶的应用中,尤其是工业生产中,对酶热稳定性的要求比较高,而天然脂肪酶的热稳定性常常难以满足生产要求。因此,对嗜热脂肪酶新酶的开发和研究一直是脂肪酶研究的重要方向之一。
嗜热微生物能够适应极端的生存环境是其不断进化的结果,其体内有较大可能会存在与高温环境相适应的各种酶类[2]。目前已从多种嗜热环境微生物的发酵产物中获得了不同的嗜热脂肪酶如Amycolatopsis mediterranei[3]、Aneurinibacillusther moaerophilus[4]、Geobacillusther moleovorans[5-6]、Bjerkandera adusta[7]、Lactobacillus plantarum[8]、Staphylococcus aureus[9]、Streptomyces thermocarboxydus[10]、Thermoanaerobacter thermohydrosulfuricus[11]、Thermosyntropha lipolytica[12]。此外,利用宏基因组技术绕过微生物培养环节,直接将环境样品中的DNA异源表达后再筛选产物的方法,同样从热泉环境中发现了多个嗜热的新脂肪酶[13-14]。
随着测序技术的发展,微生物基因组数据和各种环境的宏基因组测序数据获得急速增长。这些海量数据的绝大部分只有简单的注释,其具体功能特征并不清楚。这意味着上述测序数据里,尤其是来源于新微生物或极端环境的数据中蕴含着大量的等待挖掘的新基因和新酶。周艳杰等[15]从嗜热玫瑰红球菌ThermomicrobiumroseumDSM 5159的基因组数据中发现了新嗜热脂肪酶。有研究从基因数据库中筛选到来源于超嗜热菌Aquiexaeolicus VF5的两种嗜热新酯酶Aaeo1和Aaeo2[16]。同时,ABDELMONAEM等[17]、YADAVA等[18]研究表明脂肪酶在进化过程中具有菌属特征。因此,本文考虑从特殊的嗜热菌群中通过生物信息学手段结合异源表达技术来发现新嗜热脂肪酶。
Caldisericum exile AZM16c01是热泉新菌[19],为厌氧、嗜热、革兰氏阴性纤细热丝菌。最适生长温度为65 ℃,最适pH为6.5,对NaCl非常敏感。其基因组测序已完成,该文对其进行基因资源筛选分析,发现 BAL81435.1(GenBank ID)为酯酶,经试验证实,该酯酶具有脂肪酶的特性。据我们所知,这是首次对来源于Caldiserica新门的嗜热脂肪酶特征的描述。该研究为新酶的快速开发提供了参考。
1 材料与方法
1.1 材料
基因序列BAL81435.1由上海生物工程有限公司合成并克隆到pET28(a)载体上(在载体BamHI/XhoI之间插入该基因编码框,使其表达的重组蛋白N末端带有6×His 标签),获得pET28a-lipase-BAL质粒。DH5α, Rosetta 感受态细胞,上海唯地生物有限公司。
1.2 主要试剂
Ni-NTA Sefinose Resin试剂盒、Tube-O-DIALYZER透析柱、BCA蛋白浓度测定试剂盒、His单克隆抗体和二抗,上海生物工程有限公司。
1.3 实验方法
1.3.1 蛋白质表达、纯化和鉴定
将质粒pET28a-lipase-BAL转化入Rosetta细胞中。向100 mL新鲜LB培养基中加入2 mL过夜培养的阳性克隆菌液,于37 ℃培养2~4 h至OD600值约为0.4~0.6,然后向培养基中加入终浓度为1 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG),继续在25 ℃下诱导培养12~16 h。离心回收菌体,弃上清液,加入8 mL破碎缓冲液(含有PBS、300 mmol/L NaCl、3% Triton X-100)重悬菌体,超声破碎,4 ℃12 000 r/min离心15 min保留上清液。上清液经Ni-NTA Sefinose离子亲和层析纯化重组蛋白,操作步骤参照说明书进行。250 mmol/L咪唑洗脱得到的重组蛋白经Tube-O-DIALYZER透析柱除去咪唑后,用于鉴定和后续试验。蛋白表达样品和纯化后蛋白样品利用SDS-PAGE电泳和Western Blot(一抗为His单抗)进行鉴定。
1.3.2 脂肪酶活力测定
脂肪酶活性测定采用参考文献[20]对硝基苯酚(para-nitrophenol,p-NP)比色法进行。其过程简述如下:溶液A:称取适量的底物对硝基苯磷酸酯(para-nitrophenyl phosphate,p-NPP)溶于30 mL异丙醇中;溶液B:配制适量浓度为100 mmol/L的Tris-HCl缓冲液(pH为8.8)。将A和B溶液混匀置于适当温度下反应一定时间,通过测定OD405值,由p-NP标准曲线计算p-NP的量,换算成酶活性单位。1U表示在上述条件下,每分钟催化p-NPP释放1 μmol/L p-NP所需的酶量。
1.3.3 温度对酶活性的影响
将0.1 mL溶液A和3.7 mL溶液B充分混匀后,加入0.2 mL酶液,以等体积的灭活的酶液为对照,在不同温度下(4、18、25、37、42、50、60、70、75 ℃)反应30 min后,测定OD405值,计算酶活性。
将0.1 mL溶液A和3.7 mL溶液B充分混匀后,加入0.2 mL酶液,在不同温度下(37、42、60、70 ℃)反应90 min,并分别在不同时间段(15、30、45、60、90 min)取样测定OD405值,计算酶活性,以检测脂肪酶的温度耐受性。
1.3.4 pH对酶活性的影响
取0.1 mL溶液A至3.7 mL pH不同(6.8、8.0、8.4、8.8、9.2、9.6、10.4、10.6)的溶液B中,加入0.2 mL酶液,在最适温度下反应30 min后,测定OD405值,计算酶活性。
1.3.5 有机溶剂对酶活性的影响
将0.1 mL溶液A和3.7 mL溶液B充分混匀后,分别加入不同浓度(5%、10%),不同种类(甲醇、乙醇、丙酮、氯仿、乙氰、二甲基亚砜)的有机溶剂,充分混匀,加入0.2 mL酶液,在最适温度下反应30 min测定其OD405值,计算酶活性。
1.3.6 表面活性剂、变性剂及金属离子对酶活性的影响
将0.1 mL溶液A和3.7 mL溶液B混匀后,加入适量表面活性剂、变性剂或金属离子,加入0.2 mL酶液,在最适温度下反应30 min测定其OD405值,计算酶活性。
1.4 数据分析方法
参照JO等[5]、ABDELMONAEM等[17]、YADAVA等[18]的研究从NCBI数据库中,提取代表性lipase脂肪酶序列。首先,利用clustal W程序对得到酶蛋白序列进行多序列比对[21]。然后,利用Mega X程序基于连接法(bootstrap 自检1 000次)构建进化树[21]。
2 结果与分析
2.1 BAL81435.1序列分析
Caldisericum exile AZM16c01是海洋热泉来源的厌氧、嗜热、革兰氏阴性纤细热丝菌,最适生长温度为65 ℃,其基因组测序已经完成,目前未见与该菌有关的脂肪酶研究报道。对该菌的基因组数据(AP012051.1)筛选发现序列BAL81677和BAL81435.1的功能注释为酯酶。对BAL81435.1的进一步分析发现,其预测蛋白分子质量大小为28 kDa,等电点为6.6,无信号肽(http://smart.embl.de/)。利用blast比对发现(https://blast.ncbi.nlm.nih.gov/Blast.cgi),与BAL81435.1序列相似性高的序列分布于多个种属,大部分都来源于热泉环境。进一步选择与BAL81435.1编码蛋白Identity>40%的代表性序列进行多序列比对,结果如图1所示。这些酶存在多个保守区域,其中催化区域[Gly-X(1)-Ser-X(2)-Gly-]中绝大多数的X(1)为Lys,X(2)为Met,并且该模体后都紧邻接Gly,催化区域呈现GLSMGG模式。在DEMIRJIAN等[1]、ATALAH等[2]的研究中,分子进化分析显示脂肪酶可以分为六大类。为进一步分析BAL81435.1与其他脂肪酶的进化关系,该文选择了脂肪酶各类代表性序列和酯酶序列,以邻居连接(neighbor-joining, N-J)方法构建了进化树,结果如图2所示,BAL81435.1及其他Caldiserica来源的序列与其他脂肪酶和酯酶有着不同的进化分支。这可能表明BAL81435.1编码新的嗜热脂肪酶。为验证这个猜想,该研究对该序列的表达产物进行了研究。

图1 BAL81435.1编码蛋白与其同源性较高的蛋白多序列比对结果
Fig.1 BAL81435.1encoding protein and its high homology protein multiple sequencecomparison results
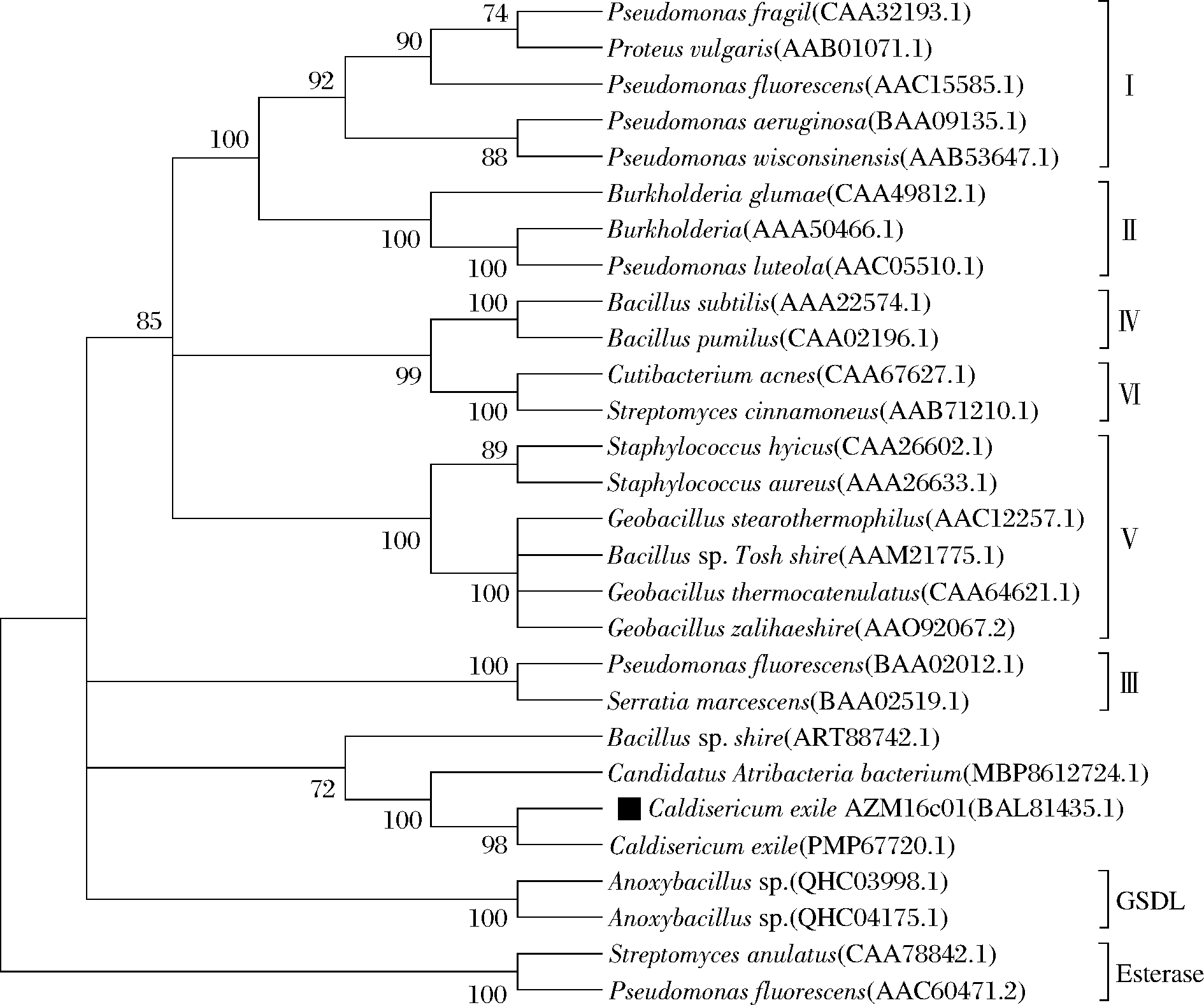
图2 脂肪酶的系统进化树
Fig.2 Phylogenetic tree of lipase 注:BAL81435.1及其相关蛋白的系统发育分析;系统进化树 采用邻居连接法(MEGAX.0软件)生成;从GenBank中提取 原细菌脂解酶的蛋白序列;节点上的数字表示1 000个重复的 bootstrap百分比;■表示该研究选择的研究对象
2.2 BAL81435.1基因的表达纯化
将含有BAL81435.1基因序列的pET28a-lipase-BAL质粒,转入Rosetta感受态细胞中并利用IPTG诱导其蛋白表达。在37 ℃诱导表达下,重组蛋白绝大部分以包涵体形式存在(结果未展示),但降低宿主表达温度,在25 ℃诱导下,重组蛋白能以可溶形式存在,结果如图3所示。通过菌体破碎,低温离心,上清液组分经过Ni-NTA 柱纯化,获得脂肪酶。由SDS-PAGE结果可见,纯化后的蛋白在25~35 kDa有唯一条带(图3),符合预期大小。利用His单抗(上海生物工程有限公司)的Western Blot检测结果表明该重组蛋白带有His标签(结果未展示)。以上结果显示成功得到了纯化的重组蛋白。
2.3 BAL81435.1基因编码脂肪酶的特征
2.3.1 脂肪酶的催化活性
利用p-NP比色法检测pET28a-lipase-BAL表达并纯化的重组蛋白的酯酶活性。该重组蛋白催化p-NPP长链底物的活性较高,尤其是C12以上的底物,而对短链底物的催化活性较弱,最适底物为C16的p-NPP,结果如图4-a所示。结果表明该重组脂肪酶为“真正”脂肪酶,而非数据库中注释的酯酶。

1-pET28a-lipase-BAL经IPTG诱导(1 mmol/L IPTG,25 ℃ 诱导12~16 h)后的总蛋白;2-破碎离心后得到的沉淀;3-破碎 离心后得到的上清液可溶蛋白;4-Ni-NTA 纯化后的蛋白
图3 pET28a-lipase-BAL在Rosetta中的表达纯化
Fig.3 Expression and purification of pET28a-lipase-BAL in Rosetta
2.3.2 温度对酶活性的影响
不同温度下(4、18、25、37、42、50、60、70、75 ℃)检测重组蛋白脂肪酶的活性,结果如图4-b所示,结果表明该酶的最适温度为60 ℃。
该脂肪酶的耐温试验表明,该重组蛋白在60 ℃及以下时,可在90 min内保持稳定,但在70 ℃蛋白的稳定性较差,酶活性明显下降(图4-c)。进一步试验表明该脂肪酶在4 ℃保存5 d,酶活性影响较少(结果未展示)。
2.3.3 pH对酶活性的影响
在不同pH值下(6.8、8.0、8.4、8.8、9.2、9.6、10.4、10.6)测定酶的活性,结果表明该脂肪酶最适pH为8.8,且pH的变化对酶的活性影响较为明显(图4-d)。
2.3.4 金属离子对酶活性的影响
金属离子常常作为酶的辅基或激活剂,通过自身化合价的变化来传递电子,完成生物体内的氧化还原反应。常见的金属离子中,一价或者二价的金属离子对酶活性影响较大[22]。该研究挑选了几种较为常见的金属离子,检测它们对该重组脂肪酶活性的影响。通过在酶反应体系中加入终浓度为10 mmol/L的不同金属离子如Ni2+、Cu2+、Ca2+、Mg2+、K+、Na+,结果表明除Ni2+、Ca2+和Na+外,其他离子都能促进酶的反应(图4-e、表1),且Na+离子浓度超过0.2 mol/L时,其抑制作用趋于稳定(图4-e)。
2.3.5 不同有机溶剂对酶活性的影响
极性有机溶剂会通过改变脂肪酶分子的构象,促进酶与底物的结合,从而提高酶的催化性能[23]。为研究有机溶剂对重组脂肪酶活性的影响,该研究选择了几种极性不同的有机溶剂:甲醇、乙醇、丙酮、氯仿、乙氰、二甲基亚砜,分别加至酶反应体系的5%或10%后测定酶活性。结果表明重组脂肪酶对低浓度有机溶剂比较耐受,甚至5%二甲基亚砜浓度对重组脂肪酶活性还有10%作用的增强作用。但是高浓度(10%)的氯仿和乙氰则明显抑制重组脂肪酶的活性,使酶活性分别降低65%和45%(表2)。
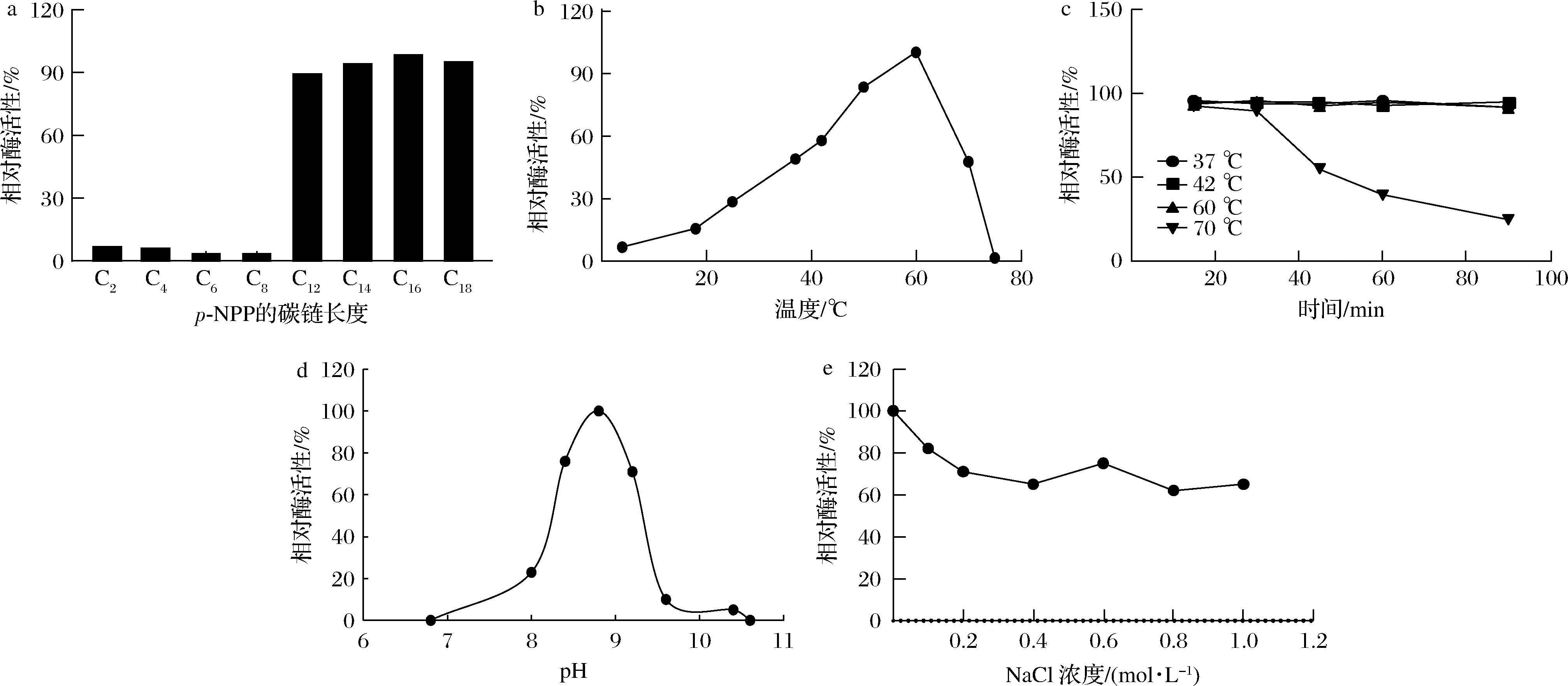
a-脂肪酶催化不同长度p-NPP底物(C2、C4、C6、C8、C12、C14、C16、C18)的活性;b-不同温度下(4、18、25、37、42、50、60、70、75 ℃)脂肪酶 的活性;c-脂肪酶对不同温度(37、42、60、70 ℃)的耐受试验;d-不同pH值下(6.8、8.0、8.4、8.8、9.2、9.6、10.4、10.6)脂肪酶的活性; e-脂肪酶对不同浓度Na+(0、0.1、0.2、0.4、0.6、0.8、1.0 mol/L)的耐受试验
图4 BAL81435.1基因编码脂肪酶的特征
Fig.4 Characteristics of lipase encoded by BAL81435.1 gene
表1 金属离子对酶活性的影响
Table 1 Effects of metal ions on enzyme activity

金属离子(10 mmol/L)相对酶活性/%对照100Ni2+54.62±0.019Cu2+118.36±0.142Ca2+80.47±0.154Mg2+133.68±0.092K+127.02±0.066
表2 有机溶剂对酶活性的影响 单位:%
Table 2 Effects of organic solvents on enzyme activity
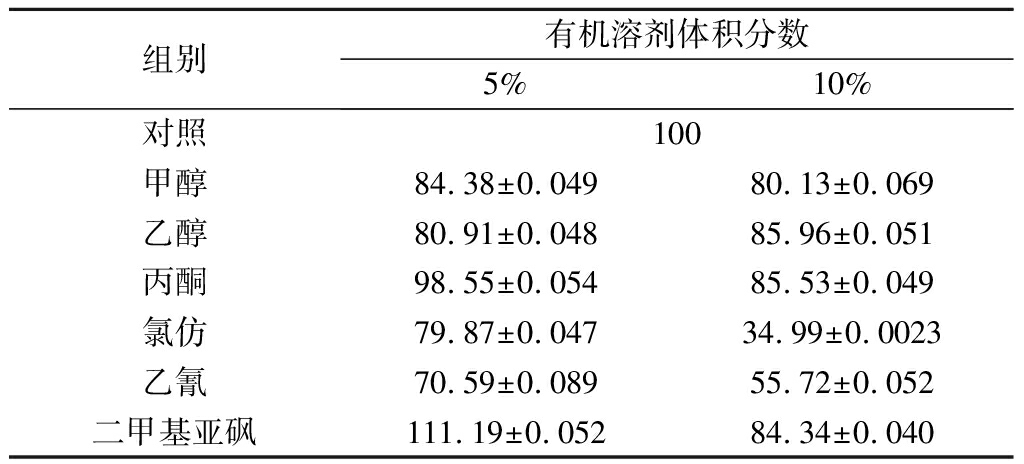
组别有机溶剂体积分数5%10%对照100甲醇84.38±0.04980.13±0.069乙醇80.91±0.04885.96±0.051丙酮98.55±0.05485.53±0.049氯仿79.87±0.04734.99±0.0023乙氰70.59±0.08955.72±0.052二甲基亚砜111.19±0.05284.34±0.040
2.3.6 表面活性剂对酶活性的影响
表面活性剂Triton X-100、Tween 80、Tween 20等都对重组脂肪酶的活性影响较大,上述试剂的1% 可使重组酶丧失绝大部分活性或完全失去活性(表3)。十二烷基硫酸钠(sodium dodecyl sulfate,SDS)、EDTA和Urea同样对酶活性影响较大,5 mmol/L的浓度可使酶活性降低近一半或丧失大部分活性(表3)。
表3 表面活性剂、变性剂对酶活性的影响
Table 3 Effects of surfactants and denaturing agents on enzyme activity
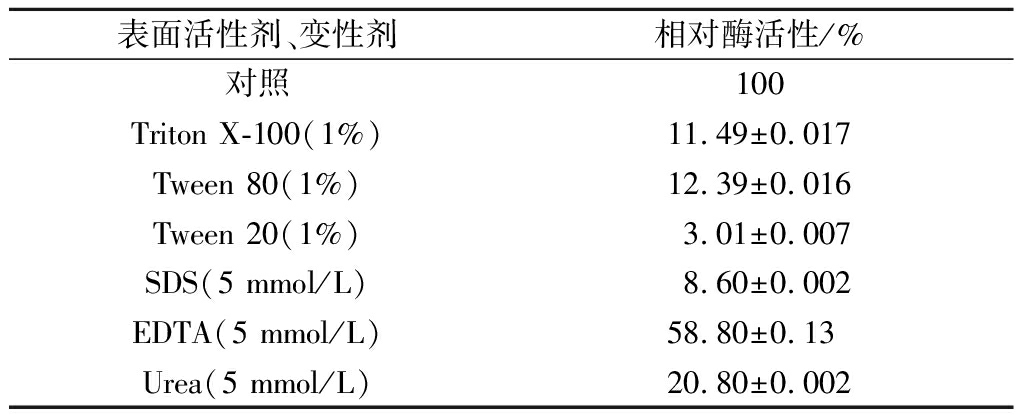
表面活性剂、变性剂相对酶活性/%对照100Triton X-100(1%)11.49±0.017Tween 80(1%)12.39±0.016Tween 20(1%)3.01±0.007SDS(5 mmol/L)8.60±0.002EDTA(5 mmol/L)58.80±0.13Urea(5 mmol/L)20.80±0.002
3 结论
Caldisericum exile AZM16c01是来源于日本热泉的嗜热性微生物,是Caldiserica新门的代表性菌株。该文以Caldisericum exile AZM16c01为研究对象,对其基因组信息进行筛选和分析,发现BAL81435.1预测为酯酶,但脂肪酶的进化树显示BAL81435.1不同于脂肪酶已有的6个进化分支,也不同于酯酶。对其重组表达产物进行功能研究,确定BAL81435.1的功能,试验结果表明BAL81435.1编码的嗜热脂肪酶,其底物为长碳链(C12以上)的p-NPP,目前最适底物为C16的长碳链,最适pH值为8.8,最适温度在60 ℃,在4~60 ℃具有较好的温度耐受性。该脂肪酶对大部分有机溶剂、金属离子、变性剂及高浓度的NaCl有较好的耐受性,但对表面活性剂敏感。
近年来微生物资源,尤其是生存环境比较特殊的极端微生物和海洋微生物,成为潜在的特性基因资源挖掘的主要对象。传统的培养方法、定向进化和功能驱动的宏基因组方法尽管可以有目的的筛选出特性基因资源,但受培养条件、筛选模式、操作繁琐等影响导致开发效率较低。该文基于已有序列的进化分析,结合异源表达等证实BAL81435.1为新嗜热脂肪酶,过程相对简洁,为快速发现特异的基因资源提供可选途径。
[1] DEMIRJIAN D C, MORIS-VARAS F, CASSIDY C S.Enzymes from extremophiles[J].Current Opinion in Chemical Biology, 2001, 5(2):144-151.
[2] ATALAH J, CACERES-MORENO P, ESPINA G, et al.Thermophiles and the applications of their enzymes as new biocatalysts[J].Bioresource Technology,2019, 280:478-488.
[3] DHEEMAN D S, FRIAS J M, HENEHAN G T.Influence of cultivation conditions on the production of a thermostable extracellular lipase from Amycolatopsis mediterranei DSM 43304[J].Journal of Industrial Microbiology & Biotechnology, 2010, 37(1):1-17.
[4] ZOTTIG X, MEDDEB-MOUELHI F, CHARBONNEAU D M, et al.Characterization of a novel alkalophilic lipase from Aneurinibacillusther moaerophilus:Lid heterogeneity and assignment to family I.5[J].The Protein Journal, 2017, 36(6):478-488.
[5] JO E, KIM J, LEE A, et al.Identification and characterization of a novel thermostable GDSL-Type lipase from Geobacillusther mocatenulatus[J].Journal of Microbiology and Biotechnology, 2021, 31(3):483-491.
[6] ABDEL-FATTAH Y R, GABALLA A A.Identification and over-expression of a thermostable lipase from Geobacillusther moleovorans Toshki in Escherichia coli[J].Microbiological Research,2008, 163(1):13-20.
[7] BANCERZ R, GINALSKA G.A novel thermostable lipase from basidiomycete Bjerkandera adusta R59:Characterisation and esterification studies[J].Journal of Industrial Microbiology & Biotechnology, 2007, 34(8):553-560.
[8] DE F TIMA SILVA LOPES M, et al.Characterization of a highly thermostable extracellular lipase from Lactobacillus plantarum[J].International Journal of Food Microbiology, 2002, 76(1/2):107-115.
TIMA SILVA LOPES M, et al.Characterization of a highly thermostable extracellular lipase from Lactobacillus plantarum[J].International Journal of Food Microbiology, 2002, 76(1/2):107-115.
[9] XIE W, KHOSASIH V, SUWANTO A, et al.Characterization of lipases from Staphylococcus aureus and Staphylococcus epidermidis isolated from human facial sebaceous skin[J].Journal of Microbiology and Biotechnology,2012, 22(1):84-91.
[10] H-KITTIKUN A, PRASERTSAN P, ZIMMERMANN W, et al.Sugar ester synthesis by thermostable lipase from Streptomyces thermocarboxydus ME168[J].Applied Biochemistry and Biotechnology,2012, 166(8):1 969-1 982.
[11] ROYTER M, SCHMIDT M, ELEND C, et al.Thermostable lipases from the extreme thermophilic anaerobic bacteria Thermoanaerobacter thermohydrosulfuricus SOL1 and Caldanaerobacter subterraneus subsp.tengcongensis[J].Extremophiles:Life under Extreme Conditions,2009, 13(5):769-783.
[12] SALAMEH M A, WIEGEL J.Purification and characterization of two highly thermophilic alkaline lipases from Thermosyntropha lipolytica[J].Applied and Environmental Microbiology,2007, 73(23):7 725-7 731.
[13] L PEZ-L
PEZ-L PEZ O, CERD
PEZ O, CERD N
N M E, GONZ
M E, GONZ LEZ SISO M I.New extremophilic lipases and esterases from metagenomics[J].Current Protein & Peptide Science,2014, 15(5):445-455.
LEZ SISO M I.New extremophilic lipases and esterases from metagenomics[J].Current Protein & Peptide Science,2014, 15(5):445-455.
[14] TIRAWONGSAROJ P, SRIPRANG R, HARNPICHARNCHAI P, et al.Novel thermophilic and thermostable lipolytic enzymes from a Thailand hot spring metagenomic library[J].Journal of Biotechnology,2008, 133(1):42-49.
[15] 周艳杰,耿鹏,时祎,等.嗜热玫瑰红球菌嗜热脂肪酶的重组表达与酶学性质研究[J].食品与发酵工业,2021,47(13):16-22.
ZHOU Y J, GENG P, SHI Y, et al.Expression and characterization of a novel thermostable lipase from Thermomicrobium roseum[J].Food and Fermentation Industries, 2021,47(13):16-22.
[16] 郭阳阳. 新型嗜热脂解酶的克隆表达及其酶学性质研究[D].无锡:江南大学, 2017.
GUO Y Y.Cloning, expression and characterization of novel thermophilic lipolytic enzymes[D].Wuxi:Jiangnan University, 2017.
[17] ABDELMONAEM M, HATEM B, IMEN G, et al.Classification of EC 3.1.1.3 bacterial true lipases using phylogenetic analysis[J].African Journal of Biotechnology,2010, 9(48):8 243-8 247.
[18] YADAVA K, DUBEYA K, YADAV S, et al.Amino acid sequences based phylogenetic and motif assessment of lipases from different organisms[J].Online Journal of Bioinformatics,2012, 13(3):400-417.
[19] MORI K, YAMAGUCHI K, SAKIYAMA Y, et al.Caldisericum exile gen.nov., sp.nov., an anaerobic, thermophilic, filamentous bacterium of a novel bacterial Phylum, Caldiserica phyl.nov., originally called the candidate phylum OP5, and description of Caldisericaceae fam.nov., Caldisericales ord.nov.and Caldisericia classis nov[J].International Journal of Systematic and Evolutionary Microbiology,2009, 59(11):2 894-2 898.
[20] TAYYAB M, RASHID N, AKHTAR M.Isolation and identification of lipase producing thermophilic Geobacillus sp.SBS-4S:Cloning and characterization of the lipase[J].Journal of Bioscience and Bioengineering, 2011, 111(3):272-278.
[21] KUMAR S, STECHER G, LI M, et al.MEGA X:Molecular evolutionary genetics analysis across computing platforms[J].Molecular Biology and Evolution, 2018, 35(6):1 547-1 549.
[22] 克曌地. 金属离子与Candidasp.99-125脂肪酶相互作用及对水解汉麻油的影响[D].郑州:郑州大学,2017.
KE Z D.Effect of metal ions on the enzymatic hydrolysis of hemp seed oilby lipase Candida sp.99-125[D].Zhengzhou:Zhengzhou University, 2017.
[23] 祖昕,杜凯,曹方,等.有机溶剂处理提高固定化脂肪酶活性及稳定性[J].食品与生物技术学报,2016,35(1):89-94.
ZU X, DU K, CAO F, et al.Treatment of organic solvents to improve activity and stability of immobilized lipases[J].Journal of Food Science and Biotechnology, 2016,35(1):89-94.