DNA甲基化是一种天然的表观遗传修饰方式,它是在DNA甲基转移酶(DNA methyltransferase,DNMTs)的作用下,以S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)为甲基供体,将其转移到基因组CpG二核苷酸胞嘧啶上,主要包括n6位腺嘌呤甲基化(6-methyladenine,6mA)、n4位胞嘧啶甲基化(4-methylcytosine,4mC)和c5位胞嘧啶甲基化(5-methylcytosine,5mC)3种类型[1]。DNMTs主要有DNMT3A和DNMT3B从头甲基转移酶和DNMT1维持性DNA甲基转移酶两类[2]。DNA甲基转移酶可以直接或间接影响小RNA在渗透胁迫下的稳定性[3]。大肠杆菌中编码腺嘌呤-N6甲基转移酶的yfiC基因可以增强细胞在渗透胁迫下的存活能力[4]。pglX是一种编码腺嘌呤特异性DNA甲基转移酶的基因,与pglY、pglZ、pglW构成了整个噬菌体生长限制系统[5],包含于新型噬菌体防疫系统(bacteriophage exclusion,BREX)中,并对BREX介导的噬菌体抗性至关重要[6]。
副干酪乳酪杆菌(Lacticaseibacillus paracasei)是一株兼性厌氧型革兰氏阳性菌株,常被作为发酵剂及辅助发酵剂用于乳制品的生产制作,尤其在干酪的生产中应用最多。副干酪乳杆菌4341作为辅助发酵剂提高了短熟Caciotta型奶酪的风味[7]。干酪的生产制作过程中菌株的生长会受到渗透胁迫的影响,在澳大利亚切达奶酪成熟期的不同阶段,副干酪乳酪杆菌GCRL163表现出良好的耐盐性[8]。TIAN等[9]通过高通量筛选技术筛选出的副干酪乳杆菌NCBIO01-M2突变体,能够通过调节不饱和脂肪酸比例和细胞内相容性溶质来调节渗透胁迫响应机制。
代谢组学是(metabonomics/metabolomics)系统生物学的重要组成部分,可以对生物体内所有代谢物进行定性和定量分析。其主要将不同层次的信息整合在一起,以确定生物体的生化反应[10-12],目前在医学和生命科学、食品和环境科学等领域应用广泛[13-14]。超高效液相色谱-四极杆飞行时间串联质谱联用技术(ultra high performance liquid chromatography-quadrupole time of flight tandem mass spectrometry,UHPLC-QTOF-MS)是目前用于代谢物鉴定和代谢组学研究较为强大的分析技术。该技术结合了UHPLC的高分离效率和QTOF-质谱的卓越结构识别能力,能够从复杂样品中检测数百甚至数千种成分[15]。
副干酪乳酪杆菌Zhang(Lacticaseibacillus paracasei Zhang)分离自酸马奶,是一株性状优良的益生菌[16-19]。在此前研究中发现副干酪乳酪杆菌Zhang中存在N6甲基腺嘌呤位点,与BREX系统的pglX基因高度相似,为了解pglX基因的生物学功能,构建了副干酪乳酪杆菌Zhang甲基化转移酶突变体(L.paracasei Zhang ΔpglX)[20-21]。本研究以副干酪乳酪杆菌Zhang甲基化转移酶突变体及其野生型为研究对象,在含有1 mol/L NaCl的培养基中进行10 h的渗透胁迫处理,从代谢组学水平上揭示DNA甲基化转移酶突变株与野生型菌株的渗透胁迫应答机制,以期获得抗逆性强的优良菌株,为干酪制品的商业发酵剂开发提供研究基础。
1 材料与方法
1.1 试验菌株
试验菌株L.paracasei Zhang和甲基化转移酶突变体L.paracasei Zhang ΔpglX,内蒙古农业大学乳品生物技术与工程教育部重点实验室。
1.2 试剂与仪器
甲酸、乙腈、氨水(LC-MS级),美国Sigma公司。亮氨酸脑啡肽(leucine-enkephalin)、ACQUITY UPLC-Xevo G2 QTOF MS超高效液相色谱-四极杆飞行时间质谱仪、MassLynx 4.1工作站、Progenesis QI软件,Waters公司;Milli-Q纯水仪,Millipore公司;电热恒温培养箱,上海一恒科技有限公司;高速控温离心机,Eppendorf公司。
1.3 试验方法
1.3.1 菌株活化
将-80 ℃冷冻保藏的L.paracasei Zhang和甲基化转移酶突变体L.paracasei Zhang ΔpglX按2%的接种量接种于5 mL已灭菌的MRS液体培养基中,于37 ℃恒温培养箱中培养24 h,即为活化菌株,需活化培养3代。
1.3.2 渗透胁迫
将活化好的L.paracasei Zhang和L.paracasei Zhang ΔpglX以2%的体积比接种于5 mL已灭菌MRS液体培养基中,于37 ℃恒温培养箱中培养到OD600值为1,按2%接种量接种于含1 mol/L NaCl溶液的培养基中,37 ℃培养10 h,收集菌液,4 ℃、4 000 r/min离心10 min获取发酵上清液。
1.3.3 UPLC-Q-TOF MS分析
1.3.3.1 样品前处理
将发酵液样品与乙腈按1∶1的比例混合均匀,4 ℃静置20 min,以沉淀蛋白质。之后振荡15 s,室温离心10 000 r/min、15 min,吸取上清液。将其经0.22 μL微孔滤膜过滤至上样瓶中,并将每个样品吸取20 μL混合成为质量控制样本(quality control,QC),以分析样本相同的检测方法,监测仪器在检测过程中的稳定性。
1.3.3.2 高效液相色谱条件
使用Waters HSS T3色谱柱(1.8 μm,2.1 mm×100 mm),柱温为35 ℃,进样量、流速分别设置为5 μL 和0.32 mL/min。正离子模式下,流动相A1为0.1%甲酸-水溶液,B1为0.1%甲酸-乙腈溶液,采用梯度洗脱,梯度洗脱条件如表1所示。负离子模式下,流动相A2为0.1%氢氧化铵-水溶液,B2为纯乙腈,梯度洗脱条件如表2所示。
表1 正离子模式下流动相梯度洗脱条件
Table 1 Gradient elution conditions of mobile phase in positive ion mode
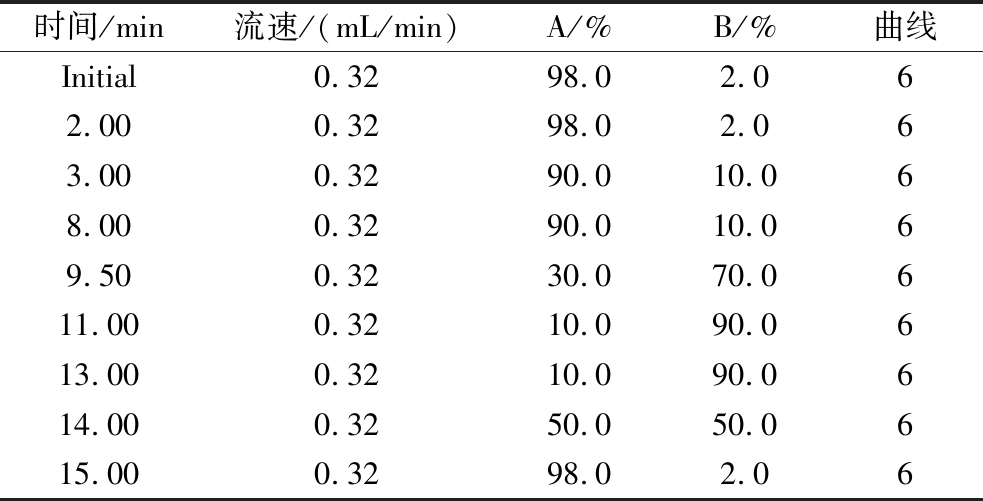
时间/min流速/(mL/min)A/%B/%曲线Initial0.3298.02.062.000.3298.02.063.000.3290.010.068.000.3290.010.069.500.3230.070.0611.000.3210.090.0613.000.3210.090.0614.000.3250.050.0615.000.3298.02.06
表2 负离子模式下流动相梯度洗脱条件
Table 2 Gradient elution conditions of mobile phase in negative ion mode

时间/min流速/(mL/min)A/%B/%曲线Initial0.3298.02.062.000.3298.02.063.000.3290.010.068.000.3230.070.069.500.3210.090.0611.000.3210.090.0613.000.3210.090.0614.000.3250.050.0615.000.3298.02.06
1.3.3.3 质谱条件
质谱采用ESI源正离子(ESI+)和负离子(ESI-)模式扫描,以氮气作为雾化气,氩气作为碰撞气体,质荷比扫描范围在50~1 200 m/z。为确保数据的准确性,在正负离子模式下采用200 ng/μL亮氨酸脑啡肽为校正液。毛细管电压为3 kV,离子源温度为120 ℃,脱溶剂气温度为500 ℃,脱溶剂气流速设置为800 L/h。
1.3.3.4 数据处理与统计学分析
将UPLC-QTOF MS采集的原始数据经Progenesis QI软件完成峰提取、峰对齐、去卷积化等处理,使用MetaboAnalyst 5.0(http://www.metaboanalyst.ca)进一步对数据进行标准化处理,之后将数据导入SIMCA 14.0软件完成多元统计分析,主要采用主成分分析(principal component analysis,PCA)和有监督的正交偏最小二乘判别分析(orthogonal partial least squares discriminant analysis,OPLS-DA)。最终结合P值≤0.05,变量投影重要性(variable importance in the projection,VIP)值≥1以及组间差异变化倍数(fold change,FC)≥2为差异代谢物筛选条件,并通过KEGG数据库进行代谢通路分析。
2 结果与分析
2.1 渗透胁迫下代谢物主成分分析
通过SIMCA 14.0软件对在渗透胁迫下L.paracasei Zhang和L.paracasei Zhang ΔpglX的代谢物进行PCA和OPLS-DA,结果如图1所示,左右两边分别为正负离子模式下的PCA图,可以看出QC样本能够很好的聚集在一起,并与突变组(M)和野生组(W)样本有明显的分离趋势,表明检测方法稳定且重复性良好。其中,第1主成分(PC1)将QC样本和两组(M组和W组)处理样本分离,并且在正负离子模式下样品总变化量分别为55.1%和58.2%;第2主成分(PC2)将QC与两组(M组和W组)处理样本分离,并且在正负离子模式下样品总变化量分别为13.7%和20.7%。
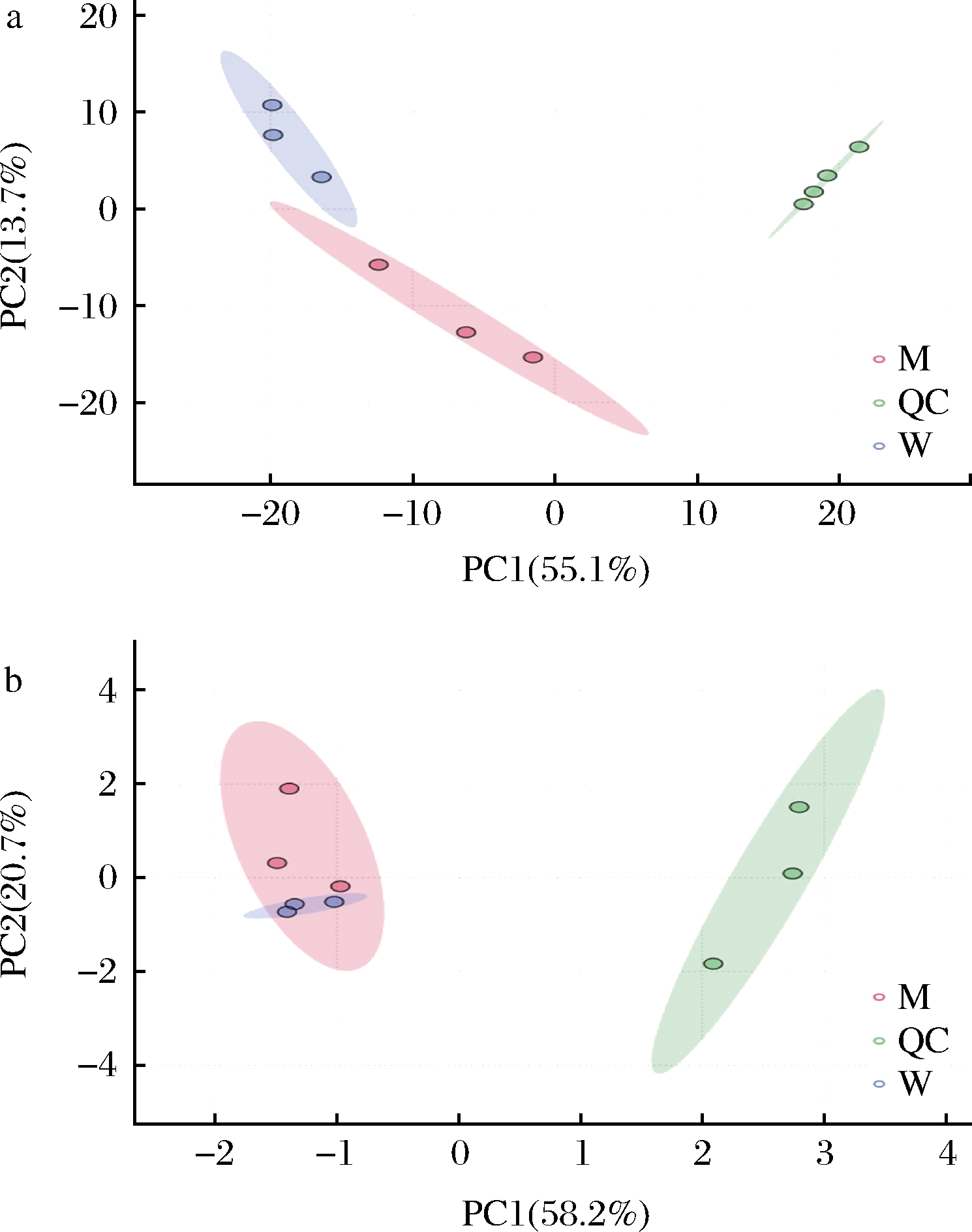
a-ESI+;b-ESI-
图1 渗透胁迫下L.paracasei Zhang和L.paracasei Zhang ΔpglX的PCA
Fig.1 PCA of L.paracasei Zhang and L.paracasei Zhang ΔpglX under osmotic stress
在OPLS-DA正负离子模式下(图2),突变组(M)与野生组(W)的3个平行样本聚集在一起,表明实验重复性较好,并且组间也有明显的区分,说明在渗透胁迫下两株菌株存在差异代谢物质。图中横坐标为主成分的得分值(Tp),可以看出组间的差异;纵坐标为正交成分得分值(TO),可以分辨出组内样本间的差异。进一步进行S-PLOT分析,结果如图3所示,图中离中心越远的点,表明其代谢物重要度越高,越能成为显著差异代谢物。因此可以确定两菌株在渗透胁迫条件下存在多个差异代谢物质。

a-ESI+;b-ESI-
图2 渗透胁迫下L.paracasei Zhang和L.paracasei Zhang ΔpglX的OPLS-DA
Fig.2 OPLS-DA of L.paracasei Zhang and L.paracasei Zhang ΔpglX under osmotic stress
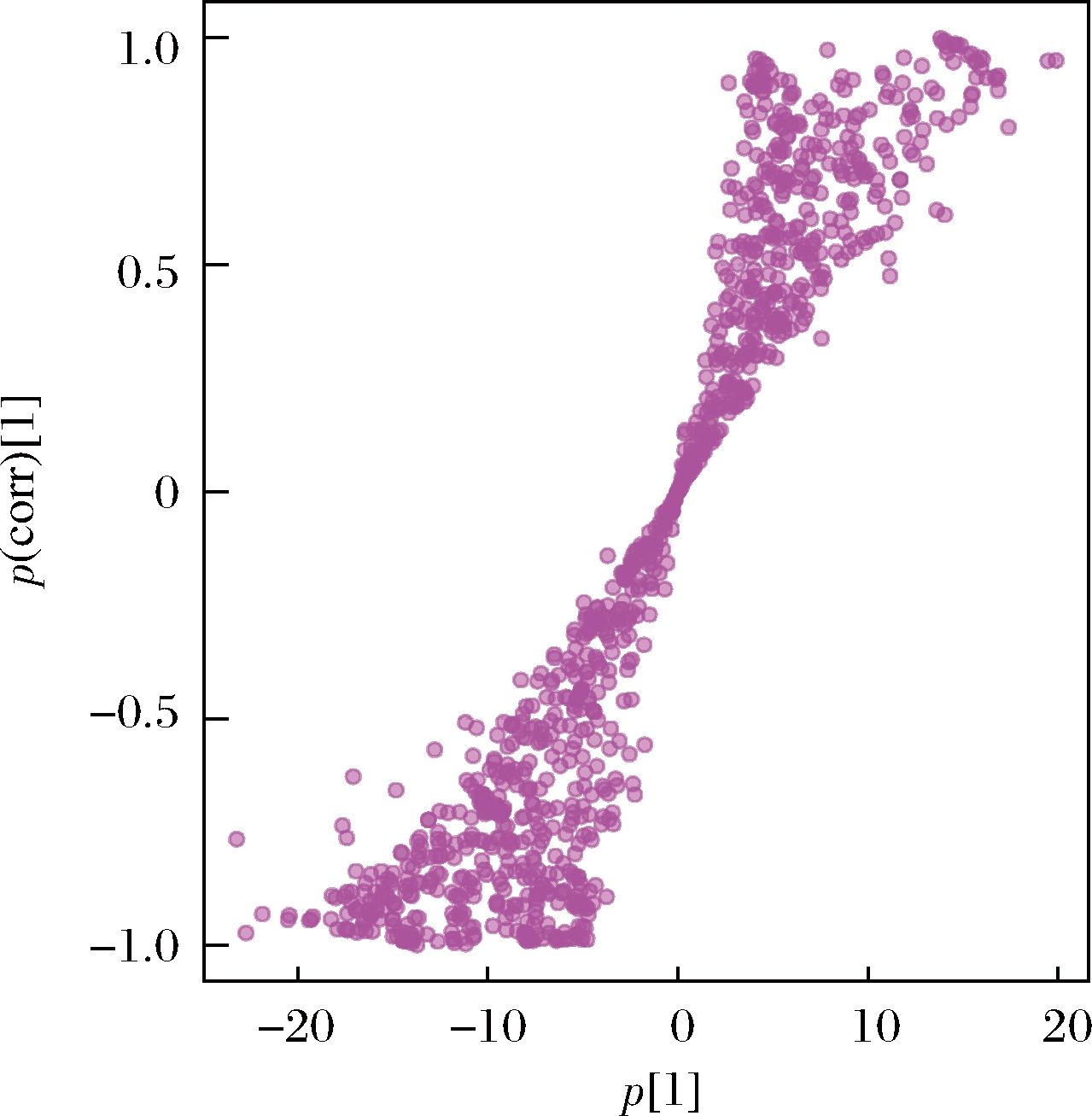
图3 渗透胁迫下L.paracasei Zhang和L.paracasei Zhang ΔpglX的S-PLOT
Fig.3 S-PLOT of L.paracasei Zhang and L.paracasei Zhang ΔpglX under osmotic stress
2.2 差异代谢物分析
根据OPLS-DA模型分析结果,结合T检验P值≤0.05、VIP值≥1和FC值≥2三个条件,筛选显著差异代谢物。结果如表3所示,突变组(M)与野生组(W)相比共筛选出10个显著差异代谢物,其中皮质醇、18-羟基皮质酮、四氢孕酮、脱氧肌苷、P1, P4-双(5′-腺苷)四磷酸盐等明显增加,甜菜碱醛、糖衣去氧胆酸呈显著下降趋势。
表3 渗透胁迫下L.paracasei Zhang和L.paracasei Zhang ΔpglX的差异代谢物
Table 3 Differential metabolites of L.paracasei Zhang and L.paracasei Zhang ΔpglX under osmotic stress
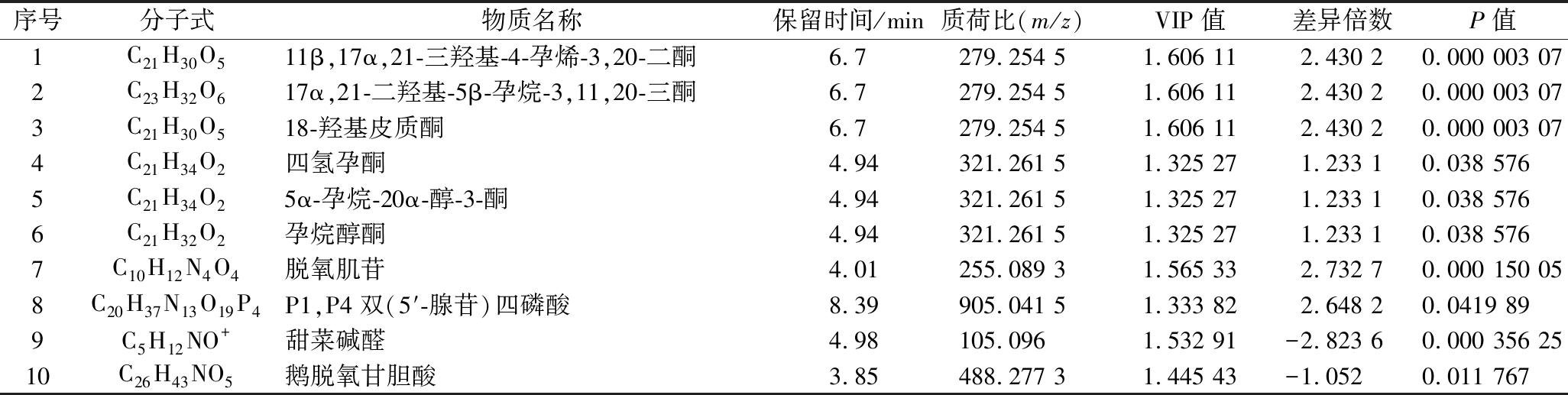
序号分子式物质名称保留时间/min质荷比(m/z)VIP值差异倍数P值1C21H30O511β,17α,21-三羟基-4-孕烯-3,20-二酮6.7279.254 51.606 112.430 20.000 003 072C23H32O617α,21-二羟基-5β-孕烷-3,11,20-三酮6.7279.254 51.606 112.430 20.000 003 073C21H30O518-羟基皮质酮6.7279.254 51.606 112.430 20.000 003 074C21H34O2四氢孕酮4.94321.261 51.325 271.233 10.038 5765C21H34O25α-孕烷-20α-醇-3-酮4.94321.261 51.325 271.233 10.038 5766C21H32O2孕烷醇酮4.94321.261 51.325 271.233 10.038 5767C10H12N4O4脱氧肌苷4.01255.089 31.565 332.732 70.000 150 058C20H37N13O19P4P1,P4双(5′-腺苷)四磷酸8.39905.041 51.333 822.648 20.0419 899C5H12NO+甜菜碱醛4.98105.0961.532 91-2.823 60.000 356 2510C26H43NO5鹅脱氧甘胆酸3.85488.277 31.445 43-1.0520.011 767
根据KEGG数据库对筛选的10个差异代谢物进行富集分析得到如图4所示的代谢通路富集分析图,主要富集到类固醇激素生物合成、嘌呤代谢、甘氨酸、丝氨酸和苏氨酸代谢以及初级胆汁酸生物合成等多条代谢通路。图5为差异代谢物富集通路图,图中能明显看出各差异代谢物的上调和下调趋势。

图4 渗透胁迫下L.paracasei Zhang和L.paracasei Zhang ΔpglX的差异代谢物富集分析
Fig.4 Differential metabolite enrichment analysis of L.paracasei Zhang and L.paracasei Zhang ΔpglX under osmotic stress
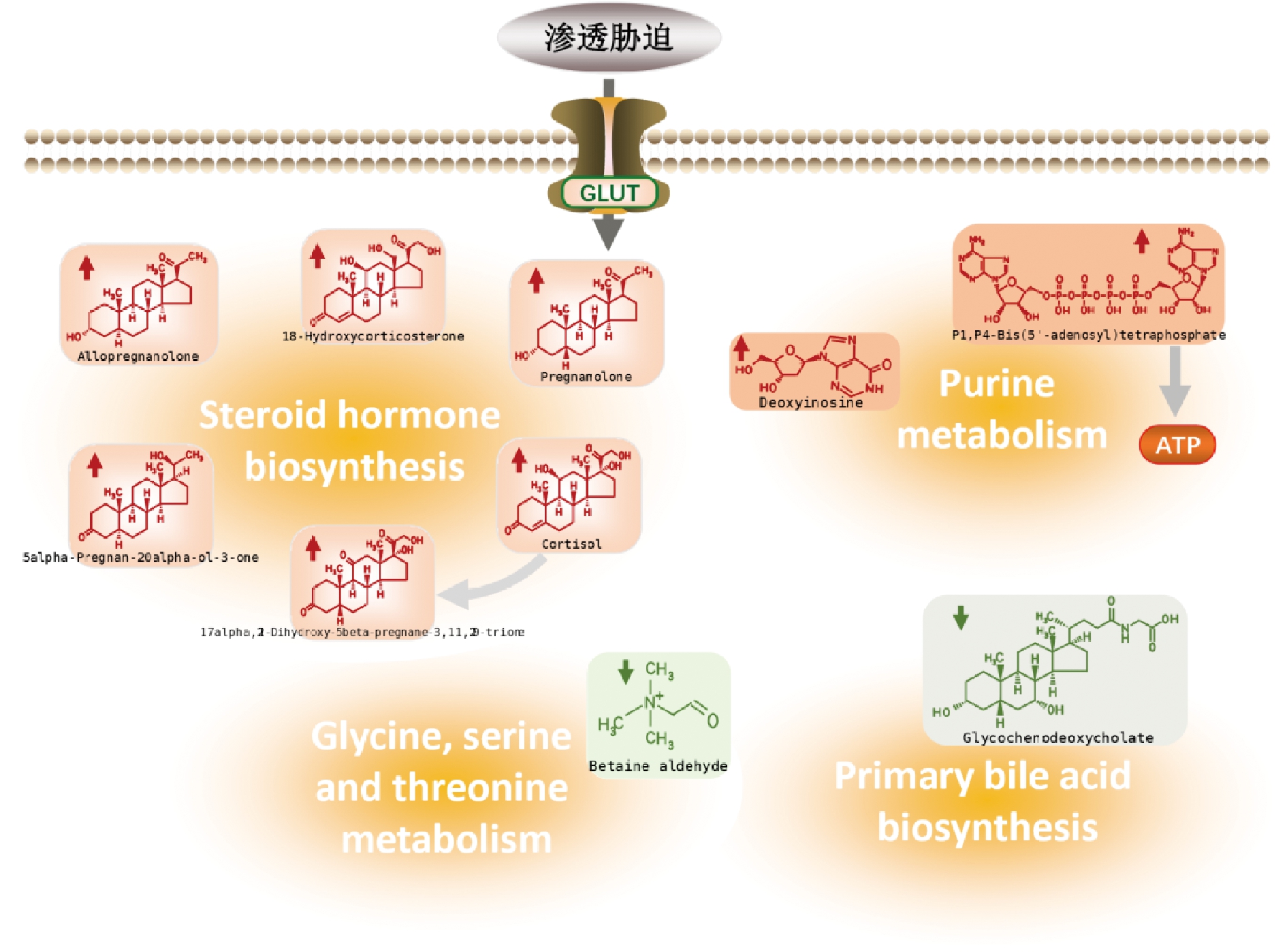
图5 渗透胁迫下L.paracasei Zhang和L.paracasei Zhang ΔpglX的差异代谢物富集通路图
Fig.5 Differential metabolite enrichment pathway of L.paracasei Zhang and L.paracasei Zhang ΔpglX under osmotic stress
2.2.1 类固醇类激素
在渗透胁迫下L.paracasei Zhang ΔpglX与L.paracasei Zhang相比,主要差异代谢物为皮质醇、18-羟基皮质酮以及四氢孕酮等类固醇激素类物质,参与了类固醇激素生物合成代谢途径。类固醇激素也称甾体激素,是一类特殊的萜类脂质,其结构包含4个环烷烃环的甾体核心。甾体可以控制细胞增殖和分化,能够通过质膜并与细胞内受体结合调节信号转导等途径,其中部分甾体在细胞与细胞的相互作用中充当信号分子[22-24]。甾体激素主要分为性激素和肾上腺皮质激素两大类,是由胆固醇衍生而来的,其具有脂溶性[25-26]。肾上腺皮质激素可以合成糖皮质激素,即皮质醇。在本研究中皮质醇存在明显上调趋势,其具有升高血糖的作用。主要机制包括拮抗胰岛素,减少葡萄糖利用,促进糖异生,增加骨骼肌蛋白和脂肪甘油三酯的分解等。同时该类物质还具有抗炎作用,但长期服用容易导致糖尿病、高血压等疾病[27]。因此,DNA甲基化转移酶突变体可以通过增加类固醇激素,提高类固醇激素生物合成来应对渗透胁迫。
2.2.2 核苷酸
DNA甲基化转移酶突变株L.paracasei Zhang ΔpglX与野生型相比,在渗透胁迫下嘌呤代谢能力增强,主要以脱氧肌苷和P1, P4-双(5′-腺苷)四磷酸盐为显著差异代谢物。脱氧肌苷也称为次黄嘌呤脱氧核苷,是由脱氧腺苷残基自发脱氨基产生的,正常的有氧呼吸产生的活性氧诱导也能生成脱氧肌苷[28-29]。在原核蛋白和真核蛋白中P1, P4-双(5′-腺苷)四磷酸盐(A[5′]P4[5′]A ammonium salt,Ap4A)与细胞分裂的控制有关[30-31]。有研究表明,将大肠杆菌(Escherichia coli)和鼠伤寒沙门氏菌(Salmonella typhimurium)暴露于氧化应激和热休克环境中,Ap4A和二(5′-核苷酸基)寡磷酸盐等相关核苷酸大量增加[32-33]。由此可以看出,与野生株相比甲基化转移酶突变体能够通过提高脱氧肌苷和Ap4A的产量,从而增强嘌呤代谢能力来应对渗透胁迫。
2.2.3 其他代谢物质
在渗透胁迫环境中,与野生株相比甲基化转移酶突变体L.paracasei Zhang ΔpglX中甘氨酸、丝氨酸和苏氨酸代谢以及初级胆汁酸生物合成显著富集,显著差异代谢物主要为甜菜碱醛和糖衣去氧胆酸。甜菜碱醛在甜菜碱醛脱氢酶(betaine aldehyde dehydrogenase,betB)或胆碱氧化酶(choline oxidase,codA)的作用下可以合成甘氨酸甜菜碱[34],甘氨酸甜菜碱的积累可以抵消细胞在高渗透压环境下水分的损失,并可以促进细胞生长,因此甘氨酸甜菜碱在多种原核生物和真核生物中被作为一种有效的渗透保护剂[35-36]。本研究中,甜菜碱醛的含量减少,表明甲基化转移酶突变株通过增加甘氨酸甜菜碱的含量来应对渗透应激反应。此外,糖衣去氧胆酸(glycochenodeoxycholic acid,GCDCA)在胆柳糖水解酶(biliosaccharide hydrolase,cbh)的作用下可以合成甘氨酸和鹅去氧胆酸盐(chenodeoxycholic acid,CDCA)。有研究表明,鹅去氧胆酸盐可以提高微胶囊的渗透稳定性[37],这就表明在本研究中,DNA甲基化转移酶突变体通过合成鹅去氧胆酸盐来提高渗透胁迫下菌株生长的稳定性。
3 结论与讨论
副干酪乳酪杆菌Zhang因其具有抗酸性以及抗胆盐性,使其在乳制品的成产制作过程中发挥重要作用。本研究采用UHPLC-QTOF-MS代谢组学分析技术,比较了DNA甲基化转移酶突变体L.paracasei Zhang ΔpglX与野生型L.paracasei Zhang在渗透胁迫下生长过程中的差异代谢物,利用主成分分析和正交偏最小二乘判别分析等多变量统计分析共筛选出10个显著差异代谢,主要为皮质醇、18-羟基皮质酮、四氢孕酮、脱氧肌苷、P1, P4-双(5′-腺苷)四磷酸盐、甜菜碱醛以及糖衣去氧胆酸等,主要涉及到类固醇激素生物合成、嘌呤代谢、甘氨酸、丝氨酸和苏氨酸代谢以及初级胆汁酸生物合成等多条代谢通路,说明DNA甲基化转移酶突变株能够通过提高类固醇激素类和核苷酸类以及甘氨酸甜菜碱来应对渗透应激反应。这一结果为揭示DNA甲基表型缺失后副干酪乳酪杆菌Zhang的渗透胁迫分子机制提供理论和方法支持。
[1] JELTSCH A P D.Beyond Watson and crick:DNA methylation and molecular enzymology of DNA methyltransferases[J].ChemBioChem, 2002, 3(4):274-293.
[2] MISHIMA Y, BRUECKNER L, TAKAHASHI S, et al.Enhanced processivity of Dnmt1 by monoubiquitinated histone H3[J].Genes to Cells, 2020, 25(1):22-32.
[3] ARYA D, KAPOOR S, KAPOOR M.Physcomitrella patens DNA methyltransferase 2 is required for recovery from salt and osmotic stress[J].FEBS Journal, 2016, 283(3):556-570.
[4] GOLOVINA A Y, SERGIEV P V, GOLOVIN A V, et al.The yfiC gene of E.coli encodes an adenine-N6 methyltransferase that specifically modifies A37 of ![]() 2009, 15(6):1134-1141.
2009, 15(6):1134-1141.
[5] SUMBY P, SMITH C M.Genetics of the phage growth limitation (Pgl) system of Streptomyces coelicolor A3(2)[J].Molecular Microbiology, 2002, 44(2):489-500.
[6] GOLDFARB T, SBERRO H, WEINSTOCK E, et al.BREX is a novel phage resistance system widespread in microbial genomes[J].The EMBO Journal, 2015, 34(2):169-183.
[7] BANCALARI E, MONTANARI C, LEVANTE A, et al.Lactobacillus paracasei 4341 as adjunct culture to enhance flavor in short ripened Caciotta-type cheese[J].Food Research International, 2020, 135:109284.
[8] SHAH S S, AL-NASERI A, ROUCH D, et al.Properties of an acid-tolerant, persistent Cheddar cheese isolate, Lacticaseibacillus paracasei GCRL163[J].Journal of Industrial Microbiology &Biotechnology, 2021, 48(9-10):kuab070.
[9] TIAN X W, WANG Y H, CHU J, et al.Exploring cellular fatty acid composition and intracellular metabolites of osmotic-tolerant mutant Lactobacillus paracasei NCBIO-M2 for highly efficient lactic acid production with high initial glucose concentration[J].Journal of Biotechnology, 2018, 286:27-35.
[10] ROBINETTE S L, HOLMES E, NICHOLSON J K, et al.Genetic determinants of metabolism in health and disease:From biochemical genetics to genome-wide associations[J].Genome Medicine, 2012, 4(4):1-10.
[11] TANG J, TAN C Y, ORESIC M, et al.Integrating post-genomic approaches as a strategy to advance our understanding of health and disease[J].Genome Medicine, 2009, 1(3):1-6.
[12] GRIFFIN J L, DES ROSIERS C.Applications of metabolomics and proteomics to the mdx mouse model of Duchenne muscular dystrophy:lessons from downstream of the transcriptome[J].Genome Medicine, 2009, 1(3):1-11.
[13] METZ T O.Metabolic Profiling:Methods and Protocols Methods in Molecular Biology[M].Heidelberg:Humana Press, 2011.
[14] VAIDYABNATHAN S, HARRIGAN G G, GOODACRE R.Metabolome Analyses:Strate-gies for Systems Biology[M].New York:Springer, 2005.
[15] KHAN H, ALI J.UHPLC/Q-TOF-MS technique:introduction and applications[J].Letters in Organic Chemistry, 2015, 12(6):371-378.
[16] YA T, ZHANG Q J, CHU F, et al.Immunological evaluation of Lactobacillus casei Zhang:A newly isolated strain from koumiss in Inner Mongolia, China[J].BMC Immunology, 2008, 9:68.
[17] WU R N, WANG L P, WANG J C, et al.Isolation and preliminary probiotic selection of lactobacilli from koumiss in Inner Mongolia[J].Journal of Basic Microbiology, 2009, 49(3):318-326.
[18] ZHANG Y, DU R T, WANG L F, et al.The antioxidative effects of probiotic Lactobacillus casei Zhang on the hyperlipidemic rats[J].European Food Research and Technology, 2010, 231:151-158.
[19] ZHU H, CAO C J, WU Z C, et al.The probiotic L.casei Zhang slows the progression of acute and chronic kidney disease[J].Cell Metabolism, 2021, 33(10):1926-1942.
[20] HUI W Y, ZHANG W Y, KWOK L Y, et al.A novel bacteriophage exclusion (BREX) system encoded by the pglX gene in Lactobacillus casei Zhang[J].Applied and Environmental Microbiology, 2019, 85(20):e01001-e01019.
[21] 惠文彦. Lactobacillus casei Zhang BREX系统鉴定及DNA甲基化与菌株生物学特性的相关性研究[D].呼和浩特:内蒙古农业大学, 2019.
HUI W Y.Identification of BREX system in Lactobacillus casei Zhang and correlation analysis between DNA methylation and biological characteristics[D].Hohhot:Inner Mongolia Agricultural University, 2019.
[22] MOON J Y, CHOI M H, KIM J.Metabolic profiling of cholesterol and sex steroid hormones to monitor urological diseases[J].Endocrine-Related Cancer, 2016, 23(10):R455-R467.
[23] BAKER M E.Origin and diversification of steroids:Co-evolution of enzymes and nuclear receptors[J].Molecular and Cellular Endocrinology, 2011, 334(1-2):14-20.
[24] WATERS C M, BASSLER B L.Quorum sensing:Cell-to-cell communication in bacteria[J].Annual Review of Cell and Developmental Biology, 2005, 21:319-346.
[25] PIIRONEN V, LINDSAY D G, MIETTINEN T A, et al.Plant sterols:Biosynthesis, biological function and their importance to human nutrition[J].Journal of the Science of Food and Agriculture, 2000, 80(7):939-966.
[26] FERNANDES P, CABRAL J M S.Phytosterols:Applications and recovery methods[J].Bioresource Technology, 2007, 98(12):2335-2350.
[27] WITCHEL S F, PINTO B, BURGHARD A C, et al.Update on adrenarche[J].Current Opinion in Pediatrics, 2020, 32(4):574-581.
[28] KARRAN P, LINDAHL T.Hypoxanthine in deoxyribonucleic acid:Generation by heat-induced hydrolysis of adenine residues and release in free form by a deoxyribonucleic acid glycosylase from calf thymus[J].Biochemistry, 1980, 19(26):6005-6011.
[29] LINDAHL T.Instability and decay of the primary structure of DNA[J].Nature, 1993, 362(6422):709-715.
[30] LEE P C, BOCHNER B R, AMES B N.Diadenosine 5′,5′″-P1, P4-tetraphosphate and related adenylylated nucleotides in Salmonella typhimurium[J].The Journal of Biological Chemistry, 1983, 258(11):6827-6834.
[31] GRUMMT F, WALTL G, JANTZEN H M, et al.Diadenosine 5′,5′″-P1, P4-tetraphosphate, a ligand of the 57-kilodalton subunit of DNA polymerase alpha[J].Proceedings of the National Academy of Sciences of the United States of America, 1979, 76(12):6081-6085.
[32] LEE P C, BOCHNER B R, AMES B N.AppppA, heat-shock stress, and cell oxidation[J].Proceedings of the National Academy of Sciences of the United States of America, 1983, 80(24):7496-7500.
[33] BOCHNER B R, LEE P C, WILSON S W, et al.AppppA and related adenylylated nucleotides are synthesized as a consequence of oxidation stress[J].Cell, 1984, 37(1):225-232.
[34] BOCH J, KEMPF B, SCHMID R, et al.Synthesis of the osmoprotectant glycine betaine in Bacillus subtilis:Characterization of the gbsAB genes[J].Journal of Bacteriology, 1996, 178(17):5121-5129.
[35] CSONKA L N.Physiological and genetic responses of bacteria to osmotic stress[J].Microbiological Reviews, 1989, 53(1):121-147.
[36] KEMPF B, BREMER E.Uptake and synthesis of compatible solutes as microbial stress responses to high-osmolality environments[J].Archives of Microbiology, 1998, 170(5):319-330.
[37] MOORANIAN A, ZAMANI N, MIKOV M, et al.Novel nano-encapsulation of probucol in microgels:Scanning electron micrograph characterizations, buoyancy profiling, and antioxidant assay analyses[J].Artificial Cells, Nanomedicine, and Biotechnology, 2018, 46(sup3):S741-S747.