腐乳是一种源于中国的传统发酵食物,由豆腐经特定的微生物如放线菌、毛霉和根霉固态发酵制成[1],不仅具有独特的风味,而且含有多种营养成分和功能性成分[2]。腐乳中的风味物质与脂肪酸和蛋白质的降解密切相关,微生物产生的脂肪酶和蛋白酶将脂肪酸和蛋白质分解成肽、氨基酸和游离脂肪酸等小分子物质,大大提高了腐乳的营养[3]。根据腐乳发酵剂种类可以将腐乳分为细菌发酵(微球菌、芽孢杆菌和乳酸菌)腐乳、霉菌发酵(放线毛霉、毛霉和根霉)腐乳及其他发酵(自然接种)腐乳[4],其中霉菌发酵腐乳是最普遍的类型。在发酵过程中,发酵菌株和其他微生物可以分泌多种酶,在它们的共同作用下将大豆蛋白和碳水化合物水解成氨基酸和小分子糖[5],这些小分子物质是腐乳风味形成的关键。由于霉菌发酵可以产生生物活性物质、有机酸、营养物质、风味物质和色素,因此它在食品发酵工业中越来越重要。如MART NEZ等[6]发现固态发酵中根霉产生的小孢子生物催化剂能持续生产油酸乙酯;MEINI等[7]报道了黑曲霉和米曲霉固态发酵的葡萄渣提取物具有抗氧化和益生特性;此外,HE等[8]将腐乳中9种细菌和6种真菌鉴定为核心功能微生物群,显著影响风味化合物的产生,尤其是游离氨基酸的产生。然而,真菌除了产生一些有益的生物分子外,也会产生危害人类健康的真菌毒素。真菌毒素是由真菌产生的极其稳定的有毒天然代谢产物,在加工和热处理过程中不会分解。因此,食物和动物饲料易受到污染,给人类和动物带来广泛的不利影响[9]。毛霉菌病由毛霉目中普遍存在的丝状真菌引起,是一种死亡率较高的侵袭性真菌[10]。随着免疫抑制、糖尿病、钝性创伤和血液恶性肿瘤等风险因素在人群中越来越常见,感染毛霉菌病的人也逐渐增加[11]。有关于全球毛霉菌病的研究调查显示根霉、毛霉和横梗霉占报告病例的80%,但仍然有不少病例与不太常见的毛霉有关,如雅致放射毛霉[12]。放射毛霉是一种自然界中普遍存在的兼性厌氧真菌,以前未被认定为侵袭性毛霉菌病的病原体,直到2001年DAVEL等[13]报道了第一例由雅致放射毛霉引起的上颌窦炎的病例,人们才意识到雅致放射毛霉也具有致病性。虽然我们未在发酵腐乳中检测到有毒物质的产生,但从消费者的心理出发,作为中国商业腐乳最常用的霉菌之一,雅致放射毛霉的安全性及其产生生物活性物质的能力仍然值得我们深入研究。目前已有许多研究将高通量测序技术应用于腐乳发酵工业,以研究不同微生物的功能特性与其基因组成的关系。然而,这些研究主要集中在腐乳发酵过程中的微生物多样性[14-16]。尽管有相关文献报道了雅致放射毛霉的生物活性、致病性和改善发酵豆制品风味的能力,但仍然迫切需要完整的雅致放射毛霉的全基因组和基因功能注释相关信息。
NEZ等[6]发现固态发酵中根霉产生的小孢子生物催化剂能持续生产油酸乙酯;MEINI等[7]报道了黑曲霉和米曲霉固态发酵的葡萄渣提取物具有抗氧化和益生特性;此外,HE等[8]将腐乳中9种细菌和6种真菌鉴定为核心功能微生物群,显著影响风味化合物的产生,尤其是游离氨基酸的产生。然而,真菌除了产生一些有益的生物分子外,也会产生危害人类健康的真菌毒素。真菌毒素是由真菌产生的极其稳定的有毒天然代谢产物,在加工和热处理过程中不会分解。因此,食物和动物饲料易受到污染,给人类和动物带来广泛的不利影响[9]。毛霉菌病由毛霉目中普遍存在的丝状真菌引起,是一种死亡率较高的侵袭性真菌[10]。随着免疫抑制、糖尿病、钝性创伤和血液恶性肿瘤等风险因素在人群中越来越常见,感染毛霉菌病的人也逐渐增加[11]。有关于全球毛霉菌病的研究调查显示根霉、毛霉和横梗霉占报告病例的80%,但仍然有不少病例与不太常见的毛霉有关,如雅致放射毛霉[12]。放射毛霉是一种自然界中普遍存在的兼性厌氧真菌,以前未被认定为侵袭性毛霉菌病的病原体,直到2001年DAVEL等[13]报道了第一例由雅致放射毛霉引起的上颌窦炎的病例,人们才意识到雅致放射毛霉也具有致病性。虽然我们未在发酵腐乳中检测到有毒物质的产生,但从消费者的心理出发,作为中国商业腐乳最常用的霉菌之一,雅致放射毛霉的安全性及其产生生物活性物质的能力仍然值得我们深入研究。目前已有许多研究将高通量测序技术应用于腐乳发酵工业,以研究不同微生物的功能特性与其基因组成的关系。然而,这些研究主要集中在腐乳发酵过程中的微生物多样性[14-16]。尽管有相关文献报道了雅致放射毛霉的生物活性、致病性和改善发酵豆制品风味的能力,但仍然迫切需要完整的雅致放射毛霉的全基因组和基因功能注释相关信息。
本文揭示了雅致放射毛霉CJ-6的全基因组从头测序和基因功能注释信息,并将其基因注释信息与米曲霉RIB40和总状毛霉B9645进行了比较分析。米曲霉RIB40是在日本传统发酵工业中广泛使用的真菌菌株,由于米曲霉在食品发酵工业中广泛使用的历史悠久,美国食品药品管理局(Food And Drug Administration,FDA)将其工业应用认证为公认安全(generally recognized as safe,GRAS)[17]。日本学者于2005年发表了米曲霉的全基因组的草图[18],标志着米曲霉基因组学全面发展时代的到来。总状毛霉是纯菌种发酵腐乳中最常用的发酵微生物之一,但总状毛霉也会引起人类和动物疾病[19]。因此,将从NCBI获得的米曲霉RIB40和总状毛霉B8645全基因组数据与雅致放射毛霉CJ-6基因注释结果进行比较分析,使我们能够深入了解雅致放射毛霉CJ-6在腐乳发酵过程中各种活性物质及毒素的合成和代谢情况,为后续的深入研究奠定理论基础,以更好地开发其作用并彻底地利用在食品发酵工业中。
1 材料与方法
1.1 样品的采集
腐乳毛坯,重庆市蔡家的市集,用无菌镊子将腐乳毛坯装入50 mL无菌离心管中,放入冷藏箱。
1.2 毛霉的分离纯化
用接种环挑取样本的菌丝置于5 mL无菌超纯水中,摇匀后进行梯度稀释至10-6,在孟加拉红平板培养基上涂布,倒置于30 ℃恒温培养箱内培养,按照菌落形态和孢子颜色进行分离纯化,重复几次后得到纯化的菌株CJ-6(图1)。

图1 CJ-6菌落形态
Fig.1 Colony morphology of CJ-6
1.3 菌株分子生物学的鉴定
参考CHEN等[20]所述的方法,使用OMEGA Fungal DNA Kit试剂盒(北京索莱宝科技有限公司)提取基因组DNA。
使用真菌通用引物ITS1和ITS4进行PCR扩增。取1 μL模板DNA、1 μL引物ITSl(5′-TCCGTAGGTGAACCTGCGG-3′)、1 μL引物ITS4(5′-TCCTCCGCTTATTGA ATGC-3′)、12.5 μL 2×Taq PCR Master Mix和9.5 μL灭菌超纯水混合得到25 μL PCR体系。扩增产物使用1.4%琼脂糖进行凝胶电泳检测,然后送华大基因测序。测序结果通过NCBI数据库中的BLAST程序进行同源性比对分析。
1.4 基因组测序和组装
应用Illumina PE150测序平台及PacBio测序平台(北京诺禾致源科技股份有限公司)对未知物种基因组从头测序的测序技术对雅致放射毛霉进行基因组测序。对于Illumina测序,用Covaris超声波破碎仪将电泳检测合格的DNA样品打断成长度约350 bp的片段,DNA片段纯化后与末端修复混合物结合,将修复后的DNA与A-Tailing混合物结合。然后将Illumina接头连接到DNA的3′端,再进行纯化、PCR扩增等步骤,完成整个文库的制备。文库构建后,使用Agilent 2100生物分析仪检测插入片段的大小,并通过实时PCR系统对文库的浓度进行定量,以确保文库的质量。合格的文库用Illumina平台测序。Pacbio测序通过构建具有20 kbp插入片段大小的SMRT Bell文库,使用Qubit确定浓度、Agilent 2100确定插入片段的大小,然后在Pacbio平台上测序来进行。得到的三代测序数据先使用SMRT Link v5.0.1过滤低质量reads,将剩下的reads初步组装成无空缺序列(Gap)的重叠群(contigs)。为降低三代测序的错误率,首先对三代数据进行三轮纠错:利用minimap2对初步组装的基因组进行mapping,然后使用racon(v1.4.13)和比对结果对初步组装的基因组进行位点校正;其次利用二代数据对其进行两轮纠错:利用bwa(0.7.8-r455)对初步组装的基因组进行mapping,然后使用samtools(v1.3.1)和比对结果对初步组装的基因组进行位点校正,最后得到纠错后的高精度Contigs。
1.5 基因组预测与功能注释
基因组预测和编码区分析由AUGUSTUS和GeneMark完成。使用基因组和RNA-Seq数据进行基因组的结构注释,分别使用RepeatMasker(Version open-4.0.5)和Tandem Repeats Finder(TRF, Version 4.07b)预测散在重复序列及串联重复序列。
预测的基因蛋白序列通过Diamond与每个功能数据库中已知注释基因的同源性注释进行比对。首先在NR(Non-Redundant Protein Database)、Pfam和SwissProtS数据库(BLASTP, e值≤1e-5)中比较预测的基因,然后基于KOG(EuKaryotic Orthologous Groups)数据库、GO(Gene Ontology)数据库和KEGG(Kyoto Encyclopedia of Genes and Genomes)进行分类。
1.6 CAZymes、Cytochrome P450和分泌蛋白
使用Diamond对基因组进行碳水化合物活性酶(carbohydrate-active enZYmes,CAZy)注释。使用BLAST注释细胞色素P450(cytochromeP450,CYP450),CYP450是一类以血红素为辅基的超基因家族蛋白,参与机体的一系列生理生化反应[21]。SignalP和TMHMM用于预测蛋白质序列和综合筛选分泌蛋白。此外,为了鉴定雅致放射毛霉的毒力或致病性,使用Diamond软件注释PHI(Pathogen Host Interactions)数据库和DFVF(Database of Fungal Virulence Factors)数据库。
1.7 次级代谢产物基因簇
使用AntiSMASH-4.0.2来预测雅致放射毛霉CJ-6基因组的次级代谢基因簇以了解其代谢真菌毒素的能力。
2 结果与分析
2.1 菌株鉴定结果
经过Genebank中的18S rDNA同源序列比较分析,根据Blast结果显示该菌株与雅致放射毛霉(Actinomucor elegans UAMH 10931)同源性达到100%(附图1,https://doi.org/10.13995/j.cnki.11-1802/ts.034416),确定该菌株为雅致放射毛霉,将其命名为雅致放射毛霉CJ-6。

附图1 CJ-6 18S rDNA基因序列对比结果
Fig.S1 CJ-6 18S rDNA gene sequence comparison results
2.2 基因组测序和组装
应用从头测序技术对雅致放射毛霉CJ-6进行基因组测序,得到2 460 521 894 bp测序数据量,使用SMRT Link v5.1.0软件进行初步组装后将组装结果进行比对评估,组装总共得到27个contigs,总长度为36 533 736 bp,其中最大的contig长度为4 995 973 bp,整个基因组组装大小约为36.53 Mbp,总GC含量为36.36%,N50长度为3 043 051 bp。上述结果表明雅致放射毛霉CJ-6基因组的组装质量较好。
雅致放射毛霉的全基因组包含多个功能区,使用AUGUSTUS等多种基因预测软件对编码基因进行预测以识别其基因功能。从头基因预测结果显示雅致放射毛霉CJ-6基因组有8 035个编码基因。推定的编码基因占雅致放射毛霉CJ-6基因组的31.35%,主要分布在200~1 700 bp。应用RepeatMasker软件预测到6 933个散在重复序列,总长1 234 984 bp,占基因组全长的3.380 4%;其中长末端重复序列和DNA转座子分别占基因全长的2.783 2%和0.653 2%。在DNA序列中,TRF预测到的串联重复序列为7 725个,占基因全长的1.232 7%,其中包括3 856个小卫星DNA和1 187个微卫星DNA。最后,基因测序显示,非编码RNA(ncRNA)包含264个tRNA,长度为19 642 bp。
2.3 基因组功能注释和比较分析
雅致放射毛霉CJ-6基因功能分析包括功能注释、效应子和致病性分析。使用不同的数据库对雅致放射毛霉CJ-6进行功能注释,编码基因在各个数据库中都有注释到,在NR数据库中注释到的基因最多。总共有7 217个,其次是GO数据库(5 843个)、KEGG数据库(3 856个)、KOG数据库(2 697个)和CAZy数据库(174个)。
2.3.1 GO数据库注释分析
GO数据库是基因功能描述的分类系统。在这个数据库中共注释到5 843个基因,注释结果由细胞定位(cellular component)、分子功能(molecular function)和生物过程(biological process)组成(图2-a)。GO注释蛋白质分类显示,这些蛋白质主要富集在“生物过程”类别的“代谢过程(metabolic process)”和“细胞进程(cellular process)”,以及“分子功能(molecular function)”类别中的“催化活性(catalytic activity)”和“绑定(binding)”。结果表明雅致放射毛霉CJ-6拥有较为旺盛的初级和次级代谢过程,同时具有对外界环境或本身的酶催化活性。

a-GO数据库注释;b-KOG数据库注释
图2 雅致放射毛霉CJ-6基因组功能注释
Fig.2 Genomic functional annotation of A.elegans CJ-6
2.3.2 KOG数据库注释分析
KOG数据库是COG(Clusters of Orthologous Groups)数据库中真核生物的直系同源数据库,可以预测蛋白质序列的功能,包括25个功能类别。雅致放射毛霉CJ-6基因组在这个数据库中共注释到2 697个基因,可分为25个功能。最丰富的类别是“转化、核糖体结构和生物合成”、“翻译后修饰、蛋白质周转、伴侣蛋白”、“仅通用功能预测”和“信号转导机制”,平均注释到300个基因。这说明雅致放射毛霉可能存在较复杂的生理生化代谢机制。同时,涉及“碳水化合物运输和新陈代谢”、“氨基酸运输和代谢”及“脂质运输和代谢”的基因相对丰富。上述结果表明,雅致放射毛霉CJ-6可能具有较好的碳水化合物、氨基酸和脂质代谢能力,这可能有助于在腐乳发酵期间产生各种风味物质(图2-b)。
2.3.3 KEGG代谢途径注释的比较分析
通过KEGG数据库注释能够帮助我们进一步掌握雅致放射毛霉CJ-6具有的代谢途径。功能注释结果包括新陈代谢(metabolism)、遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)、细胞过程(cellular processes)、生物系统(organismal systems)、人类疾病(human diseases)。
在KEGG中共注释到3 856个基因,被分为40个功能类别。从主要代谢途径分析来看,大多数注释的基因属于碳水化合物代谢(carbohydrate metabolism)和氨基酸代谢(amino acid metabolism)类别,此外脂类代谢(lipid metabolism)类别也注释到较多基因(图3-a)。腐乳中风味物质的形成与氨基酸和油脂的降解密切相关,这也在一定程度上说明,雅致放射毛霉CJ-6在传统腐乳生产过程中对腐乳风味品质的形成具有重要作用。

a-KEGG功能注释;b-3株霉菌的氨基酸代谢KEGG通路基因数量;c-3株霉菌的风味前体氨基酸KEGG通路基因占比
图3 KEGG通路注释比较分析
Fig.3 Comparative analysis of KEGG functional annotation
注:图3-c:纵坐标为各风味前体氨基酸代谢通路中所注释到的基因数量占各菌株中风味前体氨基酸总基因数量的比例,横坐标为风味前体氨基酸类别。
基于雅致放射毛霉CJ-6的氨基酸代谢对KEGG途径进行了深入的比较分析。与总状毛霉B9645(全基因组大小:65.53 Mbp)和米曲霉RIB40(全基因组大小:37.91 Mbp)相比,雅致放射毛霉CJ-6(全基因组大小:36.53 Mbp)与氨基酸代谢相关的基因较少(图3-b)。雅致放射毛霉CJ-6的氨基酸代谢KEGG注释结果显示,与支链氨基酸中的“异亮氨酸、亮氨酸和缬氨酸降解(Val、Leu and Ile degradation)”相关的基因最多,共注释到42个相关基因,其次是含硫氨酸中的“半胱氨酸和蛋氨酸代谢(Cys and Met metabolism)”,注释到39个基因。从图3-c可以看出,雅致放射毛霉CJ-6和总状毛霉B9645基因组中氨基酸代谢相关的基因分布结构较为相似,支链氨基酸“Val、Leu and Ile degradation”和含硫氨酸“Cys and Met metabolism”所占比例均较高。而米曲霉RIB40注释到较多与芳香族氨基酸“酪氨酸代谢(Tyr metabolism)”相关的基因。
基于脂质代谢(lipid metabolism)的KEGG途径注释结果显示,与脂质代谢相关的KEGG通路共分为8个类别,分别为脂肪酸降解(fatty acid degradation)、甘油酯代谢(glycerolipid metabolism)、甘油磷脂代谢(glycerophospholipid metabolism)、脂肪酸生物合成(fatty acid biosynthesis)、α-亚麻酸代谢(alpha-linolenic acid metabolism)、不饱和脂肪酸的生物合成(biosynthesis of unsaturated fatty acids)、醚酯代谢(ether lipid metabolism)及花生四烯酸代谢(arachidonic acid metabolism),相关基因的通路ID(pathway ID)及功能描述详见附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.034416)。3株霉菌中注释到的与脂质代谢相关的基因数量位于前3的代谢途径均为甘油磷脂代谢(glycerophospholipid metabolism)、脂肪酸降解(fatty acid degradation)和甘油酯代谢(glycerolipid metabolism),其中甘油磷脂代谢途径注释到的基因数量最多,雅致放射毛霉CJ-6基因组中注释到43个甘油磷脂代谢基因,米曲霉RIB40和总状毛霉B9645分别注释到40和79个基因(图4-a)。此外,由图4-b可以看出雅致放射毛霉CJ-6和总状毛霉B9645基因组中与脂肪代谢相关的基因结构分布较为相似,这一点与上述氨基酸代谢相关的基因分布结果类似,这也在一定程度上说明,雅致放射毛霉和总状毛霉在氨基酸和脂质代谢方面可能具有相似性。
附表1 雅致放射毛霉CJ-6基因组脂质代谢KEGG通路及相关基因
Table S1 Fat metabolism and degradation pathways and related genes in the genome of A. elegans CJ-6

No.通路 ID描述基因 ID1map00071Fatty acid degradationA2187, A2224, A3297, A3459, A3492.A3587, A3885, A3915, A3997, A5270, A5465, A5531, A5574, A5889, A5939, A6024, A6422, A6702, A6743, A6763, A7070, A7131, A7381, A7652, A7803, A0133, A0174, A0292, A0735, A0801, A0918, A0985, A1398, A1419, A30012map00561Glycerolipid metabo-lismA2010, A2250, A2330, A3226, A3297, A3865, A3997, A4194, A4705, A5327, A5530, A5617, A5738, A5795, A5807, A6365, A6422, A6734, A6763, A7000, A7070, A7131, A7144, A7202, A7754, A7966, A0196, A0310, A0535, A0985, A0999, A1006, A1034, A1230, A14193map00564Glycerophospholipid metabolismA2010, A2214, A2358, A2411, A2633, A2672, A3313, A3322, A3331, A3473, A3661, A3865, A4078, A4080, A4218, A4289, A4705, A5327, A5555, A5617, A5961, A6281, A6575, A6789, A7000, A7153, A7283, A7354, A7691, A7719, A0053, A0322, A0535, A0596, A0971, A0999, A1006, A1247, A1512, A31344map00061Fatty acid biosynthesisA2224, A3885, A5465, A5889, A6538, A6702, A6743, A6781, A7377, A0133, A0391, A07355map00592alpha-Linolenic acid metabolismA2010, A2187, A3587, A5531, A5574, A7381, A7803, A0292, A0801, A30016map01040Biosynthesis of unsat-urated fatty acidsA2187, A3587, A4502, A4699, A4811, A5443, A5531, A5574, A6781, A7138, A7381, A7803, A0023, A0292, A0801, A1455, A30017map00565Ether lipid metabo-lismA2010, A3473, A4080, A4218, A4289, A4554, A6698, A7283, A09718map00590Arachidonic acid me-tabolismA2010, A2205, A2422, A3872, A4692, A5595
表1 三株霉菌基因组中效应子和致病性基因预测
Table 1 Prediction of effector and pathogenic genes in the genome of three fungi
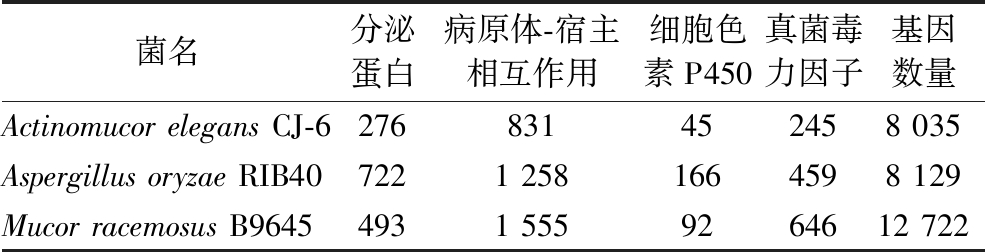
菌名分泌蛋白病原体-宿主相互作用细胞色素P450真菌毒力因子基因数量Actinomucor elegans CJ-6276831452458 035Aspergillus oryzae RIB407221 2581664598 129Mucor racemosus B96454931 5559264612 722

a-3株霉菌的脂质代谢KEGG通路基因数量;b-3株霉菌的脂质代谢KEGG通路基因占比
图4 脂质代谢KEGG通路注释比较分析
Fig.4 Comparative analysis of KEGG pathway annotation of lipid metabolism
注:图4-b:纵坐标为各脂质代谢通路中所注释到的基因数量占各菌株中脂质代谢通路总基因数量的比例,横坐标为脂质代谢类别。
2.3.4 CAZymes
CAZymes数据库是一个酶数据库资源,根据其功能将CAZymes分为糖苷水解酶(glycoside hydrolases, GHs)、糖基转移酶(glycosyl transferases, GTs)、多糖裂解酶(polysaccharide lyases, PLs)、糖酯酶(carbohydrate esterases, CEs)和氧化还原酶(auxiliary activities, AAs)五大类及一个碳水化合物结合结构域(carbohydrate binding domain,CBMs)[22]。
在雅致放射毛霉CJ-6基因组中共预测到159个潜在的碳水化合物活性酶及酶编码基因,分布在61个CAZy蛋白家族中,包含69个GHs、65个GTs、8个AAs、15个CEs和2个PLs。同时,还预测到了11个蛋白家族中24个推定的CBM酶编码基因(图5-a)。可以看出,3株霉菌均在GHs和GTs类别注释到的基因数量最多(图5-a),其中米曲霉RIB40的GHs基因占比达到54.26%。GH是一种水解糖苷键的酶,在生物体内糖和糖缀合物的水解和合成中起着不可替代的作用;GT负责磷酸激活的糖供体中糖苷键的生物合成。从图5-a还可以看出,雅致放射毛霉CJ-6和总状毛霉B9645基因组中碳水化合物活性酶的基因结构在整体上较为相似,这可能是雅致放射毛霉和总状毛霉成为腐乳发酵常用菌种的原因之一。

a-5种碳水化合物活性酶家族基因总体分布;b-碳水化合物活性酶亚家族基因分布
图5 三株霉菌中碳水化合物活性酶基因注释分析
Fig.5 Annotation analysis of CAZymes genes in three fungi
由图6可知,雅致放射毛霉注释到的碳水化合物活性酶基因总数最少,其中最高有12个基因属于GH18家族,包括几丁质酶(EC3.2.1.14)、溶菌酶(EC3.2.1.17)、内-β-N-乙酰氨基葡萄糖酶(EC3.2.1.96)、内β肽聚糖水解酶-N-乙酰氨基葡萄糖酶特异性(EC3.2.1.-)、Nod因子水解酶(EC3.2.1.-)等。在GT家族中,GT15和GT49家族均注释到8个基因,包含糖脂2-α-甘露糖基转移酶(EC2.4.1.131)、GDP-Man:α-1,2-甘露糖基转移酶(EC2.4.1.-)、β-1,3-N-乙酰氨基葡萄糖基转移酶(EC2.4.1.-)。米曲霉RIB40的CAZymes家族中GH3亚家族基因数量最高,而总状毛霉B9645则是GT2亚家族基因数量最多。3株霉菌具有不同的CAZymes家族基因,雅致放射毛霉CJ-6特有的CAZYmes家族基因包括AA2、AA5、CBM18、CBM19、CBM20、CBM43、CE2、CE6、GH5、GH8、GH13、PL14亚家族(图5-b)。上述结果说明,雅致放射毛霉、米曲霉和总状毛霉主要是依赖GH和GT家族基因来利用碳源,且3种霉菌利用碳源的能力存在一定差异,这可能与菌株的特性有关。

a-雅致放射毛霉CJ-6次级代谢基因簇及基因数量;b-3株霉菌次级代谢产物合成基因簇的比较分析
图6 真菌次级代谢基因簇分析
Fig.6 Analysis of secondary metabolic gene clusters in fungi
2.3.5 分泌蛋白和毒力相关蛋白
使用生物信息学分析方法,从雅致放射毛霉CJ-6的8 035个编码基因中预测到276个分泌蛋白。PHI数据库是一个病原体-宿主相互作用的基因数据库,涵盖许多致病基因。使用Blast序列相似性搜索功能将雅致放射毛霉CJ-6基因组中的全部序列和PHI数据库进行比对,全基因组的8 035个编码基因中注释到与病原体PHI表型突变类型相关的基因数目有831个(10.34%),在总状毛霉B9645和米曲霉RIB40中各预测到1 555(12.22%)和1 258个(15.48%)。细胞色素P450酶参与各种内源性或外源性化合物的代谢,并且通常参与丝状真菌的各种次级代谢产物的合成,与真菌的致病性也密切相关。在细胞色素P450酶数据库上进行Blast同源性搜索,注释到雅致放射毛霉CJ-6基因组中与细胞色素P450酶相关的45个基因。DFVF数据库是一个已知真菌毒力因子的综合数据库,收集了来自85个属的228种真菌携带的2 058个致病因子基因。在DFVF数据库中注释到411个雅致放射毛霉CJ-6的编码基因,其中245个编码基因的氨基酸序列能在DFVF数据库中得到具体的基因和其相对应的功能注释信息,而在米曲霉RIB40和总状毛霉B9645中注释到相对较多的致病因子基因,分别有459和646个(表1)。
2.3.6 次级代谢产物合成基因簇分析
雅致放射毛霉是食品工业应用中的重要微生物,针对雅致放射毛霉在发酵过程中可能存在的安全性隐患,主要分析了雅致放射毛霉真菌毒素相关的代谢产物合成情况。
在雅致放射毛霉基因组中预测到11个次级代谢产物合成基因簇(图6-a)。与米曲霉RIB40相比,雅致放射毛霉CJ-6基因组中仅预测到了4种类型的次级代谢基因簇,即Terpene(萜烯类)、非核糖体肽合成酶(non-ribosomal peptide synthetase, NRPS)、Siderophore(铁载体)及other,没有预测到聚酮合成酶(polyketide synthase, PKS)途径和PKS-NRPS混合代谢途径,这2种途径是大部分真菌毒素合成的关键代谢途径(图6-b)。雅致放射毛霉CJ-6基因组中预测到的次级代谢产物合成基因簇大多属于Terpene基因簇(cluster3、cluster4、cluster6、cluster7、cluster8、cluster9、cluster11),其中比对到41个基因,也有部分属于NRPS、Siderophore 和other类基因簇,分别为 cluster10、cluster1、cluster2 和 cluster5,在三类基因簇中比对到的基因数分别为10个、3个和19个(图6-b)。
3 讨论
3.1 雅致放射毛霉CJ-6的氨基酸和脂肪代谢
功能注释分析表明,雅致放射毛霉CJ-6具有复杂的代谢特性,尤其氨基酸和脂肪代谢途径在腐乳风味的形成过程中不可或缺。有研究表明,作为氨基酸代谢途径之一的转氨反应是微生物将氨基酸转化为风味物质的关键步骤[23],且支链氨基酸(leucine, isoleucine and valine)、含硫氨酸(cysteine and methionine)和芳香族氨基酸(phenylalanine, tyrosine tryptophan)是香气化合物的主要前体。基于氨基酸代谢的雅致放射毛霉CJ-6 KEGG代谢途径分析结果表明,与支链氨基酸中的“(Val、Leu and Ile degradation)”相关的基因最多,其次是含硫氨酸中的“(Cys and Met metabolism)”。支链氨基酸可降解生成3-甲基丁醛、2-甲基丁醛、2-甲基乙醛及相应的醇,含硫氨酸降解可产生挥发性物质如吲哚、氨、硫化氢等,对构成腐乳的特征风味有着重要作用。这可能是总状毛霉和雅致放射毛霉常用于腐乳发酵工业的原因。
脂类是腐乳中仅次于蛋白质的第二大类化合物,同时脂类的分解也在腐乳风味形成方面发挥着重要的作用。其中甘油三酯和磷脂能分解成游离脂肪酸、甘油二酯、甘油一酯及甘油,游离脂肪酸是甲基酮、直链醛、内酯、酯和S-硫酯的前体,这些风味芳香化合物是腐乳风味形成的关键物质[24]。基于脂肪代谢降解的KEGG途径分析结果表明,雅致放射毛霉CJ-6中有较多甘油磷脂和甘油酯代谢基因,这在一定程度上说明雅致放射毛霉CJ-6在将脂肪转化为风味化合物方面有较高潜力。
3.2 雅致放射毛霉CJ-6毒素合成能力分析
次级代谢产物合成基因簇分析表明,雅致放射毛霉CJ-6基本不具有合成真菌毒素的能力。目前的真菌毒素有400多种,有的毒素拥有同样或类似的合成途径和关键基因。合成途径主要包括PKS、NRPS、PKS-NRPS混合代谢、萜类化合物代谢、氨基酸相关代谢等,分析这些代谢途径及其关键基因,就有可能在基因水平上了解微生物是否具有合成真菌毒素的潜力。一些目前已知的真菌毒素是由萜烯类代谢产生的,如单端孢霉烯族毒素(trichothecenes)。在雅致放射毛霉基因组中注释到41个萜烯类基因簇的基因,在KEGG代谢途径中,与萜类化合物代谢相关的基因仅注释到3个,且分布在不同的代谢途径中,在萜类化合物代谢途径中并没有注释到相对完整的代谢途径基因,可认为不具有合成该物质的能力。我们在雅致放射毛霉CJ-6的基因组中没有比对到细菌、真菌中已报道的真菌毒素关键合成酶,说明在雅致放射毛霉中基本不存在PKS及PKS-NRPS混合代谢合成的相关基因及代谢途径,仅注释到少量NRPS相关基因。结果表明,雅致放射毛霉不具有合成这些真菌毒素的能力,这可能也解释了为什么在许多雅致放射毛霉发酵的腐乳中没有检测到真菌毒素。微生物基因组不仅包含与物种稳定遗传和进化多样性相关的编码基因这种功能区域,同时也有许多重要的非编码区也参与调控生物体的各个方面。基于基因序列的分析为我们探索微生物的多样性、生存环境的适应性及其复制、转录和翻译提供了详细的遗传信息[25]。
4 结论
本文利用从头测序技术对雅致放射毛霉CJ-6进行了全基因组测序、组装及基因功能注释,并结合其他2种已公开霉菌(米曲霉RIB40和总状毛霉B9645)的全基因组数据进行比较基因组学分析,以了解该菌株产生毒素的潜在能力,并验证其在腐乳生产工业中的安全性。我们从基因水平初步分析了雅致放射毛霉CJ-6的代谢途径和安全性,为进一步分析其次级代谢产物和调控其在腐乳发酵中的风味形成奠定了理论基础。然而,仅通过比较基因组学分析还不能彻底发掘雅致放射毛霉在腐乳发酵中的作用,需要进一步的分析如转录组学、基因敲除等研究方法来确定雅致放射毛霉的关键基因,从而明确其代谢机制,以便我们能将其更彻底地运用到腐乳发酵工业中。
[1] YANG J A, DING X W, QIN Y R, et al.Safety assessment of the biogenic amines in fermented soya beans and fermented bean curd[J].Journal of Agricultural and Food Chemistry, 2014, 62(31):7947-7954.
[2] MOY Y S, LU T J, CHOU C C.Volatile components of the enzyme-ripened sufu, a Chinese traditional fermented product of soy bean[J].Journal of Bioscience and Bioengineering, 2012, 113(2):196-201.
[3] MCSWEENEY P L H, SOUSA M J.Biochemical pathways for the production of flavour compounds in cheeses during ripening:A review[J].Le Lait, 2000, 80(3):293-324.
[4] HAN B Z, ROMBOUTS F M, ROBER NOUT M J.A Chinese fermented soybean food[J].International Journal of Food Microbiology, 2001, 65(1-2):1-10.
[5] FENG Z, CHEN H, LV X T, et al.Accelerated ripening of Kedong sufu with autochthonous starter cultures Kocuria rosea KDF3 and its protease KP3 as adjuncts[J].Journal of Applied Microbiology, 2014, 116(4):877-889.
[6] MART NEZ-RUIZ A, TOVAR-CASTRO L, GARC
NEZ-RUIZ A, TOVAR-CASTRO L, GARC A H S, et al.Continuous ethyl oleate synthesis by lipases produced by solid-state fermentation by Rhizopus microsporus[J].Bioresource Technology, 2018, 265:52-58.
A H S, et al.Continuous ethyl oleate synthesis by lipases produced by solid-state fermentation by Rhizopus microsporus[J].Bioresource Technology, 2018, 265:52-58.
[7] MEINI M R, CABEZUDO I, GALETTO C S, et al.Production of grape pomace extracts with enhanced antioxidant and prebiotic activities through solid-state fermentation by Aspergillus niger and Aspergillus oryzae[J].Food Bioscience, 2021, 42:101168.
[8] HE W M, CHUNG H Y.Exploring core functional microbiota related with flavor compounds involved in the fermentation of a natural fermented plain sufu (Chinese fermented soybean curd)[J].Food Microbiology, 2020, 90:103408.
[9] NATALIA A M, NATALIA C, IGNACIO L G, et al.High-resolution mass spectrometry for the determination of mycotoxins in biological samples.A review[J].Microchemical Journal, 2021, 166:106197.
[10] SERRIS A, DANION F, LANTERNIER F.Disease entities in mucormycosis[J].Journal of Fungi, 2019, 5(1):23.
[11] L PEZ-FERN
PEZ-FERN NDEZ L, SANCHIS M, NAVARRO-RODR
NDEZ L, SANCHIS M, NAVARRO-RODR GUEZ P, et al.Understanding Mucor circinelloides pathogenesis by comparative genomics and phenotypical studies[J].Virulence, 2018, 9(1):707-720.
GUEZ P, et al.Understanding Mucor circinelloides pathogenesis by comparative genomics and phenotypical studies[J].Virulence, 2018, 9(1):707-720.
[12] ALVAREZ E, SUTTON D A, CANO J, et al.Spectrum of zygomycete species identified in clinically significant specimens in the United States[J].Journal of Clinical Microbiology, 2009, 47(6):1650-1656.
[13] DAVEL G, FEATHERSTON P, FERN NDEZ A, et al.Maxillary sinusitis caused by Actinomucor elegans[J].Journal of Clinical Microbiology, 2001, 39(2):740-742.
NDEZ A, et al.Maxillary sinusitis caused by Actinomucor elegans[J].Journal of Clinical Microbiology, 2001, 39(2):740-742.
[14] 赵恒, 陈富安, 赵慧君, 等.茨河腐乳细菌与真菌多样性及其功能关联分析[J].食品与发酵工业, 2022, 48(10):212-218.
ZHAO H, CHEN F A, ZHAO H J, et al.Diversity and functional correlation analysis of bacteria and fungi in Huase sufu[J].Food and Fermentation Industries, 2022, 48(10):212-218.
[15] 陶康, 吴凌伟, 金晓芳, 等.基于高通量基因测序分析腐乳微生物多样性[J].食品科学, 2021, 42(8):143-149.
TAO K, WU L W, JIN X F, et al.Analysis of microbial diversity in sufu using high-throughput sequencing[J].Food Science, 2021, 42(8):143-149.
[16] 万红芳, 汪立平, 赵勇, 等.基于高通量测序分析2种夹江腐乳中的微生物[J].中国食品学报, 2020, 20(8):278-285.
WAN H F, WANG L P, ZHAO Y, et al.Analysis of microorganism in two kinds of Jiajiang fermented bean curd by high-throughput sequencing[J].Journal of Chinese Institute of Food Science and Technology, 2020, 20(8):278-285.
[17] MACHIDA M, YAMADA O, GOMI K.Genomics of Aspergillus oryzae:Learning from the history of koji mold and exploration of itsfuture[J].DNA Research, 2008, 15(4):173-183.
[18] MACHIDA M, ASAI K, SANO M, et al.Genome sequencing and analysis of Aspergillus oryzae[J].Nature, 2005, 438(7071):1157-1161.
[19] RIBES J A, VANOVER-SAMS C L, BAKER D J.Zygomycetes in human disease[J].Clinical Microbiology Reviews, 2000, 13(2):236-301.
[20] CHEN A J, TANG D, ZHOU Y Q, et al.Identification of ochratoxin a producing fungi associated with fresh and dry liquorice[J].PLoS One, 2013, 8(10):e78285.
[21] DENISOV I G, MAKRIS T M, SLIGAR S G, et al, Structure and chemistry of cytochrome P450[J].Chemical Reviews, 2005, 105(6):2253-2277.
[22] 王帅, 陈冠军, 张怀强, 等.碳水化合物活性酶数据库(CAZy)及其研究趋势[J].生物加工过程, 2014,12(1):102-108.
WANG S, CHEN G J, ZHANG H Q, et al.Carbohydrate-active enzyme (CAZy) database and its new prospect[J].Chinese Journal of Bioprocess Engineering, 2014,12(1):102-108.
[23] YVON M, RIJNEN L.Cheese flavour formation by amino acid catabolism[J].International Dairy Journal, 2001, 11(4-7):185-201.
[24] BERTUZZI A S, MCSWEENEY P L H, REA M C, et al.Detection of volatile compounds of cheese and their contribution to the flavor profile of surface-ripened cheese[J].Comprehensive Reviews in Food Science and Food Safety, 2018, 17(2):371-390.
[25] 谷璐婷, 李凌彦, 孔道春, 等.藤黄节杆菌ATCC21606的全基因组测序及序列分析[J].基因组学与应用生物学, 2020, 39(5):2071-2078.
GU L T, LI L Y, KONG D C, et al.Whole-genome sequencing and analysis of Arthrobacter luteus ATCC21606[J].Genomics and Applied Biology, 2020, 39(5):2071-2078.