酱香型白酒是我国独特的传统发酵饮料酒[1],其发酵微生物群落对风味特征与产品品质具有重要的作用[2-3]。研究酱香型白酒发酵过程微生物群落特征是解析酱香型白酒发酵机制的重要内容。前期对于其他香型白酒发酵过程的研究发现,在发酵过程中基于微生物群落系统发育多样性的微生物演替距离会影响代谢多样性[4],基于α多样性Shannon指数的微生物演替速率会显著影响风味物质的代谢[5],进而影响白酒风味的复杂性。因此,探究微生物群落多样性与风味物质代谢多样性的关系对酿造品质的提升具有重要作用。
白酒发酵过程理化因子可以驱动微生物群落演替。温度[6]、酸度[7]以及营养底物[8]是白酒发酵过程中影响微生物群落演替的主要驱动力。温度可以影响微生物演替速率[9],还原糖的种类差异会驱动白酒发酵过程微生物群落优势微生物组成[10],淀粉、酸度以及水分共同影响酱香型白酒堆积过程微生物群落组成以及微生物群落多样性[11]。但是,目前仍然不清楚影响酱香型白酒窖池发酵过程微生物群落多样性的主要因素,因此,有必要探究酱香型白酒发酵过程理化因子对微生物多样性的影响。另一方面,发酵过程中理化因子变化也受微生物群落的影响。研究发现,在白酒生产发酵起点添加强化菌剂,能通过增加微生物多样性,显著提升发酵过程的酸度[12];LIANG等[13]模拟葡萄酒发酵,发现初始微生物多样性能够显著影响发酵过程的乳酸含量。以上研究表明,初始微生物多样性也能够影响发酵过程理化因子的变化。然而,初始微生物多样性、发酵过程理化因子以及风味物质代谢多样性之间的相互作用规律并不清晰。
本研究以酱香型白酒2个不同车间同一生产轮次的发酵过程为研究对象,拟探究窖池发酵初始微生物多样性对发酵过程代谢多样性的作用规律。基于高通量扩增子测序技术和代谢物组学技术解析窖池发酵过程微生物群落和代谢多样性变化,并通过冗余分析判定窖池发酵过程微生物演替关键驱动力,使用结构方程模型解析窖池发酵初始微生物多样性、发酵过程理化因子和代谢多样性之间的相互作用规律。本研究将为酱香型白酒及其他发酵食品风味的定向调控提供理论指导。
1 材料与方法
1.1 实验材料
1.1.1 样品的采集
本研究针对酱香型白酒生产的3轮次酒发酵酒醅进行研究,取样时间为2022年3月,取样地点为四川省某酱香型白酒厂的2个不同产区的生产车间(L1和L4)。窖池发酵样本取发酵0、5、10、20、30、40 d的样本。每个时间点样本有3个生物平行,保存于-80 ℃冰箱用于后续检测分析。
1.1.2 主要试剂和仪器
L-薄荷醇,北京百灵威科技有限公司;其他试剂均为分析纯,国药(集团)上海化学试剂有限公司。789013-D5977B气相色谱-质谱联仪、Agilent 1260液相色谱仪,安捷伦公司;NANODROP 8000核酸浓度检测仪,赛默飞世尔科技有限公司;Mini-Bead-Beater-8细胞破碎仪,上海玉博生物科技有限公司;真空冷冻干燥仪,艾本德国际贸易有限公司;DS1921G纽扣式温度记录仪,上海沃第森电子科技有限公司。
1.2 实验方法
1.2.1 酒醅样品中风味物质的检测
于50 mL离心管中称取5.0 g酒醅,并加入25 mL超纯水充分振荡混匀,4 ℃过夜浸提。浸提后的样本于4 ℃超声波处理30 min,在4 ℃、8 000×g条件下离心5 min。取8 mL上清液和20 μL薄荷醇内标(100 μg/mL)加入至含有3.0 g NaCl的20 mL顶空瓶中。酒醅中的风味化合物的含量测定采用顶空固相微萃取技术(headspace solid phase microextraction,HS-SPME)并结合GC-MS,色谱柱为DB-FFAP(60 m×0.25 mm×0.25 μm;Agilent),检测条件参考文献[14]。
1.2.2 酒醅样品基因组DNA提取
称取7.0 g酒醅样品于50 mL无菌离心管中,后加入15 mL已灭菌且冷却后的0.1 mol/L磷酸盐缓冲液(phosphote buffer saline,PBS),并加入3颗已灭菌且冷却后的玻璃珠,300×g涡旋振荡5 min, 4 ℃离心5 min,收集上清液。重复使用PBS提取3次,收集全部的上清液。全部上清液于9 000×g,4 ℃离心3 min,弃上清液,收集细胞沉淀,并用5 mL PBS洗涤细胞沉淀3次,最后使用1 mL PBS重悬,参照文献[15]中的酚-氯仿法进行宏基因组的提取,真空冷冻干燥箱中干燥处理1 h后加入40 μL无菌ddH2O溶解DNA,用核酸浓度测定仪测定基因组的DNA浓度和A260/A280值(1.8~2.0),作为高通量测序及qPCR定量的模板。
1.2.3 高通量扩增子测序及原始序列处理
高通量扩增子测序:对于细菌,对其16S rRNA基因的V3~V4区域进行扩增,引物为338F和806R[16]。对于真菌,对其ITS2区域进行扩增,引物为ITS2和ITS3[17]。PCR产物的纯化参考文献[10]。纯化后的产物使用核酸浓度测定仪进行定量,后与同一样本中的PCR产物等分子质量混合,并根据Low Sample Protocol进行文库制备,最终在Illumina MiSeq测序平台上测序。
序列处理:通过QIIME[18]对MiSeq生成的原始序列进行处理。去除质量得分<20或长度<200 bp的序列,去除与PCR引物不完全匹配或具有未分配标签的序列[18]。使用UCHIME[19]去除嵌合体。通过DADA2将修整后的序列以100%的相似性聚类为扩增子序列变体(amplicon sequence variants,ASV)[20]。
1.2.4 酒醅理化因子检测
使用纽扣温度计对取样深度的样品进行实时温度测定。称取10.0 g样品,置于105 ℃烘箱中2 h至恒重,根据烘干前后物料的质量差计算样品的水分含量。采用滴定法检测样品中酸度和游离氨态氮含量[21]。取1.2.1节中上清液1 mL过0.22 μm有机滤膜进行过滤。通过高效液相色谱法和示差检测器(Schambeck SFD GmbH)测定滤液中的葡萄糖、乳酸、乙酸和乙醇含量,色谱柱为AminexHPX-87H(300 mm×7.8 mm,Bio-Rad)[22]。
1.2.5 风味物质代谢多样性计算
参考文献[23-24]的方法。代谢多样性是表征风味物质代谢的重要指标,各车间发酵过程不同风味物质含量归一化后的平均值即为代谢多样性。
1.2.6 统计学分析方法
主成分分析:为揭示不同车间窖池发酵过程中代谢物质变化存在差异,通过Origin 2013对发酵过程代谢物质进行主成分分析。
冗余分析:为探究窖池发酵过程理化因子对微生物结构的影响,使用R语言(version 3.2.4)中的Vegan包进行冗余分析,并同时进行蒙特卡洛检验。
结构方程模型:为探究窖池发酵初始微生物多样性对代谢多样性的影响,使用AMOS(version 20.2)进行分析,标准化的路径系数用于分析结构路径的影响关系。
2 结果与分析
2.1 不同产区生产车间窖池发酵过程代谢多样性的变化规律
酱香型白酒发酵过程风味物质的代谢对白酒风味特征以及产品品质具有重要的作用[25],本研究采用HS-SPME-GC-MS对不同车间发酵终点代谢产物进行分析,共检测到41种挥发性化合物,如图1-a所示,包括4种醛类、2种酮类、13种酯类、9种醇类、11种酸类和2种其他物质。通过主成分分析比较2个车间代谢物质的变化规律,结果如图1-b所示,发现2个车间代谢物组成从窖池发酵初始已具有差异;同时,2个车间窖池发酵样本中风味物质的组成以及变化规律均存在差异。通过归一化的方法计算车间窖池发酵过程的代谢产物多样性,如图1-c所示,窖池发酵起点车间L4的代谢多样性显著高于车间L1(P<0.01),且在整个发酵过程中,车间L4的代谢多样性均显著高于车间L1(P<0.05)。

a-不同车间窖池发酵过程代谢产物组成;b-不同车间窖池发酵过程代谢物变化规律;c-不同车间窖池发酵过程代谢多样性变化
图1 不同车间窖池发酵过程代谢多样性变化
Fig.1 The changes of metabolic diversity in liquor fermentation from different workshops
注:*P<0.05,**P<0.01,***P<0.001。
2.2 不同产区生产车间窖池发酵过程微生物群落结构
微生物群落结构会影响风味物质的代谢[9, 26]。为了探究窖池发酵过程代谢多样性差异的原因,对车间L1和L4的窖池发酵过程发酵微生物群落结构进行分析。通过Illumina MiSeq测序揭示窖池发酵过程中微生物群落的组成。在质量控制去掉低质量序列后,对于细菌,每个样品平均有51 935条高质量序列;对于真菌,每个样品平均有82 672条高质量序列。每个样品的覆盖率均在99%以上,表明测序深度足够和数据质量足够可靠。
将平均相对丰度>1%的属定义为优势微生物属,平均相对丰度>10%的属定义为绝对优势微生物属[27]。窖池发酵过程微生物群落结构的变化规律如图2所示。对于细菌群落,共获得2个优势微生物属,分别为Bacillus(平均相对丰度3.95%)和Kroppenstedtia(平均相对丰度3.49%),1个绝对优势微生物属,为Acetilactobacillus(平均相对丰度87.64%),其中,Bacillus丰度和Kroppenstedtia丰度在2个车间存在显著差异(P<0.05),在整个窖池发酵过程中,车间L4的细菌群落多样性均高于车间L1。对于真菌群落,共获得1个优势微生物属,为Byssochlamys(平均相对丰度4.35%),2个绝对优势微生物属,分别为Pichia(平均相对丰度61.31%)和Saccharomyces(平均相对丰度30.31%),其中,Pichia丰度和Saccharomyces丰度在2个车间存在显著差异(P<0.05),在整个窖池发酵过程中,车间L4的真菌群落多样性均高于车间L1。

a-细菌群落组成;b-细菌多样性;c-真菌群落组成;d-真菌多样性
图2 不同车间窖池发酵过程微生物结构和多样性变化
Fig.2 The changes of microbial structure and microbial diversity in liquor fermentation from different workshops
2.3 窖池发酵过程理化因子和微生物的关联分析
微生物群落演替易受到理化因子的影响[28],温度、水分、酸度、游离氨态氮、葡萄糖、乳酸、乙酸、乙醇是酱香型白酒发酵过程中的重要理化因子,分析结果如图3所示。窖池发酵温度呈现先上升后下降的趋势,2个车间的窖池发酵温度均在第5天达到最高值,且车间L1温度高于车间L4温度。整个窖池发酵过程车间L1水分含量高于车间L4水分含量,车间L1水分含量在44.94%~47.25%,车间L4水分含量在43.18%~45.12%。对于酸度、乳酸和乙酸,整个窖池发酵过程车间L1均高于车间L4,说明车间L1发酵酒醅中微生物群落的产酸能力要高于车间L4。

a-温度;b-水分;c-酸度;d-游离氨态氮;e-葡萄糖;f-乳酸;g-乙酸;h-乙醇
图3 不同车间窖池发酵过程理化因子动态变化
Fig.3 The dynamic changes of physical and chemical factors in liquor fermentation from two workshops
为了探究不同产区生产车间窖池发酵过程理化因子对微生物群落的影响,进行蒙特卡洛置换检验,结果如图4和表1所示。在车间L1窖池发酵过程中,理化因子共解释了微生物群落演替的53.74%,其中水分(r2=0.40,P=0.029)、酸度(r2=0.43,P=0.011)、葡萄糖(r2=0.44,P=0.026)、乙酸(r2=0.53,P=0.003)和乙醇(r2=0.37,P=0.047)与微生物群落演替显著相关。在车间L4窖池发酵过程中,理化因子共解释了微生物群落演替的77.06%,其中温度(r2=0.51,P=0.003)、水分(r2=0.42,P=0.019)、酸度(r2=0.63,P=0.002)、葡萄糖(r2=0.66,P=0.001)、乳酸(r2=0.55,P=0.002)和乙醇(r2=0.48,P=0.007)与微生物群落演替显著相关。因此,在窖池发酵过程中,酸度、葡萄糖、乙醇被确定为微生物演替的重要驱动力。其中,酸度[29]和糖类[8]可以影响微生物的多样性,乙醇可以影响发酵过程优势微生物Pichia和Lactobacillus丰度[9]。
表1 不同车间窖池发酵过程理化因子与微生物之间的蒙特卡洛置换检验结果
Table 1 Mantel test of physical and chemical factors and microbiota in liquor fermentation from two workshops

理化因子车间L1车间L4r2Pr2P温度0.160.280.510.003水分0.400.0290.420.019酸度0.430.0110.630.002游离氨态氮0.0640.60.0870.5葡萄糖0.440.0260.660.001乳酸0.290.0750.550.002乙酸0.530.0030.0860.49乙醇0.370.0470.480.007
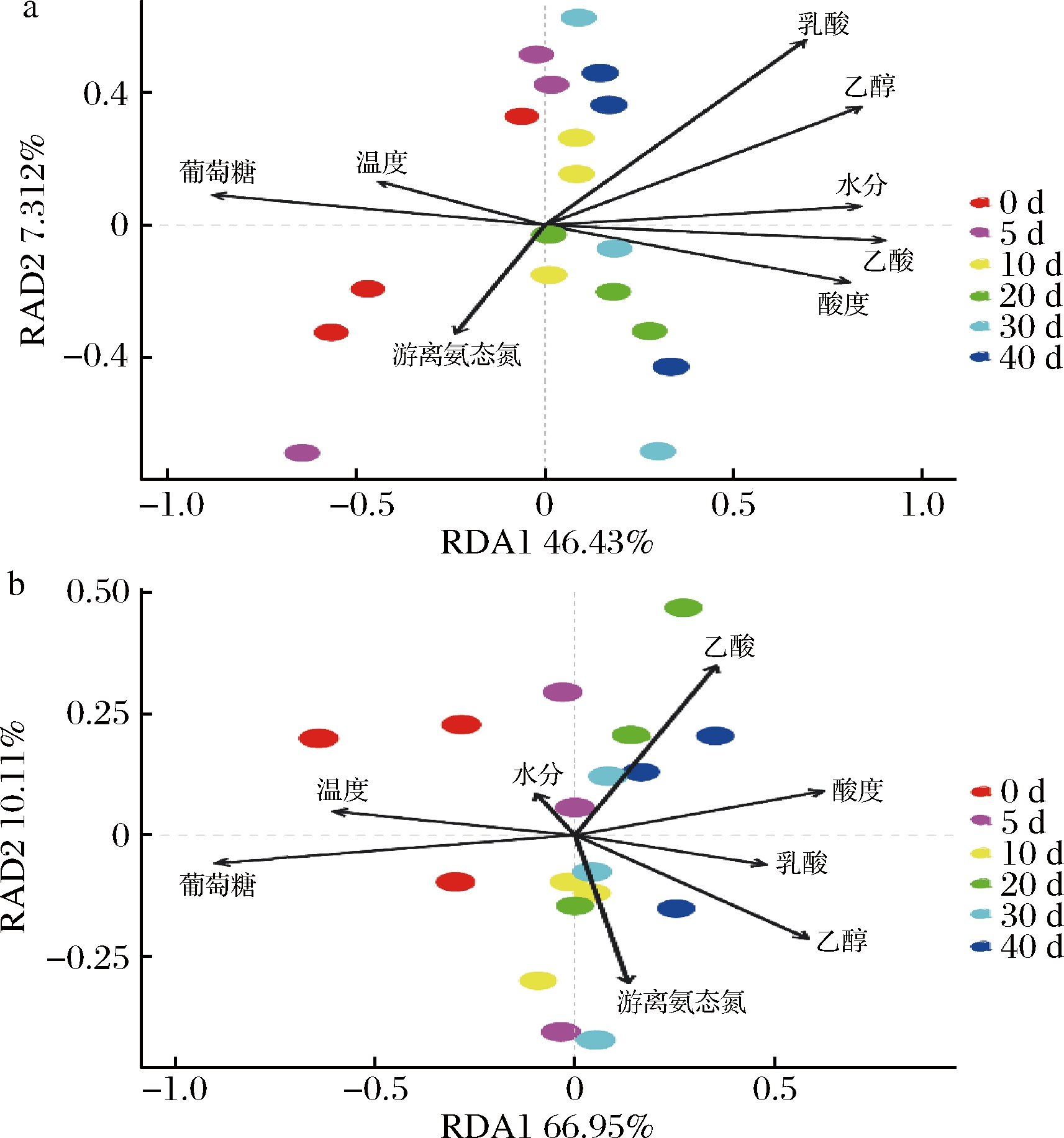
a-车间L1;b-车间L4
图4 不同车间窖池发酵过程理化因子与微生物冗余分析
Fig.4 Redundancy analysis of microbiota and physical and chemical factors in liquor fermentation from two workshops
2.4 窖池发酵初始微生物多样性对发酵过程代谢多样性的调控
为探究不同产区生产车间窖池发酵初始微生物多样性对发酵过程代谢多样性的共同影响,采用结构方程模型进行分析,该模型基于不同变量的连续回归,可以评估不同变量之间的复杂网络[30-31],结果如图5所示。窖池发酵初始微生物多样性显著影响窖池发酵过程理化因子(路径系数=-0.96,P<0.05),窖池发酵过程理化因子显著影响窖池发酵过程微生物多样性(路径系数=-0.99,P<0.001),窖池发酵过程微生物多样性显著影响窖池发酵过程代谢多样性(路径系数=1,P<0.001)。WANG等[6]和ZHANG等[9]研究了理化因子对微生物多样性的影响,HU等[32]和LIU等[33]研究了微生物多样性对风味代谢的影响,本研究初次探究了初始微生物多样性对理化因子的影响。
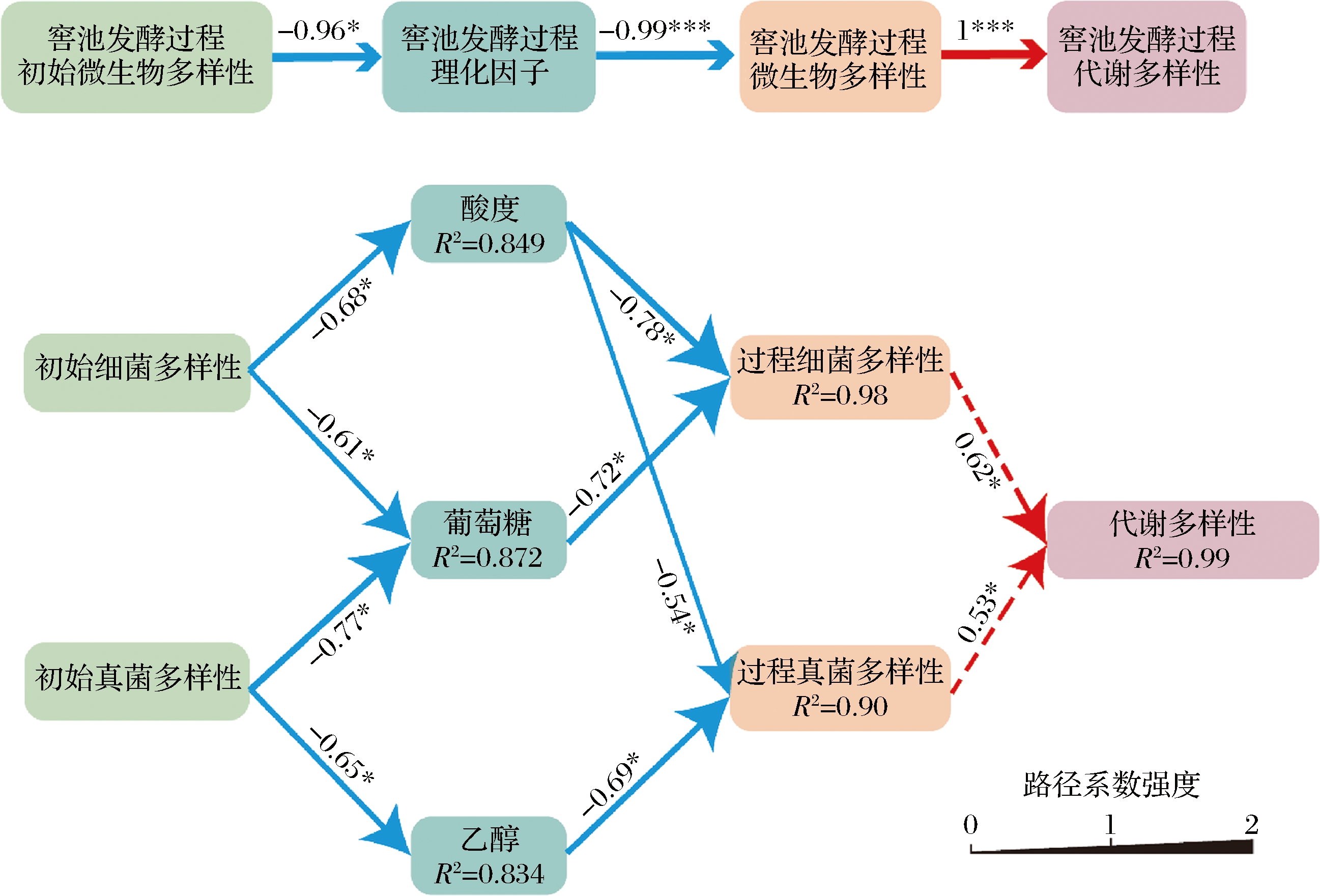
图5 发酵过程理化因子对代谢多样性的影响
Fig.5 Effects of physical and chemical factors in liquor fermentation on metabolic diversity
注:虚线和细实线箭头分别代表显著的正向和负向路径(*P<0.05,**P<0.01,***P<0.001),沿着箭头的数字表示标准化的路径系数,R2代表拟合优度
发酵过程酸度受到初始细菌多样性的影响(路径系数=-0.68,P<0.05),葡萄糖受到初始细菌多样性(路径系数=-0.61,P<0.05)和初始真菌多样性(路径系数=-0.77,P<0.05)的影响,乙醇受到初始真菌多样性的影响(路径系数=-0.65,P<0.05)。酸度在12.35~25.58 mmol/100 g显著影响发酵过程细菌多样性和真菌多样性,路径系数分别为-0.78和-0.54;葡萄糖在0.84~8.80 g/kg范围内显著影响发酵过程细菌多样性,路径系数为-0.72,乙醇在3.34~15.14 g/kg显著影响发酵过程真菌多样性,路径系数为-0.69;发酵过程细菌多样性和真菌多样性均显著影响代谢多样性,路径系数分别为0.62和0.53。之前研究中JI等[4]揭示了基于微生物多样性的演替距离会显著影响代谢多样性,TAN等[5]揭示了微生物多样性是影响微生物代谢的重要因素,WANG等[8]揭示了微生物多样性和代谢多样性呈线性正相关。说明窖池发酵过程初始细菌、真菌多样性可以通过影响窖池发酵过程理化因子,进而影响窖池发酵过程细菌、真菌多样性,最终调控发酵过程的代谢多样性。
3 结论
本研究对不同产区生产车间窖池发酵过程进行分析,揭示了初始微生物多样性对代谢多样性的调控作用。结果显示2个车间窖池发酵过程代谢多样性存在显著差异。并通过组学技术分析了窖池发酵过程微生物多样性的差异,通过冗余分析确定了发酵过程微生物群落的关键驱动力为酸度、葡萄糖和乙醇。最后,本研究使用结构方程模型揭示了初始真菌多样性对酸度和葡萄糖的显著影响,初始细菌真菌多样性对葡萄糖和乙醇的显著影响;酸度显著影响发酵过程细菌、真菌多样性,葡萄糖显著影响细菌多样性,乙醇显著影响真菌多样性;细菌多样性和真菌多样性显著影响代谢多样性。综上所述,窖池发酵初始微生物多样性可以调控代谢多样性,这为白酒及其他发酵食品的稳定生产提供了理论指导。
[1] JIN G Y, ZHU Y, XU Y.Mystery behind Chinese liquor fermentation[J].Trends in Food Science &Technology, 2017, 63:18-28.
[2] WU Q, KONG Y, XU Y.Flavor profile of Chinese liquor is altered by interactions of intrinsic and extrinsic microbes[J].Applied and Environmental Microbiology, 2015, 82(2):422-430.
[3] WANG P, WU Q, JIANG X J, et al.Bacillus licheniformis affects the microbial community and metabolic profile in the spontaneous fermentation of Daqu starter for Chinese liquor making[J].International Journal of Food Microbiology, 2017, 250:59-67.
[4] JI X A, YU X W, WU Q, et al.Initial fungal diversity impacts flavor compounds formation in the spontaneous fermentation of Chinese liquor[J].Food Research International, 2022, 155:110995.
[5] TAN Y W, ZHONG H P, ZHAO D, et al.Succession rate of microbial community causes flavor difference in strong-aroma Baijiu making process[J].International Journal of Food Microbiology, 2019, 311:108350.
[6] WANG S L, XIONG W, WANG Y Q, et al.Temperature-induced annual variation in microbial community changes and resulting metabolome shifts in a controlled fermentation system[J].mSystems, 2020, 5(4):e00555-20.
[7] WANG X S, DU H, ZHANG Y, et al.Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation[J].Applied and Environmental Microbiology, 2018, 84(4):e02369-17.
[8] WANG Z, JI X A, WANG S L, et al.Sugar profile regulates the microbial metabolic diversity in Chinese Baijiu fermentation[J].International Journal of Food Microbiology, 2021, 359:109426.
[9] ZHANG H X, WANG L, WANG H Y, et al.Effects of initial temperature on microbial community succession rate and volatile flavors during Baijiu fermentation process[J].Food Research International, 2021, 141:109887.
[10] 刘冲冲. 青稞原料对青稞酒发酵过程微生物菌群演替的影响[D].无锡:江南大学, 2019.
LIU C C.Effects of highland barley on the succession of microbial community during highland barley Baijiu fermentation[D].Wuxi:Jiangnan University, 2019.
[11] WANG H, HUANG Y G, HUANG Y L.Microbiome diversity and evolution in stacking fermentation during different rounds of Jiang-flavoured Baijiu brewing[J].LWT, 2021, 143:111119.
[12] ZHANG W Q, SI G R, DU H, et al.Directional design of a starter to assemble the initial microbial fermentation community of Baijiu [J].Food Research International, 2020, 134:109255.
[13] LIANG L H, MA Y W, JIANG Z Z, et al.Dynamic analysis of microbial communities and flavor properties in Merlot wines produced from inoculation and spontaneous fermentation[J].Food Research International, 2023, 164:112379.
[14] GAO W J, FAN W L, XU Y.Characterization of the key odorants in light aroma type Chinese liquor by gas chromatography-olfactometry, quantitative measurements, aroma recombination, and omission studies[J].Journal of Agricultural and Food Chemistry, 2014, 62(25):5796-5804.
[15] 李小龙. 芝麻香型白酒发酵过程微生物群落演替及其驱动因素[D].无锡:江南大学, 2018.
LI X L.Microbial community succession and its drivers during roasted sesame-flavor liquor fermentation[D].Wuxi:Jiangnan University, 2018.
[16] SOERGEL D A W, DEY N, KNIGHT R, et al.Selection of primers for optimal taxonomic classification of environmental 16S rRNA gene sequences[J].The ISME Journal, 2012, 6(7):1440-1444.
[17] TOJU H, TANABE A S, YAMAMOTO S, et al.High-coverage ITS primers for the DNA-based identification of ascomycetes and basidiomycetes in environmental samples[J].PLoS One, 2012, 7(7):e40863.
[18] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[19] EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics, 2011, 27(16):2194-2200.
[20] CALLAHAN B J, MCMURDIE P J, ROSEN M J, et al.DADA2:High-resolution sample inference from Illumina amplicon data[J].Nature Methods, 2016, 13(7):581-583.
[21] JIANG J, LIU Y C, LI H H, et al.Modeling and regulation of higher alcohol production through the combined effects of the C/N ratio and microbial interaction[J].Journal of Agricultural and Food Chemistry, 2019, 67(38):10694-10701.
[22] WU Q, CHEN L Q, XU Y.Yeast community associated with the solid state fermentation of traditional Chinese Maotai-flavor liquor[J].International Journal of Food Microbiology, 2013, 166(2):323-330.
[23] WAGG C, SCHLAEPPI K, BANERJEE S, et al.Fungal-bacterial diversity and microbiome complexity predict ecosystem functioning[J].Nature Communications, 2019, 10:4841.
[24] 徐炜, 井新, 马志远, 等.生态系统多功能性的测度方法[J].生物多样性, 2016, 24(1):72-84.
XU W, JING X, MA Z Y, et al.A review on the measurement of ecosystem multifunctionality[J].Biodiversity Science, 2016, 24(1):72-84.
[25] ZHANG H X, DU H, XU Y.Volatile organic compound-mediated antifungal activity of Pichia spp.and its effect on the metabolic profiles of fermentation communities[J].Applied and Environmental Microbiology, 2021, 87(9):02992-20.
[26] NICCUM B A, KASTMAN E K, KFOURY N, et al.Strain-level diversity impacts cheese rind microbiome assembly and function[J].mSystems, 2020, 5(3):e00149.
[27] WANG X S, DU H, XU Y.Source tracking of prokaryotic communities in fermented grain of Chinese strong-flavor liquor[J].International Journal of Food Microbiology, 2017, 244:27-35.
[28] BAN S B, CHEN L N, FU S X, et al.Modelling and predicting population of core fungi through processing parameters in spontaneous starter (Daqu) fermentation[J].International Journal of Food Microbiology, 2022, 363:109493.
[29] JI X A, YU X W, ZHANG L Y, et al.Acidity drives volatile metabolites in the spontaneous fermentation of sesame flavor-type Baijiu[J].International Journal of Food Microbiology, 2023, 389:110101.
[30] ARHONDITSIS G B, STOW C A, STEINBERG L J, et al.Exploring ecological patterns with structural equation modeling and Bayesian analysis[J].Ecological Modelling, 2006, 192(3-4):385-409.
[31] BANERJEE S, SCHLAEPPI K, VAN DER HEIJDEN M G A.Keystone taxa as drivers of microbiome structure and functioning[J].Nature Reviews Microbiology, 2018, 16(9):567-576.
[32] HU Y Y, ZHANG L, LIU Q, et al.The potential correlation between bacterial diversity and the characteristic volatile flavour of traditional dry sausages from Northeast China[J].Food Microbiology, 2020, 91:103505.
[33] LIU D, LEGRAS J L, ZHANG P Z, et al.Diversity and dynamics of fungi during spontaneous fermentations and association with unique aroma profiles in wine[J].International Journal of Food Microbiology, 2021, 338:108983.