鱼胶,又称花胶、鱼泡、鱼肚等,是由鱼鳔经过干制而成的制品。自古以来,鱼胶一直被视为名贵滋补品之一,有“海八珍”之誉,并与鱼翅、海参齐名[1-2]。鱼胶中含有大量的胶原蛋白,其总氨基酸含量超过70%,同时富含人体所需多种矿物元素和不饱和脂肪酸,具有止血补血、美容养颜等效用,兼具营养与药用价值[3],因此备受消费者喜爱。近年来,随着生活水平的提高,人们对营养和保健方面的需求日益增加。鱼胶作为一种优质滋补品,其供应量及种类受市场需求影响大幅增加。然而,不同品种鱼胶产品的品质和价格相差甚大,价格从每斤百元到千元不等。受利益驱动,一些不法商家通过定型处理等手段,以低质鱼胶冒充优质鱼胶,扰乱市场秩序,给消费者带来财产损失。一直以来,鱼胶鉴别主要依赖于形状、色泽、大小等感官特征,对鉴别人员的经验要求较高,常存在误判情况。因此,迫切需要建立一种权威、准确且易于操作的鱼胶品种鉴别方法来解决这一问题。
DNA条形码技术利用生物间共有、种间差异明显的一段DNA序列进行比较分析,实现物种鉴别,其核心是使用通用引物扩增目的基因片段,通过测序并比对数据库,最终确定物种信息[4-5]。DNA条形码作为一种操作过程快速简便、鉴定结果准确可靠的技术,最早由HEBERT等[6]提出,在水产品鉴别和进化分析中被广泛应用[7]。SHARRAD等[8]采用DNA条形码实现了对澳大利亚各地鲨鱼产品的物种鉴别,为鲨鱼产品的市场监管和濒危物种的保护提供了技术支持。PARDO等[9]利用DNA条形码成功对23个欧洲国家的283个海鲜样本进行鉴定,结果发现26%的样本标签错误。FERNANDES等[10]采用DNA条形码对不同种鳕鱼实现了快速鉴别和检测。
线粒体细胞色素C氧化酶亚基Ⅰ(cytochrome c oxidase subunit Ⅰ,COⅠ)基因,是一种位于线粒体DNA上的蛋白编码基因。该基因具有突变速率适中、易于扩增和高数据库覆盖率等优点,因此已成为动物物种鉴别的主要DNA条形码之一[11]。此外,除了COⅠ基因,16S核糖体RNA(16S ribosomal RNA,16S rRNA)基因同时含有高度保守与高度变异的序列区域,也广泛应用于物种鉴别和遗传多样性分析。黄权等[12]利用COⅠ和16S rRNA基因对虹鳟鱼类进行了分子鉴定;陆键萍等[13]利用COⅠ、Cyt b 和16S rRNA基因对6种金枪鱼进行了遗传差异分析和进化树分析;徐岩等[14]利用16S rRNA和COⅠ基因对中国沿海的相手蟹进行品种鉴别,并分析了其种间变异程度及亲缘关系。上述研究表明,COⅠ和16S rRNA基因已广泛应用于鱼类分类学、遗传差异分析和亲缘关系研究中,然而这些基因在鱼胶鉴别方面的适用性仍然缺乏系统性的评估和验证。本研究综合利用2种基因片段,对赤嘴鳘公胶、赤嘴鳘母胶、非洲黄花胶、东南亚黄花胶、新西兰鳕鱼胶、冰岛鳕鱼胶6种鱼胶进行分子鉴别和遗传多样性分析,初步探讨2种DNA条形码在鱼胶品种鉴别中的适用性,旨在为鱼胶产品品种来源的科学鉴别提供理论依据,促进鱼胶产业的健康发展。
1 材料与方法
1.1 试验材料
选取6个品种鱼胶,共30个样品。如表1所示,均购于汕头市颐膳美食品有限公司。
表1 鱼胶样品信息
Table 1 Information of collected fish maw samples

编号样品名称产地样品数简称1赤嘴鳘(公胶)中国4CG 1-42赤嘴鳘(母胶)中国4CM 1-43东南亚黄花胶东南亚6DH 1-64非洲黄花胶非洲6FH 1-65新西兰鳕鱼胶新西兰5XXL 1-56冰岛鳕鱼胶冰岛5BD 1-5
1.2 试剂与仪器
HiPure Tissue DNA Mini Kit试剂盒,广州美基公司;2×Taq PCR Master Mix、DL2000 DNA marker,南京诺唯赞公司;50×TAE 电泳缓冲液,北京索莱宝公司;琼脂糖,北京擎科公司;MonTrackTM Safe Red核酸染料,苏州莫纳生物公司;PCR引物,北京睿博兴科公司;T960PCR热循环仪,上海力康生物公司;Tanon 1600凝胶成像分析系统,上海天能公司;N60 Nano Photometer超微量分光光度计,德国Implen公司;H1850R高速离心机,湖南湘仪公司;DYCP-31电泳仪,北京六一公司。
1.3 实验方法
1.3.1 DNA 提取
取成品鱼胶约30 mg, 用锉刀磨成较细颗粒[15],按照HiPure Tissue DNA Mini Kit试剂盒说明书提取鱼胶DNA。用超微量分光光度计测定所提取DNA的浓度和纯度,当DNA质量浓度>50 ng/μL,A260/A280在1.8~2.0,A260/A230>2.0时,符合DNA浓度和纯度要求,于-20 ℃冰箱保存DNA。
1.3.2 PCR扩增
PCR扩增引物序列如表2所示[16-17],反应体系(总体积50 μL)为:PCR Master Mix 25 μL,上下游引物各2 μL,DNA模板1 μL,去离子水20 μL。COⅠ和16S rRNA基因的退火温度均为56 ℃,具体反应条件为:95 ℃预变性3 min;95 ℃变性15 s,56 ℃退火15 s,72 ℃延伸40 s,35个循环;72 ℃延伸10 min。将PCR扩增得到的产物进行1.0%琼脂糖凝胶电泳,通过凝胶成像分析系统观察和分析实验结果,将符合要求的PCR产物送至广州天一辉远基因科技有限公司进行序列测定。
表2 引物信息
Table 2 Information of primers

扩增区域引物名称引物序列扩增长度/bp参考文献COⅠFishF1TCAACCAACCACAAA-GACATTGGCACFishR1TAGACTTCTGGGTG-GCCAAAGAATCAFishF2TCGACTAATCATA-AAGATATCGGCACFishR2ACTTCAGGGTGAC-CGAAGAATCAGAA760760[16]16S rRNA16S-FCGCCTGTTTAT-CAAAAACAT16S-RCCGGTCTGAACT-CAGATCACGT650[17]
1.3.3 数据分析
利用NCBI网站中的BLAST功能对测序结果进行比较分析, 序列相似性达到99%的即鉴定为同一个物种[18]。根据比对结果,从NCBI网站中下载相关鱼种的参考序列。运用Mega 11软件进行多重序列比对,删除两端引物序列,统计序列碱基组成,计算序列保守位点(C)、简约信息位点(PI)和变异位点(V)。基于Kimura 2-parameter模型,计算种内和种间遗传距离,并采用邻接法(neighbor-joining,N-J)自展检验1 000次,构建系统发育进化树[19-20]。利用DnaSP 6.12软件计算样品COⅠ基因和16S rRNA基因序列的遗传多样性参数。
2 结果与分析
2.1 PCR扩增结果
30个样品经过PCR扩增和测序。其中,COⅠ基因有2个鱼胶样品无法扩增成功,16S rRNA基因全部扩增成功,原因可能是COⅠ基因片段在鱼胶制品加工过程中发生了降解[21],在鱼翅的DNA扩增过程中也有类似情况[22]。所有成功扩增的样品均呈单一片段,片段长度无多态性,经测序得知COⅠ基因片段大小约为760 bp,16S rRNA基因片段大小约为650 bp,与预期结果一致。COⅠ和16S rRNA基因的部分样品扩增电泳图如图1所示。

1~6-COⅠ基因 760 bp;7~12-16S rRNA基因650 bp;M-DNA marker;13-空白对照
图1 部分样品COⅠ和16S rRNA基因的扩增电泳图
Fig.1 Electrophoresis patterns of COⅠ and 16S rRNA genes from part of the samples
2.2 序列比对与物种溯源
基于DNA条形码技术,将获得的COⅠ基因和16S rRNA基因序列进行数据库比对及聚类分析鉴定,鉴定结果如表3所示,30个鱼胶分别来源于5个鱼种:双棘原始黄姑鱼(Protonibea diacanthus)、岬羽鼬鳚(Genypterus capensis)、大头鳕(Gadus macrocephalus)、尼罗尖吻鲈(Lates niloticus)、尖吻鲈(Lates calcarifer)。由表3可知,除COⅠ基因未扩增出的2个样品,本研究中使用的2对引物,无论是以COⅠ还是16S rRNA基因作为靶标,都能够成功地获得待测样品的准确物种信息,且其扩增鉴定结果一致。
表3 六种鱼胶样品的物种鉴定结果
Table 3 Species identification of six fish maw samples

样品编号商品名COⅠ鉴定结果16S rRNA鉴定结果结合2个靶基因的鉴定结果CG 1-4赤嘴鳘(公胶)P.diacanthusP.diacanthus双棘原始黄姑鱼(P.diacanthus)CM 1-4赤嘴鳘(母胶)P.diacanthusP.diacanthus双棘原始黄姑鱼(P.diacanthus)XXL 1-5新西兰鳕鱼胶G.capensisG.capensis岬羽鼬鳚(G.capensis)BD 1-5冰岛鳕鱼胶G.macrocephalusG.macrocephalus大头鳕(G.macrocephalus)FH 1-5非洲黄花胶L.niloticusL.niloticus尼罗尖吻鲈(L.niloticus)DH 1-5东南亚黄花胶L.calcariferL.calcarifer尖吻鲈(L.calcarifer)FH 6非洲黄花胶扩增失败L.niloticus—DH 6东南亚黄花胶扩增失败L.calcarifer—
2.3 遗传多样性分析
6种鱼胶基因片段的碱基组成见表4,其中COⅠ基因的A+T碱基平均含量为51.8%,高于G+C碱基平均含量(48.2%);16S rRNA基因中A+T平均含量为52.9%,也高于G+C平均含量(47.1%),2种基因片段都表现出A+T偏倚性,这也证实了线粒体蛋白质编码基因的一个显著特征是其核苷酸碱基组成存在偏倚性[11],在其他虹鳟、鲑科鱼类中也出现类似情况[12, 23]。
表4 六种鱼胶COⅠ和16S rRNA基因碱基组成
Table 4 Base composition of COⅠ and 16S rRNA genes in six fish maw
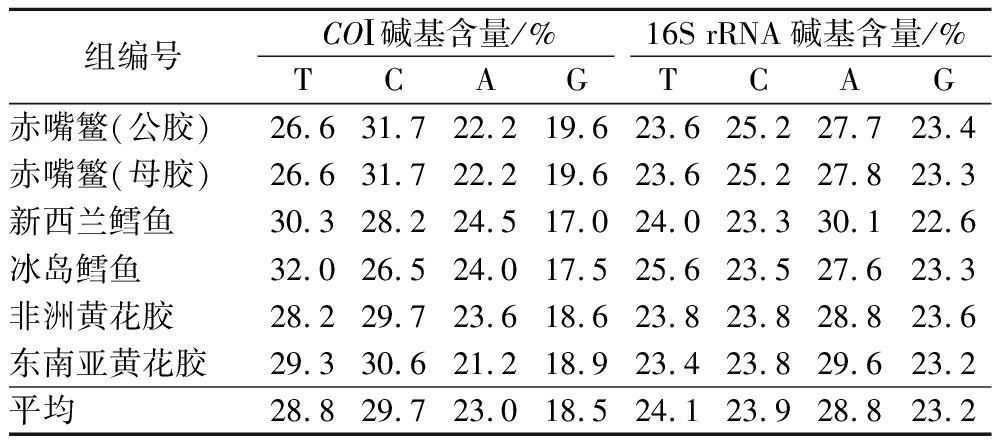
组编号COⅠ碱基含量/%16S rRNA碱基含量/%TCAGTCAG赤嘴鳘(公胶)26.631.722.219.623.625.227.723.4赤嘴鳘(母胶)26.631.722.219.623.625.227.823.3新西兰鳕鱼30.328.224.517.024.023.330.122.6冰岛鳕鱼32.026.524.017.525.623.527.623.3非洲黄花胶28.229.723.618.623.823.828.823.6东南亚黄花胶29.330.621.218.923.423.829.623.2平均28.829.723.018.524.123.928.823.2
表5为各项遗传多样性参数统计。鱼胶各物种的16S rRNA序列变异位点(V)为140,变异率为24.96%,远低于COⅠ基因34.25%的变异率。说明16S rRNA基因比COⅠ基因保守,表明了16S rRNA基因遗传物质的稳定性。16S rRNA序列转换/颠换比(R)为1.63,COⅠ序列为2.00,通常认为,当R值越大时,基因突变的序列的饱和程度越低,基因序列更适合分子进化和遗传多样性分析[24-25]。本研究中,COⅠ序列的R值大于16S rRNA序列,说明COⅠ序列更适合进行进化树分析。单倍型多样性指数(Hd)、核苷酸多样性指数(π)是衡量一个物种群体多样性非常重要的指标[26]。从Hd值来看,各物种的16S rRNA和COⅠ基因序列中均没有完全相同的序列,所以Hd值均为1.0,说明2种基因的遗传资源丰富,遗传多样性高。从π来看,16S rRNA基因的π值小于COⅠ基因,π值越小,说明核苷酸变异程度越低,综上所述,COⅠ序列相比于16S rRNA序列,具有更高的核苷酸变异程度、遗传多样性和分化程度。
表5 COⅠ和16S rRNA基因片段的遗传多样性参数
Table 5 Genetic diversity parameters for COⅠ and 16S rRNA gene fragments

基因序列长度遗传多样性参数CPIVRhHdkπCOⅠ6544291032252.0051.0131.50.201 07±0.026 4016S rRNA555415591401.6351.074.70.134 59±0.023 09
注:C-保守位点;PI-简约信息位点;V-变异位点;R-转换/颠换比;h-单倍型数;Hd-单倍型多样性指数;k-平均核苷酸差异数;π-核苷酸多样性指数。
2.4 种内与种间遗传距离
表6为COⅠ和16S rRNA基因片段在种内和种间的遗传距离。COⅠ序列的种内遗传距离为0.08%~1.15%,平均值为0.56%;种间遗传距离范围在16.41%~22.91%,平均值为20.26%。平均种间遗传距离是种内遗传距离的36.18倍。16S rRNA序列的种内遗传距离范围在0.13%~0.83%,平均值为0.37%;而种间遗传距离范围在4.17%~17.77%,平均值为13.93%。平均种间遗传距离是种内遗传距离的37.64倍。这些结果符合HEBERT等[6]提出的“10倍规则”,即种间平均遗传距离大于种内遗传距离10倍时可用于物种鉴别。
表6 COⅠ和16S rRNA基因片段的遗传距离 单位:%
Table 6 Genetic distance between COⅠ and 16S rRNA gene fragments
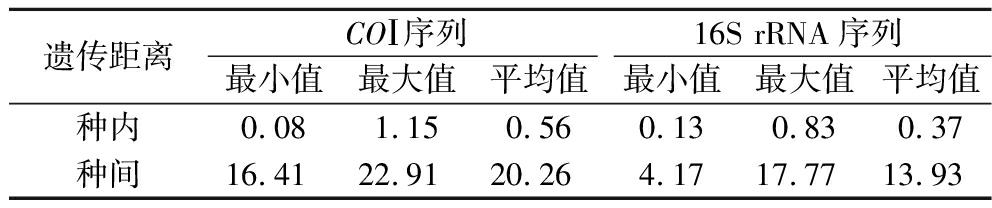
遗传距离COⅠ序列16S rRNA序列最小值最大值平均值最小值最大值平均值种内0.081.150.560.130.830.37种间16.4122.9120.264.1717.7713.93
2.5 系统进化树
2.5.1 COⅠ基因
基于COⅠ基因的N-J系统进化树见图2-a。赤嘴鳘鱼胶(CG和CM)的8个样品和双棘原始黄姑鱼种(P.diacanthus)聚为一支,新西兰鳕鱼胶(XXL)的5个样品和岬羽鼬鳚鱼种(G.capensis)聚为一支,冰岛鳕鱼胶(BD)的5个样品和大头鳕鱼种(G.macrocephalus)聚为一支,非洲黄花胶(FH)的5个样品和尼罗尖吻鲈鱼种(L.niloticus)聚为一支,东南亚黄花胶(DH)的5个样品和尖吻鲈鱼种(L.calcarifer)聚为一支,与表3的鉴定结果一致。其中,东南亚黄花胶和非洲黄花胶聚为一个单元,冰岛鳕鱼胶和新西兰鳕鱼胶聚为一个单元,表现出彼此相近的遗传关系。赤嘴鳘公胶和赤嘴鳘母胶属于同一鱼种双棘原始黄姑鱼,与理论相符。从COⅠ基因构建的系统发育树可以看出,五大类鱼胶有明显的物种群体聚类的趋势,结果表明,COⅠ基因能较好区别五大类鱼胶产品。

a-COⅠ基因;b-16S rRNA基因
图2 N-J系统进化树
Fig.2 N-J phylogenetic tree
2.5.2 16S rRNA基因
基于16S rRNA基因的N-J系统进化树见图2-b。16S rRNA基因构建的发育树的聚类结果与COⅠ基因略有不同,非洲黄花胶(FH)与东南亚黄花胶(DH)同属于一个单元(尖吻鲈属),其中包含了2个明显的分支,非洲黄花胶(FH)的5个样品聚在一个分支上,属于尼罗河尖吻鲈种(L.niloticus);东南亚黄花胶(DH)的5个样品聚在另一个分支上,属于尖吻鲈种(L.calcarifer)。结果表明,16S rRNA基因在尼罗尖吻鲈种和尖吻鲈种之间的种间遗传距离较小,但是也能单独区分出非洲黄花胶(FH)与东南亚黄花胶(DH),可以对不同品种鱼胶产品进行聚类分析。因此,相比于COⅠ基因来说,16S rRNA基因在部分同源性较高的鱼种序列上不如COⅠ基因分类明确,16S rRNA基因序列相对较为保守,这在不同品种金枪鱼的遗传分析上也有类似结果[13]。
3 结论
本研究以市场上热销的6种鱼胶30个样品为研究对象,分别采用COⅠ和16S rRNA基因序列进行进化分析,研究多基因条形码在鱼胶鉴别中的适用性。结果表明,16S rRNA基因的扩增成功率高于COⅠ基因,但16S rRNA基因序列变异率远小于COⅠ基因,16S rRNA基因遗传物质更具有稳定性。在遗传距离方面,COⅠ和16S rRNA序列的平均种间和种内遗传距离均符合10倍阈值原则,可以满足鱼胶品种间精准鉴别的需求。根据建树结果,16S rRNA基因构建的发育树与COⅠ基因的聚类结果基本一致,但是在非洲黄花胶和东南亚黄花胶的建树结果上,16S rRNA基因的分类不如COⅠ基因明确。总体而言,COⅠ基因和16S rRNA基因序列均可作为鱼胶加工品鉴别的有效DNA条形码,但COⅠ和16S rRNA基因都存在一些不足之处。COⅠ基因更容易在加工过程中发生片段降解,16S rRNA基因在部分同源性较高的物种鉴别中不如COⅠ基因分类明确。因此,在鱼胶品种鉴别应用中,建议同时使用COⅠ和16S rRNA两种基因作为DNA条形码靶标,两者联用下鉴别准确率可达100%。
[1] WEN J, ZENG L, CHEN Z M, et al.Comparison of nutritional quality in fish maw product of croaker Protonibea diacanthus and perch Lates niloticus[J].Journal of Ocean University of China, 2016, 15(4):726-730.
[2] 朱凯悦, 孙娜, 董秀萍, 等.鱼胶的研究进展[J].食品与发酵工业, 2022, 48(3):284-290.
ZHU K Y, SUN N, DONG X P, et al.Research progress of isinglass[J].Food and Fermentation Industries, 2022, 48(3):284-290.
[3] 林柏岸. 中国市场贸易中常见花胶的DNA条形码(COⅠ)鉴定[D].厦门:厦门大学, 2018.
LIN B A.Identification of common shellac in China market by DNA barcode (COⅠ)[D].Xiamen:Xiamen University, 2018.
[4] MONIZ M B J, KACZMARSKA I.Barcoding of diatoms:Nuclear encoded ITS revisited[J].Protist, 2010, 161(1):7-34.
[5] NEHAL N, CHOUDHARY B, NAGPURE A, et al.DNA barcoding:A modern age tool for detection of adulteration in food[J].Critical Reviews in Biotechnology, 2021, 41(5):767-791.
[6] HEBERT P D N, CYWINSKA A, BALL S L, et al.Biological identifications through DNA barcodes[J].Proceedings.Biological Sciences, 2003, 270(1512):313-321.
[7] TSOUPAS A, PAPAVASILEIOU S, MINOUDI S, et al.DNA barcoding identification of Greek freshwater fishes[J].PLoS One, 2022, 17(1):e0263118.
[8] SHARRAD A E, REIS-SANTOS P, AUSTIN J, et al.Umbrella terms conceal the sale of threatened shark species:A DNA barcoding approach[J].Food Control, 2023, 148:109606.
[9] PARDO M  , JIMÉNEZ E, VI
, JIMÉNEZ E, VI ARSSON J R, et al.DNA barcoding revealing mislabeling of seafood in European mass caterings[J].Food Control, 2018, 92:7-16.
ARSSON J R, et al.DNA barcoding revealing mislabeling of seafood in European mass caterings[J].Food Control, 2018, 92:7-16.
[10] FERNANDES T J R, COSTA J, OLIVEIRA M B P P, et al.COI barcode-HRM as a novel approach for the discrimination of Hake species[J].Fisheries Research, 2018, 197:50-59.
[11] 律迎春. 基于DNA条形码的分子生物学方法鉴定海参种类的研究[D].青岛:中国海洋大学, 2012.
LYU Y C.Identification of sea cucumber species by molecular biology method based on DNA barcode[D].Qingdao:Ocean University of China, 2012.
[12] 黄权, 蒋焯.基于线粒体COⅠ和16S rRNA基因序列的虹鳟DNA条形码研究[J].吉林农业大学学报, 2022, 44(2):215-220.
HUANG Q, JIANG Z.Rainbow trout(Oncorhynchus mykiss) DNA barcode based on bitochondrial COⅠ and 16S rRNA gene sequences[J].Journal of Jilin Agricultural University, 2022, 44(2):215-220.
[13] 陆键萍, 姚琳, 信红梅, 等.线粒体COⅠ、Cyt b和16S rRNA基因在6种金枪鱼鉴定中的适用性分析[J].渔业科学进展, 2020, 41(5):73-81.
LU J P, YAO L, XIN H M, et al.Applicability analysis of mitochondrial COⅠ, Cyt b and 16S rRNA genes in identification of six tuna species[J].Progress in Fishery Sciences, 2020, 41(5):73-81.
[14] 徐岩, 潘红平, 阎冰, 等.基于线粒体COⅠ和16S rRNA基因的中国沿海相手蟹系统发育研究[J].海洋学报, 2019, 41(8):63-71.
XU Y, PAN H P, YAN B, et al.Molecular phylogeny of the sesarmid crab based on the partial sequences of mitochondrial COⅠ and 16S rRNA genes from the coast of China[J].Haiyang Xuebao, 2019, 41(8):63-71.
[15] 曹卉, 田晓玲, 刘昕.鱼鳔的分子鉴别及其止血作用的药理学研究[J].中国食品学报, 2009, 9(4):170-176.
CAO H, TIAN X L, LIU X.Study on molecular identification and pharmacology of hemostasis action for isinglass[J].Journal of Chinese Institute of Food Science and Technology, 2009, 9(4):170-176.
[16] WARD R D, ZEMLAK T S, INNES B H, et al.DNA barcoding Australia’s fish species[J].Philosophical Transactions of the Royal Society of London.Series B, Biological Sciences, 2005, 360(1462):1847-1857.
[17] PALUMBI S R, MARTIN A, ROMANO S, et al.The Simple Fool’s Guide to PCR, Version 2.0, Privately Published Document Compiled by S.Palumbi[Z].Honolulu:University of Hawaii, 1991:28-29.
[18] ZENG L, ARMANI A, WEN J, et al.Molecular identification of seahorse and pipefish species sold as dried seafood in China:A market-based survey to highlight the actual needs for a proper trade[J].Food Control, 2019, 103:175-181.
[19] UTHICKE S, BYRNE M, CONAND C.Genetic barcoding of commercial Bêche-de-mer species (Echinodermata:Holothuroidea)[J].Molecular Ecology Resources, 2010, 10(4):634-646.
[20] TINACCI L, STRATEV D, STRATEVA M, et al.An authentication survey on retail seafood products sold on the Bulgarian market underlines the need for upgrading the traceability system[J].Foods, 2023, 12(5):1070.
[21] 朱崧琪, 史亚千, 黄超华, 等.鱼胶基原鱼种鉴定技术研究进展[J].食品安全质量检测学报, 2022, 13(11):3593-3601.
ZHU S Q, SHI Y Q, HUANG C H, et al.Research progress of identification techniques for original species of isinglass[J].Journal of Food Safety &Quality, 2022, 13(11):3593-3601.
[22] 熊娟, 黄启红, 陈敏儿, 等.用DNA条形码鉴定常见鱼翅的种类与真伪[J].水产学杂志, 2020, 33(6):28-33.
XIONG J, HUANG Q H, CHEN M E, et al.Identification of species and authenticity of shark fins by DNA barcoding technology[J].Chinese Journal of Fisheries, 2020, 33(6):28-33.
[23] 孙毅. 基于线粒体基因COX1、Cyt b和ND4的鲑科鱼类的系统发育[J].畜牧与饲料科学, 2015, 36(9):9-17;61.
SUN Y.Phylogenetic analysis of Salmonidae based on mitochondrial genes COX1, Cyt b and ND4[J].Animal Husbandry and Feed Science, 2015, 36(9):9-17;61.
[24] 周发林, 江世贵, 苏天凤, 等.6种笛鲷属鱼类线粒体16S rRNA基因片段的序列比较[J].中国水产科学, 2004(2):99-103.
ZHOU F L, JIANG S G, SU T F, et al.Comparative study of mtDNA 16S rRNA gene fragments among six Lutjanus fishes[J].Journal of Fishery Sciences of China, 2004(2):99-103.
[25] 郑文娟, 朱世华, 邹记兴, 等.基于16S rRNA部分序列探讨12种鲹科鱼类的分子系统进化关系[J].水产学报, 2008, 32(6):847-854.
ZHENG W J, ZHU S H, ZOU J X, et al.Molecular phylogenetic relationship of Carangidae based on partial sequence of mitochondrial 16S ribosomal RNA gene[J].Journal of Fisheries of China, 2008, 32(6):847-854.
[26] 王开杰, 徐永江, 崔爱君, 等.基于Cyt b、ND1及ND2的DNA条形码在![]() 属鱼类物种鉴定中的应用[J].渔业科学进展, 2022, 43(6):89-101.
属鱼类物种鉴定中的应用[J].渔业科学进展, 2022, 43(6):89-101.
WANG K J, XU Y J, CUI A J, et al.Application of DNA barcoding based on Cyt b, ND1 and ND2 in Seriola species identification[J].Progress in Fishery Sciences, 2022, 43(6):89-101.