果蔬汁饮料、碳酸饮料、运动饮料等酸性饮料深受各国消费者喜爱,然而随着人们健康消费意识提升,成分单一的酸性饮料已无法满足消费者营养需求。大豆蛋白是中国大宗优质植物蛋白资源,具有较高的营养价值和独特的保健功能。将大豆蛋白添加到酸性饮料中,不仅可提升产品的风味,还可改善现有酸性饮料营养成分单一的缺陷,满足消费者营养均衡的需求。然而大多数酸性饮料的pH值介于3.0~4.5,大豆蛋白的等电点在pH 4.5附近,大豆蛋白在等电点区域絮凝沉淀限制了其在酸性饮料中的应用[1-2]。国外已对大豆蛋白的改性技术和酸溶特性进行了大量深入研究,取得了系列成果,美国和日本已开发出具有良好酸溶特性的大豆蛋白产品,广泛应用于各种酸性饮料[3]。但这些技术基本没有申请专利,属于技术机密。
酶解改性是利用蛋白水解酶在温和条件下催化蛋白质中特定肽键水解断裂,所生成的小分子肽亲水性更强,分子柔顺性更好,更易与水分子发生水合作用,因此具有更好的溶解特性[4-6]。酶解改性安全高效,无氨基酸破坏和有害物质产生,故被认为是食品蛋白改性的最佳方法[4]。蛋白酶的选择对酶解改性的效果至关重要,目前的研究现状往往是随机选取蛋白酶,然后基于“试错法”进行实验筛选。然而蛋白酶的种类繁多,从大量蛋白酶中进行实验筛选,工作量大且存在一定盲目性[5-7]。
随着现代生物信息学的快速发展,各种蛋白质数据库逐步完善,许多蛋白酶的酶切位点也已比较清楚,一种基于已知蛋白质氨基酸序列的模拟酶切技术成为解决当前瓶颈的有效手段[8]。PeptideCutter是由瑞士生物信息学研究所(ExPASy)开发的蛋白模拟酶切软件,可通过输入蛋白氨基酸序列和蛋白酶酶切位点,模拟蛋白酶解;输出结果包括酶切肽段的氨基酸序列、分子质量、酶切位点和水解度等。该软件还提供了ExPASy开发的各种在线蛋白分析工具,可用于分析生成肽段的理化性质和生物活性,从而预测酶解改性的效果,指导选择适用蛋白酶[9]。如于志鹏等[10]采用PeptideCutter软件对5种鸡蛋蛋白进行模拟酶切,然后利用ExPASy提供的在线工具对生成肽段的血管紧张素转化酶抑制活性和溶解特性进行分析预测,建立了适用水解酶的高通量筛选方法。BLEAKLEY等[11]采用PeptideCutter对燕麦蛋白进行模拟酶切,以生成的寡肽(含有10个氨基酸以下的肽段)数量为指标建立肽库,从89种蛋白酶中筛选出4种优势酶,显著降低了实验成本并制备出了高二肽基肽酶抑制活性的酶解产物。值得注意的是利用模拟酶切的方法筛选适用水解酶,首先应掌握蛋白的氨基酸序列信息。目前大豆蛋白各组分中,只有7S球蛋白(β-伴大豆球蛋白)的完整氨基酸序列已经破译[12],然而基于模拟酶切指导改善7S酸溶特性的研究国内外还未见报道。因此,本研究以7S为底物蛋白,在PeptideCutter软件中采用不同酶切位点的蛋白酶对7S进行模拟酶切,然后采用ExPASy在线工具对生成肽段的酸溶特性进行分析预测,筛选出优势蛋白酶并进行实验验证。本研究为高效筛选适用于改善大豆蛋白酸溶特性的蛋白酶提供了新的思路和方法指导,也有利于实现大豆蛋白产品在酸性饮料中的应用。
1 材料与方法
1.1 材料与试剂
脱脂大豆粕(蛋白质量分数51.3%,干基),山东禹王食品有限公司;蛋白酶K(EC 3.4.21.62,3.0×105 U/g)、胰凝乳蛋白酶(EC 3.4.21.1,1.5×106 U/g)、胰蛋白酶(EC 3.4.21.4,2.5×105 U/g),上海源叶生物科技有限公司;胃蛋白酶(EC 3.4.23.1,6.0×105 U/g),北京鼎国生物技术有限公司;嗜热菌蛋白酶(EC 3.4.24.27,3.5×105 U/g)、牛血清蛋白,美国Sigma-Aldrich公司;电泳预制胶及相关试剂,美国Bio-rad公司;100%酸橙汁,美国Tropicana公司;其他化学试剂均为分析纯。
1.2 仪器与设备
PeptideCutter模拟酶切软件,瑞士生物信息学研究所;HJ-6A多头磁力搅拌器、DF-101S集热式磁力搅拌器,金坛市城东新瑞仪器厂;CR22G 高速冷冻离心机,日本日立电器公司;KDN-40A 凯氏定氮仪,上海新嘉电子有限公司;ZYS20-1000超声波粉碎机,杭州振源仪器有限公司;FA2204B电子天平,上海天美天平仪器有限公司;TIM840自动电位滴定仪,美国Radiometer公司;Mini-protean IV电泳系统,美国Bio-Rad公司;PHS-3C pH计、L6S紫外可见分光光度计,上海仪电分析仪器有限公司;Zetasizer Nano ZE-90纳米粒度及电位分析仪,英国Malvern公司;Turbiscan MA2000稳定性分析仪,法国Formulaction公司。
1.3 实验方法
1.3.1 大豆7S球蛋白的制备与纯化
参考DEAK等[13]的方法,稍加改进,过程简述如下:低温脱脂豆粕粉碎过200目筛,所得粉体加入Tril-HCl缓冲液(0.03 mol/L,下同),料液比为1∶15(g∶mL),调节pH值至8.5,45 ℃搅拌1 h;离心后(9 000×g,30 min)去渣得蛋白浸提液;在浸提液中加固体NaHSO3至0.3 mmol/L、加CaCl2至0.02 mmol/L,调pH值至6.4,4 ℃静置过夜,离心后(6 500×g,30 min)收集上清液为7S球蛋白富集组分;在7S富集组分中加固体NaCl至0.25 mol/L,调节pH值至5.5,磁力搅拌1 h,离心后(9 000×g,30 min)收集上清液;两倍体积冰水稀释上清液,搅拌1 h,调pH值至4.8(7S等电点),离心(9 000 ×g,30 min)收集沉淀并溶解于Tris-HCl缓冲液,调节pH值至6.2,离心后(9 000×g,30 min,4 ℃)收集上清液,调节pH值至7.6得到粗β-伴大豆球蛋白提取液。向提取液中加入固体(NH4)2SO4至饱和度51%,离心后(9 000×g,30 min)收集上清液,加入固体(NH4)2SO4至饱和度100%,所得沉淀用磷酸盐缓冲液(0.01 mol/L,pH 7.6)溶解,透析脱盐后冷冻干燥得到大豆7S球蛋白,研磨后过40目筛,于密封干燥容器中保藏备用。
1.3.2 大豆7S球蛋白主要成分测定
水分含量测定参照GB 5009.3—2016《食品中水分的测定》(直接干燥法);灰分测定参照GB 5009.4—2016《食品中灰分的测定》(灼烧称重法);蛋白质含量测定参照GB 5009.5—2016《食品中蛋白质的测定》(凯氏定氮法);脂肪含量测定参照GB/T 5009.6—2016《食品中脂肪的测定》(索氏抽提法)。
1.3.3 大豆7S球蛋白的模拟酶切及优势蛋白酶的选择
从UniProt数据库中(https://www.uniprot.org/)搜集大豆7S球蛋白α′、α、β亚基的氨基酸序列及相关资料(表1),整理后倒入模拟酶切软件PeptideCutter的蛋白数据库。搜集和总结国内外报道用于改善蛋白溶解特性的蛋白酶,然后选用PeptideCutter中提供的39种蛋白酶对7S中3种亚基分别进行模拟酶切,内置软件经计算得出不同酶水解释放的肽片段数量、氨基酸序列及水解度。将模拟酶切生成肽段氨基酸序列导入Compute pI/Mw在线分析软件(https://web.expasy.org/compute_pi/),得到酶切肽段的等电点(isoelectric point, pI)。根据蛋白等电点与溶解特性的关系,从生成肽段中挑选pI>pH 6.0或pI
表1 大豆7S球蛋白中α′、α和β亚基的氨基酸 序列及相关资料
Table 1 Amino acid sequences and related information of α′、α and β subunits for soy 7S globulin
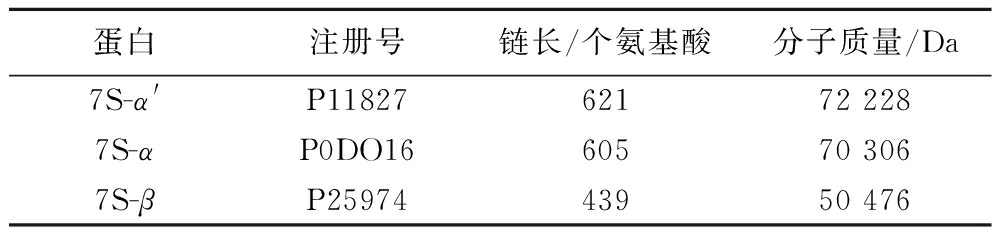
蛋白注册号链长/个氨基酸分子质量/Da7S-α'P1182762172 2287S-αP0DO1660570 3067S-βP2597443950 476
注:数据资料来自UniProt蛋白资源数据库。
1.3.4 大豆7S球蛋白的酶解及水解度(degree of hydrolysis,DH)测定
将上述制备的7S球蛋白分散到去离子水中配制成50 g/L的分散液,常温下搅拌2 h。对7S分散液进行超声波预处理以提高其酶解敏感性,超声功率400 W,处理时间10 min(每处理5 s,停1 s)。预处理后,采用1 mol/L HCl溶液和1 mol/L NaOH溶液调节样品分散液pH稳定至酶解最适值,置于恒温水浴锅中,调节水浴温度至最适酶解温度,启动磁力搅拌器,然后加入一定量的蛋白酶进行酶解(酶用量为1.0%,质量分数,蛋白酶/底物蛋白)。酶解过程中采用自动电位滴定仪自动滴加0.1 mol/L NaOH溶液入样品分散液,从而控制pH值恒定为最适值。在不同酶解时间(30~360 min)记录碱液消耗量,并根据pH-stat法计算酶解产物的DH[14]。酶解至终点时,将酶解液置于沸水浴中加热灭酶15 min。冷却至室温后在8 000×g下离心15 min后取上清液,调节pH值至7.0后冷冻干燥,研磨过40目筛,然后置于密封干燥容器中保藏备用。样品命名由底物蛋白和蛋白酶组成。例如7SH-蛋白酶K是指经蛋白酶K酶解改性后制备的7S酶解产物。
1.3.5 蛋白质溶解性的测定
将蛋白样品分散到去离子水中配制成10 g/L的分散液,常温下搅拌2 h。采用1 mol/L HCl溶液和1 mol/L NaOH溶液调节样品分散液pH稳定至目标pH(3.0~7.0),然后在8 000×g下离心15 min后取上清液。上清液中蛋白含量采用Lowry法[15]测定,以牛血清蛋白为标准物做标准曲线。蛋白质的溶解度(protein solubility,PS)表示为上清液蛋白浓度占总蛋白浓度的百分比。
1.3.6 SDS-PAGE
采用Bio-rad公司的预制胶进行电泳实验,该产品浓缩胶质量分数为4%,分离胶为质量分数4%~15%的梯度胶。将样品溶解于样品缓冲液(0.062 5 mol/L Tris-HCl buffer (pH=6.8),20 g/L十二烷基磺酸钠(sodium dodecyl sulphate, SDS-PAGE),5%(体积分数)巯基乙醇,25%(体积分数)甘油和0.1 g/L 溴酚蓝)中配制成质量浓度为10 mg/mL的分散液。95 ℃下水浴加热5 min,离心后(8 000×g,15 min)取上清液。样品上样量为10 μL。凝胶电泳在恒压下进行,在浓缩胶中电压为60 V,进入分离胶后增至150 V。凝胶染色及脱色后,在凝胶成像系统中进行成像处理,采用Quantity-One软件对电泳图谱进行分析。标准蛋白样品分子质量范围14.4~97.4 kDa。
1.3.7 蛋白溶液Zeta电位的测定
将蛋白样品分散到去离子水中配制成10 g/L的分散液,常温下搅拌2 h。采用1 mol/L HCl和1 mol/L NaOH溶液调节分散液pH稳定至目标pH(1.0~7.0),然后在8 000×g下离心15 min后取上清液。采用微孔滤膜(0.45 μm Millipore膜)滤除样品上清液中蛋白颗粒,然后将其按1∶100比例用去离子水稀释。采用Zeta电位分析仪测定蛋白样品溶液的Zeta电位(mV),蛋白和水的折光指数分别设置为1.450和1.331;吸收指数为0.001。该仪器通过测定蛋白分子在电场中移动的方向和速率,计算出蛋白溶液的Zeta电位。
1.3.8 酶解改性7S球蛋白在酸性果汁中的稳定性测试
将市售酸橙汁(pH 3.86)分成若干份,加入酶解改性后的7S样品,添加量为50 g/L,然后在室温下搅拌2 h。蛋白果汁中加入叠氮钠(0.2 g/L)抑制微生物生长,并置于4 ℃冰箱中静置贮藏24 h。贮藏后,采用Turbiscan MA2000稳定性分析仪测定蛋白果汁样品的背散射光(backscattering coefficient, BS)曲线,用于评估蛋白果汁的稳定性。测试过程中,光源检测器从样品瓶底部扫描至样品瓶顶部,扫描范围为0~45 mm,测定温度为25 ℃。
1.3.9 数据统计分析
试验结果都是3次测定的平均值,采用Duncan新复极差检验结果平均值间的显著性差异(P<0.05)。
2 结果与分析
2.1 大豆7S球蛋白的主要成分及亚基组成
本论文以脱脂豆粕为原料,采用等电点沉淀法制备大豆7S球蛋白,然后采用硫酸铵沉淀法(盐析)对产物进行纯化,7S产品的得率为12.4%,主要成分含量(质量分数)为:蛋白93.1%,水分5.7%,灰分0.9%,脂肪0.02%。采用SDS-PAGE分析该7S产品的亚基组成,结果见图1。通过与文献报道的大豆蛋白组分和7S亚基组成[16]进行比较,发现本文采用的脱脂豆粕原料(条带1)含有7S球蛋白(α′-7S、α-7S,β-7S)和11S球蛋白(A1-A5-11S,B-11S),而所制备的7S产品只含有7S球蛋白和少量杂蛋白(主要为α与β亚基之间的蛋白电泳条带)。通过对蛋白电泳条带的光密度进行计算分析,得出该产品中7S球蛋白的纯度为85.3%,其中α′亚基占15.2%,α亚基占48.4%,β亚基占21.7%。

M-蛋白标准品;1-脱脂豆粕原料;2-对照7S;3-7SH-蛋白酶K;4-7SH- 胃蛋白酶;5-7SH-嗜热菌蛋白酶;6-7SH-胰凝乳蛋白酶;7-7SH-胰蛋白酶
图1 大豆7S球蛋白及其酶解产物的SDS-PAGE图谱
Fig.1 SDS-PAGE profiles of soy 7S globulin and 7S hydrolysates prepared with various proteases
2.2 目标肽库的建立和优势蛋白酶的选择
本研究利用PeptideCutter软件对7S进行模拟酶切,然后将生成肽段的氨基酸序列导入Compute pI/Mw在线分析软件,得到肽段的pI。根据蛋白质的等电点与溶解特性的关系,在模拟酶切肽段中选择pI>pH 6.0或pI
表2 不同蛋白酶对7S球蛋白模拟酶切所得目标多肽数量
Table 2 The quantities of target peptides produced by in silico enzymatic analysis of 7S globulin using various proteases

蛋白酶酶解位点模拟酶切7S后生成的目标多肽/个目标多肽的分布/个7S-α'7S-α7S-β平均值pI
注:蛋白酶的酶切位点参考PeptideCutter软件的蛋白酶数据库;P1位是指酶切肽键C端的第1个氨基酸,P1′位是指酶切肽键N端的第1个氨基酸。
2.3 不同蛋白酶对大豆7S球蛋白的酶解特性
将上述5种优势蛋白酶分别在各自最优酶解条件下(表3)对7S进行酶解,酶解产物DH随酶解时间的变化如图2所示。在酶解反应0~180 min内,5种7S酶解产物的DH不断增加,然后随着酶解时间推移趋于平缓,并在反应300 min后DH不再出现显著变化(P>0.05)。蛋白酶K、胃蛋白酶、嗜热菌蛋白酶、胰凝乳蛋白酶和胰蛋白酶制备的7S酶解产物的实际DH分别达到了27.3%、24.8%、19.6%、13.8%和10.5%。SDS-PAGE分析结果(图1)发现,上述5种优势酶可将7S中α′、α和β亚基完全降解,7S酶解产物主要由分子质量<3 000 Da的小分子肽组成。这一发现与模拟酶切预测结果基本一致,表明5种优势蛋白酶对7S的酶解程度都较高。另一方面,实验测定的7S酶解产物的DH普遍低于模拟酶切预测的DH,这可能是由于7S球蛋白空间结构的影响,使部分酶切位点被掩蔽,难以与蛋白酶结合发生水解作用[6,15]。酶解过程中的产物抑制效应也会影响蛋白酶解反应效率,从而导致7S酶解产物实际水解程度较低[5,17]。

图2 不同蛋白酶水解7S球蛋白过程中DH 随时间的变化
Fig.2 DH of hydrolysis with time when 7S globulin were acted by various proteases
表3 不同蛋白酶对7S模拟酶切所得DH与实验测定DH对比
Table 3 Comparison of in silico predicted DH and experimental determined DH of 7S hydrolysates prepared using various proteases
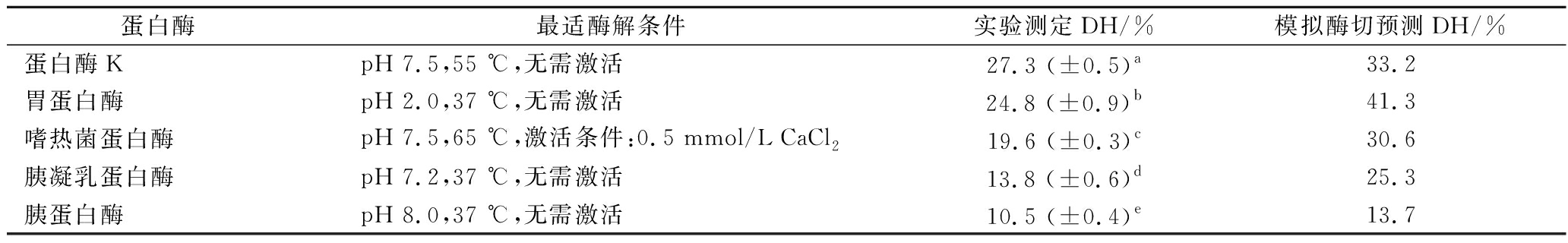
蛋白酶最适酶解条件实验测定DH/%模拟酶切预测DH/%蛋白酶KpH 7.5,55 ℃,无需激活27.3 (±0.5)a33.2胃蛋白酶pH 2.0,37 ℃,无需激活24.8 (±0.9)b41.3嗜热菌蛋白酶pH 7.5,65 ℃,激活条件:0.5 mmol/L CaCl219.6 (±0.3)c30.6胰凝乳蛋白酶pH 7.2,37 ℃,无需激活13.8 (±0.6)d25.3胰蛋白酶pH 8.0,37 ℃,无需激活10.5 (±0.4)e13.7
注:蛋白酶最适酶解条件参考酶制剂产品说明书;不同字母表示数据间有显著性差异(P<0.05)。
2.4 不同蛋白酶制备的7S酶解产物的蛋白溶解性
如图3所示,对照7S的PS-pH关系类似一条U型曲线:在pH 7.0时其PS值最高为79.6%;pH 4.5~5.0是7S的等电点区域,PS值最低,仅为6.1~6.8%;在pH 3.0~4.5(大部分酸性饮料的pH值),7S的PS值从6.8%(pH 4.5)上升至55.2%(pH 3.0)。酶解改性后,5种7S酶解产物在pH 7.0的PS值都提高到90%左右。与对照7S球蛋白相比,7S酶解产物主要由小分子肽组成,含有的极性基团数量较多,亲水性更强,且肽链长度缩短,分子柔顺性更好,因此更易于与水分子发生水合作用而具有更好的溶解特性[5-6]。随着pH降低,所有7S酶解产物的PS值在酸性条件下也有所下降,但降低的程度明显小于对照7S。据文献报道,与大分子蛋白不同,小分子肽水合作用较强,即使在其等电点区域静电排斥作用较弱时,也不易发生聚集沉淀,因此具有较好的酸溶特性[18]。除胰蛋白酶制备的7S酶解产物的最小PS值(39.4%)仍出现在7S的等电点区域(pH 4.5~5.0),其他4种7S酶解产物的最小PS值都出现在更低的pH条件。其中7SH-嗜热菌蛋白酶和7SH-胰凝乳蛋白酶的最小PS值都在pH 4.0,分别为46.2%和33.7%;7SH-胃蛋白酶的最小PS值在pH 3.0,为65.9%;7SH-蛋白酶K的PS值在pH 3.0~5.0时没有显著差异,约为81%,为所有7S酶解产物中最高。上述结果表明模拟酶切筛选出的5种优势蛋白酶都可不同程度提高7S在pH 3.0~4.5下的酸溶特性,其中蛋白酶K表现最佳。酶解改性7S酸溶特性的提高有利于改善其在酸性饮料中稳定性,从而开发出营养和口感俱佳的酸性蛋白饮料。

图3 对照7S球蛋白及其酶解产物的溶解度-pH 曲线(pH 3.0~7.0)
Fig.3 Protein solubility profiles of control 7S and its hydrolysates as a function of pH 3.0-7.0
将模拟酶切分析预测结果与实验测定结果相对照可以发现,蛋白酶K、胃蛋白酶和嗜热菌蛋白酶不仅在模拟酶切中得到较多的目标肽段,而且它们所制备的7S酶解产物的实际酸溶特性也较高。另一方面,胰凝乳蛋白酶在模拟酶切分析中得到的目标多肽数量比胰蛋白酶多,但是7SH-胰凝乳蛋白酶的酸溶特性低于7SH-胰蛋白酶。这可能是因为这两种蛋白酶模拟酶切所得目标肽段数量相差不大,而7S的酶解过程比较复杂,导致预测结果不够准确。综上所述,虽然由于7S酶解过程的复杂性,基于PeptideCutter模拟酶切的预测结果与实际情况有所差别,但对于预测酶解产物酸溶特性、筛选优势水解用酶仍然具有很好的指导作用。
2.5 不同蛋白酶制备的7S酶解产物的Zeta电位
蛋白溶解特性与其表面电荷量密切相关。7S及其酶解产物的Zeta电位-pH关系如图4所示。对照7S的Zeta电位从30.7 mV(pH 2.0)降低至-36.2 mV(pH 7.0),在pH 4.75处Zeta电位为零,为对照7S的等电点。将图3与图4对照分析可以发现在7S等电点区域,由于蛋白分子表面净电荷最少、Zeta电位最小,无法提供足够的静电排斥效应,蛋白分子间会通过疏水相互作用等次级相互作用力形成聚集物,导致其PS很低仅为6.1%;而pH值偏离等电点区域,蛋白分子表面电荷增多、Zeta电位增大,蛋白分子间的静电排斥作用抑制蛋白-蛋白聚集,宏观上表现为蛋白质的溶解度增加[19]。酶解改性对7S的Zeta电位-pH关系有重要影响,且依赖于蛋白酶的种类。5种7S酶解产物的Zeta电位-pH曲线的斜率与对照7S相比明显增大,即随着pH的变化,Zeta电位(绝对值)变化幅度增大。这可能是因为7S酶解成小分子肽后,带电基团(如—NH2+、—COO-)数量增加,使7S酶解产物表面电荷量和Zeta电位增大[18]。5种酶解产物的等电点都向酸性方向发生了偏移,这与Compute pI/Mw软件的预测结果基本一致。然而值得注意的是,除7SH-蛋白酶K外,其余4种7S酶解产物的等电点基本位于pH 3.0~4.5,而且其PS在等电点区域仍明显降低,因此不适于在酸性饮料中应用。与之相对照,7SH-蛋白酶K等电点位于pH 2.61,使其在pH 3.0~4.5带负电荷且具有较高的Zeta电位(绝对值),可提供较强的静电排斥效应,从而保持良好的溶解性。
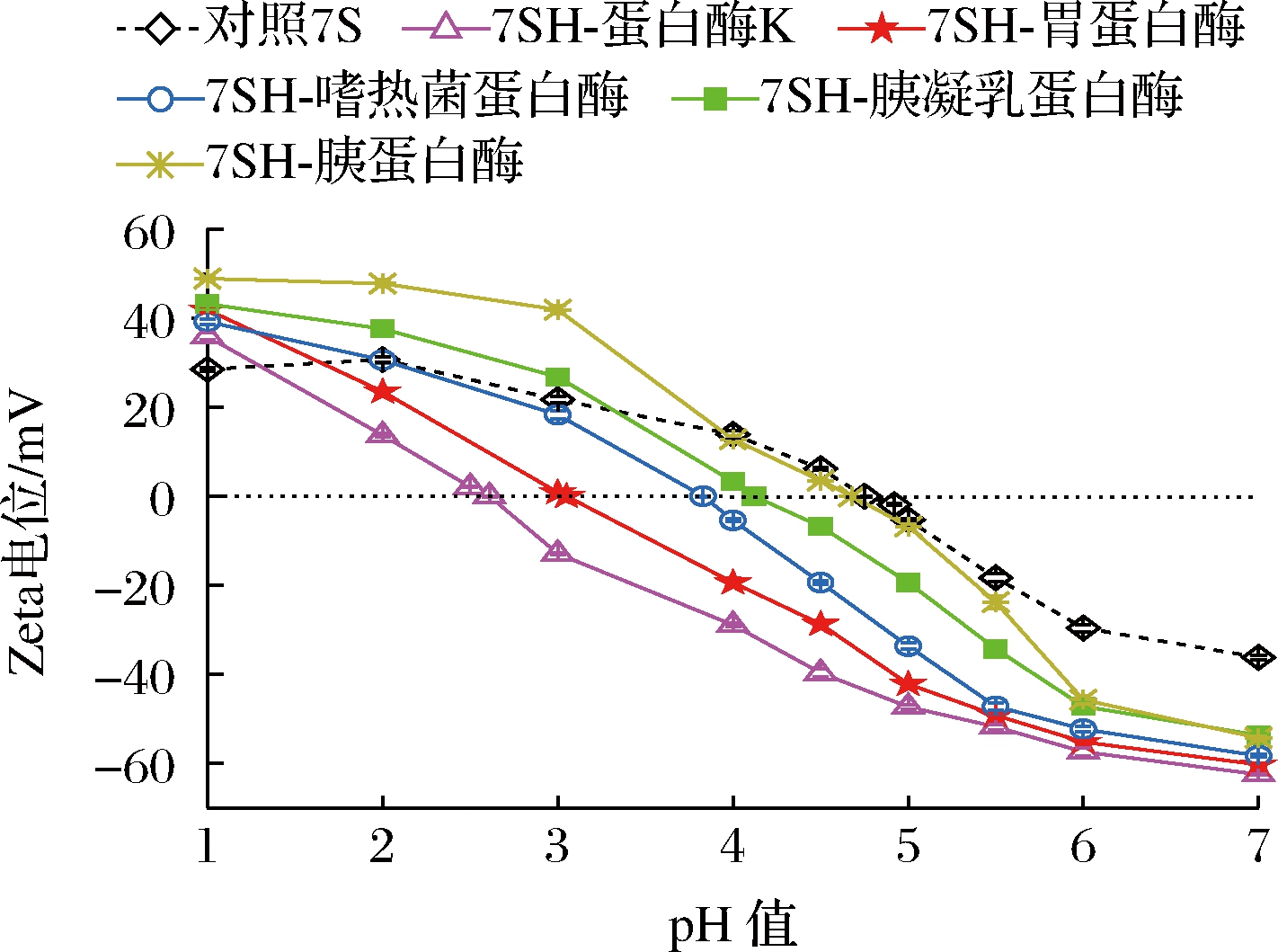
图4 对照7S球蛋白及其酶解产物的Zeta电位-pH关系图
Fig.4 Zeta potential profiles of control 7S and its hydrolysates as a function of pH 1.0-7.0
2.6 酶解改性7S球蛋白对果汁饮料稳定性的影响
Turbiscan通过测定饮料样品BS系数的分布和变化,可检测肉眼难以观察到的相分离、絮凝、沉淀等现象,从而评估饮料的稳定性[20]。图5-a为静置贮藏24 h后不同蛋白果汁样品的BS图谱,0 mm处为样品瓶底部,50 mm处为样品瓶顶部;图5-b为不同蛋白果汁样品的实物图。本实验所采用的酸橙汁(pH 3.86)在外观上呈均匀液状,其BS系数在测定高度内没有明显波动,说明该果汁中物质分布均匀。将对照7S添加到酸橙汁后,样品出现了肉眼可见的相分离现象。该样品的BS曲线底部有1个凸起峰,意味该区域反射光增强,溶液中出现沉淀;中间曲线波动较多,表明溶液中有结块或絮凝现象;顶部曲线有1个较大凸起峰,表明絮凝物漂浮在样品顶部[20-21]。由此可见,将7S直接添加到酸橙汁中会引起絮凝沉淀,严重影响饮料的外观和稳定性。与之相对照,将7SH-蛋白酶K添加到酸橙汁后,样品在外观上没有出现明显的相分离,其BS曲线底部和中部的波动较小,表明由蛋白引起的絮凝沉淀现象明显减弱;但在BS曲线上部出现1个凸起峰。推测其原因,可能是由于酸橙汁中果肉、果胶等物质通常带有负电荷[2],与7SH-蛋白酶K中少量带正电荷的酶解多肽结合,导致形成了絮状漂浮物。然而7SH-蛋白酶K的pI(pH 2.61)与7S相比向酸性方向偏移,其所含的大部分酶解多肽在酸橙汁中(pH 3.86)都带负电荷且具有较高Zeta电位(绝对值),从而使体系中多肽与多肽之间、多肽与果肉(果胶)之间都因静电排斥作用而不易发生聚集。上述结果表明7S经蛋白酶K酶解改性后酸溶特性提高,在酸橙汁中不易引起絮凝沉淀,使蛋白果汁体系的稳定性明显改善。蛋白酶K的酶切位点广泛、水解作用较强且稳定性高,在食品工业中常被用于降解蛋白生产小分子肽[22]。因此该酶制备的7S酶解产物在酸性饮料中有应用前景。

注:a-BS图谱;b-实物图
图5 添加了不同蛋白样品酸橙汁的Turbiscan BS图谱和实物图
Fig.5 BS spectra and visual appearance of orange juices added with different protein samples
3 结论与讨论
利用生物信息学和计算机科学的进步,本文以完整氨基酸序列已知的大豆7S球蛋白为底物蛋白,选用39种酶切位点已知的蛋白酶在PeptideCutter软件中对7S进行模拟酶切,以生成的目标肽段(pI>pH 6.0或pI a)实验验证表明,模拟酶切筛选出的5种优势蛋白酶(蛋白酶K、胃蛋白酶、嗜热菌蛋白酶、胰凝乳蛋白酶和胰蛋白酶)对7S的酶解程度都较高,都可不同程度提高7S在pH 3.0~4.5下的PS,其中蛋白酶K表现最佳,这说明基于模拟酶切分析预测7S酶解产物的酸溶特性对于筛选优势水解用酶具有很好的指导作用;但由于7S酶解过程的复杂性,模拟酶切预测的DH偏高,且当不同蛋白酶模拟酶切所得目标肽段数量相差不大时,预测结果不够准确。 b)蛋白酶K可将7S中α′、α和β亚基完全降解成小分子肽(分子质量<3 000 Da),所得7S酶解产物带电基团数量增多、水合作用增强,且等电点与对照7S(pI=pH 4.75)相比向酸性方向偏移(pI=pH 2.61),使其在pH 3.0~4.5具有较高的Zeta电位(绝对值),可提供较强的静电排斥效应,可能是其酸溶特性提高的主要原因。 c)7S经蛋白酶K酶解改性后酸溶特性显著提高,在酸橙汁中不易引起絮凝沉淀,使蛋白果汁体系的稳定性明显改善。 综上所述,基于计算机模拟酶切指导7S球蛋白酶解改性,不仅大大提高了水解用酶的筛选效率,而且成功制备了具有优异酸溶特性的7S酶解产物,在酸性饮料中有应用前景。 [1] 李佳笑, 石爱民, 刘红芝, 等.植物蛋白酸性条件下溶解性提高的改性方法及应用研究进展[J].中国油脂, 2019, 44(9):59-65. LI J X, SHI A M, LIU H Z, et al.Progress in modification methods to improve plant protein solubility under acidic conditions and its application[J].China Oils and Fats, 2019, 44(9):59-65. [2] 黄橙子. 酸溶性大豆蛋白的制备及其在酸性饮料中的应用[D].上海:华东师范大学, 2013. HUANG C Z.Preparation of acid soluble soybean protein and its application in acidic drinks[D].Shanghai:East China Normal University, 2013. [3] NISHINARI K, FANG Y, GUO S, et al.Soy proteins:A review on composition, aggregation and emulsification[J].Food Hydrocolloids, 2014, 39: 301-318. [4] 孙雪, 赵晓燕, 朱运平, 等.酶解大豆分离蛋白的功能性及应用研究进展[J].粮食与油脂, 2021, 34(9):14-17. SUN Y, ZHAO X Y, ZHU Y P, et al.Research progress on function and application of enzymatic hydrolysis of soy protein isolate[J].Cereals &Oils, 2021, 34(9):14-17. [5] TAVANO O L.Protein hydrolysis using proteases:An important tool for food biotechnology[J].Journal of Molecular Catalysis B:Enzymatic, 2013, 90:1-11. [6] ZEEB B, MCCLEMENTS D J, WEISS J.Enzyme-based strategies for structuring foods for improved functionality[J].Annual Review of Food Science and Technology, 2017, 8:21-34. [7] 江连洲, 佟晓红, 刘宝华, 等.酶种类对生物解离大豆蛋白酶解物功能性和苦味的影响[J].农业机械学报, 2018, 49(8):368-374. JIANG L Z, TONG X H, LIU B H, et al.Effect of enzymatic species on function and bitterness of enzyme-assisted aqueous extraction soybean protein hydrolysates[J].Transactions of the Chinese Society for Agricultural Machinery, 2018, 49(8):368-374. [8] 周亭屹, 高新昌, 党亚丽, 等.基于生物信息技术的生物活性肽研究进展[J].食品工业科技, 2019, 40(12):335-340. ZHOU T Y, GAO X C, DANG Y L, et al.Research development of the bioactive peptides based on bioinformatics[J].Science and Technology of Food Industry, 2019, 40(12):335-340. [9] GASTEIGER E, HOOGLAND C, GATTIKER A, et al.Protein Identification and Analysis Tools on the ExPASy Server[M].Totowa:Humana Press, 2005. [10] 于志鹏, 樊玥, 赵文竹, 等.鸡蛋蛋白ACE抑制肽的筛选、鉴定及其作用机制[J].食品科学, 2020, 41(12):129-135. YU Z P, FAN Y, ZHAO W Z, et al.Identification and mechanism of action of angiotensin-I converting enzyme inhibitory peptides from hen egg proteins[J].Food Science, 2020, 41(12):129-135. [11] BLEAKLEY S, HAYES M, O’SHEA N, et al.Predicted release and analysis of novel ACE-I, renin, and DPP-IV inhibitory peptides from common oat (Avena sativa) protein hydrolysates using in silico analysis[J].Foods, 2017,6(12):108. [12] 舒文涛, 耿臻, 李金花, 等.大豆7S球蛋白β-伴大豆球蛋白亚基的氨基酸序列分析[J].中国农学通报, 2018, 34(26):48-51. SHU W T, GENG Z, LI J H, et al.Amino acid sequence analysis of 7S globulin (β-conglycinin) of soybean[J].Chinese Agricultural Science Bulletin, 2018, 34(26):48-51. [13] DEAK N A, MURPHY P A, JOHNSON L A.Characterization of fractionated soy proteins produced by a new simplified procedure[J].Journal of the American Oil Chemists′ Society, 2007, 84(2):137-149. [14] ADLER-NISSEN J.Enzymic Hydrolysis of Food Proteins[M].London:Applied Science Publishers, 1986. [15] 陈林, 吴克刚, 柴向华, 等.微射流均质预处理提高大豆分离蛋白酶解效率及酶解产物乳化性能[J].农业工程学报, 2015, 31(5):331-338. CHEN L, WU K G, CHAI X H, et al.Microfluidization pretreatment improving enzymatic hydrolysis of soy isolated protein and emulsifying properties of hydrolysates[J].Transactions of the Chinese Society of Agricultural Engineering, 2015, 31(5):331-338. [16] 姜振峰, 赫卫, 汪洋, 等.大豆种子7S、11S球蛋白及7S球蛋白亚基的研究[J].中国油料作物学报, 2007, 29(2):138-141. JIANG Z F, HE W, WANG Y, et al.Study on 7S, 11S globulin and subunits of 7S globulin of soybean seed[J].Chinese Journal of Oil Crop Sciences, 2007, 29(2):138-141. [17] TSUMURA K, SAITO T, TSUGE K, et al.Functional properties of soy protein hydrolysates obtained by selective proteolysis[J].LWT-Food Science &Technology, 2005, 38(3):255-261. [18] KILARA A, PANYAM D.Peptides from milk proteins and their properties[J].Critical Reviews in Food Science and Nutrition, 2003, 43(6):607-633. [19] DICKINSON E.Hydrocolloids as emulsifiers and emulsion stabilizers[J].Food Hydrocolloids, 2009, 23, 1473-1482. [20] MENGUAL O, MEUNIER G, CAYRÉ I, et al.TURBISCAN MA 2000:Multiple light scattering measurement for concentrated emulsion and suspension instability analysis[J].Talanta, 1999, 50(2):445-456. [21] NASTAJ M, TERPI [22] 吴彤, 王瑞明, 黄磊, 等.蛋白酶K的研究进展[J].食品工业科技, 2013(14):380-384. WU T, WANG R M, HUANG L, et al.Research progress in the Proteinase K[J].Science and Technology of Food Industry, 2013(14):380-384. OWSKI K, SO
OWSKI K, SO OWIEJ B G.The effect of native and polymerised whey protein isolate addition on surface and microstructural properties of processed cheeses and their meltability determined by Turbiscan[J].International Journal of Food Science and Technology, 2020, 55(5):2179-2187.
OWIEJ B G.The effect of native and polymerised whey protein isolate addition on surface and microstructural properties of processed cheeses and their meltability determined by Turbiscan[J].International Journal of Food Science and Technology, 2020, 55(5):2179-2187.