益生菌(probiotics)的概念最早是由来自俄罗斯的诺贝尔奖获得者Elie Metchnikoff提出的,他发现保加利亚农民长寿的原因是食用了大量的发酵乳制品,于是提出肠道微生物对食物具有的依赖性有可能促使我们体内有益的微生物取代有害的微生物[1]。联合国粮农组织和世界卫生组织将益生菌定义为足量摄入后对宿主有益的活性微生物[2]。最常应用的益生菌微生物包括乳酸杆菌属(Lactobacillus)、双歧杆菌属(Bifidobacterium)和酵母菌属(Saccharomyces)的物种,其他属的益生菌包括芽孢杆菌(Bacillus)、丙酸杆菌(Propionibacterium)、链球菌(Streptococcus)和大肠杆菌(Escherichia)等[3]。益生菌具有抑制胃肠道的致病菌、降低血压和高血糖水平、改善肠道健康、降低血清胆固醇水平、降解毒素、产生辅助因子和维生素、调节免疫系统及预防肿瘤和癌症等功能[4]。其主要以个体或菌群的方式向宿主微生物群补充细菌种类、消除致病微生物或改变内源性微生物群的组成来发挥作用[5]。因此,益生菌被广泛应用于食品和膳食补充剂中。最常用的益生菌被认为是安全的,可用作一般健康人群的食品和膳食补充剂的一部分[6]。然而,有研究表明,一些益生菌微生物可能具有潜在的致病因子[7]或在临床上没有显著效果[8],导致益生菌产品的有效性和安全性不断受到质疑。因此,开发出活性稳定、功能优良且安全的益生菌产品成为食品行业的研究热点。
尽管目前发现了许多菌株具有益生特性并已被开发为适用于不同宿主的益生菌补充剂,但仍需要继续寻找比现有益生菌具有更好促进健康特性的新益生菌菌株。传统的微生物育种方法一般是从自然界、宿主或发酵食品中分离筛选菌株,其工作量大、育种周期长且效率低。微生物遗传育种解决了上述问题,它是运用遗传学原理和技术对特定生产目的菌株进行改造,增加其有益新性状,以获得遗传稳定、高产、耐受性优良和低耗的菌种,从而更好地满足工业生产和其他特定需求。为此,本文综述了原生质体融合、基因组重排技术、常压室温等离子体诱变(atmospheric and room temperature plasma,ARTP)技术和基因编辑技术[成簇规律间隔的短回文重复序列相关蛋白(clustered regularly interspaced short palindromic repeats-associated,CRISPR-Cas)技术和碱基编辑技术]等育种方法,并对这些遗传育种方法的发展现状以及在益生菌中的应用展开分析总结,同时讨论了遗传育种的安全性,也对其未来研究的发展趋势进行了展望。
1 益生菌的益生功效及生理特性
在益生菌育种过程中,菌株的益生功效是评价育种效果的重要指标。几十年来,随着人们对健康和功能食品的需求不断增加,科学家和食品药品行业人员广泛地研究了益生菌对人类的影响。目前研究表明,益生菌主要通过调节免疫系统、与肠道微生物群的相互作用、生产有机酸、竞争性排斥、改善屏障功能、制造具有系统效应的小分子物质和酶的产生等发挥益生作用[4]。乳酸杆菌和发酵乳杆菌通过产生抑菌素降低了炎症性肠病患者的炎性细胞因子(TNF-α、NF-кBp65、IL-6)的表达,减轻了炎症性肠病症状[9]。鼠李糖乳杆菌能够降低小鼠卵磷脂引起的食物过敏特异性IgE、IgG1、IgG2a和IgA的水平,使小鼠过敏性症状得到了缓解[10]。乳酸杆菌、双歧杆菌、唾液链球菌和丙酸杆菌亚种可以在回肠末端产生共轭亚油酸(cojugated linoleic acid, CLA),CLA可以被结肠细胞吸收或与结肠细胞相互作用抑制癌症的产生[11]。乳酸杆菌属的许多成员代谢产生的肽聚糖、细胞外多糖、表面蛋白和代谢产物(短链脂肪酸)以及无机多磷酸盐液体可以通过肠-肺轴和增强呼吸道局部黏膜免疫力来改善或治疗宿主的呼吸道疾病[12]。干酪乳杆菌可通过改变单链脂肪酸和烟酰胺的代谢,减轻肾损伤和肾衰竭,对急、慢性肾脏疾病有一定的缓解作用[13]。同时,干酪乳杆菌菌株Shirota对急性肝损伤具有治疗作用,可减轻肝脏和肠道损伤,降低血清γ-谷氨酰转肽酶、总胆汁酸、IL-5、IL-10、G-CSF和RANTES的升高,还可减轻肝脏中视黄醇代谢和过氧化物酶体增殖激活受体(peroxisome proliferator activated receptor, PPAR)信号转导的下调以及丙酮酸代谢途径的上调[14]。此外,益生菌在皮肤护理中的作用也引起了广泛的关注,例如用乳酸菌发酵红参会增加红参中抗氧化活性物质的含量(如尿酸、多酚和黄酮类化合物),对酪氨酸酶活性的抑制作用也得到改善,其毒性也得到降低[15]。表1总结了不同益生菌的益生功效及作用机制。然而,现有的关于有益特性的机制仍有许多是未知的,且已知的机制也未能全部在人类身上得到证实。因此,识别和建立某些负责特定健康益处的精确机制仍然是目前的研究热点。
表1 不同益生菌的益生功效及益生机制
Table 1 Probiotic efficacy and mechanism of different probiotics
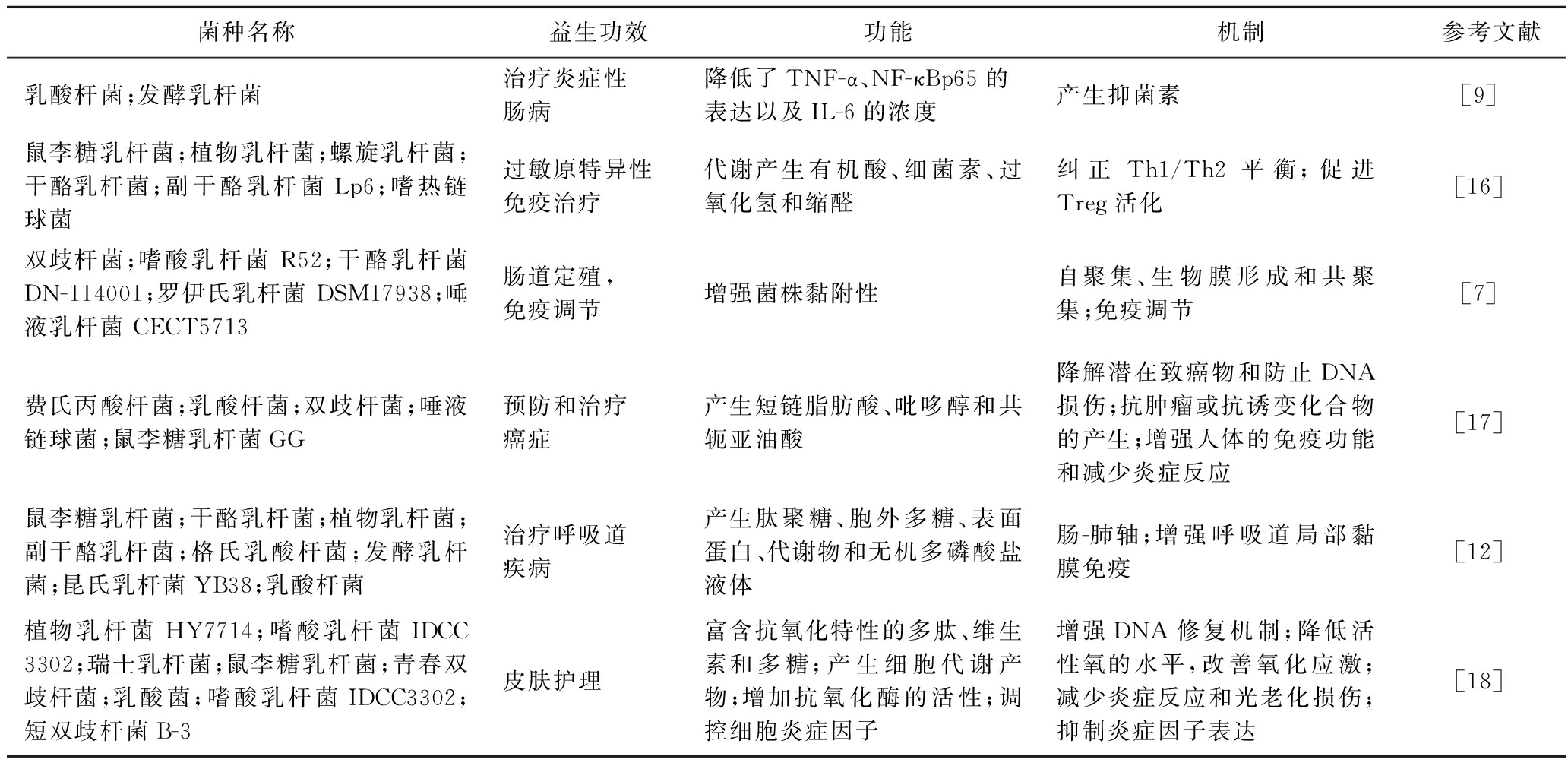
菌种名称益生功效功能机制参考文献乳酸杆菌;发酵乳杆菌治疗炎症性肠病降低了TNF-α、NF-κBp65的表达以及IL-6的浓度产生抑菌素[9]鼠李糖乳杆菌;植物乳杆菌;螺旋乳杆菌;干酪乳杆菌;副干酪乳杆菌Lp6;嗜热链球菌过敏原特异性免疫治疗代谢产生有机酸、细菌素、过氧化氢和缩醛纠正Th1/Th2平衡;促进Treg活化[16]双歧杆菌;嗜酸乳杆菌 R52;干酪乳杆菌 DN-114001;罗伊氏乳杆菌 DSM17938;唾液乳杆菌 CECT5713肠道定殖,免疫调节增强菌株黏附性自聚集、生物膜形成和共聚集;免疫调节[7]费氏丙酸杆菌;乳酸杆菌;双歧杆菌;唾液链球菌;鼠李糖乳杆菌GG预防和治疗癌症产生短链脂肪酸、吡哆醇和共轭亚油酸降解潜在致癌物和防止DNA损伤;抗肿瘤或抗诱变化合物的产生;增强人体的免疫功能和减少炎症反应[17]鼠李糖乳杆菌;干酪乳杆菌;植物乳杆菌;副干酪乳杆菌;格氏乳酸杆菌;发酵乳杆菌;昆氏乳杆菌YB38;乳酸杆菌治疗呼吸道疾病产生肽聚糖、胞外多糖、表面蛋白、代谢物和无机多磷酸盐液体肠-肺轴;增强呼吸道局部黏膜免疫[12]植物乳杆菌HY7714;嗜酸乳杆菌IDCC 3302;瑞士乳杆菌;鼠李糖乳杆菌;青春双歧杆菌;乳酸菌;嗜酸乳杆菌IDCC3302;短双歧杆菌B-3 皮肤护理富含抗氧化特性的多肽、维生素和多糖;产生细胞代谢产物;增加抗氧化酶的活性;调控细胞炎症因子增强DNA修复机制;降低活性氧的水平,改善氧化应激;减少炎症反应和光老化损伤;抑制炎症因子表达[18]
此外,在益生菌选育过程中,菌株生理特性的优化同样尤为重要,并且往往与代谢产物的产量提升相得益彰。进行益生菌生理特性筛选时一般考虑菌株对机体内胃酸、胆盐和消化液的耐受性,这是益生菌存活的基础。其次,菌株的定植和黏附能力决定了其益生作用的发挥。以提高乳酸菌在宿主肠道中的黏附力为例,植物杆菌HE-1产生的AI-2信号分子可以增强生物膜的形成,并最终增加对肠道上皮细胞的潜在黏附性[19]。luxS基因的表达水平可以影响乳酸菌的细菌素合成、耐受性和黏附特性,luxS基因的表达水平与特性效应呈正相关关系。到目前为止,luxS基因介导的乳酸菌研究内容主要包括细菌素特性、抗逆性和黏附能力3个方面,未来可以以此进行菌种选育。
2 益生菌育种技术
2.1 原生质体融合
原生质体融合技术是通过组合来自不同菌株或物种的有益等位基因来产生具有优良表型的微生物[20]。具有克服远缘杂交、提升优良性状、操作简单和应用范围广等优点[21]。原生质体融合技术已经成功应用于酵母菌、乳杆菌类、双歧杆菌类、芽孢杆菌类和链球菌类中。当所选用的菌株的基因组未被表征时,很难利用基因工程手段提高其益生性能和生理特性。而原生质体融合技术能使遗传基因高频率重组,达到高效育种的目的。比如德氏乳杆菌突变体和解淀粉芽孢杆菌通过融合将淀粉转化率增加到了96%[22];提高了嗜酸乳杆菌耐酸和耐胆盐能力[23];双歧杆菌和酿酒酵母融合得到了既具有双歧杆菌的特异性序列又具有亲本菌的生物学特性的菌株[24]。同时,嗜酸乳杆菌和地衣芽孢杆菌的融合为产芽孢乳酸菌的育种和改良提供新的思路,筛选得到了既能形成芽孢又可产蛋白酶和淀粉酶的乳酸菌[25]。此外,近期也有利用原生质体融合技术进行细菌重组动力学的研究,在将不同遗传距离的芽孢杆菌菌株进行杂交后,发现物种水平的遗传距离不影响全基因组重组[20],这将有助于更好地理解自然系统中的细菌进化,为改善细胞功能提供潜在的见解。
原生质体融合是一种简单、高效的遗传育种技术,但也存在融合效率不高的问题。同时,在进行远源物种间的原生质体融合时,可能会出现严重的不亲和性和排斥性,导致细胞无法融合[26]。因此,寻找高效的原生质体融合方法具有重要意义。
2.2 基因组重排
基因组重排是经过多轮递推的原生质体融合技术,主要基于原生质体融合。它允许多个亲本之间的重组,可获得具有多种有益突变的菌株[27]。此外,基因组重排可以突破种甚至属的限制,加速重要微生物的定向进化,且不需要全面遗传背景或代谢网络信息[22,28]。最重要的是,基于原生质体融合的自然同源重组,基因组重排获得的融合体不被视为“转基因”,克服了转基因生物应用的巨大障碍[27,29]。作为一种实用高效的育种技术,其在微生物菌种改良及代谢产物开发和产业化等研究领域得到了广泛应用[30]。
基因组重排的效率取决于原生质体的产生和融合效率、融合后原生质体的回收效率、融合后获得的二倍体或多倍体细胞中异质染色体之间的重组效率以及仅含一个基因组的真正目标生物的最终分离效率[31]。通过基因组重排技术进行菌种选育的关键是:构建具有基因和表型多样性的亲本文库、原生质体的高效制备、原生质体的理性融合和基因组重排突变菌的高效筛选和验证(图1)[30,32]。基因组重排已经广泛应用于各种乳酸菌的遗传育种中。基因组重排有效地规避了微生物基因工程改造所有的必需条件,对许多特殊种类或无法进行遗传操作的益生菌而言,仍是最有效的菌种选育方法。比如增强鼠李糖乳杆菌ATCC11443的耐酸性和乳酸产量[33]及植物乳杆菌C88的黏附性[34],提高乳酸片球菌对肠道沙门氏菌的抗菌活性[35],增加解淀粉芽孢杆菌ES-2-4的抗菌肽芬荠素产量[36],在乳酸乳球菌YF11中提高乳酸链球菌素的产量[37]。此外,目前鲜有在双歧杆菌属中应用基因组重排技术的报道。未来可在双歧杆菌属中尝试使用基因组重排技术进行遗传育种。基因组重排主要通过基因转换引起基因组保守区域的单点突变和基因重复[28]。有研究表明,菌株通过基因组重排增强的耐受性主要是由于细胞倍性调节和应激反应基因表达水平的变化[38]。如在酿酒酵母中发现泛素特异性蛋白酶基因UBP7的逆向突变有效地提高了菌株对亚硫酸盐废液的耐受性,RNA-seq分析确定其与应激反应转录抑制因子(neuregulin 1, Nrg1p)以及依赖NADPH的谷氨酸脱氢酶(glutamate derydrogenase 1, Gdh1p)有关[39]。另外,基因组重排所导致的益生菌有益性能提升不仅受到其代谢网络全局变化的调控,还涉及到许多关键生物过程和应激反应过程的交叉影响[30]。如酿酒酵母中谷胱甘肽的产量的提升涉及到mRNA转录和合成酶GSH-I的过表达,以及蛋白质合成、DNA合成和氨基酸转运[40]。
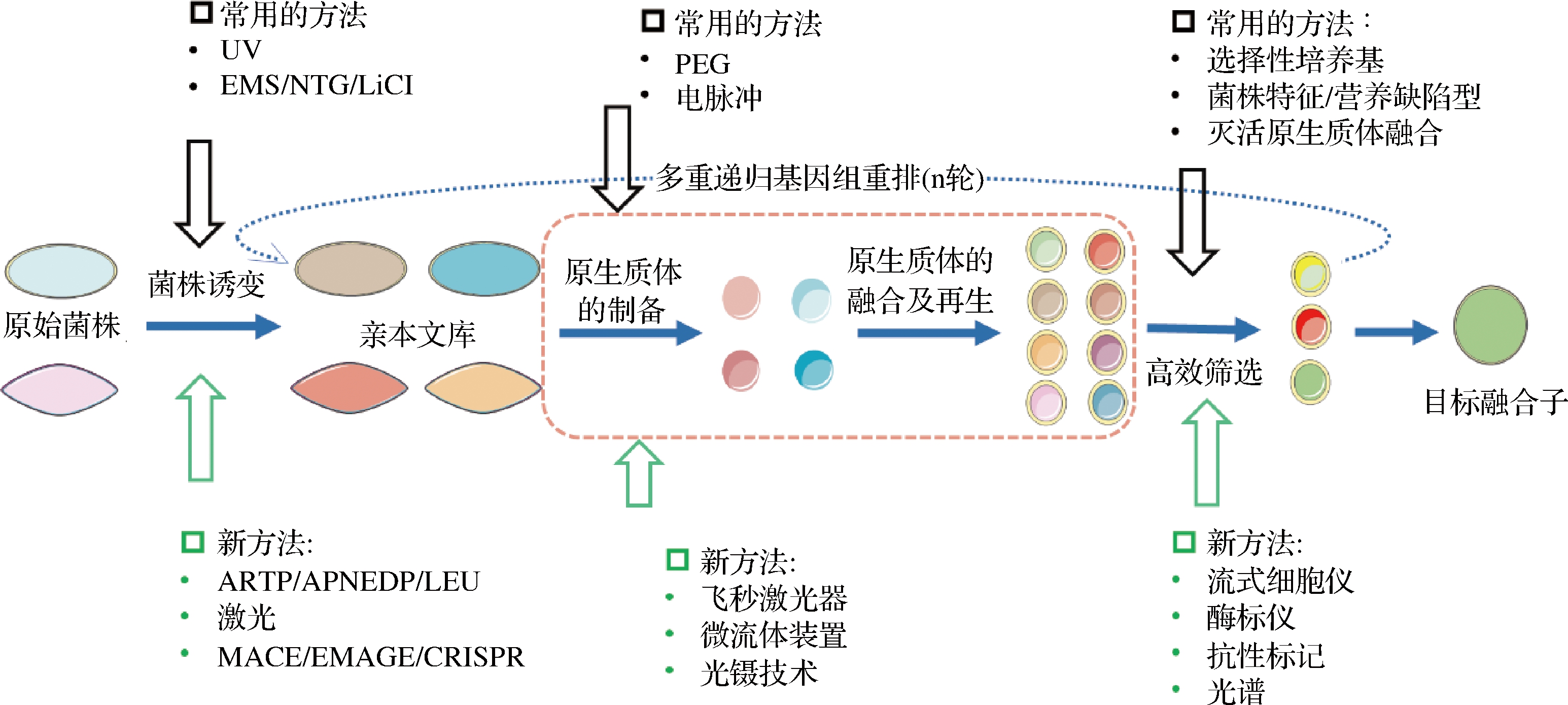
图1 基因组重排的技术流程概括[30,32]
Fig.1 The general process of genome shuffling[30,32]
基因组重排技术的难点是如何实现融合子的高通量筛选,尤其是在融合子重组片段是随机组合的情况下。目前也有研究报道了一些操作简单、高效的高通量筛选方法,但无法广泛应用。因此,建立高通量筛选方法仍是今后益生菌育种工作中的研究热点。此外,随着组学和生物信息学的快速发展,学者可以联合运用多种组学技术,对基因组重排突变菌及其亲本进行全面分析来揭示其不同表型的遗传基因和物质基础之间的相互作用关系,同时选择合适的生物信息学工具将多组学实验的结果联系起来评估菌株性能[30]。
2.3 常压室温等离子体诱变
近年来兴起的ARTP技术是基于等离子体的带电粒子、自由基、激发态中性物质、高电场、紫外辐射等多种诱变剂对微生物进行诱变的一种新型高效的育种技术[41]。ARTP技术具有高效的诱变性能、适用范围广、操作条件温和、遗传物质损伤机制多样和安全性高等优点,已被广泛应用于微生物育种中[42]。
用于微生物诱变的两种典型等离子体分别为大气介质阻挡放电(dielectric barrier discharge, DBD)等离子体和大气压射频辉光放电(radio frequency atmospheric pressure glow discharge, RF APGD)等离子体。RF APGD等离子体射流可以被用来改变生物体的基因序列,这意味着温和而均匀的RF APGD等离子体技术是一种有前途的新型微生物突变工具[43]。WANG等[44]首次报道了利用ARTP技术诱变阿维链霉菌(Streptomyces avermitilis)以产生具有高产阿维菌素B1a的突变体的成功范例。随后,这种ARTP方法引起了科研人员和企业的广泛关注,加速了ARTP技术在益生菌育种中的应用,如表2所示。ARTP将蜡样芽孢杆菌的壳聚糖酶活性提高到原菌的2.49倍[45];在罗伊氏乳杆菌中对大肠杆菌的抑菌率提高了10.64%,对沙门氏菌抑菌率提高了9.42%,对金黄色葡萄球菌抑菌率提高了9.20%[49];增加了嗜酸乳杆菌的耐酸性[51]。ARTP能够通过增加胞外多糖水平和细胞膜内K+浓度提高细菌的耐受性[52],且能够通过改变微生物的整体代谢[53]和细胞内ATP产量[54]来提高细菌的有益性能。如乳酸乳球菌主要涉及到嘌呤代谢、缬氨酸、亮氨酸和异亮氨酸生物合成代谢、ABC转运系统代谢通路,可通过群体感应机制诱导细菌素的产生,提升其抑菌活性[55]。大肠杆菌经ARTP后在葡萄糖特异性磷酸转移酶系统、丙酮酸盐裂解酶系统和发酵乳酸脱氢酶系统中发生突变,减少了副产物的形成并增加了琥珀酸和ATP的产量[54]。目前,ARTP技术的应用在乳酸菌属中的报道较多,而在双歧杆菌属的报道鲜少。在今后的工作中,可将等离子体物理学与基因组学及其他组学分析相结合在益生菌育种中开展科学研究。
表2 使用ARTP诱变的益生菌突变案例列表
Table 2 List of the probiotics mutation cases using the ARTP

益生菌正突变率/%突变结果参考文献蜡样芽孢杆菌将突变菌株的壳聚糖酶活性提高到原菌的2.49倍[45]副干酪乳杆菌62获得的突变菌株BJT-7经全脂牛乳发酵在20 d贮藏期间酸度低于70 °T,遗传稳定性良好[46]酿酒酵母28.6香豆酸的最高产量是野生型菌株的7.6倍[47]凝结芽孢杆菌与原菌株相比,改善了凝乳酶的益生性能,并显示出对pH 2.5和0.3%胆汁盐的耐受性[48]罗伊氏乳杆菌突变菌株对大肠杆菌的抑菌率提高了10.64%,对沙门氏菌抑菌率提高了9.42%,对金黄色葡萄球菌抑菌率提高了9.20%[49]唾液乳杆菌66.67筛选出的突变菌株30s-37抑菌效率提高了5%,且遗传稳定性较好[50]嗜酸乳杆菌可有效提高嗜酸乳杆菌的耐酸性(pH 2.5)[51]
到目前为止,还没有能够定量描述常压室温的等离子体作用剂量的有用变量。因此,评估和比较由不同方法产生的不同等离子体的作用强度,用于处理具有不同作用模式的生物材料,仍然非常困难。
2.4 基因编辑技术
近年来,基因组测序的价格越来越低,一些编辑和修改微生物基因组的工具也越来越强大,使我们能够按照自己的想法来设计益生菌,从而开发出个性化的益生菌[56]。
近几十年在细菌中应用的CRISPR-Cas技术预示益生菌菌株开发的有利机会。目前展示的优势是可以对顽固菌株进行工程化处理、极其有效的反选择系统、用于基因调控、减少基因组操作时间以及可以进行多重基因组编辑和调控[57]。CRISPR-Cas系统一般按CRISPR相邻的Cas蛋白基因分类。目前报道的CRISPR-Cas系统主要分为两大类、6种类型和33种亚型[58]。两类Cas蛋白家族中的核酸酶Cas9和Cas12a(Cpf1)应用较为广泛,易于编辑识别特定的DNA序列[59]。其中,Ⅱ型CRISPR-Cas9系统是目前比较常用的基因组编辑蛋白[60]。通过人工设计,crRNA和tracrRNA可以转化为小向导RNA,引导Cas9进行定点的DNA切割、基因敲除和插入[61]。CRISPR技术已经在大肠杆菌、葡萄球菌、乳酸菌、双歧杆菌等益生菌上实施[62](表3)。BERLEC等[63]开发了一个单质粒诱导型CRISPR-Cas9系统(pNZCRISPR),使CRISPR干扰介导的UPP基因沉默,用于乳酸菌的高级遗传改造。ZHOU等[66]使用CRISPR-Cas9辅助的双链DNA和单链DNA重组技术,在植物乳杆菌中进行了无缝基因组编辑,使植物乳杆菌WCFS1在不引入外源基因或质粒的情况下,通过增强GlcNAc途径产生了797.3 mg/L N-乙酰氨基葡萄糖。PAN等[72]利用内源性I-G型CRISPR-Cas系统,并采用外源性CRISPR碱基编辑技术用于双歧杆菌的基因组工程,通过在tetW中产生一个500 bp的缺失,消除了双歧杆菌的四环素抗性。CRISPR技术不仅可用于功能缺失的研究,如CRISPR敲除(CRISPRKO)和CRISPR干扰或抑制(CRISPRi),还可用于功能增益的筛选研究,如CRISPR激活(CRISPRa)[62,73]。基因编辑技术特别是CRISPR的发展为下一代益生菌的出现提供了必要的支持。
表3 CRISPR技术在益生菌中的应用
Table 3 Application of CRISPR technology in probiotics

益生菌目标靶点功能方式参考文献乳酸菌upp构建了乳链菌肽启动子使得两种模型蛋白在乳酸链球菌中平衡、诱导表达CRISPR干扰[63]乳酸菌NZ9000NICE构建了一个新的基因抑制工具,使菌株基因表达显着降低,高达99%;并鉴定出LLNZ_07335为乳酸菌NZ9000胆盐耐受功能基因CRISPR干扰[64]罗伊氏乳杆菌ATCC PTA6475RecT成功地在罗伊氏乳杆菌染色体中应用了密码子饱和诱变,可识别重组效率低的细菌中的重组细胞CRISPR抑制[65]植物乳杆菌WCFS1nagB通过增强菌株GlcNAc途径产生了797.3 mg/L N-乙酰氨基葡萄糖,且不引入外源基因或质粒CRISPR抑制[66]植物乳杆菌AR113bsh 1, bsh 3高效筛选对胆汁盐具有高耐受性的菌株CRISPR敲除[67]植物乳杆菌dnaA, EloR, KhpA初步了解植物乳杆菌关键细胞周期基因的功能及3个细胞分裂基因的表型CRISPR干扰[68]大肠杆菌Shv-18, ndm-1基于基因特征选择性消除目标菌株来调控复杂的细菌群体CRISPR抗菌剂[69]SoxS, MarA, Rob, RFP可编程控制多个基因同时激活和抑制CRISPR激活/干扰[70]葡萄球菌属Sek, mecA, aph3开发了可编程的序列特异性抗菌剂,以抗生素抗性基因为目标,可以破坏含有抗生素抗性基因的葡萄球菌质粒,并对无病毒的葡萄球菌进行免疫以防止质粒携带的抗性基因的传播CRISPR抗菌剂[71]双歧杆菌tetW消除了菌株的四环素抗性CRISPR抑制[72]
此外,基于CRISPR-Cas9技术发展而来的碱基编辑技术具有高效、安全、副产物少等显著优势,但是存在编辑窗口大小和精确度有限、脱靶效应和细胞毒性等问题[74]。碱基编辑技术主要分为基础编辑(base editors,BEs)和主要编辑(rime editors,PEs)2种。BEs是一种在单碱基分辨率下精确修改活细胞的基因组(DNA)或转录组(RNA)的新技术。碱基编辑是由催化受损的Cas核酸酶组成,该核酸酶与核苷酸脱氨酶融合,有时还与DNA修复蛋白融合。目前可用的DNA碱基编辑可以进一步分类为胞嘧啶BEs(CBEs)和腺嘌呤BEs(ABEs)[75]。CBEs和ABEs可以分别实现C到T(互补链中的G到A)和A到G(互补链中的T到C)突变。总的来说,CBEs和ABEs使我们能够实现12个可能的碱基取代中的4个,并且只催化碱基转换(嘧啶到嘧啶,嘌呤到嘌呤)。尽管BEs在引入高效的点突变方面很强大,但它们不能产生精确的插入缺失,也很难避免在其他方面的突变。相比之下,PEs可以引入所有12种可能的转换或过渡性转换突变和小缺失,以及它们的组合,并有利于对插入缺失比率进行预期编辑[76]。PEs是多功能、精确的基因组编辑工具,使用与工程逆转录酶融合的Cas9切口酶能够直接将新的遗传信息写入指定的DNA靶位点[76]。碱基编辑技术已广泛应用于植物育种、家畜育种和人类疾病治疗等方面,但是在益生菌育种中的应用鲜有报道。随着碱基编辑技术的不断优化,其编辑精确度和安全性正逐渐提高,有望广泛应用于益生菌基因编辑育种中。
通过基因编辑能够将现有的益生菌修改成所需的新益生菌,这种工程改造使我们能够直接验证这些新型微生物的遗传物质、蛋白质和功能作用是否已经发生了预期的变化。基因编辑技术的进步为益生菌的基因编辑提供了新的可能性。然而,工程益生菌的应用仍然面临着一系列的挑战。例如,许多微生物可操作的遗传工具很少,这导致益生菌的改造仅限于少数菌株。尽管工程益生菌的研究和应用存在这些局限性,但我们不能忽视它们在改善人类健康方面的潜在作用。此外,工程益生菌可能成为治疗癌症、炎症、感染和其他一些疾病的新方法。
3 益生菌遗传育种安全性
虽然益生菌的使用已经在普通大众中得到了普及,但是随着研究的不断深入,益生菌产品的安全性不断受到质疑。因此,需要对使用遗传育种的方法选育得到的益生菌菌株的安全性做出客观且全面的评估。为保证选育的益生菌菌种安全,首先需要在分类学上对益生菌菌株进行准确鉴定,其次需要确定菌种的功能特性(如安全性、抗逆性、定植性、稳定性、有效性等)[77]。针对益生菌的不同特性,粮农组织和世界卫生组织提出了多种益生菌菌株安全性评价方法,如抗生素抗性、溶血性测定、产毒能力测定和毒力因子测定等[77]。迄今为止还没有在乳酸菌[78]或双歧杆菌[79]中发现含有与致病菌相似的毒性因子。此外,由于乳酸菌安全使用的历史悠久,被美国食品药品监督管理局和欧洲食品安全局分别列为公认安全和安全合理推定[80]。因此,给予一些乳酸菌物种一般是安全的。
原生质体融合和基因组重排技术可以实现跨物种融合,一些含有抗生素抗性基因的菌株可能会将该基因转移至所选育的菌株中,可能会引发菌血症[81];此外,一些毒力基因可能会保留在所选育的菌株中,导致益生菌的安全性问题。如一些乳杆菌能够同时产生L-乳酸和D-乳酸,而D-乳酸的大量产生会导致特定高危人群(如儿童)发生短肠综合症[82],因此将能够产生D-乳酸的益生菌用于婴幼儿配方奶粉中时,其安全性需引起人们的重视。安全应用益生菌微生物的主要风险因素是对其活性缺乏了解。充分了解益生菌的结构和功能之间的关系将减少选育所带来的副作用[80]。常压室温等离子体诱变属于物理诱变,具有很高的安全性。值得注意的是,基因编辑技术筛选得到的菌株属于转基因生物范畴,它的应用在大多数国家受到严格地管制。美国食品药品监督管理局已有转基因益生菌被批准用于食品和药物,如布拉迪酵母、动物双歧杆菌Bb-12、短双歧杆菌M-16V等;澳新食品标准局批准转基因大肠杆菌K-12生产的2′-FL可用于婴儿配方食品。而欧盟目前限制使用经外源基因改造的益生菌;我国也尚未有批准转基因益生菌用于食品和药物的相关文件。以益生菌为基础的食品、保健品和药品尚无明确区分标准,不同国家之间也存在较大差异。因此,监管、立法和技术方面的复杂性成为市场增长的主要障碍[83]。另外,转基因益生菌经过灭活处理也能发挥作用[84-85],经过灭活处理的转基因益生菌不属于转基因食品,也就不受到国家法规的约束,并且容易使公众接受。肠道微生物和宿主之间的相互作用对宿主整体健康状况有重大影响。因此,在人类使用益生菌之前,应该充分了解益生菌的活动机制,还应该考虑到单一菌株或不同物种甚至属的混合菌株的活性差异[86]。
4 总结与展望
随着人们对健康和功能食品的需求不断增加,益生菌的益生特性将促使其市场价值持续增加。很显然,单一的育种方法已经很难满足益生菌的高效选育,未来应更多的联合使用多种不同育种方法或开发新型高效的育种方法来增加益生菌的突变效率。此外,遗传育种获得的菌株可能会存在潜在的安全性问题。尽管通常销售的益生菌具有良好的安全记录,但对于用于高危人群的益生菌可能存在潜在的风险[87]。由于益生菌是活的微生物,因此可能需要开发特殊的方法来验证产品安全性。
遗传育种的方法主要集中在已知的基因和已知的突变上,这些方法错过了发展优良生产特性所涉及的许多未知因素,如宿主的功能或引入的基因的相互作用。因此,它们也需要与一系列其他技术相结合来对菌株进行选育,如下一代测序、生物信息学和高通量表型等。此外,需要通过基因组学、转录组学、蛋白质组学、代谢组学和通量组学快速分析尚未完全了解其特性的菌株,以便对菌株进行全面的功能表征,并对代谢和调控网络提出新见解。
[1] SHOKRYAZDAN P, FASELEH JAHROMI M, LIANG J B, et al.Probiotics:From isolation to application[J].Journal of the American College of Nutrition, 2017, 36(8):666-676.
[2] HILL C, GUARNER F, REID G, et al.The international scientific association for probiotics and prebiotics consensus statement on the scope and appropriate use of the term probiotic[J].Nature Reviews Gastroenterology &Hepatology, 2014, 11(8):506-514.
[3] DAVID L A, MAURICE C F, CARMODY R N, et al.Diet rapidly and reproducibly alters the human gut microbiome[J].Nature, 2014, 505(7484):559-563.
[4] IDREES M, IMRAN M, ATIQ N, et al.Probiotics, their action modality and the use of multi-omics in metamorphosis of commensal microbiota into target-based probiotics[J].Frontiers in Nutrition, 2022, 9:959941.
[5] MIMEE M, CITORIK R J, LU T K.Microbiome therapeutics-advances and challenges[J].Advanced Drug Delivery Reviews, 2016, 105:44-54.
[6] SAKANDAR H A, ZHANG H P.Trends in probiotic(s)-fermented milks and their in vivo functionality:A review[J].Trends in Food Science &Technology, 2021, 110:55-65.
[7] SUEZ J, ZMORA N, SEGAL E, et al.The pros, cons, and many unknowns of probiotics[J].Nature Medicine, 2019, 25(5):716-729.
[8] GORE C, CUSTOVIC A, TANNOCK G W, et al.Treatment and secondary prevention effects of the probiotics Lactobacillus paracasei or Bifidobacterium lactis on early infant eczema:Randomized controlled trial with follow-up until age 3 years[J].Clinical and Experimental Allergy, 2012, 42(1):112-122.
[9] HEGAZY S K, EI-BEDEWY M M.Effect of probiotics on pro-inflammatory cytokines and NF-kappa B activation in ulcerative colitis[J].World Journal of Gastroenterology, 2010, 16(33):4145-4151.
[10] KIM B G, KIM J N, JANG A S, et al.Combined effects of Lactobacillus rhamnosus and egg oral immunotherapy in a mouse model of egg allergy[J].Allergy Asthma &Immunology Research, 2020, 12(4):701-711.
[11] ![]() K,
K, ![]() W.The role of probiotics in cancer prevention[J].Cancers, 2021, 13(1):20.
W.The role of probiotics in cancer prevention[J].Cancers, 2021, 13(1):20.
[12] DU T F, LEI A H, ZHANG N Y, et al.The beneficial role of probiotic Lactobacillus in respiratory diseases[J].Frontiers in Immunology, 2022, 13:908010.
[13] ZHU H, CAO C J, WU Z C, et al.The probiotic L.casei Zhang slows the progression of acute and chronic kidney disease[J].Cell Metabolism, 2021, 33(10):2091-2093.
[14] YAN R K, WANG K C, WANG Q Q, et al.Probiotic Lactobacillus casei Shirota prevents acute liver injury by reshaping the gut microbiota to alleviate excessive inflammation and metabolic disorders[J].Microbial Biotechnology, 2022, 15(1):247-261.
[15] LEE H S, KIM M R, PARK Y, et al.Fermenting red ginseng enhances its safety and efficacy as a novel skin care anti-aging ingredient:In vitro and animal study[J].Journal of Medicinal Food, 2012, 15(11):1015-1023.
[16] CAO H, GUAN L, LIU X Y, et al.Adjuvant role of probiotics in allergen-specific immunotherapy[J].Clinical Immunology, 2022, 245:109164.
[17] LU K, DONG S W, WU X Y, et al.Probiotics in cancer[J].Frontiers in Oncology, 2021, 11:638148.
[18] YU J Y, MA X M, WANG X Y, et al.Application and mechanism of probiotics in skin care:A review[J].Journal of Cosmetic Dermatology, 2022, 21(3):886-894.
[19] BUCK B L, AZCARATE-PERIL M A, KLAENHAMMER T R.Role of autoinducer-2 on the adhesion ability of Lactobacillus acidophilus[J].Journal of Applied Microbiology, 2009, 107(1):269-279.
[20] VASILEVA D P, STREICH J C, BURDICK L H, et al.Protoplast fusion in Bacillus species produces frequent, unbiased, genome-wide homologous recombination[J].Nucleic Acids Research, 2022, 50(11):6211-6223.
[21] 李亚娇, 孙国琴, 郭九峰, 等.原生质体融合技术在食用菌上应用研究进展[J].北方农业学报, 2021, 49(6):121-127.
LI Y J, SUN G Q, GUO J F, et al.Research progress on the protoplast fusion technique in edible mushroom[J].Journal of Northern Agriculture, 2021, 49(6):121-127.
[22] JOHN R P, GANGADHARAN D, MADHAVAN NAMPOOTHIRI K.Genome shuffling of Lactobacillus delbrueckii mutant and Bacillus amyloliquefaciens through protoplasmic fusion for L-lactic acid production from starchy wastes[J].Bioresource Technology, 2008, 99(17):8008-8015.
[23] 王玉华, 张桂荣, 刘景圣.原生质体融合提高嗜酸乳杆菌耐酸及耐胆盐能力[J].食品科学, 2006,27(3):96-99.
WANG Y H, ZHANG G R, LIU J S.Improvement study on acid and bile tolerance of Lactobacillus acidophilus protoplast fusion[J].Food Science, 2006, 27(3):96-99.
[24] 黎永学, 张德纯, 李代昆.双歧杆菌和酿酒酵母原生质体融合子筛选方法的探讨[J].食品科学, 2006(2):84-86.
LI Y X, ZHANG D C, LI D K.Screening of fusants derived from protoplast fusion of Bifidobacterium with Saccharomyces cerevisiae[J].Food Science, 2006(2):84-86.
[25] 高媛惠, 马林峰, 霍乃蕊.嗜酸乳杆菌-地衣芽孢杆菌融合子的制备工艺优化[J].山西农业科学, 2021, 49(2):121-125.
GAO Y H, MA L F, HUO N R.Optimization of the technique for the preparation of the fusant of Lactobacillus acidophilus and Bacillus licheniformis[J].Journal of Shanxi Agricultural Sciences, 2021, 49(2):121-125.
[26] 刘敏跃, 李鹏, 龙淼.微生物原生质体融合育种技术及其应用研究进展[J].中国饲料, 2014(7):17-21.
LIU M Y, LI P, LONG M.Research advances in microbial protoplast fusion technology in genetic breeding and its application[J].China Feed, 2014(7):17-21.
[27] GONG J X, ZHENG H J, WU Z J, et al.Genome shuffling:Progress and applications for phenotype improvement[J].Biotechnology Advances, 2009, 27(6):996-1005.
[28] LUNA-FLORES C H, PALFREYMAN R W, KRÖMER J O, et al.Improved production of propionic acid using genome shuffling[J].Biotechnology Journal, 2017, 12(2):1600120.
[29] ZHANG Y X, PERRY K, VINCI V A, et al.Genome shuffling leads to rapid phenotypic improvement in bacteria[J].Nature, 2002, 415(6872):644-646.
[30] 蒋承州, 黄勇, 段燕文, 等.后基因组时代基因组重排在微生物菌种选育中的应用及展望[J].微生物学通报, 2018, 45(11):2494-2502.
JIANG C Z, HUANG Y, DUAN Y W, et al.Progress in genome shuffling in the post genomic era for microbial strains improvement[J].Microbiology China, 2018, 45(11):2494-2502.
[31] DAI M H, ZIESMAN S, COPLEY S D, et al.Visualization of protoplast fusion and quantitation of recombination in fused protoplasts of auxotrophic strains of Escherichia coli[J].Metabolic Engineering, 2005, 7(1):45-52.
[32] BIOT-PELLETIER D, MARTIN V J J.Evolutionary engineering by genome shuffling[J].Applied Microbiology and Biotechnology, 2014, 98(9):3877-3887.
[33] WANG Y H, LI Y, PEI X L, et al.Genome-shuffing improved acid tolerance and L-lactic acid volumetric productivity in Lactobacillus rhamnosus[J].Journal of Biotechnology, 2007, 129(3):510-515.
[34] ZHAO Y J, DUAN C C, GAO L, et al.Genome-shuffling of Lactobacillus plantarum C88 improves adhesion[J].Bioscience Biotechnology and Biochemistry, 2017, 81(1):184-193.
[35] HAN G G, SONG A A, KIM E B, et al.Improved antimicrobial activity of Pediococcus acidilactici against Salmonella Gallinarum by UV mutagenesis and genome shuffling[J].Applied Microbiology &Biotechnology, 2017, 101(13):5353-5363.
[36] ZHAO J F, ZHANG C, LU J, et al.Enhancement of fengycin production in Bacillus amyloliquefaciens by genome shuffling and relative gene expression analysis using RT-PCR[J].Canadian Journal of Microbiology, 2016, 62(5):431-436.
[37] ZHANG Y F, LIU S Y, DU Y H, et al.Genome shuffling of Lactococcus lactis subspecies lactis YF11 for improving nisin Z production and comparative analysis[J].Journal of Dairy Science, 2014, 97(5):2528-2541.
[38] ZHENG D Q, CHEN J, ZHANG K, et al.Genomic structural variations contribute to trait improvement during whole-genome shuffling of yeast[J].Applied Microbiology and Biotechnology, 2014, 98(7):3059-3070.
[39] PINEL D, COLATRIANO D, JIANG H, et al.Deconstructing the genetic basis of spent sulphite liquor tolerance using deep sequencing of genome-shuffled yeast[J].Biotechnology for Biofuels, 2015, 8(1):1-16.
[40] YIN H, MA Y L, DENG Y, et al.Genome shuffling of Saccharomyces cerevisiae for enhanced glutathione yield and relative gene expression analysis using fluorescent quantitation reverse transcription polymerase chain reaction[J].Journal of Microbiological Methods, 2016, 127:188-192.
[41] ZHANG X, ZHANG X F, LI H P, et al.Atmospheric and room temperature plasma (ARTP) as a new powerful mutagenesis tool[J].Applied Microbiology and Biotechnology, 2014, 98(12):5387-5396.
[42] 唐晨旻, 张劲松, 刘艳芳, 等.常压室温等离子体诱变育种与微生物液滴培养筛选技术应用进展[J].微生物学通报, 2022, 49(3):1177-1194.
TANG C M, ZHANG J S, LIU Y F, et al.Application progress of atmospheric and room temperature plasma mutation breeding and microbial microdroplet culture screening technology[J].Microbiology China, 2022, 49(3):1177-1194.
[43] LI H P, WANG Z B, GE N, et al.Studies on the physical characteristics of the radio-frequency atmospheric-pressure glow discharge plasmas for the genome mutation of Methylosinus trichosporium[J].IEEE Transactions on Plasma Science, 2012, 40(11):2853-2860.
[44] WANG L Y, HUANG Z L, LI G, et al.Novel mutation breeding method for Streptomyces avermitilis using an atmospheric pressure glow discharge plasma[J].Journal of Applied Microbiology, 2010, 108(3):851-858.
[45] ZHANG C, LI Y, ZHANG T, et al.Increasing chitosanase production in Bacillus cereus by a novel mutagenesis and screen method[J].Bioengineered, 2021, 12(1):266-277.
[46] 周虹瑾, 霍向东, 赵丹, 等.常压室温等离子体诱变选育抗后酸化的乳酸菌[J].食品与发酵工业, 2021, 47(23):112-117.
ZHUO H J, HUO X D, ZHAO D, et al.Screening of lactic acid bacteria with anti-postacidification by atmospheric and room temperature plasma mutagenesis[J].Food and Fermentation Industries, 2021, 47(23):112-117.
[47] CAI M A, WU Y Z, QI H, et al.Improving the level of the tyrosine biosynthesis pathway in Saccharomyces cerevisiae through HTZ1 knockout and atmospheric and room temperature plasma (ARTP) mutagenesis[J].ACS Synthetic Biology, 2021, 10(1):49-62.
[48] LIU K Y, FANG H, CUI F J, et al.ARTP mutation and adaptive laboratory evolution improve probiotic performance of Bacillus coagulans[J].Applied Microbiology and Biotechnology, 2020, 104(14):6363-6373.
[49] 黄增颖. 鸭源罗伊氏乳杆菌的筛选与ARTP诱变及在番鸭上的初步应用研究[D].佛山科学技术学院, 2020.
HUANG Z Y.Screening and ARTP mutagenesis of Lactobacillus reuteri strain and its application in muscovy duck[D].Foshan University, 2020.
[50] 周荷田. 唾液乳杆菌的筛选、常压室温等离子体诱变及其初步应用[D].江西农业大学, 2021.
ZHUO H T.Screening, ARTP mutageneis of Lactobacillus salivary and its preliminary application[D].Jiangxi Agricultural University, 2021.
[51] NYABAKO B A, FANG H, CUI F J, et al.Enhanced acid tolerance in Lactobacillus acidophilus by atmospheric and room temperature plasma (ARTP) coupled with adaptive laboratory evolution (ALE)[J].Applied Biochemistry and Biotechnology, 2020, 191(4):1499-1514.
[52] HUA X F, WANG J, WU Z J, et al.A salt tolerant Enterobacter cloacae mutant for bioaugmentation of petroleum- and salt-contaminated soil[J].Biochemical Engineering Journal, 2010, 49(2):201-206.
[53] LU Y, WANG L Y, MA K, et al.Characteristics of hydrogen production of an Enterobacter aerogenes mutant generated by a new atmospheric and room temperature plasma (ARTP)[J].Biochemical Engineering Journal, 2011, 55(1):17-22.
[54] JIANG M, WAN Q, LIU R, et al.Succinic acid production from corn stalk hydrolysate in an E.coli mutant generated by atmospheric and room-temperature plasmas and metabolic evolution strategies[J].Journal of Industrial Microbiology &Biotechnology, 2014, 41(1):115-123.
[55] 李艳洁. 拮抗嗜水气单胞菌的乳酸菌分离鉴定及诱变菌株筛选[D].湖州师范学院, 2022.
LI Y J.Isolation and identification of lactic acid bacteria against Aeromonas hydrophila and screening of mutagenesis strains[D].Huzhou University, 2022.
[56] O’TOOLE P W, MARCHESI J R, HILL C.Next-generation probiotics:The spectrum from probiotics to live biotherapeutics[J].NAture Microbiology, 2017, 2(5):17057.
[57] CHARPENTIER E.CRISPR-Cas9:How research on a bacterial RNA-guided mechanism opened new perspectives in biotechnology and biomedicine[J].EMBO Molecular Medicine, 2015, 7(4):363-365.
[58] MAKAROVA K S, WOLF Y I, IRANZO J, et al.Evolutionary classification of CRISPR-Cas systems:A burst of class 2 and derived variants[J].Nature Reviews Microbiology, 2020, 18(2):67-83.
[59] LIU R M, LIANG L Y, FREED E F, et al.Directed evolution of CRISPR/Cas systems for precise gene editing[J].Trends in Biotechnology, 2021, 39(3):262-273.
[60] BURGESS D J.CRISPR screens beyond Cas9[J].Nature Reviews Genetics, 2020, 21(5):273.
[61] KNOTT G J, DOUDNA J A.CRISPR-Cas guides the future of genetic engineering[J].Science, 2018, 361(6405):866-869.
[62] MA J H, LYU Y H, LIU X, et al.Engineered probiotics[J].Microbial Cell Factories, 2022, 21(1):72.
[63] BERLEC A,  KRLEC K, KOCJAN J, et al.Single plasmid systems for inducible dual protein expression and for CRISPR-Cas9/CRISPRi gene regulation in lactic acid bacterium Lactococcus lactis[J].Scientific Reports, 2018, 8:1009.
KRLEC K, KOCJAN J, et al.Single plasmid systems for inducible dual protein expression and for CRISPR-Cas9/CRISPRi gene regulation in lactic acid bacterium Lactococcus lactis[J].Scientific Reports, 2018, 8:1009.
[64] XIONG Z Q, WEI Y Y, KONG L H, et al.Short communication:An inducible CRISPR/dCas9 gene repression system in Lactococcus lactis[J].Journal of Dairy Science, 2020,103(1):161-165.
[65] OH J H, VAN PIJKEREN J P.CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri[J].Nucleic Acids Research, 2014, 42(17):e131.
[66] ZHOU D, JIANG Z N, PANG Q X, et al.CRISPR/Cas9-Assisted seamless genome editing in Lactobacillus plantarum and its application in N-acetylglucosamine production[J].Applied and Environmental Microbiology, 2019, 85(21):e01367-e01319.
[67] WANG G Q, YU H N, FENG X, et al.Specific bile salt hydrolase genes in Lactobacillus plantarum AR113 and relationship with bile salt resistance[J].LWT, 2021, 145:111208.
[68] MYRBRÅTEN I S, WIULL K, SALEHIAN Z, et al.CRISPR interference for rapid knockdown of essential cell cycle genes in Lactobacillus plantarum[J].mSphere, 2019, 4(2):e00007-19.
[69] CITORIK R J, MIMEE M, LU T K.Sequence-specific antimicrobials using efficiently delivered RNA-guided nucleases[J].Nature Biotechnology, 2014, 32(11):1141-1145.
[70] DONG C, FONTANA J, PATEL A, et al.Synthetic CRISPR-Cas gene activators for transcriptional reprogramming in bacteria[J].Nature Communications, 2018, 9:2489.
[71] BIKARD D, EULER C W, JIANG W Y, et al.Exploiting CRISPR-Cas nucleases to produce sequence-specific antimicrobials[J].Nature Biotechnology, 2014, 32(1):1146-1150.
[72] PAN M C, MOROVIC W, HIDALGO-CANTABRANA C, et al.Genomic and epigenetic landscapes drive CRISPR-based genome editing in Bifidobacterium[J].Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(30):e2205068119.
[73] LU Y H, LI F, BAI J R, et al.Rapid screening of antimicrobial probiotics using CRISPR cascade[J].Biosensors &Bioelectronics, 2022, 216:114673.
[74] TANG J L, LEE T, SUN T.Single-nucleotide editing:From principle, optimization to application[J].Human Mutation, 2019, 40(12):2171-2183.
[75] MOLLA K A, SRETENOVIC S, BANSAL K C, et al.Precise plant genome editing using base editors and prime editors[J].Nature Plants, 2021, 7(9):1166-1187.
[76] ANZALONE A V, RANDOLPH P B, DAVIS J R, et al.Search-and-replace genome editing without double-strand breaks or donor DNA[J].Nature, 2019, 576(1785):149-157.
[77] 蒋卉. 益生菌的筛选评价与产品质量控制探讨[J].现代食品, 2019(24):8-11.
JIANG H.Discussion on screening evaluation and product quality control of probiotics[J].Modern Food, 2019, (24):8-11.
[78] VESTERLUND S, VANKERCKHOVEN V, SAXELIN M, et al.Safety assessment of Lactobacillus strains:Presence of putative risk factors in faecal, blood and probiotic isolates[J].International Journal of Food Microbiology, 2007, 116(3):325-331.
[79] OUWEHAND A C, SAXELIN M, SALMINEN S.Assessment of potential risk factors and related properties of clinical, faecal and dairy Bifidobacterium isolates[J].Bioscience and Microflora, 2004, 23(1):37-42.
[80] COLAUTTI A, ARNOLDI M, COMI G, et al.Antibiotic resistance and virulence factors in Lactobacilli:Something to carefully consider[J].Food Microbiology, 2022, 103:103934.
[81] CANNON J P, LEE T A, BOLANOS J T, et al.Pathogenic relevance of Lactobacillus:A retrospective review of over 200 cases[J].European Journal of Clinical Microbiology &Infectious Diseases, 2005, 24(1):31-40.
[82] CHIATTO F, FORSYTHE L, COLETTA R, et al.D-lactic acidosis in children with short bowel syndrome:A relevant clinical condition[J].Transplantation, 2017, 101(6):S133.
[83] AHMED F E.Genetically modified probiotics in foods[J].Trends in Biotechnology, 2003, 21(11):491-497.
[84] 邓圣庭, 闫昭明, 杨媚, 等.灭活益生菌的生理功能及其在动物生产中的应用[J].动物营养学报, 2019, 31(9):4016-4023.
DENG S T, YAN Z M, YANG M, et al.Inactivated probiotics:Physiological function and application in animal production[J].Chinese Journal of Animal Nutrition, 2019, 31(9):4016-4023.
[85] 朱秀敏, 曹萌.灭活益生菌的研究进展[J].中国微生态学杂志, 2010, 22(2):175-178.
ZHU X M, CAO M.A review of progress in researches on inactivated probiotics[J].Chinese Journal of Microecology, 2010, 22(2):175-178.
[86] ZAWISTOWSKA-ROJEK A, TYSKI S.Are probiotic really safe for humans?[J].Polish Journal of Microbiology, 2018, 67(3):251-258.
[87] SANDERS M E, MERENSTEIN D J, OUWEHAND A C, et al.Probiotic use in at-risk populations[J].Journal of the American Pharmacists Association, 2016, 56(6):680-686.