食品是人体所需的主要营养来源,调节着人体的生长发育[1-3]。食品工业的全球化促进了食品在全世界的生产、分销和贸易。同时,食品在加工、分销过程中涉及的由环境和人为因素产生的污染也在逐渐增加。近年来,有关食品中霉菌毒素、过敏性添加剂、转基因作物、多种农药残留的研究越来越多[4-7]。未来必须迎接新的食品挑战,减少可能出现的食品安全风险。
食品分析是分析化学的一个重要领域,2009年CIFUENTES[8]提出了食品组学的概念,将其定义为一门通过应用组学技术研究食品和营养领域的学科。食品组学应用于食品安全研究、质量和可追溯性以及评估食品的功能和营养。代谢组学作为食品组学的一类工具主要研究的是小分子代谢产物,核磁共振波谱(nuclear magnetic resonance spectroscopy,NMR)和高分辨质谱(high resolution mass spectrometry,HRMS)是代谢组学主要使用的2个分析平台。
目前,以质谱为基础的代谢组学研究已经比较完善。这项技术可以分析营养物质(例如氨基酸、脂质、维生素和脂肪酸)、添加剂(例如着色剂、防腐剂和香料)和食品污染物(例如杀虫剂、药物、增塑剂和表面活性剂)[9-12]。此外,代谢组学平台已经成功地应用于食品科学的各个领域,包括食品安全评估、质量管理、食品溯源、食品(生物)标志物确定、食品生物活性的研究以及食品风味化学和功能的发现[13-19]。本文综述了基于液相色谱-质谱(liquid chromatograph mass spectrometer,LC-MS)的代谢组学方法和有关代谢组学应用的最新研究现状,并展望了食品代谢组学的应用趋势。
1 基于LC-MS的食品代谢组学技术
代谢组学在医学、农业、环境科学以及食品科学领域有着不同的应用[20-21]。在食品科学领域,代谢组学与基因组学、转录组学和蛋白质组学均通过食品组学方法参与研究,其已经成为研究饮食与生物机体之间相互作用的有力工具,使研究者能够更深入地了解食品中的营养物质与疾病代谢水平之间的关系。应用代谢组学技术进行食品研究的一般工作流程包括样品收集、样品前处理、仪器分析和数据处理。图1 展示了代谢组学在食品分析中的工作流程。

图1 食品代谢组学流程
Fig.1 Schematic diagram of food metabolomics workflow
近年来,液相色谱(liquid chromatography,LC)技术发展迅速。在现代高效液相色谱中,色谱技术不断地追求分析的高速度和高分辨率,在某种程度上色谱柱的小型化是LC发展的主要方向,用于高灵敏度的分离分析[22]。纳米液相色谱(nano liquid chromatography,Nano-LC)是一种先进的色谱技术,使用柱直径≤100 μm的毛细管柱进行分离[23]。Nano-LC有很多优点,如高灵敏度、高分辨率、较低的流速(≤800 nL/min)以及试剂的消耗减少等。REINHOLDS等[24]开发了用于分析谷物和豆类中 27 种真菌毒素的Nano-LC分离技术,展现了足够的准确度和精密度。与传统方法相比,Nano-LC对包括乙腈在内的流动相的消耗显著降低。在处理非常复杂的食品基质时,传统的液相色谱法的分离度有限。二维液相色谱(two-dimensional liquid chromatography,2D-LC)技术与一维液相色谱相比,选择性好、灵敏度高、分辨率高,并且预处理简单、峰容量大,适合分离复杂样品基质。例如,在对食品的风味成分进行分析时,由于风味成分的含量从小分子到大分子变化非常大,传统的一维液相色谱法的峰容量和分辨率相对有限,不能满足对风味成分全面分析的要求。而2D-LC应运而生,成为分离复杂风味化合物的有效分析工具。
随着MS的快速发展,HRMS已在食品分析中得到充分应用[25]。HRMS分析速度快、分析精度高、具有较高的灵敏度、较宽的动态范围和较高的选择性,是许多组学科学中最强大的分析工具。在HRMS分析仪中,飞行时间质谱仪(time of flight mass spectrometer,TOF-MS)和静电场轨道阱质谱仪(orbitrap-MS)是使用最多的,而傅里叶变换离子回旋共振质谱仪(Fourier transform ion cyclotron resonance mass spectrometer,FTICR-MS)由于采集速率低,难以与快速色谱分离相结合,并且存在维护成本较高等缺点,应用较少。当MS与分离技术、串联技术和信息方法相结合时,能够提供非常有效且丰富的信息。在食品组学的分析中最常见的是多MS串联使用,如四极杆飞行时间质谱(quadrupole-time of flight mass spectrometer,Q-TOF-MS)、三重四极杆质谱(triple quadrupole mass spectrometer,QqQ-MS)、四极杆离子阱质谱(quadrupole ion trap mass spectrometer,Q-Trap-MS)、四极杆轨道阱质谱(quadrupole orbitrap mass spectrometer,Q-Orbitrap-MS)[26-29]。可以认为,MS为众多组学科学奠定了基础。
基于LC-MS的代谢组学平台已经成功地应用于食品科学的各个领域。根据所分析的化学物质在食品基质中的覆盖范围,该分析方法主要分为两类,即靶向和非靶向的代谢组学方法。非靶向方法侧重于分析样品中所有可检测的分析物,包括未知的成分。相比之下,靶向方法是对目标分析物的检测。CAJKA等[30]指出,在定量和研究结果的可比性方面,靶向或非靶向方法的应用取决于分析化学家研究问题的目的和范围[30]。
2 基于LC-MS的食品代谢组学的数据处理
数据处理可能是代谢组学的瓶颈之一,以非靶向筛选为例, LC-MS非靶向代谢组学会产生大量数据,如果没有开源的数据库,将很难根据保留时间和串联质谱信息来确定未知峰。数据处理步骤主要包括数据预处理、建立单变量或多变量分析模型和模型数据评估。
数据预处理会对结果产生影响,在进行各个步骤的时候应当避免信号被错误排除或过拟合,同时尽可能地减少由设备波动和系统测量误差等引起的差异。表1 归纳整理了常用的预处理程序的摘要。
表1 数据预处理
Table 1 Data pre-processing

预处理步骤目的相关算法及软件峰检测捕捉目标离子信号,过滤由缓冲液、溶剂产生的信号,降低假阳性结果的比例小波变换、基于centWave算法的XCMS软件[31]校准保留时间对温度或压力波动、pH值变化、基质效应等引起的保留时间的漂移进行校正动态时间归整算法32〛、相关优化规整算法33〛、参数时间规整算法34〛归一化消除系统测量的误差内标归一化、分位数归一化[35]、总和归一化[36]数据缩放消除由于分析物浓度以及不同的电离行为对信号强度的影响自动缩放、pareto缩放、范围缩放[37]数据转换减少数据的偏斜和异方差(方差的异质性),达到同方差(方差的均匀性)广义对数变换[38]、幂变换[37]
数据预处理后通常使用多变量方法进行进一步分析。多变量分析分为无监督和有监督分析方法,通过结构化、简化和图解数据集的方式来显示特征的相似性和差异性。无监督方法是一种无假设的方法,其目的是减少测量的变量并显示样本特征的最大差异。例如,不考虑特征是否取决于产地、动物研究中不考虑饲料对动物的影响、植物研究中不考虑栽培方法对植物的影响。有监督方法则需要考虑关于样本的元数据。
主成分分析(principal component analysis,PCA)是最常用的无监督多变量分析模型。PCA将多维数据降成二维或三维数据,构建新的主成分并探索数据集的整体变化,最终将不同样本之间的总代谢差异进行可视化[39]。PCA常被作为一种探索性的方法,例如在食品真实性研究中用于区分辣椒和猪肉的地理来源[40-41]。
在实际的分析中仅仅应用无监督方法是不够的,大多数情况下必须使用有监督方法来提取最相关化合物。偏最小二乘判别分析是目前最常用的有监督多变量分析模型,其旨在减少残差(实际观测值与预测值之间的偏差)并最大化样本与特征之间的协方差。正交偏最小二乘判别分析(orthogonal partial least squares-discriminant analysis,OPLS-DA)是一个相对新颖的方法,通过将类别信息纳入模型并找到类别最大化分离方向,最小化每个类别之间的分散度。目前OPLS-DA使用仍然较少,但已有研究表明了OPLS-DA的应用价值。例如,QUINTERO等[42]和BRIGANTE等[43]使用OPLS-DA确定了浓缩咖啡感官相关的标志物以及寻找奇亚、亚麻和芝麻籽真实性的标志物。监督方法往往会过度拟合,因此需要通过进一步交叉验证和结合其他模型性能标准来判别模型的可靠性。
最近,机器学习被引入食品科学领域,其最突出的特点是学习大量数据来提高模型性能,常使用的算法是随机森林和支持向量机[44]。ZHOU等[45]回顾了深度学习在食品科学中的应用,包括食品识别、热量估计、质量检测、食品溯源和食品污染。将机器学习与多源数据融合相结合,可以对食品进行更加全面地分析。因此,机器学习在食品科学领域是一个很有前景的工具,机器学习中的分类和回归模型在未来适用于食品科学领域。
3 基于LC-MS的食品代谢组学应用
现代食品分析中,食品代谢组学主要应用于食品安全(评估微生物毒素、过敏原、抗营养物质、食源性病原体、农药、兽药)、食品质量(感官特性和营养价值)、食品真实性(生物特性、地理来源、农业生产和新鲜度)和食品溯源(食品成分、加工过程)[46-47]。
3.1 食品安全
食品安全是食品分析的重要课题之一,消费者在日常的消费过程中需要在售商品都是安全可靠的,但有关食品安全的事件却层出不穷。食品安全包括很多子领域,如检测与微生物相关的食品腐败、确定过敏原、检测化学污染物和非法添加物、评估天然毒素等。
在生产、加工等关键步骤中食品可能会被污染,如食品原材料生产过程中农药、兽药的使用,食品原材料加工过程中病原体残留。根据世界卫生组织于2020年4月30日有关食品安全的报道中得知,不安全食品可导致腹泻、癌症等200多种疾病,每年全世界约有6亿人(几乎每10人中就有1人)因食用受污染的食品而患病,并约有42万人死亡[48]。
为了检测和量化食品中的污染物,需要对食品基质进行适当的前处理。QuEChERS方法快速、简便、廉价、有效、可靠和安全,作为一种两步的样品富集和脱盐方法,已被普遍用于与LC相结合的小分子检测[49],其中QuEChERS与固相萃取或固相微萃取的结合已被成功用于食品中农药残留分析。然而,考虑到一些天然毒素在非常低的浓度下仍具有极大的毒性(甚至是致命的),应该进一步改进制备方法,以便进行灵敏和高通量的检测[50]。例如,在应用HPLC-MS/MS之前,使用多抗体免疫亲和柱选择性地提取7种毒素,扩大了测定的线性范围,并将检测限降至μg/kg[51]。同样,ALTAFINI等[52]对意大利萨拉米香肠中的赭曲霉毒素A进行检测时,通过免疫亲和色谱柱进行样品制备,使用LC-MS/MS进行分析,保证平均回收率为79.4%~89.0%,检测限和定量限分别为0.10 μg/kg和0.25 μg/kg。表2 归纳整理了代谢组学在常见食品安全问题中的应用摘要。
表2 代谢组学在食品安全中的应用
Table 2 Application of metabolomics in food safety
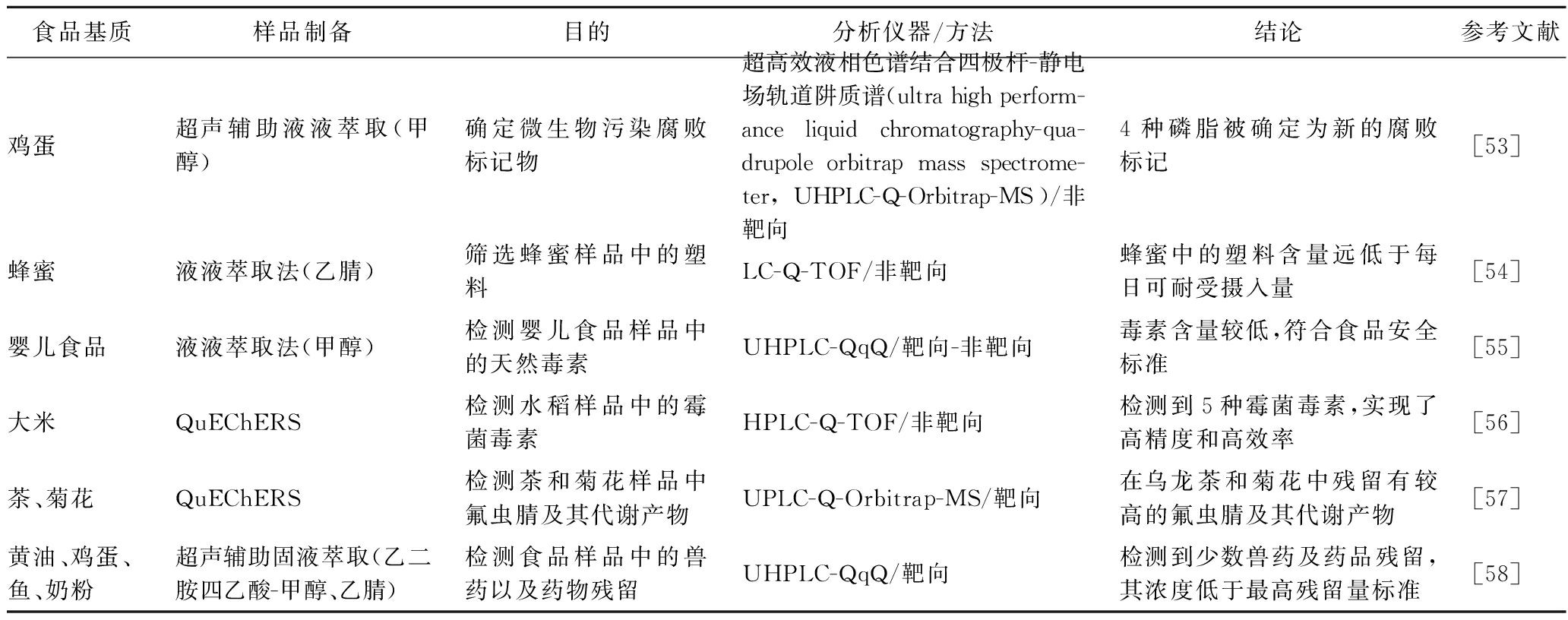
食品基质样品制备目的分析仪器/方法结论参考文献鸡蛋超声辅助液液萃取(甲醇)确定微生物污染腐败标记物超高效液相色谱结合四极杆-静电场轨道阱质谱(ultra high perform-ance liquid chromatography-qua-drupole orbitrap mass spectrome-ter,UHPLC-Q-Orbitrap-MS)/非靶向4种磷脂被确定为新的腐败标记[53]蜂蜜液液萃取法(乙腈)筛选蜂蜜样品中的塑料LC-Q-TOF/非靶向蜂蜜中的塑料含量远低于每日可耐受摄入量[54]婴儿食品液液萃取法(甲醇)检测婴儿食品样品中的天然毒素UHPLC-QqQ/靶向-非靶向毒素含量较低,符合食品安全标准[55]大米QuEChERS检测水稻样品中的霉菌毒素HPLC-Q-TOF/非靶向检测到5种霉菌毒素,实现了高精度和高效率[56]茶、菊花QuEChERS检测茶和菊花样品中氟虫腈及其代谢产物UPLC-Q-Orbitrap-MS/靶向在乌龙茶和菊花中残留有较高的氟虫腈及其代谢产物[57]黄油、鸡蛋、鱼、奶粉超声辅助固液萃取(乙二胺四乙酸-甲醇、乙腈)检测食品样品中的兽药以及药物残留UHPLC-QqQ/靶向检测到少数兽药及药品残留,其浓度低于最高残留量标准[58]
3.2 食品质量
食品质量包含多种因素,如味道、颜色、气味和营养价值。食品中的许多风味成分含量甚微,如何从中筛选和识别关键成分一直是食品感官研究的难点和焦点。经过多年的发展,代谢组学已经成为食品感官分析的重要工具,它可以在分子水平上识别食品的关键风味活性物质,揭示食品风味的形成机制,并为评价体系提供参考[59-60]。以绿茶为例,非挥发性代谢物决定着茶叶的颜色、风味质量和功能。游离氨基酸可以为茶汤带来新鲜、轻快的味道并参与香气物质的形成,类黄酮苷、咖啡因和儿茶素影响苦味,有机酸影响果味和酸度,磷脂影响香味的形成,类胡萝卜素和脂溶性叶绿素对干茶的颜色和形状有影响[61]。
代谢组学对食品营养价值分析的主要目标是评估某些化合物对生物机体可能产生的影响、不同化合物之间的协同作用以及认定与健康相关的生物标志物。这些标志物分子可以作为评价身体营养状况的指标,例如,缺乏某些脂肪酸、类胡萝卜素和维生素分子会导致脂肪沉积、胆固醇水平和胰岛素活性失调,这与慢性疾病(例如肥胖)的有关[62]。
表3归纳整理了代谢组学在常见食品质量问题中的应用摘要。
表3 代谢组学在食品质量中的应用
Table 3 Application of metabolomics in food quality

食品基质样品制备目的分析仪器/方法结论参考文献辣椒液液萃取法(丙酮、乙醚)确定红辣椒颜色的影响因素UHPLC-Q-Orbitrap-MS/非靶向脂质及其衍生物和苯丙氨酸可能影响辣椒的颜色[63]白牡丹茶液液萃取法(甲醇)测定白牡丹茶贮存过程中与感官相关的化学变化UPLC-QTRAP/靶向类黄酮、丹宁、氨基酸减少,酚酸和有机酸含量增加,导致白牡丹茶涩味和鲜味减少[29]生菜超声辅助液液萃取法(甲醇)确定生菜发生褐变的生物标记物UHPLC-HRMS/非靶向确定了12种生菜褐变的生物标志物[64]蜂蜜酸化后使用C18柱分馏确定导致蜂蜜苦味的次级代谢物LC-LTQ-Orbitrap/非靶向unedone是导致蜂蜜苦味的主要化合物[65]白酒液液萃取法确定影响烈酒口感的活性化合物LC-HRMS/非靶向白兰地丹宁A增加了白酒的甜度[66]芝麻液液萃取法(甲醇)确定抗氧化活性差异的关键代谢物LC-MS/MS/靶向芝麻的抗氧化活性可能主要与类黄酮,氨基酸及其衍生物,木酚素和萜类化合物的含量相关[67]橄榄叶、橄榄核固相支持液液萃取法测定橄榄叶和橄榄核中的生物活性成分UHPLC-Q-TOF/非靶向从叶子和果核中分别筛选出10种和7种生物酚[68]
注:unedone分子式C13H20O4,中文命名为4,8,8-三甲基-2(1,2-二羟基丙基)-1-氧杂螺[2.5]辛-4-烯-6-酮。
3.3 食品真实性
与其他行业相比,食品行业的利润率较低。为了增加利润,一些商家试图通过伪造或掺假来实现利益最大化,从而造成了食品欺诈。食品欺诈是一个日益严重的全球性问题,导致了严重的食品安全风险[69]。相关报道很多,例如2008年的中国三聚氰胺事件、2013年的欧洲马肉风波、2017年的巴西黑心肉事件等。食品欺诈使得食品的真实性备受质疑,如生物特性、地理来源、农业生产和新鲜度等。近年来代谢组学已经成为了打击食品欺诈的一个强有力的武器,一些代谢物、脂类作为分子标志物在确定食品真实性方面发挥着重要作用。与遗传和蛋白质标志物相比,代谢标志物(代谢物、脂类)具有样品制备简单、应用范围广的优势。
不同生物身份的食品很难进行区分,而加工食品尤其容易造成食品欺诈的风险。对于食品生物特性的鉴别,目前常采用LC-MS非靶向代谢组学的方法来探索食品生物特性的标志物。牛奶、山羊奶、水牛奶是饮食中3种消费量很大的奶类,由于生产和饲养成本等各种因素的影响,水牛奶、山羊奶的价格高于牛奶,因此产生了食品欺诈的风险。JIA等[70]利用UHPLC-Q-Orbitrap-MS进行非靶向代谢组学在3种动物的奶中寻找标志物,从11个潜在标记物中筛选出5个标记物来区分3种产品。其中β-胡萝卜素仅在牛奶中发现,其定量检测灵敏度能够检测出含有5%的牛奶掺假,维生素D2仅在水牛奶中发现;山羊奶中壬酸、癸酸和辛酸的含量高于牛奶和水牛奶中的含量。
地理原产地有助于提高食品的竞争力,特别是对于那些带有原产地保护、受地理标志保护和传统特产保证的食品[71-72]。因此,迫切需要保护特定地理来源的食品,防止可能的食品欺诈。原材料的地理来源信息至关重要。CAVANNA等[73]基于UHPLC-Q-Orbitrap-MS的非靶向代谢组学方法用来检测来自意大利、欧洲和欧洲以外国家的硬质小麦样品中的代谢物,最终确定了14个“非意大利”标志物、2个“非欧洲”标志物和9个“意大利”标志物。
农业生产是指培养动、植物或真菌的生产系统。市场上的一些食品被贴上有机、野生、散养、草饲等标签,不同的饲养条件对产品产生不同的影响。因此,有必要关注不同饲养条件产品的鉴别。在农业生产中常关注的是有机食品和常规食品的鉴别。与常规食品相比,有机食品对人体健康更加有益,其往往以更高的价格出售从而造成了食品欺诈的风险。CUBERO-LEON等[74]使用UHPLC-TOF对有机和常规胡萝卜进行非靶向代谢组学分析,选择了区分2组样品的24个生物标志物,其中有5个标记物显示出了显著差异。如果在预测模型(OPLS-DA)中把收获年份排除在训练集之外,则分类正确率为76%~100%,这说明该方法受收获时间的影响较小,方法稳定性高。
食品的新鲜度能够反映出食品自身的特点,不仅影响食品的风味和营养还会影响有害物质的含量。此外,新鲜度将直接影响食品原料的销售、加工价值以及加工食品的质量和安全。鱼类很容易变质,为解决把产品冷冻后再解冻然后当作新鲜产品销售的问题,STELLA等[75]提出了一种基于UHPLC-Q-Orbitrap-MS的非靶向代谢组学方法,对冷冻后再解冻和新鲜欧洲海鲈鱼片的代谢组进行分析,最终把二十碳五烯酸和二十二碳六烯酸确定为具有高度区分性的新鲜度标志物。
3.4 食品溯源
食品溯源是指在整个供应链中对食品进行持续监测。毋庸置疑,食品溯源与食品质量、食品安全和公共健康密切相关。由于在初级生产、加工、分销和零售阶段对有关食品来源和食品成分信息的精确度要求越来越高,食品溯源对食品工业和消费者均很重要。因此,对食品的加工方法、成分和地理来源的了解是食品可溯源性研究的主要目的。
可使用代谢组学可以对食品工业的主要传统方法(如加热、干燥、浓缩和发酵)进行评估。例如,ROCCHETTI等[76]研究了在制造干式发酵萨拉米香肠过程中,加入3种不同的微生物发酵剂(即戊糖片球菌、木糖葡萄球菌和清酒乳杆菌)促进发酵的过程。基于UHPLC-Q-Orbitrap非靶向代谢组学分析结果,每种微生物发酵剂在香肠成熟末期都有独特的代谢组学特征,涉及脂质氧化(包括脂肪酸的羟基和环氧衍生物)和γ-谷氨酰肽的积累,有助于产品的感官质量。表4 归纳整理了一些代谢组学在食品真实性和食品追溯中的应用摘要。
表4 代谢组学在食品真实性和食品溯源中的应用
Table 4 Application of metabolomics in food authenticity and food traceability
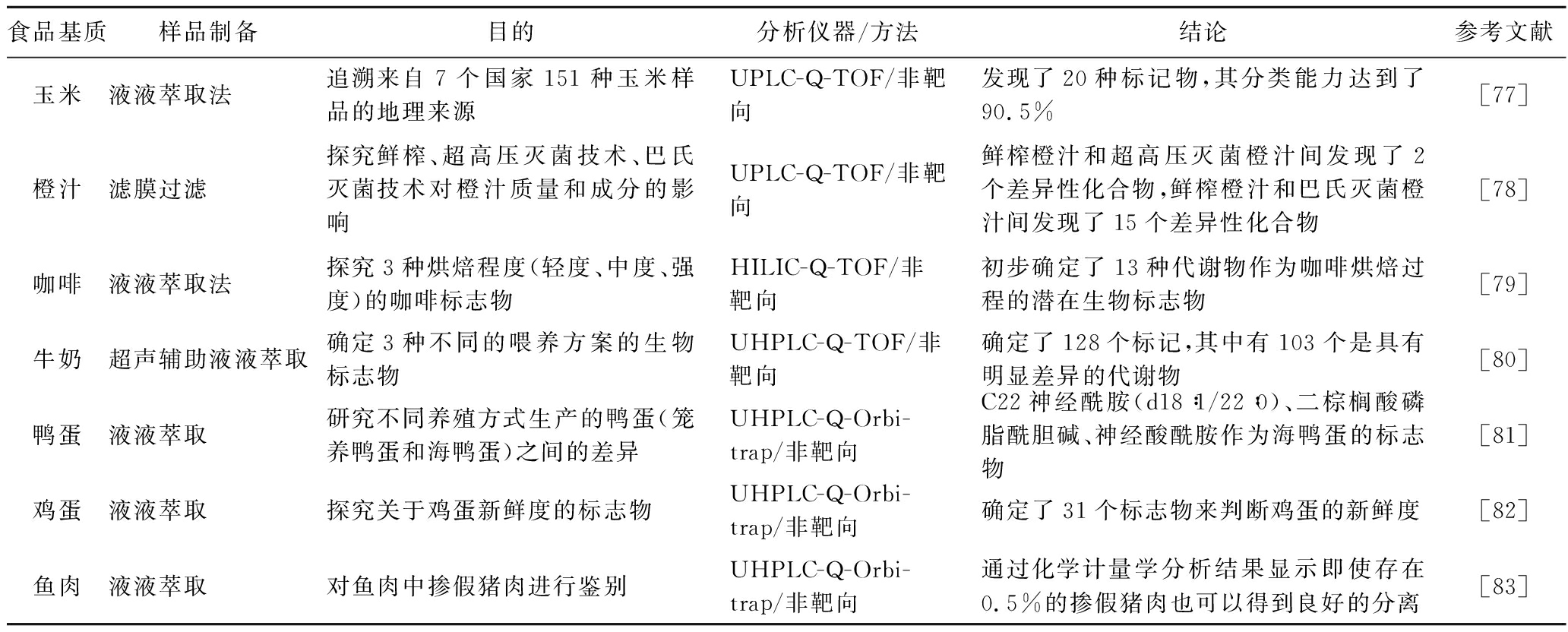
食品基质样品制备目的分析仪器/方法结论参考文献玉米液液萃取法追溯来自7个国家151种玉米样品的地理来源UPLC-Q-TOF/非靶向发现了20种标记物,其分类能力达到了90.5%[77]橙汁滤膜过滤探究鲜榨、超高压灭菌技术、巴氏灭菌技术对橙汁质量和成分的影响UPLC-Q-TOF/非靶向鲜榨橙汁和超高压灭菌橙汁间发现了2个差异性化合物,鲜榨橙汁和巴氏灭菌橙汁间发现了15个差异性化合物[78]咖啡液液萃取法探究3种烘焙程度(轻度、中度、强度)的咖啡标志物HILIC-Q-TOF/非靶向初步确定了13种代谢物作为咖啡烘焙过程的潜在生物标志物[79]牛奶超声辅助液液萃取确定3种不同的喂养方案的生物标志物UHPLC-Q-TOF/非靶向确定了128个标记,其中有103个是具有明显差异的代谢物[80]鸭蛋液液萃取研究不同养殖方式生产的鸭蛋(笼养鸭蛋和海鸭蛋)之间的差异UHPLC-Q-Orbi-trap/非靶向C22神经酰胺(d18∶1/22∶0)、二棕榈酸磷脂酰胆碱、神经酸酰胺作为海鸭蛋的标志物[81]鸡蛋液液萃取探究关于鸡蛋新鲜度的标志物UHPLC-Q-Orbi-trap/非靶向确定了31个标志物来判断鸡蛋的新鲜度[82]鱼肉液液萃取对鱼肉中掺假猪肉进行鉴别UHPLC-Q-Orbi-trap/非靶向通过化学计量学分析结果显示即使存在0.5%的掺假猪肉也可以得到良好的分离[83]
4 总结和展望
本篇综述展示了色谱与质谱的最新发展,并介绍了基于 LC-MS 的靶向和非靶向代谢组学在食品科学领域的应用。代谢组学已经在食品安全、食品质量、食品真实性以及食品溯源方面进行了全面的研究,为食品科学领域提供了可靠的数据。NMR是一种重现性好、无损的表征方法,通过原子核的固有磁性检测分子特征,常用于靶向代谢组学分析,但其维护成本高、灵敏度低并在数据采集和数据处理方面受到限制。相对而言,HRMS灵敏度高、分辨率高、动态范围宽,使得靶向和非靶向方法的融合成为了可能,是食品代谢组学分析中的常用技术,其使用局限性是代谢物数据库少、样品需要繁琐的前处理。在整个非靶向代谢组学分析程序中,数据处理是一个非常巨大且重要的工程,如果没有强大的统计软件工具,将难以处理大量的实验数据并进行精确和可靠的量化。
在今后的食品代谢组学分析中应注重以下工作:a)优化从复杂食品基质中提取目标分析物的方法;b)将靶向和非靶向方法进行融合以实现更有效的代谢组学分析;c)开发用于数据处理的生物信息学算法,以自动组合大量代谢组学数据;d)不断拓展食品成分数据库和用于代谢物鉴定的串联质谱数据库;e)将代谢组学与其他组学相结合,例如代谢组学与宏基因组学相结合可以更好地了解影响食品质量各因素之间的关联和相互作用,并确认不同生物水平(基因型-表型)的特征。
[1] UAUY R, KURPAD A, TANO-DEBRAH K, et al.Role of protein and amino acids in infant and young child nutrition:Protein and amino acid needs and relationship with child growth[J].Journal of Nutritional Science and Vitaminology, 2015, 61(Supplement):S192-S194.
[2] SPERRINGER J E, ADDINGTON A, HUTSON S M.Branched-chain amino acids and brain metabolism[J].Neurochemical Research, 2017, 42(6):1697-1709.
[3] THOMASSET B, SARAZIN C.Biodiversity of lipid species-Benefit for nutrition and effects on health[J].Biochimie, 2020, 178:1-3.
[4] DUAN Y, RAMILAN T, LUO J H, et al.Risk assessment approaches for evaluating cumulative exposures to multiple pesticide residues in agro-products using seasonal vegetable monitoring data from Hainan, China:A case study[J].Environmental Monitoring and Assessment, 2021, 193(9):578.
[5] VALLUZZI R L, FIERRO V, ARASI S, et al.Allergy to food additives[J].Current Opinion in Allergy and Clinical Immunology, 2019, 19(3):256-262.
[6] ARPAIA S.Environmental risk assessment in agro-ecosystems:Revisiting the concept of receiving environment after the EFSA guidance document[J].Ecotoxicology and Environmental Safety, 2021, 208:111676.
[7] AYOFEMI OLALEKAN ADEYEYE S.Aflatoxigenic fungi and mycotoxins in food:A review[J].Critical Reviews in Food Science and Nutrition, 2020, 60(5):709-721.
[8] CIFUENTES A. Food analysis and foodomics[J]. Journal of Chromatography A, 2009, 1216(43): 7109.
[9] JIA W, WANG H, SHI L, et al.High-throughput foodomics strategy for screening flavor components in dairy products using multiple mass spectrometry[J].Food Chemistry, 2019, 279:1-11.
[10] IB
 EZ C, SIM
EZ C, SIM C, GARC
C, GARC A-CA
A-CA AS V, et al. The role of direct high-resolution mass spectrometry in foodomics[J]. Analytical and Bioanalytical Chemistry, 2015, 407(21): 6275-6287.
AS V, et al. The role of direct high-resolution mass spectrometry in foodomics[J]. Analytical and Bioanalytical Chemistry, 2015, 407(21): 6275-6287.
[11] CHANG W C W, WU H Y, YEH Y, et al.Untargeted foodomics strategy using high-resolution mass spectrometry reveals potential indicators for fish freshness[J].Analytica Chimica Acta, 2020, 1127:98-105.
[12] HERRERO M, SIM C, GARC
C, GARC A-CA
A-CA AS V, et al.Foodomics:MS-based strategies in modern food science and nutrition[J].Mass Spectrometry Reviews, 2012, 31(1):49-69.
AS V, et al.Foodomics:MS-based strategies in modern food science and nutrition[J].Mass Spectrometry Reviews, 2012, 31(1):49-69.
[13] BRAGAGNOLO F S, FUNARI C S, IB
 EZ E, et al.Metabolomics as a tool to study underused soy parts:In search of bioactive compounds[J].Foods, 2021, 10(6):1308.
EZ E, et al.Metabolomics as a tool to study underused soy parts:In search of bioactive compounds[J].Foods, 2021, 10(6):1308.
[14] CERRATO A, AITA S E, CAVALIERE C, et al.Comprehensive identification of native medium-sized and short bioactive peptides in sea bass muscle[J].Food Chemistry, 2021, 343:128443.
[15] AFZAAL M, SAEED F, HUSSAIN M, et al.Proteomics as a promising biomarker in food authentication, quality and safety:A review[J].Food Science &Nutrition, 2022,10(7):2333-2346.
[16] WANG K W, XU L, WANG X, et al.Discrimination of beef from different origins based on lipidomics:A comparison study of DART-QTOF and LC-ESI-QTOF[J].LWT, 2021, 149:111838.
[17] 王永芳,娄婷婷,温华蔚,等.LC-MS/MS测定牛奶中氟虫腈及代谢物残留量[J].中国乳品工业,2021,49(3):47-51.
WANG Y F, LOU T T, WEN H W, et al.Determination of fipronil and its metabolites residues in milk by liquid chromatography-tandem mass spectrometry[J].China Dairy Industry, 2021, 49(3):47-51.
[18] 贾玮,杨颖欢,曾桥,等.基于代谢组学方法分析14种辐照香辛料中的斑蝥素[J].陕西科技大学学报,2022,40(3):66-71.
JIA W, YANG Y H, ZENG Q, et al.Analysis of cantharidin of 14 irradiated spices based on metabonomics methods[J].Journal of Shaanxi University of Science &Technology, 2022, 40(3):66-71.
[19] 安婷婷,祖仲骐,陈琪,等.基于UPLC-Q-exactive orbitrap MS代谢组学分析冠突散囊菌发酵对黑茶代谢物和滋味品质的影响[C].中国食品科学技术学会第十八届年会摘要集,2022:514-515.
[20] MURITHI J M, OWEN E S, ISTVAN E S, et al.Combining stage specificity and metabolomic profiling to advance antimalarial drug discovery[J].Cell Chemical Biology, 2020, 27(2):158-171.e3.
[21] ALMUHAYAWI M S, HASSAN A H A, AL JAOUNI S K, et al.Influence of elevated CO2 on nutritive value and health-promoting prospective of three genotypes of Alfalfa sprouts (Medicago Sativa)[J].Food Chemistry, 2021, 340:128147.
[22] WILSON S R, OLSEN C, LUNDANES E.Nano liquid chromatography columns[J].The Analyst, 2019, 144(24):7090-7104.
[23]  EST
EST K J, MORAVCOV
K J, MORAVCOV D, KAHLE V.Instrument platforms for nano liquid chromatography[J].Journal of Chromatography A, 2015, 1421:2-17.
D, KAHLE V.Instrument platforms for nano liquid chromatography[J].Journal of Chromatography A, 2015, 1421:2-17.
[24] REINHOLDS I, JANSONS M, FEDORENKO D, et al.Mycotoxins in cereals and pulses harvested in Latvia by nanoLC-Orbitrap MS[J].Food Additives &Contaminants:Part B, 2021, 14(2):115-123.
[25] 赖博文,刘玢,梁永康.基于高分辨质谱的非靶向代谢组学在食品造假鉴定中的研究进展[J].生物技术通报,2019,35(2):192-197.
LAI B W, LIU B, LIANG Y K.Research progress on food fraud using non-targeted metabolomics based on high-resolution mass spectrometry[J].Biotechnology Bulletin, 2019, 35(2):192-197.
[26] RIVERA-PÉREZ A, ROMERO-GONZ LEZ R, GARRIDO FRENICH A.Application of an innovative metabolomics approach to discriminate geographical origin and processing of black pepper by untargeted UHPLC-Q-Orbitrap-HRMS analysis and mid-level data fusion[J].Food Research International, 2021, 150:110722.
LEZ R, GARRIDO FRENICH A.Application of an innovative metabolomics approach to discriminate geographical origin and processing of black pepper by untargeted UHPLC-Q-Orbitrap-HRMS analysis and mid-level data fusion[J].Food Research International, 2021, 150:110722.
[27] ZHANG X N, LI X, SU M S, et al.A comparative UPLC-Q-TOF/MS-based metabolomics approach for distinguishing peach (Prunus persica (L.) Batsch) fruit cultivars with varying antioxidant activity[J].Food Research International, 2020, 137:109531.
[28] ALEM N-JIMÉNEZ C, DOM
N-JIMÉNEZ C, DOM NGUEZ-PERLES R, FANTI F, et al.Unravelling the capacity of hydroxytyrosol and its lipophenolic derivates to modulate the H2O2-induced isoprostanoid profile of THP-1 monocytes by UHPLC-QqQ-MS/MS lipidomic workflow[J].Microchemical Journal, 2021, 170:106703.
NGUEZ-PERLES R, FANTI F, et al.Unravelling the capacity of hydroxytyrosol and its lipophenolic derivates to modulate the H2O2-induced isoprostanoid profile of THP-1 monocytes by UHPLC-QqQ-MS/MS lipidomic workflow[J].Microchemical Journal, 2021, 170:106703.
[29] FAN F Y, HUANG C S, TONG Y L, et al.Widely targeted metabolomics analysis of white peony teas with different storage time and association with sensory attributes[J].Food Chemistry, 2021, 362:130257.
[30] CAJKA T, FIEHN O.Toward merging untargeted and targeted methods in mass spectrometry-based metabolomics and lipidomics[J].Analytical Chemistry, 2016, 88(1):524-545.
[31] TAUTENHAHN R, BÖTTCHER C, NEUMANN S.Highly sensitive feature detection for high resolution LC/MS[J].BMC Bioinformatics, 2008, 9:504.
[32] RAMAKER H J, VAN SPRANG E N M, WESTERHUIS J A, et al.Dynamic time warping of spectroscopic BATCH data[J].Analytica Chimica Acta, 2003, 498(1-2):133-153.
[33] SKOV T, VAN DEN BERG F, TOMASI G, et al.Automated alignment of chromatographic data[J].Journal of Chemometrics, 2006, 20(11-12):484-497.
[34] EILERS P H C.Parametric time warping[J].Analytical Chemistry, 2004, 76(2):404-411.
[35] LEE J, PARK J, LIM M S, et al.Quantile normalization approach for liquid chromatography-mass spectrometry-based metabolomic data from healthy human volunteers[J].Analytical Sciences, 2012, 28(8):801-805.
[36] DI GUIDA R, ENGEL J, ALLWOOD J W, et al.Non-targeted UHPLC-MS metabolomic data processing methods:A comparative investigation of normalisation, missing value imputation, transformation and scaling[J].Metabolomics, 2016, 12(5):93.
[37] VAN DEN BERG R A, HOEFSLOOT H C J, WESTERHUIS J A, et al.Centering, scaling, and transformations:Improving the biological information content of metabolomics data[J].BMC Genomics, 2006, 7:142.
[38] AMBROISE J, BEARZATTO B, ROBERT A, et al.Impact of the spotted microarray preprocessing method on fold-change compression and variance stability[J].BMC Bioinformatics, 2011, 12:413.
[39] INOUE K, TANADA C, HOSOYA T, et al.Principal component analysis of molecularly based signals from infant formula contaminations using LC-MS and NMR in foodomics[J].Journal of the Science of Food and Agriculture, 2016, 96(11):3876-3881.
[40] MI S, SHANG K, LI X, et al.Characterization and discrimination of selected China′s domestic pork using an LC-MS-based lipidomics approach[J].Food Control, 2019, 100:305-314.
[41] BARBOSA S, SAURINA J, PUIGNOU L, et al.Classification and authentication of paprika by UHPLC-HRMS fingerprinting and multivariate calibration methods (PCA and PLS-DA)[J].Foods, 2020, 9(4):486.
[42] QUINTERO M, SANTANDER M J, VEL SQUEZ S, et al.Exploring chemical markers related to the acceptance and sensory profiles of concentrated liquid coffees:An untargeted metabolomics approach[J].Foods, 2022, 11(3):473.
SQUEZ S, et al.Exploring chemical markers related to the acceptance and sensory profiles of concentrated liquid coffees:An untargeted metabolomics approach[J].Foods, 2022, 11(3):473.
[43] BRIGANTE F I, PODIO N S, WUNDERLIN D A, et al.Comparative metabolite fingerprinting of chia, flax and sesame seeds using LC-MS untargeted metabolomics[J].Food Chemistry, 2022, 371:131355.
[44] LIEBAL U W, PHAN A N T, SUDHAKAR M, et al.Machine learning applications for mass spectrometry-based metabolomics[J].Metabolites, 2020, 10(6):243.
[45] ZHOU L, ZHANG C, LIU F, et al.Application of deep learning in food:A review[J].Comprehensive Reviews in Food Science and Food Safety, 2019, 18(6):1793-1811.
[46] COOK P W, NIGHTINGALE K K.Use of omics methods for the advancement of food quality and food safety[J].Animal Frontiers, 2018, 8(4):33-41.
[47] PINU F R.Early detection of food pathogens and food spoilage microorganisms:Application of metabolomics[J].Trends in Food Science &Technology, 2016, 54:213-215.
[48] 世界卫生组织.食品安全[EB/OL].(2020-4-30)[2022-6-5].https://www.who.int/zh/news-room/fact-sheets/detail/food-safety.
World Health Organization.Food Safety[EB/OL].(2020-4-30)[2022-6-5].https://www.who.int/zh/news-room/fact-sheets/detail/food-safety.
[49] GONZ LEZ-CURBELO M
LEZ-CURBELO M  , SOCAS-RODR
, SOCAS-RODR GUEZ B, HERRERA-HERRERA A V, et al.Evolution and applications of the QuEChERS method[J].TrAC Trends in Analytical Chemistry, 2015, 71:169-185.
GUEZ B, HERRERA-HERRERA A V, et al.Evolution and applications of the QuEChERS method[J].TrAC Trends in Analytical Chemistry, 2015, 71:169-185.
[50] MAN Y, LIANG G, LI A, et al.Analytical methods for the determination of Alternaria mycotoxins[J].Chromatographia, 2017, 80(1):9-22.
[51] ZHANG Z W, HU X F, ZHANG Q, et al.Determination for multiple mycotoxins in agricultural products using HPLC-MS/MS via a multiple antibody immunoaffinity column[J].Journal of Chromatography B, 2016, 1021:145-152.
[52] ALTAFINI A, GUERRINI A, CAPRAI E, et al.Ochratoxin A contamination of the casing and the edible portion of artisan Salamis produced in two Italian regions[J].World Mycotoxin Journal, 2020, 13(4):553-562.
[53] CHANG W C W, WU H Y, KAN H L, et al.Discovery of spoilage markers for chicken eggs using liquid chromatography-high resolution mass spectrometry-based untargeted and targeted foodomics[J].Journal of Agricultural and Food Chemistry, 2021, 69(14):4331-4341.
[54] VON EYKEN A, RAMACHANDRAN S, BAYEN S.Suspected-target screening for the assessment of plastic-related chemicals in honey[J].Food Control, 2020, 109:106941.
[55] BRAUN D, EISER M, PUNTSCHER H, et al.Natural contaminants in infant food:The case of regulated and emerging mycotoxins[J].Food Control, 2021, 123:107676.
[56] WANG T T, DUEDAHL-OLESEN L, LAURITZ FRANDSEN H.Targeted and non-targeted unexpected food contaminants analysis by LC/HRMS:Feasibility study on rice[J].Food Chemistry, 2021, 338:127957.
[57] CHEN H P, GAO G W, LIU P X, et al.Development and validation of an ultra performance liquid chromatography Q-Exactive Orbitrap mass spectrometry for the determination of fipronil and its metabolites in tea and chrysanthemum[J].Food Chemistry, 2018, 246:328-334.
[58] DASENAKI M E, THOMAIDIS N S.Multi-residue determination of 115 veterinary drugs and pharmaceutical residues in milk powder, butter, fish tissue and eggs using liquid chromatography-tandem mass spectrometry[J].Analytica Chimica Acta, 2015, 880:103-121.
[59] SHU N X, CHEN X Y, SUN X, et al.Metabolomics identify landscape of food sensory properties[J].Critical Reviews in Food Science and Nutrition, 2022:1-11.
[60] 陈丽君, 邓吉斯, 金子涵, 等. 基于代谢组学研究蔬菜面条在高温贮藏环境中的变化[J]. 食品与发酵工业, 2023, 49(16): 206-213.
CHEN L J, DENG J S, JIN Z H, et al. Metabolomics-based study of changes in vegetable noodles during storage under high-temperature conditions[J]. Food and Fermentation Industries, 2023, 49(16): 206-213.
[61] WANG H J, HUA J J, YU Q Y, et al.Widely targeted metabolomic analysis reveals dynamic changes in non-volatile and volatile metabolites during green tea processing[J].Food Chemistry, 2021, 363:130131.
[62] TOFFANO R, HILLESHEIM E, MATHIAS M, et al.Validation of the Brazilian healthy eating index-revised using biomarkers in children and adolescents[J].Nutrients, 2018, 10(2):154.
[63] FENG X Y, YU Q Q, LI B, et al.Comparative analysis of carotenoids and metabolite characteristics in discolored red pepper and normal red pepper based on non-targeted metabolomics[J].LWT, 2022, 153:112398.
[64] LIU Z H, SUN J H, TENG Z, et al.Identification of marker compounds for predicting browning of fresh-cut lettuce using untargeted UHPLC-HRMS metabolomics[J].Postharvest Biology and Technology, 2021, 180:111626.
[65] MONTORO P, D′URSO G, KOWALCZYK A, et al.LC-ESI/LTQ-Orbitrap-MS based metabolomics in evaluation of bitter taste of Arbutus unedo honey[J].Molecules, 2021, 26(9):2765.
[66] WINSTEL D, CAPELLO Y, QUIDEAU S, et al.Isolation of a new taste-active brandy tannin A:Structural elucidation, quantitation and sensory assessment[J].Food Chemistry, 2022, 377:131963.
[67] SEGLA KOFFI DOSSOU S, XU F T, YOU J, et al.Widely targeted metabolome profiling of different colored sesame (Sesamum indicum L.) seeds provides new insight into their antioxidant activities[J].Food Research International, 2022, 151:110850.
[68] KRITIKOU E, KALOGIOURI N P, KOLYVIRA L, et al.Target and suspect hrms metabolomics for the determination of functional ingredients in 13 varieties of olive leaves and drupes from greece[J].Molecules, 2020, 25(21):4889.
[69] MARVIN H J P, BOUZEMBRAK Y, JANSSEN E M, et al.A holistic approach to food safety risks:Food fraud as an example[J].Food research international, 2016, 89:463-470.
[70] JIA W, DONG X Y, SHI L, et al.Discrimination of milk from different animal species by a foodomics approach based on high-resolution mass spectrometry[J].Journal of Agricultural and Food Chemistry, 2020, 68(24):6 638-6 645.
[71] DIAS C, MENDES L.Protected designation of origin (PDO), protected geographical indication (PGI) and traditional speciality guaranteed (TSG):A bibiliometric analysis[J].Food Research International, 2018, 103:492-508.
[72] 任欣, 张敏, 关丽娜, 等. 东北稻谷挥发性有机化合物代谢组学指纹图谱的地理鉴别[C].中国食品科学技术学会第十八届年会摘要集, 2022:538-539.
[73] CAVANNA D, LOFFI C, DALL′ASTA C, et al.A non-targeted high-resolution mass spectrometry approach for the assessment of the geographical origin of durum wheat[J].Food Chemistry, 2020, 317:126366.
[74] CUBERO-LEON E, DE RUDDER O, MAQUET A.Metabolomics for organic food authentication:Results from a long-term field study in carrots[J].Food Chemistry, 2018, 239:760-770.
[75] STELLA R, MASTRORILLI E, PRETTO T, et al.New strategies for the differentiation of fresh and frozen/thawed fish:Non-targeted metabolomics by LC-HRMS (part B)[J].Food Control, 2022, 132:108461.
[76] ROCCHETTI G, REBECCHI A, DALLOLIO M, et al.Changes in the chemical and sensory profile of ripened Italian salami following the addition of different microbial starters[J].Meat Science, 2021, 180:108584.
[77] SCHÜTZ D, ACHTEN E, CREYDT M, et al.Non-targeted LC-MS metabolomics approach towards an authentication of the geographical origin of grain maize (Zea mays L.) samples[J].Foods, 2021, 10(9):2160.
[78] WANG K W, XU Z Z.Comparison of freshly squeezed, Non-thermally and thermally processed orange juice based on traditional quality characters, untargeted metabolomics, and volatile overview[J].Food Chemistry, 2022, 373:131430.
[78] WANG K W, XU Z Z.Comparison of freshly squeezed, Non-thermally and thermally processed orange juice based on traditional quality characters, untargeted metabolomics, and volatile overview[J].Food Chemistry, 2022, 373:131430.
[79] PÉREZ-M GUEZ R, CASTRO-PUYANA M, S
GUEZ R, CASTRO-PUYANA M, S NCHEZ-L
NCHEZ-L PEZ E, et al.Untargeted HILIC-MS-based metabolomics approach to evaluate coffee roasting process:Contributing to an integrated metabolomics multiplatform[J].Molecules, 2020, 25(4):887.
PEZ E, et al.Untargeted HILIC-MS-based metabolomics approach to evaluate coffee roasting process:Contributing to an integrated metabolomics multiplatform[J].Molecules, 2020, 25(4):887.
[80] ROCCHETTI G, GALLO A, NOCETTI M, et al.Milk metabolomics based on ultra-high-performance liquid chromatography coupled with quadrupole time-of-flight mass spectrometry to discriminate different cows feeding regimens[J].Food Research International, 2020, 134:109279.
[81] DONG X Y, WANG X J, XU X L, et al.An untargeted metabolomics approach to identify markers to distinguish duck eggs that come from different poultry breeding systems by ultra high performance liquid chromatography-high resolution mass spectrometry[J].Journal of Chromatography B, 2021, 1179:122820.
[82] DANIELE C, DANTE C, CHIARA D, et al.Egg product freshness evaluation:A metabolomic approach[J].Journal of Mass Spectrometry, 2018, 53(9):849-861.
[83] WINDARSIH A, WARMIKO H D, INDRIANINGSIH A W, et al.Untargeted metabolomics and proteomics approach using liquid chromatography-Orbitrap high resolution mass spectrometry to detect pork adulteration in Pangasius hypopthalmus meat[J].Food Chemistry, 2022, 386:132856.