麦芽是啤酒酿造的主要原料,其制备从原料大麦开始,包括浸麦、发芽和干燥3个环节。工业制麦对原料大麦的需求量巨大,多数情况下制麦企业需要从全球不同国家和地区进口大麦原料,如俄罗斯、德国、法国、乌克兰、澳大利亚等。大麦产区分布广泛,覆盖范围从接近北极圈的斯堪的维纳半岛到赤道附近的埃塞俄比亚和南美洲,从低于水平面的死海到安第斯山和喜玛拉雅山,从湿润温暖的西欧到北美、非洲和澳大利亚的部分干燥区域等[1]。
不同产区大麦的微生物组成千差万别。大麦表面被纤维质的谷壳包裹,成为众多细菌、酵母和丝状真菌的理想“栖息地”[2]。在制麦过程中,这些微生物会持续生长繁殖,并影响大麦的发芽性能以及麦芽和最终啤酒的品质。与细菌相比,真菌对制麦的影响会更明显,因为制麦过程中所需的水解大麦淀粉、蛋白质和半纤维素等高分子物质的酶,除了来源于大麦,还可由大麦籽粒表面的真菌提供[3]。目前,大麦原料中微生物具体的种属与胞外酶活之间的关系尚不清楚。另外,真菌污染也会降低大麦的发芽率、引发啤酒酵母超前絮凝和啤酒喷涌等问题[4-5]。因此,控制真菌污染一直是制麦行业的研究热点。
近年来,高通量分子测序技术因其具有测序深度大、准确度高等特点,已在不同食品领域的微生物群落分析中得到广泛应用[6]。本研究采用MiSeq高通量测序技术,并结合可培养的优势真菌分离技术对制麦过程中的真菌进行分析;同时结合理化指标分析和可培养真菌的产酶情况,揭示制麦过程真菌菌群结构演替规律及其对麦芽品质的影响,以期为工业制麦过程微生物的控制及制麦工艺优化提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
大麦:La Trobe(澳大利亚产区)。
1.1.2 培养基
孟加拉红培养基、PDA培养基和WL培养基,北京陆桥技术有限责任公司;YPD培养基(g/L):酵母粉10,蛋白胨20,葡萄糖20,琼脂20,调节pH至 6.0,于121 ℃、20 min条件灭菌后备用。
1.1.3 试剂
乳酸酚棉蓝染液,北京陆桥技术有限责任公司;E×Taq酶、dNTPs Mixture、10×Buffer I,TaKaRa公司;真菌DNA提取试剂盒,北京天根生化科技有限公司;羧甲基纤维素钠、酪蛋白、可溶性淀粉、刚果红(分析纯),上海沪试公司;木聚糖,阿拉丁试剂有限公司;NaCl、葡萄糖、(NH4)2SO4、MgSO4·7H2O、K2HPO4、KCl、NaNO3、FeSO4(分析纯),国药集团化学试剂有限公司。
1.2 仪器与设备
VB-7101-3/4微型制麦系统,德国SEEGER公司;S1000 TM PCR仪,美国Bio-Rad公司;Illumina HiSeq 2500测序平台,美国Illumina公司;Scope A1正置相差显微镜,德国ZEISS公司;IPP260低温培养箱,德国Memmert公司;Scan1200自动菌落计数仪,法国Interscience公司;AC2-4S1生物安全柜,新加坡ESCO公司;SQ510C高压蒸汽灭菌锅,日本YAMATO公司。
1.3 实验方法
1.3.1 制麦流程及样品采集
麦芽制备流程如下所示:
原料大麦→浸麦(15 ℃、30 h,浸麦度43%~44%)→发芽(15 ℃、96 h)→排潮(45~55 ℃、6 h)→干燥(65~85 ℃、11 h,降低水分至3%~5%)→除根(成品麦芽)
利用多点采样法分别对不同制麦工艺阶段的样品进行无菌取样,用于优势真菌的分离鉴定及真菌群落结构和多样性分析。大麦原料样品编号为M1、浸麦结束样品编号为M2、发芽24 h样品编号为M3、发芽结束样品编号为M4、排潮结束样品编号为M5、成品麦芽编号为M6。
1.3.2 菌种的分离与纯化
称取样品25 g,置于盛有225 mL灭菌生理盐水及玻璃珠的三角瓶中,于振荡培养箱中150 r/min振摇30 min,制得1∶10稀释度的样品匀液。将此样品依次进行10倍梯度稀释至10-6。取各梯度样品稀释液1 mL均匀涂布在孟加拉红培养基和YPD平板培养基上,28 ℃恒温倒置培养3~5 d。菌落计数后,根据菌落形态、大小及颜色等特点,挑选不同的单菌落划线接种至WL培养基和PDA培养基中。重复此纯化过程至少3次。将所得单菌落接种于YPD培养基和PDA培养基试管斜面,培养结束后,4 ℃保存备用。
1.3.3 真菌的形态观察
将保存的丝状真菌接种于PDA培养基上,28 ℃培养7 d;酵母菌接种于WL培养基上培养,28 ℃培养2~3 d。根据菌落生长情况及菌落形态,挑取单个酵母菌落制成水浸片,用于菌体形态观察;挑取丝状真菌菌落用乳酸酚棉蓝染液染色,用于菌丝及产孢结构观察。
1.3.4 真菌的分子生物学鉴定
取单个酵母菌落和单个丝状真菌菌落,接种于液体培养基中,28 ℃培养3~7 d。取1 mL菌液,4 000 r/min离心10 min后,弃去上清液,收集菌体沉淀,使用真菌DNA提取试剂盒提取菌株DNA。使用超微量紫外分光光度计检测所得基因组DNA的浓度和纯度。使用引物ITS1(5′-TCCGTAGGTGAACCTGCGG-3′)和ITS4(5′-TCCTCCGCTTATTGATATGC-3′)扩增真菌的ITS区。将PCR产物进行纯化后送至北京新时代众合科技有限公司进行测序。
将测序获得的有效序列在美国国立生物技术信息中心(National Center of Biotechnology Information, NCBI)的GenBank数据库中进行BLAST(Basic Local Alignment Search Tool)同源序列比对。根据比对结果,导出同源性≥98%的菌株ITS基因序列区。借助MEGA 11.0软件,采用邻接法(neighbor-joining method, NJ)构建系统发育树。
1.3.5 真菌多样性测定
采用E.Z.N.A.® soil DNA kit试剂盒提取样品中的微生物总DNA。使用引物ITS3F(5′-GCATCGATGAAGAACGCAGC-3′)和ITS4R(5′-TCCTCCGCTTATTGATATGC-3′)对总DNA进行PCR扩增。扩增体系与程序参照文献[7]。将扩增产物送至上海美吉生物医药科技有限公司利用Illumina MiSeq PE300平台进行双端测序。采用美吉云计算平台分析,对序列进行操作分类单位(operational taxonomic unit,OTU)聚类,对每条序列进行物种分类注释,比对UNITE 8.0数据库,基于分类学信息,在不同分类学水平上进行群落结构统计分析。测序原始数据已上传至NCBI数据库,SRA登录号为PRJNA907033。
1.3.6 蛋白酶、淀粉酶、木聚糖酶和纤维素酶活性检测
使用透明圈法检测所得菌株产胞外蛋白酶、淀粉酶、木聚糖酶和纤维素酶的能力。以3点接种法将活化的菌株接种到相应筛选培养基上,25 ℃培养3~7 d后观察菌落周围透明圈的出现情况[8-11]。
1.3.7 产酶菌株对麦芽品质的影响验证
将产酶菌株孢子悬液喷洒在浸麦结束后的大麦上,进入发芽环节。接种方法参照赵志超等[12]的描述并稍作修改:选取产胞外酶能力较强的菌株接种到PDA培养基中,28 ℃培养至孢子长出。将其冲洗到已灭菌的培养基中并调整孢子浓度至106个/mL,30 ℃萌发6 h。按照2%(mL/100 g)的接种量将孢子萌发液均匀喷洒在浸麦结束的大麦上进行发芽。麦芽浸出率和糖化力的测定参照QB/T 1686—2008中规定的方法。
1.3.8 数据处理
所有检测试验重复3次,使用Excel软件进行数据计算和分析。利用R软件(v3.5.3)和Origin 2018软件进行图表绘制。
2 结果与分析
2.1 制麦过程中酵母及丝状真菌数量变化情况
不同制麦阶段样品中的可培养酵母和丝状真菌的数量如图1所示。原料大麦(M1)自身携带一定数量的酵母和丝状真菌,其数量分别为1.4×104、7.1×103 CFU/g。随着制麦过程的进行,酵母和丝状真菌均出现先升高后下降的趋势。这与制麦过程中水分和温度的变化密切相关[13]。发芽结束时(M4)样品中酵母和丝状真菌数量达到峰值,分别为6.2×107、3.2×105 CFU/g。随着温度的上升和水分含量的下降,排潮阶段(M5)样品中微生物的数量下降。在成品麦芽(M6)中酵母和丝状真菌数量分别为2.1×105、1.1×104 CFU/g,约为制麦过程中微生物峰值的1/3。
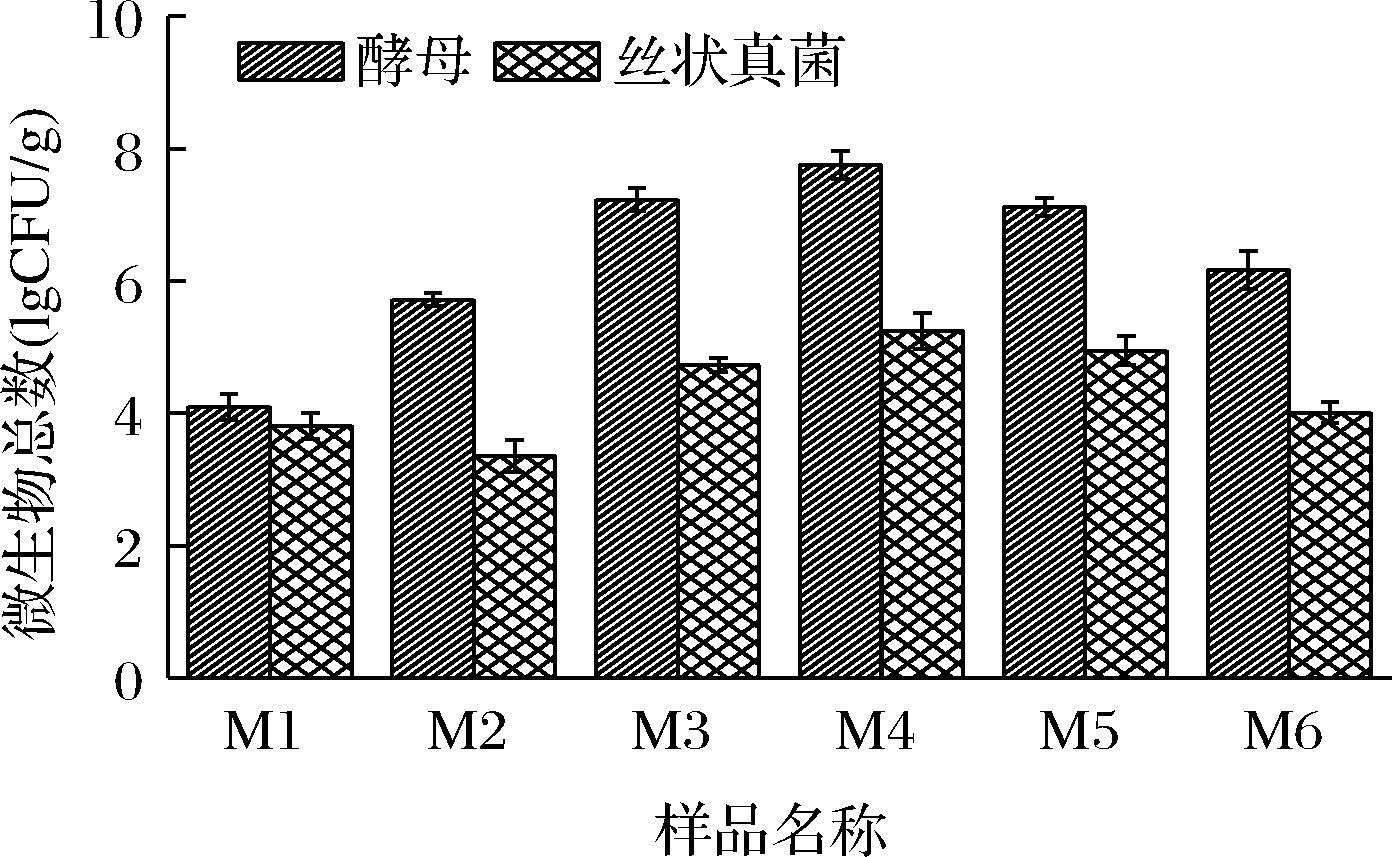
图1 制麦过程中酵母及丝状真菌数量变化情况
Fig.1 Amount changes in yeast and filamentous fungi during malting
2.2 制麦过程中可培养优势真菌的分离与鉴定
2.2.1 制麦过程中可培养真菌的分离与纯化
从制麦工艺的6个阶段样品中共分离得到174株真菌,包括酵母及酵母样真菌93株、丝状真菌81株。酵母及酵母样真菌菌落表现为表面光滑、不透明和黄油质的平坦菌落;蜡状、不透明,乳白色至奶油白;奶油质地和粉红色或红色等。菌落直径为1~4 mm,小部分菌落直径小于1 mm。光学显微镜下酵母菌的细胞形态主要有球形、近球形、卵圆形、椭圆形、柠檬形等形状,直径为1.5~5 μm。生长于PDA培养基上的丝状真菌,菌落形态为放线状皱纹或中心有脐状突起,质地绒状,略带絮状,也有茸毛状、絮状菌落,菌落颜色为浅黄色、褐色、灰色等。部分真菌菌落形态和显微结构如图2所示。

图2 制麦过程中部分可培养真菌菌落及显微结构图
Fig.2 Colony and microstructure photos of some fungi from malting
2.2.2 可培养真菌的分子鉴定
根据菌落形态及显微结构对分离得到的真菌进行初步分组,将菌落形态及显微结构相类似的菌株划分为一组。选取菌落形态差异较大的代表菌株进行ITS-rRNA基因序列测序分析,共测序78个菌株。如表1所示,ITS基因序列一致性比对结果表明,78株真菌的ITS基因序列与数据库中2个门(Basidiomycota和Ascomycota)10个目的25个模式菌株ITS序列的一致性均大于98%。其中,丝状真菌有10种,酵母状真菌有15种。丝状真菌主要分离自大麦原料样本,这可能与大麦原料水分过低,不适合酵母类微生物生长相关[14]。浸麦之后,大麦水分增加明显,促进了酵母快速繁殖和真菌休眠孢子萌发。因此,在浸麦结束和发芽初期(M2和M3)样品中的真菌以酵母为主,特别是担子菌门的酵母。这与担子菌门酵母在低于20 ℃的环境中生长良好密切相关[13-15]。与浸麦和发芽初期不同,发芽结束样本(M4)至成品麦芽(M6)中的优势真菌为子囊菌,与LAITILA等[14]的研究结果一致。
表1 制麦过程中可培养真菌ITS序列同源比对结果
Table 1 Results of ITS sequence homology alignment of culturable fungi from malting
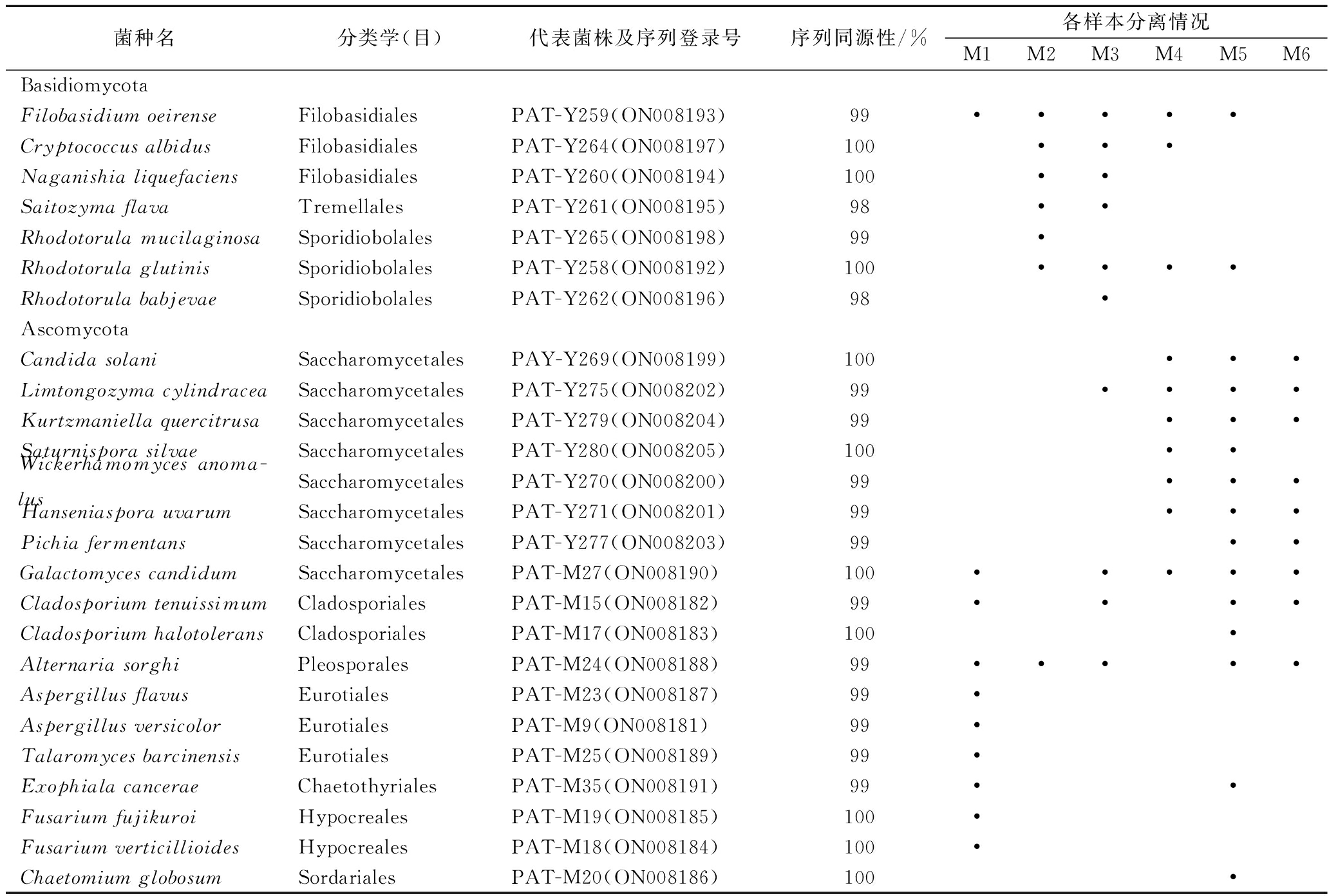
菌种名分类学(目)代表菌株及序列登录号序列同源性/%各样本分离情况M1M2M3M4M5M6BasidiomycotaFilobasidium oeirenseFilobasidialesPAT-Y259(ON008193)99•••••Cryptococcus albidusFilobasidialesPAT-Y264(ON008197)100•••Naganishia liquefaciensFilobasidialesPAT-Y260(ON008194)100••Saitozyma flavaTremellalesPAT-Y261(ON008195)98••Rhodotorula mucilaginosaSporidiobolalesPAT-Y265(ON008198)99•Rhodotorula glutinisSporidiobolalesPAT-Y258(ON008192)100••••Rhodotorula babjevaeSporidiobolalesPAT-Y262(ON008196)98•AscomycotaCandida solaniSaccharomycetalesPAY-Y269(ON008199)100•••Limtongozyma cylindraceaSaccharomycetalesPAT-Y275(ON008202)99••••Kurtzmaniella quercitrusaSaccharomycetalesPAT-Y279(ON008204)99•••Saturnispora silvaeSaccharomycetalesPAT-Y280(ON008205)100••Wickerhamomyces anoma-lusSaccharomycetalesPAT-Y270(ON008200)99•••Hanseniaspora uvarumSaccharomycetalesPAT-Y271(ON008201)99•••Pichia fermentansSaccharomycetalesPAT-Y277(ON008203)99••Galactomyces candidumSaccharomycetalesPAT-M27(ON008190)100•••••Cladosporium tenuissimumCladosporialesPAT-M15(ON008182)99••••Cladosporium halotoleransCladosporialesPAT-M17(ON008183)100•Alternaria sorghiPleosporalesPAT-M24(ON008188)99•••••Aspergillus flavusEurotialesPAT-M23(ON008187)99•Aspergillus versicolorEurotialesPAT-M9(ON008181)99•Talaromyces barcinensisEurotialesPAT-M25(ON008189)99•Exophiala canceraeChaetothyrialesPAT-M35(ON008191)99••Fusarium fujikuroiHypocrealesPAT-M19(ON008185)100•Fusarium verticillioidesHypocrealesPAT-M18(ON008184)100•Chaetomium globosumSordarialesPAT-M20(ON008186)100•
注:“•”表示这个样本中该真菌。
以25株代表菌株的ITS-rRNA序列(GenBank登录号为ON008181-ON008205)及与其相似度最高的模式菌株构建系统发育树。如图3所示,所得菌株与GenBank内相应同源性参考菌株聚为一支,且大多数菌株与同源性参考菌株聚为一支的支持率为100%,显示分离菌株与模式菌株存在很强的亲缘关系。

图3 基于ITS-rRNA序列构建的可培养真菌系统发育树
Fig.3 Phylogenetic tree of the culturable fungi based on the ITS-rRNA gene sequences
2.3 制麦过程中真菌菌群的Alpha多样性分析
制麦过程中真菌菌群的Alpha多样性分析结果如表2所示。每个样品的Coverage值均达到0.999,表明各样品文库的覆盖率高,测序结果足够体现样品中真菌菌群多样性的真实情况。发芽24 h样品(M3)的Shannon指标达到最高值1.58,意味着真菌的物种多样性在此阶段最高。Chao和Ace是用来评价菌群丰富度的指标,数值越大,丰富度越高[16]。Chao和Ace指数均呈现先下降后升高的趋势。在原料大麦(M1)中,Ace和Chao的数值分为52.5和54.0,均高于发芽结束的样品(M4),说明大麦原料中的真菌丰富度非常高。因此,有学者将大麦种子视为一个珍贵的天然菌种库[17]。浸麦后样品(M2)的Ace和Chao值分别降低至30.6和32.0,这可能是浸麦操作洗掉了大麦表面附着的真菌所致。随后,真菌的丰富度呈现上升趋势,直至最终成品麦芽(M6),Ace和Chao的值达到55.0和53.0的水平。这表明较高的焙焦温度未降低成品麦芽中真菌的丰富度。这与BIANCO等[18]的研究结果一致。
表2 制麦过程中真菌的Alpha多样性分析
Table 2 Alpha diversity analysis on the fungi from malting
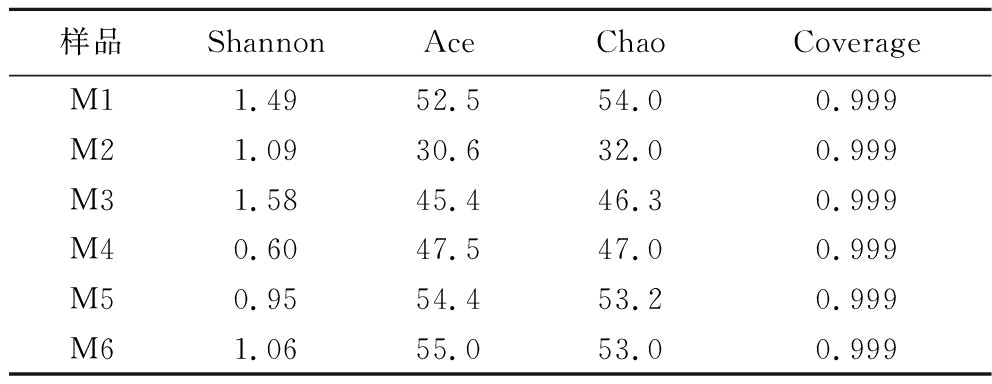
样品ShannonAceChaoCoverageM11.4952.554.00.999M21.0930.632.00.999M31.5845.446.30.999M40.6047.547.00.999M50.9554.453.20.999M61.0655.053.00.999
2.4 制麦过程中真菌菌群的Beta多样性分析
制麦过程6个不同阶段样品中真菌OTUs的聚类分析和主成分分析结果如图4所示。基于OTUs的聚类分析显示了不同样本中真菌菌群结构的相似性或差异关系[19]。如图4-A所示,大麦(M1)菌群单独为一支。浸麦结束后,大麦水分含量上升,酵母和休眠孢子萌发,真菌菌群相似性增加,因此,浸麦结束样品(M2)和发芽24 h样品(M3)真菌菌群聚为一支。发芽结束后,样品中真菌的种类和数量达到峰值。随后的排潮和焙焦环节因温度较高,不利于真菌种类和数量的增加,样品中的真菌群落具有较高相似度。因此,发芽结束样品(M4)、排潮结束样品(M5)和成品麦芽(M6)聚为一支。

A-聚类分析;B-主成分分析
图4 制麦过程6个不同阶段样品中真菌OTUs的 聚类分析及主成分分析
Fig.4 Cluster analysis and principal component analysis on fungus OTUs from the 6 different stages of malting
如图4-B所示,对制麦过程样品进行主成分分析,以样品点之间的距离来衡量其真菌群落结构组成的相似或差异程度。分析发现,大麦原料(M1)与各个样品距离相对较远,即真菌群落差异较大;浸麦结束样品(M2)和发芽24 h样品(M3)距离较近,发芽结束样品至成品麦芽(M4、M5、M6)聚在一起,与图4-A聚类分析结果相一致。聚类分析与主成分分析结果相互佐证,进一步表明制麦前期样品(M2和M3)与制麦后期样品(M4~M6)真菌群落差异明显,但各相邻样品的真菌群落结构差异性不明显,即进入制麦后真菌群落结构的变化是一个逐步演替的过程。
2.5 制麦过程中真菌菌群结构多样性分析
为深入揭示制麦过程中真菌菌群在各分类学水平上的组成情况,对不同制麦阶段样品中真菌菌群结构的门水平和属水平的特征进行分析(图5)。在门水平上,所有样品中真菌群落共包含2个门(图5-A)。其中,子囊菌门为大麦原料样品(M1)中的优势菌门,其相对丰度为81.79%,随后呈现先下降后上升的趋势,并保持在95.33%~97.82%的相对丰度,为整个制麦过程中的优势菌门。担子菌门在浸麦(M2)和发芽24 h(M3)阶段为优势菌门,浸麦阶段相对丰度达到98.17%,随后呈下降趋势。

A-门水平;B-属水平
图5 不同制麦阶段真菌群落结构图
Fig.5 Community structure of the fungi from different stage in the malting process
从不同制麦阶段的样品中共鉴定出57个真菌属,其中未被分类的酵母目(unclassified_o_Saccharomycetales)、线黑粉酵母属(Filobasidium)、链格孢属(Alternaria)等13个真菌属平均相对丰度≥1%,为优势菌属(图5-B)。大麦原料(M1)中相对丰度最高的优势真菌属为Alternaria,相对丰度为68.03%,浸麦结束(M2)后下降至1.3%,可能是由于浸麦水的冲洗,或是浸麦温度相对较低不适于该属的真菌生长所致。Filobasidium是浸麦结束(M2)和发芽前期(M3)样品中相对丰度最高的优势菌属,在大麦原料中该属的相对丰度仅为6.99%,但在浸麦结束和发芽前期分别上升至84.51%和69.79%。浸麦阶段和发芽初期为制麦过程中湿度条件最大的2个阶段,推测Filobasidium的生长可能和环境较高的湿度有关[20]。发芽结束(M4)、排潮结束(M5)和成品麦芽(M6)阶段的绝对优势菌属是unclassified_o_Saccharomycetales,并一直保持着较高的丰度。
2.6 制麦过程中可培养真菌产胞外酶分析
将分离得到的真菌接种至蛋白酶、淀粉酶、木聚糖酶及纤维素酶筛选培养基上,考察各菌株产胞外酶的情况,结果如表3所示。使用透明圈法筛选到了7株产蛋白酶的真菌、6株产淀粉酶的真菌、7株产木聚糖酶的真菌和12株产纤维素酶的真菌菌株。至少产1种胞外酶的真菌共有16株,其中,6株真菌具有同时产3种胞外酶的能力,2株真菌具有同时产4种胞外酶的能力。
表3 制麦过程中可培养真菌产胞外酶的能力
Table 3 The ability of the culturable fungi from malting to produce extracellular enzymes
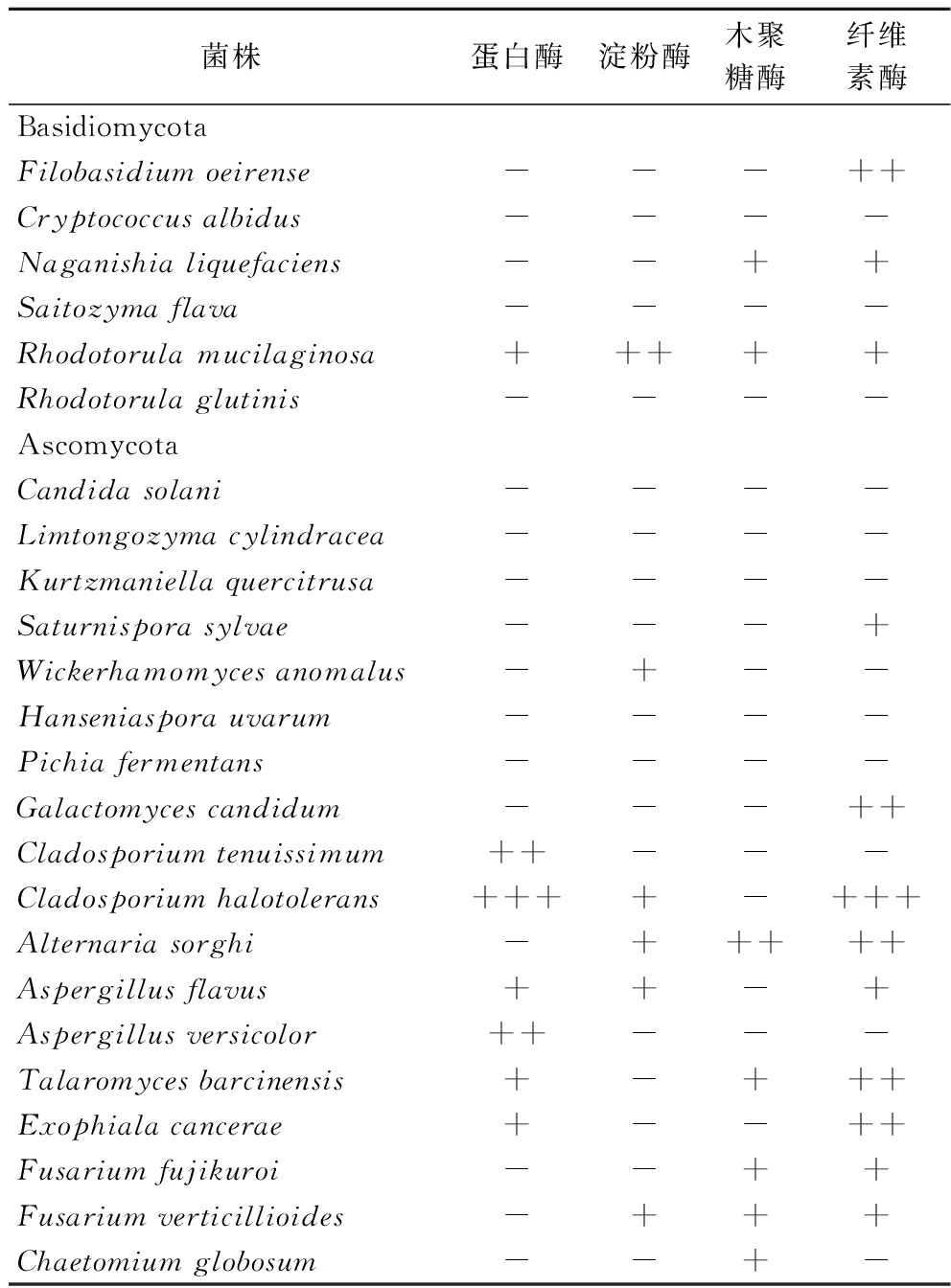
菌株蛋白酶淀粉酶木聚糖酶纤维素酶BasidiomycotaFilobasidium oeirense---++Cryptococcus albidus----Naganishia liquefaciens--++Saitozyma flava----Rhodotorula mucilaginosa+++++Rhodotorula glutinis----AscomycotaCandida solani----Limtongozyma cylindracea----Kurtzmaniella quercitrusa----Saturnispora sylvae---+Wickerhamomyces anomalus-+--Hanseniaspora uvarum----Pichia fermentans----Galactomyces candidum---++Cladosporium tenuissimum++---Cladosporium halotolerans++++-+++Alternaria sorghi-+++++Aspergillus flavus++-+Aspergillus versicolor++---Talaromyces barcinensis+-+++Exophiala cancerae+--++Fusarium fujikuroi--++Fusarium verticillioides-+++Chaetomium globosum--+-
注:“-”代表无透明圈产生;“+”代表有透明圈产生;“++”代表透明圈清晰;“+++”代表透明圈非常清晰。
2.7 产酶菌株对麦芽品质的影响
糖化力是指麦芽中酶分解自身胚乳淀粉的能力。浸出率是指每百克无水麦芽中可溶性物质的含量,在一定程度上反映了大麦籽粒发芽过程中干物质的损耗情况[21]。在制麦过程中接种产酶菌株后,麦芽糖化力和浸出率的变化情况如图6所示。结果表明,接种产胞外酶的菌株后,麦芽的糖化力均有所提升,以接种PAT-Y265(R.mucilaginosa)菌株的提升最为显著,这可能是因为该菌株产生的胞外淀粉酶提高了麦芽糖化力。由图6-B可知接种产胞外酶菌株也提升了麦芽的浸出率,特别是PAT-Y265(R.mucilaginosa)、PAT-M17(C.halotolerans)和PAT-M25(T.barcinensis)对麦芽浸出率的提升最为明显,达到了显著的水平。这可能是因为它们分泌的胞外纤维素酶、蛋白酶、木聚糖酶促进了麦芽胚乳细胞壁、蛋白质等生物大分子的水解[22],进而提高了麦芽的浸出率。产胞外酶菌株接种麦芽的结果再次表明,合理控制制麦过程中的真菌组成有助于提升麦芽糖化力、浸出率等品质指标。
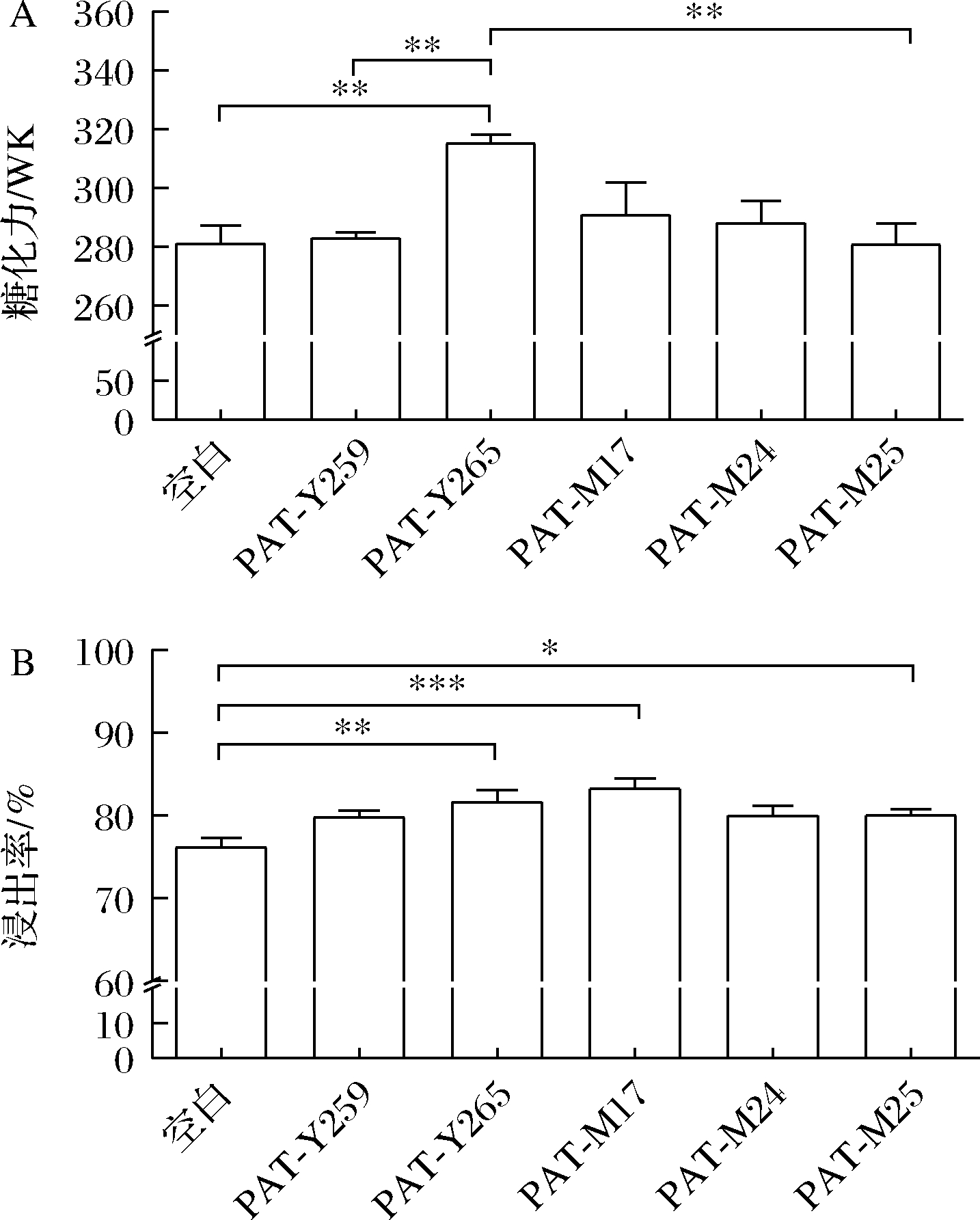
A-糖化力;B-浸出率
图6 产酶菌株对麦芽糖化力和浸出率的影响情况(P<0.05)
Fig.6 Effect of extracellular enzyme producing strains on malt diastatic power and malt extract (P<0.05)
3 结论
本研究利用传统分离培养方法,对制麦过程中不同阶段样品的真菌群落进行了分析。结果表明,制麦过程中酵母菌数量远高于丝状真菌,且酵母菌和丝状真菌呈现先升高后下降的趋势。利用分离培养法在不同制麦阶段采集的样品中分离到174株真菌,形态学及ITS-rRNA序列分析结果表明,这些真菌属于2门、17属、25种。其中,丝状真菌主要分离自大麦原料,在浸麦结束和发芽初期的样品中主要分离到担子菌门酵母,而发芽结束到成品麦芽样品中以子囊菌酵母为优势菌群。利用平板筛选法对制麦过程中可培养真菌的蛋白酶、淀粉酶、木聚糖酶和纤维素酶4种水解酶活性进行筛选,结果表明制麦过程中真菌群落对麦芽水解酶系有一定贡献作用。此外,选取了5株产胞外酶能力较高的菌株并将其应用到制麦过程中,所得成品麦芽的糖化力和浸出率均有所提升,进一步说明这些微生物在制麦过程中具有促进麦芽溶解的作用。
对样品的高通量测序结果表明,在浸麦结束(M2)时真菌物种丰富度最低,随后呈上升趋势。聚类分析与主成分分析结果相互佐证,制麦前期样品(M2和M3)与制麦后期样品(M4~M6)的真菌群落有明显差异,但各相邻样本的真菌群落结构差异性不明显,说明进入制麦后真菌群落结构的变化是一个逐步演替的过程。从制麦样品中共检出2真菌门、58真菌属。门水平上,大麦原料(M1)及制麦后期(M4~M6)样本以Ascomycota为优势门,而Basidiomycota为浸麦结束(M2)和发芽前期(M3)样本中的优势门,此结果与平板分离培养结果一致。制麦过程的优势菌属主要有unclassified_o_Saccharomycetales、Filobasidium和Alternaria等13属。
本研究利用分离培养结合高通量测序技术揭示了制麦过程中丰富的真菌群落多样性及其演替情况,这些真菌在制麦生态系统中扮演着重要的角色,对麦芽产品的质量有着重要影响,其具体功能性及安全性有待深入研究。此外,通过对制麦过程中真菌群落的分析,在丰富了制麦微生物资源的同时,也为麦芽质量控制以及麦芽食品安全控制提供了重要的理论依据。
[1] KAUR M, BOWMAN J P, STEWART D C, et al.The fungal community structure of barley malts from diverse geographical regions correlates with malt quality parameters[J].International Journal of Food Microbiology, 2015, 215:71-78.
[2] FLANNIGAN B.The Microbiota of Barley and Malt[M].3rd ed.New York:Kluwer Academic/Plenum Publishers, 2003.
[3] BECCARI G, SENATORE M T, TINI F, et al.Fungal community, Fusarium head blight complex and secondary metabolites associated with malting barley grains harvested in Umbria, central Italy[J].International Journal of Food Microbiology, 2018, 273:33-42.
[4] 王晋, 钱竹, 石维忱, 等.梨孢镰刀霉菌的疏水蛋白对啤酒喷涌的影响[J].食品与发酵工业, 2009, 35(2):31-34.
WANG J, QIAN Z, SHI W C, et al.The effect of Fusarium poae on beer-gushing[J].Food and Fermentation Industries, 2009, 35(2):31-34.
[5] 尹木. 制麦过程中霉菌对大麦发芽代谢谱的影响[D].大连:大连工业大学, 2018.
YIN M.The influence of mould on metabolism spectrum of barley germination during malting[D].Dalian:Dalian Polytechnic University, 2018.
[6] 向凡舒, 折米娜, 何萌, 等.基于DGGE和Illumina MiSeq技术解析恩施地区米酒细菌多样性[J].食品与发酵工业, 2019, 45(14):41-46;62.
XIANG F S, ZHE M N, HE M, et al.Bacterial community in Enshi rice wines analyzed by DGGE and Illumina MiSeq high-throughput sequencing[J].Food and Fermentation Industries, 2019, 45(14):41-46;62.
[7] CAO D M, LOU Y H, JIANG X J, et al.Fungal diversity in barley under different storage conditions[J].Frontiers in Microbiology, 2022, 13:895975.
[8] SUMARDI, EKOWATI C N, RISMAYANTI.The activity assay of protease, cellulase, amylase, xylanase and mannanase from Bacillus sp.as a candidate of probiotics[J].World Journal of Pharmaceutical and Life Sciences, 2019, 5(3):88-93.
[9] CARRASCO M, VILLARREAL P, BARAHONA S, et al.Screening and characterization of amylase and cellulase activities in psychrotolerant yeasts[J].BMC Microbiology, 2016, 16(1):1-9.
[10] HANKIN L, ANAGNOSTAKIS S L.The use of solid media for detection of enzyme production by fungi[J].Mycologia, 1975, 67(3):597-607.
[11] CARDER J H.Detection and quantitation of cellulase by Congo red staining of substrates in a cup-plate diffusion assay[J].Analytical Biochemistry, 1986, 153(1):75-79.
[12] 赵志超, 贠建民, 艾对元, 等.毛霉F-32产β-葡聚糖酶发酵条件优化及其对麦芽溶解性的影响[J].食品科学, 2013, 34(1):199-204.
ZHAO Z C, YUN J M, AI D Y, et al.Optimization of fermentation conditions for β-glucanase production by Mucor F-32 and its effect on malt solubility[J].Food Science, 2013, 34 (1):199-204.
[13] JUSTÉ A, MALFLIET S.Microflora during malting of barley:Overview and impact on malt quality[J].Brewing Science, 2011, 64(3):22-31.
[14] LAITILA A, WILHELMSON A, KOTAVIITA E, et al.Yeasts in an industrial malting ecosystem[J].Journal of Industrial Microbiology and Biotechnology, 2006, 33(11):953-966.
[15] LAITILA A, SARLIN T, RAULIO M, et al.Yeasts in malting, with special emphasis on Wickerhamomyces anomalus (synonym Pichia anomala)[J].Antonie Van Leeuwenhoek, 2011, 99(1):75-84.
[16] 万宁威, 雷帮星, 李彪, 等.白酸汤发酵过程中微生物多样性及有机酸动态变化研究[J].食品与发酵工业, 2022, 48(9):65-70.
WAN N W, LEI B X, LI B, et al.Microbial diversity and dynamic changes of organic acids during the fermentation of white sour soup[J].Food and Fermentation Industries, 2022, 48(9):65-70.
[17] BZIUK N, MACCARIO L, STRAUBE B, et al.The treasure inside barley seeds:Microbial diversity and plant beneficial bacteria[J].Environmental Microbiome, 2021, 16(1):20.
[18] BIANCO A, FANCELLO F, BALMAS V, et al.The microbiome of Sardinian barley and malt[J].Journal of the Institute of Brewing, 2018, 124(4):344-351.
[19] MA Y B, WANG W W, ZHANG H J, et al.Supplemental Bacillus subtilis DSM 32315 manipulates intestinal structure and microbial composition in broiler chickens[J].Scientific Reports, 2018, 8:15358.
[20] VAUGHAN A, O'SULLIVAN T, SINDEREN D.Enhancing the microbiological stability of malt and beer-A review[J].Journal of the Institute of Brewing &Distilling, 2005, 111(4):355-371.
[21] 董文斌. 大麦品质性状的差异性及籽粒品质与麦芽品质的相关性分析[D].扬州:扬州大学, 2018.
DONG W B.Differences of barley quality traits and correlations between grain and malting qualities[D].Yangzhou:Yangzhou University, 2018.
[22] CONTRERAS-JIMÉNEZ B, DEL REAL A, MILLAN-MALO B M, et al.Physicochemical changes in barley starch during malting[J].Journal of the Institute of Brewing, 2018, 125(1):10-17.