绿豆芽是绿豆种子经浸泡萌发后产生的具有丰富营养的蔬菜,含有大量的维生素、矿物质以及多种植物营养素(如硫代葡萄糖苷、酚类物质和异黄酮),兼具减少抗营养因子(如植酸、单宁酸和草酸盐)、增加矿物质的生物可及性的功效[1]。另外,在绿豆萌发为绿豆芽的过程中,部分蛋白质会分解为人体所需的氨基酸,为食用者提供丰富的营养物质。生物硒强化正是利用种子萌发过程以提高芽苗菜有机硒含量的一种简单而又高效的富硒食品开发技术[2]。种子萌发期间无机硒会转化为硒蛋白、硒多糖等有机硒,而其中硒蛋白的生物利用度可高达90%以上[3]。
当前,已有众多研究探讨了各类有机硒化合物的提取工艺,响应面(response surface methodology,RSM)和人工神经网络(artificial neural network,ANN)等技术被广泛应用于工艺优化和过程模拟之中[4]。RSM是一种通过一系列确定性实验,用多项式函数模拟真实极限状态曲面的方法。但RSM模型中的所有试验是基于单因素选择的试验点,当试验点的选择不合适时,则得不到理想的优化效果[5]。ANN是一种类似于生物结构的建模工具,通过寻找输入和输出数据中复杂的非线性关系,进而输出预测数据的方法[6]。遗传算法(genetic algorithm,GA)是“适者生存”概念启发式方法的优化算法之一,可对已构建的神经网络进行全局搜索与优化,进而获得最优解[7]。迄今为止,遗传算法-人工神经网络(GA-ANN)已被应用于如甜叶菊叶提取物[8]、多酚[9]、类黄酮[10]、硒蛋白[11]和多糖[12]等各种天然产物的提取工艺优化。有研究表明,与RSM方法相比,GA-ANN的应用可实现更高的预测准确性,并且更接近实验数据[13]。
本研究在单因素实验基础上,分别应用RSM和GA-ANN模型对蛋白提取工艺进行优化,并从多角度评估了2种模型的预测性能,在此基础上分析了富硒绿豆芽蛋白的氨基酸含量和有机硒形态,本研究为人工智能技术在功能性富硒食品的开发提供一定的理论参考依据。
1 材料与方法
1.1 材料与试剂
绿豆(内蒙古赤峰市所产),沈阳信昌粮食贸易有限公司。
牛血清蛋白,上海蓝季生物有限公司;考马斯亮蓝G250、氯化钠、氢氧化钠、乙醇、甲酸、盐酸、柠檬酸钠,国药集团化学试剂有限公司;乙腈,美国ACS公司;氨基酸混标,和光纯药工业株式会社;硒代甲硫氨酸,上海麦克林生化科技有限公司;硒代胱氨酸,上海源叶生物科技有限公司。
1.2 仪器与设备
CB-A330豆芽机,佛山市顺德区嘉壕实业有限公司;LD-Y500A粉碎机,上海顶帅电器有限公司;CARY300Conc紫外可见分光光度计,美国Varina公司;SH220F石墨消解仪,山东海能科学仪器有限公司;K9860全自动凯氏定氮仪,山东海能科学仪器有限公司;LA8080氨基酸自动分析仪,日本株式会社日立高新技术科学;1290-6470液相色谱仪质谱仪,美国Agilent公司。
1.3 实验方法
1.3.1 富硒绿豆芽粉的制备
选取大小均匀、完好无缺的绿豆,用分析天平称重后清洗干净[14]。然后使用10 mg/L的硒溶液浸泡绿豆,料液比为1∶3(g∶mL)。将浸泡后的绿豆置于豆芽机中进行避光培养3 d,取出放入45 ℃的电热鼓风干燥箱内烘干24 h,用粉碎机粉碎,过60目筛后放入自封袋于干燥器内保存。
1.3.2 富硒绿豆芽蛋白提取溶剂的选择
参考徐亚等[15]的方法测定上清液中蛋白含量,富硒绿豆芽粉中的蛋白含量参考GB 5009.5—2016《食品安全国家标准 食品中蛋白质的测定》中的凯氏定氮法。称取4份富硒绿豆芽粉各1 g于4个离心管中,按液料比20∶1(mL∶g)分别向离心管中加入纯水、NaCl溶液(0.5 mol/L)、乙醇溶液(体积分数75%)、碱液(0.1 mol/L NaOH溶液),在50 ℃下提取60 min后,10 000 r/min离心10 min,测定上清液内蛋白含量,根据4种溶剂的蛋白提取率来选择提取溶剂。蛋白质提取率计算如公式(1)所示:
蛋白质提取率![]()
(1)
式中:M1为上清液中蛋白含量,%,M2为富硒绿豆芽粉中蛋白含量,%。
1.3.3 单因素试验设计
选取碱液浓度(0.01、0.05、0.10、0.15、0.20 mol/L)、液料比(20∶1、30∶1、40∶1、50∶1、60∶1,mL∶g)、提取温度(30、40、50、60、70 ℃)、提取时间(30、60、90、120、150 min)和提取次数(1、2、3次)5个因素进行单因素试验,探究其对蛋白质提取率的影响。
1.3.4 响应面试验设计
根据单因素实验结果,设计4因素3水平的响应面实验,探究因素水平组合对蛋白质提取率的影响,确定最优提取条件。
1.3.5 人工神经网络建模
选择响应面模型中的89个数据构建ANN模型,其中模型训练使用70%的数据,验证训练使用15%,其余的用于测试。计算模型各参数以评估模型的预测和优化性能,其主要系数包括:相关系数(R2)、均方根误差(root mean square error,RMSE)、绝对平均偏差(absolute average deviation,AAD)和预测标准误差(standard error of prediction,SEP),R2越高,RMSE、AAD和SEP越低,则表明所建立的模型越可靠[16]。R2、RMSE、AAD和SEP的计算分别如公式(2)~公式(5)所示:
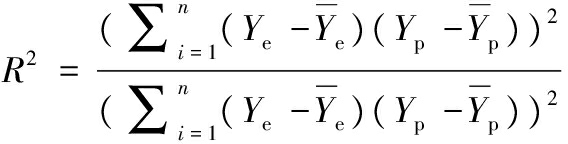
(2)
(3)
(4)
(5)
式中:n代表样本数,Ye代表实验值,Yp代表预测值,变量上的“-”表示相关变量值的平均值。
1.3.6 GA优化人工神经网络模型
以ANN的输出值(蛋白质提取率)为适应度函数值,根据适应度值从每一代的个体中不断地进行选择、交叉和变异,直至获得最优个体,并预测相应的提取条件(碱液浓度、提取温度、提取时间、液料比)。隐含层神经元的个数设为3~12,遗传算法的最大进化代数设为100,种群大小为50,交叉概率与变异概率分别为0.8和0.2[11]。在最优提取工艺下提取蛋白的得率参考YANG等[11]的方法。
1.3.7 绿豆芽蛋白中氨基酸含量的测定
氨基酸含量参照GB 5009.124—2016中的方法进行测定。
1.3.8 绿豆芽蛋白中硒代氨基酸含量的测定
硒代氨基酸含量采用液相色谱仪质谱仪进行检测。首先称取一定量样品于离心管中,然后加入5 mL 的Tris-HCl缓冲溶液(30 mmol/L)和20 mg胃蛋白酶XIV,混匀,放入37 ℃恒温箱中孵育24 h,取出冷却后10 000 r/min离心10 min,取上清液过0.45 μm 微孔滤膜后上机检测。仪器条件如下:色谱柱:C18(4.6 mm×250 mm,5 μm);采集模式:ESI+;柱温:30 ℃;进样量:2 μL;流速:0.3 mL/min;流动相:0.1%甲酸水溶液(A相)和乙腈(B相);梯度洗脱:0 min:67% A+33% B;0.6 min:67% A+33% B;1.3 min:63% A+37% B;2.8 min:62.5% A+37.5% B;3.0 min:57% A+43% B;3.8 min:90% A+10% B;4.5 min:90% A+10% B;4.8 min:67% A+33% B。
1.4 数据处理
使用IBM SPSS statistic 26.0软件进行单因素方差分析。采用Design-Expert ver 13.0软件设计响应面试验,并使用Matlab R 2016 a构建人工神经网络模型。实验结果用Graphpad prism 8.0.2和Origin 2021软件进行绘图。
2 结果与分析
2.1 蛋白提取溶剂的选择
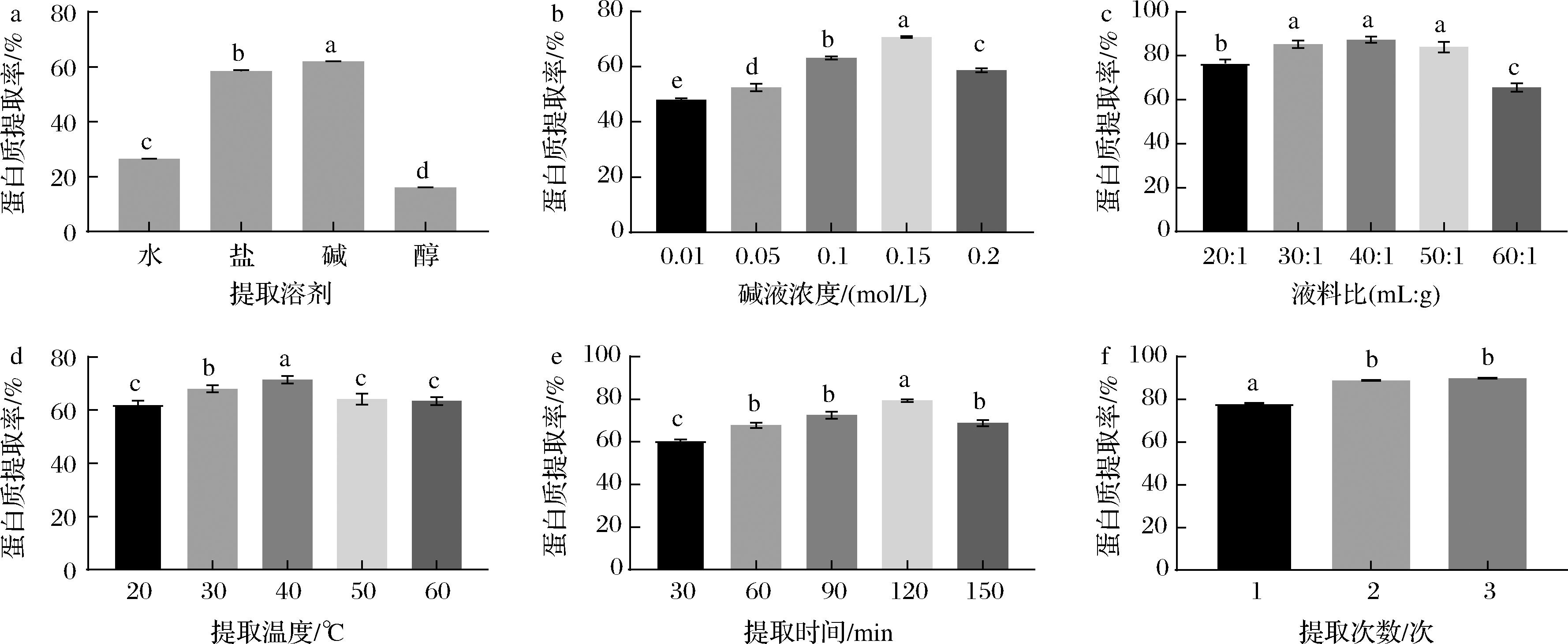
由图1-a可知,富硒绿豆芽粉经纯水、NaCl溶液(0.5 mol/L)、碱液(0.1 mol/L NaOH溶液)、乙醇溶液(体积分数75%)提取,蛋白质提取率分别为26.44%、58.50%、61.87%、15.99%。由以上数据可知,在4种提取溶剂中,碱液的蛋白提取率最高,其次是NaCl溶液,然后为纯水,而醇溶液的蛋白提取率最低,且不同溶剂组蛋白质提取率间存在显著性差异(P<0.05)。结合前人相关研究推测,之所以碱液的蛋白提取率最高,可能主要是因为绿豆芽粉中谷蛋白含量较高,而谷蛋白难溶于纯水、乙醇或中性盐溶液,但较易溶于碱[17]。因此,基于当前的研究结果,后续蛋白的提取均选择碱液为提取溶剂以获得更高的蛋白质得率。
a-提取溶剂;b-碱液浓度;c-液料比;d-提取温度;e-提取时间;f-提取次数
图1 各单因素对蛋白质提取率的影响
Fig.1 Effects of single factors on protein extraction rate
注:不同小写字母表示差异显著(P<0.05)。
2.2 富硒绿豆芽蛋白提取单因素实验
由图1-b可知,随着碱液浓度的增加,蛋白质提取率先升高后降低,在碱液浓度为0.15 mol/L时,蛋白质提取率最高,各组间蛋白质提取率存在显著性差异(P<0.05)。在碱液浓度为0.01~0.15 mol/L时,可能是由于蛋白被水解,增加了蛋白的亲水性,因此提取率升高,但高碱浓度下会加强美拉德反应,造成部分氨基酸的降解,甚至会产生有毒物质[18]。因此选择碱液浓度为0.1、0.15、0.2 mol/L进行响应面实验。
由图1-c可知,蛋白质提取率随着液料比的增加先升高后降低,且各组间蛋白质提取率存在显著性差异(P<0.05)。当液料比为40∶1(mL∶g)时,蛋白质提取率最高。可能的原因是,当液料比较低时,溶液的黏度和蛋白的浓度比较大,固相与液相浓度差较小,流动性小,导致蛋白质提取率较低;当液料比增加到40∶1时,绿豆芽粉与提取溶剂接触面积增大,扩散程度就会增大;但当液料比大于40∶1后,蛋白质提取率表现出降低的趋势,可能是因为过高的液料比降低了提取体系的黏度,使得溶出的蛋白在离心过程中更易沉降,一定程度上降低了上清液中的蛋白浓度,导致最终偏低的蛋白质提取率[11]。因此,选择最佳液料比为30∶1、40∶1、50∶1进行响应面实验。
如图1-d可知,富硒绿豆芽蛋白质提取率随着温度的升高先增加后降低,在提取温度为40 ℃时,蛋白质提取率最高。可能是因为适当的升温使蛋白质分子结构舒展、运动速度加快,有助于蛋白质与水的相互作用,蛋白质提取率有所提高。但高温会使部分蛋白结构发生改变而产生沉淀,溶解在溶液中的蛋白就会降低[19]。因此,选择最佳提取温度为30、40、50 ℃进行响应面实验。
如图1-e所示,当提取时间在30~120 min时,随着提取时间的延长,蛋白质提取率逐渐升高,但继续延长至150 min时,蛋白质提取率开始降低,且存在显著性差异(P<0.05)。可能的原因是前期溶液中蛋白含量低,随着提取时间的延长,样品中的蛋白逐渐溶出;但过度延长提取时间,有可能导致充分溶出的部分蛋白因变性而沉降,或发生一定程度的降解,最终致使上清液中的蛋白浓度降低[11]。因此,选择最佳提取时间为60、90、120 min进行响应面实验。
如图1-f所示,随着提取次数的增加,蛋白质提取率逐渐升高,第2次的蛋白提取率显著高于第1次(P<0.05),但第3次与第2次的蛋白质提取率已无显著性差异(P>0.05),表明第3次只能提取少量蛋白质,这与YANG等[11]的研究结果一致。因此,选择蛋白的最佳提取次数为2次。
2.3 响应面试验与人工神经网络训练结果
2.3.1 响应面优化蛋白提取条件
在单因素的实验基础上,探究自变量及各因素间的交互作用对蛋白质提取率的影响。采用Design-Expert 13.0软件进行多元回归方程拟合,得到回归方程:Y=81.16+0.58A-7.38B-1.82C+3.88D+3.30AB-6.02AC-3.67AD-0.87BC-1.28BD-4.26CD-5.65A2-8.82B2-2.31C2-5.56D2。响应面试验设计方案及结果见表1。
对蛋白质提取率的回归模型进行方差分析,回归模型方差分析结果见表2。由表2可知,该模型的F值为43.14,P值小于0.000 1,表示模型极显著,失拟项P值为0.334 7(P>0.05),说明实验受失拟因素的影响较小。模型的相关系数R2为0.9773,调整后的R2与预测的R2相差小于0.2,表明该模型可以对富硒绿豆芽蛋白的提取条件进行分析。表3中B、C、D、A2、B2、C2、D2、AB、AC、AD、CD具有极显著性(P<0.01),而BC、BD对应的P值大于0.05,表明BC和BD的交互作用对蛋白质提取率的影响不显著。比较F值的大小可以得出各因素对蛋白质提取率的影响大小依次为B>D>C>A。图2体现了各因素之间交互作用对蛋白质提取率的影响。由图2可知,AB、AC、AD和CD的曲面较为陡峭,等高线形状趋近于椭圆形,说明两两因素交互作用明显。BC、BD的曲面较平滑,因此交互作用不显著。
表1 响应面试验设计方案及结果
Table 1 The design scheme and its results of the response surface test

实验号因素蛋白质提取率/%碱液浓度(A)/(mol/L)提取温度(B)/℃提取时间(C)/min液料比(D)/(mL∶g)实际值预测值10.15409030∶166.1966.9720.203012040∶156.5656.5930.104012030∶160.1361.8340.153012030∶156.6654.2450.10409040∶170.5468.4260.155012030∶171.0871.5770.20409040∶181.5781.6280.204012050∶171.2170.7390.15409050∶181.8683.25100.15309040∶162.7063.60110.154012040∶182.1381.16120.15509040∶181.1180.11130.104015040∶178.2876.82140.154012040∶181.0681.16150.105012040∶170.0570.20160.153012050∶166.4564.56170.155012050∶175.7576.76180.15015040∶159.4761.70190.154012040∶179.5281.16200.154015050∶171.6871.08210.104012050∶176.3576.92220.154015030∶173.0671.86230.154012040∶180.0281.16240.103012040∶160.8862.03250.204015040∶165.2265.93260.205012040∶178.9377.95270.204012030∶169.6670.32280.155015040∶174.4074.72290.154012040∶183.0681.16
表2 回归模型方差分析
Table 2 Regression model analysis of variance
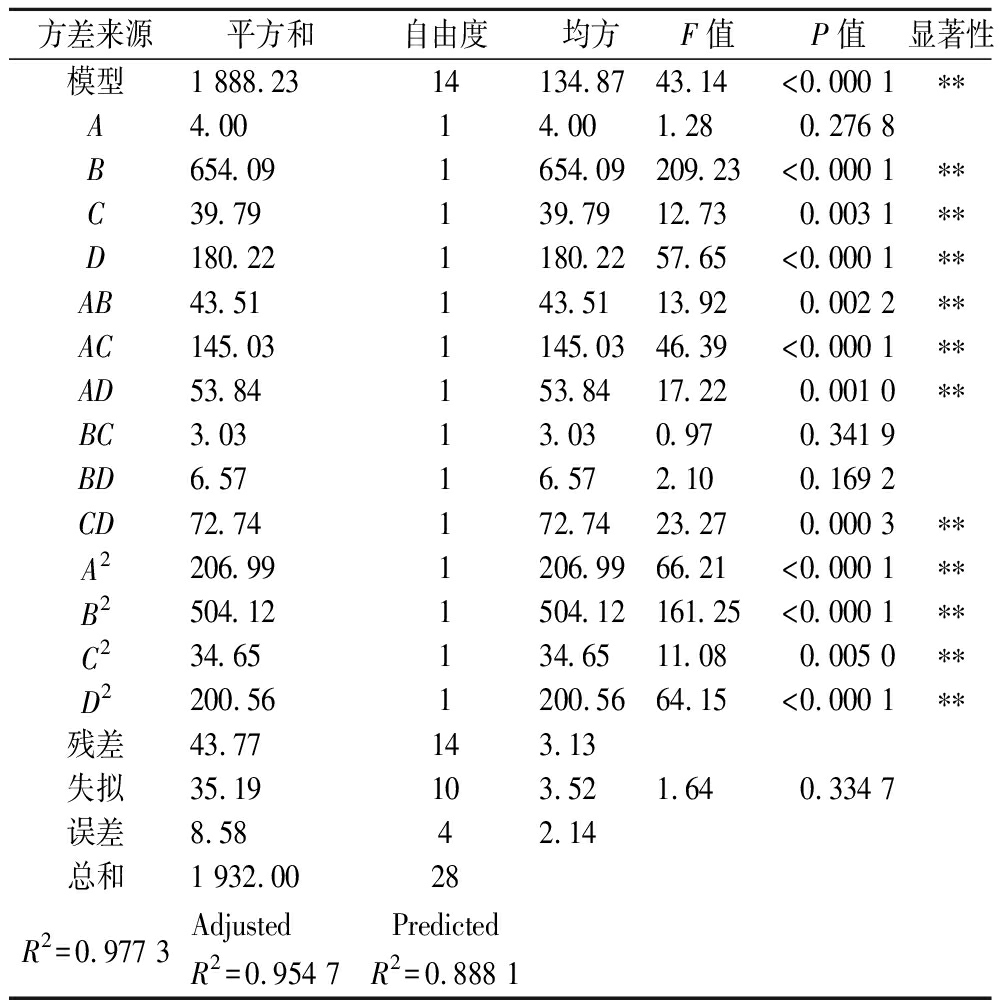
方差来源平方和自由度均方F值P值显著性模型1 888.2314134.8743.14<0.000 1∗∗A4.0014.001.280.276 8B654.091654.09209.23<0.000 1∗∗C39.79139.7912.730.003 1∗∗D180.221180.2257.65<0.000 1∗∗AB43.51143.5113.920.002 2∗∗AC145.031145.0346.39<0.000 1∗∗AD53.84153.8417.220.001 0∗∗BC3.0313.030.970.341 9BD6.5716.572.100.169 2CD72.74172.7423.270.000 3∗∗A2206.991206.9966.21<0.000 1∗∗B2504.121504.12161.25<0.000 1∗∗C234.65134.6511.080.005 0∗∗D2200.561200.5664.15<0.000 1∗∗残差43.77143.13失拟35.19103.521.640.334 7误差8.5842.14总和1 932.0028R2=0.977 3AdjustedR2=0.954 7PredictedR2=0.888 1
注:**代表数值有极显著差异,P<0.01。
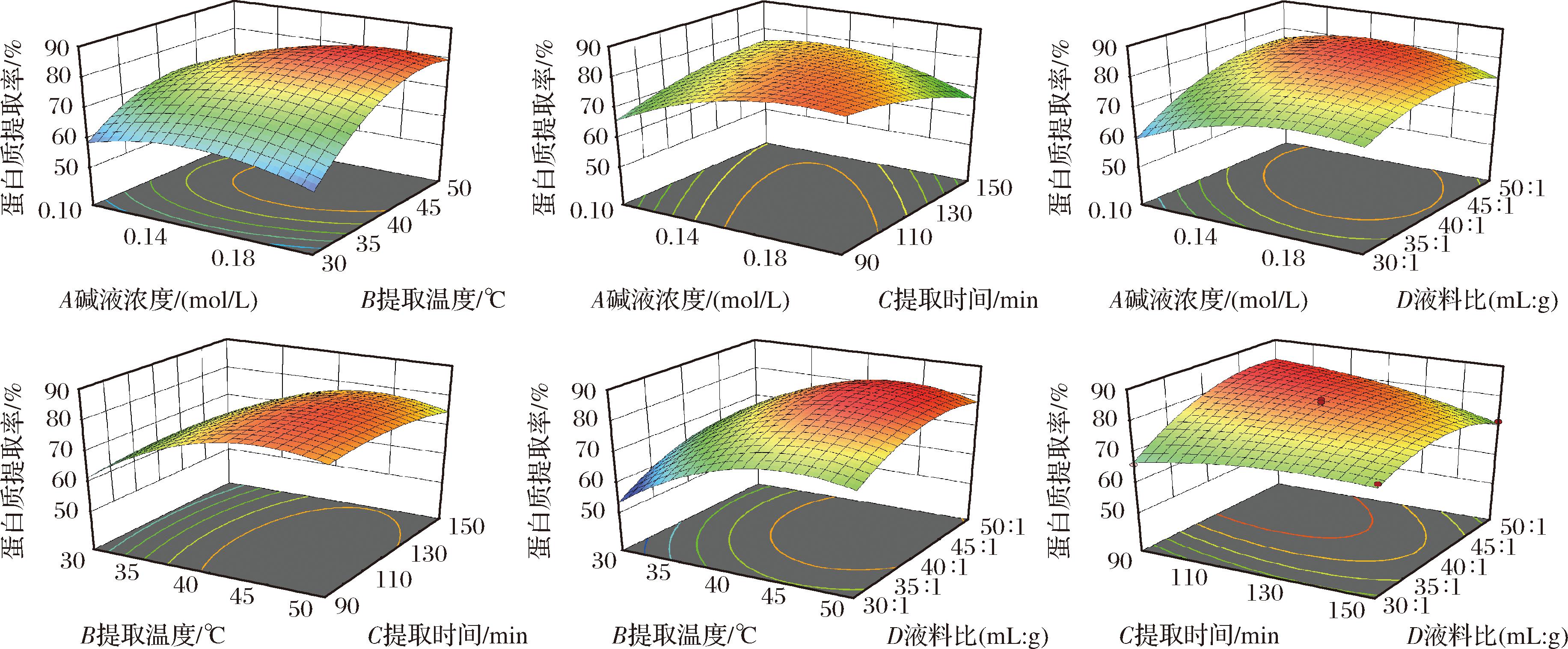
图2 各实验因素交互作用对蛋白质提取率的影响
Fig.2 Effects of interaction of various experimental factors on protein extraction rate
响应面模型分析得到最佳提取条件为:碱液浓度0.15 mol/L、提取温度45.46 ℃、提取时间102.72 min、液料比46.28∶1(mL∶g)。预测蛋白质提取率为84.69%。为便于实际操作,将提取条件调整为:碱液浓度0.15 mol/L、提取温度45 ℃、提取时间103 min、液料比46∶1(mL∶g)。在此条件下经过3次重复实验,得到蛋白质提取率为86.34%,预测值与实际值相差1.65%。
2.3.2 人工神经网络模型训练相关性评价
通过人工神经网络模型训练发现隐含层的个数为8时,均方误差最小,因此确定神经网络的最佳拓扑结构为4-8-1,如图3所示。由图4可知,训练集(0.996)、验证集(0.996 31)、测试集(0.992 82)和全部数据集(0.995)的相关系数(R2)值表明,ANN模型具有很好的回归和拟合能力。因此,说明人工神经网络对训练、验证和测试的预测能力和模型在预测响应方面的精确度较高。
2.3.3 利用GA优化及验证蛋白质提取工艺结果
由图5可知,随着迭代次数的增加,蛋白质提取率呈现曲折上升的趋势,种群迭代进化87代以后,蛋白质提取率达到最大值,适应度函数趋于平稳。预测最优蛋白质提取率为91.62%,提取条件为碱液浓度0.15 mol/L,提取温度42.52 ℃,提取时间133.36 min,液料比49.99∶1(mL∶g)。为便于实际操作,将各参数值调整为:碱液浓度0.15 mol/L,提取温度43 ℃,提取时间133 min,液料比50∶1(mL∶g)。重复提取3次后蛋白质提取率为90.32%,与预测值相差1.30%。
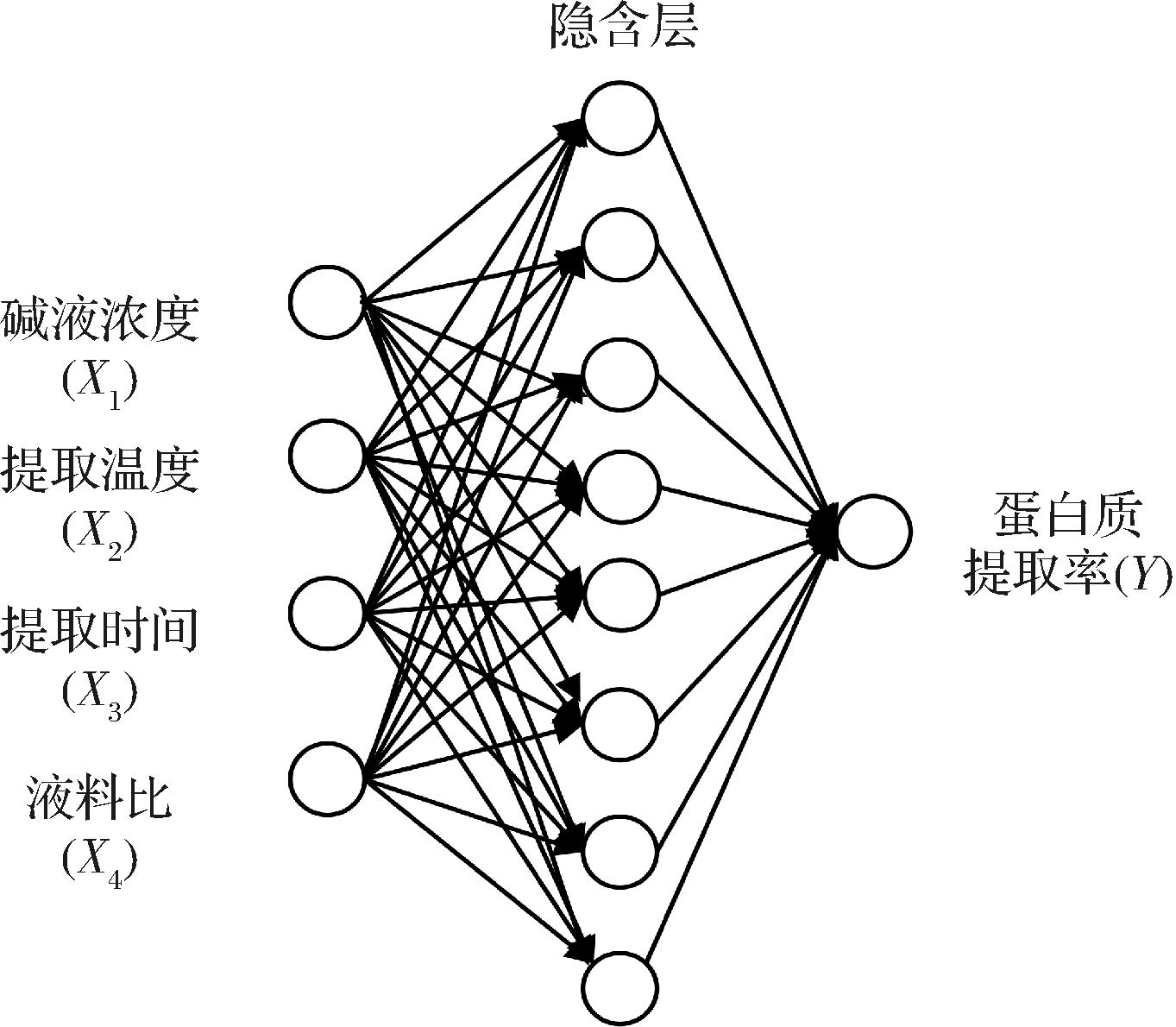
图3 神经网络拓扑结构
Fig.3 Neural network topology architecture
2.4 响应面与人工神经网络对比分析
2.4.1 模型参数的比较以及模型预测值与实验值的比较
表3为RSM和ANN的模型参数,由表3可以看出,2种模型的相关系数R2均超过0.95,表明模型具有较好的拟合度,但ANN的R2要高于RSM,且RMSE、AAD、SEP的值更低。因此,与RSM模型相比,人工神经网络模型具有更好的建模能力。图6描述了RSM和ANN的实验值和预测值。由图6可以看出,ANN的预测值与实验值更接近,表明了ANN模型的预测精度更高。
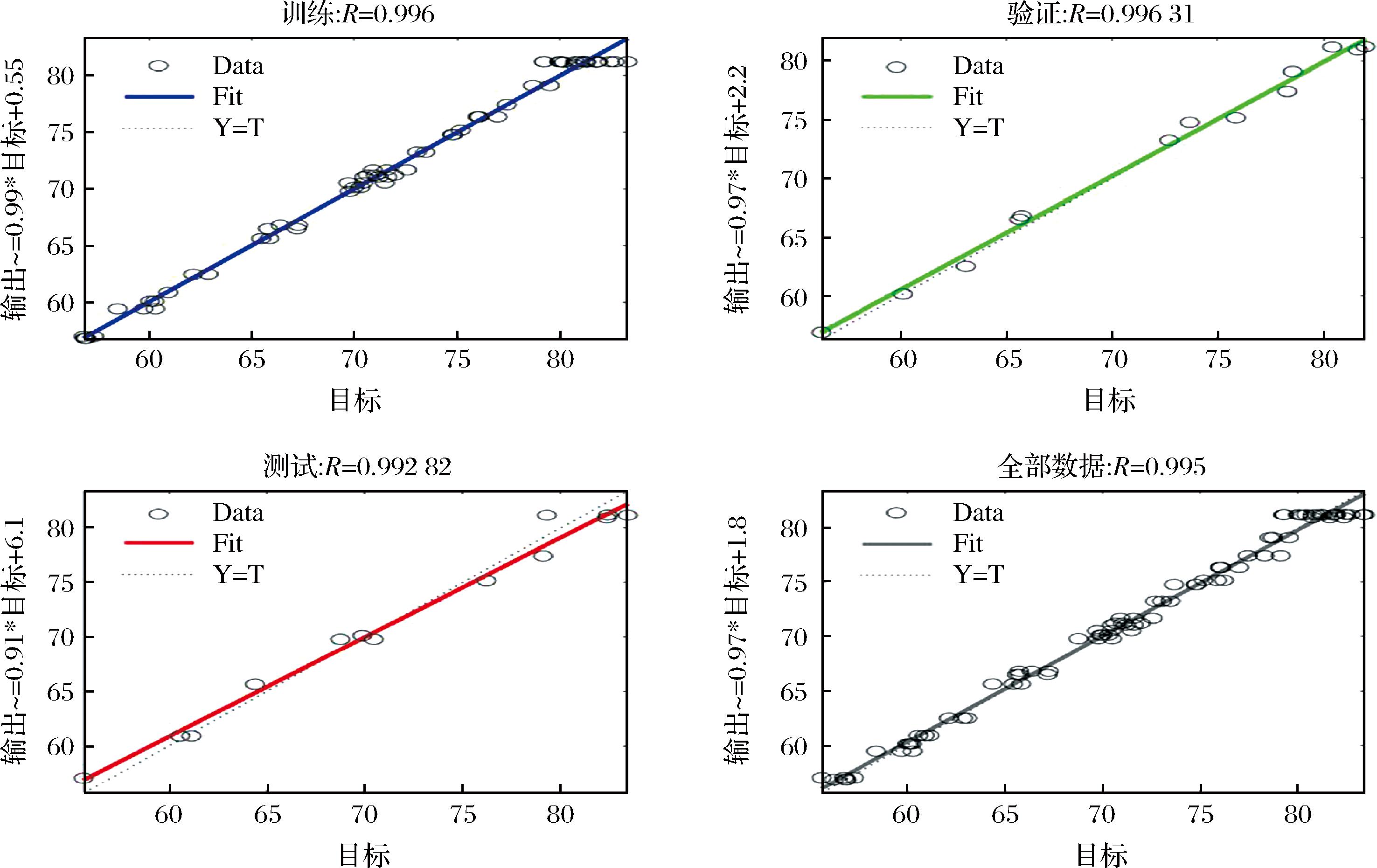
a-训练集;b-验证集;c-测试集;d-全部数据集
图4 蛋白质提取率的目标输出和网络输出的回归分析
Fig.4 Regression analysis of target output and network output of protein extraction rate
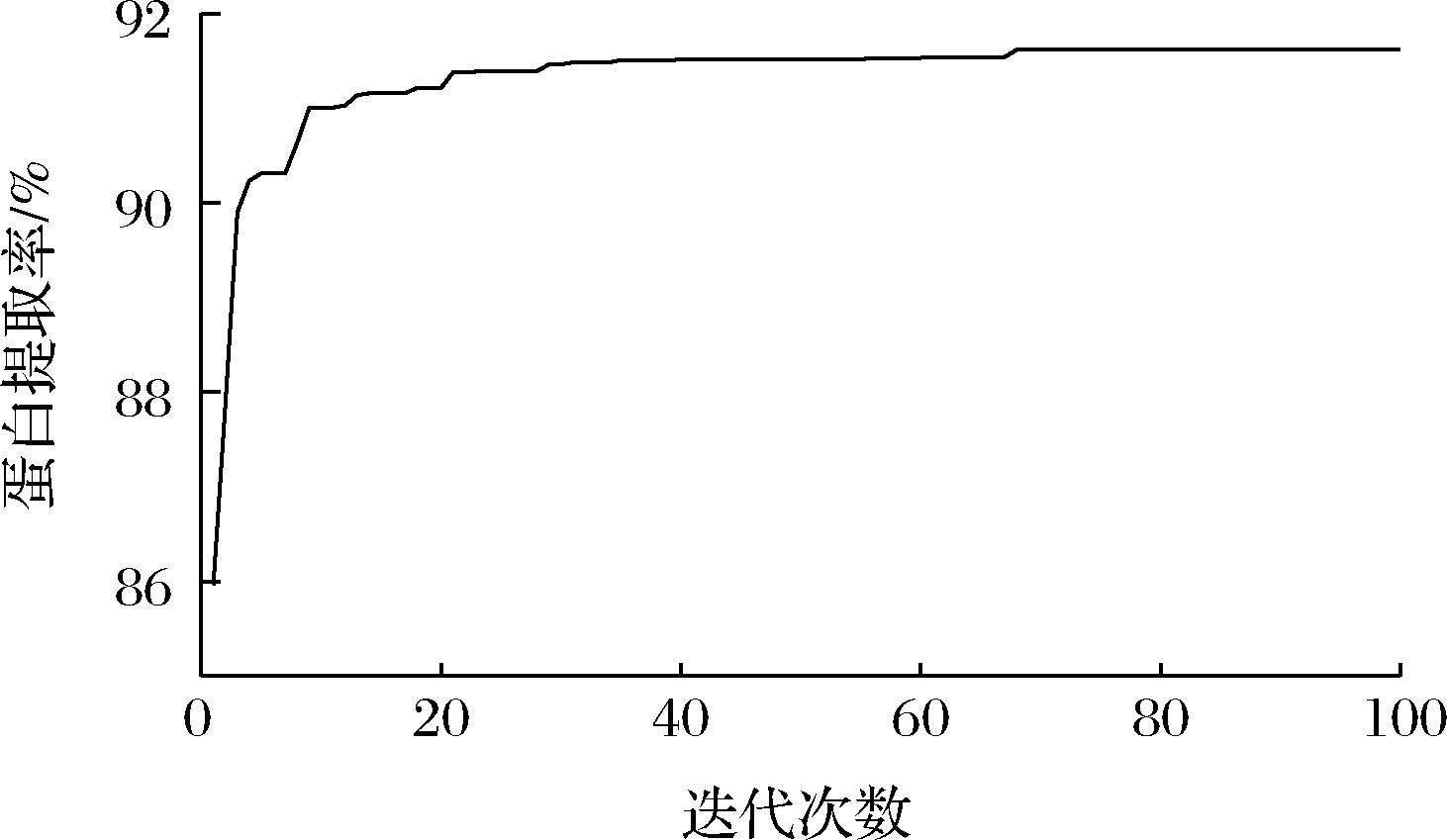
图5 种群适应度值(蛋白质提取率)迭代变化曲线
Fig.5 Iteration curve of population fitness value (protein extraction rate)
表3 RSM和ANN模型参数的比较
Table 3 Comparison of RSM and ANN model parameters
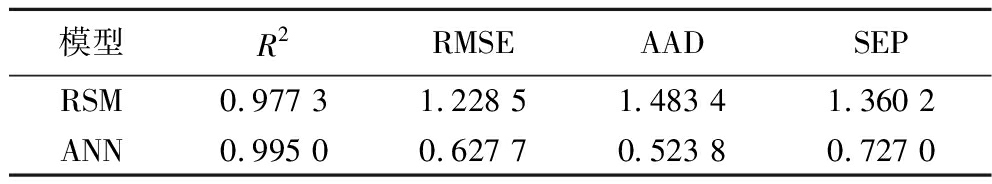
模型R2RMSEAADSEPRSM0.977 31.228 51.483 41.360 2ANN0.995 00.627 70.523 80.727 0

图6 RSM和ANN的预测值与实验值的对比
Fig.6 Comparison of predicted and experimental values of RSM and ANN
2.4.2 模型最优解的比较
RSM和GA-ANN分析得到最佳提取条件如表4所示,由表4可以看出,RSM的预测提取率与实验提取率相差1.65%,GA-ANN的预测提取率与实验提取率相差1.30%,GA-ANN预测值与实验值的差值较RSM更小。表明了与RSM相比,GA-ANN模型更适合用来预测富硒绿豆芽蛋白的提取条件,最佳提取条件为:碱液浓度0.15 mol/L,提取温度43 ℃,提取时间133 min,液料比50∶1(mL∶g)。在此条件下提取蛋白的得率为62.23%。
表4 两种模型预测最优条件下的对比
Table 4 Comparison of the two models under optimal conditions
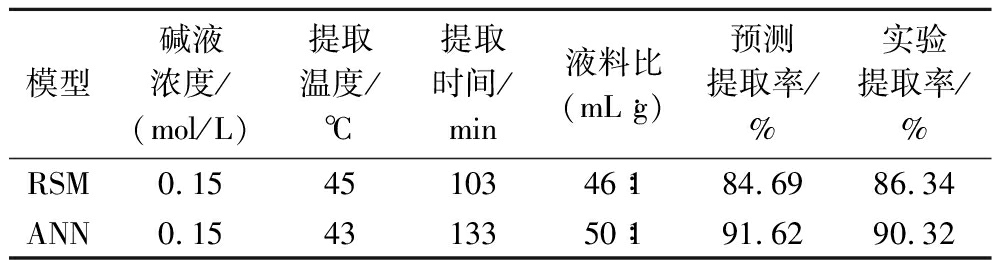
模型碱液浓度/(mol/L)提取温度/℃提取时间/min液料比(mL∶g)预测提取率/%实验提取率/%RSM0.154510346∶184.6986.34ANN0.154313350∶191.6290.32
2.5 绿豆芽蛋白中氨基酸的营养评价
蛋白质中氨基酸的种类、数量及组成比例决定着其营养价值的高低[20]。联合国粮农组织/世界卫生组织(Food and Agriculture Organization/World Health Organization, FAO/WHO)提出理想蛋白质中必需氨基酸含量与氨基酸总量的比值为0.4左右,必需氨基酸与非必需氨基酸含量的比值大于0.6时,表明该蛋白质的营养价值比较高[21]。由表5可知,绿豆芽蛋白的氨基酸种类较为齐全,有17种氨基酸,其中7种为必需氨基酸。含硒组氨基酸总量和必需氨基酸的含量分别为48.89 g/100 g、19.82 g/100 g,对照组氨基酸总量和必需氨基酸的含量分别为55.81 g/100 g、22.03 g/100 g,对比发现含硒组蛋白中氨基酸含量比对照组低。含硒组蛋白中的必需氨基酸含量与氨基酸总量的比值为0.4,高于对照组(0.39),且含硒组与对照组必需氨基酸与非必需氨基酸含量的比值都大于0.6,含硒组(0.66)略高于对照组(0.65),符合FAO/WHO提出的理想蛋白质条件。另外,富硒绿豆芽蛋白中含量最高的氨基酸为谷氨酸(7.85 g/100 g),而谷氨酸具有风味增强作用,因此,富硒绿豆芽蛋白及其水解物有望作为功能性蛋白用于食品加工以改善食品风味。
表5 绿豆芽蛋白的氨基酸组成
Table 5 Amino acid composition of mung bean sprout protein
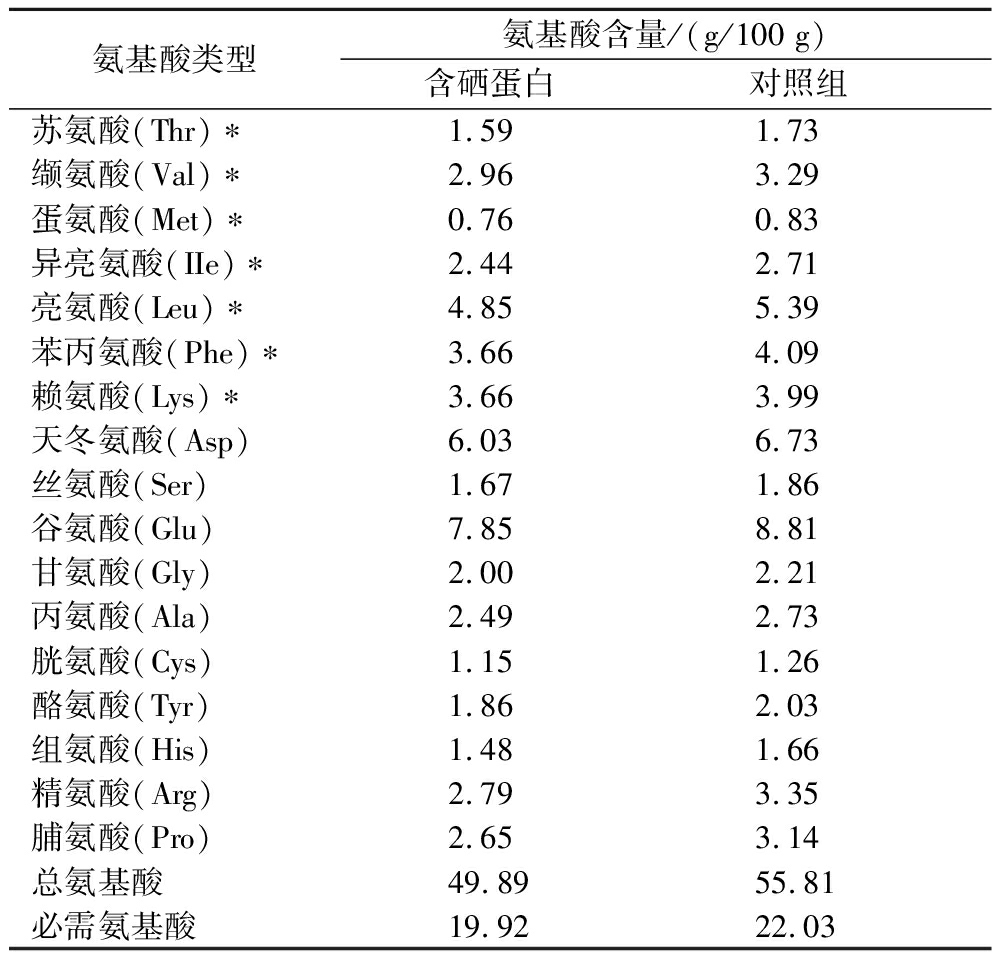
氨基酸类型氨基酸含量/(g/100 g)含硒蛋白 对照组 苏氨酸(Thr)∗1.591.73缬氨酸(Val)∗2.963.29蛋氨酸(Met)∗0.760.83异亮氨酸(IIe)∗2.442.71亮氨酸(Leu)∗4.855.39苯丙氨酸(Phe)∗3.664.09赖氨酸(Lys)∗3.663.99天冬氨酸(Asp)6.036.73丝氨酸(Ser)1.671.86谷氨酸(Glu)7.858.81甘氨酸(Gly)2.002.21丙氨酸(Ala)2.492.73胱氨酸(Cys)1.151.26酪氨酸(Tyr)1.862.03组氨酸(His)1.481.66精氨酸(Arg)2.793.35脯氨酸(Pro)2.653.14总氨基酸49.8955.81必需氨基酸19.9222.03
注:*代表必需氨基酸。
2.6 绿豆芽蛋白中硒代氨基酸分析
由表6可知,含硒组蛋白中硒代胱氨酸(selenocystine,SeCys)和硒代甲硫氨酸(selenomethionine,SeMet)含量分别为340.56 μg/kg、568.63 μg/kg,对照组蛋白中SeCys和SeMet含量分别为278.88 μg/kg、240.77 μg/kg,含硒组蛋白中的SeCys和SeMet含量分别为对照组的1.22、2.36倍,SeCys和SeMet含量的增加可能是因为硒与硫的代谢方式相似,进而代替了氨基酸中的硫元素。另外对照组SeMet含量低于SeCys的含量,而含硒组SeMet含量高于SeCys含量。因此,绿豆芽中氨基酸与硒的主要结合方式为SeMet。这与CHENG等[1]的研究结果一致,其研究表明,绿豆在萌发过程中将Se(IV)转化为Se(VI),并运输到茎、叶和根。Se(VI)或Se(IV)通过亚硫酸盐还原酶和半胱氨酸合成酶催化转化为SeCys,大部分SeCys通过胱硫醚β裂解酶转化为硒同型半胱氨酸,转运到质体中,最后通过甲硫氨酸合成酶转化为SeMet。但高俣[22]的研究结果显示,硒甲基硒代半胱氨酸为绿豆芽的主要硒代氨基酸,原因可能是由于绿豆的种类、硒的来源、培养条件以及提取方法的不同。
表6 富硒绿豆芽蛋白及对照组中氨基酸组成
Table 6 Amino acid composition of selenium-enriched mung bean sprout protein and control group
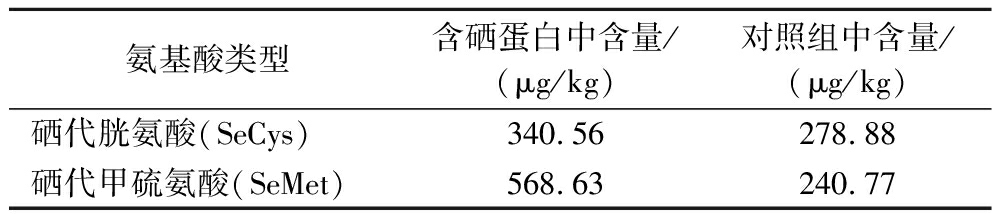
氨基酸类型含硒蛋白中含量/(μg/kg)对照组中含量/(μg/kg)硒代胱氨酸(SeCys)340.56278.88硒代甲硫氨酸(SeMet)568.63240.77
3 结论
本文从不同的角度比较了RSM与GA-ANN模型对富硒绿豆芽蛋白提取工艺的预测性能,结果表明,GA-ANN模型具有更高的准确性和预测能力,可以作为更好的替代方案;对富硒绿豆芽中的氨基酸和硒代氨基酸的含量进行检测,含硒组蛋白和对照组的必需氨基酸含量与氨基酸总量的比值分别为0.4、0.39,且必需氨基酸与非必需氨基酸含量的比值都大于0.6,基本符合FAO/WHO提出的理想蛋白质条件,表明该硒蛋白作为优质硒蛋白对于促进人体健康有一定意义。另外,富硒绿豆芽中氨基酸与硒的主要结合方式为SeMet,具有更高的安全性,可作为缺硒地区人群的营养补充剂。本研究结果可为GA-ANN模型用于优化硒蛋白的提取工艺以及富硒功能性食品的开发提供一定参考。
[1] CHENG H, LI L, DONG J Z, et al. Transcriptome and physiological determination reveal the effects of selenite on the growth and selenium metabolism in mung bean sprouts[J]. Food Research International, 2023, 169:112880.
[2] HUANG Y T, FAN B, LEI N Y, et al. Selenium biofortification of soybean sprouts: Effects of selenium enrichment on proteins, protein structure, and functional properties[J]. Frontiers in Nutrition, 2022, 9:849928.
[3] DENG B L, TIAN S, LI S P, et al. A simple, rapid and efficient method for essential element supplementation based on seed germination[J]. Food Chemistry, 2020, 325:126827.
[4] GIRMA A E, ABADR A, RAMACHANDRAN K, et al. Artificial neural network and response surface methodology for modeling and optimization of activation of lactoperoxidase system[J]. South African Journal of Chemical Engineering, 2021, 37(prepublish): 12-22.
[5] 薛宏坤, 刘成海, 刘钗, 等. 响应面法和遗传算法-神经网络模型优化微波萃取蓝莓中花青素工艺[J]. 食品科学, 2018, 39(16):280-288. XUE H K, LIU C H, LIU C, et al. A comparative study of the optimization of microwave extraction of anthocyanins from blueberry by response surface methodology and genetic algorithm-artificial neural network[J]. Food Science, 2018, 39(16):280-288.
[6] KUO C H, LIU T A, CHEN J H, et al. Response surface methodology and artificial neural network optimized synthesis of enzymatic 2-phenylethyl acetate in a solvent-free system[J]. Biocatalysis and Agricultural Biotechnology, 2014, 3(3):1-6.
[7] YU Z X, ZHANG Y Y, ZHAO X X, et al. Simultaneous optimization of ultrasonic-assisted extraction of Danshen for maximal tanshinone IIA and salvianolic acid B yields and antioxidant activity: A comparative study of the response surface methodology and artificial neural network[J]. Industrial Crops and Products, 2021, 161:113199.
[8] AMEER K, AMEER S, KIM Y M, et al. A hybrid RSM-ANN-GA approach on optimization of ultrasound-assisted extraction conditions for bioactive component-rich Stevia rebaudiana (bertoni) leaves extract[J]. Foods, 2022, 11(6):883.
[9] CIRIC A, KRAJNC B, HEATH D, et al. Response surface methodology and artificial neural network approach for the optimization of ultrasound-assisted extraction of polyphenols from garlic[J]. Food and Chemical Toxicology, 2020, 135:110976.
[10] CHOI H J, NAZNIN M, ALAM M B, et al. Optimization of the extraction conditions of Nypa fruticans Wurmb. using response surface methodology and artificial neural network[J]. Food Chemistry, 2022, 381:132086.
[11] YANG T, LAI H K, CAO Z M, et al. Comparison of an artificial neural network and a response surface model during the extraction of selenium-containing protein from selenium-enriched Brassica napus L[J]. Foods, 2022, 11(23):3823.
[12] QIU J J, SHI M L, LI S Q, et al. Artificial neural network model- and response surface methodology-based optimization of Atractylodis Macrocephalae Rhizoma polysaccharide extraction, kinetic modelling and structural characterization[J]. Ultrasonics Sonochemistry, 2023, 95:106408.
[13] HUANG S M, KUO C H, CHEN C N, et al. RSM and ANN modeling-based optimization approach for the development of ultrasound-assisted liposome encapsulation of piceid[J]. Ultrasonics Sonochemistry, 2017, 36:112-122.
[14] 李蕾, 董静洲, 程水源. 6种豆科植物芽苗的富硒能力比较筛选与富硒条件研究[J]. 食品科技, 2022, 47(5):102-108. LI L, DONG J Z, CHENG S Y. Comparative screening of selenium-enriched ability and study on selenium-enriched conditions of six species of leguminous plants sprouts[J]. Food Science and Technology, 2022, 47(5):102-108.
[15] 徐亚, 范会芬, 赵玎玲, 等. 考马斯亮蓝法测定大豆水溶性蛋白提取方法的优化[J]. 大豆科学, 2022, 41(2):196-202. XU Y, FAN H F, ZHAO D L, et al. Optimization of extraction method for water-soluble protein determination by coomassie bright blue method[J]. Soybean Science, 2022, 41(2):196-202.
[16] 刘开放, 席志文, 黄林娜, 等. 布拉酵母高密度发酵培养基及发酵工艺优化[J]. 食品科学, 2019, 40(8):56-62. LIU K F, XI Z W, HUANG L N, et al. Optimization of high cell density fermentation of Saccharomyces boulardii for enhanced biomass production[J]. Food Science, 2019, 40(8):56-62.
[17] 代晹鑫, 徐莹, 毕爽, 等. 核桃粕蛋白提取纯化工艺优化及其功能性质分析[J]. 食品工业科技, 2023, 44(2):241-252. DAI Y X, XU Y, BI S, et al. Extraction and purification process optimization and functional properties analysis of walnut meal protein[J]. Science and Technology of Food Industry, 2023, 44(2):241-252.
[18] HOU F R, DING W H, QU W J, et al. Alkali solution extraction of rice residue protein isolates: Influence of alkali concentration on protein functional, structural properties and lysinoalanine formation[J]. Food Chemistry, 2017, 218:207-215.
[19] QU W J, FAN W, FENG Y T, et al. Preparation of heat-sensitivity proteins from walnut meal by sweep frequency ultrasound-assisted alkali extraction[J]. Journal of Food Quality, 2021, 2021:1-12.
[20] 吕青遥, 周敏, 周慧玲, 等. 不同产地雪菊氨基酸组成分析及综合评价[J]. 食品与机械, 2022, 38(11):27-32. LU Q Y, ZHOU M, ZHOU H L, et al. Analysis and comprehensive evaluation of amino acid composition of Coreopsis tinctoriafrom different habitats[J]. Food &Machinery, 2022, 38(11):27-32.
[21] 刘哲, 叶英, 罗黎霞, 等. 狭果茶藨子营养成分分析与氨基酸提取工艺优化及评价[J]. 食品与发酵工业, 2022, 48(13):188-195. LIU Z, YE Y, LUO L X, et al. Analysis of nutritional components and optimization of amino acid extraction process of Ribes stenocarpum Maxim[J]. Food and Fermentation Industries, 2022, 48(13):188-195.
[22] 高俣. 绿豆芽富硒特性及HPLC-MS联用法测定绿豆芽中硒代氨基酸[D]. 沈阳: 辽宁大学, 2016. GAO Y. Accumulation characteristics of selenium in selenium-enriched mung bean sprouts and analysis of seleno-amino acids by HPLC-MS[D]. Shenyang: Liaoning University, 2016.