脂肪酶(EC 3.1.1.3)可以催化酯化、转酯、水解、酸解等反应[1-3]。在众多脂肪酶中,南极假丝酵母脂肪酶B(Candida antarctic lipase B,CALB)具有高对映选择性及广泛的底物范围[4],在生物柴油的生产、对映体的拆分、甘油单酯的生产[5-8]等领域均有应用。由于酶的反应速率会随着温度的升高呈指数增加[9],且在工业中通常要求酶在严苛的条件下仍具有较好的稳定性,所以酶在应用时需要考虑其热稳定性。KÖSE等[10]采用Novozym 435(商业化的固定化CALB)催化棉籽油和醇类生成甲酯,当温度高于50 ℃时,甲酯产量会降低4.2%。KIM等[11]等采用商业化的CALB在60 ℃下催化甘油和碳酸二甲酯转化为碳酸甘油酯的效率降低7.3%。提高CALB的热稳定性成为其应用于工业化需要解决的问题。
为了提高酶的稳定性,理性设计、半理性设计、定向进化等[12]策略常被使用。半理性设计中的共识设计操作较简单,在不需要准确的蛋白质三维结构的条件下就能进行酶学性质的改造[13]。其基于以下原则:在进化过程中,随机突变很难提高蛋白的稳定性,而同源蛋白中出现频率较高的氨基酸(即保守氨基酸)对蛋白结构稳定性的贡献要高于非保守氨基酸[14-15]。通过同源序列比对寻找保守氨基酸,这些氨基酸组成的序列称为共识序列。将非保守氨基酸突变为共识序列中的保守氨基酸,从而提高酶的稳定性。STEVENS等[16]通过共识设计获得一条共识快速DNA聚合酶III内含子序列(Cfa),其剪切速率随着温度的增加而增加,且始终高于野生型的来源于Nostoc punctiforme PCC73102的断裂蛋白质内含子Npu(30 ℃下是Npu的2.5倍),80 ℃时虽然Cfa的剪切产物产量降低,但Npu在该温度下已无活性。PAATERO等[17]对小鼠富含亮氨酸的跨膜蛋白LRRTM2进行保守突变获得cLRRTM2,其Tm值相比于野生型LRRTM提高了32 ℃。
为满足工业生产的需要,提高CALB的热稳定性,本研究将南极假丝酵母来源的脂肪酶B(GenBank:Z30645.1)进行多序列比对,选择对CALB热稳定性有潜在影响的点进行定点突变,并进行有序叠加,筛选热稳定性提高的突变体。通过Discovery Studio 2022进行分子动力学模拟和作用力分析,从结构上解释最优突变体热稳定性提高的原因。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
质粒pET22b、菌株E.coli JM109、E.coli BL21(DE3)均由本实验室保存,质粒pET22b-calb由上海生工合成构建。
1.1.2 主要试剂
质粒提取试剂盒、柱回收试剂盒、异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactoside,IPTG)、一步克隆试剂盒、Dpn I、氨苄霉素,生工生物工程(上海)股份有限公司;Prime STAR Max Premix DNA聚合酶、大肠杆菌感受态细胞制备试剂盒,大连TaKaRa公司;蛋白marker、蛋白电泳Loading buffer、Bis-Tris预制凝胶,Invitrogen公司;Ni-NTA亲和层析预装柱,美国通用电气公司;Bradford蛋白浓度测定试剂盒,碧云天生物;对硝基苯酚丁酸酯,上海百灵威化学技术有限公司;其他常规试剂及药品为国产或进口分装。
1.1.3 培养基及缓冲液
LB培养基(g/L):酵母提取物5,蛋白胨10,NaCl 5,pH值7.0。
TB培养基(g/L):酵母提取物24,蛋白胨12,甘油5,KH2PO4 2.31,K2HPO4·3H20 16.4。
25 mmol/L对硝基苯酚丁酸酯(p-nitrophenol butyrate,pNPB):0.523 g pNPB中加入250 μL的Triton X-100,加100 mL超纯水定容后超声5 min使其均一。
50 mmol/L Tris-HCl缓冲液:3.028 g Tris加超纯水定容至500 mL,浓HCl调pH值至8.0。
2.5 mmol/L对硝基苯酚(p-nitrophenol,pNP)母液的配制:0.034 8 g pNP溶于100 mL 50 mmol/L Tris-HCl缓冲液中。
A液:Tris 50 mmol/L,NaCl 200 mmol/L,浓HCl调pH值至7.5~8.0。
B液:Tris 50 mmol/L,NaCl 200 mmol/L,咪唑500 mmol/L,浓HCl调pH值至7.5~8.0。
C液:Tris 50 mmol/L,浓HCl调pH值至7.5~8.0。
固体培养基中添加1.5%~2%(质量分数)的琼脂。
1.2 实验方法
1.2.1 突变体的构建
以质粒pET22b-calb为模板,采用表1中的引物对进行反向PCR。PCR体系为:模板1 μL,上游引物1 μL,下游引物1 μL,ddH2O 22 μL,Prime STAR Max Premix DNA聚合酶25 μL。PCR条件:98 ℃预变性3 min,98 ℃变性10 s,55 ℃退火10 s,72 ℃延伸1 min 30 s,30个循环,72 ℃继续延伸10 min,4 ℃保温。将PCR后的产物加入Dpn I消化模板,消化后进行柱回收。将回收后的片段转入E.coli JM109感受态中,挑取长出的转化子过夜培养,提质粒送测序。将测序正确的转化子转入E.coli BL21(DE3)得到重组菌。
表1 本研究使用的引物
Table 1 Primers used in this study

引物名称引物序列(5′-3′)A25G-FGGCCTGACCTGCCAAGGTGGTAGTCCTTCTTCCGTTA25G-RCTTGGCAGGTCAGGCCGGCATCGAGV30Q-FAGGTGCTAGTCCTTCTTCCCAATCTAAGCCCATTTTGTV30Q-RAAGAAGGACTAGCACCTTGGCAT80A-FAATGATACTCAGGTCAATGCTGAGTACATGGTCAACT80A-RTGACCTGAGTATCATTAAGCATGAAAGGAGM83I-FCTCAGGTCAATACTGAGTACATTGTCAACGCTATCACCM83I-RACTCAGTATTGACCTGAGTATCATTAAGCATI87V-FGAGTACATGGTCAACGCTGTCACCGCTCTTTATGI87V-RCGTTGACCATGTACTCAGTATTGACCTGAGL99V-FGGCTCAGGTAATAACAAGGTACCAGTTCTAACATGL99V-RGTTATTACCTGAGCCTGCATAAAGAGCV110A-FGGTCGCAGGGAGGGCtAGCAGCACAATGGGGV110A-RAGCCCTCCCTGCGACCATGTTAGAACTG114A-FGGCTAGTAGCACAATGGGCCCTGACCTTCTTCCCTAGG114A-RATTGTGCTACTAGCCCTCCCTGCGA148G-FCCCTCGATGCCCTCGGCGTCAGCGCCCCA148G-RCGAGGGCATCGAGGGGGCCGGCGAGGT159A-RGGTCTGCTGCCAGACGGAGGGGGCT159A-FTCTGGCAGCAGACCGCCGGCTCCGCCCTCAG207A-FATTCCTCGTACCTGTTCAACGCCAAGAACGTCCAGGCACAG207A-RTGAACAGGTACGAGGAATCCAGGGGGGAGTTGA214S-FAACGTCCAGGCACAGTCCGTCTGCGGCCA214S-RCTGTGCCTGGACGTTCTTGCCGTTGAACAGP280V-FGCCCTGCTGGCCGTTGCCGCCGCCGCCATP280V-RCGGCGGCGGCAACGGCCAGCAGGGCA284 N-FGCCGCCGCCAACATCGTCGCCA284 N-RCGGCGACGATTTCGGCGGCGGCAG
注:下划线为酶切位点或一步克隆对应的同源臂序列。
1.2.2 CALB及突变体的表达
将带有正确质粒的转化子接入加有终质量浓度为100 μg/mL氨苄青霉素的液体LB中过夜培养,将种子按3%的接种量接种至装有30 mL TB的发酵培养基中,37 ℃,220 r/min培养至OD600≈0.8~1.0后,添加终浓度为0.1 mmol/L的IPTG 20 ℃诱导,24 h后结束发酵。
1.2.3 CALB及突变体的纯化、脱盐
将发酵液在10 000 r/min,4 ℃下离心10 min获得发酵上清液,采用0.22 μm的水系滤膜过滤后进行镍柱亲和层析获得目的蛋白。上样完成后先用A液平衡,再用不同浓度的B液洗脱。洗脱后的纯蛋白通过Sephadex G-25脱盐柱采用C液进行脱盐。
1.2.4 CALB及突变体性质测定
残余酶活的测定:将纯化后的野生型和突变体(质量浓度均为0.15 mg/mL)置于50 ℃的金属浴中处理5 min,冰上冷却后测定酶活力,以未处理的酶活力为100%,计算残余酶活力。
半衰期的测定:将野生型和突变体的纯蛋白(质量浓度为0.005 mg/mL)分装后置于50 ℃水浴中,测定不同时间下的酶活力,以未处理前的酶活力为100%,ln(残余酶活力)为纵坐标,t为横坐标进行线性拟合,计算kd,得到t1/2。
Tm值的测定:采用Nano-DSC测定CALB及突变体的Tm。先将样品脱气,上样后等热流信号和压力信号稳定后开始运行,以1 ℃/min的速率将温度从30 ℃上升至80 ℃对样品进行扫描。利用软件Nano Analysis对结果进行拟合,得出Tm。
动力学参数的测定 配制0、5、10、15、20、25、30、35 mmol/L的pNPB,测定不同底物浓度下CALB及突变体的酶活。利用Origin 2021进行非线性拟合,计算Km和kcat。
1.2.5 SDS-PAGE分析和蛋白浓度测定
将发酵液10 000 r/min,4 ℃离心后获得发酵上清液,菌体采用C液重悬后进行超声破碎,将破碎后的样品4 ℃离心,上清即为胞内可溶部分,沉淀采用C液重悬后获得胞内不溶部分。将重组菌BL21/pET22b和BL21/pET22b-calb的发酵上清液、胞内可溶和不溶部分进行SDS-PAGE。V(蛋白样品)∶V(4×SDS上样缓冲液)=3∶1,98 ℃煮沸10 min,上样20 μL恒压120 V进行SDS-PAGE。蛋白浓度采用碧云天Bradford蛋白浓度测定试剂盒。
1.2.6 CALB酶活力测定[18]
pNP标准曲线的绘制:配制0、0.025、0.05、0.075、0.1、0.125、0.15、0.17、0.2、0.225、0.25 mmol/L的pNP,测定OD405值。以pNP的浓度为横坐标,OD405值为纵坐标绘制标曲。
CALB酶活力测定:将900 μL的Tris-HCl 缓冲液和50 μL适当稀释的酶液在45 ℃下预热5 min后,加入50 μL 25 mmol/L pNPB反应5 min,最后加入预冷的乙醇终止反应,测定OD405值,每分钟催化生成1 μmol pNP所需的酶量为1 U。
1.2.7 分子动力学模拟和机制分析
利用在线软件I-TASSER(https://zhanggroup.org/I-TASSER/)对CALB和突变体进行蛋白质建模。采用软件Discovery Studio 2022进行分子动力学模拟,力场选择CHARMm,将CALB和突变体放于一个立体水盒子中,并添加Na+和Cl-中和体系中的电荷以使能量最小化。随后在平衡200 ps后在330 K下进行50 ns的模拟。CALB及突变体的分子结构和作用力分析借助软件Discovery Studio 2022和Pymol 2.5.4完成。底物pNPB的构象从PubChem(https://pubchem.ncbi.nlm.nih.gov/)中获得,通过Discovery Studio 2022转为PDB格式后和CALB及突变体进行对接,选择能量最低的构象进行分析。
1.2.8 生物信息学分析
采用NCBI(https://www.ncbi.nlm.nih.gov/)进行检索,获得同源序列。采用软件Jalview 2.11.2.0进行同源性比对后得到共识序列,分别计算共识序列和CALB中每个氨基酸在所有比对序列中出现频率百分比,两个百分比的比值即为相对频率,通过相对频率确定突变位点。
2 结果与分析
2.1 CALB在大肠杆菌中的表达及纯化
大肠杆菌遗传背景清晰,操作成熟,周期短,生长环境简单[19],故本研究选择其作为宿主。将calb连接至pET22b中pelB信号肽的下游,得到重组表达质粒pET22b-calb(图1-a)。在该质粒转入E.coli BL21(DE3)后得到重组菌BL21/pET22b-calb。将重组菌发酵24 h后测定发酵液上清液、胞内可溶部分和不溶部分的酶活,并进行SDS-PAGE分析。与pET22b相比,重组菌BL21/pET22b-calb的发酵上清液和胞内可溶部分中CALB酶活分别为2.68 U/mL和1.05 U/mL(图1-b),且均有明显条带(理论分子质量为33 kDa)(图1-c)。胞内不溶部分未检测到CALB酶活为且无对应蛋白条带。这些结果表明,CALB在E.coli BL21(DE3)中实现可溶性表达,但并未全部分泌至胞外。
将重组菌BL21/pET22b-calb的发酵上清液进行镍柱亲和层析。分别采用10%,15%,20%,40%,60%,100% B液进行梯度洗脱,收集洗脱样品并进行SDS-PAGE分析。如图1-d所示,10% B液洗脱出杂蛋白,40% B液洗脱出目标蛋白,以上结果表明,镍柱亲和层析可以获得单一的目标蛋白。
2.2 共识序列分析
共识序列改造是使用同源序列之间的信息获得共识序列,将改造序列中保守性低的氨基酸突变为共识序列中的保守氨基酸,以此提高酶的稳定性[20]。将由317个氨基酸编码的CALB序列输入至NCBI(https://www.ncbi.nlm.nih.gov/)中进行比对,并获得100条序列。选择一致性在30%~60%[21]的48条序列作为同源序列,采用软件Jalview 2.11.2.0对这些序列进行同源比对后获得共识序列(图2),计算相对频率和相对频率的平均值和标准差,分别为 0.64和0.38。一般认为,当相对频率低于平均值-1.5×标准差时,该位置氨基酸的保守性较低,需要突变为共识序列中对应的氨基酸[13]。CALB大多数位置的相对频率>0.26(相对频率的均值和标准差的差值),但有14个点(图2)的相对频率<0.07(平均值-1.5×标准差)。为了评估这些点对CALB稳定性的影响,将其全部突变为共识序列中的氨基酸。
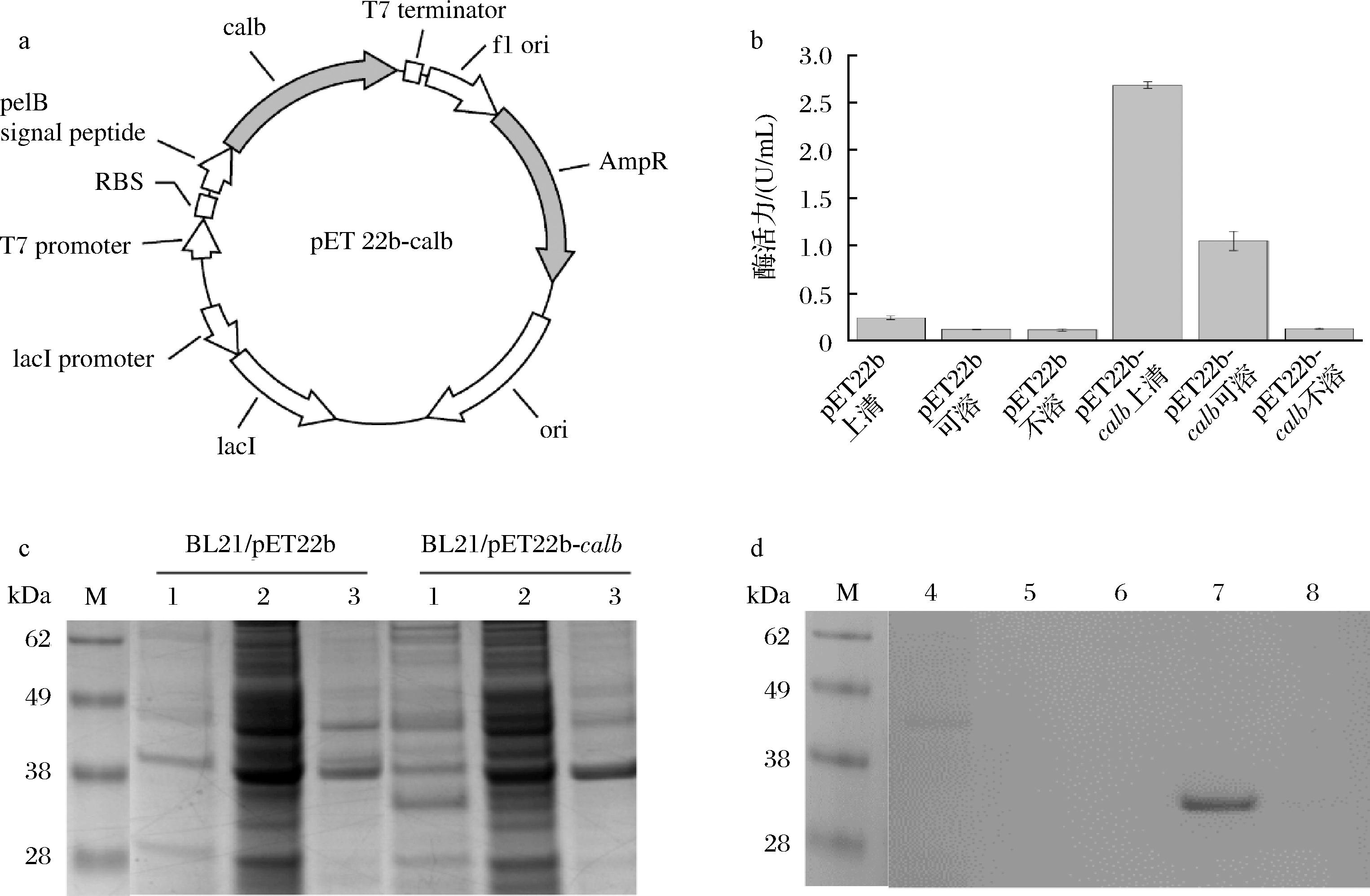
a-用于CALB表达的质粒pET22b-calb;b-诱导24 h后BL21/pET22b和BL21/pET22b-calb的发酵液上清液、胞内可溶和不溶部分酶活力; c-诱导24 h后BL21/pET22b和BL21/pET22b-calb的发酵液上清液、胞内可溶和不溶部分SDS-PAGE分析(M-Marker;1-BL21/pET22b 发酵上清;2-BL21/pET22b 胞内可溶;3-BL21/pET22b 胞内不溶);d-不同浓度B液洗脱后蛋白的SDS-PAGE验证(M-Marker; 4-10% B液洗脱;5-15%B液洗脱;6-20% B液洗脱;7-40% B液洗脱;8-60% B液洗脱)
图1 重组CALB的表达情况分析和SDS-PAGE分析
Fig.1 Expression analysis and SDS-PAGE analysis of recombinant CALB
2.3 共识序列改造单点突变对CALB热稳定性及催化活性的影响
将共识设计得到的位点进行定点突变后获得正确突变体,突变体经IPTG诱导24 h后结束发酵,测定发酵上清液中CALB的酶活。其中,V30Q,T80A,T280V基本未正常表达(数据未显示)。将剩余的突变体进行蛋白纯化(图3-a),测定纯化后CALB和突变体的比酶活及在50 ℃保温5 min后的残余酶活力。有研究表明,共识序列改造只有部分会提高酶的稳定性,而其他的会破坏稳定性或不会产生任何影响[22]。和CALB相比,I87V、G114A、A148G、T159A和A284 N残余酶活力分别提高了10%,10.1%,2.6%,10.5%和13.4%,A25G,L99V,G207A残余酶活力无明显变化,M83I,V110A,A214S分别下降11.8%,21.4%,17.1%。T159A比酶活增加13.7%,G114A,G207A无明显变化,其他突变体的比酶活均有不同程度的下降(图3)。
2.4 共识序列改造组合突变对CALB热稳定性及催化活性的影响
为了获得更优突变体,将单点突变中有正向效果的点进行组合突变。以残余酶活力最高的A284 N为基础,分别将I87V、G114A、A148G、T159A进行叠加,得到突变体A284 N/I87V、A284 N/G114A、A284 N/A148G、A284 N/T159A,测定上述突变体的比酶活和残余酶活力。相比于A284 N,A284 N/G114A和A284 N/I87V残余酶活力提高了16.1%和14.5%,A284 N/A148G和A284 N/T159A下降了44.8%和47.7%。和CALB相比,仅A284 N/A148G的比酶活提高了22.5%,其他突变体的比酶活均有不同程度的降低(图4)。最后将I87V、G114A、A284 N进行组合,得到A284 N/I87V/G114A,该突变体的残余酶活力并未高于A284 N/G114A(图4)。经组合突变,得到最优突变体A284 N/G114A(CALBm)。PARK等[4]通过分子动力学模拟和RosettaDesign算法获得的突变体A251E虽然热稳定性提高了2.5倍,但比酶活却下降了50%(CALBm下降了36%)。

图2 CALB和共识序列比对结果
Fig.2 Alignment result of CALB and consensus sequence
注:序列右侧数字:氨基酸编号;●:突变氨基酸;-:此位置氨基酸出现空缺;+:此位置氨基酸无保守性;% cal res(CALB residues): CALB每个氨基酸出现频率占比对序列百分比;% con res(consensus sequence residues):共识序列中每个氨基酸出现频率占比对序列百分比; % Rel.Freq.(Relative Frequency):% cal res与% con res的比值。
为进一步了解突变体的热稳定性变化,测定了野生型和突变体的熔解温度及50 ℃下的半衰期,结果如表2所示。和CALB相比,CALBm在50 ℃下的t1/2为14.5 min,提高了74.7%。Tm为57.9 ℃,提高了1.3 ℃。KIM等[23]通过B-因子和RosettaDesign获得突变体R249L,其Tm值(56.8 ℃)低于CALBm的Tm值(57.9 ℃)。LE等[24]通过二硫键的引入获得的突变体A162C-K308C Tm值(55.9 ℃)同样也低于CALBm。温露文等[25]通过PoPMuSiC和FoldX预测获得突变体A146G-L278M和A146G-L278M-A151P,其在50 ℃下的半衰期分别是野生型的1.35倍和1.5倍,提升幅度低于CALBm(1.7倍)。同时测定了CALB和CALBm对不同浓度pNPB的催化效率,和CALB相比,CALBm的Km增加了26%,kcat/Km下降了23%。XIE等[26]通过选取活性位点附近高B-factor值的点进行突变后获得D223G/L278M,其Km增加了35%,对底物对硝基苯酚辛酸酯(p-nitrophenol caprylate,pNPC)的亲和力降低,kcat/Km无明显变化。KIM等[23]通过B-因子和RosettaDesign获得突变体R249L,其Km增加了7%,Vmax/Km下降了8%。和其他突变体比较,CALBm催化效率下降较多,可能是由于底物种类和浓度不同造成。
2.5 分子动力学模拟和底物对接
研究表明,蛋白结构刚性的增加是酶稳定的主要因素之一,通常表现为突变蛋白主链较低的RMSD和RMSF值[27]。将CALB和CALBm在330 K下进行分子动力学模拟。和CALB相比,CALBm的平均RMSD值(3.54)和最终RMSD值(4.7)均低于CALB(均值4.61,最终值6.35)(图5-a)同时,CALBm在区域I(100~150)和区域II(250~300)的RMSF值均低于CALB(图5-b),说明G114A和A284 N的突变可能提高蛋白的局部刚性,从而影响整体的稳定性。

a-CALB及突变体纯化后SDS-PAGE验证; b-CALB及突变体纯化后的残余酶活和比酶活
图3 CALB和突变体的SDS-PAGE分析及残余酶活和比酶活
Fig.3 SDS-PAGE analysis and residual enzyme activity and specific enzyme activity of CALB and mutants

图4 组合突变体的残余酶活和比酶活
Fig.4 Residual enzyme activity and specific enzyme activity of combined mutants
表2 CALB和CALBm性质比较
Table 2 Properties comparison of CALB and CALBm

组别t1/2/minTm/℃Km/(mmol/L)kcat/(103/min)Kcat/Km/[mmol/(L·min)]CALB8.356.615.6120.27 716.6CALBm14.557.919.7116.85 932.3

a-RMSD分析;b-RMSF分析;c-突变位点的相互作用力分析
图5 CALB和CALBm的RMSD和RMSF分析及突变位点的相互作用力变化
Fig.5 RMSD and RMSF analysis of CALB and CALBm and interaction changes in mutation sites
注:绿色:CALB的局部结构;白色:CALBm的局部结构;黄色虚线:氢键;紫色虚线:疏水相互作用。
进一步对突变前后氨基酸与周围氨基酸之间的相互作用进行分析(图5-c),G114突变后,在原来氢键(G114 vs F118,G114 vs V110)的基础上,新增加了3个疏水相互作用(G114 vs M83,G114 vs V84,A114 vs I121)。A284突变后,虽然少了1个疏水相互作用(A284 vs F71),但新形成了2个氢键(A284 vs G41,A284 vs A281)。突变后新形成的键在一定程度上增加了CALB的刚性,促使热稳定性的提升。
将CALB和CALBm与底物pNPB分别进行对接后,分析底物和氨基酸残基间的相互作用力,pNPB和CALB之间的作用力由突变前的2个氢键和4个疏水相互作用变为1个氢键和3个疏水相互作用(图6),底物和酶分子之间作用力的减少可能影响了底物和酶的结合,导致催化效率下降。

a-CALB和pNPB相互作用力;b-CALBm和pNPB相互作用力
图6 CALB和CALBm与pNPB分子对接
Fig.6 Molecular docking of CALB and CALBm with pNPB
注:绿色:CALB的局部结构;白色:CALBm的局部结构; 绿色和浅蓝色虚线:氢键;紫色虚线:疏水相互作用。
3 结论
本研究通过多序列比对,得到对CALB热稳定性有潜在影响的点进行突变,获得热稳定性提升的突变体CALBm。和CALB相比,CALBm在50 ℃下的残余酶活提升了25.9%,50 ℃下的半衰期(t1/2=14.5 min)增加了6.2 min,Tm值提高了1.3 ℃。但比酶活下降了36.3%,Km增加了26%,对底物的亲和力降低,kcat/Km也存在一定程度的下降。动力学分析和局部相互作用分析表明,CALBm热稳定性提升的原因可能是由于突变后新增加的氢键和疏水相互作用使蛋白局部刚性增强,从而影响整体的稳定性。
[1] GOTOR-FERN NDEZ V, BUSTO E, GOTOR V.Candida antarctica lipase B:An ideal biocatalyst for the preparation of nitrogenated organic compounds[J].Advanced Synthesis &Catalysis, 2006, 348(7-8):797-812.
NDEZ V, BUSTO E, GOTOR V.Candida antarctica lipase B:An ideal biocatalyst for the preparation of nitrogenated organic compounds[J].Advanced Synthesis &Catalysis, 2006, 348(7-8):797-812.
[2] NAMAL SENANAYAKE S P J, SHAHIDI F.Incorporation of docosahexaenoic acid (DHA) into evening primrose (Oenothera biennis L.) oil via lipase-catalyzed transesterification[J].Food Chemistry, 2004, 85(4):489-496.
[3] 李站胜, 颜晨麟, 江宁, 等.脂肪酶处理白酒丢糟提升复糟酒的品质[J].现代食品科技, 2019, 35(5):191-197;123. LI Z S, YAN C L, JIANG N, et al.Lipase treatment of distiller's grains wine to promote its quality[J].Modern Food Science and Technology, 2019, 35(5):191-197;123.
[4] PARK H J, PARK K, KIM Y H, et al.Computational approach for designing thermostable Candida antarctica lipase B by molecular dynamics simulation[J].Journal of Biotechnology, 2014, 192(Part A):66-70.
[5] DU W, XU Y Y, LIU D H, et al.Comparative study on lipase-catalyzed transformation of soybean oil for biodiesel production with different acyl acceptors[J].Journal of Molecular Catalysis B:Enzymatic, 2004, 30(3-4):125-129.
[6] BARBOSA O, ORTIZ C, TORRES R, et al.Effect of the immobilization protocol on the properties of lipase B from Candida antarctica in organic media:Enantiospecifc production of atenolol acetate[J].Journal of Molecular Catalysis B:Enzymatic, 2011, 71(3-4):124-132.
[7] TAMAYO J J, LADERO M, SANTOS V E, et al.Esterification of benzoic acid and glycerol to α-monobenzoate glycerol in solventless media using an industrial free Candida antarctica lipase B[J].Process Biochemistry, 2012, 47(2):243-250.
[8] KAPOOR M, GUPTA M N.Obtaining monoglycerides by esterification of glycerol with palmitic acid using some high activity preparations of Candida antarctica lipase B[J].Process Biochemistry, 2012, 47(3):503-508.
[9] PETERSON M E, DANIEL R M, DANSON M J, et al.The dependence of enzyme activity on temperature:Determination and validation of parameters[J].The Biochemical Journal, 2007, 402(2):331-337. [10] KÖSE Ö, TÜTER M, AKSOY H A.Immobilized Candida antarctica lipase-catalyzed alcoholysis of cotton seed oil in a solvent-free medium[J].Bioresource Technology, 2002, 83(2):125-129.
[11] KIM S C, KIM Y H, LEE H, et al.Lipase-catalyzed synthesis of glycerol carbonate from renewable glycerol and dimethyl carbonate through transesterification[J].Journal of Molecular Catalysis B:Enzymatic, 2007, 49(1-4):75-78.
[12] LIU Q, XUN G H, FENG Y.The state-of-the-art strategies of protein engineering for enzyme stabilization[J].Biotechnology Advances, 2019, 37(4):530-537.
[13] RATH A, DAVIDSON A R.The design of a hyperstable mutant of the Abp1p SH3 domain by sequence alignment analysis[J].Protein Science, 2000, 9(12):2457-2469.
[14] TOKURIKI N, TAWFIK D S.Stability effects of mutations and protein evolvability[J].Current Opinion in Structural Biology, 2009, 19(5):596-604.
[15] STEIPE B, SCHILLER B, PLÜCKTHUN A, et al.Sequence statistics reliably predict stabilizing mutations in a protein domain[J].Journal of Molecular Biology, 1994, 240(3):188-192.
[16] STEVENS A J, BROWN Z Z, SHAH N H, et al.Design of a split intein with exceptional protein splicing activity[J].Journal of the American Chemical Society, 2016, 138(7):2162-2165.
[17] PAATERO A, ROSTI K, SHKUMATOV A V, et al.Crystal structure of an engineered LRRTM2 synaptic adhesion molecule and a model for neurexin binding[J].Biochemistry, 2016, 55(6):914-926.
[18] 张玲敏. 南极假丝酵母脂肪酶B在黑曲霉中的分泌表达及硅藻土固定化应用研究[D].广州:华南理工大学, 2019. ZHANG L M.Secretory expression of Candida antarctica lipase B in Aspergillus niger and its application in diatomite immobilization[D].Guangzhou:South China University Of Technology, 2019.
[19] HAYAT S M G, FARAHANI N, GOLICHENARI B, et al.Recombinant protein expression in Escherichia coli (E.coli):What we need to know[J].Current Pharmaceutical Design, 2018, 24(6):718-725.
[20] STERNKE M, TRIPP K W, BARRICK D.The use of consensus sequence information to engineer stability and activity in proteins[J].Methods in Enzymology, 2020, 643:149-179.
[21] ANBAR M, GUL O, LAMED R, et al.Improved thermostability of Clostridium thermocellum endoglucanase Cel8A by using consensus-guided mutagenesis[J].Applied and Environmental Microbiology, 2012, 78(9):3458-3464.
[22] LEHMANN M, LOCH C, MIDDENDORF A, et al.The consensus concept for thermostability engineering of proteins further proof of concept[J].Protein Engineering, 2002, 15(5):403-411.
[23] KIM H S, LE Q A T, KIM Y H.Development of thermostable lipase B from Candida antarctica (CalB) through in silico design employing B-factor and RosettaDesign[J].Enzyme and Microbial Technology, 2010, 47(1-2):1-5.
[24] LE Q A T, JOO J C, YOO Y J, et al.Development of thermostable Candida antarctica lipase B through novel in silico design of disulfide bridge[J].Biotechnology and Bioengineering, 2012, 109(4):867-876.
[25] 温露文, 徐岩, 喻晓蔚.理性设计提高酯合成催化反应脂肪酶的热稳定性[J].微生物学通报, 2020, 47(7):2106-2118. WEN L W, XU Y, YU X W.Rational design to improve lipase thermostability for ester synthesis[J].Microbiology, 2020, 47(7):2106-2118.
[26] XIE Y, AN J, YANG G Y, et al.Enhanced enzyme kinetic stability by increasing rigidity within the active site[J].Journal of Biological Chemistry, 2014, 289(11):7994-8006.
[27] SU B M, WU D Y, XU X Q, et al.Design of a PL18 alginate lyase with flexible loops and broader entrance to enhance the activity and thermostability[J].Enzyme and Microbial Technology, 2021, 151:109916.
[28] SIDDIQUI K S.Defying the activity-stability trade-off in enzymes:taking advantage of entropy to enhance activity and thermostability[J].Critical Reviews in Biotechnology, 2017, 37(3):309-322.