腈水合酶(nitrile hydratase,NHase;EC4.2.1.84)[1-2]是一种金属酶,可以在温和的反应条件下催化腈类物质生成对应的酰胺[3]。虽然腈水合酶已经成功应用于烟酰胺和丙烯酰胺的大规模工业生产[4-5],但是其完整的催化机制仍然不为人知,学者们已经为此进行大量的实验和理论研究[6]。
常规的腈水合酶有异源的α和β两个亚基[7-8],其可以结合成异源二聚体、四聚体、或者更高的聚合形式[9]。存在于腈水合酶基因簇下游的调控蛋白基因几乎存在于已知的所有腈水合酶,它对腈水合酶的金属摄取和翻译后修饰是必不可少的[10-12](唯一例外的是来自于M.brevicollis的CtNHase,至今没有关于它的调控蛋白的报道[13])。根据活性中心金属离子的不同可以将腈水合酶分为铁型腈水合酶(Fe-NHase)和钴型腈水合酶(Co-NHase)。所有已知腈水合酶的金属结合位点都存在于α亚基上[14],它们都含有一段共同的金属结合基序[CXLC(SO2H)SC(SOH)],这段序列包含了2个氧化的半胱氨酸[7, 15]。质谱已经证明2个氧化的半胱氨酸和金属离子组成了一个独特的配位结构,被称为“爪形”结构[16-17]。
硫氰酸水解酶(SCNase)是一种主要来自于硫杆菌属的含钴酶,能够催化焦化废水中的主要成分-硫氰酸盐降解(SCN-+2H2O→COS+NH3+OH-),它由α,β,γ 3个亚基组成[18]。γ亚基与腈水合酶的α亚基有很高的序列相似度,α亚基和β亚基分别与腈水合酶β亚基的N端和C端相似。硫氰酸水解酶与腈水合酶同属一个超家族,虽然SCNase在结构上与Co-NHase有很高的相似性,包括一个独特的低自旋三价Co催化中心和2个氧化的半胱氨酸配体,但是它们只作用于各自的底物[19]。研究表明它们的底物结合口袋有差异,这可能是其活性不互通的原因[20]。SCNase可以降解硫氰酸盐,这种物质通常在采矿、纺织的工业废水中大量存在,不仅对环境造成污染,对人类也有严重的毒害作用。因此对硫氰酸盐的研究可能帮助解决工业废水的排污问题。
通过在公共数据库里搜寻,我们发现了一种来源于Roseomonas stagni的新基因型的SCNase,其3个亚基的Genbank编号分别为SAMN02745775_103231、SAMN02745775_103232和SAMN02745775_103230。这3个亚基中SAMN02745775_103231所编码的亚基与腈水合酶α亚基有较高的相似度,其余2个亚基分别与常规腈水合酶β亚基的N端和C端同源。本研究表达、纯化并且对这个特殊的腈类降解酶(Rose.s SCNase)进行了表征。有趣的是,Rose.s SCNase不仅能催化腈类底物,对硫氰酸钾也有催化活性,这是迄今为止发现的第一个存在活性互通的腈类降解酶。Rose.s SCNase可能对含氰基底物降解研究有巨大的帮助。
1 材料与方法
1.1 实验材料
由金唯智生物技术有限公司合成玫瑰单胞菌Rose.s SCNase基因,克隆到pET-Duet质粒上。Rose.s NHase在大肠杆菌BL21(DE3)中异源表达。吡啶和酰胺购自TCI(上海)发展有限公司。所有其他化学品都是通过商业途径获得的。
100 mmol/L KPB缓冲液:磷酸氢二钾13.95 g、磷酸二氢钾10.92 g,用磷酸二氢钾调节pH为7.4,定容至1 L。蛋白纯化用Binding buffer:10 mmol/L KPB缓冲液。Washing buffer:在上述Binding buffer中加入终浓度为500 mmol/L的氯化钠。
1.2 实验方法
1.2.1 生物信息分析
利用NCBI-blast工具,将含有CTLCSC基序的酶进行初筛。调查研究背景,利用DNAMAN对酶活性中心的氨基酸序列进行复筛。最终选择来源于玫瑰单胞菌Roseomonas stagni的腈水合酶(Rose.s SCNase)进行研究。并利用AlphaFold对所选酶进行三维结构预测。
1.2.2 表达载体的构建及表达
将Rose.s SCNase进行密码子优化,将优化好的序列送至金唯智公司进行基因合成。各亚基前添加T7启动子和优化过的RBS序列(AAGGAGATATAGAT)使其共表达。选择pET-Duet-1为连接载体,基因插入位置为NdeⅠ和EcoRⅠ2个酶切位点。将连接成功的质粒pETDuet-1-Rose.s SCNase转化至BL21(DE3)感受态细胞,涂布于含有100 μg/mL氨苄青霉素的固体LB平板上。37 ℃倒放静置过夜培养。挑取单菌落接于含有100 μg/mL氨苄青霉素的5 mL LB液体培养基的试管中。37 ℃,200 r/min培养8 h左右。将试管种子液以1%的接种量转接至含有100 μg/mL氨苄青霉素的2YT培养基摇瓶中。37 ℃、200 r/min培养至OD600=0.6~0.8时[21],添加不同终浓度为0.2、0.4、0.6、0.8 mmol/L 异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)和0.05、0.1、0.2、0.3 g/L Co2+的诱导剂,25 ℃,200 r/min诱导16 h。离心收菌并用10 mmol/L KPB重悬、破碎细胞、获取上清液并通过SDS-PAGE分析蛋白表达情况。
1.2.3 酶的分离纯化及浓度检测
因标签纯化该酶效果较差,故采用传统硫酸铵沉淀、阴离子层析交换、凝胶柱过滤的方法分布进行纯化。具体步骤为将细胞破碎获取上清液定容至30 mL。参照硫酸铵饱和度计算表(0 ℃),逐步缓慢加入质量分数依次为20%、40%、60%及80%的硫酸铵粉末,边缓慢加入边搅拌直至硫酸铵充分溶解。过程中要严格监测pH值和温度变化,防止蛋白到达等电点或高温变性沉淀。过程中可利用氨水和磷酸二氢钾缓冲对调节pH值。每加完一个浓度梯度后继续搅拌30 min,然后用离心机以12 000 r/min离心 20 min,重悬沉淀并收集上清液用于下一个梯度的硫酸铵沉淀。如此往复,直到硫酸铵溶液的饱和度为80%。每次收集的沉淀用适量10 mmol/L KPB缓冲液重悬,用于SDS-PAGE分析,由此确定目的蛋白所在的沉降区间,为下一次操作简化步骤。获得的含有目的蛋白的沉淀用10 mmol/L KPB缓冲液重悬至10 mL,并在4 ℃层析柜中透析12 h,期间每4 h更换一次透析液来去除高浓度的盐离子。
Rose.s SCNase的理论等电点在5左右,所以选取DEAE阴离子交换柱,进一步纯化透析好的蛋白[22]。首先用Binding-Buffer平衡,然后上样,再用Washing-Buffer进行线性洗脱(0~500 mmol/L NaCl),将不同UV吸收峰的蛋白进行收集,SDS-PAGE电泳鉴定目的蛋白的洗脱范围[23]。
凝胶过滤层析对以上获得的蛋白样品中含有分子质量差异较大杂蛋白的样品进行分离。本研究中使用的凝胶柱为Superdex G200。首先用10 mmol/LKPB缓冲液以0.5 mL/min的流速平衡柱子,通过Loop环注入少量样品(300~500 μL),切换流动线路使样品进入凝胶柱中进行分离。最后根据吸收值再进行SDS-PAGE电泳分析。得到的蛋白用Brandford法测定蛋白质量浓度后稀释至质量浓度1 mg/mL备用。
1.2.4 酶活力检测
本研究中腈水合的活性通过催化腈类生成相应的酰胺量来计算[24]。反应体系为490 μL烟腈(200 mmol/L)及 10 μL酶溶液,在不同温度的金属浴中反应10 min,然后加入500 μL纯乙腈终止反应。反应液使用0.22 μm的有机微孔过滤膜过滤,用HPLC检测样品。HPLC使用的色谱柱为C18反相色谱柱,检测波长为215 nm,流动相为体积比为1∶2的乙腈与水的混合溶液。根据不同浓度烟酰胺标准品的对应吸收值作标准曲线,并依此计算样品中的烟酰胺浓度。催化烟腈的酶活力单位定义为:在相应条件下每分钟催化烟腈生成1 μmol烟酰胺所需要的酶量。催化丙烯腈的酶活力单位定义为:在相应条件下每分钟催化丙烯腈生成1 μmol丙烯酰胺所需要的酶量。
水解硫氰酸盐的活性通过催化硫氰酸钾生成的铵盐的量来计算,铵盐检测使用铵盐检测试剂盒(美国Sigma公司)。反应体系为450 μL硫氰酸钾(50 mmol/L)及50 μL酶溶液,在不同温度的金属浴中反应10 min,然后100 ℃加热20 min终止反应。硫氰酸水解酶的酶活力单位定义为:在相应条件下每分钟催化硫氰酸钾生成1 μmol铵盐所需要的酶量。
1.2.5 酶学性质
最适温度:分别测定该酶在20、30、40、50、60、70 ℃下的酶活力。
最适pH:配制含有终浓度为200 mmol/L烟腈、丙烯腈和终浓度为50 mmol/L硫氰酸钾的不同pH梯度(pH=4、5、6、7、8、9)的10 mmol/L磷酸钾缓冲液,测定该酶在各pH底物缓冲溶液下的酶活力,分析其随pH的变化情况。
2 结果与分析
2.1 Rose.s NHase基因的异源表达及其分离纯化
图1为用于表达Rose.s SCNase的质粒示意图。pETDuet-1作为载体,RBS是核糖体结合位点,质粒上带有氨苄青霉素抗性基因。
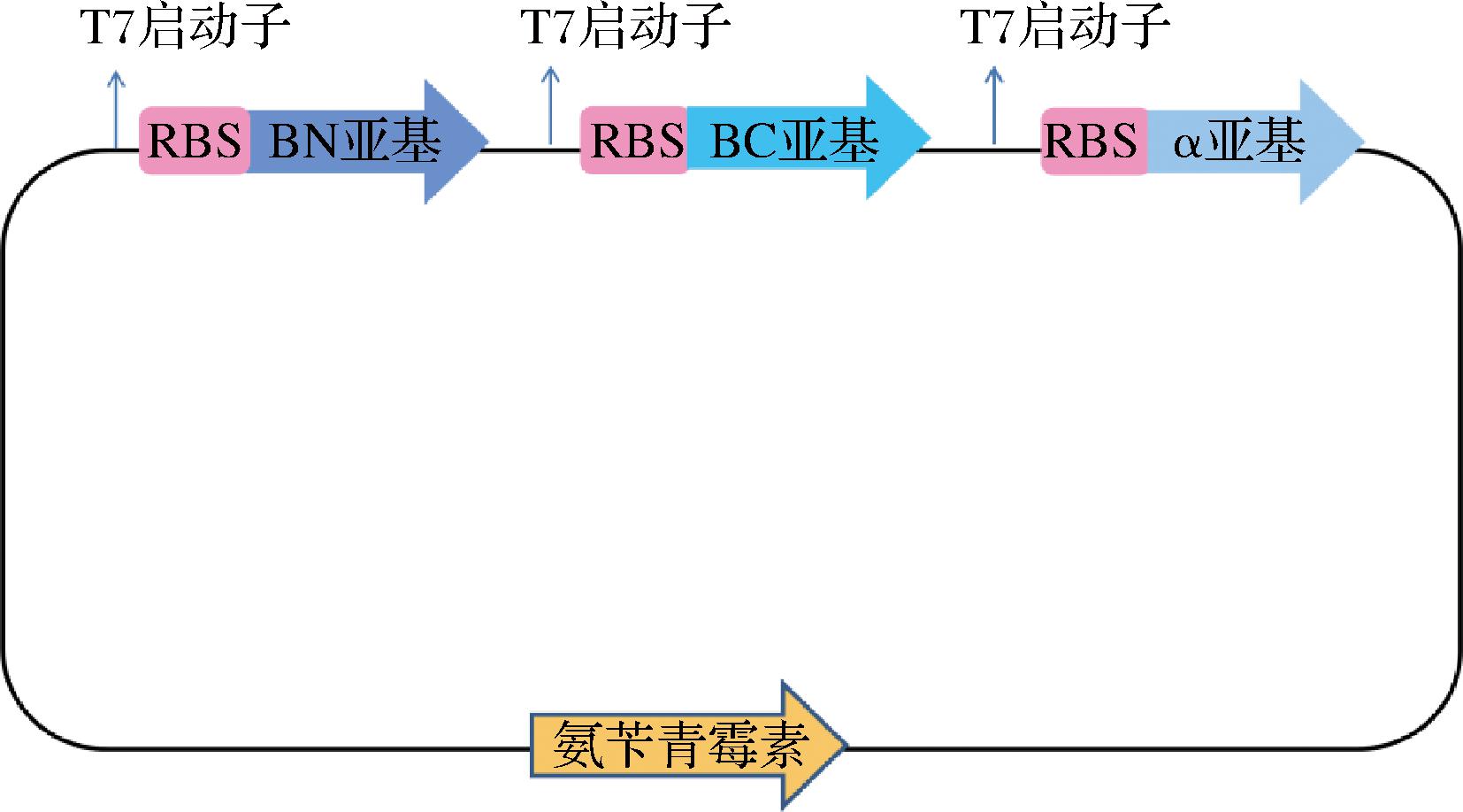
图1 表达含有βN、βC、α亚基的Rose.s SCNase的质粒
Fig.1 Expression plasmid of Rose.s SCNase containing βN, βC and α subunits
优化剂浓度初步的确定参照文献[25-26]的要求。Co2+对腈水合酶翻译后修饰和发挥活性具有重要作用,过高浓度的Co2+会抑制菌体生长从而影响蛋白的表达。IPTG也对酶的诱导表达有影响。因此对Co2+ 和IPTG浓度进行优化,结果如图2所示,从SDS-PAGE图中确定最佳诱导条件为Co2+ 0.1 g/L、IPTG 0.6 mmol/L。

图2 优化诱导表达条件的SDS-PAGE分析
Fig.2 SDS-PAGE analysis of optimized induced expression conditions
因Strep亲和层析纯化效果不佳,因此在纯化策略上选择硫酸铵沉淀-阴离子交换-凝胶柱分步的方法。分级硫酸铵沉淀的结果如图3-a所示,目的蛋白在饱和度20%~40%析出最多,通过硫酸铵沉淀,基本去除了杂蛋白。该酶的理论等电点为5.2,因此采用DEAE阴离子柱交换进行进一步纯化,最后经过凝胶柱分离,得到了纯度>95%的蛋白,成功实现了其纯化(图3-b)。

a-Rose.s SCNase分级硫铵沉淀样品的SDS-PAGE分析; b-Rose.s SCNase纯化的SDS-PAGE分析
图3 Rose.s SCNase纯化过程的SDS-PAGE分析
Fig.3 SDS-PAGE analysis of the purification process of Rose.s SCNase
2.2 酶学性质的分析
不同的反应温度对Rose.s SCNase酶活的影响如图4-a所示,该酶对腈类和硫氰酸钾底物的最适温度均为40 ℃。催化腈类底物的最适pH为7.0,降解硫氰酸钾的最适pH为6.0(图4-b)。这表明Rose.s SCNase是中性的。
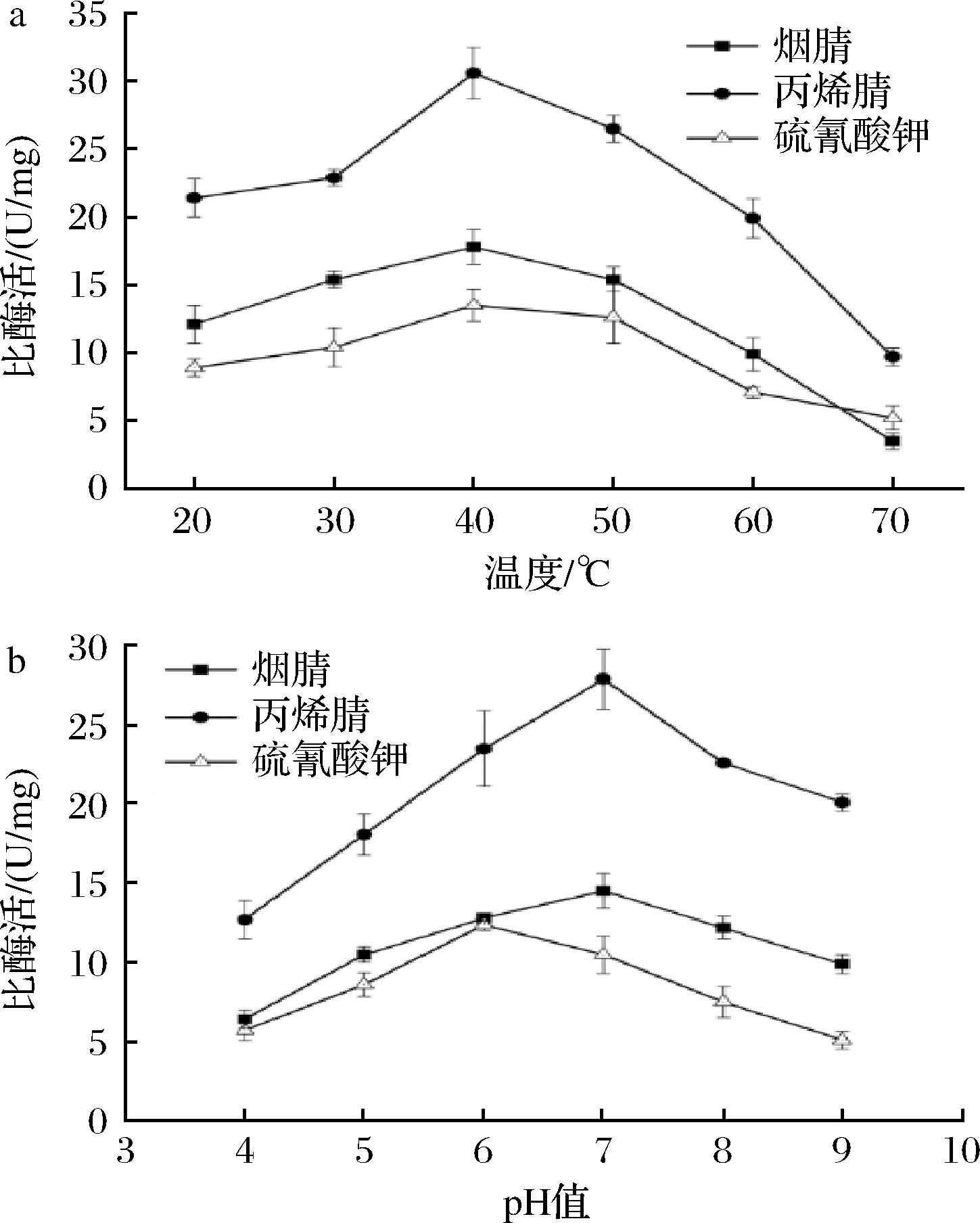
a-不同底物的最适温度;b-不同底物的最适pH
图4 Rose.s SCNase的酶学性质
Fig.4 Enzymatic properties of Rose.s SCNase
与其他腈类降解酶相比,Rose.s SCNase最为独特的是它同时拥有硫氰酸水解酶和腈水合酶2种酶的活性。该酶在最适pH和温度下对烟腈的比酶活为(17.8±1.3) U/mg,对丙烯腈的比酶活为(30.6±1.9) U/mg,对硫氰酸钾的比酶活为(13.5±0.9) U/mg。具体见表1。
表1 Rose.s Nhase最适条件下的活性
Table 1 activity of Rose.s Nhase under optimum conditions

底物比酶活/(U/mg)烟腈17.8±1.3丙烯腈30.6±1.9硫氰酸钾13.5±0.9
2.3 序列比对
将Rose.s SCNase的氨基酸序列与Th-Scnase、Pt-NHase的序列进行比对,结果如图5所示。Rose.s SCNase与Th-Scnase和Pt-NHase α亚基的相似度为65.05%,β亚基的相似度为36.32%。且均含有钴离子结合位点CTLCSC。

a-α亚基序列对比;b-β亚基序列比对
图5 Rose.s SCNase与Th-SCNase和ptNhase的序列比对
Fig.5 Enzymatic properties of Rose.s SCNase
注:红色表示在该位置有相同的氨基酸。
2.4 结构分析
图6为使用AlphaFold预测得到的Rose.s SCNase的结构。用Verify 3D服务器对获得的模型进行评估,最高得分为99.16%,表明所得的三维结构比较可靠。用所获得的结构进行下一步结构比对。

图6 Rose.s NHase模拟结构图
Fig.6 Model diagram of Rose.s NHase
根据之前的报道,腈水合酶和硫氰酸水解酶虽属于同一家族但活性并不互通。2014年有学者研究发现是其底物结合口袋不同导致了不同的底物选择性[20]。作者将硫氰酸水解酶底物结合口袋顶部2个精氨酸分别突变成了苯丙氨酸Phe和色氨酸Trp,这2个突变体丧失了降解硫氰酸盐的活性,同时却产生了腈水合酶的活性。基于这一研究,我们将Rose.s SCNase与嗜热假诺卡氏菌P.thermophila JCM 3095腈水合酶(Pt-NHase)和Thiobacillus thioparus THI115来源的硫氰酸水解酶(Th-SCnase)进行结构比对,二者的RMSD值分别为0.96和0.88,这表明这株新的腈类降解酶与腈水合酶和硫氰酸水解酶均有较高的结构相似度。结构对比上更偏向硫氰酸水解酶,所以本研究中命名其为Rose.s SCNase。
比对结果显示三者的底物结合口袋非常相似。不同之处在于活性金属上方顶部空间的氨基酸(图7)。Rose.s SCNase和Th-SCnase在此位置是精氨酸Arg,精氨酸侧链向口袋延伸,可使底物结合口袋顶部空间呈正电,更有利于结合硫氰酸盐底物。而Pt-NHase对应的位置是色氨酸Trp,这个疏水基团可能更有利于其捕获腈类底物。且色氨酸向口袋顶部延伸,增高了顶部空间,更有利于腈类底物进入活性中心。
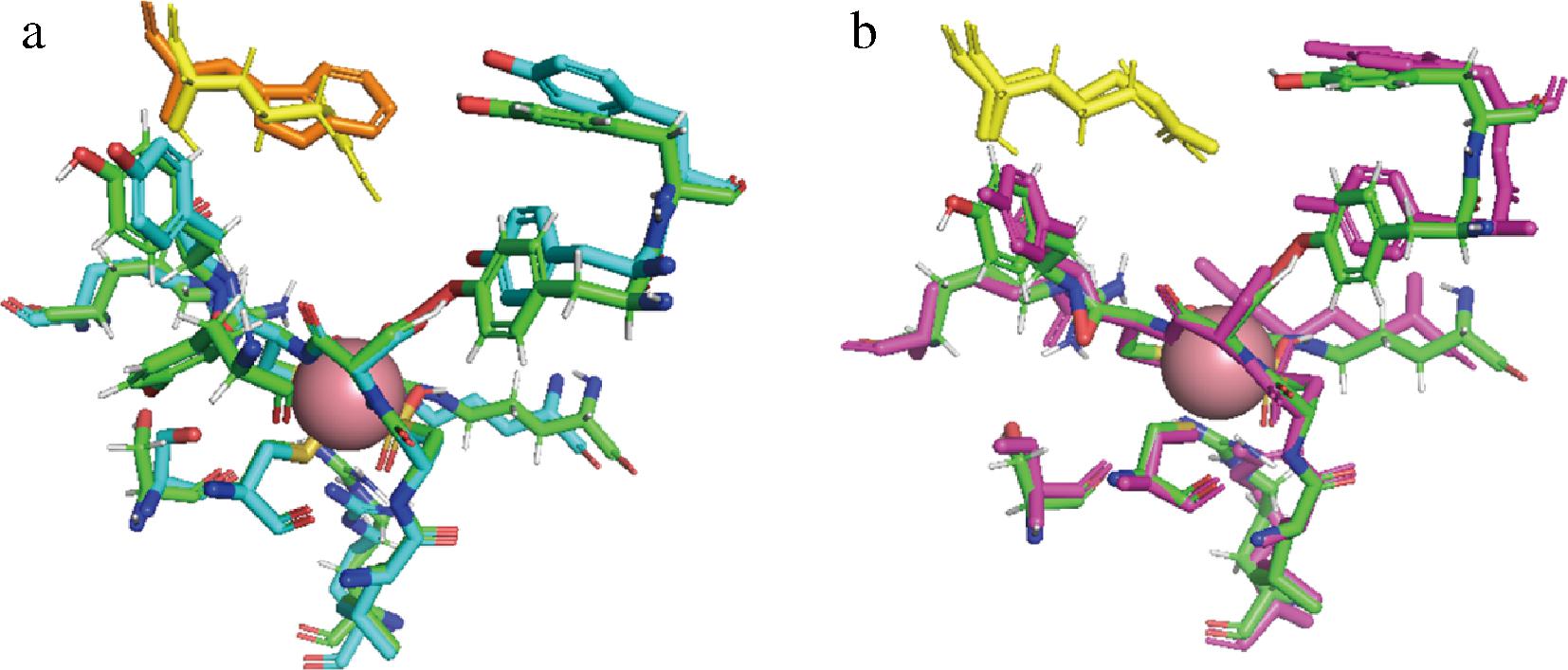
a-Rose.s SCNase与Pt-NHase结合口袋对比图; b-Rose.s SCNase与Th-SCnase结合口袋对比图
图7 Rose.s SCNase底物结合口袋比对图
Fig.7 Comparison of substrate binding pocket of Rose.s NHase
注:绿色为Rose.s SCNase,青色为Pt-NHase,紫色为Th-SCNase, 黄色代表关键位置上精氨酸Arg,橙色代表关键位置上的色氨酸Trp, 紫色球代表活性中心的Co。
3 结论
本文成功优化了Rose.s SCNase的异源表达,并通过硫酸铵沉淀、阴离子交换和凝胶柱过滤的方法实现了其纯化。与传统的腈水合酶不同的是Rose.s SCNase对腈类底物和硫氰酸钾均具有催化活性。Rose.s SCNase在最适条件下对烟腈的比酶活为(17.8±1.3) U/mg,对丙烯腈的比酶活为(30.6±1.9) U/mg,对硫氰酸钾的比酶活为(13.5±0.9) U/mg。比对Rose.s SCNase、Pt-NHase和Th-SCnase的底物结合口袋的氨基酸,发现活性金属顶部的氨基酸有差异,这可能控制了酶的选择性。
[1] KOBAYASHI M, SHIMIZU S.Metalloenzyme nitrile hydratase:Structure, regulation, and application to biotechnology[J].Nature Biotechnology, 1998, 16(8):733-736.
[2] ASANO Y, TANI Y, YAMADA H.A new enzyme “nitrile hydratase” which degrades acetonitrile in combination with amidase[J].Agricultural and Biological Chemistry, 1980, 44(9):2251-2252.
[3] CHEN J, ZHENG R C, ZHENG Y G, et al.Microbial transformation of nitriles to high-value acids or amides[J].Advances in Biochemical Engineering/Biotechnology, 2009, 113:33-77.
[4] NAGASAWA T, MATHEW C D, MAUGER J, et al.Nitrile hydratase-catalyzed production of nicotinamide from 3-cyanopyridine in Rhodococcus rhodochrous J1[J].Applied and Environmental Microbiology, 1988, 54(7):1766-1769.
[5] PRASAD S, BHALLA T C.Nitrile hydratases (NHases):At the interface of academia and industry[J].Biotechnology Advances, 2010, 28(6):725-741.
[6] NELP M T, SONG Y, WYSOCKI V H, et al.A protein-derived oxygen is the source of the amide oxygen of nitrile hydratases[J].Journal of Biological Chemistry, 2016, 291(15):7822-7829.
[7] KOVACS J A.Synthetic analogues of cysteinate-ligated non-heme iron and non-corrinoid cobalt enzymes[J].Chemical Reviews, 2004, 104(2):825-848.
[8] YAMADA H, KOBAYASHI M.Nitrile hydratase and its application to industrial production of acrylamide[J].Bioscience, Biotechnology, and Biochemistry, 1996, 60(9):1391-1400.
[9] HOURAI S, MIKI M, TAKASHIMA Y, et al.Crystal structure of nitrile hydratase from a thermophilic Bacillus smithii[J].Biochemical and Biophysical Research Communications, 2003, 312(2):340-345. [10] WU S, FALLON R D, PAYNE M S.Over-production of stereoselective nitrile hydratase from Pseudomonas putida 5B in Escherichia coli:Activity requires a novel downstream protein[J].Applied Microbiology and Biotechnology, 1997, 48(6):704-708.
[11] NOJIRI M, YOHDA M, ODAKA M, et al.Functional expression of nitrile hydratase in Escherichia coli:Requirement of a nitrile hydratase activator and post-translational modification of a ligand cysteine[J].J The Journal of Biochemistry, 1999, 125(4):696-704.
[12] HASHIMOTO Y, NISHIYAMA M, HORINOUCHI S, et al.Nitrile hydratase gene from Rhodococcus sp.N-774 requirement for its downstream region for efficient expression[J].Bioscience, Biotechnology, and Biochemistry, 1994, 58(10):1859-1865.
[13] MARTINEZ S, YANG X H, BENNETT B, et al.A cobalt-containing eukaryotic nitrile hydratase[J].Biochimica et Biophysica Acta.Proteins and Proteomics, 2017, 1 865(1):107-112.
[14] MURAKAMI T, NOJIRI M, NAKAYAMA H, et al.Post-translational modification is essential for catalytic activity of nitrile hydratase[J].Protein Science:A Publication of the Protein Society, 2000, 9(5):1024-1030.
[15] CHENG Z Y, XIA Y Y, ZHOU Z M.Recent advances and promises in nitrile hydratase:From mechanism to industrial applications[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:352.
[16] TSUJIMURA M, DOHMAE N, ODAKA M, et al.Structure of the photoreactive iron center of the nitrile hydratase from Rhodococcus sp.N-771.Evidence of a novel post-translational modification in the cysteine ligand[J].The Journal of Biological Chemistry, 1997, 272(47):29454-29459.
[17] NOGUCHI T, NOJIRI M, TAKEI K I, et al.Protonation structures of Cys-sulfinic and Cys-sulfenic acids in the photosensitive nitrile hydratase revealed by Fourier transform infrared spectroscopy[J].Biochemistry, 2003, 42(40):11642-11650.
[18] ARAKAWA T, KAWANO Y, KATAOKA S, et al.Structure of thiocyanate hydrolase:A new nitrile hydratase family protein with a novel five-coordinate cobalt(III) center[J].Journal of Molecular Biology, 2007, 366(5):1497-1509.
[19] KATAYAMA Y, HASHIMOTO K, NAKAYAMA H, et al.Thiocyanate hydrolase is a cobalt-containing metalloenzyme with a cysteine-sulfinic acid ligand[J].Journal of the American Chemical Society, 2006, 128(3):728-729.
[20] YAMANAKA Y, ARAKAWA T, WATANABE T, et al.Two arginine residues in the substrate pocket predominantly control the substrate selectivity of thiocyanate hydrolase[J].Journal of Bioscience and Bioengineering, 2013, 116(1):22-27.
[21] XIA Y Y, PEPLOWSKI L, CHENG Z Y, et al.Metallochaperone function of the self-subunit swapping chaperone involved in the maturation of subunit-fused cobalt-type nitrile hydratase[J].Biotechnology and Bioengineering, 2019, 116(3):481-489.
[22] ZHOU Z M, HASHIMOTO Y, CUI T W, et al.Unique biogenesis of high-molecular mass multimeric metalloenzyme nitrile hydratase:Intermediates and a proposed mechanism for self-subunit swapping maturation[J].Biochemistry, 2010, 49(44):9638-9648.
[23] GUO J L, CHENG Z Y, BERDYCHOWSKA J, et al.Effect and mechanism analysis of different linkers on efficient catalysis of subunit-fused nitrile hydratase[J].International Journal of Biological Macromolecules, 2021, 181:444-451.
[24] CHENG Z Y, JIANG S J, ZHOU Z M.Substrate access tunnel engineering for improving the catalytic activity of a thermophilic nitrile hydratase toward pyridine and pyrazine nitriles[J].Biochemical and Biophysical Research Communications, 2021, 575:8-13.
[25] 张晓欢, 崔文璟, 周哲敏.高分子量腈水合酶在大肠杆菌中的表达策略及重组菌的细胞催化[J].微生物学通报, 2016, 43(10):2121-2128. ZHANG X H, CUI W J, ZHOU Z M.Strategy of high molecular mass nitrile hydratase expression in Escherichia coli and the whole-cell catalysis by the recombinant strains[J].Microbiology China, 2016, 43(10):2121-2128.
[26] 张赛兰, 李婷, 程中一, 等.新型耐热腈水合酶的异源表达及其催化工艺研究[J].食品与发酵工业, 2020, 46(14):108-113. ZHANG S L, LI T, CHENG Z Y, et al.Heterologous expression of a novel thermostable nitrile hydratase and its catalytic process[J].Food and Fermentation Industries, 2020, 46(14):108-113.