肉类和肉制品掺假问题已成为世界各地的共同问题,2014年欧洲报道的“马肉丑闻”更是揭露了多国肉制品的掺假现象[1]。牛肉造假的主要原因是部分商家为节省成本,通过添加牛肉香精、染料等化学物质,将价格较低的猪鸡鸭等肉全部或部分代替价格较高的牛肉。牛肉造假主要分为生牛肉造假、牛肉生制品造假(如肥牛卷、调制牛排等)以及牛肉熟制品造假(如牛肉干、牛肉丸等)。由于掺假过程不标明掺假成分,从而导致消费者面临接触病原体和过敏原等健康风险[2-3]。因此,发展牛肉制品的掺假测定及真伪鉴别等安全检测手段对于规范肉类市场秩序及震慑不法分子具有重要的意义,更是对习总书记要求保护民众“舌尖上的安全”的践行。
常见的肉类鉴别技术主要有免疫学检测方法[4]、光谱技术[5]及聚合酶链式反应技术[6]。其中,免疫学检测基于抗原抗体特异结合的原理,具有灵敏性高的特点,但测试过程耗时长、操作繁琐,难以实现待检测物质的高通量鉴别[7];光谱技术检测基于被检测原子或分子在激发状态下发射特征光谱的原理,虽然能够快速、无损地进行肉制品掺假鉴别,但是由于光谱数据容易受到外加添加剂的干扰,存在不法分子借此逃过检查的可能,且模型的稳定性易受光谱采集参数和环境因素影响[5],因此重现性差;聚合酶链式反应技术依靠核酸来鉴别肉及肉制品是否掺假,具有稳定性高、灵敏度高的特点,检测更准确[8],但是反应耗时较长、步骤复杂、成本较高,易出现假阳性的风险[9]。最重要的是,经过熟加工,由于蛋白质、脂肪、核酸受热变性,食品添加剂(如调味料、防腐剂、色素)的加入,上述方法对熟肉制品进行检测的成本与难度随之增大。
基质辅助激光解析电离飞行时间质谱(matrix-assisted laser desorption/ionization time of flight mass spectrometry,MALDI TOF-MS)是一种生物质谱技术,具有高灵敏度、高通量、快速、准确等优点[10-12],利用软电离,能在保持大分子完整性的前提下得到质谱图。由于其能快速、无损地获得分析物质谱图,因此常被用于非靶向性分析,通过综合多个特征峰完成鉴别,具有高稳定性、高抗污染性的优点,且相较于其他手段,质谱能直接获得样品信息,使得造假行为难以遁形。目前,该项技术已在癌症[13]、心血管疾病[14]等临床领域研究和食品微生物检测[15]中取得显著进展。近年来在肉类的鉴别中也有了初步探索,如对猪、牛、马、鸡的鲜肉和烹制后肉制品,以及鱼类肉制品进行MALDI TOF-MS表征后,利用聚类分析(hierarchical cluster analysis, HCA)进行了鉴别[16-17]。
针对牛肉制品的掺假鉴别,本研究将MALDI TOF-MS 与机器学习算法随机森林和主成分分析(principal component analysis, PCA)相结合,充分发挥非靶向性分析的优势,在模型建立过程中:通过MALDI TOF-MS表征得到生肉和熟肉蛋白质质谱图(包括鸡、鸭、猪、牛的不同食用部位),经过 R 语言MALDIquant包[18]完成数据预处理后,利用随机森林算法对生、熟条件下4种肉进行分析,筛选能代表在生熟不同状态下4种肉的特征蛋白质指纹图谱,结合PCA构建4种肉的分类模型,以达到鉴别牛肉掺假的目的。通过HCA验证分类模型的正确性,最后通过检测鸡、鸭、猪分别以不同比例混入牛肉的掺假样本,以验证牛肉掺假鉴别的正确性。
1 材料与方法
1.1 实验材料
本实验所用肉类样品(牛、猪、鸡和鸭),汕头鮀浦当地市场;硫脲、尿素、三羟基氨基甲烷(Tris)、乙腈,均为色谱级,上海阿拉丁生化科技股份有限公司;三氟乙酸(质谱级),芥子酸(98%)、MD25-1000型透析袋,Sigma-Aldrich;有机相滤头(PVDF, 0.45 μm),津隆;一次性针器注射器,江苏治宇医疗器械。
1.2 仪器与设备
LC-SFJ-10手持匀质匀浆机,上海力辰科技有限公司;autoflex speed飞行时间质谱仪,布鲁克(北京)科技有限公司;TGL-16Gr台式冷冻离心机,上海安亭科学仪器厂;PHS-3E酸度计,上海仪电科学仪器。
1.3 实验方法
1.3.1 样品信息
肉类样品从当地市场购得后立即放入冰袋,转入4 ℃下冷藏保鲜。测试前,随机切取10.0 g肌肉部位组织,避免切入筋膜及脂肪。样品切碎混匀后,称取0.50 g肉样,共收集到如表1所示136个样本。
1.3.2 肉类样品蛋白质提取与纯化
0.50 g肉类样品放入玻璃匀浆器,加入10 mL蛋白质提取液(6 mol/L尿素, 1 mol/L硫脲, 50 mmol/L Tris,pH=8.2)[19],8 000 r/min匀浆2 min至无明显组织,4 ℃、12 000×g离心1 min,取上清液于0.45 μm 滤头过滤,将滤液装入透析袋,用100倍体积蒸馏水透析2 h。
表1 本实验所用的肉类样品信息(共136个鲜肉样品)
Table 1 Different tissue from 4 meat species adopted in the study (136 fresh meat samples)
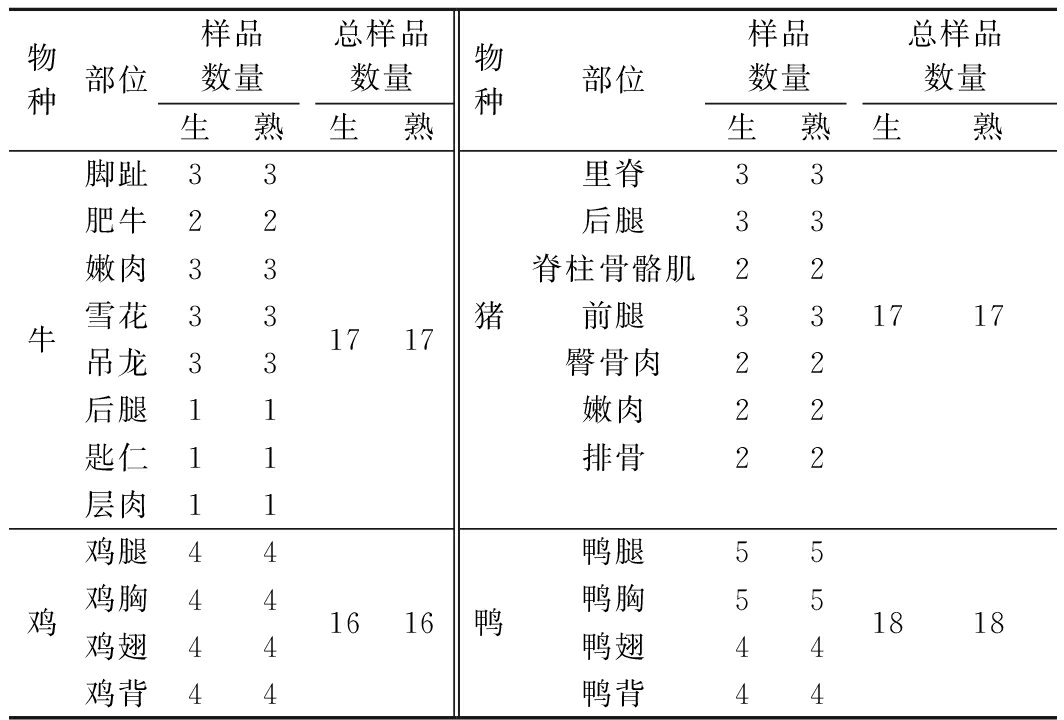
物种部位样品数量总样品数量生熟生熟物种部位样品数量总样品数量生熟生熟牛脚趾33肥牛22嫩肉33雪花33吊龙33后腿11匙仁11层肉111717猪里脊33后腿33脊柱骨骼肌22前腿33臀骨肉22嫩肉22排骨221717鸡鸡腿44鸡胸44鸡翅44鸡背441616鸭鸭腿55鸭胸55鸭翅44鸭背441818
1.3.3 MALDI-TOF MS表征
按照V(乙腈)∶V(0.1%三氟乙酸)=3∶7的比例配制TA30溶液,将透析除盐后的蛋白质溶液与TA30饱和芥子酸溶液等比例混合,取2.5 μL点在靶板上,每个样品重复点3次,干燥后用MALDI-TOF MS采集数据。平行实验2次。MALDI-TOF MS具体参数如下:激光频率为500 Hz,能量为99.6%,在正离子线性模式下扫描范围为4 000~22 000 Da。
1.3.4 数据预处理
利用R语言MALDIquant[18]包完成质谱数据的预处理,包括平滑、基线矫正、峰对齐、峰提取等,最终将质谱图谱转化为二维数据矩阵,之后利用基于Microsoft Excel 的归一化方法(MS total useful signal, MSTUS)[20]对数据矩阵进行归一化处理。
1.3.5 随机森林算法分析
随机森林算法是以决策树为基础构建的,与决策树类似,随机森林算法是通过多个“节点”,实现多次二分类,直至不能再分裂,而随机森林通过数据和特征的随机抽取构建多个彼此间不相关的决策树,共同完成分类预测,其随机性使得模型的泛化能力和抗过拟合性均得到了很大提升[21]。
利用R语言中的Random forest包[22]对所得的272个数据进行分类建模,训练集占比为80%(217),测试集占比为20%(55),筛选生或熟状态下均能代表牛肉、鸡肉、鸭肉或猪肉的重要变量。通过在质谱图中确认重要变量对应离子峰存在(信噪比>3),则确认其为特征蛋白质,并在Uniprot(蛋白质数据库)中比对检索特征蛋白质。随后将特征蛋白质作为新的变量进行随机森林分类模型的构建,以判断所筛选特征蛋白质的有效性。R语言版本为4.1.2,Random forest包的版本为4.6~14。通过一系列建模实验确定模型的参数ntree=50,mtry=24。
1.3.6 PCA和HCA
PCA将多元变量投影分布到几个不相关的主成分上,以达到数据降维的目的,实现同类数据的聚集和不同类数据的分离,同时实现数据可视化。将随机森林算法筛选的特征蛋白质作为PCA的新变量,构建4种肉的分类模型,随后通过HCA以验证分类模型,最后通过鉴别包括生熟的鸡肉、鸭肉和猪肉分别以25%、50%、75%质量比与牛肉混合制备的掺假样品,完成对模型鉴别能力的验证。PCA和HCA利用SIMCA-P14.1 软件完成。
2 结果与分析
2.1 四种肉的MALDI -TOF MS图谱
牛鸡鸭猪4种肉蛋白质的代表性MALDI-TOF MS图谱如图1所示,4个物种间质谱图离子峰存在较大差异,说明存在可以区分4种肉的特征蛋白质;而相同物种的肉在经过烹煮后,离子峰总数目均增大,同时也伴随一些离子峰的消失,说明在烹煮过程中,部分热不稳定的蛋白质发生水解。烹煮前后存在许多重合的离子峰,说明存在一些不易水解、性质稳定的蛋白质,具有作为能区分生熟状态下4种肉类特征蛋白质的潜力。
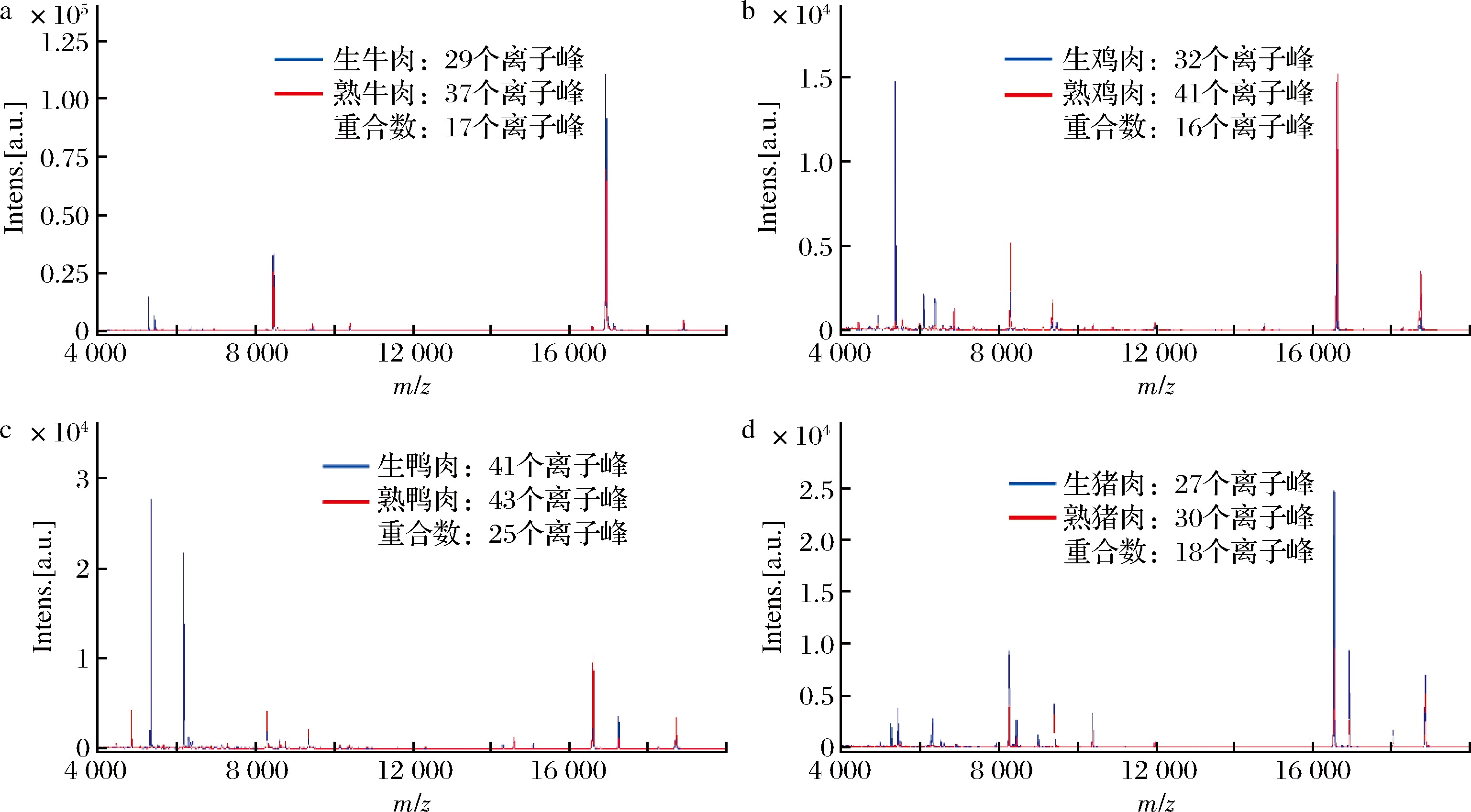
a-牛肉;b-鸡肉;c-鸭肉;d-猪肉
图1 4种肉(生熟)的MALDI-TOF MS图谱
Fig.1 MALDI-TOF MS spectra of 4 species meats (including fresh and cooked)
注:重合数代表同种肉生肉和熟肉间重合峰的数目
2.2 重要变量的筛选
在R语言中利用Random forest包在ntree=50、mtry=24进行分类模型的构建。通过这种方法建立的模型分类正确率为100%,袋外误差为0;在训练集内部与测试集的准确率均为100%, 受试者工作特征曲线(receiver operating characteristic curve, ROC)下与坐标轴围成的面积(area under curve, AUC)值均为1,表示训练集内部数据与测试集数据均能被正确分类。证实随机森林模型具有很好的分类能力,筛选出的重要变量可信度较高。
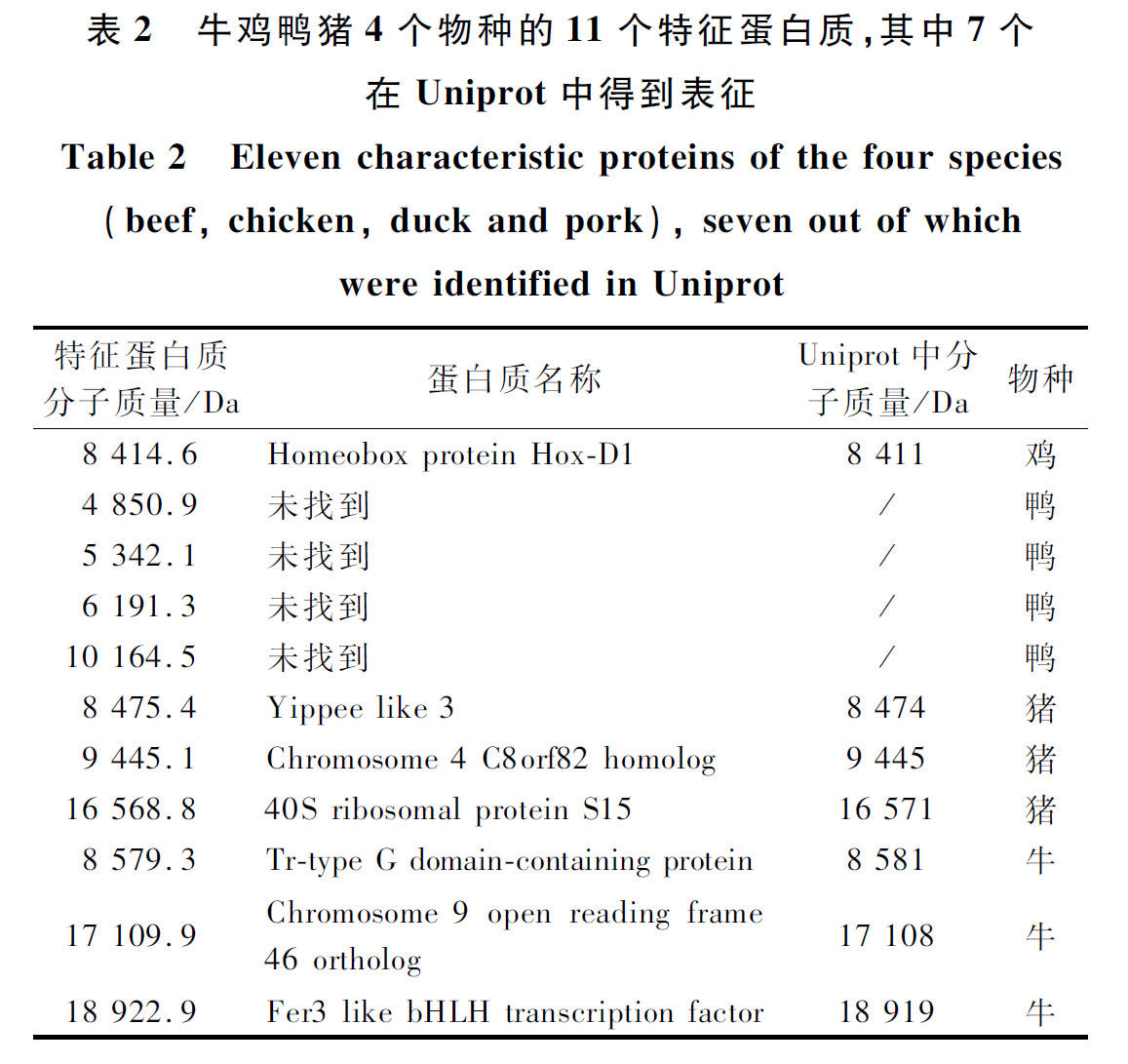
调用该模型的变量重要性输出,选取变量重要性较大的前11个变量(图2)作为重要变量。最终选取的变量为4 850.9、5 342.1、6 191.3、8 414.6、8 475.4、8 579.3、9 445.1、10 164.5、16 568.8、17 109.9及18 922.9。通过判断其在MALDI-TOF MS谱图中对应的离子峰的存在,确认这11个重要变量均为特征蛋白质。随后,通过在Uniprot中比对检索,如表2所示,共找到7个特征蛋白质。
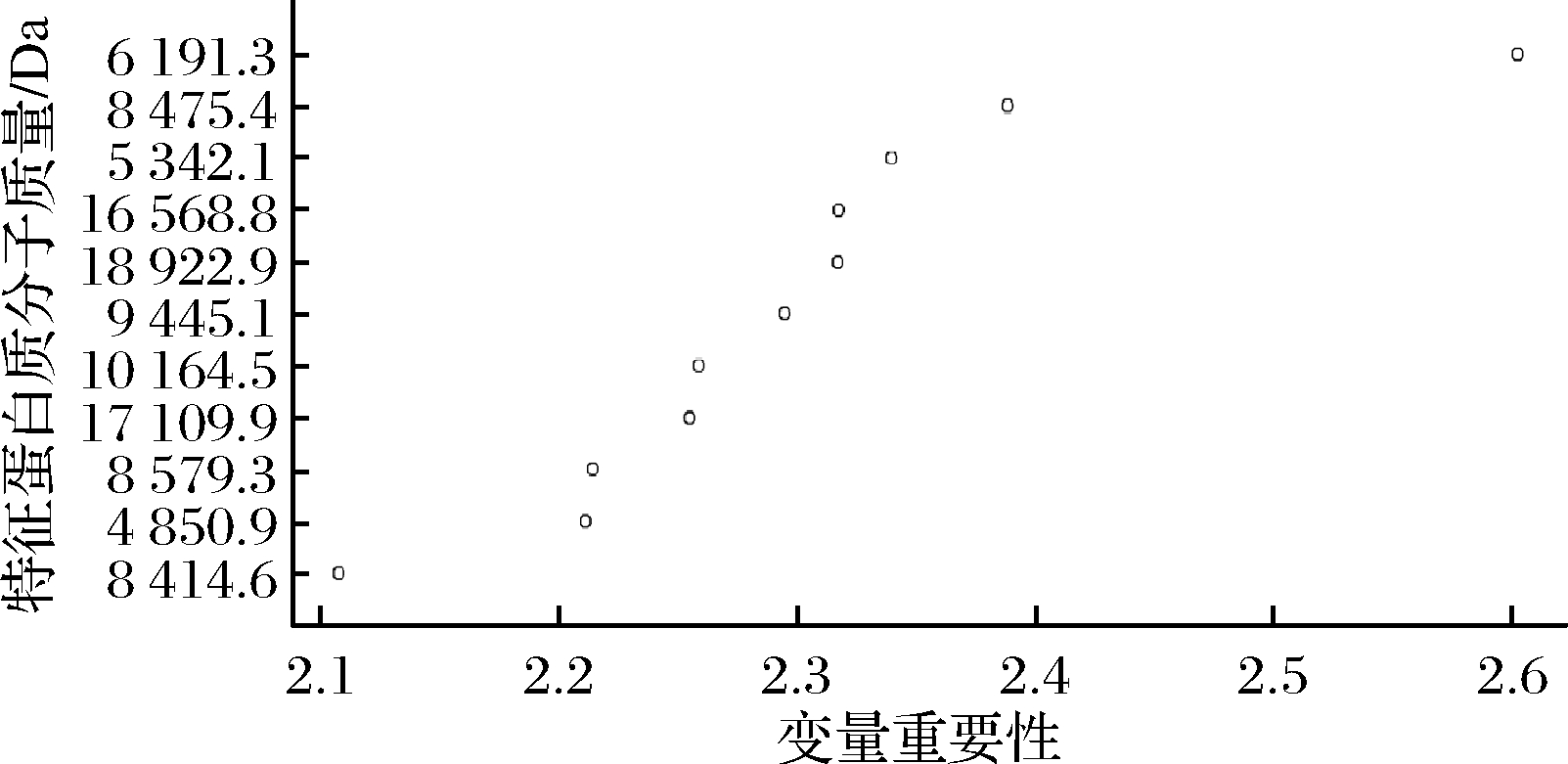
图2 随机森林算法的前11个重要变量
Fig.2 The 11 important variables of random forest
将上述11个特征蛋白质作为新的变量进行随机森林分类模型的构建。超参数设定为ntree=50、mtry=3, 训练集占比为80%(217),测试集占比为20%(55)。在新的模型中,用于构建模型的训练集被100%正确分类,袋外误差率为0,同时在训练集内部预测正确率为100%、AUC值为1、95%置信区间为(0.988 1,1),且对于4种肉的特异度、敏感度均达到了1;在测试集预测中,分类的正确率为100%、AUC值为1、95%置信区间为(0.952 6,1),特异度和敏感度均为1。所得到的模型仍然具有较好的分类能力。由此,可以验证选取的特征蛋白质具有很好的区分4种肉类的能力。
2.3 掺假判别模型的构建
2.3.1 PCA与HCA
4种肉质谱数据所有变量的PCA得分如图3-a所示,前2个主成分的积累贡献为R2=23.0%,说明模型对数据的拟合程度较差。各种肉间均有一定的聚类趋势,而牛肉和猪肉之间、鸭肉和鸡肉之间比较接近,反映了物种间亲缘关系的接近程度。将随机森林算法筛选得到的11个特征蛋白质作为变量进行PCA,其得分如图3-b所示,前2个主成分积累贡献率为R2=69.0%,模型对数据的拟合程度较好,特征蛋白质的选择使得主成分对变量变异性的解释能力得到了很大的提高。各物种肉均有明显的聚类趋势,能在2个主成分下得到分离。

a-生熟4种肉;b-11个特征蛋白质作为变量下的生熟4种肉
图3 四种肉类样品的PCA得分图
Fig.3 PCA scoring plot of four species meats
通过对数据进行HCA,结果显示不同物种的肉被正确地分为4类(图4),验证了PCA的可信性。
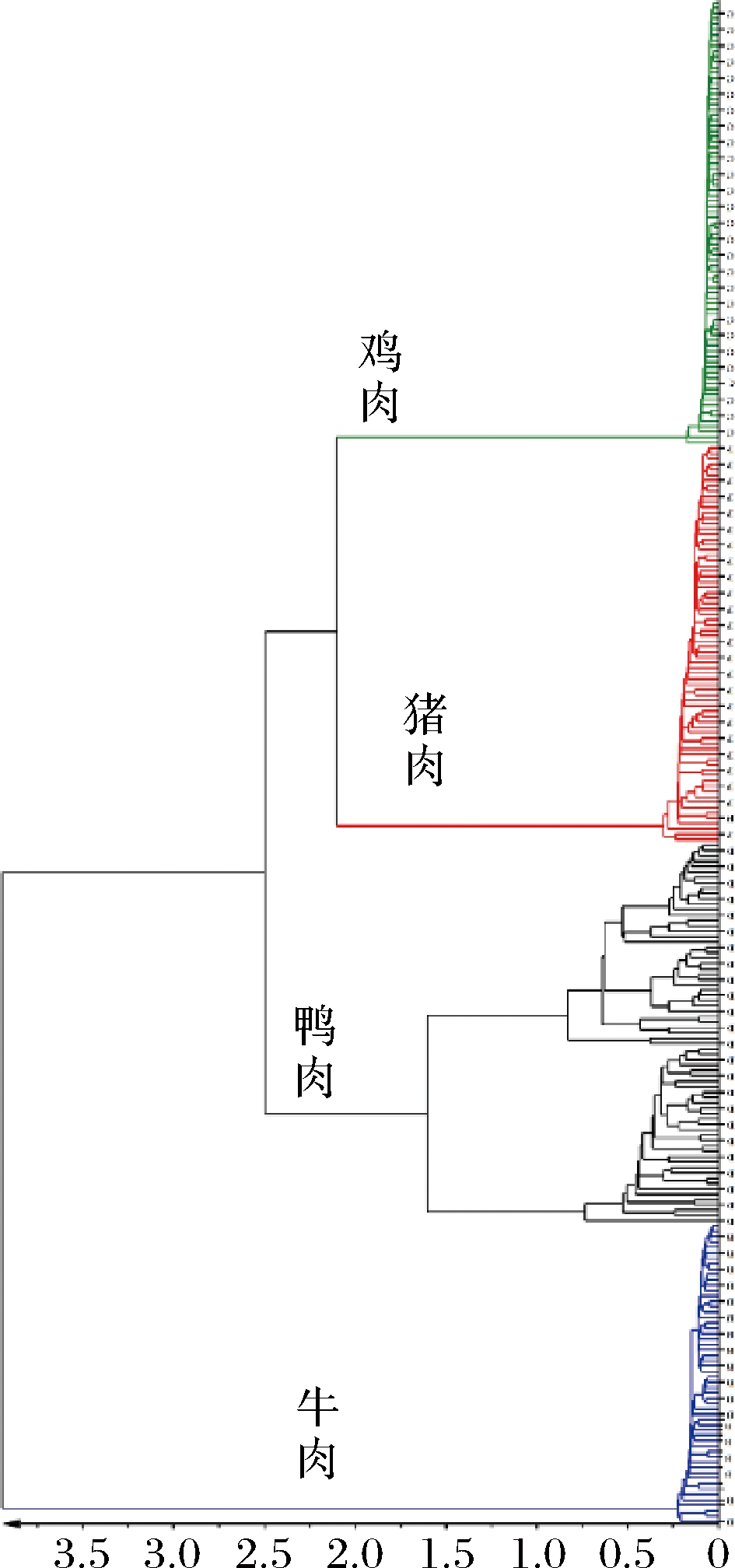
图4 十一个特征蛋白质作为变量下的生熟4种肉的HCA
Fig.4 HCA using the 11 characteristic proteins of the four species meats (including fresh and cooked)
2.3.2 牛肉掺假样品验证
包括生熟的鸡肉、鸭肉和猪肉分别以25%、50%、75%质量比与牛肉混合制备的掺假样品被用于鉴别模型的验证。其结果如图5所示,掺假样品均能与纯肉分离,且随着3种掺假肉加入牛肉的比例增大,掺假牛肉样品逐渐从纯牛肉向对应掺假纯肉的方向移动。该结果表明模型能较好地完成对生熟牛肉掺假样品的定性检验。
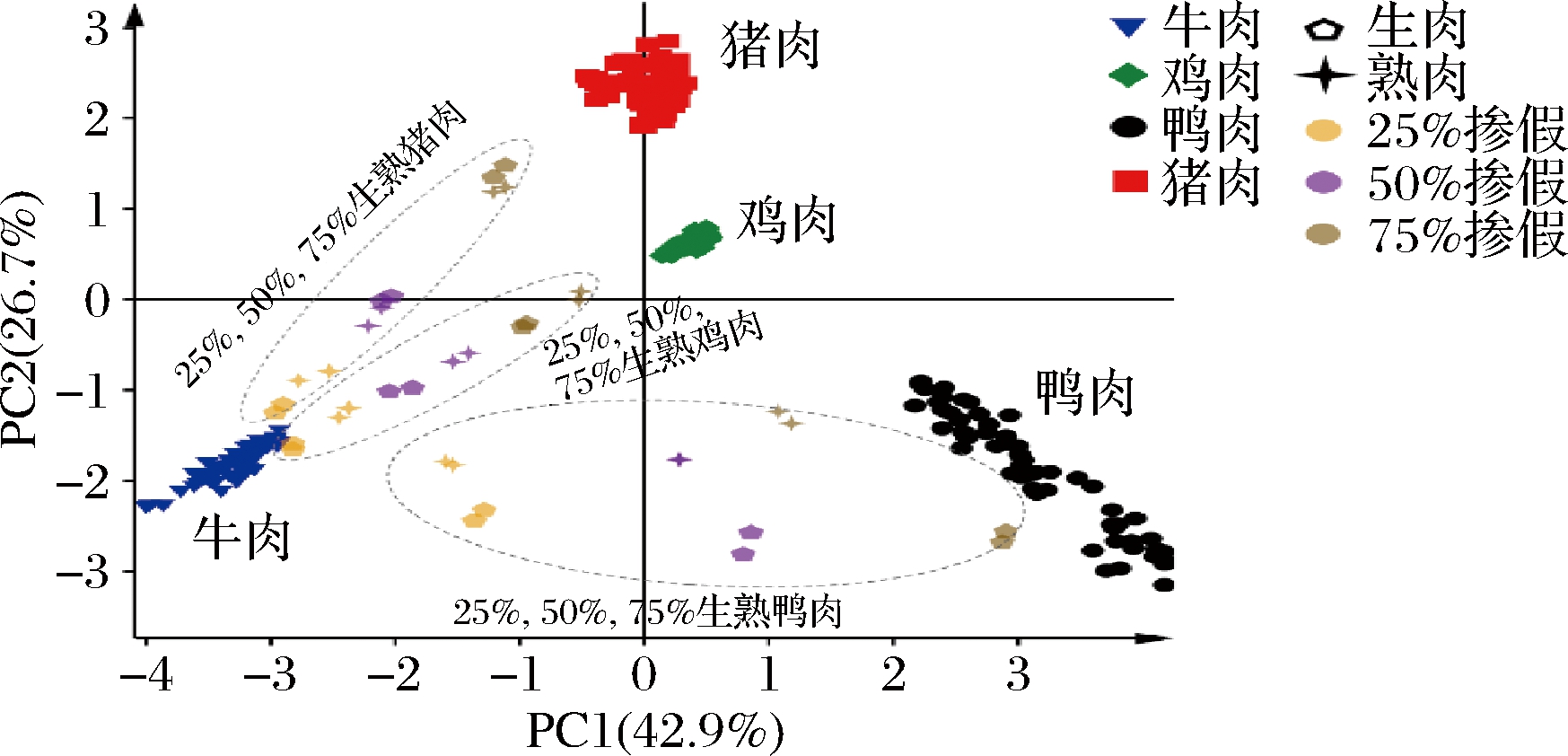
图5 生熟纯肉和鸡肉、鸭肉和猪肉与牛肉混合制备的掺假样品PCA得分图
Fig.5 PCA scoring plot of pure beef, chicken, duck and pork meats, and adulterated beef samples
3 结论
通过对牛、鸡、鸭和猪的不同食用部位的肉样,分别在生熟状态下进行简单、快速的蛋白质提取,并通过 MALDI-TOF MS进行表征,得到了4种肉的蛋白质质谱图。经预处理后得到的数据矩阵进行随机森林算法分析,共找到11个能代表生熟4种肉的特征蛋白质,其中7个在Uniprot中得到表征,将这些特征蛋白质作为PCA的变量以构建牛肉掺假鉴别模型。最后通过HCA和牛肉掺假样品的鉴别验证了该模型,说明通过本方法,可以完成掺假牛肉的初步筛查。依托于MALDI-TOF MS的高通量、简便等优势,可在3 h内完成对384个样品的检测,证明该方法可应用于市场及海关等检测现场,维护肉制品市场的安全与秩序。
[1] 袁淑辉,蔡军,王忠才,等.4种肉类成分多重PCR的鉴定方法[J].中国兽医杂志, 2020, 56(11):65-69.
YUAN S H, CAI J, WANG Z C, et al.A multiplex PCR method for the identification of four meat ingredients[J]. Chinese Journal of Veterinary Medicine, 2020, 56(11):65-69.
[2] VELIOGLU H M, SEZER B, BILGE G, et al.Identification of offal adulteration in beef by laser induced breakdown spectroscopy (LIBS)[J].Meat Science, 2018, 138:28-33.
[3] BALLIN N Z.Authentication of meat and meat products[J].Meat Science, 2010, 86(3):577-587.
[4] 马永征,马冬,白娣斯,等.免疫学检测肉类制品掺假研究进展[J].肉类研究, 2012,26(9):26-29.
MA Y Z, MA D, BAI D S,et al.Recent advances in immunological detection of meat adulteration[J].Meat Research, 2012, 26(9):26-29.
[5] 郎玉苗,杨春柳,李翠,等.光谱技术在肉品掺杂掺假鉴别中的应用研究进展[J].肉类研究, 2019, 33(2):72-77.
LANG Y M, YANG C L, LI C, et al.Application of spectroscopic techniques in identification of meat adulteration:A review[J].Meat Research, 2019, 33(2):72-77.
[6] 胡馨予, 黄朱梁, 汤海凤, 等.基于PCR技术的肉类成分溯源鉴定方法研究进展[J].食品安全质量检测学报, 2020, 11(11):3 385-3 390.
HU X Y, HUANG Z L, TANG H F, et al.Research progress in species identification methods of meat ingredients based on PCR technology[J].Journal of Food Safety & Quality, 2020, 11(11):3 385-3 390.
[7] PALMER H M.Using antibodies:A laboratory manual[J].Journal of Antimicrobial Chemotherapy, 2000, 45(3):413.
[8] 任君安, 黄文胜, 葛毅强, 等.肉制品真伪鉴别技术研究进展[J].食品科学, 2016, 37(1):247-257.
REN J A, HUANG W S, GE Y Q, et al.Progress in meat adulteration detection techniques[J].Food Science, 2016, 37(1):247-257.
[9] REN J A, DENG T T, HUANG W S, et al.A digital PCR method for identifying and quantifying adulteration of meat species in raw and processed food[J].PLoS One, 2017, 12(3):e0173567.
[10] DE RAAD M, FISCHER C R, NORTHEN T R.High-throughput platforms for metabolomics[J].Current Opinion in Chemical Biology, 2016, 30:7-13.
[11] LAGACÉ-WIENS P R S, ADAM H J, KARLOWSKY J A, et al.Identification of blood culture isolates directly from positive blood cultures by use of matrix-assisted laser desorption ionization-time of flight mass spectrometry and a commercial extraction system:Analysis of performance, cost, and turnaround time[J].Journal of Clinical Microbiology, 2012, 50(10):3 324-3 328.
[12] LAAKMANN S, GERDTS G, ERLER R, et al.Comparison of molecular species identification for North Sea calanoid copepods (Crustacea) using proteome fingerprints and DNA sequences[J].Molecular Ecology Resources, 2013, 13(5):862-876.
[13] 孙克娜, 许小雨, 朱小双, 等.WCX-MB联合MALDI-TOF MS技术建立宫颈癌诊断预测模型及初步验证[J].生物技术通讯, 2020, 31(5):547-553.
SUN K N, XU X Y, ZHU X S, et al.Establishment and preliminary validation of a diagnostic prediction model for cervical cancer using WCX-MB and MALDI-TOF MS[J].Letters in Biotechnology, 2020, 31(5):547-553.
[14] MESAROS C, BLAIR I A.Mass spectrometry-based approaches to targeted quantitative proteomics in cardiovascular disease[J].Clinical Proteomics, 2016, 13:20.
[15] 顾春华. MALDI-TOF MS技术及其在食品微生物检测方面的应用[J].中国酿造, 2019, 38(9):24-27.
GU C H.MALDI-TOF MS technique and its application challenges in food microbiology detection[J].China Brewing, 2019, 38(9):24-27.
[16] FLAUDROPS C, ARMSTRONG N, RAOULT D, et al.Determination of the animal origin of meat and gelatin by MALDI-TOF-MS[J].Journal of Food Composition and Analysis, 2015, 41:104-112.
[17] STAHL A, SCHRÖDER U.Development of a MALDI-TOF MS-based protein fingerprint database of common food fish allowing fast and reliable identification of fraud and substitution[J].Journal of Agricultural and Food Chemistry, 2017, 65(34):7 519-7 527.
[18] GIBB S, STRIMMER K.MALDIquant:a versatile R package for the analysis of mass spectrometry data[J].Bioinformatics, 2012, 28(17):2 270-2 271.
[19] VON BARGEN C, BROCKMEYER J, HUMPF H U.Meat authentication:A new HPLC-MS/MS based method for the fast and sensitive detection of horse and pork in highly processed food[J].Journal of Agricultural and Food Chemistry, 2014, 62(39):9 428-9 435.
[20] WU Y M, LI L.Sample normalization methods in quantitative metabolomics[J].Journal of Chromatography A, 2016, 1430:80-95.
[21] BIAU G.Analysis of a random forests model[J].The Journal of Machine Learning Research, 2012, 13(1):1 063-1 095.
[22] BREIMAN L.Random forests[J].Machine Learning, 2001, 45:5-32.