我国古代就有将发霉的谷物作引子来发酵熟谷物使其转变为酒的记载,形成早期的传统发酵食品。与工业生产所用的直投式发酵剂不同,传统发酵食品所用的发酵剂菌种更丰富且来源更广泛,这使得传统发酵食品具有更加独特的风味和丰富的营养成分,因此很多企业希望将其进行规模化生产,但目前效果不够理想,主要原因是传统发酵食品中复杂且数量庞大的微生物群体使得发酵过程难以控制,导致产品质量不稳定并存在一定安全风险。为了扩大传统发酵食品的市场,应对其微生物种群、发酵过程以及微生物间的相互作用进行更深入的研究。多数传统发酵食品中都含有乳酸菌和酵母菌,两者在发酵食品中占据极为重要的地位,对两者的研究能够解决目前传统发酵食品发展所遇到的一些问题。
乳酸菌是一类可发酵碳水化合物产乳酸的无芽胞革兰氏阳性菌,也是食品中公认的益生微生物,具有促进消化吸收、维持肠道菌群平衡、免疫调节等作用[1-3],而酵母菌是一类典型的单细胞微生物,能将糖发酵成酒精和CO2,在有氧和无氧环境下都能生存,属于兼性厌氧菌[4]。两者共同参与发酵制得的产品主要有酸马奶酒、酸面团、乳酪等,其相互作用机制较为复杂,目前研究发现两者间存在的互作机制可分为以下3类:(1)代谢产物互补;(2)群体感应现象;(3)影响细胞生长,其中代谢产物间的互补是发现较早也是最为常见的。
1 传统发酵食品及其菌种
传统发酵食品在生产过程中所涉及到的微生物种类繁多,大多包含了十几种到几十种微生物,这些微生物的共同作用赋予了传统发酵食品独特的风味、色泽和质地。如表1所示,多数传统发酵食品中都含有乳酸菌和酵母菌,且往往不以单一菌种的形式存在。已有报道证实多菌种的复杂发酵体系是传统发酵食品风味的重要来源[5]。其中对产品风味影响较突出的是乳酸菌和酵母菌2类。因此,在对多种复杂微生物菌群间互作关系研究较为困难的情况下,探究乳酸菌和酵母菌二者间存在的互作关系,能够在一定程度上解析发酵微生物与食品品质的相关性,为传统发酵食品的工业化发展提供理论依据。

2 乳酸菌与酵母菌互作机制研究方法
乳酸菌与酵母菌间存在的互作关系多为协同作用,包括互相利用代谢产物、互相修复细胞结构、调节生长状态、提升抗热能力等,但也有研究表明二者在一些条件下还存在拮抗作用,DA SILVA等[14]发现希氏乳杆菌(Lactobacillus hilgardii)和布氏乳杆菌(Lactobacillus buchneri)会显著抑制酵母菌生长, LVAREZ-MART
LVAREZ-MART N等[15]也发现副干酪乳杆菌(Lactobacillus paracasei)和乳酸乳球菌(Lactococcus lactis)在与酵母菌共培养中生长被抑制。这说明乳酸菌与酵母菌间的互作关系并不是单纯的协同或拮抗,而有着更加复杂的互作机制,随着研究的深入,对二者互作机制的研究方法也在逐步改进,可归纳为传统方法和新兴的代谢组学、转录组学等。
N等[15]也发现副干酪乳杆菌(Lactobacillus paracasei)和乳酸乳球菌(Lactococcus lactis)在与酵母菌共培养中生长被抑制。这说明乳酸菌与酵母菌间的互作关系并不是单纯的协同或拮抗,而有着更加复杂的互作机制,随着研究的深入,对二者互作机制的研究方法也在逐步改进,可归纳为传统方法和新兴的代谢组学、转录组学等。
2.1 传统方法在互作机制中的应用
传统方法通常是将二者混合发酵的部分代谢产物、生长状态等指标作为分析目标,间接地推测乳酸菌和酵母菌间存在的互作机制。王美霞[16]将筛选得到存在代谢互补的乳酸菌和酵母菌共培养后,分离出能够促进乳酸菌生长的酵母菌代谢产物,以此来分析2株菌间的互作原理,经鉴定认为该促进物质是蛋白或肽类物质的混合物。贺银凤等[17]则将乳酸菌和酵母菌的无菌代谢产物交叉培养,发现乳酸菌的无菌发酵液能够对酵母菌的生长起到促进作用,而酵母菌的无菌发酵液能够显著增强乳酸菌的产酸能力。通过分析乳酸菌和酵母菌共同培养时的代谢产物和生长状态能够分离并鉴定出影响两者生长代谢的物质,以此推测二者间的互作机制,这类方法在早期研究中应用广泛且效果良好,但微生物发酵过程中产生的代谢产物数量庞大,通过逐个分离鉴定的方式工作量较大且不系统,解析微生物的相互作用机制需要大量数据作为支撑,代谢组学技术可以达到这一目的。
2.2 代谢组学在互作机制中的应用
代谢组学技术是对微生物发酵过程中全部的小分子代谢产物进行鉴定,并利用生物信息数据库分析出发酵过程中所涉及到的代谢通路,进而解析微生物间的互作机制,为精准调节传统食品的发酵作用提供依据。代谢组学是研究某一细胞、组织、器官或生物体中所有小分子代谢组分集合的科学,是系统生物学的重要分支,可分为靶向代谢组学和非靶向代谢组学,前者是对某一类代谢产物进行分析而后者范围更宽泛,目标是从生物样本中同时分析尽可能多的代谢物[18],能够对发酵过程中微生物代谢产物变化进行更全面的研究。
孙哲航[19]利用代谢组学比较了干酪乳杆菌(Lactobacillus casei,M2-L2)和单孢酿酒酵母(Kazachstania unispora,M4-Y2)单菌发酵和混菌发酵过程中代谢产物变化情况,发现相较于单菌发酵,混菌发酵产生了更多的长链脂肪酸,这解释了混菌发酵产酸速率更快的现象。而混菌发酵体系中更多的甲硫氨酸、苯丙氨酸、酪氨酸和精氨酸,证明氨基酸可能是促进乳酸菌增殖的重要因子。BLASCHE等[20]通过代谢组学发现产马乳酒乳杆菌(Lactobacillus kefiranofaciens)和肠膜明串珠菌(Leuconostoc mesenteroides)共培养时,肠膜明串珠菌乳酸产量增加并消耗了更多的氨基酸,同样证实了两者间存在氨基酸代谢互补机制。KADYAN等[21]利用非靶向代谢组学确定植物乳杆菌(Lactobacillus plantarum)和乳酸克鲁维酵母(Kluyveromyces lactis)单菌发酵和混菌发酵时的关键代谢产物,其中混菌体系中糖类含量更低,推测是混菌发酵提升了糖类的利用率。
代谢产物是微生物代谢过程的最后环节,代谢物质的变化是由于微生物自身调控引起还是其他因素对物质本身的影响需要结合基因层面共同分析。
2.3 转录组学在互作机制中的应用
转录组学是针对功能基因的一种研究方法,通过筛选混菌发酵过程中表达差异显著的相关基因,利用功能注释和通路分析等手段解析微生物之间的互作机制。转录组学研究目的不仅是针对转录组样本中每个基因的表达水平的变化,也包括转录组的定位和注释及每个基因的功能和结构测定[22]。转录组学通过对微生物总mRNA进行提取分析,可以了解到微生物在发酵过程中代谢、生长、结构等方面的变化,以此反应发酵中微生物间的互作机制。
如表2所示,转录组学技术在解析各类发酵食品的微生物互作现象中已有应用,通过筛选出发酵前后表达差异性显著的基因,再利用生物信息数据库得到相关通路,来挖掘二者的互作机制。其中杨浣漪[23]利用转录组学技术对比酿酒酵母(Saccharomyces cerevisiae)单菌发酵和与旧金山乳杆菌(Lactobacillus Sanfranciscensis)混菌发酵面团的差异基因,发现酿酒酵母共有625个基因上调,571个基因下调,其中编码甘油-3-磷酸酶的基因HOR2明显上调,说明混菌发酵能够增加甘油合成。而编码磷酸烯醇式丙酮酸羧激酶的基因PCK1表达明显下调,解释了混菌发酵可增加有机酸合成。与转录组学相比,宏转录组学以样品中全部微生物RNA为研究对象,能够从整体上分析样本中优势微生物和基因表达情况,张国华等[24]采用宏转录组学技术分析酸面团不同发酵时期菌种的差异表达基因,发现细胞过程、发展过程、代谢过程转运活性方面的基因丰度超过10%,说明乳酸菌和酵母菌之间存在代谢产物互补机制。研究发现混合发酵所影响的相关通路主要包括氨基酸代谢、丙酮酸代谢等,其中氨基酸代谢在较多实验中都有发现,说明乳酸菌和酵母菌的互作与氨基酸的合成和利用具有一定相关性。但转录组学只能体现微生物将要进行的生理活动,而这些生理活动是否发生还需要结合相关代谢产物进行验证。
表2 转录组学技术在解析微生物互作机制中的应用
Table 2 Application of transcriptomics in the analysis of microbial interaction mechanism
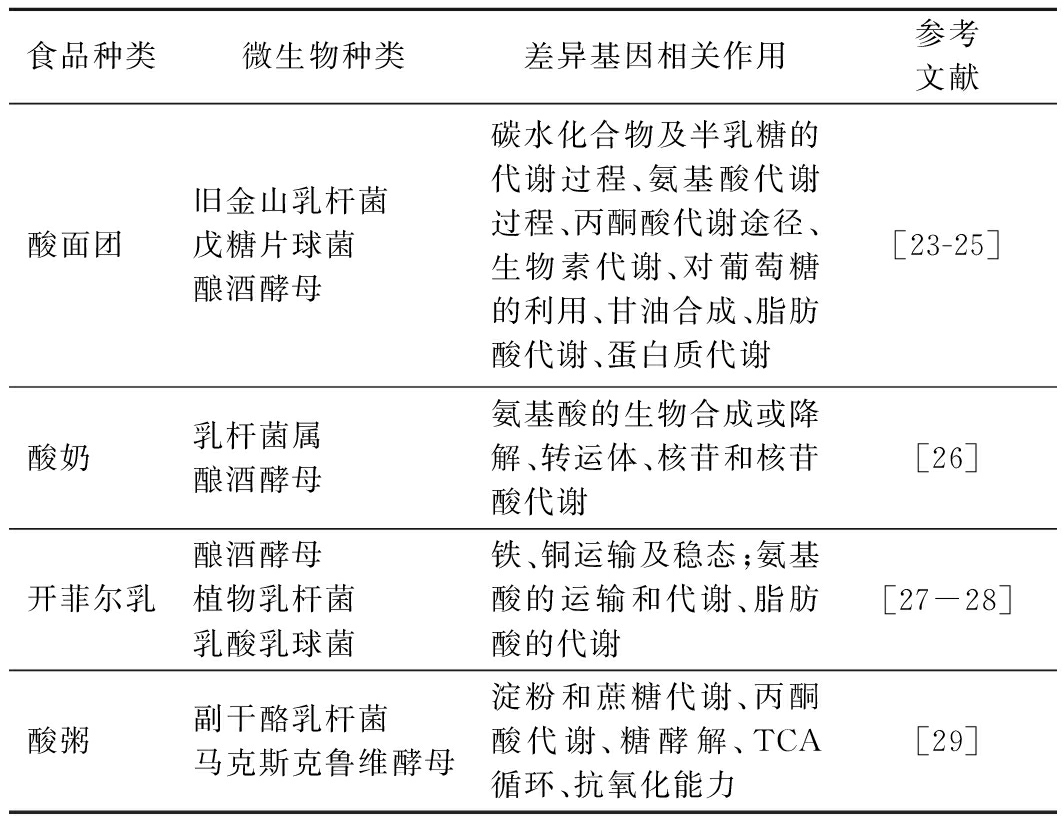
食品种类微生物种类差异基因相关作用参考文献酸面团 旧金山乳杆菌戊糖片球菌酿酒酵母碳水化合物及半乳糖的代谢过程、氨基酸代谢过程、丙酮酸代谢途径、生物素代谢、对葡萄糖的利用、甘油合成、脂肪酸代谢、蛋白质代谢[23-25]酸奶 乳杆菌属酿酒酵母氨基酸的生物合成或降解、转运体、核苷和核苷酸代谢[26]开菲尔乳酿酒酵母植物乳杆菌乳酸乳球菌铁、铜运输及稳态;氨基酸的运输和代谢、脂肪酸的代谢[27-28]酸粥 副干酪乳杆菌马克斯克鲁维酵母淀粉和蔗糖代谢、丙酮酸代谢、糖酵解、TCA循环、抗氧化能力[29]
3 乳酸菌与酵母菌的互作机制
乳酸菌和酵母菌间复杂的相互作用关系使得二者相较于单独生长时,在生长状态、代谢产物、菌群结构等方面存在诸多差异。范维等[30]发现在乳酸菌发酵牛乳的过程中添加马克斯克鲁维酵母(Kluyveromyces marxiamus)能够促进乳酸菌的生长,并使保加利亚乳杆菌(Lactobacillus bulgaricus)提前进入对数期,且酵母菌的存在还可以延迟嗜热链球菌(Streptococcus thermophilus)的衰亡。较多乳酸菌和酵母菌间的相互作用效果都向着对生长代谢有利的方向进行,而如何进一步加以利用则需要更加深入地研究二者间的相互作用机制。
3.1 代谢产物互补
代谢互补机制是在乳酸菌和酵母菌间广泛存在的一类相互作用机制,较常见的是乳酸菌在代谢过程中产生乳糖酶分解乳糖为半乳糖,为非乳糖代谢型酵母提供碳源,而酵母菌代谢产生的氨基酸等物质又能被乳酸菌作为营养物质利用[31]。因此,在以乳糖为碳源时,乳酸菌和酵母菌间更容易发生互惠作用,常见的代谢互补等现象多发生于乳制品中。如表3所示,各类发酵食品中存在已发现的代谢互补机制,这类机制存在的主要原因是不同微生物所具备的酶系不同,由于乳酸菌和酵母菌各自缺乏分解某种碳源所需要的特异性酶,因此需要利用对方所代谢的酶解产物进一步反应。如图1所示,当以乳糖为唯一碳源时,由于多数酵母菌不具有乳糖酶,无法直接利用乳糖,因此需要乳酸菌所具备的α-半乳糖苷酶将乳糖分解为葡萄糖和半乳糖,葡萄糖可作为碳源直接进入EMP途径被大多数乳酸菌和酵母菌利用,但乳酸菌缺乏足够的半乳糖激酶,无法进一步利用半乳糖。马克斯克鲁维酵母的参与能够将半乳糖转化为6-磷酸葡萄糖进入EMP途径,最终形成丙酮酸,在乳酸脱氢酶的作用下产生乳酸,这一机制也解释了乳酸菌和酵母菌共同发酵能够产生更多乳酸这一现象。
表3 部分发酵食品中代谢互补机制
Table 3 Metabolic complementation mechanism in partially fermented foods

食品种类代谢互补机制参考文献发酵乳 乳酸菌利用乳糖产生半乳糖,酵母菌利用半乳糖产生丙酮酸,乳酸菌和酵母菌共同利用丙酮酸形成乳酸[32]发酵酒 酵母菌利用乳酸菌乳糖酶分解产生的葡萄糖和半乳糖[33]发酵面团酵母菌产生氨基酸、维生素等为乳酸菌提供营养因子,乳酸菌生成的葡萄糖被酵母菌作为营养来源利用[24]发酵蔬菜酵母菌产生氨基酸被乳酸菌所利用,乳酸菌代谢乳酸形成乳酸盐为酵母菌提供碳源,醋酸菌利用酵母菌产生乙醇形成乙酸[34]
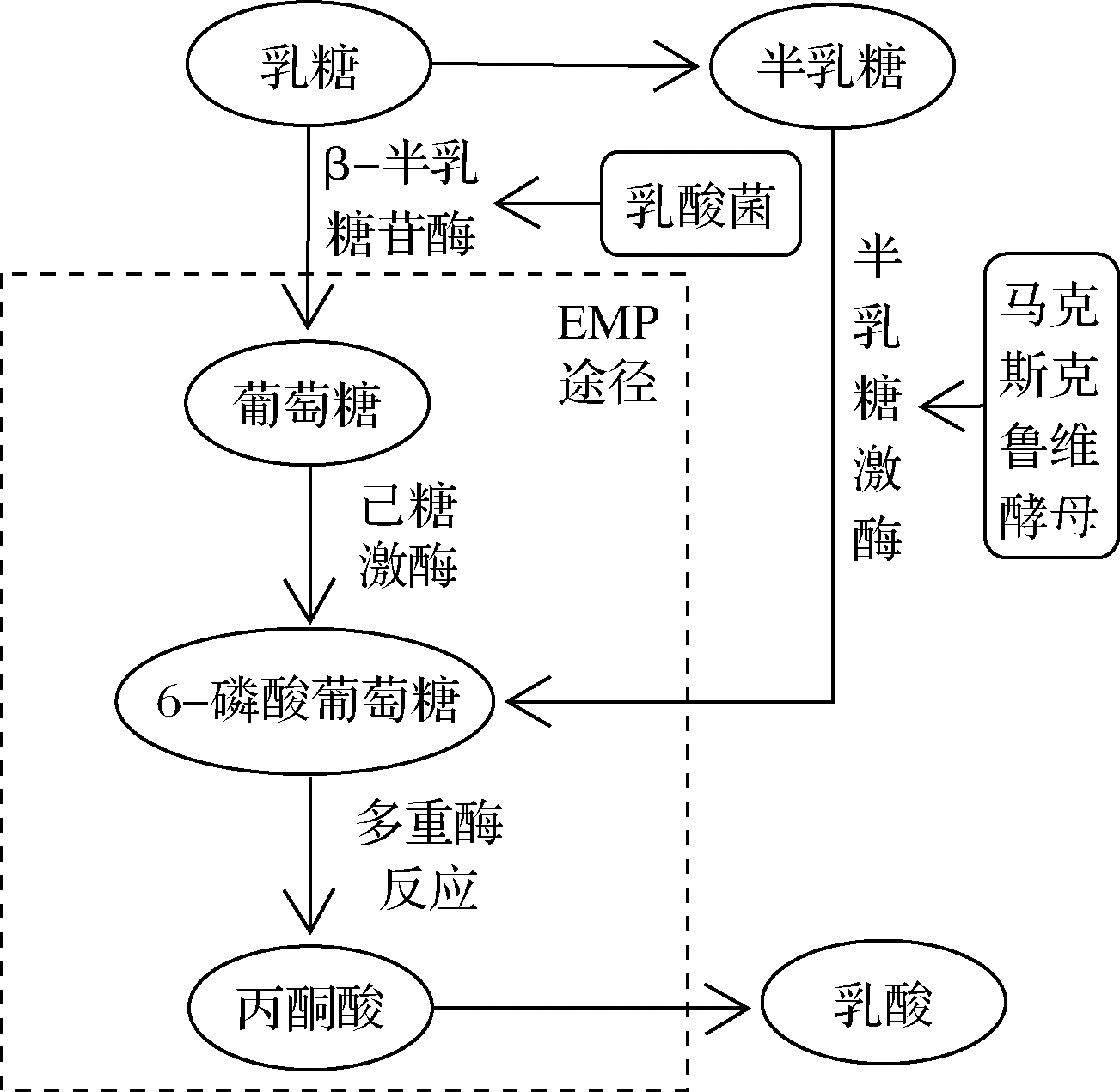
图1 乳酸菌和酵母菌共同利用乳糖
Fig.1 Lactic acid bacteria and yeasts use lactose together
乳酸菌的生长对营养物质需求较苛刻,除糖类外还需要氨基酸、维生素等,酵母菌生长所需物质条件则较为简单。因此在发酵过程中,酵母菌还扮演着为乳酸菌提供生长所需营养物质的角色,如图2所示,在富含氮源的生长环境中,酵母菌的TORC1信号通路受到刺激,调节产生氨基酸,为乳酸菌提供必备的生长因子,而乳酸菌分解乳糖所产生的葡萄糖同时可被酵母菌利用。这一代谢互补机制使得乳酸菌在酵母菌的参与下即使处于营养物质较匮乏的发酵环境中仍然能够生长。

图2 乳酸菌和酵母菌对氮的代谢互补[27]
Fig.2 Lactic acid bacteria and yeast complement the metabolism of nitrogen
3.2 影响细胞生长
乳酸菌和酵母菌在共同培养下,其生长情况相较于单独培养有着显著差异,王小标等[35]发现马克斯克鲁维酵母可以显著促进干酪乳杆菌的生长,而乳酸乳球菌可以显著促进马克斯克鲁维酵母的生长。洪家丽等[36]则发现在红曲黄酒酿造过程中,不同种类的乳酸菌对酵母菌细胞的生长情况影响不同,其中干酪乳杆菌会显著抑制酿酒酵母的生长。这样的现象主要由乳酸菌和酵母菌通过直接或间接途径影响彼此细胞生长所造成,其作用途径包括竞争作用和分泌影响细胞生长的代谢产物等。
竞争是微生物间常见的一种互作关系,MENDES等[28]发现乳酸菌和酵母菌间存在对脂肪酸的竞争关系,以此影响细胞生长。而在青贮饲料中,乳酸菌与酵母菌的营养竞争现象同样存在,在发酵前期,乳酸菌作为优势菌种与酵母菌进行营养竞争使得酵母菌生长缓慢[37]。
代谢产物对细胞生长的影响在发酵过程中更为普遍,乳酸菌代谢产生大量乳酸,过度积累将导致环境pH降低从而抑制酵母细胞生长,而酵母菌所分泌的乙醇也会影响乳酸菌的细胞结构,乙醇与乳酸还能进行酯化反应,在一定程度上降低对二者自身细胞的损伤。此外,还有研究发现酵母菌代谢产生的CO2、维生素B6等物质均能够刺激乳酸菌生长,而乳酸菌产生的乳酸对阳离子具有螯合作用,能够减轻阳离子对酵母菌细胞的影响[38-40]。张文娟等[41]则发现,酵母菌产生乙醇可以与乳酸菌代谢的乳酸进行酯化反应,以防止乳酸堆积造成乳酸菌自身生长减缓。
3.3 群体感应现象
早期群体感应定义为细菌随着菌体密度的增大和生长周期的变化分泌出一种或几种化学信号分子,从而导致其生理和生化特性的变化,显示出少量菌体或单个菌体所不具备的特征[42],但进一步研究发现真菌和昆虫群体中也存在这类感应现象,其中食源性微生物的群体感应调控性状包括生物膜形成、耐酸胁迫、细菌素代谢和定向生长等[43]。乳酸菌作为革兰氏阳性菌具有的群体感应调节系统如图3所示,包括种间群体感应系统和G+群体感应系统,其信号分子主要为呋喃硼酸二酯(AI-2)以及寡肽类(AIPs)2种,种间群体感应系统由LuxS基因和Pfs基因调控,G+群体感应系统主要包括ABC(ATP-binding cassette transporter)转运系统和双组分系统(two component signaling system,TCS)2个部分,ABC转运系统负责将寡肽类信号分子转运到细胞外,双组分系统则负责检测细胞外的信号分子浓度,并加以调控。酵母菌中所检测出的信号分子包括色氨酸以及一些醇类、酸类物质,如苯乙醇等,已有报道指出,乙酸可被作为信号分子调控酿酒酵母W141产生2,3-丁二醇[44],但部分酵母菌无法产生群体感应信号分子,而是利用其代谢产物影响乳酸菌分泌信号分子,以达到影响乳酸菌生长代谢的效果。
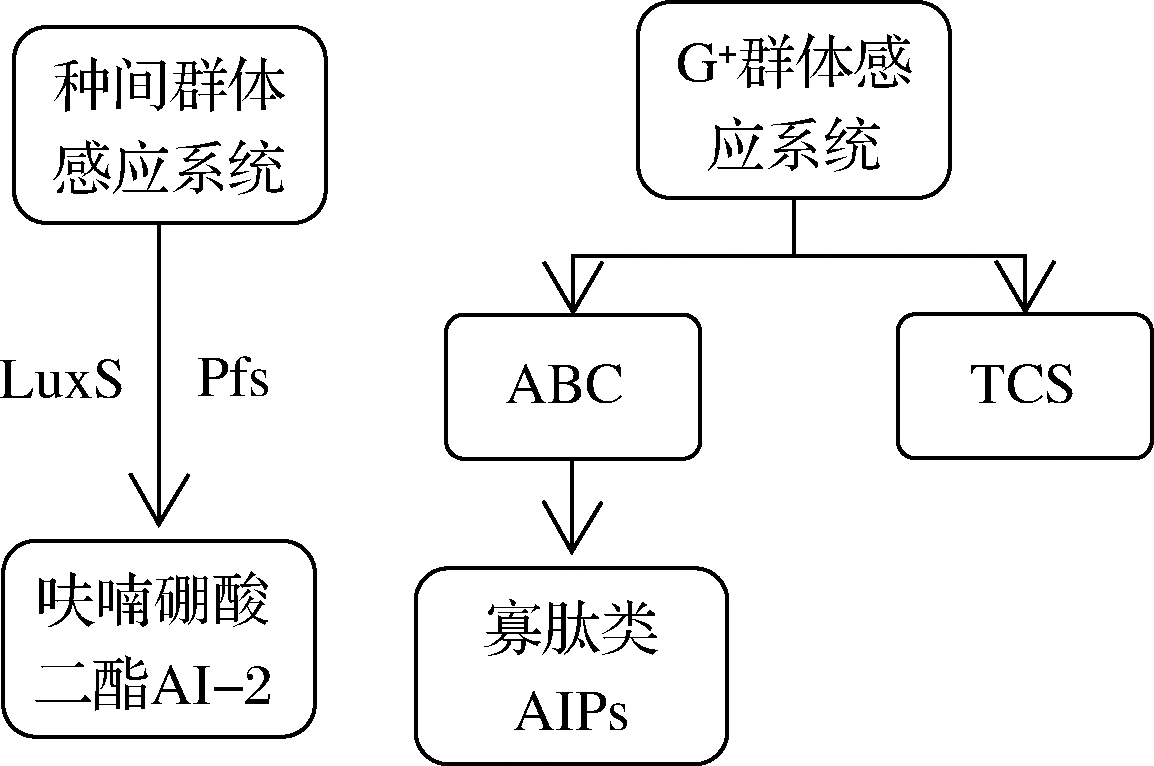
图3 种间群体感应系统及革兰氏阳性群体感应系统
Fig.3 Interspecies quorum sensing system and Gram-positive quorum sensing system
廉雪花[45]将4株酵母菌与2株乳酸菌分别进行共培养,发现其中一株酵母菌能够促进乳酸菌分泌AI-2信号分子,而其余3株酵母菌均抑制AI-2信号分子的产生,但4株酵母菌的代谢产物均能促进乳酸菌分泌AI-2信号分子。PROUST等[46]将酵母菌的代谢产物添加到嗜热链球菌的培养环境中,检测到shCDS显著表达,推测是酵母菌代谢产物中的部分多肽物质刺激了乳酸菌的群体感应调节机制,顾悦[47]也发现酿酒酵母的无菌代谢产物能够刺激屎肠球菌8-3产生AI-2信号分子,进一步研究发现其中部分代谢产物具有抑制LuxS基因转录,促进Pfs基因转录的作用。
4 总结与展望
乳酸菌和酵母菌作为传统发酵食品中常见的微生物,二者的互作机制一直是研究热点。近年来组学技术的快速发展为解析微生物互作机制提供了新的思路,从基因组学、转录组学到蛋白组学和代谢组学,针对微生物生长代谢的每个阶段都有较为全面的研究手段,但各种组学技术都存在自身的局限性,无法对微生物从基因层面到分泌代谢物的整个过程进行分析,因此便需要多种组学技术共同使用,在技术上互补可使对互作机制的解析更加系统。随着组学技术的成熟,将多组学技术结合传统方法对关键代谢产物和微生物生长状态的分析将会成为解析微生物间的相互作用机制的有效手段。
解析乳酸菌和酵母菌的互作机制目的在于掌握并调节二者的混菌发酵过程,工业生产上一般通过优化发酵底物和控制发酵条件来调节发酵过程,而解析出二者互作机制作为理论依据后便可进行更加精准的调控,如针对传统发酵食品风味难以控制的问题,可以通过调节关键风味物质的前体物质以达到精准调节发酵产品风味的目的。这样的调控方式更易操作,目的性也更强,可以作为解决传统发酵食品工业化问题的一种思路。
[1] GAO J, LI Y B, WAN Y, et al.A novel postbiotic from Lactobacillus rhamnosus GG with a beneficial effect on intestinal barrier function[J].Frontiers in Microbiology, 2019, 10:477.
[2] LI S J, CHEN T T, XU F, et al.The beneficial effect of exopolysaccharides from Bifidobacterium bifidum WBIN03 on microbial diversity in mouse intestine[J].Journal of the Science of Food and Agriculture, 2014, 94(2):256-264.
[3] LIU H, ZHANG J, ZHANG S H, et al.Oral administration of Lactobacillus fermentum I5007 favors intestinal development and alters the intestinal microbiota in formula-fed piglets[J].Journal of Agricultural and Food Chemistry, 2014, 62(4):860-866.
[4] MACORI G, COTTER P D.Novel insights into the microbiology of fermented dairy foods[J].Current Opinion in Biotechnology, 2018, 49:172-178.
[5] THOMA I, LOEFFLER C, SINHA A K, et al.Cyclopentenone isoprostanes induced by reactive oxygen species trigger defense gene activation and phytoalexin accumulation in plants[J].The Plant Journal:For Cell and Molecular Biology, 2003, 34(3):363-375.
[6] BAYILI G R, JOHANSEN P, NIELSEN D S, et al.Identification of the predominant microbiota during production of lait caillé, a spontaneously fermented milk product made in Burkina Faso[J].World Journal of Microbiology & Biotechnology, 2019, 35(7):100.
[7] AKABANDA F, OWUSU-KWARTENG J, GLOVER R L K, et al.Microbiological characteristics of Ghanaian traditional fermented milk product,Nunu[J].Nature And Science, 2010, 8(9):178-187.
[8] 杨俊俊. 西藏牦牛奶渣中微生物的分离鉴定及优良乳酸菌的筛选[D].杨凌:西北农林科技大学, 2014.
YANG J J.Isolation and identification of microflora in yak milk dreg of Tibet in China and screening of fine lactic acid bacteria[D].Yangling:Northwest A & F University, 2014.
[9] CAI H Y, ZHANG T, ZHANG Q, et al.Microbial diversity and chemical analysis of the starters used in traditional Chinese sweet rice wine[J].Food Microbiology, 2018, 73:319-326.
[10] ASTUDILLO-MELGAR F, OCHOA-LEYVA A, UTRILLA J, et al.Bacterial diversity and population dynamics during the fermentation of palm wine from Guerrero Mexico[J].Frontiers in Microbiology, 2019, 10:531.
[11] 努尔古丽·热合曼, 华长春, 朱晓莹, 等.新疆柯尔克孜族传统发酵饮料博扎中微生物群落结构的PCR-DGGE分析[J].食品科学, 2012, 33(1):111-114.
RAHMAN N, HUA C C, ZHU X Y, et al.PCR-DGGE analysis of microbial community structure of Bozaa, a traditional Kyrgyz fermented beverage[J].Food Science, 2012, 33(1):111-114.
[12] LU Z H, PENG H H, CAO W, et al.Isolation, characterization and identification of lactic acid bacteria and yeasts from sour Mifen, a traditional fermented rice noodle from China[J].Journal of Applied Microbiology, 2008, 105(3):893-903.
[13] 乌云, 高增丽, 曹文慧, 等.传统列巴面团中潜在共生菌的筛选[J].中国食品添加剂, 2020, 31(10):85-94.
WU Y, GAO Z L, CAO W H, et al.Screening of potential symbiotic bacteria in traditional leba dough[J].China Food Additives, 2020, 31(10):85-94.
[14] DA SILVA E B, SMITH M L, SAVAGE R M, et al.Effects of Lactobacillus hilgardii 4785 and Lactobacillus buchneri 40788 on the bacterial community, fermentation and aerobic stability of high-moisture corn silage[J].Journal of Applied Microbiology, 2021, 130(5):1 481-1 493.
[15]  LVAREZ-MART
LVAREZ-MART N P, FL
N P, FL REZ A B, HERN
REZ A B, HERN NDEZ-BARRANCO A, et al.Interaction between dairy yeasts and lactic acid bacteria strains during milk fermentation[J].Food Control, 2008, 19(1):62-70.
NDEZ-BARRANCO A, et al.Interaction between dairy yeasts and lactic acid bacteria strains during milk fermentation[J].Food Control, 2008, 19(1):62-70.
[16] 王美霞. 具有共生作用乳酸菌与酵母菌培养基优化及促生物质的分离[D].呼和浩特:内蒙古农业大学, 2013.
WANG M X.Optimization medium of symbiotic lactic acid bacteria and yeasts and the separation of the promote biomass[D].Hohhot:Inner Mongolia Agricultural University, 2013.
[17] 贺银凤, 刘敏敏, 李燕军, 等.酸马奶酒中乳酸菌和酵母菌互作关系的研究[J].食品科技, 2011, 36(5):32-36.
HE Y F, LIU M M, LI Y J, et al.Interactions between lactic acid bacteria and yeasts from koumiss[J].Food Science and Technology, 2011, 36(5):32-36.
[18] RALLI E, AMARGIANITAKI M, MANOLOPOULOU E, et al.NMR spectroscopy protocols for food metabolomics applications[J].Methods in Molecular Biology (Clifton, N.J.), 2018, 1 738:203-211.
[19] 孙哲航. 酸马奶源优势菌群与特征风味的探讨及代谢组学研究[D].杭州:浙江工商大学, 2019.
SUN Z H.Characterization of dominant bacteria and volatile flavors in koumiss and study on its metabolomics[D].Hangzhou:Zhejiang Gongshang University, 2019.
[20] BLASCHE S, KIM Y, MARS R A T, et al.Metabolic cooperation and spatiotemporal niche partitioning in a kefir microbial community[J].Nature Microbiology, 2021, 6(2):196-208.
[21] KADYAN S, RASHMI H M, PRADHAN D, et al.Effect of lactic acid bacteria and yeast fermentation on antimicrobial, antioxidative and metabolomic profile of naturally carbonated probiotic whey drink[J].LWT, 2021, 142:111059.
[22] LOCKHART D J, WINZELER E A.Genomics, gene expression and DNA arrays[J].Nature, 2000, 405(6 788):827-836.
[23] 杨浣漪. 传统酸面团中酿酒酵母和旧金山乳杆菌的种内多样性及其互作研究[D].杭州:浙江大学, 2018.
YANG H Y.Intraspecific diversity of Saccharomvces cerevisiae and Lactobacillus sanfranciscensis isolates from Chinese traditional sourdough and their interaction[D].Hangzhou:Zhejiang University, 2018.
[24] 张国华, 王伟, 涂建, 等.基于宏转录组学技术解析传统酸面团中微生物代谢机理[J].中国粮油学报, 2019, 34(11):10-16.
ZHANG G H, WANG W, TU J, et al.Analysis of microbial metabolism mechanism in traditional sourdough fermentation based on macro-transcriptomics technology[J].Journal of the Chinese Cereals and Oils Association, 2019, 34(11):10-16.
[25] FU W H, LIU C L, MENG X, et al.Co-culture fermentation of Pediococcus acidilactici XZ31 and yeast for enhanced degradation of wheat allergens[J].International Journal of Food Microbiology, 2021, 347:109190.
[26] MALIGOY M, MERCADE M, COCAIGN-BOUSQUET M, et al.Transcriptome analysis of Lactococcus lactis in coculture with Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2008, 74(2):485-494.
[27] PONOMAROVA O, GABRIELLI N, SÉVIN D C, et al.Yeast creates a niche for symbiotic lactic acid bacteria through nitrogen overflow[J].Cell Systems, 2017, 5(4):345-357.
[28] MENDES F, SIEUWERTS S, DE HULSTER E, et al.Transcriptome-based characterization of interactions between Saccharomyces cerevisiae and Lactobacillus delbrueckii subsp.bulgaricus in lactose-grown chemostat cocultures[J].Applied and Environmental Microbiology, 2013, 79(19):5 949-5 961.
[29] LIU N, QIN L K, MAZHAR M, et al.Integrative transcriptomic-proteomic analysis revealed the flavor formation mechanism and antioxidant activity in rice-acid inoculated with Lactobacillus paracasei and Kluyveromyces marxianus[J].Journal of Proteomics, 2021, 238:104158.
[30] 范维, 李路, 姜铁民, 等.PCRDGGE技术分析混菌发酵乳中马克斯克鲁维酵母与乳酸菌的相互作用[J].食品工业科技, 2015, 36(11):147-152.
FAN W, LI L, JIANG T M, et al.Application of PCR- DGGE to analyze the interaction between Kluyveromyces marxianus and the lactic acid bacteria during fermentation and storage process[J].Science and Technology of Food Industry, 2015, 36(11):147-152.
[31] STADIE J, GULITZ A, EHRMANN M A, et al.Metabolic activity and symbiotic interactions of lactic acid bacteria and yeasts isolated from water kefir[J].Food Microbiology, 2013, 35(2):92-98.
[32] SUDUN, WULIJIDELIGEN, ARAKAWA K, et al.Interaction between lactic acid bacteria and yeasts in airag, an alcoholic fermented milk[J].Animal Science Journal, 2013, 84(1):66-74.
[33] 范维, 张咚咚, 张彧, 等.马克斯克鲁维酵母对发酵乳中糖代谢的影响[J].食品科学, 2015, 36(15):128-134.
FAN W, ZHANG D D, ZHANG Y, et al.Effect of Kluyveromyces marxianus on lactose metabolism in traditional fermented milk[J].Food Science, 2015, 36(15):128-134.
[34] 李啸. 我国传统泡菜自然发酵与单菌发酵微生物及代谢特性研究[D].南昌:南昌大学, 2014.
LI X.The study on microbiological and metabolic property in natural and stater culture fermentation of Chinese sauerkraut[D].Nanchang:Nanchang University, 2014.
[35] 王小标, 武运, 王璐, 等.酸马乳发酵过程中乳酸菌与酵母菌生长的相互影响[J].食品科学, 2015, 36(11):125-129.
WANG X B, WU Y, WANG L, et al.Interaction between lactic acid bacteria and yeast during koumiss fermentation[J].Food Science, 2015, 36(11):125-129.
[36] 洪家丽, 陈敏, 周文斌, 等.乳酸菌对红曲黄酒酿造过程酵母菌生长及风味物质形成的影响[J].中国食品学报, 2020, 20(1):91-100.
HONG J L, CHEN M, ZHOU W B, et al.Effects of lactic acid bacteria on the growth of yeast and the formation of flavor compounds in the fermentation of Hongqu glutinous rice wine[J].Journal of Chinese Institute of Food Science and Technology, 2020, 20(1):91-100.
[37] 侯楠楠, 谢全喜, 雷春红, 等.不同微生物青贮剂对全株玉米青贮饲料的影响[J].中国酿造, 2019, 38(11):63-66.
HOU N N, XIE Q X, LEI C H, et al.Effect of different microbial silage agents on whole crop corn silage[J].China Brewing, 2019, 38(11):63-66.
[38] SIEUWERTS S, BRON P A, SMID E J.Mutually stimulating interactions between lactic acid bacteria and Saccharomyces cerevisiae in sourdough fermentation[J].LWT, 2018, 90:201-206.
[39] LEROI F, PIDOUX M.Detection of interactions between yeasts and lactic acid bacteria isolated from sugary kefir grains[J].Journal of Applied Bacteriology, 1993, 74(1):48-53.
[40] ABBOTT D A, SUIR E, VAN MARIS A J A, et al.Physiological and transcriptional responses to high concentrations of lactic acid in anaerobic chemostat cultures of Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2008, 74(18):5 759-5 768.
[41] 张文娟, 陈安特, 韩宇琴, 等.酿酒酵母对萝卜泡菜发酵的影响[J].食品与发酵工业, 2017, 43(8):134-137.
ZHANG W J, CHEN A T, HAN Y Q, et al.Effects of Saccharomyces cerevisiae on the fermentation of radish pickle[J].Food and Fermentation Industries, 2017, 43(8):134-137.
[42] MILLER M B, BASSLER B L.Quorum sensing in bacteria[J].Annual Review of Microbiology, 2001, 55:165-199.
[43] ZHANG C, ZHOU X H, TONG T Q, et al.Acetic acid acting as a signaling molecule in the quorum sensing system increases 2, 3-butanediol production in Saccharomyces cerevisiae[J].Preparative Biochemistry & Biotechnology, 2022, 52(5):487-497.
[44] JOHANSEN P, JESPERSEN L.Impact of quorum sensing on the quality of fermented foods[J].Current Opinion in Food Science, 2017, 13:16-25.
[45] 廉雪花. 酸马奶酒中乳酸菌产AI-2信号分子的研究[D].呼和浩特:内蒙古农业大学, 2014.
LIAN X H.Study of AI-2Signal molecule in lactic acid bacteria from koumiss[D].Hohhot:Inner Mongolia Agricultural University, 2014.
[46] PROUST L, HAUDEBOURG E, SOURABIÉ A, et al.Multi-omics approach reveals how yeast extract peptides shape Streptococcus thermophilus metabolism[J].Applied and Environmental Microbiology, 2020, 86(22):e01446-e01420.
[47] 顾悦. 环境胁迫及酵母菌对乳酸菌LuxS/AI-2群体感应系统的影响[D].呼和浩特:内蒙古农业大学, 2017.
GU Y.The effects of environmental stresses and yeast on LuxS/AI-2 quorum sensing system of lactic acid bacteria[D].Hohhot:Inner Mongolia Agricultural University, 2017.