酱油是一种发源自我国的传统酿造调味品,又称“酱汁”或“清酱”,距今已有2 500多年历史,深受人们喜爱[1]。酱油发酵是一个半开放的混菌共同发酵体系,参与的微生物主要是米曲霉、酵母菌和乳酸菌。乳酸菌和酵母菌是制曲和发酵过程中从环境中带入的微生物,微生物种群是酱油的发酵和风味物质合成的重要影响因素之一[2]。酱醪发酵是酱油制作中最重要的阶段之一,在酱油酿造中起到关键作用。酱醪发酵阶段主要是以酵母菌和乳酸菌为主进行液体发酵,这个阶段生成大量的醇类、酯类和酚类物质以及芳香族化合物,赋予酱油特殊的香气[3-4]。
目前微生物多样性的研究方法主要分为传统可培养方法和免培养方法。传统可培养方法是最常见的研究方法之一,利于了解分离纯化得到的微生物的形态特征和生理特性[5]。YAN等[6]利用传统微生物培养法对日本酱油发酵过程中微生物变化分析得出乳酸菌为优势菌群,其次为酵母菌和霉菌。随着分子生物学的进步,免培养技术逐渐趋于成熟。近年来利用免培养技术研究酱油(醪)发酵过程中微生物多样性的报道明显增多,谢显华[7]利用变性梯度凝胶电泳技术对3个月发酵周期的酱油细菌群落结构演变规律进行了分析,发现魏斯氏菌属为优势菌属。WANG等[8]使用宏基因组学研究酱油中细菌群落多样性,发现酱醪阶段存在34个细菌属,其中以库特菌属、葡萄球菌属和肠球菌属为优势菌。阮志强等[9]采用高通量测序研究酱油发酵过程中真菌群落结构变化及优势真菌,整个发酵过程优势菌为曲霉属、柯达酵母属和结合酵母属。
目前国内外对酱油(醪)发酵大多采用单一分析方法对微生物多样性进行研究,或是针对细菌或真菌的群落结构动态变化研究其与风味物质的相关性。但是,鲜有使用传统可培养技术和免培养技术结合的方法探索酱醪发酵过程中细菌及真菌群落结构和多样性的研究。本研究采用高通量测序技术和传统可培养技术结合的手段,对酱醪在发酵过程中细菌和真菌群落的动态变化进行研究,解析不同时期酱醪中主要微生物群落的构成,为企业提高酱油品质及酱醪发酵时间的调控提供理论依据。
1 材料与方法
1.1 材料与设备
1.1.1 样品采集
利用无菌袋分别对发酵时间为0、1、2、3、4、5、6个月的酱醪进行取样,样品采用高盐稀态酿造工艺,四川清香园调味品有限公司。
1.1.2 实验试剂
Bacterial DNA Isolation Kit,2× PCR Mix,成都福际生物技术公司;Biomiga EZgene TM Fungal gDNA Miniprep Kit,美国Biomiga;PCR引物,成都擎科梓熙生物技术有限公司;无水乙醇,成都市科龙化工有限公司;琼脂糖,北京索莱宝科技有限公司;MRS培养基、高盐MRS培养基(含100 g/L食盐)、孟加拉红培养基和PDA培养基均现配现用,其他试剂均为分析纯。
1.1.3 实验设备
T960梯度PCR热循环仪,杭州晶格科学仪器有限公司;JY04S-3E凝胶成像系统,北京君意东方电泳设备有限公司;DYCP-31DN琼脂糖水平电泳仪,北京市六一仪器厂;THZ-C-1台式恒温振荡器,江苏太仓实验设备厂;DHP-9080B恒温培养箱,上海琅玕实验设备有限公司;YXQ-LS-50SII立式压力蒸汽灭菌锅,上海博迅实业有限公司;SW-CJ-1FD超净工作台,江苏苏净集团有限公司;ABI GeneAmp® 9700 PCR仪,美国应用生物系统公司;TGL-20BR台式高速冷冻离心机,上海安亭科学仪器厂;DNP-9162电热恒温培养箱,长沙精开仪器设备有限公司。
1.2 实验方法
1.2.1 酱醪中微生物计数
将1 mL酱醪加入到9 mL质量浓度8.5 g/L无菌生理盐水中,按照10倍梯度稀释,并选取合适的稀释度和培养基,将0.1 mL稀释液注入90 mm培养皿中,采用涂布法分别对乳酸菌和酵母菌活菌计数。非嗜盐乳酸菌选择MRS培养基(添加30 g/L纳他霉素),嗜盐乳酸菌选择高盐MRS培养基(含100 g/L食盐),待涂布完毕后,置于37 ℃恒温培养箱中,培养24~48 h后进行计数;酵母菌选择孟加拉红培养基,置于28 ℃恒温培养箱中,培养48~72 h后计数。
1.2.2 酱醪中微生物的分离、纯化
非嗜盐乳酸菌:在乳酸菌计数平板中,按菌落形态差异(形状、颜色、大小、凸起度、干湿度、透明度、边缘完整性)挑选菌落于MRS平板上划线,并记录该菌株在平板上的相似菌株数量。将菌株反复纯化3~4次后,进行革兰氏染色与接触酶试验,筛选出革兰氏阳性、接触酶阴性的无芽孢纯培养物;将筛选出的菌株接入MRS液体培养基,并在37 ℃恒温培养箱中培养18~24 h,然后与甘油溶液以体积比1∶1加入甘油管中(甘油最终体积分数20%),保存于-80 ℃超低温冰箱。
嗜盐乳酸菌:选用添加食盐(100 g/L)的MRS培养基进行嗜盐乳酸菌计数。按相同步骤进行稀释涂布后,于30 ℃下培养6~7 d进行计数。分离纯化,镜检后,将筛选出的菌株接入MRS液体培养基,并在37 ℃恒温培养箱中培养18~24 h,然后与甘油溶液以体积比1∶1加入到甘油管中(甘油最终体积分数20%),保存于-80 ℃超低温冰箱备用。
酵母菌:将计数平板中挑选的单菌落(按形状、颜色等培养特征)进行3~4次划线分离纯化,于PDA培养基上纯化。将纯化后的菌株进行形态描述并进行镜检,观察是否有杂菌,若有,则还需进一步划线分离,直至为纯培养物。将纯化后的菌株接种PDA液体培养基中28 ℃培养24~36 h后,将菌液按体积比1∶1加入到已灭菌的甘油管中(甘油最终体积分数20%),放入-80 ℃超低温冰箱保藏。
1.2.3 细菌16S rDNA分析
使用细菌通用引物27F和1492R对16S rDNA全长进行基因扩增,扩增后的PCR产物由成都擎科梓熙生物技术有限公司进行测序,采用双向测序法,即每株菌得到2条800~1 000 bp的片段。利用BLAST进行比对,寻找基因序列同源性最高的已知分类地位的菌种。从美国国家生物技术信息中心(NCBI)上查找到相关细菌模式菌株16S rDNA序列,利用Mega 5.0软件将测序的菌株与模式菌株序列多重对比后进行系统进化亲缘关系研究,采用邻接法构建系统发育树。
1.2.4 真菌26S rDNA分析
采用NL1和NL4引物扩增26S rDNA片段,PCR扩增后的产物由成都擎科梓熙生物技术有限公司进行测序,将测序后的结果利用BLAST进行比对,寻找基因序列同源性最高的已知分类地位的菌种;从NCBI的GenBank中查找代表性序列,将其与测序序列进行多重对比后作系统进化亲缘关系研究,利用软件Mega 5.0构建系统发育树。
1.2.5 高通量测序分析
样品的高通量测序采用上海美吉生物医药科技有限公司MiSeq测序平台,按照97%相似性对非重复序列(不含单序列)进行操作分类单元(operational taxonomic units,OTU)聚类,在聚类过程中去除嵌合体,得到OTU的代表序列,将所有优化序列map至OTU代表序列,选出与OTU代表序列相似性在97%以上的序列,16S细菌使用Silva数据库,ITS真菌使用Unite真菌数据库进行比对。最后采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,并统计每个样品的群落组成[10]。
2 结果与分析
2.1 酱醪发酵过程中微生物菌变化分析
在酱醪发酵过程中,米曲霉通常在发酵初期由人工接种,第0月时高达108CFU/mL,但其在酱醪发酵1个月时就已消失,主要是其产生的蛋白酶参与后续发酵。酱醪中酵母菌和乳酸菌主要由环境带入,故其发酵初期含量较高[11]。如图1所示,发酵第0月时,非嗜盐乳酸菌含量达到108 CFU/mL,发酵第0~1月数量急剧下降,可能是酱醪中盐含量较高,导致体系中非嗜盐乳酸菌迅速死亡[12];发酵第1~4月,非嗜盐乳酸菌数量下降缓慢,可能是由于总酸上升,pH下降,抑制了非嗜盐乳酸菌的生长;发酵第5月时非嗜盐乳酸菌已消失。酵母菌发酵初期数量远低于乳酸菌,只有106 CFU/mL左右,随着发酵的进行,数量下降,但在发酵第5月和第6月时略有升高。同非嗜盐乳酸菌相比较,嗜盐乳酸菌在发酵初期数量较少,但发酵1个月时,数量骤增至108CFU/mL,而后嗜盐乳酸菌数量逐渐减少,但在发酵后期仍是酱醪中的优势微生物菌群,发挥主要作用,这与ZHANG等[13]的研究结果一致,并且嗜盐乳酸菌和酵母菌存在于整个酱醪发酵过程。
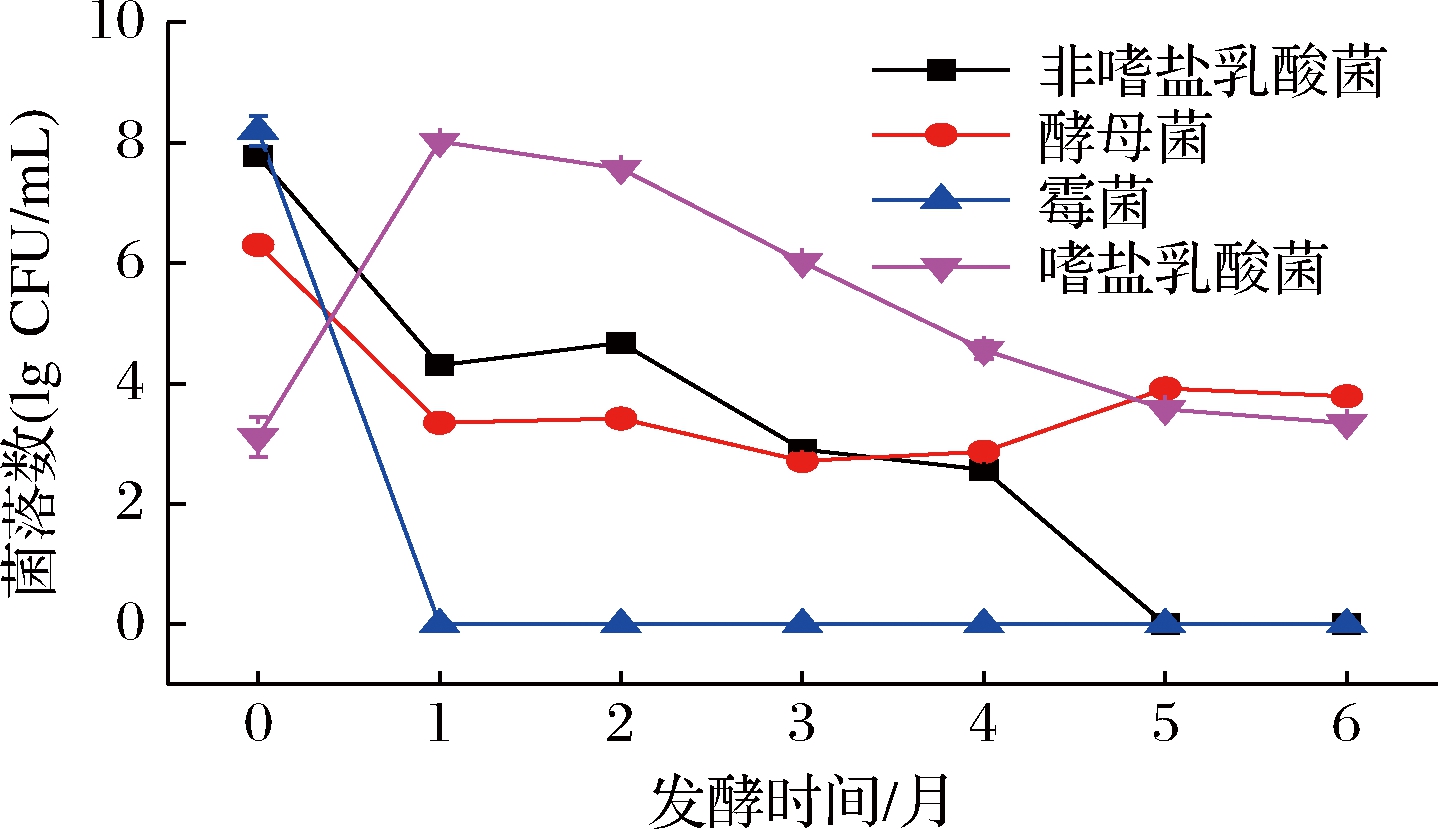
图1 酱醪发酵过程中微生物菌相变化
Fig.1 Microbial phase changes during soy sauce fermentation
2.2 传统可培养方法鉴定酱醪发酵过程中的非嗜盐乳酸菌
2.2.1 非嗜盐乳酸菌分离鉴定
从酱醪发酵各阶段的样品中分离纯化得到非嗜盐乳酸菌共计42株。测序结果通过BLAST软件在GenBank数据库中比对,比对结果见图2。

图2 非嗜盐乳酸菌系统发育树
Fig.2 Phylogenetic tree of non-halophilic lactic acid bacteria
通过比对乳酸菌的16S rDNA序列和构建系统发育树发现,JY1R1等与模式菌株Pediococcus lolii/acidilactici的序列非常相近,同源性非常高,因此将其鉴定结果记为Pediococcus lolii/acidilactici;同理,JY1R5等菌株鉴定为Lactobacillus acidipiscis/pobuzihi,JY1R2等菌株鉴定为Enterococcus sp.,JY0R6鉴定为Leuconostoc pseudomesenteroides,JY0R13等菌株鉴定为Weissella cibaria,JY0R4鉴定为Weissella thailandensis,JY0R2鉴定为Weissella paramesenteroides/hellenica。
2.2.2 酱醪发酵过程中非嗜盐乳酸菌群落结构以及数量变化
如图3所示,酱醪发酵过程中,前期乳酸菌种类较后期丰富,发酵初期,Weissella比例较高,W.thailandensis,W.paramesenteroides/hellenica,W.cibaria/confusa相对丰度较高,也有一部分Leuconostoc pseudomesenteroides/mesenteroides存在。发酵第1月时,Weissella和Leuconostoc均消失,Enterococcus相对丰度最高,其次是P.lolii/acidilactici,L.acidipiscis/pobuzihi也存在;发酵2~4月,仅存在L.acidipiscis/pobuzihii和Enterococcus,而L.acidipiscis/pobuzihii相对丰度远高于Enterococcus。酱醪发酵5个月之后,未分离鉴定到非嗜盐乳酸菌。

图3 可培养方法中非嗜盐乳酸菌群落结构
Fig.3 Community structure of non-halophilic lactic acid bacteria in culturable medium
如图4所示,Weissella在酱醪发酵初期,数量高达5×107CFU/mL,但在发酵第1月时就已消失,可能原因是这类菌株对高盐环境较为敏感;P.lolii/acidilactici仅出现在酱醪发酵第1个月,数量约7×103CFU/mL,发酵第2个月时就已消失。Enterococcus和L.acidipiscis/pobuzihii为酱醪发酵中期的主要乳酸菌,发酵2月时数量达到最大值,随后开始下降;发酵第1月时,Enterococcus sp.数量高于L.acidipiscis/pobuzihii;发酵2~4月,L.acidipiscis/pobuzihii数量则比Enterococcus高1个数量级。酱醪发酵5~6月非嗜盐乳酸菌消失。
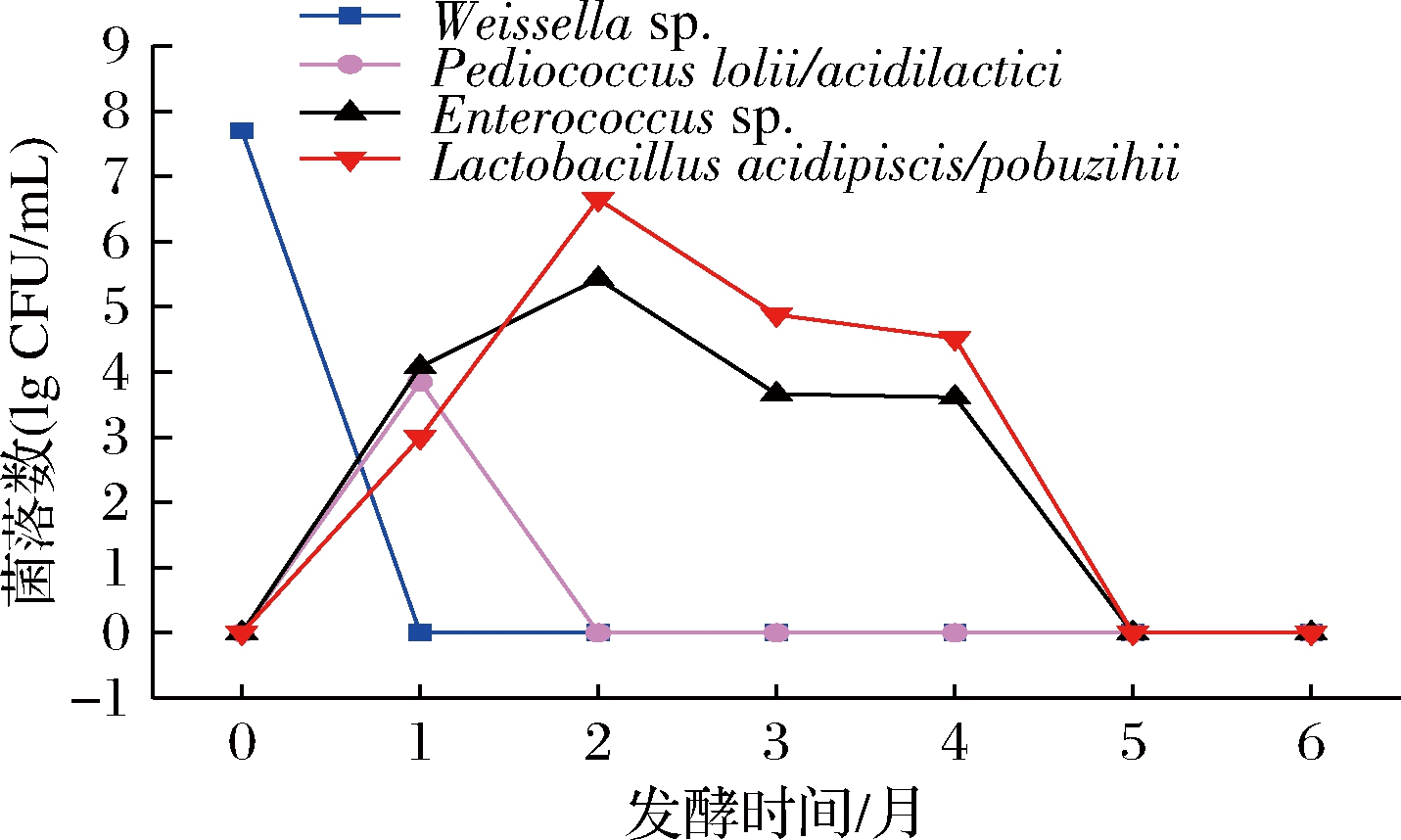
图4 可培养技术优势非嗜盐乳酸菌的数量变化
Fig.4 Quantitative changes of dominant non-halophilic lactic acid bacteria in culturable medium
2.3 传统可培养方法鉴定酱醪发酵过程中的酵母菌
2.3.1 酱醪发酵酵母菌分离鉴定
从酱醪发酵各阶段的样品中分离、纯化后得到酵母菌共计24株,测序结果通过BLAST软件在GenBank数据库中比对,比对结果见图5。
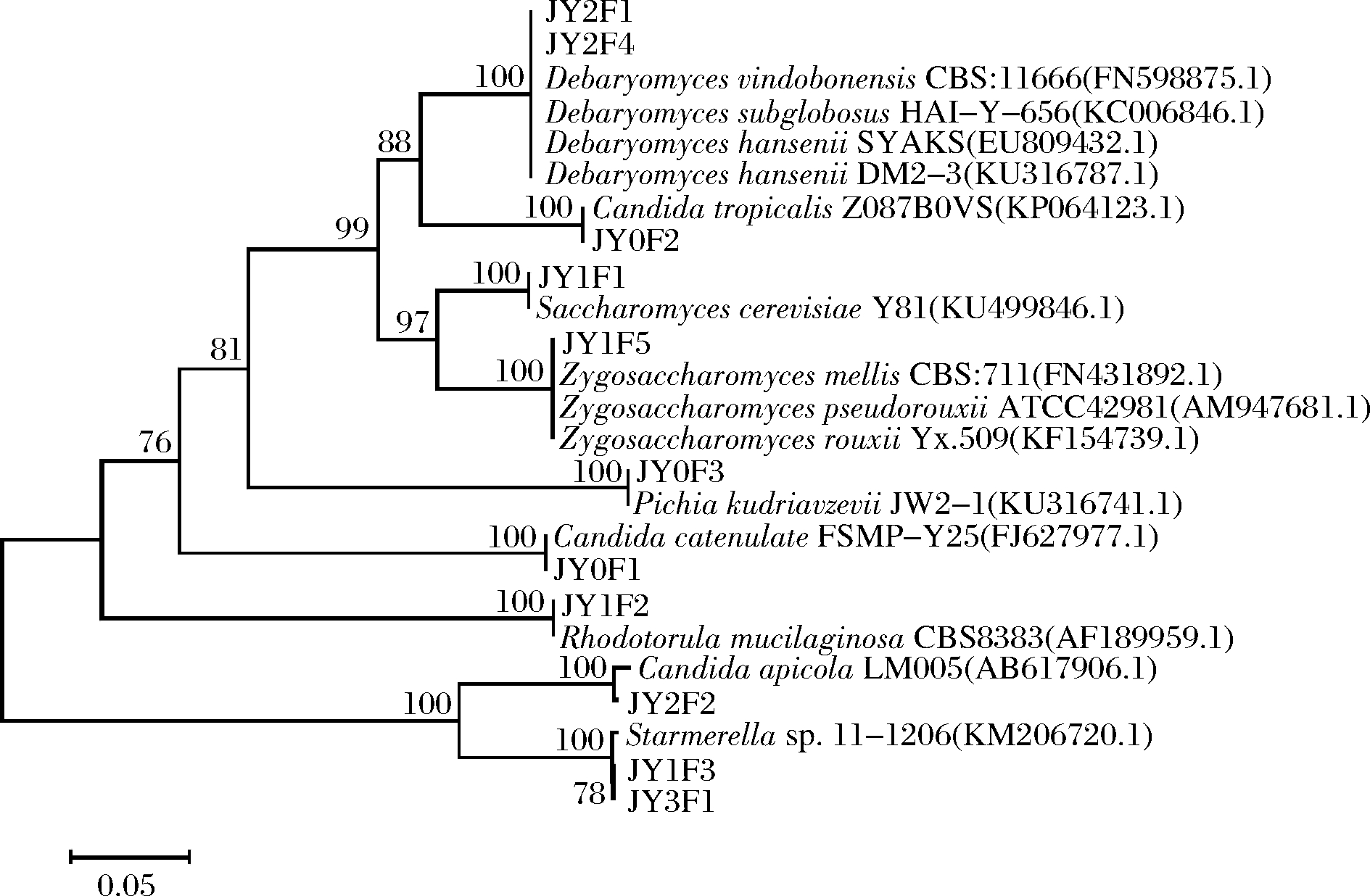
图5 酵母菌系统发育树
Fig.5 Yeast phylogenetic tree
通过比对酵母菌的26S rDNA序列和构建系统发育树发现,JY2F1、JY1F4与Debaryomyces hansenii,Debaryomyces vindobonensis,Debaryomyces subglobosus等有效菌株序列同源性都较高,系统发育树聚为一支,故鉴定为Debaryomyces sp.;JY0F2鉴定为Candida tropicalis;JY1F1鉴定为Saccharomyces cerevisiae;JY1F5鉴定为Zygosaccharomyces sp.;JY0F3鉴定为Pichia kudriavzevii;JY0F1鉴定为Candida catenulata;JY1F2鉴定为Rhodotorula mucilaginosa;JY2F2鉴定为Candida apicola;JY1F3鉴定为Starmerella sp.。
2.3.2 酱醪发酵过程中酵母菌群落结构
如图6所示,酵母菌存在于酱醪发酵的整个过程,刚入池发酵时存在P.kudriavzevii、C.tropicalis、C.catenulate 3种酵母,其中P.kudriavzevii相对丰度最高,P.kudriavzevii被发现普遍存在于果酒等传统发酵食品中,是异戊醇、乙酸乙酯等风味化合物的主要来源之一[14]。发酵第3月和第6月时Debaryomyces也占有较高的丰度,ZHANG等[15]发现D.hansenii仅通过蛋氨酸分解代谢产生挥发性硫化合物,能赋予酱醪特殊的风味。发酵1~6月,Starmerella的丰度都很高,为整个酱醪发酵过程的优势真菌属;而R.mucilaginosa、Zygosaccharomyces、S.cerevisiae、C.apicola等可能是发酵前期环境中偶然出现的菌种,在随后的发酵中消失。
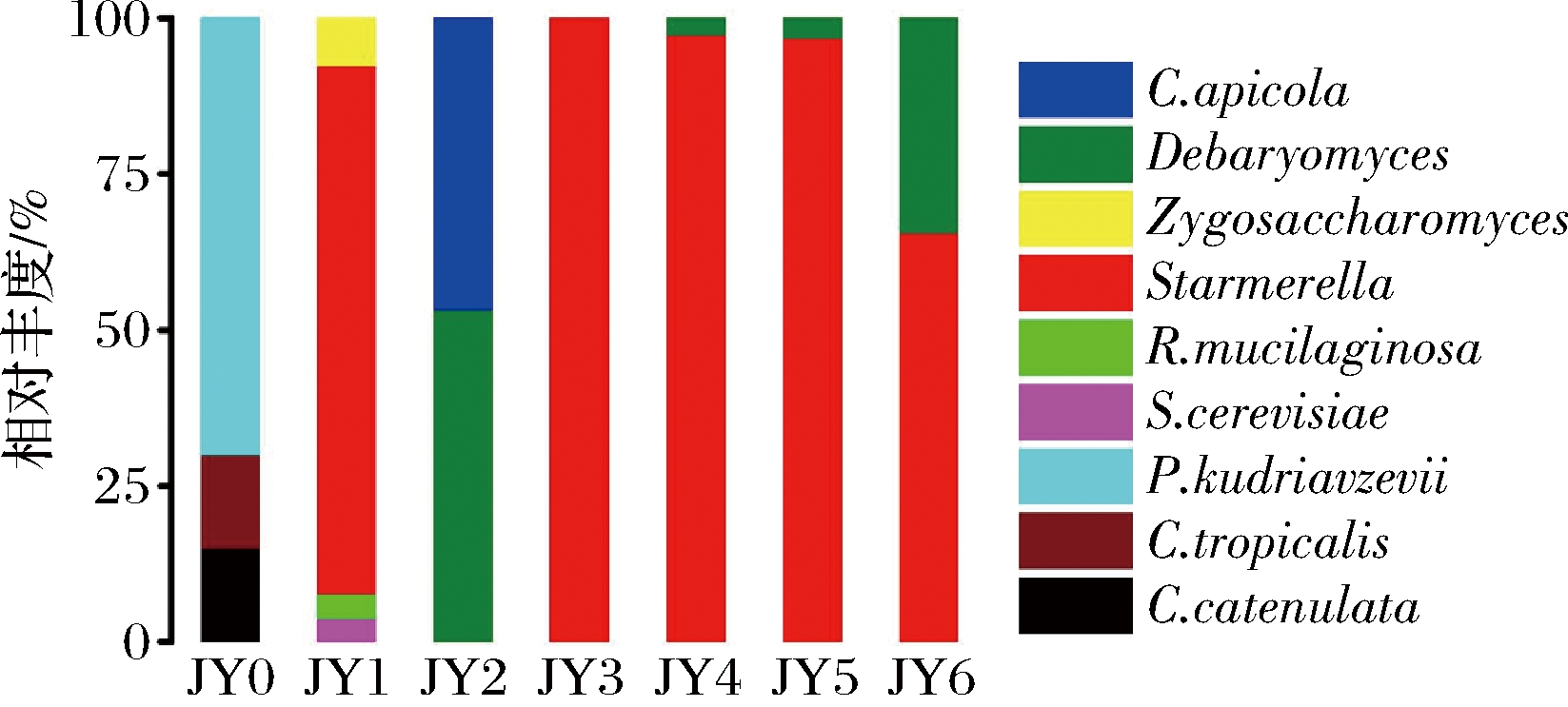
图6 酵母菌群落结构
Fig.6 Yeast community structure
2.4 高通量测序解析酱醪发酵过程中主要优势微生物
2.4.1 样品细菌序列信息和α多样性分析
利用MiSeq对酱醪发酵过程中样品进行测序,根据测序比对结果,7个样品中含有454 470条序列,共得到838个OTUs。如表1所示,7个样品的Coverage均在99.9%以上,说明样品覆盖率较高。Chao1指数和Ace代表物种丰富度,而Shannon度量表示观察到的OTU丰度,其能同时反映丰富度和均匀度[16]。其中,样品JY2的Ace指数和Chao 1指数最高,分别为155和161,说明发酵第2月时细菌物种丰富度最高;样品JY3的OTU指数和Shannon指数最高,分别为138和2.55,说明发酵第3月时细菌多样性最高。由此可见,不同发酵阶段样品之间的微生物多样性以及物种丰度有一定程度的差异。酱醪发酵初期物种相对丰富,多样性呈现明显的下降趋势,到第1月时物种丰富度和多样性到达最低值,随后在发酵第2月开始升高并且恢复到较高水平,表明细菌群落适应了发酵环境并处于动态平衡。
表1 不同样品中α细菌多样性指数
Table 1 Alpha diversity index of bacterial community in different samples
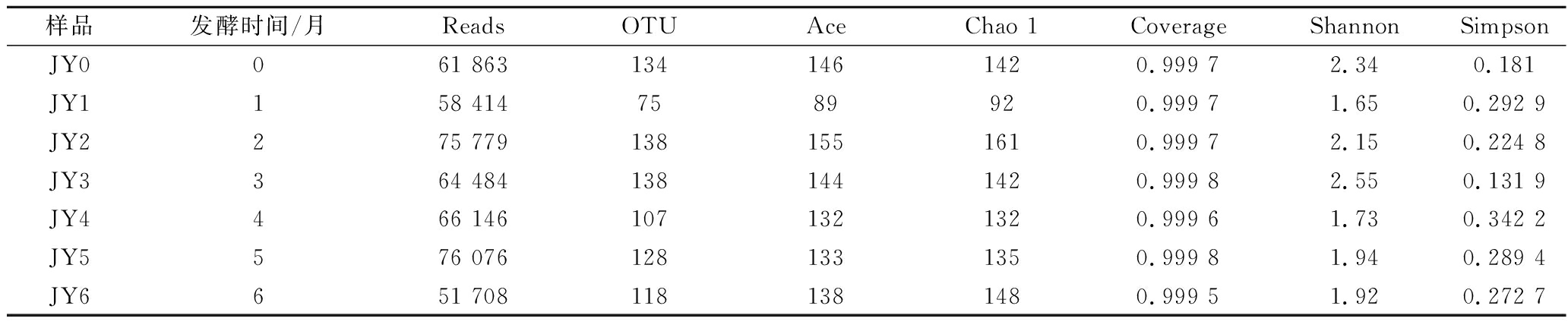
样品发酵时间/月ReadsOTUAceChao 1CoverageShannonSimpsonJY0061 8631341461420.999 72.340.181JY1158 4147589920.999 71.650.292 9JY2275 7791381551610.999 72.150.224 8JY3364 4841381441420.999 82.550.131 9JY4466 1461071321320.999 61.730.342 2JY5576 0761281331350.999 81.940.289 4JY6651 7081181381480.999 51.920.272 7
2.4.2 细菌多样性分析
如图7所示,从门水平看,样品包含7个门,其中糖精细菌门(Saccharibacteria)、Spirochaetae丰度较低,不是酱醪发酵的优势菌群,而厚壁菌门(Firmicutes)占总序列的比例达到了94.04%,是优势菌群。
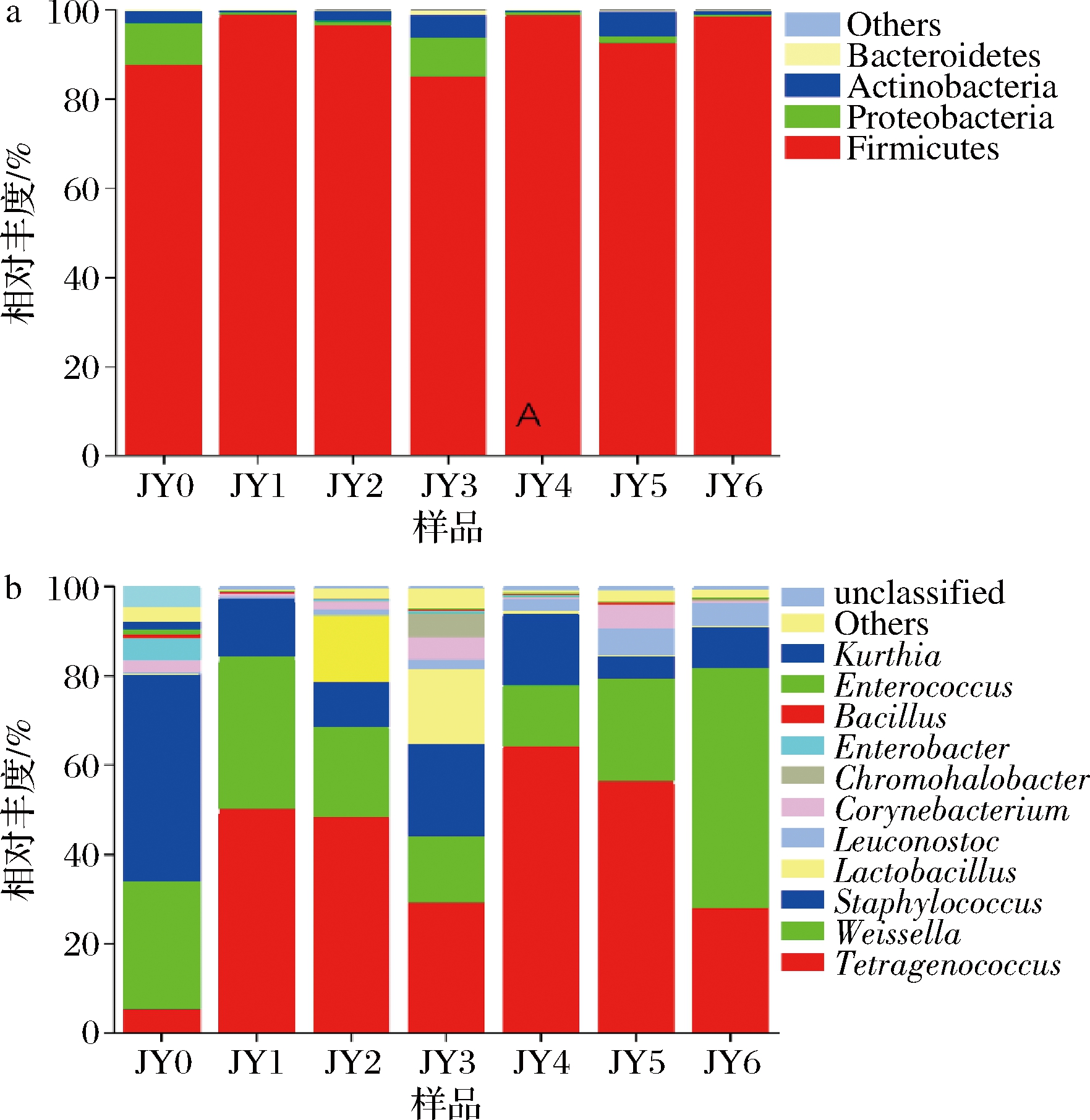
a-门;b-属
图7 酱醪发酵过程中细菌群落在门和属水平上的结构变化
Fig.7 Changes of bacterial communities at phylum and genus levels during soy sauce fermentation
从属水平看,样品包含86个属,其中四联球菌属(Tetragenococcus)和魏斯氏菌属(Weissella)为酱醪发酵中优势菌属,分别占总序列的41.55%和25.84%,Tetragenococcus的丰度呈现先增长后下降的趋势,且在4个月时最高,占64.43%。而Weissella在0个月时占28.65%,相对丰度较高,其后随着发酵的进行,有减少趋势,这与文献报道一致[17-18]。OGASAWARA等[19]的研究中发现Tetragenococcus与碱性蛋白酶或风味蛋白酶结合,能够提高挥发性物质和游离氨基酸的含量以提升酱醪的风味,TRAN等[20]的研究指出,Weissella在酱醪发酵过程中能产生一些有机酸,例如乙酸,以增加酱醪的特殊风味。值得注意的是,Lactobacillus在发酵2个月、3个月时丰度增高,分别为15.03%和15.69%,其后又急减,几乎消失,原因可能是由于大多数Lactobacillus对高盐环境耐受性低所致。
2.4.3 样品真菌序列信息和α多样性分析
如表2所示,利用MiSeq测序方法对酱醪发酵过程中样品进行测序,根据测序比对结果,7个样品中含有237 086条序列,共得到104个OTUs。7个样品的coverage均在99%以上,说明样品覆盖率较高。样品真菌群落的Ace指数和Chao1指数呈现波动变化且逐渐降低,可能是由于随着酱醪发酵的进行,发酵前期带入的环境真菌中有部分不适应高盐环境而逐渐消亡,耐盐真菌开始生长繁殖所致。Shannon指数和Simpson 指数同样呈现出相似的动态变化规律。结合图1真菌数量变化,发酵前期霉菌急剧减少,酵母菌也呈现先下降后呈平稳的趋势,说明前期开始生长的耐盐真菌逐步成为优势菌,而不耐盐真菌则出现消亡,真菌数量处于动态平衡,但物种多样性降低。
表2 不同样品中真菌α多样性指数
Table 2 Alpha diversity index of fungi community in different samples
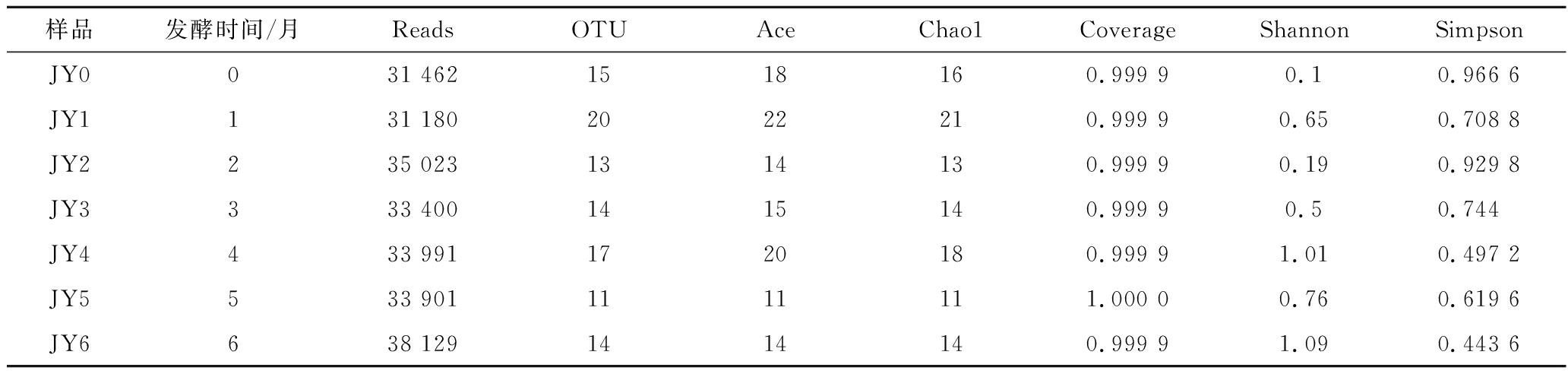
样品发酵时间/月ReadsOTUAceChao1CoverageShannonSimpsonJY0031 4621518160.999 90.10.966 6JY1131 1802022210.999 90.650.708 8JY2235 0231314130.999 90.190.929 8JY3333 4001415140.999 90.50.744JY4433 9911720180.999 91.010.497 2JY5533 9011111111.000 00.760.619 6JY6638 1291414140.999 91.090.443 6
2.4.4 真菌多样性分析
如图8所示,从门水平看,Ascomycota(子囊菌门)占总序列的99.83%,为优势菌群。WEI等[21]所检测出的真菌种类皆属于Ascomycota,与本研究结果相似。从属水平看,Aspergillus 0个月时相对丰富度很高,此时人工接种的米曲霉刚入发酵池;其后在发酵过程中逐渐减少,直至几乎消失。而Starmerella相对丰度逐渐增高,在发酵3月、4月时最高,到6月又降低,此时未有分类信息的环境微生物占据了62.75%。Candida的丰富度也呈现先增后降的趋势,但规律不是很明显。其余菌属占比较少,无规律可循。从整个发酵过程中可以看出Starmerella为优势菌属。目前鲜有文献对酱油发酵过程中的Starmerella
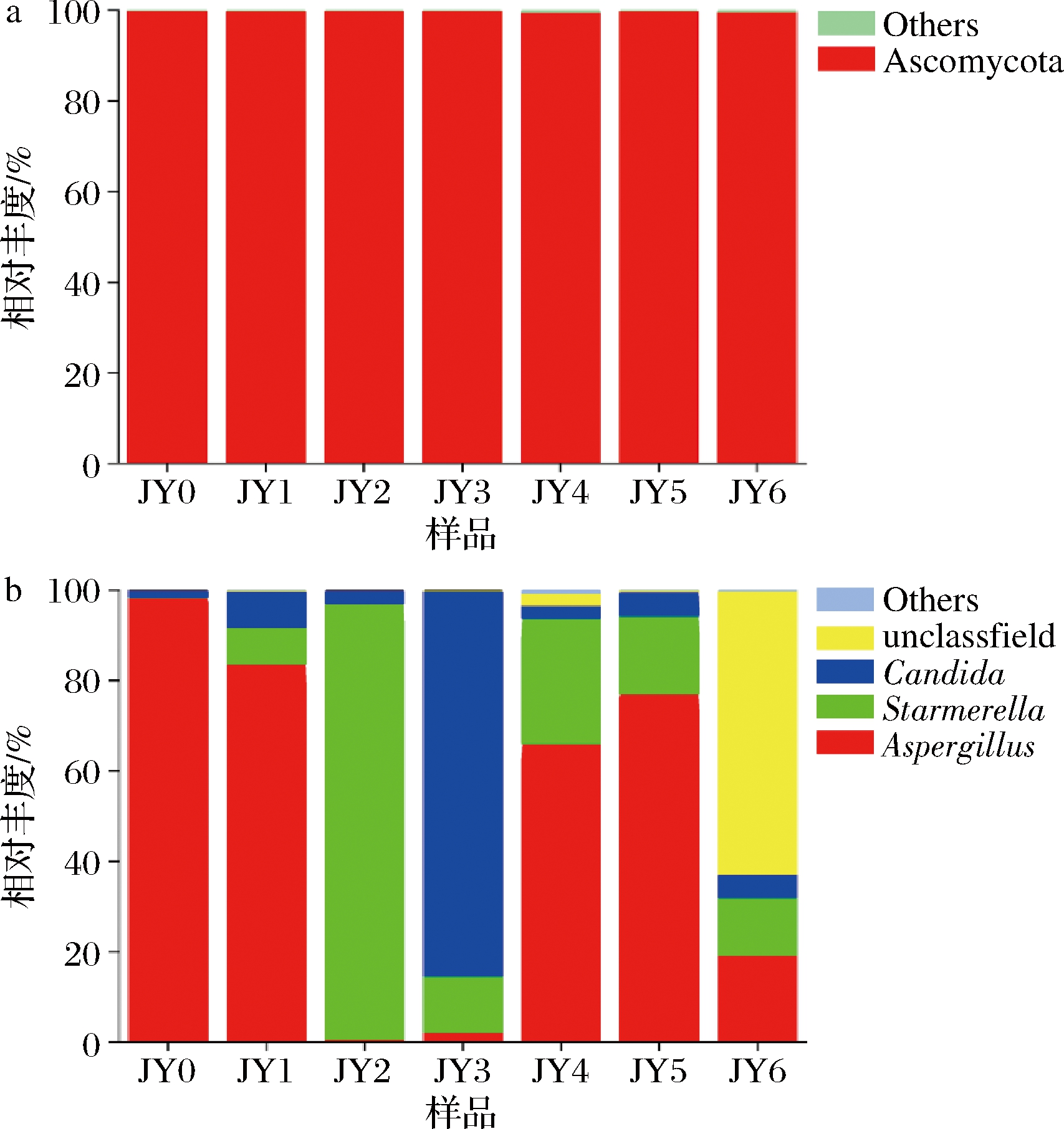
a-门;b-属
图8 酱醪发酵过程中细菌群落在门和属水平上的结构变化
Fig.8 Changes of fungi communities at phylum and genus levels during soy sauce fermentation
进行报道,SULAIMAN等[22]用鸟枪宏基因组方法分析马来西亚的中国传统酱油厂0~6个月的酱醪中微生物变化,发现真菌主要为Candida、Starmerella和Wickerhamiella,而其中Candida为优势菌属。值得一提的是,本研究是首次发现Starmerella在酱醪中作为优势真菌属,其在酱醪发酵过程中的作用特性有待进一步探索。
3 结论
应用Illumina Miseq高通量测序和传统可培养相结合的手段研究酱醪不同发酵阶段微生物群落结构变化及优势菌群,从而揭示酱醪发酵过程中微生物的演替规律,得出以下结论:
(1)基于可培养方法,可将整个酱醪发酵过程分为3个时期。0~1月为发酵前期,此间,霉菌逐渐消亡,非嗜盐乳酸菌含量最高,约108CFU/mL,种类也比较丰富,其中魏斯氏菌属(Weissella sp.)数量较高,认为启动了发酵;2~4个月为发酵中期,乳酸菌种类和数量逐渐减少,其中肠球菌属(Enterococcus)、乳杆菌属(Lactobacillus)和四联球菌属(Tetragenococcus)为优势菌;5~6个月为发酵后期,非嗜盐乳酸菌逐渐消亡,嗜盐乳酸菌和酵母菌成为主要菌群。
(2)高通量测序也可得到相似的结果,酱醪中细菌包含7个门86个属,其中,四联球菌属(Tetragenococcus)占总序列的41.55%,魏斯氏菌属(Weissella)占25.84%,葡萄球菌属(Staphylococcus)占16.81%,乳杆菌属(Lactobacillus)占5.04%。因此,Tetragenococcus和Weissella在酱醪发酵中为优势细菌属。酱醪中真菌主要为子囊菌门(Ascomycota),属水平上以Starmerella、Candida和Aspergillus为主。从整个发酵过程中可以看出Starmerella为优势菌属。当前对以Starmerella为优势真菌的报道几乎没有,其在酱醪发酵过程中的作用特性也不得而知,有待进一步研究。
通过掌握酱醪中微生物的演替规律,探究不同发酵阶段微生物菌群与挥发性风味物质、氨基酸、有机酸等理化指标的相关性。实际酱油生产中可通过人工添加乳酸菌、酵母菌等对发酵过程进行调控,从而达到提高酱油风味和品质的目的。
[1] 胡传旺. 酱油发酵过程微生物群落解析及功能研究[D].无锡:江南大学, 2017.
HU C W.Dynamics of microbial community during soy sauce fermentation and functional analysis strains isolated from moromi[D].Wuxi:Jiangnan University, 2017.
[2] WEI Q Z, WANG H B, CHEN Z X, et al.Profiling of dynamic changes in the microbial community during the soy sauce fermentation process[J].Applied Microbiology and Biotechnology, 2013, 97(20):9 111-9 119.
[3] YANG Y, DENG Y, JIN Y L, et al.Dynamics of microbial community during the extremely long-term fermentation process of a traditional soy sauce[J].Journal of the Science of Food and Agriculture, 2017, 97(10):3 220-3 227.
[4] WANG J W, ZHAO M M, XIE N Y, et al.Community structure of yeast in fermented soy sauce and screening of functional yeast with potential to enhance the soy sauce flavor[J].International Journal of Food Microbiology, 2022, 370:109652.
[5] 张书泰, 周斌, 童星, 等.酱油酿造过程中微生物多样性分析方法研究进展[J].中国调味品, 2019, 44(2):193-200.
ZHANG S T, ZHOU B, TONG X, et al.Research progress on analysis methods of microbial diversity in soy sauce brewing process[J].China Condiment, 2019, 44(2):193-200.
[6] YAN Y Z, QIAN Y L, JI F D, et al.Microbial composition during Chinese soy sauce koji-making based on culture dependent and independent methods[J].Food Microbiology, 2013, 34(1):189-195.
[7] 谢显华. 酱油发酵过程微生物群落结构的动态研究[D].广州:华南理工大学, 2010.
XIE X H.Study on the dynamic changes of microbial community during fermentation process of soy sauce[D].Guangzhou:South China University of Technology, 2010.
[8] WANG H B, WEI Q Z, GUI S Q, et al.Metagenomic profiling of the bacterial community changes from koji to mash stage in the brewing of soy sauce[J].Polish Journal of Microbiology, 2017, 66(4):537-541.
[9] 阮志强, 董玺梅, 蒋雪薇, 等.高盐稀态酱油发酵优势真菌与风味物质相关性分析[J].食品科学, 2022, 43(10):172-179.
RUAN Z Q, DONG X M, JIANG X W, et al.Correlation between dominant fungi and variation of flavor compounds during high-salt liquid-state soy sauce fermentation[J].Food Science, 2022, 43(10):172-179.
[10] 李恒, 陈功, 伍亚龙, 等.高通量测序方法研究传统四川泡菜母水中微生物群落的动态变化[J].食品科学, 2018, 39(24):131-138.
LI H, CHEN G, WU Y L, et al.Analysis of microbial community dynamics of traditional Sichuan Paocai brine by high-throughput sequencing[J].Food Science, 2018, 39(24):131-138.
[11] 燕平梅, 马雁飞, 倪玲.发酵食品中微生物多样性研究方法进展[J].中国酿造, 2011, 30(2):12-15.
YAN P M, MA Y F, NI L.Research advance in methods of microbial diversity in fermented food[J].China Brewing, 2011, 30(2):12-15.
[12] YANG Y, DENG Y, JIN Y L, et al.Dynamics of microbial community during the extremely long-term fermentation process of a traditional soy sauce[J].Journal of the Science of Food and Agriculture, 2017, 97(10):3 220-3 227.
[13] ZHANG L Q, ZHOU R Q, CUI R Y, et al.Characterizing soy sauce moromi manufactured by high-salt dilute-state and low-salt solid-state fermentation using multiphase analyzing methods[J].Journal of Food Science, 2016, 81(11):C2639-C2646.
[14] HU L L, WANG J, JI X A, et al.Selection of non- Saccharomyces yeasts for orange wine fermentation based on their enological traits and volatile compounds formation[J].Journal of Food Science and Technology, 2018, 55(10):4 001-4 012.
[15] ZHANG L, HUANG C C, JOHANSEN P G, et al.The utilisation of amino acids by Debaryomyces hansenii and Yamadazyma triangularis associated with cheese[J].International Dairy Journal, 2021, 121:105135.
[16] KIM B R, SHIN J, GUEVARRA R, et al.Deciphering diversity indices for a better understanding of microbial communities[J].Journal of Microbiology and Biotechnology, 2017, 27(12):2 089-2 093.
[17] 王博, 周朝晖, 李铁桥, 等.嗜盐四联球菌及其在发酵食品中的应用[J].食品与发酵工业, 2017, 43(8):267-272.
WANG B, ZHOU Z H, LI T Q, et al.Tetragenococcus halophilus and its application in fermented foods[J].Food and Fermentation Industries, 2017, 43(8):267-272.
[18] LIU X Y, QIAN M, SHEN Y X, et al.An high-throughput sequencing approach to the preliminary analysis of bacterial communities associated with changes in amino acid nitrogen, organic acid and reducing sugar contents during soy sauce fermentation[J].Food Chemistry, 2021, 349:129131.
[19] OGASAWARA M, YAMADA Y, EGI M.Taste enhancer from the long-term ripening of miso (soybean paste)[J].Food Chemistry, 2006, 99(4):736-741.
[20] TRAN Q N M, MIMOTO H, KOYAMA M, et al.Lactic acid bacteria modulate organic acid production during early stages of food waste composting[J].Science of the Total Environment, 2019, 687:341-347.
[21] WEI C L, CHAO S H, TSAI W B, et al.Analysis of bacterial diversity during the fermentation of inyu, a high-temperature fermented soy sauce, using nested PCR-denaturing gradient gel electrophoresis and the plate count method[J].Food Microbiology, 2013, 33(2):252-261.
[22] SULAIMAN J, GAN H M, YIN W F, et al.Microbial succession and the functional potential during the fermentation of Chinese soy sauce brine[J].Frontiers in Microbiology, 2014, 5:556.