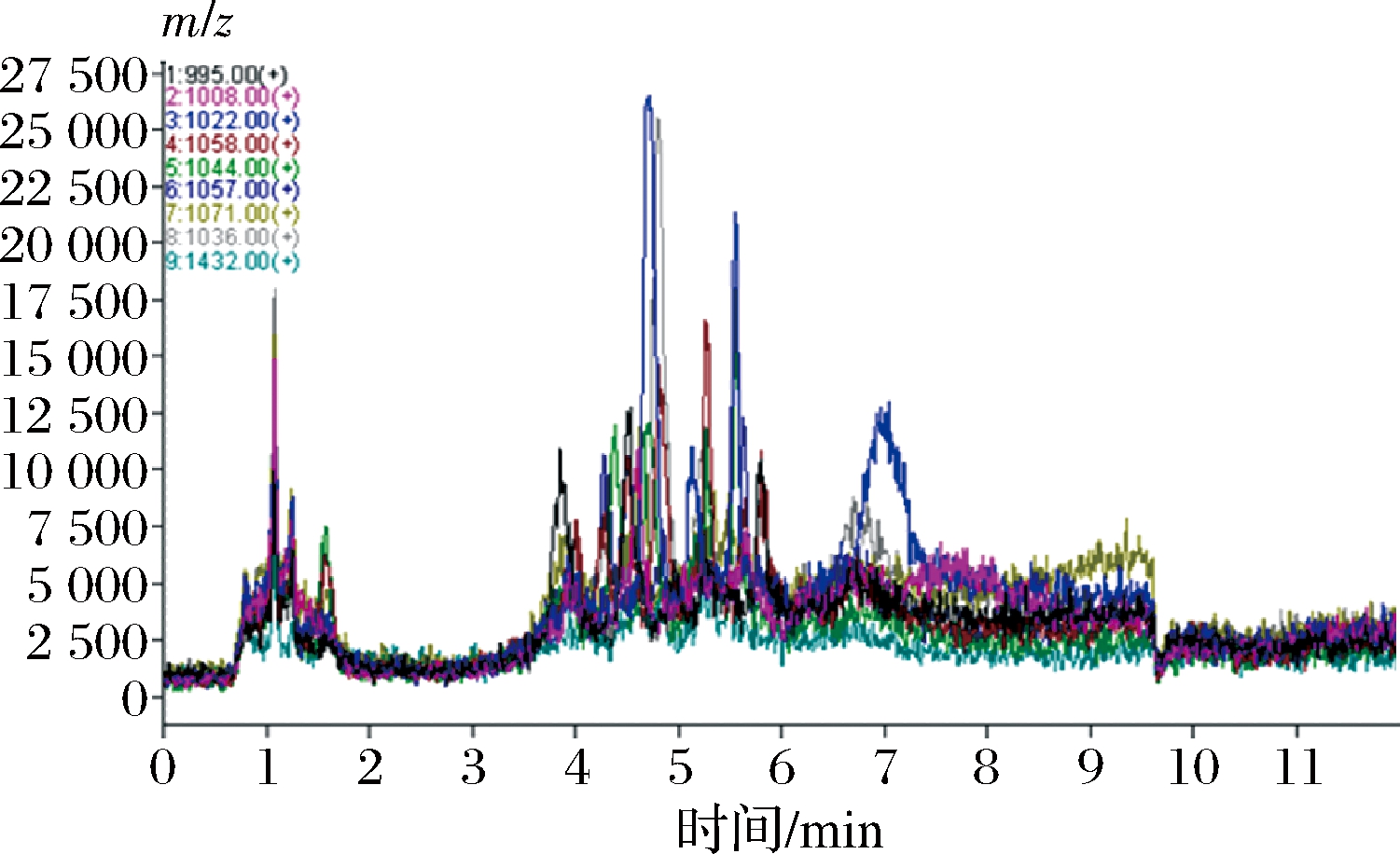
图1 CMT-9发酵产物的总离子流色谱图
Fig.1 Total ion chromatogram of CMT-9 fermentation products
抗菌脂肽是芽孢杆菌中的非核糖体多肽合成酶催化合成的小分子脂肽,主要包括表面活性素(surfactin)、伊枯草菌素(iturin)和芬荠素(fengycin)3大家族化合物[1-2]。抗菌脂肽抗菌谱广,对大多数细菌和真菌都有抑制作用,且具有绿色、高效、低毒、无污染等优点[3-5]。因此,在食品、医药、农业等领域具有广泛的发展前景[6-9]。
芽孢杆菌种类繁多,枯草芽孢杆菌、解淀粉芽孢杆菌和贝莱斯芽孢杆菌等均能产生抗菌脂肽[10-12]。不同芽孢杆菌的脂肽谱有所差异,主要与其合成酶及关键基因的数量和结构相关[13-14]。DENG等[15]对B.velezensis CMT-6进行高通量测序发现,合成酶基因srfA、ituA、fenA的完整性分别是菌株合成脂肽(surfactin、iturin和fengycin)的前提条件,而核心基因发生的InDels和SNP情况是造成脂肽种类和产量差异的主要原因。此外,脂肽是由前体物质氨基酸残基和脂肪酸链组成的大环内酯类物质[16-17],但目前关于前体物质合成途径中功能基因和代谢通路中关键基因的表达情况及其对脂肽谱的影响鲜有报道。
本课题组从海洋红树林生境中筛选到一株芽孢杆菌B.amyloliquefaciens CMT-9,该菌株具有生长速度快、营养需求简单且抗逆性强、抗菌谱广泛等特点,展现出良好的应用潜力。为探明海洋源CMT-9产脂肽的组分及其分子机理,本研究拟以高效液相色谱串联质谱(liquid chromatography tandem mass spectrometry,LC-MS/MS)技术鉴定CMT-9的发酵液组分,采用全基因组测序技术探明CMT-9基因组中合成脂肽的代谢通路和关键基因,同时通过比较基因组学分析其与不同种属的陆地源芽孢杆菌B.velezensis CMT-6(可合成surfactin、iturin和fengycin)、B.velezensis FZB42(可合成surfactin、fengycin)、B.amyloliquefaciens DSM7T(可合成surfactin、iturin)和B.subtilis 168(无脂肽合成能力)之间的脂肽谱差异性,为解析CMT-9产脂肽的调控机制和指导其实际生产提供理论参考。
1.1.1 实验菌株
Bacillus amyloliquefaciens CMT-9,广东海洋大学食品科技学院水产品绿色控制实验室保藏。
1.1.2 培养基
改良Landy培养基(g/L):葡萄糖20.0,L-谷氨酸氢钠5.0,MgSO4·7H2O 0.5,KCl 0.5,KH2PO4·3H2O 1.0,FeSO4·7H2O 0.001 5,MnSO·5H2O 0.05,CuSO4·5H2O 0.001 6,pH 7.0。
LB培养基(g/L):蛋白胨10.0,酵母粉5.0,NaCl 5.0,pH 7.0。
HHS恒温水浴锅、SPX-250B-Z生化培养箱,上海博迅实业有限公司;PHS-3E型pH计,上海雷磁仪器有限公司;DC-12H氮吹仪,上海安谱科学仪器有限公司;Invitrogen Qubit 4荧光定量仪,美国赛默飞世尔公司。
1.3.1 脂肽发酵条件
挑取菌株CMT-9在50 mL LB培养基中37 ℃、150 r/min培养14 h进行活化复壮。按体积分数为5%的接种量加入100 mL改良Landy培养基中,30 ℃、160 r/min发酵培养36 h。
1.3.2 脂肽组分检测
将发酵液置于4 ℃,8 000 r/min离心30 min,获得上清液。加入甲酸调节pH至2.0,静置沉淀8 h。8 000 r/min、4 ℃离心20 min,向沉淀物加入少量的无菌水,用氨水调pH至7.0,甲醇萃取2~6 h,然后10 000 r/min、4 ℃离心20 min,取上清液。同样步骤萃取2次,合并上清液并经旋转蒸发仪浓缩至近干,加入无菌水稀释,用无菌0.22 μm滤膜过滤。采用LC-MS/MS技术测定发酵液主要成分,参照DENG等[18]进行参数设置。
1.3.3 RNA提取和检测
采用DNeasy plantMini kit试剂盒提取CMT-9的基因组RNA,通过1%琼脂糖凝胶电泳检测技术和荧光定量仪检测样品基因组RNA的完整性、总量和浓度。
1.3.4 基因组测序与基因功能注释
将合格的RNA样品送到深圳华大基因研究院进行富集纯化、反转录文库的构建及测序。将通过高通量测序获得的每个样品的转录组文库转化为原始测序序列数据,对其进行CASAVA碱基序列鉴定分析。通过软件HTseq(版本:0.12.4)来处理高通量测序所产生的数据,将这些reads高效、准确地比对到基因上去,使不同基因、不同实验间估计的基因表达水平具有可比性。用DESeq 2R软件分析和比较差异表达基因。采用Benjamini & Hochberg方法调整P值,将P<0.05和∣log2fold change∣≥2设置为差异表达基因。利用GO数据库(http://geneontology.org/)和KEGG数据库(https://www.kegg.jp/kegg/pathway.html)分别对差异表达基因进行分析;利用COG数据库(https://www.ncbi.nlm.nih.gov/research/cog-project/)、CAZy数据库(http://www.cazy.org/)分别对差异表达基因的代谢途径、碳水化合物酶类进行注释。
1.3.5 比较基因组学分析
将B. amyloliquefaciens CMT-9与B.velezensis CMT-6、B.amyloliquefaciens DSM7T、B.velezensis FZB42和B.subtilis168进行比较基因组学分析。
CMT-9发酵产物的LC-MS/MS离子流图如图1所示。

图1 CMT-9发酵产物的总离子流色谱图
Fig.1 Total ion chromatogram of CMT-9 fermentation products
CMT-9菌株发酵产物的主要质荷比为995、1 008、1 022、1 058、1 044、1 057、1 071、1 036、1 432,共9种组分。这些组分的质荷比与文献报道的在相同发酵工艺和纯化条件下检测到的surfactin(m/z为995、1 008、1 022、1 036、1 058、1 044)和iturin(m/z为1 057、1 071)的分子质量相一致[18]。由此初步判定CMT-9菌株所产生的抗菌物质为surfactin和iturin[18]。此外,邓旗等[19]研究发现fengycin A的质荷比在1 431~1 492,由此推测质荷比为1 432的组分可能是fengycin A的一种。
由图2可得,将菌株CMT-9的基因序列与GO数据库进行比对共注释到9 763个基因,其中与分子功能相关的基因数量为4 617个,主要富集在催化活性(catalytical activity),转运能力(transport activity);与细胞组成相关的基因数量为2 189个,主要富集在高分子聚乳酸(macromolecular complex);与生物过程相关的基因数量为2 957个,主要富集在代谢过程(metabolic process),细胞过程(cellular process),生物调节(biological regulation)。

图2 CMT-9差异基因的GO功能分类与富集
Fig.2 GO functional classification and enrichment of CMT-9 differential genes
由图3可得,KEGG分析表明CMT-9发酵培养后的差异基因在代谢(metabolism)、环境信息过程(environmental information processing)和基因信息过程(genetic information processing)通路显著富集,其中最丰富的路径为代谢过程中的碳水化合物代谢(carbohydrate metabolism)和氨基酸代谢(amino acid metabolism),它们的差异基因个数分别为506和411。
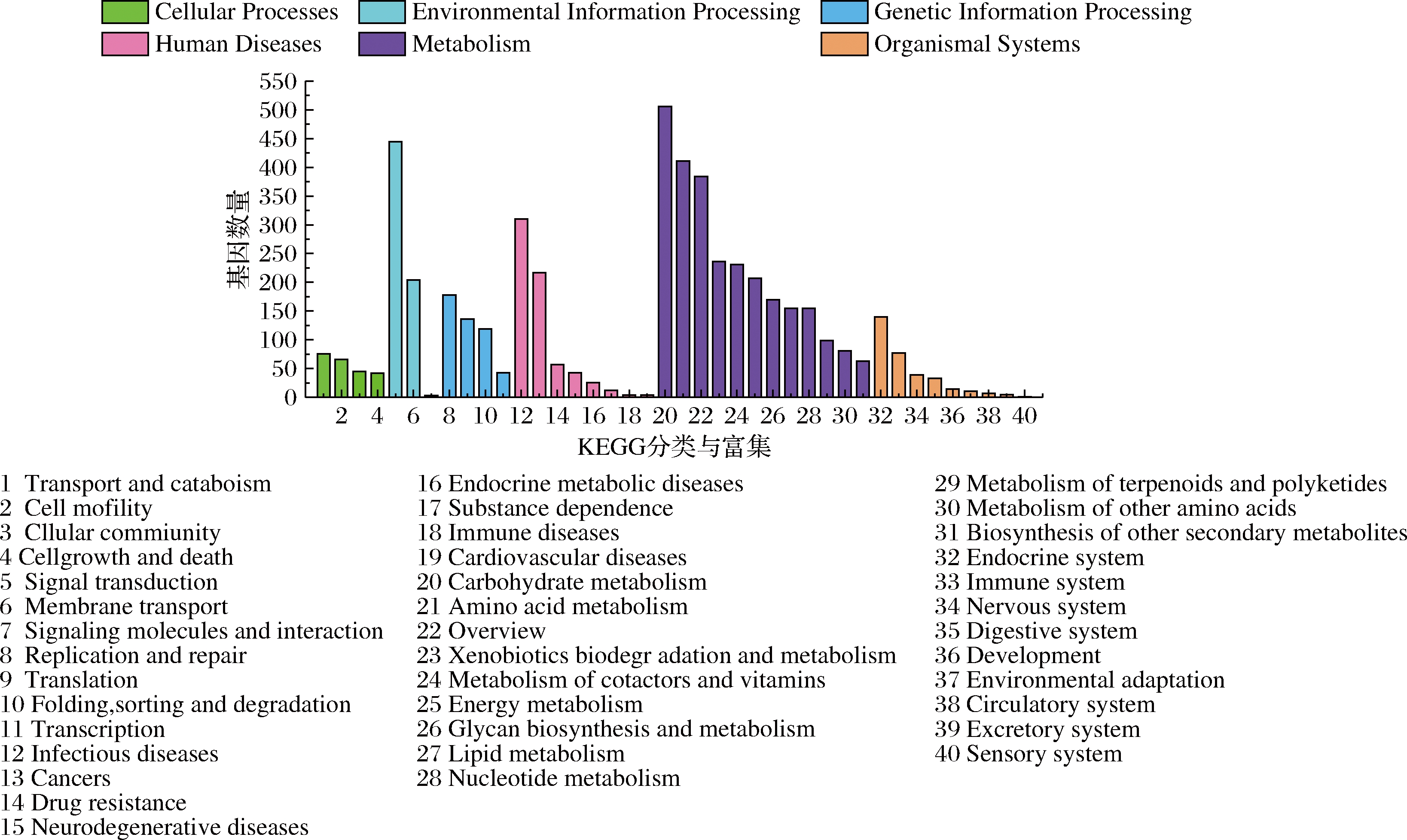
图3 CMT-9差异基因的KEGG通路分类与富集
Fig.3 KEGG pathway classification and enrichment of CMT-9 differential genes
由图4可得,通过COG功能分析发现差异基因主要富集在一般功能预测(general function prediction only)、氨基酸运输与代谢(amino acid transport and metabolism)、碳水化合物运输与代谢(carbohydrate transport and metabolism)。其中,参与氨基酸运输与代谢(amino acid transport and metabolism)的基因个数为333个;参与碳水化合物运输与代谢(carbohydrate transport and metabolism)的基因个数为230个;参与次级代谢物的生物合成、转运和分解代谢(secondary metabolites biosynthesis, transport and catabolism)的基因个数为97个。

图4 CMT-9差异基因的COG功能分类与富集
Fig.4 COG functional classification and enrichment of CMT-9 differential genes
碳水化合物活性酶主要由5类催化酶和一类非催化模块组成,催化酶包括糖苷水解酶(glycoside hydrolases, GHs)、多糖裂解酶(polysaccharidelyases, PLs)、碳水化合物酯酶(carbohydrate esterases, CEs)、糖基转移酶(glycosyltransferases, GTs)以及辅助氧化还原酶(auxiliary activities, AAs),非催化模块即碳水化合物结合模块(carbohydrate-binding modules, CBMs)[20]。由图5可得,菌株CMT-9中含有138个碳水化合物活性酶,其中尤以GH最多,数量为46个,所占比例为33.33%;其次是GT,数量为36,所占比例为26.09%,CBM、CE、AA、PL的数量分别为31、17、5、3,所占比例分别为22.46%、12.32%、3.62%、2.17%。
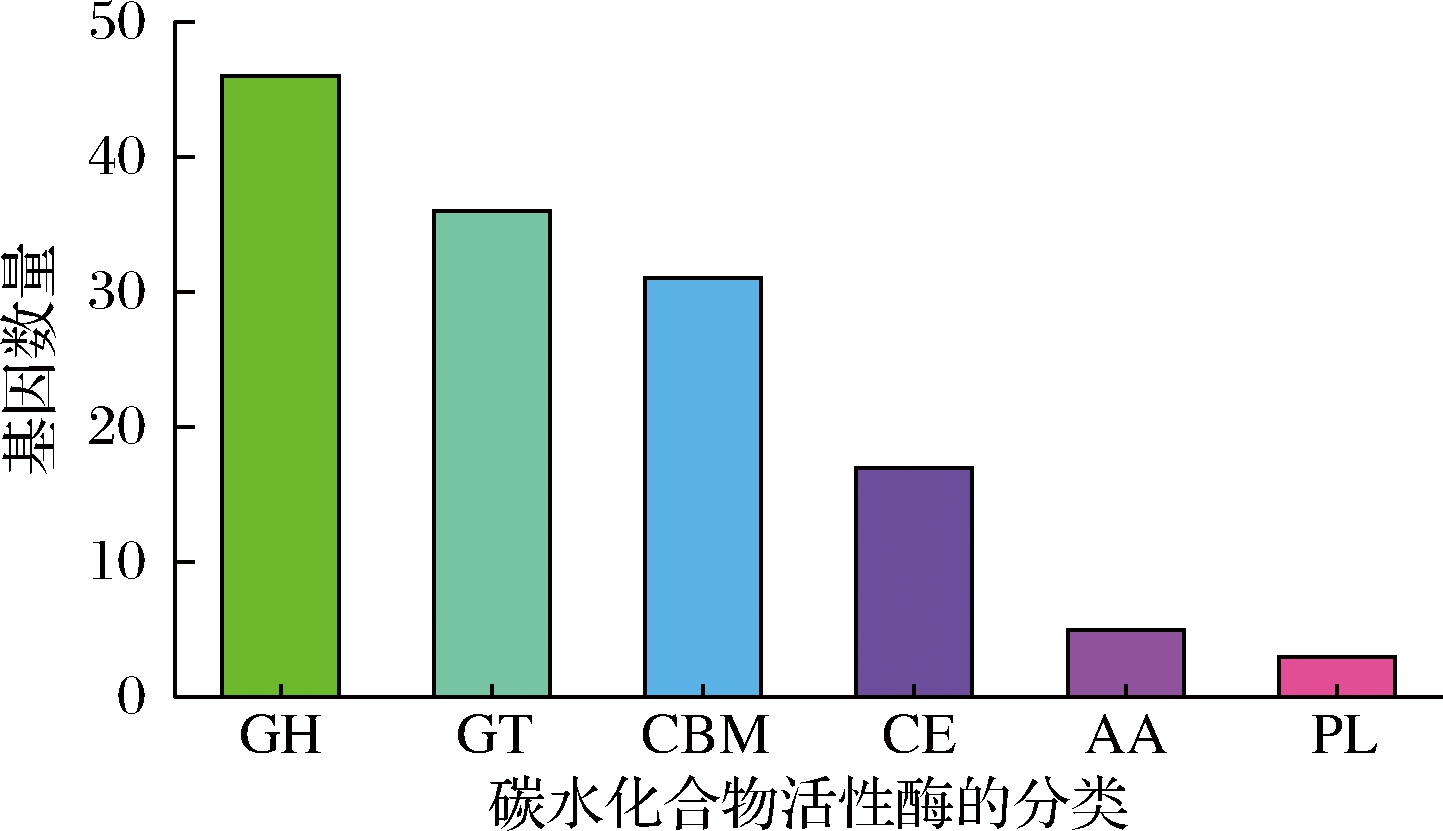
图5 CMT-9碳水化合物活性酶(CAZymes)的分布情况
Fig.5 Distribution of CMT-9 CAZymes
由表1可知,CMT-9的基因组大小为3 928 149 bp,G+C含量为46.39%,蛋白质编码序列长度为4 080,tRNA的数量为83,rRNA的数量为9。CMT-9的基因组大小、G+C含量与CMT-6、FZB42和DSM7T的相似度较高;蛋白质编码序列长度与CMT-6相似度较高;tRNA的数量与CMT-6、FZB42和168的相似度较高。因此,CMT-9与4株菌株的基因组组分的相似度从高到低依次为CMT-6、FZB42、DSM7T、168。
表1 基因组组分比较
Table 1 Genome component comparison
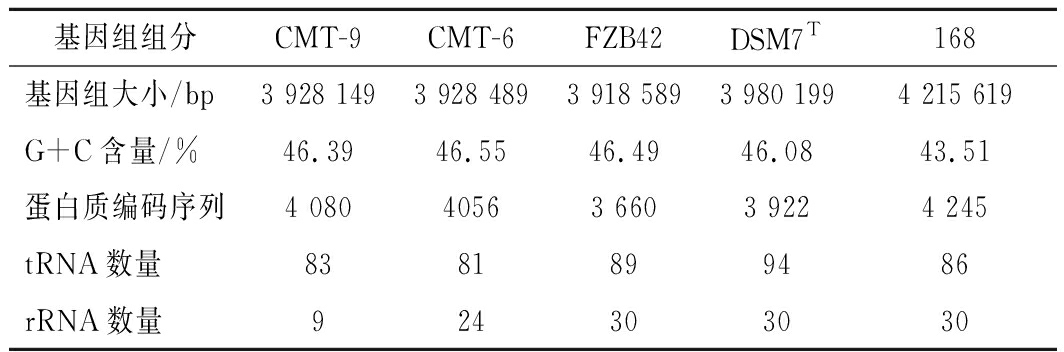
基因组组分CMT-9CMT-6FZB42DSM7T168基因组大小/bp3 928 1493 928 4893 918 5893 980 1994 215 619G+C含量/%46.3946.5546.4946.0843.51蛋白质编码序列4 08040563 6603 9224 245tRNA数量8381899486rRNA数量924303030
2个物种之间的共线性关系可以作为衡量物种之间进化距离的尺度,进而判断其亲缘关系[21]。由图6可知,CMT-9与CMT-6、FZB42均表现出高度的共线性;与DSM7T的共线性较差,在1~1.75 Mb片段出现较明显的缺失和易位;与168的共线性最差,存在显著的插入、易位、倒位和等基因重排现象,在1.3~1.4、1.85~1.95和2.65~2.8 Mb片段最为明显。
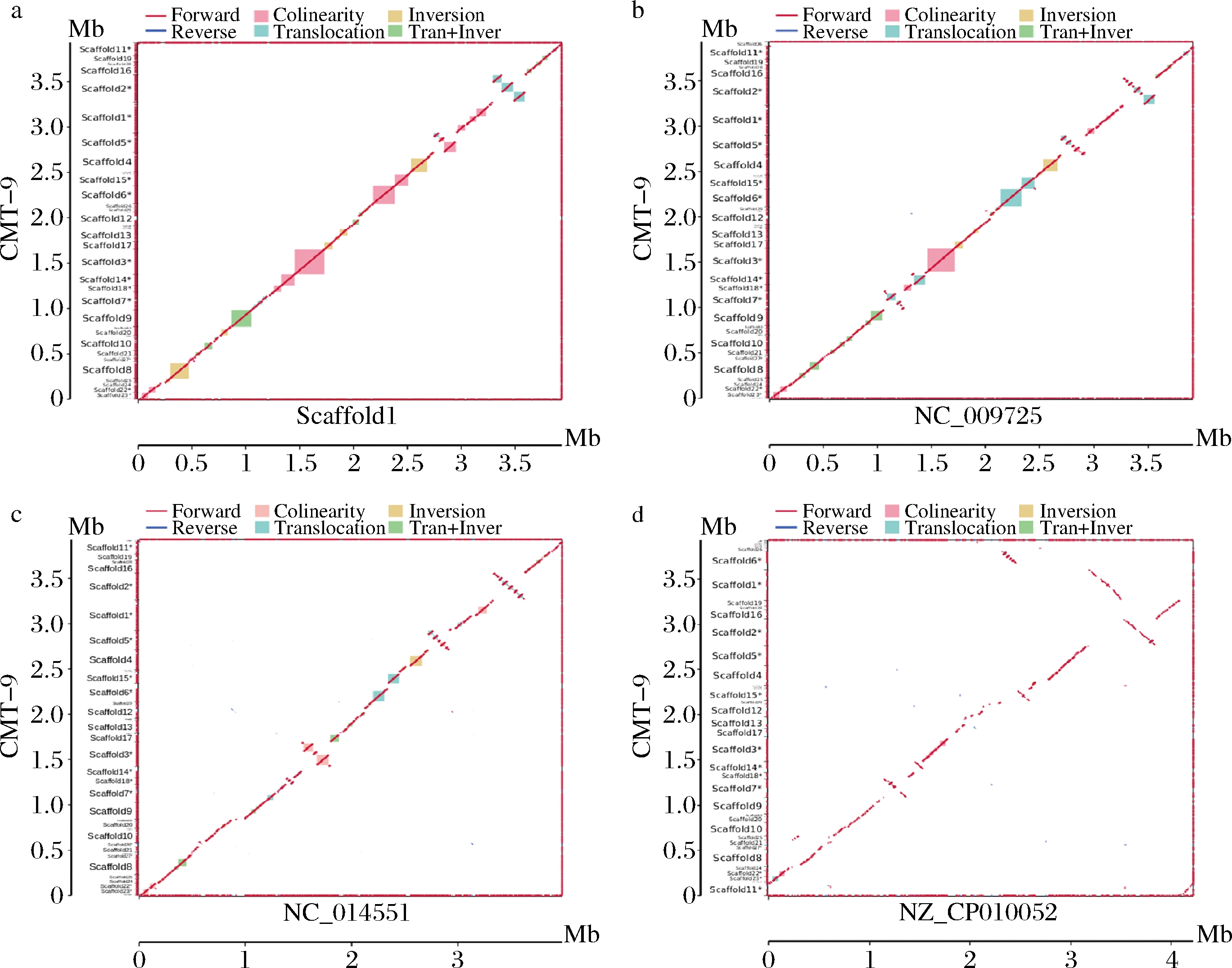
a-CMT-6;b-FZB42;c-DSM7T;d-168
图6 全基因组二维共线性比较
Fig.6 Two-dimensional collinearity comparison of genomes
将CMT-9的Reads比对到4株芽孢杆菌的基因组序列上,其中与CMT-6的覆盖率最高,覆盖区域总长度为3 724 736 bp,占CMT-6基因组的94.81%;其次是FZB42,覆盖区域总长度为3 918 589 bp,占FZB42基因组的90.68%;而CMT-9与DSM7T、168的覆盖率较低,覆盖区域总长度分别为2 036 571、104 859 bp,各占其基因组的51.17%和2.49%(表2)。
表2 测序长度和覆盖度比较
Table 2 Comparison of sequencing length and coincidence
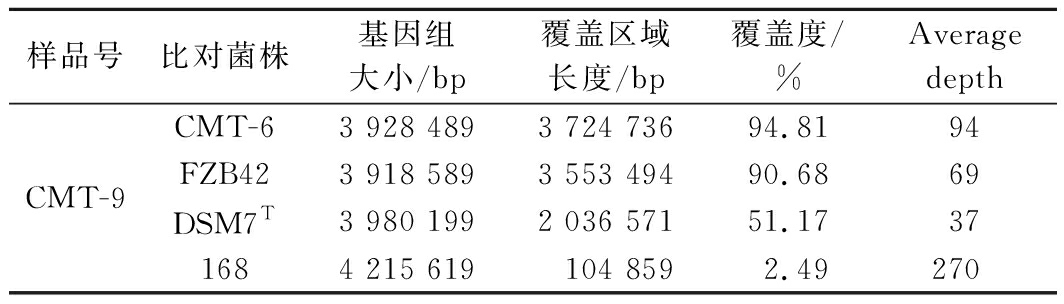
样品号比对菌株基因组大小/bp覆盖区域长度/bp覆盖度/%Average depthCMT-9CMT-63 928 4893 724 73694.8194FZB423 918 5893 553 49490.6869DSM7T3 980 1992 036 57151.17371684 215 619104 8592.49270
同时,将菌株CMT-9与CMT-6、168、DSM7T、FZB42基因组的蛋白序列进行比较,构建基因组间的Core-Pan基因集。将5株菌株的基因进行同源性分析,5株菌株的共有基因为2 675个,CMT-9与CMT-6的共有基因数量最多(3 545个),与FZB42、DSM7T、168的共有基因数量分别为3 166、3 090、2 882(图7)。
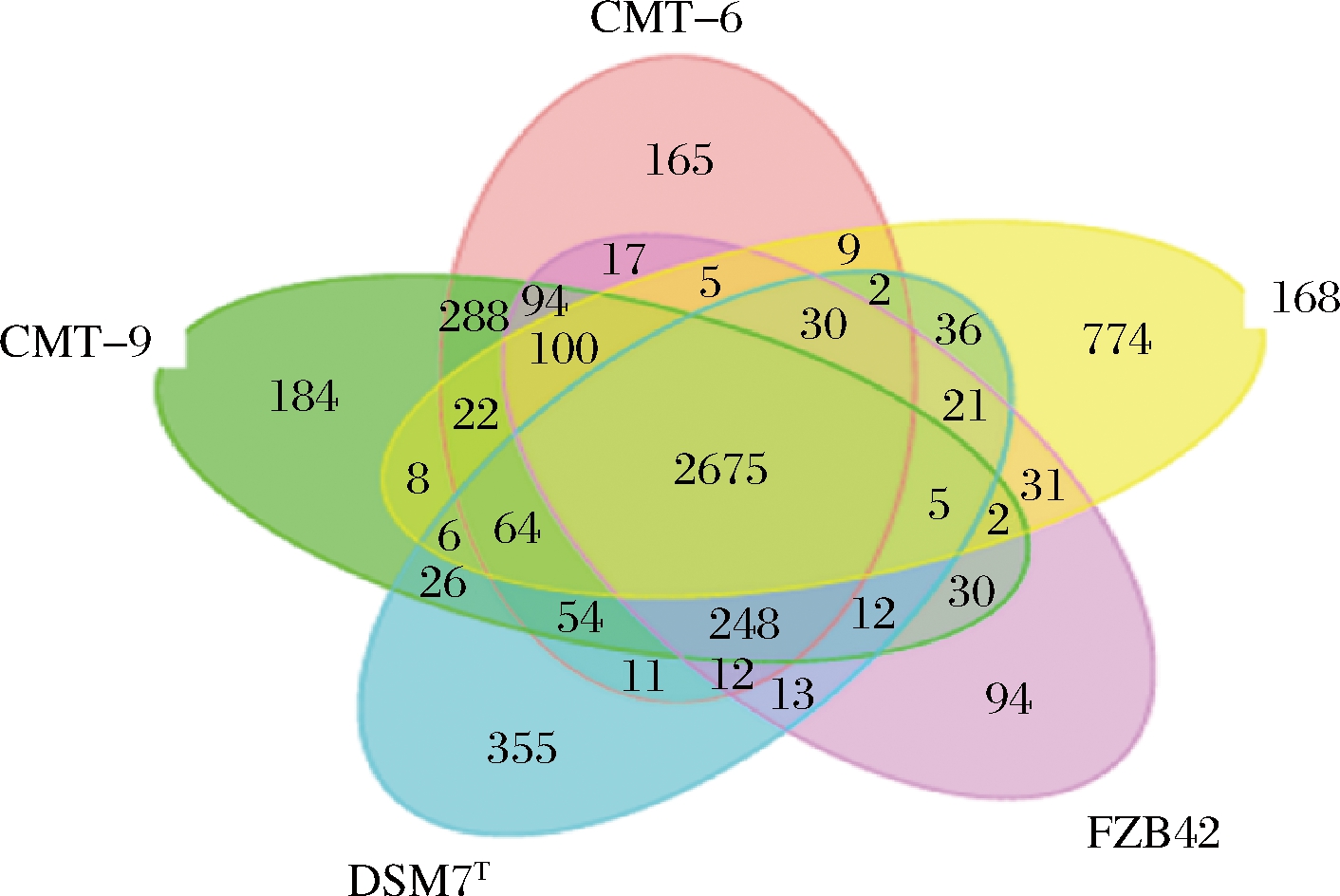
图7 基因组的Core-Pan基因数目维恩图
Fig.7 Venn diagram of the number of Core-Pan genes in the genome
本研究采用LC-MS/MS方法检测菌株CMT-9的发酵产物组分,发现其质荷比与张宁等[22]的研究中的surfactin和iturin相一致,说明CMT-9可以发酵产生抗菌脂肽。同时还检测到一个质荷比为1 432的组分,该值在fengycin质荷比(1 431~1 492)范围内,因此推测其可能为fengycin[23],但具体结构仍需进一步的探索和验证。
全基因组测序技术可从基因层面挖掘活性菌株次级代谢产物的合成潜力。本研究对菌株CMT-9进行全基因组测序和通路分析,发现其差异表达基因主要富集在碳水化合物代谢、氨基酸代谢通路。碳水化合物代谢过程中产生的丙酮酸会影响乙酰CoA的合成,乙酰CoA可进一步合成脂肪酸[24-25]。因此,这2条通路可为脂肽的合成提供脂肪酸和氨基酸等重要前体物质,前体物质的含量是菌株高效合成脂肽的关键。WU等[26]研究发现添加复合氨基酸可显著提高解淀粉芽胞杆菌Ba-BPD1的iturin产量。LU等[27]研究碳水化合物对解淀粉芽胞杆菌合成抗菌脂肽的影响,发现果糖通过提升氨基酸的浓度,进而增加抗菌脂肽相关合成酶基因的表达,最终提高抗菌脂肽的产量。因此,脂肽前体物质合成通路中基因的数量和表达水平与脂肽产量呈正相关性。
前期研究发现,菌株CMT-6具备合成surfactin、iturin和fengycin 3大类脂肽的合成酶基因,且数量较多,可合成高水平的抗菌脂肽[15]。根据已公布的CMT-6菌株的全基因组序列,通过比较基因组学发现CMT-9与CMT-6菌株的基因组大小、G+C含量、蛋白质编码序列和tRNA数量均表现高度的一致性。此外,在共线性分析中,发现菌株CMT-9与CMT-6具有高度共线性,与FZB42、DSM7T和168相比存在不同程度的差异,甚至出现线性缺失现象。同源性分析中CMT-9与CMT-6菌株的同源性较FZB42、DSM7T、168最高,达到94.81%。因此,芽孢杆菌基因组组分、共线性和同源性决定了其脂肽谱的相似度。
本研究采用LC-MS/MS技术发现CMT-9的发酵液中含有surfactin、iturin和fengycin 3种脂肽组分,进一步的全基因组测序和比较基因组学分析发现,前体物质合成基因数量与脂肽产量密切相关,基因组组分、共线性和同源性的差异是脂肽谱多样化的重要原因。研究结果为脂肽产生菌的筛选及脂肽定向合成工程菌株的改造提供理论依据。
[1] BENITEZ L B, VELHO R V, LISBOA M P, et al.Isolation and characterization of antifungal peptides produced by Bacillus amyloliquefaciens LBM5006[J].Journal of Microbiology (Seoul, Korea), 2010, 48(6):791-797.
[2] BEHARY N, PERWUELZ A, CAMPAGNE C, et al.Adsorption of surfactin produced from Bacillus subtilis using nonwoven PET (polyethylene terephthalate) fibrous membranes functionalized with chitosan[J].Colloids and Surfaces B:Biointerfaces, 2012, 90(1):137-143.
[3] 张可可, 席宇, 吴少雄, 等.甲基营养型芽孢杆菌的分离鉴定及其广谱抗菌性能初步研究[J].中国调味品, 2019, 44(10):31-34;45.
ZHANG K K, XI Y, WU S X, et al.Isolation and identification of Bacillus methylotrophicus and preliminary study on its broad-spectrum antibacterial activity[J].China Condiment, 2019, 44(10):31-34;45.
[4] 周涛, 满文曾, 吴晓营, 等.广谱抑菌性多粘类芽孢杆菌的筛选及其细菌素理化特性[J].食品工业科技, 2019, 40(24):99-103;109.
ZHOU T, MAN W Z, WU X Y, et al.Screening of broad-spectrum antibacterial Paenibacillus polymyxa and physicochemical properties of its bacteriocin[J].Science and Technology of Food Industry, 2019, 40(24):99-103;109.
[5] 陈燕萍, 陈梅春, 郑梅霞, 等.脂肽复合保鲜剂对龙眼的保鲜效果研究[J].福建农业科技, 2022, 53(1):1-5.
CHEN Y P, CHEN M C, ZHENG M X, et al.Study on the preservation effect of lipopeptide compound preservatives on longan[J].Fujian Agricultural Science and Technology, 2022, 53(1):1-5.
[6] 张楠楠. 产脂肽枯草芽孢杆菌的发酵优化以及在面包中的应用[D].无锡:江南大学, 2017.
ZHANG N N.Study on the production optimization of lipopepetide from Bacillus subtilis and application in bread[D].Wuxi:Jiangnan University, 2017.
[7] CHEN K, TIAN Z H, HE H, et al.Bacillus species as potential biocontrol agents against citrus diseases[J].Biological Control, 2020, 151:104419.
[8] PARK S C, KO C, HYEON H, et al.Imaging and targeted antibacterial therapy using chimeric antimicrobial peptide micelles[J].ACS Applied Materials & Interfaces, 2020, 12(49):54 306-54 315.
[9] 杨秀文, 邓旗, 陶森, 等.一株拮抗海鱼干优势耐盐真菌芽孢杆菌HY-5筛选及其抗菌成分鉴定[J].食品工业科技, 2018, 39(16):120-125.
YANG X W, DENG Q, TAO S, et al.Identification of antagonistic Bacillus HY-5 and its antimicrobial compositions from mangrove against specific salt resistance fungi of marine dried fish[J].Science and Technology of Food Industry, 2018, 39(16):120-125.
[10] 杨胜远, 韦锦, 李云, 等.一株产抗菌活性物质解淀粉芽孢杆菌的筛选及鉴定[J].食品科学, 2010, 31(21):208-212.
YANG S Y, WEI J, LI Y, et al.Screening and identification of a Bacillus amyloliquefaciens strain producing antimicrobial substances[J].Food Science, 2010, 31(21):208-212.
[11] 朱亚珠, 夏率博, 陈琳, 等.一株贝莱斯芽孢杆菌的生长特性及抑菌活性研究[J].食品科学技术学报, 2022, 40(1):85-92.
ZHU Y Z, XIA S B, CHEN L, et al.Study on growth characteristics and antimicrobial activities of Bacillus velezensis[J].Journal of Food Science and Technology, 2022, 40(1):85-92.
[12] 钱荣, 续晓琪, 许宗奇, 等.枯草芽孢杆菌KC-WQ发酵液中抗菌脂肽的分离鉴定及发酵条件优化[J].食品工业科技, 2022, 43(15):123-131.
QIAN R, XU X Q, XU Z Q, et al.Isolation and identification of antibacterial lpopeptides from Bacillus subtilis KC-WQ fermentation broth and optimization of fermentation conditions[J].Science and Technology of Food Industry, 2022, 43(15):123-131.
[13] ABDALLAH D B, TOUNSI S, GHARSALLAH H, et al.Lipopeptides from Bacillus amyloliquefaciens strain 32a as promising biocontrol compounds against the plant pathogen Agrobacterium tumefaciens[J].Environmental Science and Pollution Research International, 2018, 25(36):36 518-36 529.
[14] CHEN M C, WANG J P, ZHU Y J, et al.Antibacterial activity against Ralstonia solanacearum of the lipopeptides secreted from the Bacillus amyloliquefaciens strain FJAT—2349[J].Journal of Applied Microbiology, 2019, 126(5):1 519-1 529.
[15] DENG Q, WANG R D, SUN D F, et al.Complete genome of Bacillus velezensis CMT-6 and comparative genome analysis reveals lipopeptide diversity[J].Biochemical Genetics, 2020, 58(1):1-15.
[16] 李辉, 方志锴, 郭霞凌, 等.支链氨基酸代谢在糖肽类和环脂肽类抗生素产量提高与组分优化中的应用[J].中国抗生素杂志, 2022, 47(3):263-269.
LI H, FANG Z K, GUO X L, et al.The application of branched-chain amino acids metabolism regulation to increase the yields and optimizing components of glycopeptides and cyclopeptide fermentation[J].Chinese Journal of Antibiotics, 2022, 47(3):263-269.
[17] 王世伟, 王卿惠, 翟丽萍, 等.解淀粉芽胞杆菌抗真菌活性研究进展[J].中国微生态学杂志, 2020, 32(8):971-978;982.
WANG S W, WANG Q H, ZHAI L P, et al.Advances in research on antifungal activity of Bacillus amyloliquefaciens[J].Chinese Journal of Microecology, 2020, 32(8):971-978;982.
[18] DENG Q, WANG W J, SUN L J, et al.A sensitive method for simultaneous quantitative determination of surfactin and iturin by LC-MS/MS[J].Analytical and Bioanalytical Chemistry, 2017, 409(1):179-191.
[19] 邓旗, 花梅芳, 杨秀文, 等.基于PCR和TOF-MS海洋源产脂肽芽孢杆菌筛选[J].广东海洋大学学报, 2021, 41(1):72-80.
DENG Q, HUA M F, YANG X W, et al.Screening of Bacillus spp.producing lipopeptide from marine environment based on PCR and TOF-MS[J].Journal of Guangdong Ocean University, 2021, 41(1):72-80.
[20] 许俊杰, 蔡中华, 周进.海洋细菌CAZymes的研究进展[J].微生物学报, 2022, 62(4):1 286-1 307.
XU J J, CAI Z H, ZHOU J.Research progress of carbohydrate-active enzymes on marine bacteria[J].Acta Microbiologica Sinica, 2022, 62(4):1 286-1 307.
[21] 姜玉梅, 姜如云, 李俊畅, 等.小麦BES1基因家族的比较基因组学分析[J].麦类作物学报, 2021, 41(10):1 181-1 188.
JIANG Y M, JIANG R Y, LI J C, et al.Comparative genomics analysis of wheat BES1 gene family[J].Journal of Triticeae Crops, 2021, 41(10):1 181-1 188.
[22] 张宁, 孙力军, 王雅玲, 等.纳豆芽孢杆菌NT-6固态发酵罗非鱼下脚料条件优化及产脂肽组分分析[J].食品科学, 2016, 37(21):144-150.
ZHANG N, SUN L J, WANG Y L, et al.Component analysis of antimicrobial lipopeptides from solid-state fermentation of tilapia processing byproducts with Bacillus natto NT-6 and optimization of fermentation conditions[J].Food Science, 2016, 37(21):144-150.
[23] 刘宇帅, 张杰, 钟瑾, 等.解淀粉芽孢杆菌TF28抗菌脂肽芬芥素的分离鉴定及抑菌作用[J].中国生物工程杂志, 2018, 38(10):20-29.
LIU Y S, ZHANG J, ZHONG J, et al.Isolation and identification of antibacterial lipopeptides fengycin produced by Bacillus amyloliquefaciens TF28 and its anti-fungal mechanism studies[J].China Biotechnology, 2018, 38(10):20-29.
[24] 乐易林, 傅毓, 倪黎, 等.热稳定性丙酮酸:铁氧还蛋白氧化还原酶异源表达及其在乙酰辅酶A合成中的应用[J].中国生物工程杂志, 2020, 40(3):72-78.
LE Y L, FU Y, NI L, et al.Expression and characterization of a thermostable pyruvate ferredoxin oxidoreductase from the hyperthermophile thermotoga neapolitana and its application in acetyl-Co A production[J].China Biotechnology, 2020, 40(3):72-78.
[25] 于爽, 田海莹, 李生梅, 等.花生乙酰辅酶A羧化酶家族基因在不同胁迫下的响应模式分析[J].植物生理学报, 2021, 57(10):2 011-2 023.
YU S, TIAN H Y, LI S M, et al.Response expression patterns analysis to different stresses of acetylCo A carboxylase family genes in peanut[J].Plant Physiology Journal, 2021, 57(10):2 011-2 023.
[26] WU J Y, LIAO J H, SHIEH C J, et al.Kinetic analysis on precursors for iturin A production from Bacillus amyloliquefaciens BPD1[J].Journal of Bioscience and Bioengineering, 2018, 126(5):630-635.
[27] LU H, QIAN S, MUHAMMAD U, et al.Effect of fructose on promoting fengycin biosynthesis in Bacillus amylolique fmb-60[J].Journal of Applied Microbiology, 2016, 121(6):1 653-1 664.