肉中富含优质蛋白质、矿物质、脂肪和维生素(维生素B6、维生素B12和维生素D)等,此外,它含有维持基本生命活动所需的必需氨基酸。近年来,肉及肉制品掺伪掺假事件频繁发生,已对消费者的权益造成伤害,扰乱社会秩序,侵犯宗教信仰。随着部分商贩及企业掺假手段不断提高,使得肉制品的多样性和复杂性日益增加,传统鉴别技术已不能适应市场监管要求,快速、高通量、准确的动物源性成分检测方法,不仅能为政府部门监管工作提供技术支持,也为消费者权益提供技术保障。目前国内外已研发多种鉴别肉类掺假的检测方法,如基于形态学特征的感官鉴定技术、以光谱学为基础的化学鉴别法、以蛋白质为基础的免疫学方法及色谱质谱联用技术。
DNA是生物体组织和细胞内基因储存、复制和传递的重要物质基础,与蛋白质相比,其稳定性和种间多态性更强,利于物种鉴别。因此,以核酸分子为基础的生物学技术在物种识别中具有重要意义,已成为肉类种属鉴别最常用的方法,如普通PCR、数字PCR、多重PCR、实时荧光PCR、限制性片段长度多态性PCR,除此之外,研究人员还开发出环介导等温扩增、DNA条形码、高通量测序(next-generation sequencing, NGS)等快速、高通量的检测方法。目前,主要选择线粒体DNA和核基因组作为肉制品源性成分的鉴定基因,线粒体DNA是一种双链核外遗传物质,具有分子质量小、分子进化速度快、物种间差异大,种间稳定性好等特点,广泛应用于物种鉴定领域[1],常用的目标基因有cytb、ATP6、12S rRNA、D-loop区、16S rRNA 和ATP8[2]等。
1 PCR技术
PCR检测技术是目前国内肉类成分鉴定的标准方法,我国已陆续颁布一些国家标准、行业标准和地方标准,以荧光PCR技术为主,可用于绝大多数动物源性成分的鉴定,也能满足食品安全监管部门对检测技术简便、准确、高效的基本需求。
1.1 普通PCR定性检测技术
PCR是一种在生物体外进行的特殊DNA复制,其最显著的特征是能大量增加DNA的数量,并能根据不同物种的特定基因比对分析,设计适合的特异性引物,实现对物种种源的鉴别[3]。普通PCR是用一对特异性引物对目的片段扩增,是一种常规的肉制品鉴别技术。CALVO等[4]筛选猪的特异性引物,从香肠、馅饼、生熟肉、烟熏肉等猪肉制品中成功检测出了猪肉。普通PCR方法的应用为物种鉴别开辟了PCR技术的新时代,但普通PCR技术一次只能检出一个物种,不能满足高通量的检测需求。
多重PCR技术(multiplex PCR)又称复合PCR技术,是一种基于聚合酶链式反应的新PCR技术,加入的两对或以上引物在同一反应体系中扩增不同模板,获得不同的目标片段。如表1列举多重PCR技术用于牛、羊、鸡、鸭等成分的检测,检出限达0.01 ng/μL,最高可同时检测6种动物源性成分,多重PCR法不仅特异性强、灵敏度高,同时又减少试剂用量,提高检测效率,节约检测费用,被广泛用于动物源性成分的检测,但该技术引物设计是关键,数量过多可能引起交叉反应。
表1 多重PCR检测技术
Table 1 Multiplex PCR detection technology

物种靶基因名称最低检出限参考文献牛、羊、鸡、猪、鸭、驴cytb0.1 ng/μL[5]大鼠、狐狸、鸭、羊16S rRNA、ATPase 6、COX2、tRNA及ND1、xytb0.05 ng/μL[6]牛、猪、鸡、马cytb10×10-3ng/μL[7]鸡、猪、牛、山羊、马、鸵鸟cytb,COI和12S rRNA12 500线粒体拷贝[8]牛、羊、猪、鸡、鸭cytb25 pg gDNA[9]牛、鸡、驴ATPase 6、12S rRNA、ND20.01 ng/μL[10]
1.2 限制性片段长度多态性PCR技术
限制性片段长度多态性PCR(PCR-restriction fragment length polymorphism,PCR-RFLP)将RFLP与PCR两项技术相结合,经同一限制性内切酶对扩增产物进行酶切,获得不同长度的酶切片段,用琼脂糖凝胶电泳法或毛细管电泳法分析酶切片段,从而鉴别样品中的动物源性成分。已有大量研究将该技术应用于肉制品的鉴别中,如ISLAM等[11]基于PCR-RFLP法,采用4种限制性内切酶分别对64个肉类样品的12S rRNA基因进行分析,结果表明,所有酶剪切位点在这些样品中均是保守的,该技术可用于肉类的检测与鉴别。同时UDDIN等[12]建立了牛、水牛、鸡、鸭、山羊、绵羊和猪的七重PCR-RFLP检测方法,PCR产物经FatⅠ、BfaⅠ、HPY 188I酶切鉴定,得到混合肉类的检出限为0.5%(质量分数),该方法的灵敏度高,稳定性好,利于生鲜肉及加工肉制品中目标物种的鉴别。RFLP方法虽然有很好的检测效果,但由于内切酶位点随机变异,使实验结果的重现性较差,因此设计合适的酶切位点至关重要。
1.3 实时荧光PCR相对定量技术
实时荧光PCR是一种以PCR为基础的检测技术,指将荧光基团加入反应体系中,通过连续监测PCR扩增过程中荧光信号的强度来实时监测特定产物的量,并利用标准曲线对其进行定量检测的过程,可分为荧光探针法和荧光染料法,如图1所示,此技术已广泛应用于肉类掺假鉴定[14-15]。基于实时荧光PCR技术的定量分析策略主要通过构建标准质粒、引入校正系数等方案实现动物源成分的准确定量。徐琼等[16]基于Taq Man实时荧光PCR技术,将朊蛋白基因(PRNP)作为猪特异性基因,使用ΔCt法进行相对定量,猪源性的检出限为0.25%(质量分数),所配制的猪肉含量为31%、52%、59%和79%的仿冒羊肉样品,其检测值与真值差异较小,表明此法能够精准、稳定地检测出在羊肉中所掺的猪肉含量。ZHU等[17]建立了常规多重PCR和标准化实时PCR同时检测5种常见肉类及定量检测食品中驴肉含量的方法,能快速准确区分驴、羊、牛、猪和马5种常见的掺假品种。IWOBI等[18]利用肌生长抑制素的通用序列和双重量化性质,建立一种定量检测肉糜中牛肉和猪肉组分的三重实时荧光PCR方法,其检测限为20个基因组当量,能为肉糜的量化提供可靠依据。与数字PCR相比,实时荧光PCR技术具有成本低、仪器平台广泛等优势,但也存在一些不足之处,定量过程需要构建标准曲线,但无法获得定量准确的标准品。其次,以Ct值作为中间系数,无法准确计算用于定量的靶基因拷贝数与样品质量分数之间的对应关系,因此一定程度阻碍了该方法在肉类定量检测中的应用[19]。

图1 实时荧光PCR 技术原理图[13]
Fig.1 Real-Time PCR technology schematic diagram
1.4 数字PCR绝对定量技术
微滴数字PCR技术(droplet digital PCR, ddPCR)是一种核酸定量检测方法,通过微滴生成仪把反应体系分散到不同的反应单元中,独立进行PCR,并依据阳性比例和泊松分布计算出DNA起始浓度,能实现核酸的绝对定量[20],如图2所示。肉制品定量检测的关键是实现DNA拷贝数与样品质量分数的转换,吴海晶等[21]基于微滴式数字PCR建立羊肉中鸭肉组分与拷贝数之间的线性关系,通过测定混合样品中羊肉与鸭肉的DNA拷贝数,利用结合建立的标准曲线计算出羊肉与鸭肉的质量比,结果表明,该方法准确度高,经多重验证,与标准样品的绝对误差为0.35%;REN等[22]采用微滴数字PCR定量测定绵羊和山羊肉产品中鸡肉的含量,并在检验过程中引入校正系数k,将动物源成分质量比例转换为拷贝数的比例,研究结果表明,原料肉的不同部位对所建立方法的定量准确性无影响,且检测经高温高压处理的样品时,该方法表现出良好的重复性和稳定性。与传统PCR、荧光定量PCR相比,数字PCR技术准确度和灵敏度更高,不依赖扩增阈值(Ct值),无需考虑不同样品的引物扩增效率差异造成定量检测不准确,人为操作因素少,对模板浓度进行精准定量,但实际检测时,不同来源的肉制品中核基因组DNA或线粒体DNA拷贝数及丰度存在差异,线粒体DNA丰度高且拷贝数大,易造成定量不准确,而核基因组DNA拷贝数恒定,不易受到不同组织细胞的影响,更适用于动物源成分的定量检测。
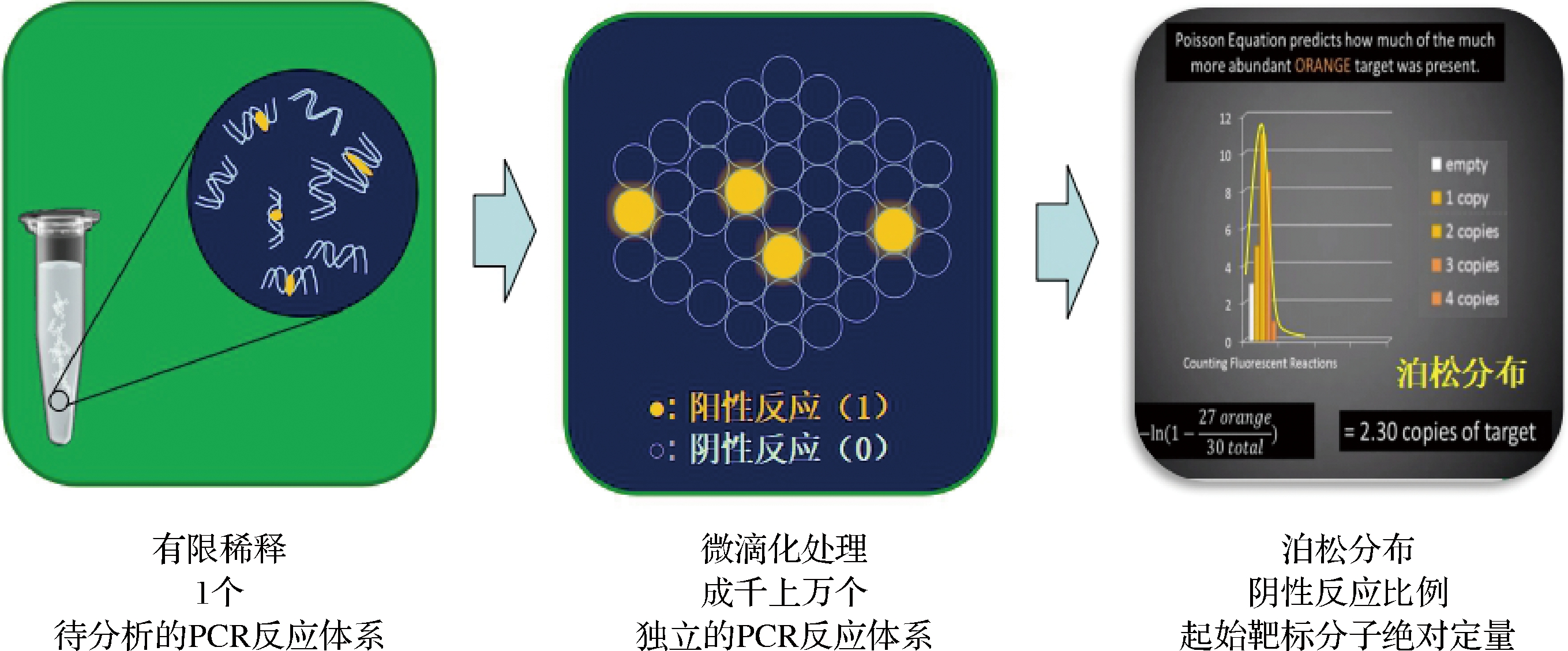
图2 微滴数字PCR技术原理图
Fig.2 Droplet digital PCR technology schematic diagram
2 快速检测技术
随着全球对肉类鉴别技术越来越高的要求,开发准确高效的现场检测技术是监管部门执法的关键,环介导等温扩增技术(loop-mediated isothermal amplification,LAMP)是一种新型核酸体外扩增技术,基本原理是DNA在4条特异性引物(内外引物各2条)及链置换DNA聚合酶作用下可不断合成链置换DNA,具有可以在恒定温度下(65 ℃左右)快速高效地扩增目的片段的优势,由于该方法无DNA变性步骤,因此摆脱了复杂的温度控制装备及精密仪器的限制,能满足现场检测及执法的需求,耗时较少,结果肉眼可判别,应用于肉类快速检测具有较好的前景。CAI等[23]研发了一种基于实时LAMP的测定法,此方法可在20 min内检测出肉制品中猪源成分,检测限达1.76 pg/μL,比PCR灵敏度高1 000倍,这是首次应用LAMP法检测商业产品中的猪源成分,其实际应用将有助于打击食品工业的掺假行为;ZAHRADNIK等[24]采用LAMP法检测加工食品中马肉含量,以马的线粒体基因组设计了特定扩增引物,引物特异性好,未观察到牛肉、猪肉和鸡肉的交叉反应,方法灵敏度高,能从制备的模型香肠中检测到0.1%的马肉。于媛媛等[25]以猪、鸡、鸭线粒体DNA为靶基因设计特异性引物,对3种源性成分的模拟深加工样品检测分析,分析时间为36 min,可实现生鲜肉、加工肉制品肉源成分的快速筛查。目前国内外已有大量研究者将LAMP应用于肉类成分鉴别中。
3 新型分子检测技术
目前,我国相关标准涉及的方法主要是针对已知目标物种,而在实际检测中,对于更多未知成分的筛查,传统方法不适用,为满足这一需求,有研究者开发了非目标成分检测的DNA测序技术,该技术可以通过一次测序得到混合样品尤其是未知样品中的全部目标片段序列,随后与基因数据库进行比对,从而确定物种种类。现存在许多包含核酸序列信息的动物DNA数据库,有学者实验室建立了包含2 700多个种的动物数据库[26],即使没有参考样本或参考基因序列,通过数据库序列信息比对也能确定动物源性。该技术准确、快速、通量高,在肉类掺假检测中具有较好的发展潜力。
3.1 DNA条形码技术
DNA条形码(DNA barcode)技术是由HEBERT等[27]提出并在物种鉴别中开始应用的,其工作原理是使用标准的或已知的基因组DNA基因片段作为分子靶标,在种级水平进行物种鉴定,与超市里识别商品的条形码相似,DNA barcode主要编码线粒体COI基因,线粒体COI基因已被证明可作为动物公认条形码[28],DNA条形码技术能够实现对多种未知掺杂成分的快速、高通量的检测,真正突破了常规分子方法定向测定的局限,它有不可替代的优越性,是一个通用、非定向靶标的检测方法。而国内关于牛、羊、猪、鸭等肉类DNA条形码的研究仍处于初级阶段[29]。AHMED等[30]以印度不同烹饪方式加工的肉制品为研究对象,测试DNA条形码的适用性,研究显示,经烹饪加工的肉制品虽经复杂处理及含有大量香料,但对应用DNA条形码技术进行物种鉴定无影响,表明其可作为一种可靠的动物源性追溯方法;田晨曦等[31]采用COI-2引物进行PCR扩增及序列比对,建立以COI基因为基础的肉制品掺假鉴别方法,对牛-猪、羊-猪、牛-鸭和羊-鸭模型掺假物的检出限分别为5%、8%、1%和4%,方法简便快速有效;励炯等[32]基于COI序列建立并优化可鉴别11种生鲜肉的DNA条形码技术,研究以低价值肉(鸡、鸭、马等)掺入高价值肉类中(牛、羊、兔等)制备18种掺假模型,结果显示,在18种掺假模型中,牛羊肉与低价值肉掺混的最低检出率为5%,该方法灵敏度高,可作为高低价值肉类掺杂的有效鉴别方法。目前国际上对于DNA条形码技术在鉴定杂交物种种属信息方面存在争议,但可通过联合其他分子检测方法来解决,例如,在某种程度上,采用复合条形码技术、微型条形码技术解决了含有多个未知物种的混合样品的鉴定问题[33]。
3.2 高通量测序技术
21世纪NGS的出现使动物源性成分检测取得了新突破。NGS可分为靶向测序、全基因组测序和转录组测序,NGS可以通过聚合酶或连接酶直接体外合成和测序同时进行,具有高通量、快速、可定量、低成本和高分辨率等优点。一次高通量测序可以读取400 000~4 000 000条序列,不同的测序平台可读取到的碱基数不等[34],目前NGS的主要代表平台有Illumina公司的Solexa基因组分析仪和ABI的SOLiD测序仪,不同平台的核心技术有所区别,图3为BGISEQ原理图。Solexa聚合酶合成测序的关键技术是:“DNA簇”和“可逆性末端终结”[36];SOLiD连接酶测序取代了传统PCR,可对单拷贝DNA片段进行大规模扩增和高通量测序[37]。

图3 高通量测序技术原理图[35]
Fig.3 Next-generation sequencing technology schematic diagram
该项技术已广泛应用于环境中微生物多样性分析[38]、病毒鉴别[39]、临床诊断[40]等。蔡一村等[41]利用二代测序和DNA条形码技术对哺乳动物、禽类及其他肉类混合样品进行鉴定,通过扩增线粒体基因16S rRNA区,使用Illumina Miseq二代测序技术获取所有序列信息,并与GenBank数据库进行比对,结果显示,混合样品中鹅、鸭成分被正确识别,且检测下限为0.5 ng/μL,该方法准确度高,用途广泛,实现了混合DNA片段序列的高通量测定;PAN等[42]提出一种能准确识别加工肉制品中猪、牛、家禽等物种的NGS结合DNA微条形码的方法,基于51种动物的140条序列设计引物,鉴定包含12个物种的鲜肉混合物,经测序、序列比对分析显示,12个物种序列与目标序列完全一致,说明此方法检测效率较高。ZHANG等[43]采用NGS结合包含动物2 354个完整的线粒体基因组序列数据库,对不同含量的猪、牛、羊、鸡、兔和小鼠成分的模拟样品鉴定研究,结果表明该方法能够检测肉类中1%的动物源成分,方法的准确率达97.65%,对肉制品中混合动物源性成分或未知成分的识别具有良好的应用前景。NGS具有快捷、成本低、通量高等特点,能一次实现多种动物源成分的检测,将为食品安全执法监管、掺杂掺假、清真食品的检测提供新思路。
4 总结与展望
近年来,屡次出现的肉制品掺假事件存在较大的食品安全风险,严重影响消费者,破坏社会经济秩序,研究快速、准确、高效的动物源性成分检测方法,对保障国家食品安全、社会稳定、人民健康至关重要。目前国内外学者开展的基于分子生物学技术已广泛应用于肉制品的检测,不同技术各有优缺点,详见表2,实验人员可根据具体条件选择适当的的方法。未来,在现有检测技术基础上,开发低成本的快检试剂盒、丰富动物基因组序列数据库、细化杂交物种的DNA条形码、优化数据分析方法、采用多核苷酸多态性标记等是研究的趋势,同时,NGS一次可检测几百份样品,具有通量高、快速、准确性好等优势,为动物源性成分鉴定的发展提供了新方向,必将在大批量、未知掺假成分的肉制品检测中发挥重要作用。
表2 检测技术应用场景及特点比较
Table 2 Comparison of application scenarios and characteristics of detection technologies

注:*表示程度,*越多表明准确性更好,灵敏度更高
检测技术准确性灵敏度特点应用文献来源普通PCR**操作简便,但易因操作污染而出现假阳性鲜肉、热加工肉制品[44]实时荧光PCR****可实现相对定量,但需专门的PCR实验室鲜肉组织、香肠、风干肉制品、卤肉[45]数字PCR******能分析复杂混合物,但成本高,对食品检验人员技术要求较高鲜肉、经深加工的肉制品,如香肠、鸡粉[46]LAMP****快速、简便,结果可视化,无需昂贵的仪器设备鲜肉、香肠、烟熏肉[47]DNA条形码******非靶标鉴定,但鉴定周期长,基因参考数据库不够完善香肠、肉丸等肉糜制品,罐头等高度加工肉制品[48]NGS******通量高、快速、无靶标鉴定肉丸、肉肠等肉糜制品、酱卤肉、熏烤肉、罐头制品等经高温高压处理的肉制品[49]
[1] 史莹莹. 基于核酸分子水平鉴定肉及肉制品中动物种源成分[D].扬州:扬州大学, 2020.
SHI Y Y.Identification of animal provenance components in meat and products based on nucleic acid molecular level[D].Yangzhou:Yangzhou University, 2020.
[2] CAHYADI M, WIBOWO T, PRAMONO A, et al.A novel multiplex-PCR assay to detect three non-halal meats contained in meatball using mitochondrial 12S rRNA gene[J].Food Science of Animal Resources, 2020,40(4):628-635.
[3] 邵博宇, 徐宁, 迟凯月, 等.基于多重PCR定性鉴别鹿肉掺假[J].食品与发酵工业, 2022,48(24):282-288.
SHAO B Y, XU N, CHI K Y, et al.Qualitative identification of adulterated venison based on multiplex PCR[J].Food and Fermentation Industries, 2022,48(24):282-288.
[4] CALVO J H, OSTA R, ZARAGOZA P.Quantitative PCR detection of pork in raw and heated ground beef and paté[J].Journal of Agricultural and Food Chemistry, 2002,50(19):5 265-5 267.
[5] 赵志勇, 朱少华, 赵晓燕, 等.基于多重PCR技术同时检测肉制品中6种动物源性成分[J].上海农业学报, 2021,37(4):117-124.
ZHAO Z Y, ZHU S H, ZHAO X Y, et al.Simultaneous determination of six animal-derived ingredients in meat product by multiple PCR method[J].Acta Agriculturae Shanghai, 2021,37(4):117-124.
[6] LIU W W, WANG X N, TAO J, et al.A multiplex PCR assay mediated by universal primers for the detection of adulterated meat in mutton[J].Journal of Food Protection, 2019,82(2):325-330.
[7] 袁淑辉, 蔡军, 王忠才, 等.4种肉类成分多重PCR的鉴定方法[J].中国兽医杂志, 2020,56(11):65-69.
YUAN S H, CAI J, WANG Z C, et al.A multiplex PCR method for the identification of four meat ingredients[J].Chinese Journal of Veterinary Medicine, 2020,56(11):65-69.
[8] KITPIPIT T, SITTICHAN K, THANAKIATKRAI P.Direct-multiplex PCR assay for meat species identification in food products[J].Food Chemistry, 2014,163:77-82.
[9] 王金斌, 白蓝, 李文, 等.同步检测动物源性成分的五重PCR的条件优化和检出限分析[J].核农学报, 2018,32(3):506-514.
WANG J B, BAI L, LI W, et al.Optimization of five-plx PCR method for the identification of five animal species and analysis of its detection limit[J].Journal of Nuclear Agricultural Sciences, 2018,32(3):506-514.
[10] GALAL-KHALLAF A.Multiplex PCR and 12S rRNA gene sequencing for detection of meat adulteration:A case study in the Egyptian markets[J].Gene, 2021,764:145062.
[11] ISLAM A, HALDER J, RAHMAN A M, et al.Meat origin differentiation by polymerase chain reaction-restriction fragment length polymorphism[J].International Journal of Food Properties, 2021,24(1):1 022-1 033.
[12] UDDIN S M K, HOSSAIN M A M, CHOWDHURY Z Z, et al.Detection and discrimination of seven highly consumed meat species simultaneously in food products using heptaplex PCR-RFLP assay[J].Journal of Food Composition and Analysis, 2021,100:103938.
[13] 王玉倩, 薛秀花.实时荧光定量PCR技术研究进展及其应用[J].生物学通报, 2016,51(2):1-6.
WANG Y Q, XUE X H.Research progress and application of real-time quantitative PCR technology[J].Bulletin of Biology, 2016,51(2):1-6.
[14] CHEN X Y, LU L X, XIONG X H, et al.Development of a real-time PCR assay for the identification and quantification of bovine ingredient in processed meat products[J].Scientific Reports, 2020,10(1):2052.
[15] 胡悦, 刘艳艳, 任金瑞, 等.牛、羊肉中水貂、猪、鼠混杂成分的多重荧光PCR鉴定方法的建立[J].农业生物技术学报, 2018,26(9):1 621-1 630.
HU Y, LIU Y Y, REN J R, et al.Establishment of multiplex real-time fluorescence PCR for detection of mink (Mustela lutreola), pig (Sus scrofa) and rat (Mus musculus) derived components in beef and mutton[J].Journal of Agricultural Biotechnology, 2018,26(9):1 621-1 630.
[16] 徐琼, 张奕南, 顾文佳, 等.TaqMan实时荧光PCR法定量检测生肉中猪源性成分的建立[J].食品科技, 2016,41(2):309-313.
XU Q, ZHANG Y N, GU W J, et al.Establishment of quantitative Taq Man real-time PCR targeting the swine-derived in raw meat[J].Food Science and Technology, 2016,41(2):309-313.
[17] ZHU T Y, ZHOU X F, ZHANG W, et al.Multiplex and real-time PCR for qualitative and quantitative donkey meat adulteration[J].Journal of Food Measurement and Characterization, 2021,15(2):1 161-1 168.
[18] IWOBI A, SEBAH D, KRAEMER I, et al.A multiplex real-time PCR method for the quantification of beef and pork fractions in minced meat[J].Food Chemistry, 2015,169:305-313.
[19] 马炳存, 崔学文, 李琰歆, 等.基于实时荧光聚合酶链式反应技术定量检测肉制品中动物源性成分研究进展[J].肉类研究, 2021,35(7):44-49.
MA B C, CUI X W, LI Y X, et al.A review of the application of real-time polymerase chain reaction for quantitative detection of animal-derived ingredients in meat products[J].Meat Research, 2021,35(7):44-49.
[20] KÖPPEL R, GUERTLER P, WAIBLINGER H U.Duplex droplet digital PCR (ddPCR) method for the quantification of common wheat (Triticum aestivum) in spelt (Triticum spelta)[J].Food Control, 2021,130:108382.
[21] 吴海晶, 冯秋实, 刘延, 等.基于微滴式数字PCR的羊肉中鸭源性成分定量检测[J].食品科技, 2021,46(12):288-293.
WU H J, FENG Q S, LIU Y, et al.A quantitative assay for measuring duck adulterated into mutton products by droplet digital PCR[J].Food Science and Technology, 2021,46(12):288-293.
[22] REN J N, DENG T T, HUANG W S, et al.A digital PCR method for identifying and quantifying adulteration of meat species in raw and processed food[J].PLoS One, 2017,12(3):e0173567.
[23] CAI S X, KONG F D, XU S F.Detection of porcine-derived ingredients from adulterated meat based on real-time loop-mediated isothermal amplification[J].Molecular and Cellular Probes, 2020,53:101609.
[24] ZAHRADNIK C, MARTZY R, MACH R L, et al.Loop-mediated isothermal amplification (LAMP) for the detection of horse meat in meat and processed meat products[J].Food Analytical Methods, 2015,8(6):1 576-1 581.
[25] 于媛媛, 曲勤凤, 张奕南.环介导等温扩增法快速检测牛羊肉中的掺杂肉[J].食品工业, 2019,40(3):325-330.
YU Y Y, QU Q F, ZHANG Y N.Meat species authenticity rapid identification by loop-mediated isothermal amplification assay in beef and mutton[J].The Food Industry, 2019,40(3):325-330.
[26] 甘永琦, 卢曼曼, 赖青鸟, 等.高通量测序技术在肉类掺假检测中的应用进展[J].生物工程学报, 2022,38(2):411-426.
GAN Y Q, LU M M, LAI Q N, et al.Application and progress in high-throughput sequencing technology for meat adulteration detection[J].Chinese Journal of Biotechnology, 2022,38(2):411-426.
[27] HEBERT P D N, CYWINSKA A, BALL S L, et al.Biological identifications through DNA barcodes[J].Proceedings Biological Sciences, 2003,270(1 512):313-321.
[28] NEHAL N, CHOUDHARY B, NAGPURE A, et al.DNA barcoding:A modern age tool for detection of adulteration in food[J].Critical Reviews in Biotechnology, 2021,41(5):767-791.
[29] 邱德义, 胡佳, 刘德星, 等.DNA条形码技术在肉品防欺诈鉴别中的应用[J].肉类研究, 2013,27(4):40-43.
QIU D Y, HU J, LIU D X, et al.Application of DNA barcoding in anti-fraud identification of aquatic products[J].Meat Research, 2013,27(4):40-43.
[30] AHMED N, SANGALE D, TIKNAIK A, et al.Authentication of origin of meat species processed under various Indian culinary procedures using DNA barcoding[J].Food Control, 2018,90:259-265.
[31] 田晨曦, 周巍, 王爽, 等.基于DNA条形码技术常见肉类掺假鉴别技术的研究[J].现代食品科技, 2016,32(8):295-301.
TIAN C X, ZHOU W, WANG S, et al.Techniques for identifying common meat adulterations based on DNA barcoding[J].Modern Food Science and Technology, 2016,32(8):295-301.
[32] 励炯, 吴琼, 扈明洁, 等.基于细胞色素C氧化酶亚基Ⅰ序列的DNA微条形码技术鉴别11种生鲜肉制品掺假的研究[J].浙江大学学报(农业与生命科学版), 2021,47(1):52-59.
LI J, WU Q, HU M J, et al.Identification of adulteration in 11 fresh meat products by DNA mini-barcoding methods based on cytochrome C oxidase subunit I (CO I) sequence[J].Journal of Zhejiang University(Agriculture and Life Sciences), 2021,47(1):52-59.
[33] SCALES Z M, NARBAY E, HELLBERG R S.Use of DNA barcoding combined with PCR-SFLP to authenticate species in bison meat products[J].Foods (Basel, Switzerland), 2021,10(2):347.
[34] 李妍, 徐兴祥.高通量测序技术的研究进展[J].中国医学工程, 2019,27(3):32-37.
LI Y, XU X X.Research progress of high-throughput sequencing technology[J].China Medical Engineering, 2019,27(3):32-37.
[35] 华大基因. 高通量测序原理及其发展简介[DB/OL].2022-03-24.https://www.bilibili.com/read/cv6521433.
The Beijing Genomics Institute.An introduction to the principles of high-throughput sequencing and its development[DB/OL].2022-03-24.https://www.bilibili.com/read/cv6521433.
[36] LIU L, LI Y H, LI S L, et al.Comparison of next-generation sequencing systems[J].Journal of Biomedicine and Biotechnology, 2012,2012:251364.
[37] MARDIS E R.The impact of next-generation sequencing technology on genetics[J].Trends in Genetics, 2008,24(3):133-141.
[38] WANG Z S, ZHU Y Q, LI N, et al.High-throughput sequencing-based analysis of the composition and diversity of endophytic bacterial community in seeds of saline-alkali tolerant rice[J].Microbiological Research, 2021,250:126794.
[39] GAAFAR Y Z A, ZIEBELL H.Comparative study on three viral enrichment approaches based on RNA extraction for plant virus/viroid detection using high-throughput sequencing[J].PLoS One, 2020,15(8):e0237951.
[40] DHINGRA A, GÖTTING J, VARANASI P R, et al.Human cytomegalovirus multiple-strain infections and viral population diversity in haematopoietic stem cell transplant recipients analysed by high-throughput sequencing[J].Medical Microbiology and Immunology, 2021,210(5-6):291-304.
[41] 蔡一村, 林颖峥, 刘群秀, 等.高通量二代测序基因条形码技术在动物源性制品物种鉴定中的应用[J].现代食品科技, 2020,36(3):252-259.
CAI Y C, LIN Y Z, LIU Q X, et al.Application of high-throughput sequencing and gene barcode technology in species identification of animal-derived products[J].Modern Food Science and Technology, 2020,36(3):252-259.
[42] PAN Y Y, QIU D Y, CHEN J, et al.Combining a COI mini-barcode with next-generation sequencing for animal origin ingredients identification in processed meat product[J].Journal of Food Quality, 2020,2020:1-9.
[43] ZHANG Y N, QU Q F, RAO M Z, et al.Simultaneous identification of animal-derived components in meats using high-throughput sequencing in combination with a custom-built mitochondrial genome database[J].Scientific Reports, 2020,10:8965.
[44] AFIFA KHATUN M, HOSSAIN A, HOSSAIN M S, et al.Detection of species adulteration in meat products and Mozzarella-type cheeses using duplex PCR of mitochondrial cyt b gene:A food safety concern in Bangladesh[J].Food Chemistry:Molecular Sciences, 2021,2:100017.
[45] WU Q Q, XIANG S N, WANG W J, et al.Species identification of fox-, mink-, dog-, and rabbit-derived ingredients by multiplex PCR and real-time PCR assay[J].Applied Biochemistry and Biotechnology, 2018,185(1):1-12.
[46] TEMISAK S, THANGSUNAN P, BOONNIL J, et al.Accurate determination of meat mass fractions using DNA measurements for quantifying meat adulteration by digital PCR[J].International Journal of Food Science & Technology, 2021,56(12):6 345-6 358.
[47] THANGSUNAN P, TEMISAK S, JAIMALAI T, et al.Sensitive detection of chicken meat in commercial processed food products based on one-step colourimetric loop-mediated isothermal amplification[J].Food Analytical Methods, 2022, 15(5):1 341-1 355.
[48] BÖHME K, CALO-MATA P, BARROS-VEL ZQUEZ J, et al.Review of recent DNA-based methods for main food-authentication topics[J].Journal of Agricaltural and Food Chemistry, 2019,67(14):3 854-3 864.
ZQUEZ J, et al.Review of recent DNA-based methods for main food-authentication topics[J].Journal of Agricaltural and Food Chemistry, 2019,67(14):3 854-3 864.
[49] COTTENET G, BLANCPAIN C, CHUAH P F, et al.Evaluation and application of a next generation sequencing approach for meat species identification[J].Food Control, 2020,110:107003.