禽肉加工业的快速发展产生了大量角蛋白质废弃物,如羽毛、爪、喙、蹄、角等,该类蛋白具有大量二硫键及交联性结构,在自然环境中很难被降解[1]。角蛋白的粗蛋白含量超过85%,是一个巨大的可再生蛋白资源库。然而,目前仍然缺乏有效的回收方式。传统的处理方法如高压水解、微波消解、酸水解、碱水解,存在反应条件苛刻、二次污染、产物低营养等问题[2]。角蛋白酶(keratinase)是一种能够特异性降解角蛋白的蛋白酶,可水解角蛋白产生可溶性蛋白、氨基酸和多肽等营养物质。角蛋白酶的来源广泛,其中来自芽孢杆菌属(Bacillus sp.)的细菌为主要的生产菌株[3]。近年来,已有许多微生物发酵法与酶法处理羽毛及羊毛等的相关研究报道[4-9],但降解效果难以满足大规模应用的要求。特别是在酶法处理羽毛的过程中,较低的角蛋白酶浓度显著限制了羽毛降解能力。因此提高微生物角蛋白酶分泌能力是提升角蛋白酶应用性的关键问题之一。
通过信号肽筛选增强分泌能力是提升异源蛋白胞外表达量的常用策略。WATANABE等[10]通过对谷氨酸棒杆菌Corynebacterium glutamicum R全基因组系统筛选,得到405条信号肽序列,其中11条信号肽能够表达嗜热脂肪地热芽孢杆菌Geobacillus stearothermophilus来源的α-淀粉酶,表达量相比常见的棒状细菌分泌蛋白PS2增加了50~150倍不等;GUO等[11]在解淀粉芽孢杆Bacillus amyloliquefaciens 111018中以增强绿色荧光蛋白(enhanced green fluorescent protein,EGFP)为融合标签,对裂解多糖单加氧酶(MtC1LPMO)进行重组表达,并选取宿主内源的20个信号肽对分泌进行优化,最终其胞外荧光强度最高提升4.1倍。此外,核糖体结合位点(ribosomebinding site,RBS)优化也是提升异源蛋白胞外表达量的常用方法。MAO等[12]通过RBS优化,将在枯草芽孢杆菌Bacillus subtilis中异源表达二乙酰基化二糖脱乙酰酶的胞外酶活力提升至4 807.6 U/mL,相较优化前提升了30%。
在前期研究中,本实验室已将从自然环境中筛选得到1株地衣芽孢杆菌B.licheniformis BBE11-1的角蛋白酶基因ker,并在枯草芽孢杆菌B.subtilis WB600系统中进行了重组表达,其胞外角蛋白酶活力为10.4 kU/mL。但在SDS-PAGE检验中发现发酵上清液中的胞外蛋白表达量较低,无法满足工业应用的需求。本研究中,通过将角蛋白酶原有信号肽替换为枯草芽孢杆菌内源信号肽,并对RBS位点进行基于半理性设计的饱和突变,从而提高角蛋白酶的胞外表达量。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
大肠杆菌Escherichia coli JM109,枯草芽孢杆菌B.subtilis strain 168,枯草芽孢杆菌B.subtilis WB600,质粒p43NMK均为本实验室保藏。
1.1.2 酶与试剂
DNA聚合酶(PrimeSTAR Max),TaKaRa公司;DNA聚合酶(2×Rapid Taq Master Mix、2×Phanta Max Master Mix),Vazyme公司;超级感受态细胞制备试剂盒、SanPrep柱式质粒DNA小量抽提试剂盒,上海Sangon Biotech公司;柱纯化与回收试剂盒,Thermo fisher公司;Gibson assembly反应所需T5外切酶、Phusion聚合酶、Taq连接酶等,NEB公司;SDS-PAGE所用预制胶,Invitrogen公司,相关试剂,Life公司;胰蛋白胨与酵母提取物,OXOID公司;角蛋白酶(部分磺化,5%水溶液),TCI梯希爱(上海)化成工业发展有限公司;其他常规试剂,国药集团化学试剂有限公司。
1.1.3 培养基与溶液
LB培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 5,pH 7.0;LB固体培养基需加入2%琼脂粉。
发酵培养基(g/L):胰蛋白胨20,酵母提取物5,蔗糖30,KH2PO4 3,Na2HPO4 6,MgSO4 0.3,自然pH。
1.2 实验方法
1.2.1 培养方法
菌体活化:菌体保存在-80 ℃的甘油管中,用接种环取少量菌液在固体LB培养基平板上划线,在37 ℃条件下培养12~14 h。
种子培养:从平板上挑取直径在1~2 mm的单菌落,接种至装液3 mL的12 mL摇菌管中的LB培养基中,在37 ℃、220 r/min条件下培养12~14 h。
摇瓶发酵:将培养好的种子液按5%的接种量接种至装液量50 mL的250 mL挡板摇瓶中的发酵培养基中,在37 ℃、220 r/min条件下培养24 h。
96孔板培养:从平板上挑取直径在1~2 mm的单菌落接种至装液量700 μL每孔的96孔板中的发酵培养基,在37 ℃、220 r/min条件下培养24 h。
1.2.2 信号肽序列选择及替换
利用Signal Peptide Database (http://www.signalpeptide.de/index.php),检索到枯草芽孢杆菌来源的信号肽序列。使用SignalP 5.0 (https://services.healthtech.dtu.dk/service.php?SignalP-5.0)[13]和PSORTb 3.0 (https://www.psort.org/psortb/)[14]预测信号肽对应的蛋白分泌途径和亚细胞位置,选取分泌途径预测为Sec途径且亚细胞定位在胞外的信号肽序列。
以枯草芽孢杆菌B.subtilis 168基因组为模板,PCR获得目的信号肽片段SPx。以质粒p43NMK为模板,PCR获得不含信号肽片段的线性质粒p43NMK-SP0。将不同的信号肽片段SPx与p43NMK-SP0通过Gibson assembly反应连接,如图1所示,过程中涉及引物见附表1(https://kns.cnki.net/kcms/detail/11.1802.TS.20220616.1104.003.html,下同)。

a-质粒结构;b-信号肽序列替换
图1 信号肽序列替换
Fig.1 Signal peptide sequence replacement
按照感受态制备试剂盒操作手册转化大肠杆菌E.coli JM109,经菌落PCR及测序验证后得到含有不同信号肽的重组质粒,将测序正确的重组质粒使用常规化学转化方法转入枯草芽孢杆菌B.subtilis WB600。培养方法见1.2.1。
1.2.3 半理性设计RBS突变文库构建与筛选
利用RBS Library Calculator(https://salislab.net/software/predict_rbs_library_calculator)预测对翻译起始速率影响最大的区域,并通过含有简并碱基的引物对选中位点进行饱和突变,利用96孔板进行高通量筛选,所用引物见附表2。转化流程见1.2.2,培养方法见1.2.1。
1.2.4 角蛋白酶活力测定方法
将发酵液离心(12 000 r/min,2 min)后取上清液,用0.22 μm滤膜过滤,即为粗酶液。
反应阶段:向反应体系中依次加入pH 10.0的Gly-NaOH(0.05 mol/L)缓冲液150 μL、25 g/L底物100 μL、经适度稀释的粗酶液50 μL,空白对照中应在加入酶液前提前加入200 μL三氯乙酸(0.4 mol/L)。将样品在60 ℃条件下保温20 min。反应结束后,加入200 μL三氯乙酸终止反应。
显色阶段:将终止反应的反应体系于12 000 r/min下离心2 min。在已加入1 mL 50 g/L Na2CO3溶液的1.5 mL EP管中加入200 μL反应体系上清液,并加入200 μL福林酚。将样品在50 ℃保温10 min显色,并在660 nm下测定吸光值。96孔板高通量测定时,显色体系中各组分加入量分别为100 μL 50 g/L Na2CO3溶液、20 μL反应体系上清液、20 μL福林酚。
酶活力定义:在上述反应体系中,使吸光度值上升0.001所需酶量定义为1个酶活力单位。
2 结果与分析
2.1 信号肽序列选择及发酵验证
在Signal Peptide Database中以枯草芽孢杆菌来源为限制条件检索,共得到信号肽序列244条。排除由于命名不同及其他原因导致的序列重复后,使用SignalP 5.0和PSORTb 3.0预测信号肽对应的蛋白分泌途径和亚细胞位置,选取分泌途径预测为Sec途径且亚细胞定位在胞外的信号肽序列共20条。
Sec途径信号肽通常被划分为3个区域:蛋白N端区域(N区)、疏水性区域(H区)、切割识别位点(C区)。目前存在多种在线预测方法划分信号肽内部区域,如Phobius[15]和SignalP等,但各服务器对于疏水核心范围的定义和预测方法不同,以及由于部分信号肽不符合信号肽结构的一般规律,导致无法预测信号肽内部区域。
TJALSMA等[16]通过首先定义信号肽末端信号肽酶的识别切割位点3个氨基酸为C区,定义N端带有正电荷的部分为N区,将C区与N区中间包含大量疏水性氨基酸的疏水核心区选择出来。在其他定义中,靠近H区疏水核心但不包含大量疏水氨基酸的区域往往被划分到C区中,该方法有利于信号肽中部序列整体序列保守性的分析。因此,本研究参照该方法对本研究中所选取的信号肽序列中的信号肽区域进行划分,如表1所示。
表1 本研究中选择的信号肽序列
Table 1 Signal peptide sequences selected in this study

基因N区H区C区pelCMKKIVSILFMFGLVMGFSQFQPSTVFAvprMKKGIIRFLLVSFVLFFALSTGITGVQAxynAMFKFKKNFLVGLSAALMSISLFSATASAmprMKLVPRFRKQWFAYLTVLCLALAAAVSFGVPAKAbsnMTKKAWFLPLVCVLLISGWLAPAASAbdbDMKKKQQSSAKFAVILTVVVVVLLAAIVIINNKTEQGnprEMGLGKKLSVAVAASFMSLSISLPGVQAdacBMRIFKKAVFVIMISFLIATVNVNTAHAyncMMAKPLSKGGIL-VKKVLIAGAVGTAVLFGTLSSGIPGL-PAADAQVAKAytfIMVYVLTAILILIGIVLLLRMKIHVALEYLHAcsnMKISMQKADF-WKKAAISLLVFTMFFTLMMSETVFAbglSMPYLKRVLLLLVTGLFMSLFAVTATASApelMKKVMLATALFLGLTPAGANAggtMKRTWNVCLTALLSVLLVAGSVPFHAEAsacBMNIKKFAKQATVLTFTTALLAGGATQAFAnucBMKKWMAGLFLAAAVLLCLMVPQQIQGASyfhKMKKKQVMLALTAAAGLGLTALHSAPAAKAyetMMKHMLIAGGGIGGLSAAISLRKAGFSVTLCEAamyEMFAKRFKTSLLPLFAGFLLLFHLVLAGphoBMKKFPKKLLPIAVLSSIAFSSLASGSVPEASA
经摇瓶发酵验证,20株菌株中,有10株的酶活力有不同程度的提高,如图2所示。其中胞外角蛋白酶活力最高的重组菌株为WB600-p43 NMK-Ker-SPdacB,其胞外酶活力达到84.3 kU/mL,为出发菌株的8.1倍。

图2 不同信号肽对胞外角蛋白酶活力的影响
Fig.2 Effects of different signal peptides on the activity of extracellular keratinase
对4株含有高效信号肽重组菌株的发酵上清液进行SDS-PAGE分析,如图3所示,角蛋白酶条带所在位置如箭头所示。4株含有高效信号肽的重组菌株发酵上清液中的角蛋白酶条带颜色较对照组更深,且根据观察,颜色加深程度与各菌株胞外酶活力总体呈正相关,这与实验结果基本吻合。

1-B.subtilis WB600(对照);2-SPdacB;3-SPpelC;4-SPbsn;5-SPmpr;6-初始信号肽
图3 高效信号肽重组菌株发酵上清液SDS-PAGE
Fig.3 Efficient signal peptide recombinant strain fermentation supernatant SDS-PAGE
2.2 信号肽性质分析
对所选取的20个信号肽性质进行了分析,统计了其长度、N区正电荷、疏水性和mRNA折叠Gibbs自由能(ΔG)。
信号肽N区正电荷的计算方法为,将精氨酸(arginine, R)和赖氨酸(lysine, K)带电量定义为+1,天冬氨酸(aspartic acid, D)和谷氨酸(glutamic acid, E)带电量为-1,其他氨基酸带电量均视为0,计算N区的总带电量。信号肽的疏水性计算方法为计算丙氨酸(alanine, A)、异亮氨酸(isoleucine, I)、亮氨酸(leucine, L)、缬氨酸(valine, V)、苯丙氨酸(phenylalanine, F)、色氨酸(tryptophan, W)和酪氨酸(tyrosine, Y)8种疏水性氨基酸个数占信号肽总氨基酸个数的比例。20条所选取的信号肽性质见表2。信号肽mRNA折叠ΔG的计算方法为在mfold(http://www.unafold.org/mfold/applications/rna-folding-form-v2.php),输入所选取的20个信号肽包含RBS序列在内向后共150 bp的序列。
表2 信号肽性质分析
Table 2 Analysis of signal peptide character
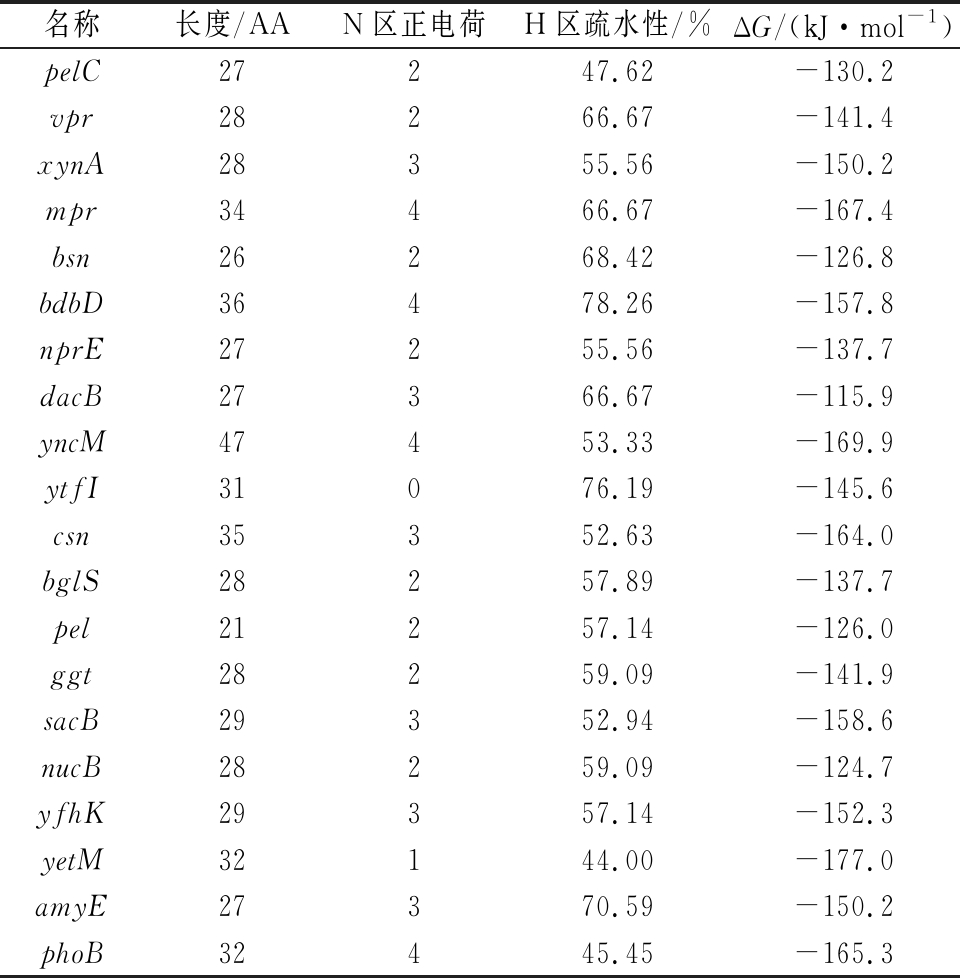
名称长度/AAN区正电荷H区疏水性/%ΔG/(kJ·mol-1)pelC27247.62-130.2vpr28266.67-141.4xynA28355.56-150.2mpr34466.67-167.4bsn26268.42-126.8bdbD36478.26-157.8nprE27255.56-137.7dacB27366.67-115.9yncM47453.33-169.9ytfI31076.19-145.6csn35352.63-164.0bglS28257.89-137.7pel21257.14-126.0ggt28259.09-141.9sacB29352.94-158.6nucB28259.09-124.7yfhK29357.14-152.3yetM32144.00-177.0amyE27370.59-150.2phoB32445.45-165.3
选取胞外酶活力较高的菌株对其各项性质与胞外酶活力的相关性进行分析。
信号肽文库中的信号肽长度集中在2~35 AA,随着信号肽的增长其胞外酶活力总体上呈现下降趋势,证明信号肽长度增加对于角蛋白酶的分泌是不利的,其最佳的长度为25~30 AA左右。此外,高效信号肽的N区正电荷量普遍分布在在2~4,但随着N端电荷的增加,信号肽的角蛋白酶分泌能力并未显示出同步的上升。分泌能力最高的4条信号肽序列SPdacB、SPbsn、SPmpr和SPbdbD相对正电荷量分别为50%、50%、40%、40%,以上结果说明信号肽中N端相对正电荷量处于一个特定的范围内时,信号肽的分泌能力更高,这与以往的研究中提出的观点相似[17]。
信号肽H区疏水性与胞外酶活力分析的结果表明,在筛选得到的高效信号肽中,角蛋白酶的胞外酶活力水平与H区疏水性有显著的正相关性,及随着H区疏水性的提高,角蛋白酶的胞外酶活力提高。此外,有相关研究报道了H区内的疏水性氨基酸会形成疏水核心,通过影响信号肽的蛋白二级结构的方式影响其跨膜时的效率[18]。
如图4所示,信号肽mRNA折叠的ΔG与胞外酶活力分析的结果表明,在筛选得到的高效信号肽中,角蛋白酶的胞外酶活力水平与ΔG有显著的正相关性,即随着ΔG的提高,角蛋白酶的胞外酶活力提高,这与此前相关研究的结果基本一致[11]。

a-长度;b-N区电荷;c-H区疏水性;d-mRNA折叠的ΔG
图4 信号肽性质与胞外酶活性相关性分析
Fig.4 Correlation analysis of signal peptide properties and extracellular enzyme activity
总体而言,许多研究已经发现通常被认为高效且应用于异源蛋白表达的信号肽,如SPaprE、SPpel、SPyoaW等,对于其他蛋白在枯草芽孢杆菌中的表达并不一定是最优的[19-21]。事实上,已有研究提出并不存在广泛适用于多种蛋白分泌的高效信号肽[22]。
已有研究中[11,16-17]对N区、H区、C区的序列分析表明,N区通常含有较多带正电荷的氨基酸,如R和K;H区通常疏水性氨基酸较多,如A、V、L等;C区最显著的特点是信号肽末端切割位的A-X-A保守序列。对表达效率最高的5条信号肽序列进行了类似的分析,如图5所示,发现高效信号肽表现出的性质符合上述中的总体规律。

a-N区序列;b-H区序列;c-C区序列
图5 高效信号肽序列保守性分析
Fig.5 Efficient signal peptide sequence conservation analysis
此外,LOW等[23]的研究发现N区的MKK序列和H区的甘氨酸对于蛋白的高效易位可能是有利的。本研究中的结果显示,对胞外酶活力提升最显著的SPdacB并不具备N区的MKK和H区的甘氨酸这2个特点;含有MKK序列的信号肽,如SPbdbD、SPnucB、SPpel和SPvpr,其胞外酶活力未表现出与该序列的相关性。SPbdbD的H区含有强疏水性区域5V片段,这显著提高了H区的疏水性。
综上所述,对于特定蛋白的表达,信号肽筛选仍是必要的,但通过分泌途径和亚细胞定位预测少量筛选信号肽而筛选宿主全信号肽文库的信号肽优化方案,能够显著地提高筛选效率。
2.3 半理性设计选择RBS突变位点
利用RBS Library Calculator(https://salislab.net/software/predict_rbs_library_calculator)预测对翻译起始速率影响最大的区域。通过比较从RBS起始位点逐步向翻译起始位点移动的方式进行预测。比较了不同位点预测翻译起始速率前50的RBS序列的分布,如图6所示。
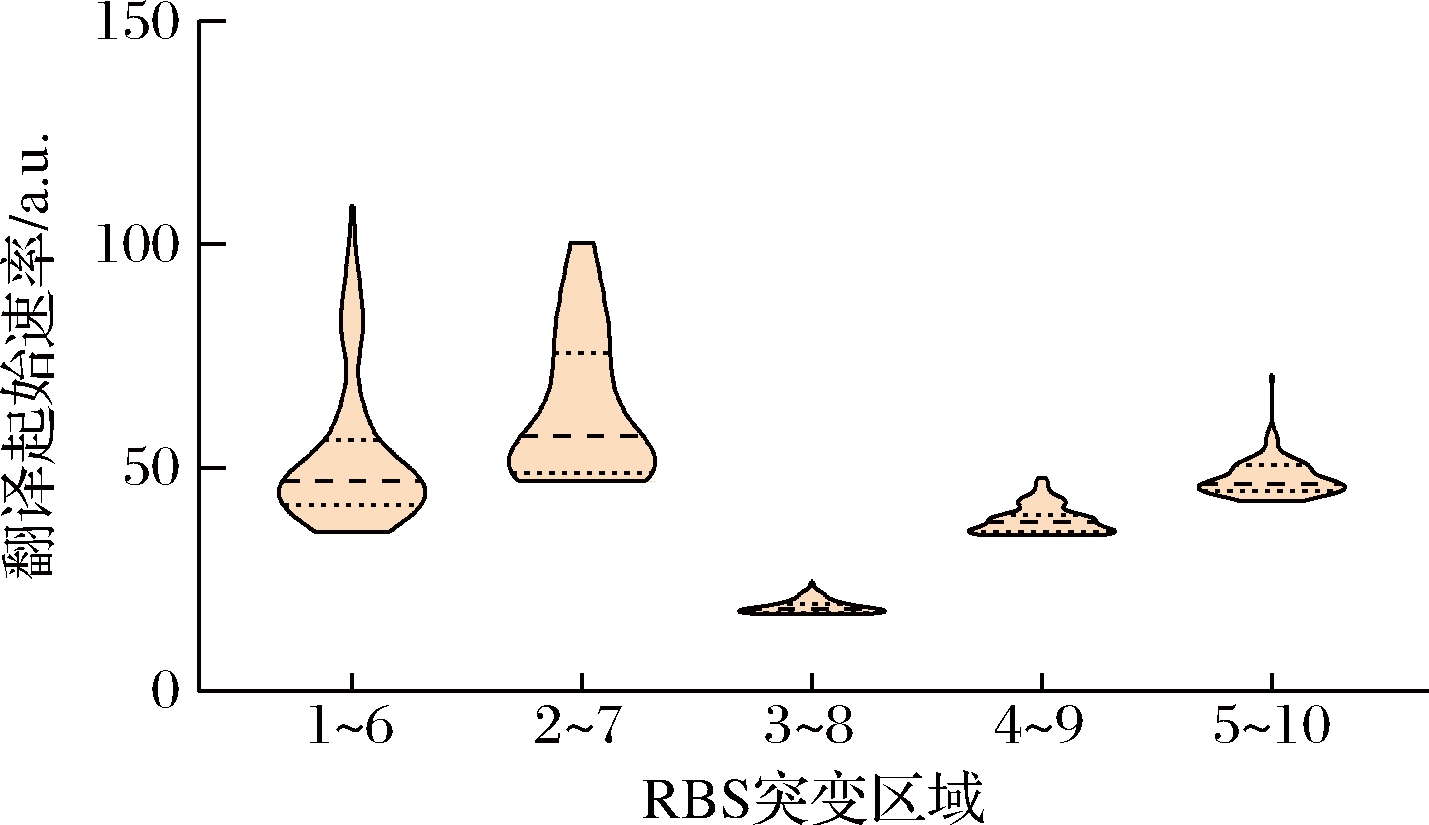
图6 RBS半理性设计突变区域对翻译起始速率的影响
Fig.6 Effects of RBS semi-rational design of mutant regions on translation initiation rate
我们发现在1~6位和2~7位时的饱和突变对理论起始翻译速率提升最显著,且1~6位相较2~7位具有更高的最大翻译起始速率,有利于高通量筛选的进行。因此,选择RBS 1~6位进行饱和突变,构建了含有4 096个突变体的突变文库,选择理论数量3倍的转化子进行筛选以达到95%的覆盖率。
2.4 RBS饱和突变筛选
通过96孔板发酵初筛得到7株较优化前酶活力有提高的重组菌株,如表3所示。
表3 RBS饱和突变筛选
Table 3 RBS Saturation Mutation Screening

名称序列翻译起始速率/a.u.ΔG/(kJ·mol-1)WTGTAAGAGAGG10.45-18.0R14C10GGGAGAGAGG32.20-20.8R16G2GAGAAAGAGG10.00-17.7R16D12GAGCTGGAGG10.10-17.8R18F7GAGGGGGAGG35.10-17.3R22D5GAGAGAGAGG7.10-20.8R126G2CGGTGAGAGG14.65-28.6R221C10GAAGGGAAGG4.74-15.8
摇瓶复筛后,其中仅3株胞外酶活力较优化前菌株有显著提高,如图7所示。

图7 RBS突变对胞外酶活力的影响
Fig.7 Effects of RBS mutation on extracellular enzyme activity
其中菌株R16D12的胞外酶活力达到109.1 kU/mL,较优化前提高了29%,是出发菌株胞外酶活力的10.5倍。对其进行了SDS-PAGE分析,结果如图8所示,发现其表达量提升并不显著,与实验结果基本吻合。结合以上研究结果,我们发现预测得到的mRNA折叠ΔG和翻译起始速率与胞外酶活力的相关性并不显著,在RBS Library Calculator预测中得到具有较高的翻译起始速率或较低的吉布斯自由能并不能说明RBS序列是高效的,其数据并不能作为RBS选择的直接依据,但基于RBS饱和突变的高通量筛选对于RBS优化仍是有效的策略。

1-SPdacB;2-R16D12
图8 RBS饱和突变菌株发酵上清液SDS-PAGE
Fig.8 RBS saturation mutant strain fermentation supernatant SDS-PAGE
3 结论
本研究结合信号肽筛选和RBS优化策略,提高了重组枯草芽孢杆菌Bacillus subtilis WB600-p43NMK-Ker的角蛋白酶胞外表达量。通过筛选来自枯草芽孢杆菌的20条信号肽序列,得到带有SPdacB的重组菌株其胞外角蛋白酶活力达到84.3 kU/mL,约为出发菌株胞外酶活力的8.1倍。在此基础上,通过对RBS进行基于半理性设计的饱和突变,筛选得到的1株高产菌株R16D12,其胞外酶活达到109.1 kU/mL,较RBS优化前提高了29%,是出发菌株胞外酶活力的10.5倍。研究结果表明,信号肽筛选和RBS优化的组合策略显著提高了角蛋白酶在枯草芽孢杆菌中的胞外表达量,所得重组菌株在摇瓶发酵条件下的角蛋白酶胞外酶活力在目前已报道的研究中处于较高水平。
[1] QIU J W, WILKENS C, BARRETT K, et al.Microbial enzymes catalyzing keratin degradation:Classification, structure, function[J].Biotechnology Advances, 2020, 44:107607.
[2] LI Q X.Progress in microbial degradation of feather waste[J].Frontiers in Microbiology, 2019, 10:2717.
[3] GUPTA R, RAJPUT R, SHARMA R, et al.Biotechnological applications and prospective market of microbial keratinases[J].Applied Microbiology and Biotechnology, 2013, 97(23):9 931-9 940.
[4] PENG Z, MAO X Z, ZHANG J, et al.Biotransformation of keratin waste to amino acids and active peptides based on cell-free catalysis[J].Biotechnology for Biofuels, 2020,13:61.
[5] SOBUCKI L, RAMOS R F, DAROIT D J.Protease production by the keratinolytic Bacillus sp. CL18 through feather bioprocessing[J].Environmental Science and Pollution Research International, 2017, 24(29):23 125-23 132.
[6] PENG Z, MAO X Z, ZHANG J, et al.Effective biodegradation of chicken feather waste by co-cultivation of keratinase producing strains[J].Microbial Cell Factories, 2019,18(1):84.
[7] 李光磊, 张娟, 方真, 等.嗜麦芽寡养单胞菌角蛋白酶基因在毕赤酵母中的表达[J].生物技术通报, 2016, 32(8):152-160.
LI G L, ZHANG J, FANG Z, et al.Expression of keratinase gene derived from Stenotrophomonas maltophilia in Pichia pastoris[J].Biotechnology Bulletin, 2016, 32(8):152-160.
[8] FANG Z, ZHANG J, LIU B H, et al.Biochemical characterization of three keratinolytic enzymes from Stenotrophomonas maltophilia BBE11-1 for biodegrading keratin wastes[J].International Biodeterioration & Biodegradation, 2013, 82:166-172.
[9] LIU B H, ZHANG J, LI B, et al.Expression and characterization of extreme alkaline, oxidation-resistant keratinase from Bacillus licheniformis in recombinant Bacillus subtilis WB600 expression system and its application in wool fiber processing[J].World Journal of Microbiology & Biotechnology, 2013, 29(5):825-832.
[10] WATANABE K, TSUCHIDA Y, OKIBE N, et al.Scanning the Corynebacterium glutamicum R genome for high-efficiency secretion signal sequences[J].Microbiology, 2009, 155(Pt3):741-750.
[11] GUO X, CHAI C C, AN Y J, et al.Rational design of signal peptides for improved MtC1LPMO production in Bacillus amyloliquefaciens[J].International Journal of Biological Macromolecules, 2021, 175:262-269.
[12] MAO X Z, HUANG Z Y, SUN G Y, et al.High level production of diacetylchitobiose deacetylase by refactoring genetic elements and cellular metabolism[J].Bioresource Technology, 2021, 341:125836.
[13] ALMAGRO ARMENTEROS J J, TSIRIGOS K D, SØNDERBY C K, et al.SignalP 5.0 improves signal peptide predictions using deep neural networks[J].Nature Biotechnology, 2019, 37(4):420-423.
[14] YU N Y, WAGNER J R, LAIRD M R, et al.PSORTb 3.0:Improved protein subcellular localization prediction with refined localization subcategories and predictive capabilities for all prokaryotes[J].Bioinformatics, 2010, 26(13):1 608-1 615.
[15] K LL L, KROGH A, SONNHAMMER E L L.Advantages of combined transmembrane topology and signal peptide prediction-the Phobius web server[J].Nucleic Acids Research, 2007, 35:429-432.
LL L, KROGH A, SONNHAMMER E L L.Advantages of combined transmembrane topology and signal peptide prediction-the Phobius web server[J].Nucleic Acids Research, 2007, 35:429-432.
[16] TJALSMA H, BOLHUIS A, JONGBLOED J D, et al.Signal peptide-dependent protein transport in Bacillus subtilis:A genome-based survey of the secretome[J].Microbiology and Molecular Biology Reviews, 2000, 64(3):515-547.
[17] PENG C, SHI C, CAO X, et al.Factors influencing recombinant protein secretion efficiency in gram-positive bacteria:Signal peptide and beyond[J].Frontiers in Bioengineering and Biotechnology, 2019, 7:139.
[18] CHOU M M, KENDALL D A.Polymeric sequences reveal a functional interrelationship between hydrophobicity and length of signal peptides[J].Journal of Biological Chemistry, 1990, 265(5):2 873-2 880.
[19] TSUJI S, TANAKA K, TAKENAKA S, et al.Enhanced secretion of natto phytase by Bacillus subtilis[J].Bioscience Biotechnology and Biochemistry, 2015, 79(11):1 906-1 914.
[20] FU G, LIU J L, LI J S, et al.Systematic screening of optimal signal peptides for secretory production of heterologous proteins in Bacillus subtilis[J].Journal of Agricultural and Food Chemistry, 2018, 66(50):13 141-13 151.
[21] HEINRICH J, DREWNIOK C, NEUGEBAUER E, et al.The YoaW signal peptide directs efficient secretion of different heterologous proteins fused to a StrepII-SUMO tag in Bacillus subtilis[J].Microbial Cell Factories, 2019, 18(1):31.
[22] PARK S, SCHUMANN W.Optimization of the secretion pathway for heterologous proteins in Bacillus subtilis[J].Biotechnology and Bioprocess Engineering, 2015, 20(4):623-633.
[23] LOW K O, JONET M A, ISMAIL N F, et al.Optimization of a Bacillus sp. signal peptide for improved recombinant protein secretion and cell viability in Escherichia coli[J].Bioengineered, 2012, 3(6):334-338.


