ε-聚赖氨酸(ε-poly-L-lysine,ε-PL)主要是从链霉菌发酵液中分离纯化得到的一种阳离子同型氨基酸聚合物,一般是由25~35个L-赖氨酸残基通过ε-氨基和α-羧酸基之间的酰胺键连接而成[1]。由于其多阳离子性质可以通过离子吸附与携带负电荷的细胞表面相互作用,故对于包括革兰氏阳性和阴性细菌、酵母菌、霉菌甚至病毒在内的多种微生物均表现出良好的抑菌抗病毒活性[2]。目前,ε-PL及其盐酸盐已获得日本、韩国、美国、中国等国家和地区批准用作天然食品防腐剂[3],被认为是天然防腐剂取代化学防腐剂的关键品种。此外,ε-PL还在药物载体、纳米粒、基因载体、脂质体、干扰素诱导剂、脂肪酶抑制剂和水凝胶等领域有着广阔的应用前景[4]。由此可见,ε-PL是一种具有广泛应用领域的生物技术产品。
提高ε-PL发酵产量以降低其生产成本是推动ε-PL在上述领域广泛应用的重要前提。然而,获得一株生产性能好、遗传稳定的ε-PL高产菌株是实现其高效发酵生产的先决条件。为此,过去40多年,ε-PL生产菌株改造一直是学术界和产业界的研究重点。早在20世纪90年代,HIRAKI等[5]通过物理/化学诱变结合L-赖氨酸结构类似物和甘氨酸双重抗性筛选策略,获得了一株高产突变株Streptomyces albulus 11011A,其摇瓶产量达到2.11 g/L,较出发菌株提高了10倍。日本JNC公司(原Chisso公司)正是利用该突变株建立了年产千吨级的生产线,从而成为目前全球最大的ε-PL制造商。在该育种思路启发下,国内学者纷纷建立了基于诱变和抗性筛选的菌种改造策略,实现拥有自主知识产权的ε-PL产生菌Kitasatospora sp.PL6-3、S.diastatochromogenes、S.albulus C-18,ε-PL的产量从而提高了40%~300%。本团队从2010年开始率先尝试应用基因组重排育种技术提高产生菌合成ε-PL的能力,实现摇瓶ε-PL产量达到2.42 g/L[6];继续利用核糖体工程育种技术,实现S.albulus AS3-14的摇瓶ε-PL产量提高到2.91 g/L[7]。随后,通过多种育种方法和不同抗生素抗性叠加,筛选到一株最高产的突变株S.albulus R6,其摇瓶ε-PL产量达到4.41 g/L,是已报道最高产菌株(S.albulus 11011A)的2.1倍[4]。由此可见,诱变、基因组重排、核糖体工程等育种策略是提高ε-PL产生菌产物合成能力的有效手段。然而,大量的工作实践表明,传统育种方法已不再能够继续显著提升生产菌的ε-PL合成能力。可能的原因在于,反复的诱变、基因组融合和抗生素抗性筛选使得生产菌产生了“疲劳效应”。
近年来,国内外学者积极探索利用代谢工程手段提高菌株的ε-PL产量。例如,HAMANO等[8]为了消除S.albulus NBRC14147中天门冬氨酸激酶受L-赖氨酸和/或L-苏氨酸反馈抑制作用,对天门冬氨酸激酶进行了定点突变改造,使得工程菌在5 L发酵罐中ε-PL产量达到15.2 g/L,提高了26.7%;南京工业大学徐虹教授课题组分别将铵转运蛋白基因(amtB)和血红蛋白基因(vgb)在S.albulus PD-1中进行过表达,ε-PL产量均提高了50%,达到35 g/L左右[3,9];YAMANAKA等[10]通过敲除S.albulus 中tetramycins和tetrins合成基因簇(95 kb),将更大代谢流导向ε-PL生物合成,将ε-PL产量提升了约20%。WANG等[11]通过在S.albulus CICC11022中过表达ε-PL合成酶(Pls),结合柠檬酸钠的流加,实现72 h补料分批发酵条件下ε-PL产量达到20.1 g/L,较出发菌株提高了3.2倍。LI等[12]在S.diastatochromogenes 6#-7中过表达二氢二吡啶酸合成酶基因(dapA),工程菌ε-PL产量达到30.54 g/L,较出发菌株提高了19.8%。因此,借助分子操作手段改造ε-PL高产菌株是继传统育种之后第二种有效提高ε-PL产生菌株的有效手段。然而,目前对于ε-PL生物合成关键代谢节点和调控机制的认识还不清晰,因此还缺乏有效的靶基因。
已有研究表明,ε-PL生物合成过程是以L-赖氨酸作为前体、ATP作为辅因子,在Pls催化下形成ε-PL[13]。因此,本研究试图聚焦ε-PL生物合成前体L-赖氨酸的合成途径(还原力NADPH及碳骨架),以期通过提高L-赖氨酸合成能力来提高ε-PL合成效率。为此,基于L-赖氨酸代谢工程改造文献[14-18],我们选取了丙酮酸羧化酶基因(pyc)、磷酸烯醇式丙酮酸羧化酶基因(ppc)、6-磷酸葡萄糖脱氢酶基因(zwf)、二氢吡啶二羧酸合成酶基因(dapA)、二氨基庚二酸脱羧酶基因(lysA)和ε-PL合成酶基因(pls)等6个靶基因,通过基因过表达手段,评估它们对ε-PL生物合成影响,为后续代谢工程系统改造ε-PL生产菌种提供有效靶基因。
1 材料与方法
1.1 菌株及质粒
本研究所用的菌株和质粒见表1。所用引物由苏州金唯智生物科技有限公司合成,见表2。其中Streptomyces albulus WG608为出发菌株,其余系列菌株均为S.albulus WG608的突变株。E.coli DH5α主要用于重组质粒的克隆,E.coli ET12567/pUZ8002则用于产生非甲基化DNA。整合型质粒pIB139用于过表达菌株的构建。
表1 本研究所用菌株及质粒
Table 1 Strains and plasmids used in this study
菌株/质粒特征来源Streptomyces albulusWG608M-Z18选育而来的ε-PL高产菌株实验室保藏OE-ppcWG608中ppc基因过表达菌株本研究OE-zwfWG608中zwf基因过表达菌株本研究OE-dapAWG608中dapA基因过表达菌株本研究OE-lysAWG608中lysA基因过表达菌株本研究OE-pycWG608中pyc基因过表达菌株本研究OE-plsWG608中pls基因过表达菌株本研究control-pIB139WG608中过表达菌株对照菌株本研究E.coliDH5α用于重组质粒的制备实验室保藏ET12567(pUZ8002)DNA甲基化缺陷型,用于接合转移实验室保藏质粒p1B139整合型过表达质粒王浩鑫课题组惠赠p1B139-ppcppc基因过表达质粒本研究p1B139-zwfzwf基因过表达质粒本研究p1B139-dapAdapA基因过表达质粒本研究p1B139-lysAlysA基因过表达质粒本研究p1B139-pycpyc基因过表达质粒本研究p1B139-plspls基因过表达质粒本研究
1.2 试剂与仪器
限制性内切酶,TaKaRa (大连)有限公司;一步克隆试剂盒、高保真DNA聚合酶预混液、基因组提取试剂盒、质粒提取试剂盒、片段纯化试剂盒、反转录试剂盒、荧光定量PCR试剂盒,诺唯赞生物科技(南京)有限公司;Trizol总RNA抽提试剂盒、50×TAE缓冲液、卡那霉素、安普霉素、氯霉素、萘啶酮酸,生工生物工程(上海)股份有限公司;ε-PL标准品,郑州拜纳佛生物工程股份有限公司。
表2 本研究所用到的引物
Table 2 Primers used in this study

引物名称引物序列(5′-3′)基因过表达引物ppc-FggttggtaggatccacatatgCCCCGCCAAGACGTGCTCppc-RctatgacatgattacgaattcTCAGCCGGTGTTGCGCAGzwf-FggttggtaggatccacatatgGCCGTCGGGCCTGGTCATzwf-RctatgacatgattacgaattcTCATGGCCGACGCCAGCTdapA-FggttggtaggatccacatatgCTTGACCGGGCACCCGCGdapA-RctatgacatgattacgaattcTCAGCCGGTGGGGCGCAGlysA-FggttggtaggatccacatatgTCCCGGAACAACCAGCTACGlysA-RctatgacatgattacgaattcTCAGAGGCCGATGTCGCGpyc-FggttggtaggatccacatatgATGGTCTCGTCACCCGGCpyc-RcgcggccgcggatcctctagaTCAGGCGAGTTGGACGAGAA验证基因过表达的引物p1B139-FTTGCGCCCGATGCTAGTCGp1B139-RGCACGACAGGTTTCCCGACTGO-pyc-FGACTCCGTGGTCGGCTTCCTO-pyc-RACCGCTACATACTCGCTCTGCTAO-pls-FCGCGATGCTGTTGTGGGO-pls-RAACCGTTCGGCGTCCTT实时定量-PCR引物Q-ppc-FGCCATCCCGTGGGTGTTQ-ppc -RAGGAAGTTGCGGAAGAAGTGQ-zwf-FAGTGGGAGGACGAGGACTTQ-zwf-RTTGCCCTGGACGAACCQ-dapA-FTGGCACGCTGATGATTGGTQ-dapA-RCTCCCTCACCCGACTGGTCTQ-lysA-FCCACTTGGGCTCCCAGATCQ-lysA-RAGGGTGATGCCGTGTTGCQ-pyc-FTCGAGGCGATGAAGATGGAQ-pyc-RGGCGAGTTGGACGAGAAGATQ-pls-FTCACCTACCGCGACCTGTGQ-pls-RAGGACGGCGAGGATGGA
DYCP-31DN电泳仪,北京市六一仪器厂;C1000 Touch PCR仪、Gel Doc EZ凝胶成像仪,Bio-Rad;K5800微量分光光度,北京凯奥科技发展有限公司;UV2100分光光度,优尼科仪器有限公司;T&J-Miniskid 1L×4型1 L发酵罐,迪必尔生物工程(上海)有限公司;BLBIO-XM型5 L发酵罐,上海百仑生物科技有限公司。
1.3 主要培养基和培养条件
大肠杆菌在37 ℃的LB固体/液体培养基(胰蛋白胨10 g/L,酵母粉5 g/L,NaCl 10 g/L,琼脂粉15 g/L,pH 7.0)中培养,需要添加抗生素时,卡那霉素、安普霉素和氯霉素添加的最终质量浓度分别为25、50、25 μg/mL。小白链霉菌(S.albulus)的产孢培养基为BTN固体培养基(葡萄糖10 g/L,鱼粉蛋白胨2 g/L,酵母粉1 g/L,pH 7.5),在30 ℃下进行培养。S.albulus和大肠杆菌种间接合转移的培养基为MS固体培养基(甘露醇20 g/L,大豆粉20 g/L,琼脂粉20 g/L,pH 7.0,灭菌后加入MgCl2至最终摩尔浓度10 mmol/L),在30 ℃下进行培养,覆盖抗生素安普霉素和萘啶酮酸素最终质量浓度分别为50、25 μg/mL。S.albulus种子培养基为M3G培养基[葡萄糖50 g/L,酵母粉5 g/L,(NH4)2SO4 10 g/L,MgSO4·7H2O 0.5 g/L,FeSO4·7H2O 0.03 g/L,KH2PO4 1.36 g/L,K2HPO4 0.8 g/L,ZnSO4·7H2O 0.04 g/L,pH 6.8],在30 ℃ 200 r/min下进行培养。S.albulus发酵培养基为RSM培养基[葡萄糖60 g/L,酵母粉10 g/L,(NH4)2SO4 10 g/L,MgSO4·7H2O 0.8 g/L,FeSO4·7H2O 0.05 g/L,KH2PO4 4 g/L,pH 6.8],在30 ℃ 200 r/min下进行培养。
1.4 过表达菌株的构建
pIB139质粒是由整合型pSET152质粒改造而来的大肠杆菌-链霉菌穿梭型质粒,其中包含了红霉素强启动子、pUC18复制子、安普霉素抗性基因以及Ф31(attp)的整合酶基因和整合位点,因此可以通过整合位点高效的将过表达质粒整合到链霉菌的基因组中,从而实现目的基因的过表达。过表达质粒构建原理如图1所示。本研究选取NdeⅠ和EcoRⅠ作为酶切位点,以提取的S.albulus WG608和S.lividans全基因组为模板分别进行PCR扩增得到ppc、zwf、dapA、lysA、pls和pyc基因片段,将片段纯化后分别与线性化pIB139载体进行连接,然后转化进E.coli DH5α感受态细胞,在含有安普霉素抗性的平板上挑取转化子进行过夜培养,提取质粒进行双酶切验证并测序。将验证正确的过表达质粒转化入E.coli ET12567,然后与S.albulus WG608进行接合转移。为了得到过表达菌株,在添加了安普霉素和萘啶酮酸的BTN固体培养基上进行接合子的筛选,待菌丝长出后,进行菌落PCR验证。将验证正确的菌株的孢子进行传代,然后提取基因组和RNA进行PCR验证和实时定量PCR(quantitative real-time PCR,qRT-PCR)验证。将验证成功的过表达菌株进行传代保菌,并进行发酵验证。
1.5 RNA提取和qRT-PCR
首先,按照Trizol总RNA抽提试剂盒说明书操作步骤提取RNA,用核酸定量仪测定其质量浓度和纯度后,利用核酸电泳检测RNA的完整性。其次,参考反转录试剂盒说明书取800 ng RNA进行反转录(先消化残留gDNA)得到cDNA,置于-80 ℃保存。最后,将cDNA样品稀释10倍作为模板,按照qRT-PCR试剂盒说明书进行qRT-PCR。所有反应重复3次,RT-qPCR结果采用2-ΔΔCt法进行内参基因hrdB的相对定量。对于每个样本中的目的基因,将对照组(S.albulus WG608)中该基因的转录水平定义为1,结果显示为相对于对照组中的变化倍数。相关基因引物设计见表2。

图1 过表达质粒构建流程
Fig.1 Construction procedure of overexpression plasmid
1.6 小白链霉菌的发酵
种子培养:从活化的新鲜平板上挑取3~4环孢子接种至M3G培养基,30 ℃、200 r/min培养24 h左右。
摇瓶发酵:种子液按照体积分数8%的量转接到RSM培养基,30 ℃、200 r/min培养72 h左右。
1 L分批发酵:在接种之前,温度、通气和搅拌分别保持在30 ℃、0.8 L/min和300 r/min,通过氨水的流加将初始pH值控制在6.8。将培养24 h种子液按照体积分数8%的量转接到RSM培养基,在发酵过程中将搅拌(300~1 500 r/min)与溶氧联动,保证溶氧在30%左右,并流加氨水维持pH 4.0。
5 L补料分批发酵:前期同1 L发酵罐的分批发酵,待pH值降至5.5时,流加氨水维持pH值5.5预培养9.5 h。随后不控制pH值,待其自然下降至4.0开始计时,6 h后将pH值调回4.0,之后将搅拌维持在900 r/min,同时手动调节通气保证溶氧在20%左右,每隔6 h取样实时测定菌体干重(dried cell weight,DCW)、ε-PL浓度、葡萄糖浓度![]() 浓度和L-赖氨酸浓度,通过蠕动泵流加料液维持葡萄糖质量浓度10 g/L左右
浓度和L-赖氨酸浓度,通过蠕动泵流加料液维持葡萄糖质量浓度10 g/L左右![]() 质量浓度0.2~0.4 g/L,L-赖氨酸质量浓度2 g/L左右。
质量浓度0.2~0.4 g/L,L-赖氨酸质量浓度2 g/L左右。
1.7 分析方法
发酵参数的测定:取10 mL发酵液10 000 r/min离心5 min。ε-PL浓度的测定采用甲基橙比色![]() 浓度的测定采用奈斯勒试剂比色法[20];葡萄糖和L-赖氨酸的浓度利用生物传感分析仪(SBA-40C)进行测定;DCW采用滤纸差量测重法,将离心的沉淀倒入干燥并称重的滤纸上抽滤,105 ℃烘干至恒重,称重并计算菌体量。碳源转化率计算如公式(1)所示:
浓度的测定采用奈斯勒试剂比色法[20];葡萄糖和L-赖氨酸的浓度利用生物传感分析仪(SBA-40C)进行测定;DCW采用滤纸差量测重法,将离心的沉淀倒入干燥并称重的滤纸上抽滤,105 ℃烘干至恒重,称重并计算菌体量。碳源转化率计算如公式(1)所示:
碳源转化率![]()
(1)
2 结果与分析
2.1 过表达质粒的构建
按照1.4节所述方法,利用PCR技术从Streptomyces.lividans基因组中扩增出pyc(3 575 bp),从S.albulus WG608基因组中分别扩增出ppc(2 819 bp)、zwf(1 465 bp)、dapA(1 052 bp)、lysA(1 461 bp)和pls(4 069 bp),结果如图2-a所示。随后,通过一步克隆试剂盒将上述6个基因分别连接到载体pIB139上,得到重组质粒pIB139-ppc、pIB139-zwf、pIB139-dapA、pIB139-lysA、pIB139-pls和pIB139-pyc。为保证重组质粒携带的基因正确,首先利用NdeⅠ 和EcoRⅠ双酶切验证各重组质粒携带的基因大小,结果如图2-b所示,表明6个双酶切基因片段大小与实际大小一致;再将重组质粒进行测序,结果表明重组质粒携带的基因序列与目标基因序列完全一致(测序结果未给出)。因此,6个重组质粒均已构建成功,可用于下一步过表达菌株构建。

a-基因片段PCR;b-重组质粒的双酶切验证
图2 过表达质粒的构建
Fig.2 Construction of overexpression plasmids
2.2 过表达菌株的构建
将上述构建成功的重组质粒分别转化到E.coli DH5α感受态细胞,然后通过接合转移分别导入到S.albulus WG608菌株中,筛选具有安普霉素抗性的菌株,待菌丝长出后,进行菌落PCR验证,将验证正确的菌株的孢子进行传代。将菌落PCR正确的菌株孢子接入种子培养基培养24 h,然后分别提取它们的基因组和RNA进行PCR验证和qRT-PCR监测,结果如图3所示。
基因组PCR结果(图3-a)显示,6个过表达菌株中携带的基因大小与预期基因大小一致,表明6个基因的过表达质粒均已成功整合到S.albulus WG608基因组。qRT-PCR结果(图3-b)表明,ppc和dapA在S.albulus WG608中获得了15倍左右的表达,lysA和pls分别获得了50倍和40倍的表达,而pyc则获得了12 000倍的表达。令人意外的是,zwf未获得过表达,可能原因是S.albulus WG608未正常转录zwf,也可能zwf作为管家基因已经获得了高表达[21]。总之,除zwf外,其余5个基因ppc、dapA、lysA、pls和pyc均在S.albulus WG608中成功实现了过表达。
2.3 过表达菌株发酵生产ε-PL性能评价
将出发菌株S.albulus WG608、含空载质粒菌株Control-pIB139和过表达菌株按照1.6节所述的方法进行摇瓶发酵,结果如图4-a所示。发酵结果表明,control-pIB139和原始菌株S.albulus WG608的菌体量和ε-PL产量均保持一致,表明pIB139质粒不会影响S.albulus WG608的菌体生长和ε-PL合成。然而,过表达zwf、dapA和lysA均未实现ε-PL产量增加,只有OE-ppc(3.08 g/L)和OE-pls(2.96 g/L)两个过表达菌株的ε-PL产量较原始菌株S.albulus WG608(2.72 g/L)有所提升,分别提高了14.9%和10.3%。值得注意的是,异源表达pyc使得ε-PL产量下降了10.3%,推测可能是异源表达pyc为S.albulus WG608正常代谢带来了负荷,影响了ε-PL的正常合成。

a-基因组PCR验证;b-qRT-PCR验证
图3 菌株的过表达验证
Fig.3 Overexpression validation of six strains
由于pH值是影响ε-PL生物合成最重要的环境条件,故在1 L发酵罐中进行了恒定pH 4.0的分批发酵,以考察过表达菌株分批发酵生产ε-PL情况,结果如图4-b所示。从发酵结果可以看出,与摇瓶发酵结果相类似,pH 4.0分批发酵中control-pIB139和原始菌株S.albulus WG608的菌体量和ε-PL产量仍均保持一致,过表达zwf、dapA和lysA仍不能增加ε-PL产量,而过表达ppc可以使得ε-PL产量提高15.2%。然而,与摇瓶发酵结果不同的是,在pH 4.0分批发酵中,过表达pyc实现ε-PL产量达到6.39 g/L,较出发菌株提高了14.1%,同时单位菌体ε-PL合成能力也提高了23.1%;过表达pls却未实现ε-PL产量增加,表明过表达pyc和pls在pH 4.0分批发酵和摇瓶自然发酵两种条件下对ε-PL合成的影响是不同的,这可能和两种培养条件下菌体生理状态有关。LI等[12]在S.diastatochromogenes中过表达dapA后,摇瓶发酵ε-PL产量达到0.97 g/L,较原始菌株提高17.5%。zwf、dapA和lysA没有达到预期的效果也有可能是单个基因过表达的效果有限,无法展现出ε-PL产量上的差异。

a-过表达菌株摇瓶发酵;b-过表达菌株恒定pH 4.0分批发酵
图4 过表达菌株利用摇瓶发酵和恒定pH 4.0 分批发酵生产ε-PL的结果
Fig.4 Fermentation results of the overexpressed strains in shake flask fermentation and batch fermentation at constant pH 4.0 for ε-PL production
令人意外的是,过表达pls可以在摇瓶水平提高ε-PL产量,但却不能在pH 4.0的1 L分批发酵条件下提高ε-PL产量,推测可能是在pH 4.0分批发酵条件下前体L-赖氨酸供应不足限制了ε-PL产量提升。因此,在分批发酵进行到24 h时,一次性添加4 g/L L-赖氨酸,最终发酵结果如图4-b所示。添加L-赖氨酸后,出发菌和OE-pls的ε-PL产量分别达到8.52 g/L 和10.02 g/L,较未添加L-赖氨酸的分批发酵,分别提高了54.5%和81.9%,表明前体L-赖氨酸供应不足是限制出发菌和pls过表达菌株合成ε-PL的瓶颈,这种限制作用在OE-pls菌株中表现得更为明显。
2.4 过表达菌株补料分批发酵生产ε-PL
补料分批发酵方式是生产ε-PL的主流形式。为了评估过表达菌株OE-ppc、OE-pyc和过表达菌株OE-pls联合L-赖氨酸流加在补料分批发酵条件下对ε-PL产量提升作用,对这3个过表达菌株进行了5 L发酵罐的补料分批发酵,结果如图5和表3所示。出发菌S.albulus WG608经过192 h发酵,ε-PL产量为35.5 g/L、菌体量64.7 g/L、单位菌体合成能力0.55 g/g、葡萄糖消耗速率7.15 g/h、碳源转化率9.57%,而过表达菌株OE-ppc、OE-pyc和OE-pls发酵192 h的ε-PL产量分别为36.5、39.0、41.3 g/L,较出发菌株分别提高了2.8%、9.9%和16.3%;菌体量分别为59.1、58.5、47.8 g/L,较出发菌株分别下降了8.7%、9.6%和26.1%;单位菌体合成能力分别为0.62、0.67、0.86 g/g,较出发菌株分别提高了12.7%、21.8%和56.4%;葡萄糖消耗速率分别为6.54、7.13、6.70 g/h,与出发菌无显著差异;葡萄糖转化率分别为10.75%、10.55%和11.88%,较出发菌株分别提升了12.3%、10.2%和24.1%。由此可以看出,在补料分批发酵过程中,过表达ppc、pyc、pls均能实现ε-PL产量、单位菌体合成能力、葡萄糖转化率提高。过表达ppc提高ε-PL产量不明显,可能是由于磷酸烯醇式丙酮酸羧化酶受到了天冬氨酸和苹果酸的刚性反馈抑制[22],CHEN等[18]也遇到了类似情况,于是在ppc中引入了一个点突变解除了磷酸烯醇式丙酮酸羧化酶受到的反馈抑制,增加了回补途径通量从而促进L-赖氨酸的合成。后期,也可以考虑从这个角度出发来提高L-赖氨酸的合成能力。从菌体量来看,3株过表达菌株的菌体量与出发菌相比都有一定的下降,而对于插入片段最长的OE-pls而言,它的菌体量下降尤为严重,这可能是因为过表达质粒的引入给菌体生长带来了一定负荷,最终导致了菌体量下降。
为了进一步提高OE-pls菌株的ε-PL产量,在补料分批发酵过程中进行了L-赖氨酸的连续流加,结果如图5和表3所示。与不流加L-赖氨酸相比,出发菌株的ε-PL产量提升到了45.7 g/L,提高了28.7%;菌体量降低到了52.6 g/L,降低了18.7%;单位菌体合成能力提升到了0.87 g/g,提高了58.2%;葡萄糖消耗速率为7.23 g/h,与出发菌相当;碳源转化率提升到了11.4%,提高了19.5%;OE-pls菌株的ε-PL产量提升到了61.9 g/L,提高了49.9%;菌体量提高到了64.3 g/L,增加了34.5%;单位菌体合成能力提升到了0.96 g/g,提高了11.6%;葡萄糖消耗速率提升到了7.5 g/h,提高了11.9%;碳源转化率提升到了14.5%,提高了22.1%。在流加L-赖氨酸后与出发菌相比,OE-pls菌株的L-赖氨酸消耗量提高了52.2%,ε-PL产量提高了35.4%,单位菌体合成能力提高了10.3%,碳源的转化率提高了26.8%。可见,在补料分批发酵过中流加L-赖氨酸可以提高菌株的ε-PL产量和碳源转的化率,促进OE-pls菌株葡萄糖的消耗及菌体的生长。而pls的过表达可以显著提高菌株的ε-PL产量和L-赖氨酸及葡萄糖的利用能力,显著提高菌株的整体发酵水平。pls的过表达结合L-赖氨酸流加,使得ε-PL产量提升的同时也增加了菌株对于ATP的需求,从而导致了葡萄糖消耗速率的增加,而葡萄糖消耗速率的增加也促进了菌体的生长。该发酵工艺的ε-PL产量与当前报道的5 L发酵罐中最高ε-PL产量70.3 g/L[4]还有一定的差距,但当前并未对该菌株的发酵培养基及发酵工艺进行系统优化,后续若对培养基进行优化并结合先进的发酵工艺可能会展现出工程菌株的最大优势。

a-发酵过程中ε-PL产量变化情况; b-发酵过程中菌体量变化情况
图5 过表达菌株在5 L发酵罐中补料分批发酵生产 ε-PL的结果
Fig.5 Fermentation results of the overexpressed strains used for ε-PL production by fed-batch fermentation in 5 L fermentor
表3 过表达菌株补料分批发酵生产ε-PL的过程参数
Table 3 Process parameters of the overexpressed strains used for ε-PL production by fed-batch fermentation
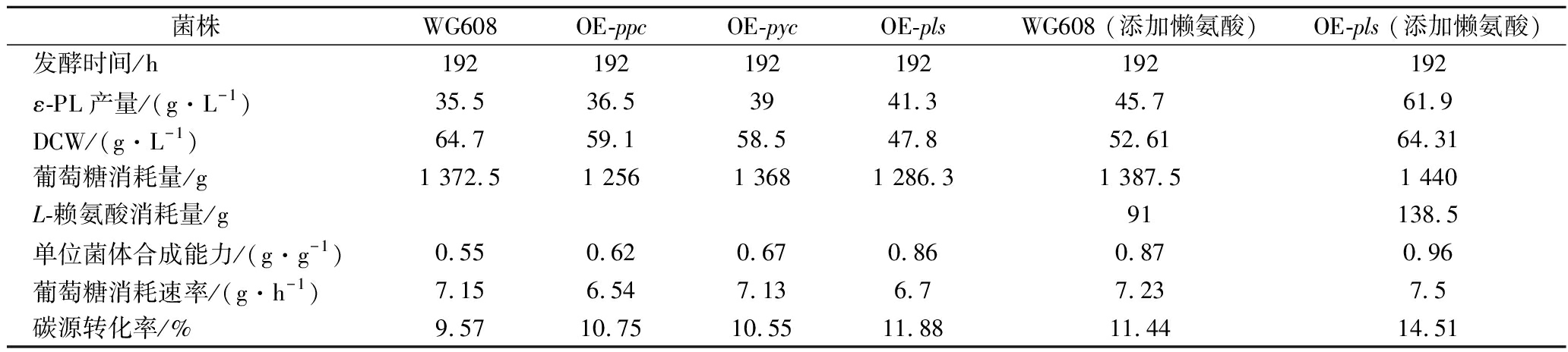
菌株WG608OE-ppcOE-pycOE-plsWG608 (添加懒氨酸)OE-pls (添加懒氨酸)发酵时间/h192192192192192192ε-PL产量/(g·L-1)35.536.53941.345.761.9DCW/(g·L-1)64.759.158.547.852.6164.31葡萄糖消耗量/g1 372.51 2561 3681 286.31 387.51 440L-赖氨酸消耗量/g91138.5单位菌体合成能力/(g·g-1)0.550.620.670.860.870.96葡萄糖消耗速率/(g·h-1)7.156.547.136.77.237.5碳源转化率/%9.5710.7510.5511.8811.4414.51
利用遗传改造技术提高ε-PL产量有关于改造氮代谢[3](转氨酶基因amtB)、二氨基庚二酸途径[8,12](天冬氨酸激酶基因ask和二氢吡啶二羧酸合成酶基因dapA)和ε-PL合成酶基因pls[11,23]的研究,但同时考虑到ε-PL合成的碳代谢途径(二氨基庚二酸途径、草酰乙酸回补途径、戊糖磷酸途径和ε-PL合成途径)上多个基因的文章鲜有报道。本研究关于二氨基庚二酸途径上的2个基因dapA和lysA及戊糖磷酸途径的关键酶基因zwf的过表达并没有达到预期提高ε-PL产量的效果,草酰乙酸回补途径的2个基因ppc和pyc的过表达也没有较显著的效果,但却在一定程度上改善了菌株的生产性能,同时ε-PL合成酶基因pls的过表达也达到了比较好的效果,这与WANG等[11]的报道一致,在摇瓶发酵时ε-PL产量达到了0.85 g/L,较出发菌株提高88%。本研究的结果表明,通过改造碳代谢途径的基因来提高ε-PL产量这一遗传操作方法是可行的,而草酰乙酸回补途径的两个基因和ε-PL合成酶基因在以后的代谢工程改造中可以作为有效的改造靶点。
3 结论
为了发掘影响ε-PL生物合成的关键基因,本研究通过基因过表达的手段,考察了L-赖氨酸合成途径关键基因(ppc、zwf、dapA、lysA、pyc)和ε-PL合成酶基因(pls)在摇瓶发酵、分批发酵和补料分批发酵中对ε-PL合成的影响。研究结果表明,回补途径的2个基因(ppc和pyc)和ε-PL合成酶基因(pls)的过表达可以有效促进S.albulus WG608合成ε-PL。在分批发酵及补料分批发酵过程中添加L-赖氨酸可以有效促进S.albulus WG608发酵生产ε-PL,表明前体L-赖氨酸供应不足是限制出发菌和pls过表达菌株合成ε-PL的瓶颈,这种限制作用在OE-pls菌株中表现得更为明显。ppc和pyc是位于草酰乙酸回补途径上的两个关键酶基因,它们可以促进草酰乙酸的补充,而草酰乙酸又为L-赖氨酸的合成提供碳骨架,这2个基因的过表达可以促进ε-PL的合成,归根结底还是促进了前体L-赖氨酸的供应。但ppc和pyc单个基因的过表达却并未达到出发菌S.albulus WG608外源添加L-赖氨酸时的效果,表明在L-赖氨酸合成途径中一定还存在其他重要节点。位于二氨基庚二酸途径上的2个基因dapA和lysA的过表达没有达到促进ε-PL合成的目的,这很有可能是因为单基因的过表达的作用有限。而位于戊糖磷酸途径上的关键酶基因zwf的过表达也没达到预期效果,可能是因为在S.albulus WG608中并不缺乏NADPH,但是在过表达了其它L-赖氨酸合成途径的关键酶后也许会增强菌株对于NADPH的需求。如果将ppc、zwf、pyc、dapA、lysA和pls组合过表达,也许可以取得更加显著的效果。
[1] SHIMA S, SAKAI S.Polylysine produced by Streptomyces[J].Agricultural and Biological Chemistry, 1977,41(9):1 807-1 809.
[2] SHIH I L, SHEN M H, VAN Y T.Microbial synthesis of poly(epsilon-lysine) and its various applications[J].Bioresource Technology, 2006, 97(9):1 148-1 159.
[3] XU D L, YAO H Q, CAO C H, et al.Enhancement of ε-poly-L-lysine production by overexpressing the ammonium transporter gene in Streptomyces albulus PD-1[J].Bioprocess and Biosystems Engineering, 2018, 41(9):1 337-1 345.
[4] WANG L, LI S, ZHAO J J, et al.Efficiently activated epsilon-poly-L-lysine production by multiple antibiotic-resistance mutations and acidic pH shock optimization in Streptomyces albulus[J].Microbiologyopen, 2019, 8(5):e00728.
[5] HIRAKI J, HATAKEYAMA M, MORITA H, et al.Improved epsilon-poly-L-lysine production of an S-(2-aminoethyl)-L-cysteine resistant mutant of Streptomyces albulus[J].Seibutsu-Kogaku Kaishi, 1998, 76(12):487-493.
[6] 刘春梅, 李树, 董传亮, 等.Genome shuffling技术改造ε-聚赖氨酸重组菌Streptomyces sp.FeeL-1[J].食品与发酵工业, 2012, 38(8):36-41.
LIU C M, LI S, DONG C L, et al.Genome shuffling enhanced ε-poly-L-lysine production of a recombinant Streptomyces sp.FeeL-1[J].Food and Fermentation Industries, 2012, 38(8):36-41.
[7] WANG L, CHEN X S, WU G Y, et al.Improved ε-poly-L-lysine production of Streptomyces sp. FEEL-1 by atmospheric and room temperature plasma mutagenesis and streptomycin resistance screening[J].Annals of Microbiology, 2015, 65(4):2 009-2 017.
[8] HAMANO Y, NICCHU I, SHIMIZU T, et al.ε-Poly-L-lysine producer, Streptomyces albulus, has feedback-inhibition resistant aspartokinase[J].Applied Microbiology and Biotechnology, 2007, 76(4):873-882.
[9] XU Z X, CAO C H, SUN Z Z, et al.Construction of a genetic system for Streptomyces albulus PD-1 and improving poly(ε-L-lysine) production through expression of Vitreoscilla hemoglobin[J].Journal of Microbiology and Biotechnology, 2015, 25(11):1 819-1 826.
[10] YAMANAKA K, HAMANO Y, OIKAWA T.Enhancement of metabolic flux toward ε-poly-L-lysine biosynthesis by targeted inactivation of concomitant polyene macrolide biosynthesis in Streptomyces albulus[J].Journal of Bioscience and Bioengineering, 2020, 129(5):558-564.
[11] WANG A, TIAN W, CHENG L, et al.Enhanced epsilon-poly-L-lysine production by the synergistic effect of ε-poly-L-lysine synthetase overexpression and citrate in Streptomyces albulus[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:288.
[12] LI W C, LYU J G, DONG T Y, et al.Effects of amino acids and overexpression of dapA gene on the production of ε-poly-L-lysine by Streptomyces diastatochromogenes strains[J].Current Microbiology, 2021, 78(7):2 640-2 647.
[13] YAMANAKA K, KITO N, IMOKAWA Y, et al.Mechanism of ε-poly-L-lysine production and accumulation revealed by identification and analysis of an epsilon-poly-L-lysine-degrading enzyme[J].Applied and Environmental Microbiology, 2010, 76(17):5 669-5 675.
[14] XU J Z, WU Z H, GAO S J, et al.Rational modification of tricarboxylic acid cycle for improving L-lysine production in Corynebacterium glutamicum[J].Microbial Cell Factories, 2018, 17(1):105.
[15] YOKOTA A, SAWADA K, WADA M.Boosting anaplerotic reactions by pyruvate kinase gene deletion and phosphoenolpyruvate carboxylase desensitization for glutamic acid and lysine production in Corynebacterium glutamicum[J].Advances in Biochemical Engineering/Biotechnology, 2017,159:181-198.
[16] NAERDAL I, NETZER R, ELLINGSEN T E, et al.Analysis and manipulation of aspartate pathway genes for L-lysine overproduction from methanol by Bacillus methanolicus[J].Applied and Environmental Microbiology, 2011, 77(17):6 020-6 026.
[17] BECKER J, KLOPPROGGE C, HEROLD A, et al.Metabolic flux engineering of L-lysine production in Corynebacterium glutamicum-over expression and modification of G6P dehydrogenase[J].Journal of Biotechnology, 2007, 132(2):99-109.
[18] CHEN Z, BOMMAREDDY R R, FRANK D, et al.Deregulation of feedback inhibition of phosphoenolpyruvate carboxylase for improved lysine production in Corynebacterium glutamicum[J].Applied and Environmental Microbiology, 2014, 80(4):1 388-1 393.
[19] ITZHAKI R F.Colorimetric method for estimating polylysine and polyarginine[J].Analytical Biochemistry, 1972, 50(2):569-574.
[20] 谢志鹏, 徐志南, 郑建明, 等.靛酚蓝反应测定发酵液中的氨态氮[J].浙江大学学报(工学版), 2005, 39(3):437-439;444.
XIE Z P, XU Z N, ZHENG J M, et al.Determination of ammonium nitrogen in fermentation broth through indophenol blue reaction[J].Journal of Zhejiang University(Engineering Science), 2005, 39(3):437-439;444.
[21] ARENZ A, STOJICIC N, LAU P, et al.Suitability of commonly used housekeeping genes in gene expression studies for space radiation research[J].Advances in Space Research, 2007, 39(6):1 050-1 055.
[22] O‘REGAN M, THIERBACH G, BACHMANN B, et al.Cloning and nucleotide sequence of the phosphoenolpyruvate carboxylase-coding gene of Corynebacterium glutamicum ATCC13032[J].Gene, 1989, 77(2):237-251.
[23] 汪泽, 王开方, 胡扬帆, 等.过表达pls基因结合前体流加提高小白链霉菌ε-聚赖氨酸产量[J].食品与发酵工业, 2020, 46(14):1-6.
WANG Z, WANG K F, HU Y F, et al.Enhanced ε-poly-L-lysine production through overexpression of pls gene combined with precursor feeding in Stretomyces albulus[J].Food and Fermentation Industries, 2020, 46(14):1-6.