克罗诺杆菌(Cronobacter.spp)是肠杆菌科的革兰氏阴性菌,一般来说,克罗诺杆菌抗逆性较强,可在酸(pH<3.9)、高盐浓度(10%NaCl)和干燥等环境存活,因此,在食品加工环境和食品如奶粉、米粉、肉制品、奶酪、香料、果蔬制品中都有检出[1]。研究表明,在细菌演化过程中,一些菌种变得对人类具有致病性,如C.sakazakii可导致脑膜炎、败血症和坏死性小肠结肠炎等疾病,主要影响免疫力低下的人群,特别是婴儿、老年人和一些有免疫缺陷的人[2]。现在已知的克罗诺杆菌的致病途径主要是通过促进组织粘连,宿主的入侵以及与宿主细胞的损伤等,许多相关毒力基因相继被报道[3-4],如编码外膜蛋白X(outer membrane protein X)的基因OmpX,外膜蛋白X参与宿主细胞的侵袭,抵御宿主细胞防御机制,编码具有溶血功能的蛋白溶血素(hemolysin)的基因hly、血浆纤溶酶原激活物(Cronobacter plasminogen activator)的基因cpa、免疫相关蛋白(siderophore-interacting protein)的基因sip等。此外,食品链多个环节过度使用或滥用抗生素引发的细菌耐药性也是不容忽视的问题,直接影响细菌感染后的治疗效果[5],进一步研究克罗诺杆菌分离株的毒力基因和耐药性情况十分必要。
西双版纳州位于云南省南端,处于澜沧江-湄公河的中心位置,与老挝、缅甸接壤,和泰国、越南近邻,是我国面向东南亚的重要枢纽,拥有国家级重点开发开放试验区、跨境经济合作区、对外开放口岸。边境贸易也是西双版纳州经济发展的一大特色,目前由“边民互市贸易”和“边境小额贸易”两种形式组成,西双版纳州的进口食品来源广、销售渠道复杂(进口食品店、农贸市场、边民互市点、集市和路边摊等场所),在运输、销售和贮存过程中可能导致食品品质下降或微生物污染[6]。本研究通过对西双版纳进口食品进行克罗诺杆菌的检测,探究其毒力基因携带情况和耐药性,以期为克罗诺杆菌引起的食源性疾病的防控监测提供参考。
1 材料与方法
1.1 材料与仪器
根据昆明海关对边贸进口食品监测数据,通过随机采样的方式采集西双版纳进口食品店、批发市场、口岸农贸市场等地所售进口的即食食品,共计13类食品172份。本次采样的样品中以糖果、水果制品、饼干3类样品为主,占样品的比例分别为31.81%,22.09%和15.11%。
1.1.1 试验试剂
血琼脂平板、胰酪大豆胨琼脂培养基(TSA),广东环凯生物科技有限公司;阪崎肠杆菌显色培养基,法国科玛嘉;GN鉴定卡,梅里埃诊断产品(上海)有限公司;细菌DNA提取试剂盒,北京卓诚惠生公司;药敏纸片,杭州滨河微生物试剂有限公司。
1.1.2 试验设备
AC2-5S1生物安全柜,ESCO Airstream 公司;LTI-700 W恒温培养箱,BOXUN公司;Bagmixer 400cc均质器,法国INTERSCIENCE公司;PREVI Color Gram全自动革兰氏染片仪、VITEK 2 compact system全自动微生物鉴定系统,生物梅里埃公司。
1.2 实验方法
1.2.1 克罗诺杆菌分离和生化鉴定
样品均在洁净条件下进行前处理,根据GB 4789.40—2016《食品安全国家标准 食品微生物学检验 克罗诺杆菌属(阪崎肠杆菌) 检验》,再挑取经形态学鉴定后的可疑单菌落,用无菌盐水制备成浊度为5.0~6.3的菌悬液,使用全自动微生物鉴定系统 VITEK 2 compact 的GN检验卡进行鉴定。
1.2.2 菌株16S rDNA分析
将分离得到的克罗诺杆菌菌株进行16S rDNA测序,引物序列:27F(5′-AGAGTTTGATCCTGGCTCAG-3′),1492R(5′-GGCTACCTTGTTACGACTT-3′),PCR反应体系50μL,其中,DNA模板1μL,MIX 45μL,上下游引物各2μL。PCR反应条件为:98 ℃预变性2 min,(98 ℃,10 s,56 ℃,30 s,72 ℃,10 s)×35,72 ℃,5 min。PCR产物纯化后用相应的扩增引物进行双向测序,用ContigExpress软件拼接测序结果,得到细菌16S rDNA基因序列。所得序列提交GENBANK进行BLAST比对,对种属鉴定结果进一步验证。
1.2.3 体外毒力基因检测
以提取的DNA为模板,对克罗诺杆菌的4种毒力基因进行扩增,引物序列及扩增片段大小见表1。引物均由北京擎科生物科技有限公司昆明分公司合成。
表1 毒力基因引物信息
Table 1 The primers of virulence gene

目的基因引物引物序列(5′-3′)产物长度/bpompXompX-FCACCATGAAAAAAATTGCATGTCTTTCAG700ompX-RGAAGCGGTAACCCACACCTGCcpacpa-FCTAGGGCGATGATAGCTGCCTCCG1 015cpa-RCTAGGGGGAACAGCCACGAGGAAAhlyhly-FCTAGGGTAACGGACTGTCACAGAT880hly-RCTAGGAAGAAGCGTAAGCGTCTGAsipsip-FCTAGGCAAAAGAATCGACAAAGGG934sip-RCTAGGTTGTTGTCTTTATCCGTTC
1.2.4 药物敏感实验
根据美国临床实验室标准研究所(Clinical and Laboratory Standards Institute,CLSI)的标准指南 CLSI M100[9],采用Kirby-Bauer纸片扩散法对分离出的13株克罗诺杆菌进行药物敏感性试验,所用的抗生素有5类14种,以大肠杆菌 ATCC 25922 和金黄色葡萄球菌 ATCC 25923 作为质控菌,不同抗生素的抑菌圈直径标准如表2所示。
表2 不同抗生素的抑菌圈直径标准
Table 2 Antibacterial zone diameter interpretive criteria of different antibiotics

种类抗生素纸片药物含量/μg抑菌圈直径/mm敏感/S中介/I耐药/R氨苄西林(ampicillin,AMP)10> 2928~29<28阿莫西林(amoxicillin,AMX)20>1813~18<13头孢他啶(ceftazidime,CAZ)30>2118~21<18β-内酯酰胺类亚胺培南(imipenem,IPM)10>1613~16<13头孢噻肟(cefotaxime,CTX)30>2622~26<22头孢噻吩(cefalotin,CFT)30>2214~22<14替卡西林-克拉维酸(ticarcillin-clavulanate,TIC)10>2014~20<14喹诺酮类环丙沙星(ciprofloxacin,CIP)5>2115~21<15萘啶酸(nalidixic acid,NAL)30>1913~19<13氯霉素类氯霉素(chloramphenicol,CHL)30>1812~18<12磺胺类复方新诺明(sulfamethoxazole/trimethoprim SXT)23.75>1610~16<10卡那霉素(kanamycins,KAN)30>1813~18<13氨基糖苷类链霉素(streptomycin,SM)25>1511~15<11庆大霉素(gentamicin,GEN)10>1512~15<12
1.2.5 全基因组测序及分析
按照试剂盒的说明书提取细菌基因组DNA,采用分光光度法和琼脂糖凝胶电泳法对基因组DNA浓度、纯度以及完整性进行检验,结果达到要求后再进行建库,库检合格后,按照目标下机数据量,使用Sequel Binding Kit 2.0、Sequel Sequencing Kit 2.1与Sequel SMRT Cell 1M v2进行 PacBio测序,并用SMRT LINK 5.0软件进行数据的处理。使用Canu软件纠正读取和修剪可疑区域,将数据组装成高质量的基因组序列。将基因与已知的数据库(NR、Swiss-Prot、COG、KEGG、GO、pfam、TrEMBL、VFDB、CARD)比对(挑选最好的比对结果,最高的identity和最多hit)对现有蛋白做功能预测和注释。
2 结果与分析
2.1 克罗诺杆菌的检出情况
从所采集的172份样品中检出阳性样品5份,分别从糖果制品、糕点、饼干和饮料中检出,检出率为2.9%(5/172),共分离出13株菌株(表3)。如图1所示,这些菌株在阪崎肠杆菌显色培养基生长情况良好,菌落呈蓝绿色,且革兰氏镜检结果为革兰氏阴性杆菌,使用VITEK 2 compact GN卡对菌株的鉴定结果为克罗诺杆菌,每株菌的鉴定结果的置信栏均显示为“极好的鉴定”。综上,可判定其为克罗诺杆菌,由此可见,西双版纳市售进口食品中存在一定的克罗诺杆菌污染风险。

a-菌株在培养基的生长状况;b-革兰氏染色结果
图1 克罗诺杆菌菌株形态学结果
Fig.1 Morphological results of Cronobacter spp. isolates
表3 各类样品中克罗诺杆菌的检出情况
Table 3 Detection results of Cronobacter spp.in samples

样品种类样品数量/份构成比/%阳性数量/份检出率/%检出菌株数/株饼干2615.1213.82炒货食品及坚果制品63.4900.00方便食品31.7400.00蜂产品21.1600.00糕点126.9818.34粮食加工品52.9100.00薯类和膨化食品116.400.00水产制品52.9100.00水果制品3822.0900.00糖果制品5330.8123.85饮料116.419.12总计172-52.913
2.2 16S rDNA分子鉴定结果
将拼接好的序列与NCBI数据库进行对比,与西双版纳进口食品中分离得到的克罗诺杆菌序列相似度最高的菌株如表4所示,13株菌株与参考菌株基因覆盖率均为100%,E值也都为0.0,BQ1、BQ2、BQ4、BQ5、BQ6和BQ12与C.sakazakii 菌株的序列一致性为100%,其他6株菌株序列与C.sakazakii的相似度也>99.79%,初步推定13株菌株为C.sakazakii。
表4 16S rDNA 分子鉴定结果
Table 4 The identification result of the 16S rDNA PCR

菌株编号序列长度/bp相似度最高菌株(登录号)覆盖率/%相似度/%BQ11 360Cronobacter sakazakii strain crj03(JF330153.1)100100BQ21 370Cronobacter sakazakii strain 05CHPL18(GU122187.1)100100BQ31 397Cronobacter sakazakii strain WJ0121(KC818142.1)10099.79BQ41 381Cronobacter sakazakii strain G4083(HQ880405.1)100100BQ51 380Cronobacter sakazakii strain WJ2280(KC818219.1)100100BQ61 365Cronobacter sakazakii strain KYU34(GU227668.1)100100BQ71 372Cronobacter sakazakii strain PM107(KF360280.1)10099.93BQ81 343Cronobacter sakazakii strain BW190102(MT476371.1)10099.93BQ91 373Cronobacter sakazakii strainWJ2469(KC818231.1)10099.93BQ101 361Cronobacter sakazakii strainFCC061(KF360269.1)10099.93BQ111 366Cronobacter sakazakii strain BDCSS035(KU364486.1)10099.93BQ121 293Cronobacter sakazakii strain PM468(KF360281.1)100100BQ131 382Cronobacter sakazakii strain BDCSS014(KU364465.1)10099.93
2.3 体外毒力基因检测结果
毒力基因PCR检测结果如表5所示,样品中分离出的13株克罗诺杆菌均检测出毒力基因,毒力基因携带率为100%,13株分离菌株BQ1~BQ13的毒力基因携带模式均为ompX-cpa-hly,且都未检出免疫相关蛋白编码基因sip。
表5 克罗诺杆菌分离菌株毒力基因分布
Table 5 Distribution of virulence genes of Cronobacter spp. isolates
菌株ompXcpahlysipBQ1~BQ13+++-
注:“+”表示检出阳性,“-”表示检出阴性
2.4 药物敏感性检测结果
根据抑菌圈的大小可以判断菌株对本次供试抗生素的耐药情况。从表6和表7可知,在13株克罗诺杆菌菌株中,有9株细菌对本次实验使用的抗生素存在耐药性,其中,对头孢噻吩(CFT)的耐药性最强,耐药率为69.23%(9/13),对卡那霉素(KAN)、链霉素(SM)和庆大霉素(GEN)表现为中介的菌株较多,而对氨苄西林(AMP)、阿莫西林(AMX)、替卡西林(TIC)、氯霉素(CHL)等抗生素,菌株则普遍表现敏感。如表7所示,存在3种耐药谱,有7株菌株的耐药谱为CFT,占比53.85%,其他2株菌株BQ2和BQ10的耐药谱分别为AMP-CFT和CTX+CFT+KAN。
表6 菌株药物敏感性检测结果
Table 6 The result of drug sensitivity test of the isolates

菌株编号AMPAMXTICCTXCFTCAZIPMGENKANSMSXTCHLNALCIPBQ119.3123.1226.0426.767.1625.223.9415.1415.3415.1323.7921.6823.6826.84SSSSRSSSISSSSSBQ28.0623.7126.3424.787.1221.2229.0914.2514.314.0823.5723.6220.8225.95RSSIRSSIIISSSSBQ318.9123.826.8525.27.1420.6328.7914.81615.0322.121.9421.9325.16SSSIRISIISSSSSBQ418.8625.1825.4328.887.1325.2827.0216.716.7616.6525.6423.9724.1330.31SSSSRSSSISSSSSBQ518.7625.0924.8426.4424.8823.9330.1315.0914.2515.5923.4618.8322.5927.96SSSSSSSSISSSSSBQ619.2924.1527.5726.619.2423.0428.1514.4514.6213.9823.5719.7324.9629.92SSSSRSSIIISSSSBQ718.9623.6728.2325.87.1322.4128.413.4514.9814.2325.7723.8728.6734.6SSSIRSSIIISSSSBQ820.4724.9126.6526.347.1221.5528.8617.0816.3116.526.5621.5427.3133.23SSSSRSSSISSSSSBQ919.4327.0126.3324.797.1521.4330.3917.617.3116.4124.6219.8426.7634.09SSSIRSSSISSSSSBQ1018.2524.7225.3420.777.1221.1726.3313.9112.9513.2720.3621.5222.7630.44SSSRRSSIRISSSSBQ1120.5725.6827.4826.0923.1619.8126.614.4316.9415.3123.4721.6124.32786.3SSSSSISIISSSSSBQ1218.5424.4928.0324.9426.1120.726.3315.4216.9513.2824.5222.126.2432.94SSSISISSIISSSSBQ1318.3422.924.5425.9923.1524.826.5613.9514.9214.6524.7721.5222.6823.26SSSISSSIIISSSS
注:抑菌圈大小单位为mm;“S”代表敏感,“I”代表中介,“R”代表耐药
表7 克罗诺杆菌菌株耐药谱
Table 7 Resistance spectrum of Cronobacter spp. isolates
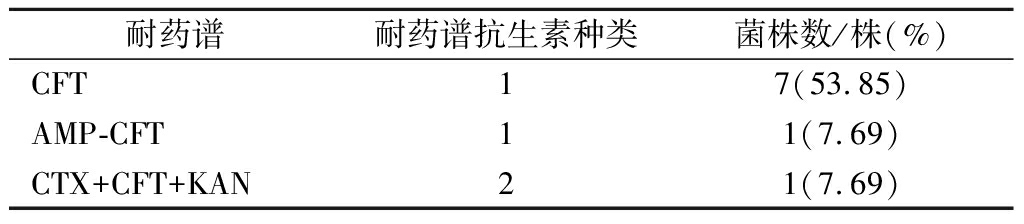
耐药谱耐药谱抗生素种类菌株数/株(%)CFT17(53.85)AMP-CFT11(7.69)CTX+CFT+KAN21(7.69)
2.5 BQ10全基因组分析
为进一步了解本次试验菌株的分子特征,对BQ10进行全基因组测序。组装后的BQ10Z基因组大小为4.75 Mb,GC 含量为54.69%,共预测出4 499个编码基因,平均长度946 bp,预测得到17个rRNA (包括5S、16S、23S)和83个tRNA。另外还含有3个质粒,大小分别为0.085、0.053、0.023 Mb,通过MLST在线网站(https://cge.cbs.dtu.dk/services/MLST/)分析,BQ10的序列型为ST431。菌株BQ10共有3 464个基因具有COG功能分类,2 534个基因在GO数据库中被注释到,KEGG分析显示,共3 059个基因富集在109条代谢通路中。
2.5.1 BQ10的毒力基因分析
通过与VFDB数据库比对,BQ10基因组中预测到612个毒力基因,如表8所示,其中发现了157个黏附和入侵相关基因,如OmpA、OmpX、fliA、flgC等基因,OmpA和OmpX基因可改善阪崎肠杆菌的细胞黏附能力,细菌鞭毛相关基因在黏附和侵袭中起着重要作用[7]。有32个分泌系统类别的基因,其中,Type VI相关的基因最多,有21个,而VI型分泌系统(T6SS)是一种多功能蛋白质分泌机制,能向靶细胞输送溶菌效应物,可破坏宿主细胞结构[8]。在铁摄取类别中,发现了103个负责产生金属结合蛋白的基因,如HitABC、FbpABC、Ent等,铁是影响克罗诺杆菌在血液中存活的重要因素,对其致病性至关重要[4]。此外,还发现了29个与毒素相关的基因,如hlyD、dppF、mdlA,仍未发现sip基因,这与体外毒力基因实验结果一致。
表8 BQ10 的毒力基因预测分类统计表
Table 8 Virulence genes of strain BQ10 predicted by VFDB
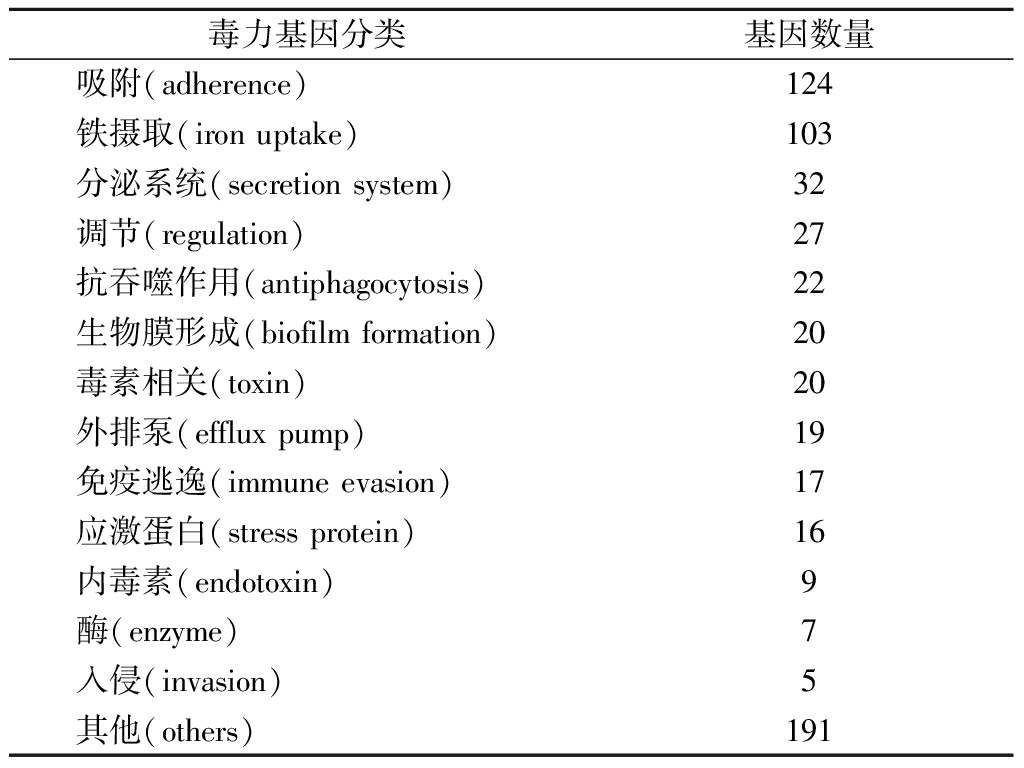
毒力基因分类基因数量吸附(adherence)124铁摄取(iron uptake)103分泌系统(secretion system)32调节(regulation)27抗吞噬作用(antiphagocytosis)22生物膜形成(biofilm formation)20毒素相关(toxin)20外排泵(efflux pump)19免疫逃逸(immune evasion)17应激蛋白(stress protein)16内毒素(endotoxin)9酶(enzyme)7入侵(invasion)5其他(others)191
2.5.2 BQ10的毒力基因分析
通过对与CARD数据库进行比对,对菌株BQ10携带多种类型的耐药基因进行预测,结果如表9所示,基因组上与磷霉素、β内酰胺类、氨基糖苷类、喹诺酮类等抗生素相关基因的核酸一致性为85.25%~100%,主要的耐药机制为抗生素外排(antibiotic efflux),如与其相关的耐药基因有ACT-12、CRP、baeR、H-NS、msbA、emrR、oqxA、rsmA,也有部分基因通过编码使抗生素失活(antibiotic inactivation)的酶类如ACTβ-内酰胺酶(ACTbeta-lactamase)、磷霉素硫醇转移酶(fosfomycin thiol transferase)从而产生耐药性,还有通过编码细菌孔蛋白以降低对β-内酰胺类抗生素的渗透性(reduced permeability to antibiotic)从而达到耐药效果的基因marA。
表9 克罗诺杆菌BQ10的耐药基因情况
Table 9 Basic information of antibiotic resistance genes in BQ10

耐药基因耐药基因家族相关抗生素耐药机制核酸一致性/%FosA2磷霉素硫醇转移酶类(fosfomycin thiol transferase)磷霉素抗生素失活100ACT-12ACTβ-内酰胺酶类(ACT beta-lactamase)碳青霉烯类,头孢菌素类,头孢霉素类抗生素失活99.48CRP耐药结节细胞分化超家族(resistance-nodulation-cell division, RND)大环内酯类,氟喹诺酮类抗生素外排99.05baeR耐药结节细胞分化超家族氨基糖苷类,氨基香豆素类抗生素外排95.82H-NS主要协同转运蛋白超家族(major facilitator super family, MFS)大环内酯类,氟喹诺酮类,头孢菌素,头孢霉素,四环素类抗生素外排95.59msbAATP-结合盒转运蛋白家族(ATP-binding cassette, ABC)硝基咪唑类抗生素外排94.67emrR主要协同转运蛋白超家族氟喹诺酮类抗生素外排94.29marA耐药结节细胞分化超家族, 对β-内酰胺的渗透性低的一般细菌孔蛋白(general bacterial porin with reduced permeability to beta-lactams)氟喹诺酮类,碳青霉烯类、头孢菌素类,甘草环素类,头孢霉素,四环素类,利福霉素,青霉烯类抗生素外排, 降低抗生素渗透性93.55oqxA耐药结节细胞分化超家族氟喹诺酮类,甘草环素类,四环素类,二氨基嘧啶类,硝基呋喃类抗生素外排91.05
3 结论与讨论
近几年,西双版纳州与周边国家贸易往来密切的同时,进口食品贸易量也在不断提升,由于进口食品量大,监管范围广,技术、标准、地域差异等因素,进口食品安全仍是难题。克罗诺杆菌广泛分布于自然界中,具有较强的生存能力,在多种食品中被检出。本文对西双版纳进口食品中的克罗诺杆菌进行了检测,从5份阳性样品中分离得到13株克罗诺杆菌疑似菌,通过VITEK 2 compact和16S rDNA进一步分析鉴定,确定13株菌均为克罗诺杆菌,初步推定为Cronobacter sakazakii,从饼干、糕点、糖果等食品中检出克罗诺杆菌,表明这些食品存在被污染的风险,赵丽娜等[9]在上海口岸采集七大洲不同国家包括生肉、冰鲜水产品、乳粉、预包装食品等4类1 511份进口食品中,在乳粉中检出克罗诺杆菌,检出率为0.99%(3/302),预包装食品未检出,进口食品致病菌的污染问题仍需要引起一定的重视。
对10株分离菌株进行毒力基因检测的结果显示,所有菌株全部携带有毒力基因,具有一定的致病潜力,其中ompX、cpa和hly基因全部检出,检出率高达100%,而免疫相关蛋白编码基因sip未检出,该结果与罗梦幽等[10]研究结果一致,而HOLY等[11]对36 株临床克罗诺杆菌菌株的毒力基因(ompA、inv、sip、aut、hly、fliC、cpa)进行了检测,基因ompA和fliC的携带率为100%,其他基因的检出情况为sip(97%), hly(92%), aut(94%),cpa(67%)和inv(69%),这与本文的结果有些不同,但即使样品种类存在差异、采集样品的地区不同,但是克罗诺杆菌毒力基因分布仍会存在一定相似性。菌株BQ10全基因测序结果也可印证菌株毒力基因的情况。虽然人们对其致病性已有一定的了解,如外膜蛋白是克罗诺杆菌的主要毒力标志物,但对克罗诺杆菌的致病机理和病原学等特性研究尚浅,如各菌株对宿主吸附、侵袭力如何,致病过程是怎样等问题仍需继续研究。
目前,我国食源性耐药菌引发的疾病和耐药率呈现快速上升趋势,给人体健康造成潜在健康风险[12]。目前在多种食品中都已检测到抗生素耐药菌的存在,如研究人员从多种食品中分离出了多种耐药菌如副溶血性弧菌、金黄色葡萄球菌、沙门氏菌等致病菌[13-14]。本次西双版纳进口食品中分离得到的菌株主要是对头孢噻吩(CFT)表现出明显的耐药性,对庆大霉素(GEN)、卡那霉素(KAN)和链霉素(SM)药物敏感性则表现为中介的菌株较多,对本次试验的其他抗生素菌株普遍表现为敏感,本实验虽未发现多重耐药菌株,但仍然需要对克罗诺杆菌的耐药性问题保持关注,LI等[15]对从水产品中分离出的33株克罗诺杆菌药敏性结果显示菌株对头孢菌素的耐药性最高(84.8%),其次是四环素(6.1%)、甲氧苄啶(3.0%)和氯霉素(3.0%)。CARVALHO等[16]对分离自婴儿配方奶粉的72株克罗诺杆菌分离株进行了测试,发现有68株菌对头孢唑林耐药,7株菌对阿莫西林耐药,4株菌对头孢噻肟耐药,1株菌对链霉素耐药。大量证据表明,抗生素耐药菌和抗生素耐药基因会破坏肠道菌群的稳定,改变菌群组成和影响免疫功能[17-18]。食品的全球化贸易进一步加剧了细菌耐药性的全球性传播,在世界多个国家如美国、中国、日本、尼日利亚等地检测到含有喹诺酮类、β-内酰胺类、磺胺类耐药基因的致病菌[19-21]。菌株BQ10全基因组测序的结果表明,菌株BQ10含有多种毒力基因,抗生素外排为主要耐药机制。通过对食源性致病菌进行全基因组测序,可以对菌株进行更深入的研究,如预测其毒力基因和耐药基因携带情况,为云南进口食品中食源性致病菌风险评估、预警以及采取相关防控措施提供一定依据。
[1] LING N, JIANG Y J, ZENG H Y, et al.Advances in our understanding and distribution of the Cronobacter genus in China [J].Journal of Food Science, 2021, 86(2):276-283.
[2] COSTA P V, DE SIQUEIRA R M, ROSA GUIMAR ES A C, et al.Cytotoxicity profile of Cronobacter species isolated from food and clinical specimens in Brazil [J].Journal of Applied Microbiology, 2021, 130(5):1 758-1 769.
ES A C, et al.Cytotoxicity profile of Cronobacter species isolated from food and clinical specimens in Brazil [J].Journal of Applied Microbiology, 2021, 130(5):1 758-1 769.
[3] ARSLAN S, ERTÜRK H G.Occurrence, virulence and antimicrobial susceptibility profiles of Cronobacter spp.from ready-to-eat foods [J].Current Microbiology, 2021, 78(9):3 403-3 416.
[4] JANG H, GOPINATH G R, ESHWAR A, et al.The secretion of toxins and other exoproteins of Cronobacter: Role in virulence, adaption, and persistence [J].Microorganisms, 2020, 8(2):229.
[5] COSTA P V, VASCONCELLOS L, DA SILVA I C, et al.Multi-locus sequence typing and antimicrobial susceptibility profile of Cronobacter sakazakii and Cronobacter malonaticus isolated from corn-based farinaceous foods commercialized in Brazil [J].Food research international, 2020, 129:108805.
[6] 魏善君. 德宏口岸进口食品安全监管工作中的问题及对策研究 [D].昆明:昆明理工大学, 2020.
WEI S J. Study on the problems and countermeasures in the safety supervision of imported food at Dehong Port[D]. Kunming: Kunming University of Science and Technology, 2020.
[7] PARRA-FLORES J, ![]() O, RIFFO F, et al.Profiling the virulence and antibiotic resistance genes of Cronobacter sakazakii strains isolated from powdered and dairy formulas by whole-genome sequencing[J].Frontiers in Microbiology, 2021, 12: 694922.
O, RIFFO F, et al.Profiling the virulence and antibiotic resistance genes of Cronobacter sakazakii strains isolated from powdered and dairy formulas by whole-genome sequencing[J].Frontiers in Microbiology, 2021, 12: 694922.
[8] CUI J H, HU J R, DU X L, et al.Genomic analysis of putative virulence factors affecting cytotoxicity of Cronobacter [J].Frontiers in Microbiology, 2020,10:3104.
[9] 赵丽娜, 申进玲, 宁雪,等.2017—2019年我国进口食品食源性致病菌污染状况分析 [J].食品安全质量检测学报, 2020, 11(9):2 930-2 935.
ZHAO L N, SHEN J L, NING X,et al.Analysis on the contamination status of food-borne pathogens of imported food in China from 2017 to 2019 [J].Journal of Food Safety and Quality, 2020, 11(9):2 930-2 935.
[10] 罗梦幽, 柯旭泽, 贺苏皖,等.食品中克罗诺杆菌分离菌株生物被膜形成、耐药性及毒力基因检测 [J].食品工业科技, 2019, 40(4):106-111.
LUO M Y, KE X Z, HE S W,et al.Biofilm formation, antimicrobial susceptibility and virulence gene detection in Cronobacter spp.isolated from food sources[J].Science and Technology of Food Industry, 2019,40(4):106-111.
[11] ![]() O, CRUZ-C
O, CRUZ-C RDOVA A, XICOHTENCATL-CORTES J, et al.Occurrence of virulence factors in Cronobacter sakazakii and Cronobacter malonaticus originated from clinical samples [J].Microbial Pathogenesis, 2019, 127: 250-256.
RDOVA A, XICOHTENCATL-CORTES J, et al.Occurrence of virulence factors in Cronobacter sakazakii and Cronobacter malonaticus originated from clinical samples [J].Microbial Pathogenesis, 2019, 127: 250-256.
[12] 沙国萌, 陈冠军, 陈彤, 等. 抗生素耐药性的研究进展与控制策略[J]. 微生物学通报, 2020, 47(10):3 369-3 379.
SHA G M, CHEN G J, CHEN T, et al. Strategies for controlling antibiotic resistance: A review[J]. Microbiology China, 2020, 47(10):3 369-3 379.
[13] 李丽倩,姚琳,林才云,等.2018年贝类来源副溶血性弧菌毒力基因及耐药性分析 [J].中国食品卫生杂志, 2020, 32(4):364-370.
LI L Q, YAO L, LIN C Y, et al.Analysis of virulence genes and antimicrobial resistance of Vibrio parahaemolyticus isolated from shellfish in 2018 [J].Chinese Journal of Food Hygiene, 2020, 32(4):364-370.
[14] WANG Y T, LIN Y T, WAN T W, et al.Distribution of antibiotic resistance genes among Staphylococcus species isolated from ready-to-eat foods [J].Journal of Food and Drug Analysis, 2019, 27(4):841-848.
[15] LI C S, ZENG H Y, ZHANG J M, et al.Cronobacter spp.isolated from aquatic products in China:Incidence, antibiotic resistance, molecular characteristic and CRISPR diversity [J].International Journal of Food Microbiology, 2020, 335, 108857.
[16] CARVALHO G G, CALARGA A P, TEODORO J R, et al.Isolation, comparison of identification methods and antibiotic resistance of Cronobacter spp.in infant foods [J].Food Research International, 2020, 137: 109643.
[17] 段宇婧,吴新颜,陈则友,等.人体肠道耐药基因组的研究进展 [J].生态毒理学报, 2020, 15(4):1-10.
DUAN Y J, WU X Y, CHEN Z Y, et al.Advances in human gut resistome [J].Asian Journal of Ecotoxicology, 2020, 15(4):1-10.
[18] CHEN J, YING G G, DENG W J.Antibiotic residues in food:Extraction, analysis, and human health concerns [J].Journal of Agricultural and Food Chemistry, 2019, 67(27):7 569-7 586.
[19] MUNKHOLM L, RUBIN O.The global governance of antimicrobial resistance:A cross-country study of alignment between the global action plan and national action plans [J].Globalization and Health, 2020, 16(1):109.
[20] INNES G K, RANDAD P R, KORINEK A, et al.External societal costs of antimicrobial resistance in humans attributable to antimicrobial use in livestock [J].Annual Review of Public Health, 2020, 41: 141-157.
[21] AABED K, MOUBAYED N, ALZAHRANI S.Antimicrobial resistance patterns among different Escherichia coli isolates in the Kingdom of Saudi Arabia [J].Saudi Journal of Biological Sciences, 2021, 28(7):3 776-3 782.