潜江地处湖北省中南部、江汉平原腹地,是湖北“两圈两带”战略的重要节点城市,独特的生态经济环境使潜江地区拥有丰富的菌种资源,为该区域发酵食品的产生创造得天独厚的自然环境。鲊广椒是以二荆条辣椒和小子黄苞谷为原料,添加适量盐和胡椒发酵而成的,具有独特的风味。除了以辣椒和苞谷为原料制作的鲊广椒,潜江地区还存在一种以莲藕和苞谷为主要原料发酵制成的食品,这种发酵食品在当地被称为藕鲊。本研究中将以苞谷为原料,添加辣椒或莲藕等蔬菜混合发酵的食品统称为蔬菜鲊。目前关于鲊广椒中细菌多样性的研究相对较多,采用Illumina MiSeq高通量测序技术对当阳[1]、咸丰[2]和洪湖[3]鲊广椒进行研究,发现鲊广椒主要由隶属于厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)的细菌组成。关于藕鲊中细菌群落多样性的研究报道较少,其与鲊广椒之间相关性分析的理论基础匮乏。本研究基于潜江地区鲊广椒和藕鲊样品中细菌群落多样性与滋味品质的关联性进行分析,以期为后续鲊广椒和藕鲊之间的分析提供一定的理论基础和数据支撑。
以Illumina MiSeq高通量测序技术为代表的第二代高通量测序技术在发酵食品微生物群落结构的研究中得到广泛应用,能实现宏基因组测序、小型基因组测序和扩增子测序等[4]应用。ZANG等[5]采用Illumina MiSeq技术对样品16S rRNA和ITS1(Internal Transcribed Spacer, ITS)基因进行测序分析,研究不同发酵阶段酸鱼中微生物群落多样性,结果表明酸鱼发酵过程中微生物结构呈动态变化。田建军等[6]采用Illumina MiSeq测序技术对风干肉细菌群落组成和多样性进行研究,为保证风干肉制品的质量安全、提高风干肉制品的品质、优化风干肉的生产工艺提供理论依据。王玉荣等[7]采用MiSeq测序技术对鲊广椒的扩增区域进行研究,发现选择引物515F/907R扩增16S rRNA基因的V4~V5区较合适。电子舌作为一种模拟人体舌头的仿生仪器,可以对样品信息进行检测、识别和分类,之后用多元统计的方法对测得的数据进行分析,从而达到对不同产品的相对味觉强度进行比较分析的目的。目前,电子舌技术已广泛应用于白酒[8]、腐乳[9]和辣椒酱[10]等发酵产品的研究中。
本研究将Illumina MiSeq高通量测序技术和电子舌技术相结合,在对样品中微生物菌落结构和滋味品质进行解析的同时,进一步将两者进行关联分析。同时,采用传统分离培养的方法对样品中含有的乳酸菌进行分析。以期为后续潜江地区蔬菜鲊样品中微生物群落多样性和滋味品质的相关性分析提供一定的科学研究和菌株支持。
1 材料与方法
1.1 材料与试剂
样品采集:从湖北省潜江市菜市场采集5 个鲊广椒和5 个藕鲊样品,编号为ZG1~ZG5和OZ1~OZ5。采集的样品来自不同个体,根据其不同的生活方式与饮食习惯发酵制成,具有一定的当地特色。每份样品采集300 g左右装入无菌采样袋中,置于含有冰袋的样品采集箱中低温运回实验室。
QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒,德国QIAGEN公司;引物27F/1495R和338F/806R[其中正向引物338F前端加入7 个核苷酸标签(barcode)],武汉天一辉远生物科技有限公司合成;FastPfu Fly DNA Polymerase和5×TransStartTM FastPfu Buffer,北京全式金生物技术有限公司;电子舌内部溶液、极性溶液和参比溶液,日本Insent公司;10×PCR buffer、dNTPs Mix、rTaq DNA聚合酶、pMD18-T克隆载体、DL15000 Marker和DL2000 Marker,宝生物工程(大连)有限公司。
1.2 仪器与设备
5810R型台式高速冷冻离心机,德国Eppendorf公司;Miseq PE300型高通量测序平台,美国Illumina公司;R920型机架式服务器,美国DELL公司;SA 402B电子舌,日本Insent公司;vetiri梯度基因扩增仪,美国AB公司;DYY-12型电泳仪,北京六一仪器厂;UVPCDS8000凝胶成像分析系统,美国ProteinSimple公司。
1.3 实验方法
1.3.1 细菌宏基因组DNA提取
在样品采集后的48 h内,根据QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒中说明书的约束步骤对样品进行宏基因组DNA的提取,并使用琼脂糖凝胶电泳对其进行分析,将检测合格的DNA置于-20 ℃备用。
1.3.2 细菌16S rRNA序列扩增与Miseq测序
利用正向引物338F(5′-ACTCCTACGGGAGGCAGCA-3′)和反向引物806R(5′-GGACTACHVGGGTWTCTAAT-3′)对细菌16S rRNA基因的V4~V5区进行扩增[7],参考李大飞等[11]研究中的PCR扩增体系和扩增条件。将检测合格的PCR产物准确定量并用无菌ddH2O将产物浓度稀释至100 nmol/L后置于干冰中,冷链寄往上海美吉生物医药科技有限公司使用Miseq PE300平台进行高通量测序。
1.3.3 序列拼接和质量控制[12]
经高通量测得的原始序列(reads),根据barcode分配到对应样品并进行校正,得到各样本的有效序列。对下机数据进行拼接处理,去除其末端低质量序列,将成对的reads拼接成一条序列。选择成对序列的重叠区≥10 bp、最大错配比率≤0.2、barcode碱基无错配且引物碱基错配数≤2 bp的优质序列,其余不合格的序列予以剔除。
1.3.4 生物信息学分析
采用QIIME(V1.70)分析平台对质控后的优质序列进行生物信息学分析。通过Py NAST[13]进行校准、两步UCLUST法[14]建立分类操作单元(operational taxonomic units,OTU)聚类和Chimera Slayer去除含嵌合体序列OTU[15]等步骤,挑选细菌OTU代表性序列,根据RDP(Ribosomal Database Project,Release 11.5)[16]和Greengenes(Release 13.8)[17]数据库进行同源性比对,明确其不同分类学地位的相对含量,同时使用Fast Tree软件绘制其系统发育树,并对样品中微生物香农指数(Shannon index)和超1指数(Chao1 index)等α多样性指标进行计算;基于UniFrac距离进行主坐标分析(principal coordinate analysis,PCoA)和聚类分析(cluster analysis,CA),进而对不同样品间细菌群落结构进行β多样性分析。
1.3.5 基于电子舌技术对样品滋味品质的评价
参考王玉荣等[18]的方法对本研究中蔬菜鲊样品进行处理,基于电子舌技术得到样品鲜味、咸味、酸味、涩味和苦味5 个基本味及后味A(涩味的回味)、后味B(苦味的回味)和丰度(鲜味的回味)3个基本味回味的相对强度值。每个样品测定4 次,取后3 次的测定数据作为此次研究的原始数据。
1.3.6 乳酸菌的分离与鉴定
采用传统培养方法[19]分离潜江蔬菜鲊样品中的乳酸菌。使用含1.5%(质量分数)CaCO3的MRS固体培养基分离纯化乳酸菌,30 ℃条件下厌氧培养,挑选含有透明圈且菌落形态不同的单菌落进行3次划线,之后将过氧化氢酶实验阴性且革兰氏染色阳性的菌株利用甘油保藏法进行保藏。采用十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB)法[20]对纯化菌株进行DNA提取,参照KHEMARIYA等[21]PCR扩增方法对菌株进行16S rRNA扩增,扩增产物经过检测、清洁、连接、转化和鉴定,挑取阳性克隆子送往武汉天一辉远生物科技有限公司进行测序,然后在NCBI数据库(https://www.ncbi.nlm.nih.gov/)中进行同源性比对并申请登录号。
1.3.7 多元统计学分析
使用R4.0.0软件绘制样品中优势细菌群落相对含量气泡图、乳酸菌含量热图、α多样性小提琴图和优势细菌属与滋味品质的普式分析图,使用Origin 9.8软件绘制样品间PCoA图和滋味品质箱型图,使用Mega7.0软件绘制样品间CA图,其他数据在Excel中进行分析处理。
2 结果与分析
2.1 蔬菜鲊优势细菌门和优势细菌属分析
若隶属于某一门或属的细菌在其对应样品中的平均相对含量>1%,则将其定义为优势细菌门或属[22],平均相对含量<1%的定义为其他(other),无法鉴定到门或属的定义为不可鉴定(unclassified)。蔬菜鲊样品中优势细菌门和优势细菌属的相对含量如图1所示。
由图1-A可知,在门水平上,鲊广椒中的细菌类群主要隶属于Firmicutes和Proteobacteria,其平均相对含量分别为97.99%和1.40%;藕鲊中的细菌类群主要隶属于Firmicutes、Actinobacteria和Proteobacteria,其平均相对含量分别为72.09%、18.78%和8.82%。
由图1-B可知,在属水平上,鲊广椒中优势细菌主要隶属于乳酸杆菌属(Lactobacillus)和片球菌属(Pediococcus),其平均相对含量分别为95.58%和1.52%;藕鲊中优势细菌主要隶属于Lactobacillus、葡萄球菌属(Staphylococcus)、嗜热油菌属(Thermoleophilum)、棒状杆菌属(Corynebacterium)和魏斯氏菌属(Weissella),其平均相对含量分别为49.37%、11.19%、9.44%、8.32%和5.21%。

A-优势细菌门;B-优势细菌属
图1 蔬菜鲊中优势细菌门和属相对含量的气泡图
Fig.1 Bubble plot of relative contents of dominant bacterial phyla and genera in vegetables Zha-Chili samples
为进一步探究鲊广椒和藕鲊样品中优势细菌门和优势细菌属的差异性,本研究对鲊广椒和藕鲊样品中优势细菌类群进行差异分析,结果如图2所示。
由图2-A可知,在门水平上,鲊广椒和藕鲊样品中优势细菌类群Firmicutes和Proteobacteria差异显著(P<0.05),细菌类群Actinobacteria差异非常显著(P<0.01)。由图2-B可知,在属水平上,鲊广椒和藕鲊样品中优势细菌类群Lactobacillus和Staphylococcus差异非常显著(P<0.01),而优势细菌类群Thermoleophilum、Corynebacterium、Weissella和Pediococcus差异不显著(P > 0.05)。由此表明,蔬菜鲊样品中细菌群落结构丰富,且不同原料发酵的蔬菜鲊样品中部分细菌类群存在一定的差异。
综上可知,潜江地区蔬菜鲊样品中的优势细菌门为Firmicutes和Proteobacteria,优势细菌属为Lactobacillus,这与当阳[1]、咸丰[2]和洪湖[3]等地区的研究结果基本一致,揭示了潜江地区蔬菜鲊样品的优势细菌群落结构。值得关注的是,以不同原料发酵制成的蔬菜鲊,基于属水平细菌类群的Lactobacillus和Staphylococcus差异非常显著(P<0.01),且以辣椒和苞谷为主要原料制成的鲊广椒中特有的优势细菌属仅存有Pediococcus,以莲藕和苞谷为主要原料制成的藕鲊中特有的优势细菌属分别为Staphylococcus、Thermoleophilum、Corynebacterium和Weissella。由此推测,蔬菜鲊样品中发酵原料的不同导致其细菌群落结构存在差异,从而可能与蔬菜鲊的滋味品质存在一定的相关性。

A-门水平;b-属水平
图2 基于蔬菜鲊样品中优势细菌类群相对含量的箱型图
Fig.2 Box diagram based on the relative content of dominant bacterial groups in vegetables Zha-Chili
注:“**”代表差异极显著,“*”代表差异显著,“NS”代表差异不显著(下同)
2.2 蔬菜鲊样品差异分析
为进一步探究微生物群落结构对样品的影响,本研究利用UniFrac进行β多样性的评估分析,通过样品间的比较分析,计算其Unifrac距离并得到相关矩阵,从而比较环境样品在特定的进化谱系中是否有显著的微生物群落差异。本研究基于分类操作单元的加权UniFrac距离对样品间的差异性进行分析,结果如图3所示。

A-主坐标分析;B-聚类分析
图3 基于分类操作单元的加权UniFrac距离的 主坐标和聚类分析
Fig.3 Principal coordinates and cluster analysis based on weighted UniFrac distance of operational taxonomic units
由图3-A可知,第一主成分(PCoA1,51.28%)和第二主成分(PCoA1,20.41%)累计贡献率达71.69%,说明加权UniFrac距离的主坐标分析可以较好地反映样品的整体信息。编号为ZG1、ZG2、ZG3、ZG4和ZG5的5个鲊广椒样品分布较集中,均位于第二象限;藕鲊样品空间分布较为分散,呈一定趋势分布于第一、三、四象限。由图3-B可知,ZG1、ZG3、ZG4和ZG5两两形成一个分支且位于同侧,表明这4个鲊广椒样品细菌群落结构相似;OZ1、OZ3、OZ4和OZ5两两形成一个分支且位于同侧,表明这4个藕鲊样品细菌群落结构相似;而ZG2和OZ2各自形成分支,说明这两个样品的细菌群落结构与其他样品间存在较为明显的差异。
由此可知,主坐标分析和聚类分析结果基本一致,表明鲊广椒和藕鲊样品间微生物群落结构存在差异,且鲊广椒样品间细菌群落结构相似度更高。其中编号为ZG1、ZG3、ZG4和ZG5的蔬菜鲊样品细菌群落结构相似性较高,编号为OZ4和OZ5、OZ1和OZ3的蔬菜鲊样品细菌群落结构存在一定的相似性。
从总体上来说,蔬菜鲊样品间形成较为明显聚类还可能与某些平均相对含量较低的细菌群落相关[23]。将鲊广椒和藕鲊样品中特有的OTU和所有样品中均存在的OTU统称为独特OTU,鲊广椒和藕鲊样品中独特OTU的平均相对含量如表1所示。
由表1可知,鲊广椒和藕鲊样品中核心OTU[24]有两个,鉴定结果为Lactobacillus和Thermoleophilum,累积平均相对含量分别为4.64%和2.58%。鲊广椒中特有OTU[24]有4个,鉴定结果为Lactobacillus,累积平均相对含量为0.73%;藕鲊中特有OTU有1 个,鉴定结果为Staphylococcus,累积平均相对含量为0.09%。
表1 不同样品中独特OTU的平均相对含量
Table 1 Average relative content of unique OTU in different samples
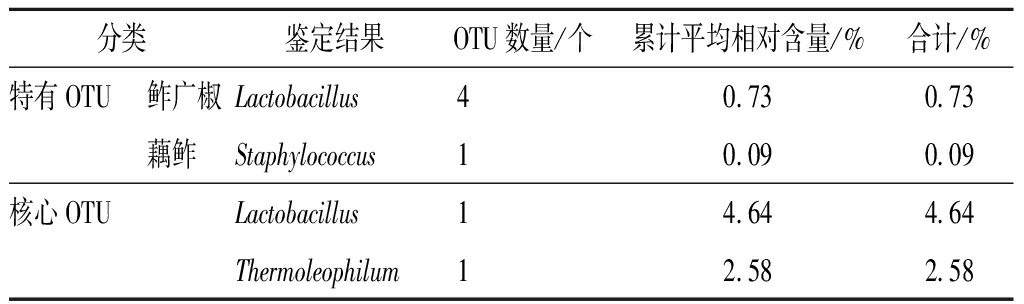
分类鉴定结果OTU数量/个累计平均相对含量/%合计/%特有OTU鲊广椒Lactobacillus40.730.73藕鲊 Staphylococcus10.090.09核心OTULactobacillus14.644.64Thermoleophilum12.582.58
由此可知,Lactobacillus存在于所有蔬菜鲊样品中,且由图1可知,Lactobacillus是鲊广椒与藕鲊样品中共有的优势细菌属,由图3可知,蔬菜鲊样品间存在较为明显的聚类趋势,这可能与Lactobacillus的相对含量有关。Thermoleophilum存在于所有OTU中,且为藕鲊中的优势细菌属,平均相对含量高于鲊广椒中;Staphylococcus为藕鲊中特有OTU的细菌属,且为藕鲊中的优势细菌属,表明Staphylococcus仅存在于藕鲊样品中。然而,目前关于Thermoleophilum和Staphylococcus的相关研究较少,其在食品中的作用有待进一步研究。
2.3 蔬菜鲊样品间滋味指标相对强度分析
为了探究样品中微生物群落结构与滋味品质的相关性,采用电子舌技术对样品的滋味品质进行可视化分析,通过Origin 9.8软件绘制箱型图。箱型图主要包含数据的6个数据节点,用以反映数据的分散情况,广泛应用于产品研究和品质评价等方面。结果如图4所示。

图4 基于电子舌分析鲊广椒与藕鲊的箱型图
Fig.4 Boxplot of Zha-Chili and lotus root based on electronic tongue analysis
图4表明,鲊广椒和藕鲊样品各滋味相对强度差异性均不显著。其中,与鲊广椒样品相比,藕鲊样品在酸味和咸味指标上的差异较为明显,可能对消费者的影响较大[25]。结合上述分析,与鲊广椒样品相比,藕鲊样品中细菌微生物群落结构呈现多样性分布,且存在相对含量较低的优势菌属和特有菌属,由此可能对样品的滋味品质产生一定的影响。
2.4 蔬菜鲊样品中分离乳酸菌与细菌群落结构分析
为进一步探究样品中菌种的分布情况,本研究使用传统培养方法对潜江地区样品中的乳酸菌进行分离鉴定,共鉴定出42 株乳酸菌,样品中不同乳酸菌含量如图5所示。

图5 鲊广椒和藕鲊样品中乳酸菌含量图
Fig.5 Content of lactic acid bacteria in Zha-Chili and Lotus root samples
由图5可知,鲊广椒中含有8 种乳酸菌,分别为L.pentosus、L.plantarum、L.farciminis、L.reuteri、W.cibaria、W.confusa 、L.mesenteroides和P.acidilactici;藕鲊中含有6 种乳酸菌,分别为L.pentosus、L.plantarum、W.paramesenteroides、W.viridescens、L.mesenteroides和L.citreum。
由于传统培养方法的条件约束[26],可能导致本研究样品中某些乳酸菌无法适应培养条件和培养温度,为避免因实验方法造成的误差,本研究进一步利用Illumina MiSeq 高通量测序技术对蔬菜鲊样品中细菌群落的丰度和多样性进行统计,并与样品中分离乳酸菌种的丰度和多样性进行比较分析,其Chao1指数和Shannon指数的小提琴图如图6所示。

A、C-基于乳酸菌分离株结果计算;B、D-基于高通量测序技术计算
图6 基于Chao1指数和Shannon指数的小提琴图
Fig.6 Violin plot based on Chao1 index and Shannon index
如图6-A和图6-B所示,基于分离乳酸菌种和细菌群落结构的水平对鲊广椒和藕鲊样品的丰度进行比较分析,结果显示一致,鲊广椒和藕鲊样品的丰度差异性不显著(P>0.05)。如图6-C和图6-D所示,基于分离乳酸菌类群和细菌群落结构的水平对鲊广椒和藕鲊样品的多样性进行比较分析,结果显示一致,鲊广椒和藕鲊样品的多样性差异不显著(P>0.05)。由此可知,本研究中由于传统培养方法的条件约束对细菌群落的丰度和多样性研究方面的影响不大,鲊广椒和藕鲊样品在丰度和多样性上的差异性均不显著(P>0.05)。
由图5亦可知,通过传统培养方法分离出P.acidilactici为ZG5中特有的乳酸菌,P.acidilactici隶属于Pediococcus,由图1可知,Pediococcus为潜江地区鲊广椒中特有的优势细菌属,由图3可知,ZG1、ZG3、ZG4和ZG5呈现较为明显的聚类趋势,表明本研究中采用传统培养方法在分离P.acidilactici的过程中存在一定的不足,P.acidilactici可能存在于多个蔬菜鲊样品中。P.acidilactici是具有益生效果的肠道益生菌[27],其在鲊广椒样品中的存在表明,鲊广椒可能对人体肠道具有一定的益生特性。
2.5 蔬菜鲊样品滋味品质与细菌群落结构分析
为进一步探究蔬菜鲊中细菌群落结构与滋味品质的相关性,本研究利用普氏分析法[28](Procrustes analysis)对优势细菌属与样品滋味品质进行可视化分析。结果如图7所示。
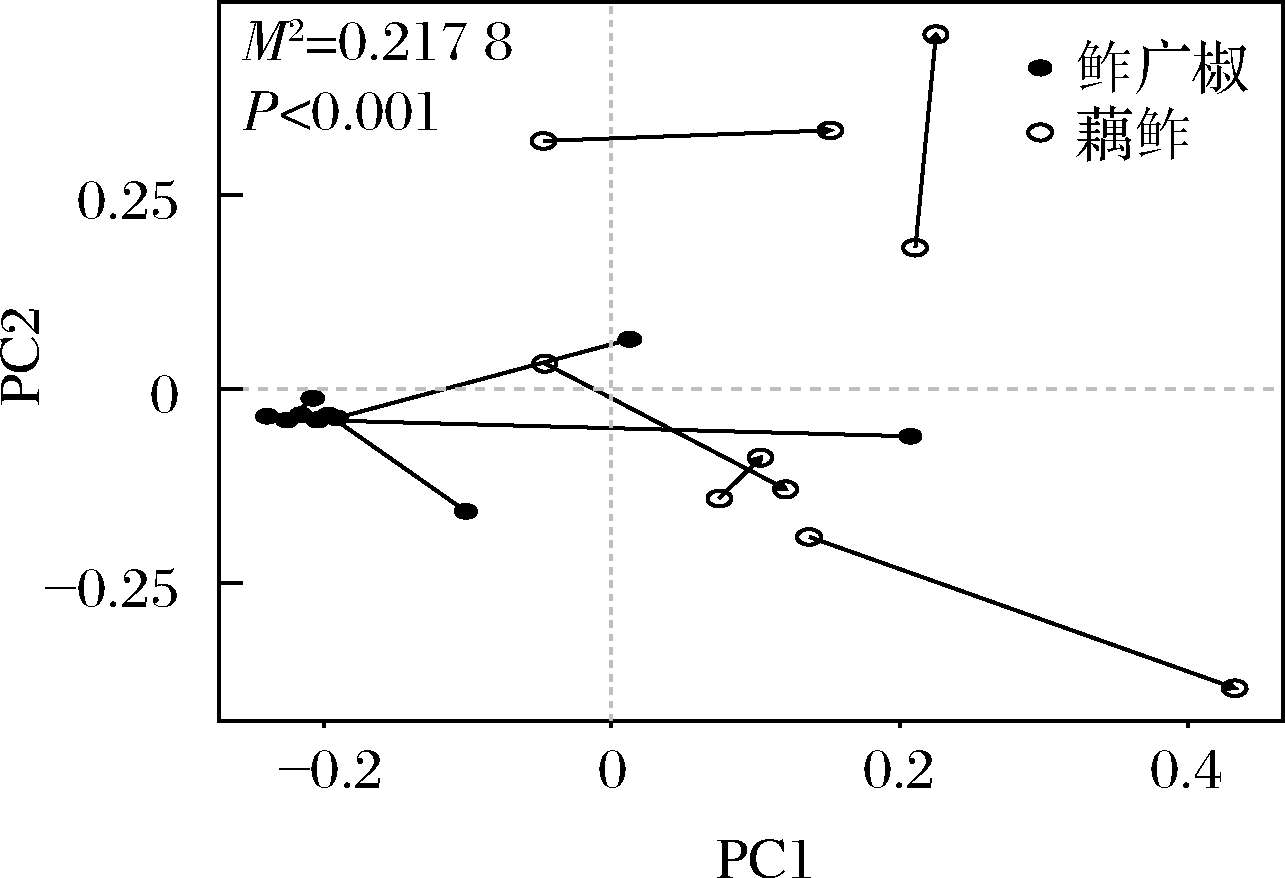
图7 基于蔬菜鲊中优势细菌属与滋味品质的普氏分析
Fig.7 Procrustes analysis based on dominant bacteria genus and taste quality in vegetables Zha-Chili
由图7可知,蔬菜鲊样品中优势细菌属与其滋味品质之间相关性极显著(P<0.001)。且由以上分析可知,以不同原料制成的蔬菜鲊,基于属水平优势细菌类群的Lactobacillus和Staphylococcus差异非常显著(P<0.01),基于电子舌技术分析其各滋味品质之间无显著差异。与鲊广椒样品相比,藕鲊样品中细菌微生物群落结构呈现多样性分布,且存在相对含量较低的优势菌属和特有菌属,由此可能对样品的滋味品质产生一定的影响。由此表明,蔬菜鲊样品中低丰度的优势菌属具有至关重要的作用,对其滋味品质的影响不可忽视。综上所述,优势细菌属对蔬菜鲊的品质,及其分类学地位具有极其重要的作用,为蔬菜鲊的产业化发展具有一定的指导作用。
3 结论
通过本研究发现鲊广椒中的优势细菌门为Firmicutes和Proteobacteria,优势细菌属主要为隶属于Firmicutes的Lactobacillus和Pediococcus。藕鲊中的优势细菌门为Firmicutes、Actinobacteria和Proteobacteria,优势细菌属为Lactobacillus、Staphylococcus、Thermoleophilum、Corynebacterium和Weissella。鲊广椒和藕鲊样品形成一定的聚类,其样品丰度和多样性差异性均不显著(P>0.05)。蔬菜鲊样品中优势细菌属与其滋味品质之间相关性极显著(P<0.001)。由此表明,潜江地区蔬菜鲊中含有丰富的细菌群落结构,对蔬菜鲊的滋味品质具有重要的影响,同时为蔬菜鲊的研究及其产业化发展提供支撑。
[1] 王玉荣, 沈馨, 董蕴, 等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械, 2018, 34(4):25-30.
WANG Y R, SHEN X, DONG Y, et al.Characterization of bacterial microflora and their functions on flavor quality [J].Food & Machinery, 2018, 34(4):25-30.
[2] 李娜, 张苗苗, 舒娜, 等.咸丰和当阳地区鲊广椒细菌群落结构差异性研究[J].中国酿造, 2020, 39(10):48-53.
LI N, ZHANG M M, SHU N, et al.Difference of bacterial community structure of Zhaguangjiao in Xianfeng and Dangyang region [J].China Brewing, 2020, 39(10):48-53.
[3] 李娜, 崔梦君, 马佳佳, 等.基于Illumina MiSeq测序和传统可培养方法的洪湖鲊广椒乳酸菌多样性研究[J].食品与发酵工业, 2021, 47(4):110-115;122.
LI N, CUI M J, MA J J, et al.Lactic acid bacterial diversity of Zha-chili in Honghu county based on Illumina Miseq sequencing and traditional culture method [J].Food and Fermentation Industries, 2021, 47(4):110-115;122.
[4] AN F Y, SUN H J, WU J R, et al.Investigating the core microbiota and its influencing factors in traditional Chinese pickles[J]. Food Research International, 2021, 147:110543.
[5] ZANG J H, XU Y S, XIA W S, et al.Dynamics and diversity of microbial community succession during fermentation of Suan yu, a Chinese traditional fermented fish, determined by high throughput sequencing[J].Food Research International, 2018, 111:565-573.
[6] 田建军, 张开屏, 杨明阳, 等.应用Illumina MiSeq测序技术比较风干肉中细菌多样性和微生物安全性[J].食品科学, 2019, 40(8):33-40.
TIAN J J, ZHANG K P, YANG M Y, et al.Comparative bacterial diversity analysis and microbial safety assessment of air-dried meat products by Illumina Miseq sequencing technology[J].Food Science, 2019, 40(8):33-40.
[7] 王玉荣, 杨成聪, 葛东颖, 等.扩增区域对鲊广椒细菌MiSeq测序的影响[J].食品科学, 2019, 40(10):134-140.
WANG Y R, YANG C C, GE D Y, et al.Influence of different amplified region on results of bacterial diversity in Zhaguangjiao, a Chinese traditional fermented chili product, by MiSeq sequencing[J].Food Science, 2019, 40(10):134-140.
[8] HU Y L, LEI X Y, ZHANG X M, et al. Characteristics of the microbial community in the production of Chinese rice-flavor Baijiu and comparisons with the microflora of other flavors of Baijiu[J]. Frontiers in Microbiology, 2021, 12:673670.
[9] 樊艳, 李浩丽, 郝怡宁.基于电子舌与SPME-GC-MS技术检测腐乳风味物质[J].食品科学, 2020, 41(10):222-229.
FAN Y, LI H L, HAO Y N.Analysis of characteristic flavor compounds of fermented bean curd using electronic tongue and solid-phase microextraction combined with gas chromatography-mass spectrometry[J].Food Science, 2020, 41(10):222-229.
[10] 沈馨, 王艳, 代凯文, 等.基于Miseq高通量测序技术的辣椒酱核心细菌类群研究[J].食品研究与开发, 2018, 39(10):151-157.
SHEN X, WANG Y, DAI K W, et al.Characterization of core bacterial microflora in chilli sauce by miseq high throughput sequencing technologies[J].Food Research and Development, 2018, 39(10):151-157.
[11] 李大飞, 赵承鑫, 贾利蓉, 等.贵州不同地区番茄红酸理化、风味和细菌群落比较分析[J].中国调味品, 2020, 45(4):59-63;68.
LI D F, ZHAO C X, JIA L R, et al.Comparative analysis of physicochemicalproperty, flavor and bacterial community of tomato sour soup(TSS)from different regions of guizhou province[J].China Condiment, 2020, 45(4):59-63;68.
[12] RODRIGUES D C, MUFTEEV M, ELLIS J.Quantification of mRNA ribosomal engagement in human neurons using parallel translating ribosome affinity purification (TRAP) and RNA sequencing[J]. STAR Protocols, 2021, 2(1):100229.
[13] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al.PyNAST:A flexible tool for aligning sequences to a template alignment[J].Bioinformatics, 2010, 26(2):266-267.
[14] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics, 2010, 26(19):2 460-2 461.
[15] HO M, MOON D, PIRES-ALVES M, et al.Recovery of microbial community profile information hidden in chimeric sequence reads[J].Computational and Structural Biotechnology Journal, 2021, 19:5 126-5 139.
[16] COLE J R, CHAI B, FARRIS R J, et al.The ribosomal database project (RDP-Ⅱ):Introducing myRDP space and quality controlled public data[J].Nucleic Acids Research, 2007, 35(1):169-172.
[17] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al.Greengenes, a chimera checked 16S rRNA gene database and workbench compatible with ARB[J].Applied and Environmental Microbiology, 2006, 72(7):5 069-5 072.
[18] 王玉荣, 代凯文, 沈馨, 等.鲊广椒真菌多样性及其对滋味品质影响的评价[J].食品科学, 2018, 39(18):173-178.
WANG Y R, DAI K W, SHEN X, et al.Characterization of fungal microflora and its influence on taste quality of zhaguangjiao, a Chinese traditional fermented chili product[J].Food Science, 2018, 39(18):173-178.
[19] WU Y, WU D D, LIU D Y, et al.The isolation and identification of Lactobacillus from naturally fermented yoghurt[J]. IOP Conference Series: Earth and Environmental Science, 2020, 565(1):012055.
[20] LAVANYA R, ARUN V.Standardization of DNA extraction protocol and screening of begomovirus in chilli and tomato plants[J]. Journal of Plant Diseases and Protection, 2019, 126(4):343-349.
[21] KHEMARIYA P, SINGH S, JAISWAL N, et al. Isolation and identification of Lactobacillus plantarum from vegetable samples[J]. Food Biotechnology, 2016, 30(1):49-62.
[22] 王琳, 胡小霞, 黄永光.茅台镇酱香型白酒不同生产轮次酿造环境的细菌菌群结构特征[J].食品科学, 2021, 42(22):185-192.
WANG L, HU X X, HUANG Y G, et al.Characteristics of bacterial microflora structure in environment samples from different rounds of Maotai-flavor liquor fermentation in Maotai town[J].Food Science, 2021, 42(22):185-192.
[23] RANGJAROEN C, RERKASEM B, TEAUMROONG N, et al.Comparative study of endophytic and endophytic diazotrophic bacterial communities across rice landraces grown in the highlands of northern Thailand[J].Archives of Microbiology, 2014, 196(1):35-49.
[24] 张振东, 尚雪娇, 董蕴, 等.丢糟窖窖泥细菌多样性评价[J].中国微生态学杂志, 2018, 30(8):882-888.
ZHANG Z D, SHANG X J, DONG Y, et al.The diversity of bacterial microflora in vinasse [J].Chinese Journal of Microecology, 2018, 30(8):882-888.
[25] 葛东颖, 王玉荣, 向凡舒, 等.荆州地区鲊广椒乳酸菌多样性解析及其分离株发酵特性的评价[J].中国食品添加剂, 2019, 30(4):72-79.
GE D Y, WANG Y R, XIANG F S, et al.Analysis of lactic acid bacterial diversity in Zhaguangjiao from Jingzhou and evaluation of fermentation characteristics of isolated strains [J].China Food Additives, 2019, 30(4):72-79.
[26] 林莹, 代道芳, 辛志平, 等.传统培养结合分子生物学法分析传统发酵食品中乳酸菌的多样性[J].中国酿造, 2011,30(2):52-54.
LIN Y, DAI D F, XIN Z P, et al.Diversity of lactic acid bacteria in traditional fermented food analyzed by conventional culture and molecular biology methods [J].China Brewing, 2011,30(2):52-54.
[27] 舒中玉. 乳酸片球菌S204益生特性、发酵及喷雾干燥工艺的研究[D].武汉:华中农业大学, 2018.
SHU Z Y.Research on biological characteristics, fermentation process and spray drying process of Pediococcus acidilactici S204 [D].Wuhan:Huazhong Agricultural University, 2018.
[28] CAI W C, TANG F X, WANG Y R, et al.Bacterial diversity and flavor profile of Zha-Chili, a traditional fermented food in China[J].Food Research International, 2021, 141(1):110112.