近年来,植物蛋白因其对机体健康有益及对环境友好等,越来越广泛地被应用于食品工业中。但部分植物蛋白质(如小麦面筋蛋白、大豆分离蛋白、玉米蛋白等)具有较高含量的谷氨酰胺残基,通过疏水或氢键相互作用诱导蛋白质聚集和沉淀[1],限制了植物蛋白在食品工业中的应用[2]。脱酰胺作用可将蛋白质的酰胺基转化为羧基,增加负电荷、降低蛋白质等电点,从而改善蛋白质的功能[3]。目前,可采用物理、化学以及酶解的方法进行脱酰胺处理。相较于前两者,酶解法脱酰胺具有反应速率高、反应条件温和、底物特异性强、成本低、更安全等特点[4],更适用于食品工艺。
蛋白质谷氨酰胺酶(protein glutaminase, PG)可水解短肽或蛋白质中的谷氨酰胺残基,且不影响天冬酰胺残基或游离的谷氨酰胺,从而催化蛋白质的脱酰胺作用,改善蛋白功能特性[5]。目前,PG对许多蛋白已起到改性作用,如大豆蛋白经PG处理后溶解度从约25%提升至65%以上[6];玉米蛋白经PG处理后,溶液浊度增加,沉淀物的量明显减少,溶解度从极低提升至85%左右[7]。但其生产菌株解朊金黄杆菌的产量较低,仅为0.258 U/mL;同时由于其基因操作效率低,分析其调控机制、提高PG的表达水平具有挑战性[8]。建立外源表达系统,可分析其激活机制并提高其表达水平。
酵母异源表达系统具有诸多优点,如可将目标产物分泌到细胞外,内源性分泌蛋白少、易于纯化等;同时,与细菌相比,还具有生长速度快、翻译后修饰、易于遗传操作等优势[9-10]。线性化的外源DNA通过交叉重组可高效插入酵母细胞的染色体中,从而产生稳定的细胞系[11]。
本文构建了重组质粒pPIC9K-Pro-PG,将其转化至毕赤酵母GS115细胞中,实现PG在真核细胞中的异源表达,并对重组酶的酶学性质进行了探究。
1 材料与方法
1.1 材料
1.1.1 菌株及质粒
大肠杆菌Escherichia coliDH5α、酵母表达菌株(Pichia pastoris)GS115及pPIC9K-ProK质粒均为实验室保存。
1.1.2 主要试剂
分子克隆试剂盒,天根生化科技有限公司;Not I-HF酶、EcoR I-HF酶,NEB公司;QuickcutTMSalI酶,宝日成生物技术有限公司。
1.1.3 培养基
LB![]() 脂培养基、YPD培养基、BMGY培养基、BMMY培养基、MD
脂培养基、YPD培养基、BMGY培养基、BMMY培养基、MD![]() 脂培养基的配制均参照文献[12]。
脂培养基的配制均参照文献[12]。
1.2 实验方法
1.2.1 目的基因的扩增及载体构建
利用细菌基因组抽提试剂盒提取解朊金黄杆菌基因组,以基因组DNA[13]为模板,在Pro-PG序列的5′端加上EcoR I酶切位点及保护碱基,3′加上NotI酶切位点及保护碱基,PCR扩增合成目的基因。
Forward:5′-CCGGAATTCGATTCCAACGGGAATCA-
Reverse:3′-AAGGAAAAAAGCGGCCGCTTAAAA-TCCACAGCTGGATAC-
用EcoR I和NotI两种限制性内切酶分别双酶切pPIC9K-ProK及目的基因,用1%(质量分数)琼脂糖凝胶电泳检测,回收目的片段,将回收产物通过T4连接酶进行连接后,转化至E.coliDH5α,并于LB![]() 脂培养基上培养,挑取单菌落经鉴定获得pPIC9K-Pro-PG表达载体。
脂培养基上培养,挑取单菌落经鉴定获得pPIC9K-Pro-PG表达载体。
1.2.2 毕赤酵母转化
pPIC9K-Pro-PG表达载体经SalI限制性内切酶37 ℃条件下处理20 min后,通过电转法将其转入毕赤酵母GS115感受态细胞中[14],电击参数为1 500 V、250 Ω、25 μF。电转结束后,30 ℃、220 r/min振荡培养1 h,适当浓缩后涂到MD![]() 脂平板上,30 ℃培养2~3 d,直到长出转化子[15]。
脂平板上,30 ℃培养2~3 d,直到长出转化子[15]。
1.2.3 高拷贝转化子的筛选
随机挑选转化子,重悬于无菌水中,混匀后涂布于含不同质量浓度G418(0.5、1.0、2.0、3.0 mg/mL)的YPD培养板上,30 ℃培养3~4 d。从3.0 mg/mL G418浓度的平板上,挑取长出的单菌落进行PCR鉴定。
1.2.4 Pro-PG的诱导表达
将3.0 mg/mL G418的YPD平板上长出的单菌落接种于10 mL YPD液体培养基中,在30 ℃、220 r/min条件下振荡培养至OD600=3.0时,吸取1 mL接种于40 mL BMGY培养基中,30 ℃、220 r/min继续振荡培养至OD600=2.0~6.0,将发酵液离心后用40 mL BMMY培养基重悬,使OD600=1.0,30 ℃、220 r/min继续振荡培养,每24 h添加1%终体积的甲醇。同时,每24 h取1 mL发酵液,离心后取上清液进行SDS-PAGE分析。
1.2.5 胰蛋白酶处理Pro-PG及PG活力测定
Pro-PG消化实验:用pH 7.4的PBS配制3 mg/mL胰蛋白酶液,取500 μL发酵上清液,加入适当体积的胰蛋白酶母液、PBS,使终体积为750 μL,胰蛋白酶终质量浓度分别为0、0.15、0.30、0.45 mg/mL。37 ℃水浴12 h后,进行SDS-PAGE分析和酶活力测定。
1.2.6 摇瓶培养Pro-PG表达条件的优化
为了探究摇瓶发酵时重组毕赤酵母产Pro-PG的最佳条件,对诱导过程中的pH (4.0~8.0)、甲醇体积分数(0.5%~3.0%)进行了优化。诱导120 h后,进行SDS-PAGE通过灰度分析来确定最佳诱导条件。
1.2.7 酶学性质分析
Pro-PG经胰蛋白酶消化激活,对PG进行酶学性质分析,分别在不同pH(4.0~9.0)和不同温度(30~80 ℃)下,将PG孵育1 h后,测定酶活力,探究PG的最适酶反应条件。
2 结果与分析
2.1 目的基因的获取
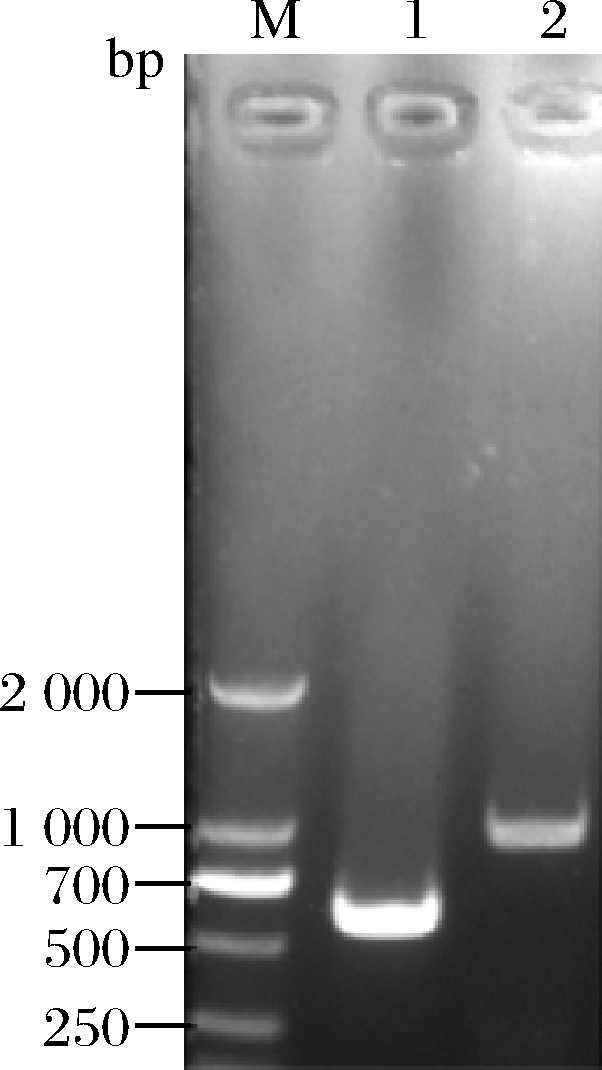
利用细菌基因组抽提试剂盒提取解朊金黄杆菌基因组,以其为模板,通过PCR扩增得到约900 bp的片段,基本符合目的基因片段大小,如图1所示。

M-DL2000 Marker;1-阴性对照;2、3-Pro-PG
图1 PCR鉴定Pro-PG基因
Fig.1 PCR analysis of Pro-PG gene
按1.2.1的方法,对载体pPIC9K、目的基因进行双酶切和回收酶连后,转化至DH5α中,37 ℃过夜培养后,进行菌落PCR并送至金唯智生物有限公司测序,结果表明pPIC9K-Pro-PG重组表达载体构建成功(图2)。
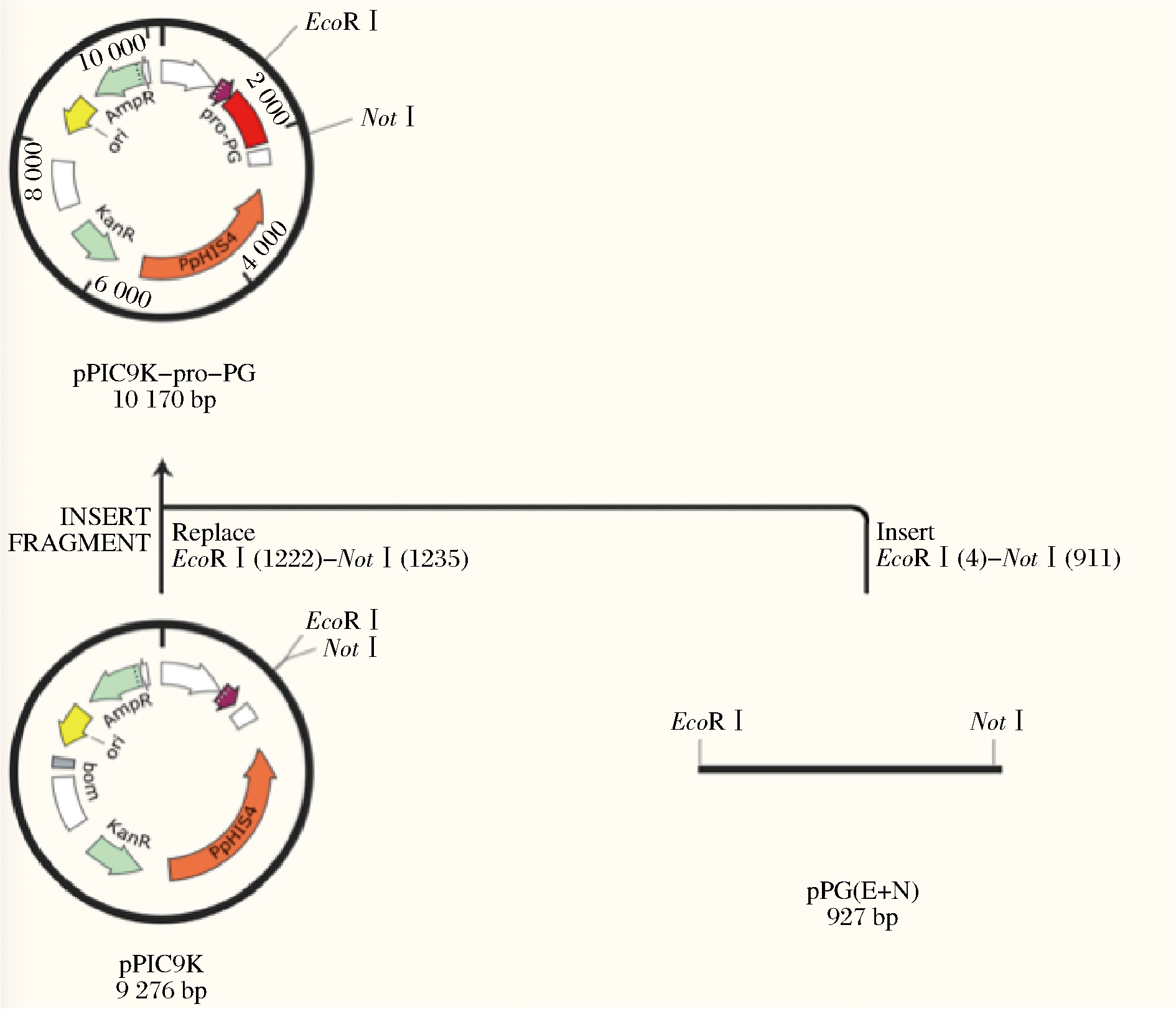
图2 pPIC9K-Pro-PG质粒重组策略
Fig.2 Construction strategy of recombinant plasmid pPIC9k-Pro-PG
2.2 重组酵母pPIC9K-Pro-PG阳性高拷贝转化子的筛选
pPIC9K载体上含有氨苄青霉素和卡那霉素抗性,高拷贝的插入可提高转化子对G418的抗性。挑取转化子,经过不同质量浓度G418(0.5、1.0、2.0、3.0 mg/mL)的YPD培养板筛选;从含3 mg/mL G418的YPD平板上,挑取单菌落扩培提取基因组,进行PCR扩增鉴定。结果如图3所示,说明pPIC9K-pPG质粒已经整合到毕赤酵母基因组中。
M-DL2000 Marker;1-PG基因;2-Pro-PG基因
图3 PCR鉴定转化子
Fig.3 PCR analysis of transformants
2.3 重组酵母的诱导表达
经过甲醇诱导表达后,每隔取24 h取1 mL发酵液,离心后取上清液对其进行SDS-PAGE分析。结果如图4所示,随着时间增加,在35 kDa附近有达标条带,分子质量大小与Pro-PG理论分子质量一致,表达量随培养时间增加而逐渐增多。
2.4 胰蛋白酶加工Pro-PG及PG活力测定
上清液超滤浓缩3倍,脱盐。Pro-PG经胰蛋白酶酶解激活,如图5所示,胰蛋白酶处理12 h时,Pro-PG逐渐减少,在25 kDa附近出现新条带(PG),此时PG的酶活力大约为0.382 U/mL;经胰蛋白酶酶切处理24 h时,Pro-PG完全降解,此时测得的PG活力约为0.878 U/mL。
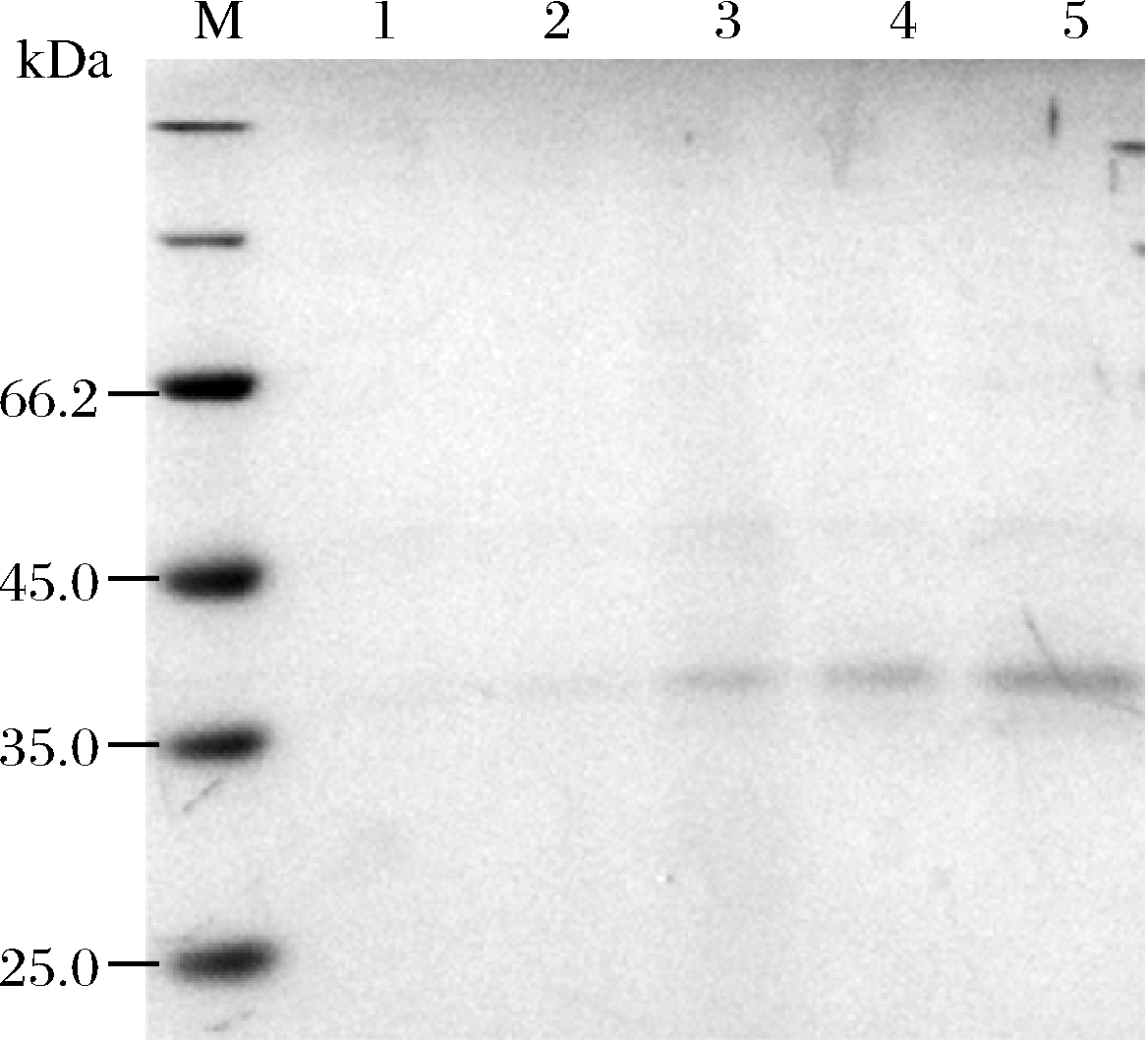
M-Marker;1~5-培养24、48、72、96、120 h发酵液上清液
图4 凝胶电泳分析Pro-PG表达
Fig.4 SDS-PAGE analysis of Pro-PG expression
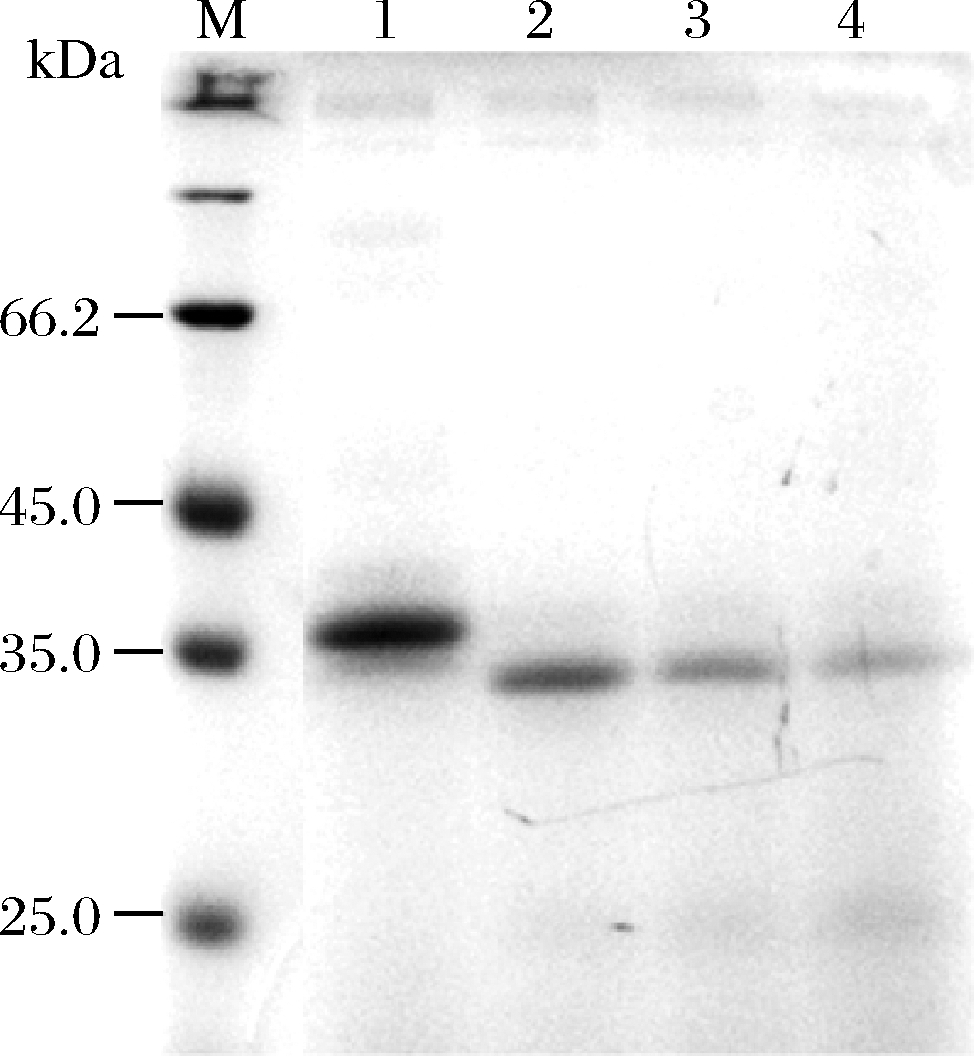
M-Marker;1-3倍浓缩发酵液;2-0.15 mg/mL胰蛋白酶酶解; 3-0.3 mg/mL 胰蛋白酶酶解;4-0.45 mg/mL胰蛋白酶酶解
图5 胰蛋白酶酶解激活Pro-PG
Fig.5 Trypsin digestion of Pro-PG
2.5 摇瓶发酵诱导条件的优化
对诱导pH、甲醇添加量进行优化,结果如图6所示。对SDS-PAGE结果(图6)经灰度分析处理后,从图7可知发酵培养基的初始pH为8.0时,Pro-PG的表达情况最好;每间隔24 h添加甲醇体积分数为终体积的2%时,Pro-PG的表达量最高。
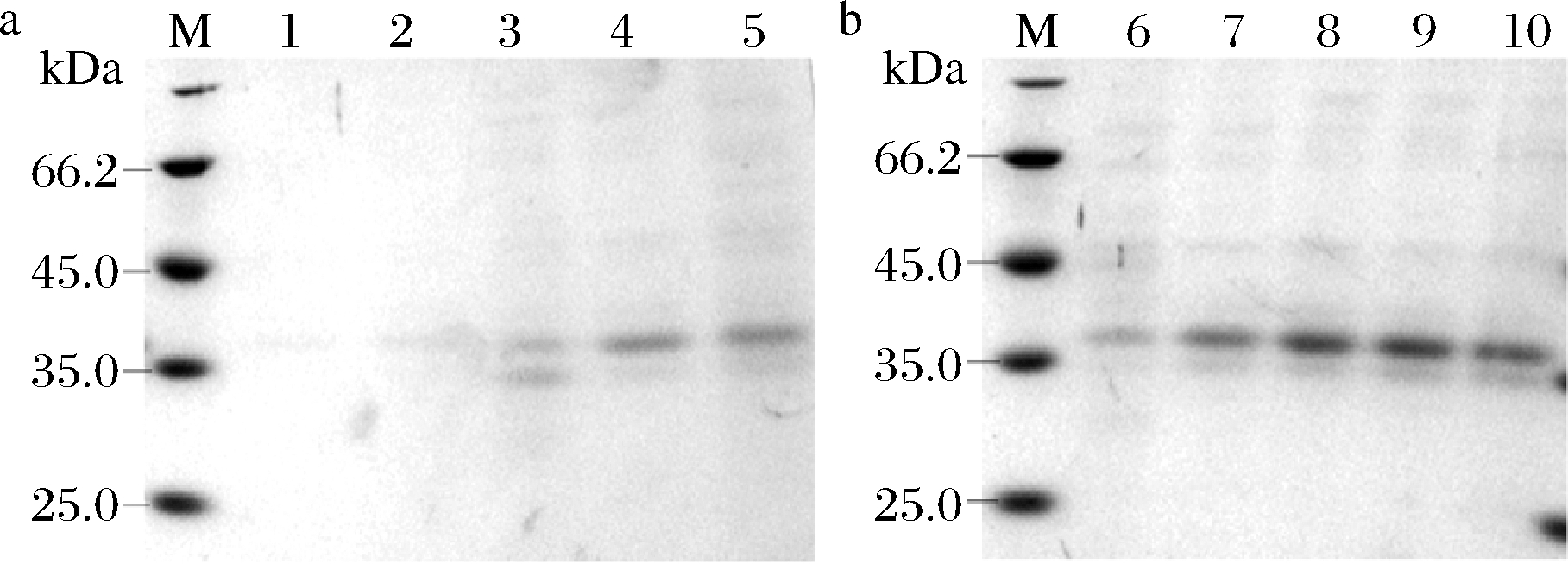
M-Marker;1-pH 4.0;2-pH 5.0;3-pH 6.0;4-pH 7.0; 5-pH 8.0;6-0.5%;7-1.0%;8-1.5%;9-2%;10-3% a-甲醇体积分数为1%;b-pH-7.0
图6 Pro-PG表达条件优化
Fig.6 Optimization of fermentation conditions for Pro-PG production
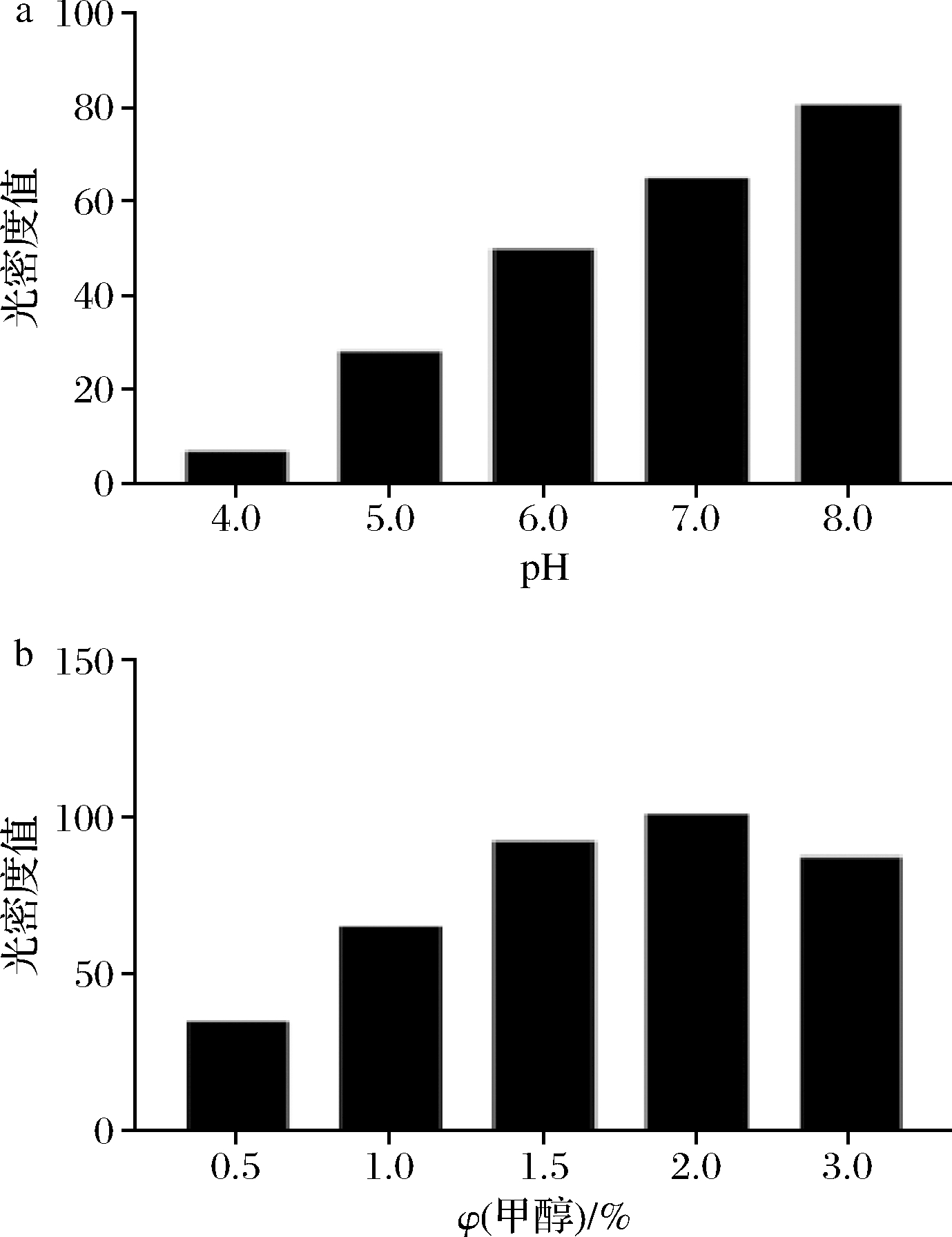
a-pH;b-甲醇终体积分数
图7 Pro-PG表达量(灰度扫描)
Fig.7 Expresion of Pro-PG by grey scanning
2.6 PG的酶学性质分析
PG的最适反应温度为60 ℃(图8-a),在不超过60 ℃条件下,1 h其相对酶活力可保持在80%以上,在1 h内,该酶在不超过50 ℃条件下热稳定性良好(图8-c),当温度升高至70 ℃时,1 h内PG完全失活。
PG的最适反应pH为6.0(图8-b)。在pH 5.0~8.0间PG均表现出较高酶活力水平。PG在较广的pH范围内都表现出较高的稳定性(图8-d),在pH 3.0~8.0间,其酶活力在1 h内可保持70%以上的活性。尤其是对酸性条件有较强的耐性,但在碱性条件下,其酶活力有较大的丧失。
3 讨论与结论
本实验通过构建pPIC9K-Pro-PG/GS115菌株,实现了Pro-PG在真核系统中的异源表达,并且在优化发酵条件后获得0.878 U/mL的最高酶活力。
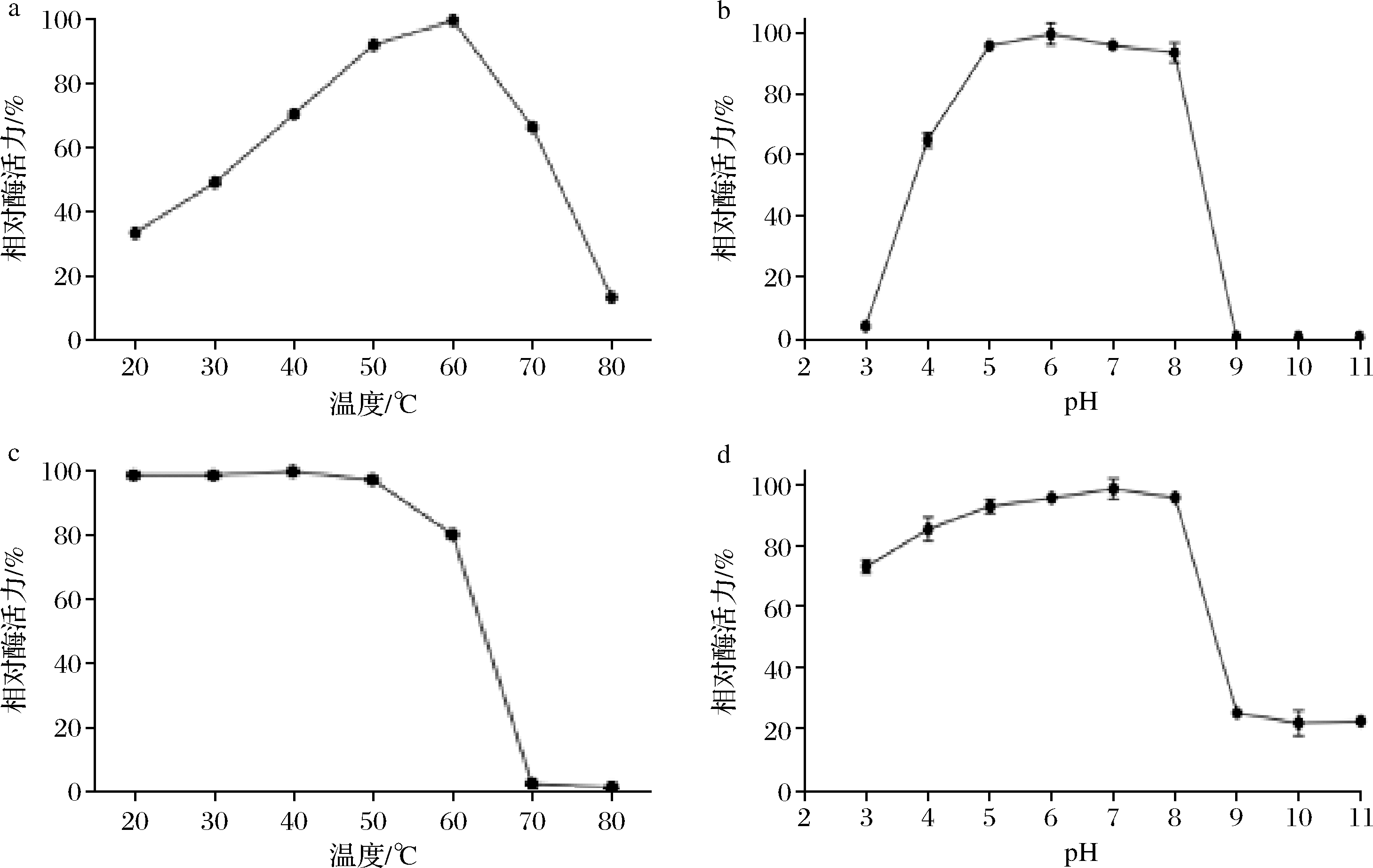
a-最适反应温度;b-最适pH;c-温度稳定性;d-pH稳定性
图8 成熟PG酶学特性
Fig.8 Enzymatic properties of active PG
目前毕赤酵母表达出的最高酶活力仍较低,较工业生产要求(10 U/mL)相比仍有较大差距。但研究表明通过密码子优化的方式,可进一步提高蛋白的表达量[16],如LI等[17]指出密码子优化过的乙酰胆碱酯酶的表达水平较野生型菌株提升约3.14倍。可对Pro-PG的基因序列进行优化,以进一步研究其在毕赤酵母中的表达情况。
苗杨利等[18]通过软件预测出9种具有分泌潜力的毕赤酵母内源信号肽序列,经验证PRY2、DSE4、FRE2介导的EGFP分泌水平显著优于pPIC9K中携带的MF-α,可尝试调整信号肽以进一步优化Pro-PG的表达。
此外,高密度发酵的菌体产量和产物表达量可达非高密度发酵的10~50倍[19]。毕赤酵母是好氧性微生物,适合扩大培养,可通过分批发酵积累到一定的生物量,再用甲醇进行诱导,期间进行分批补料、优化发酵条件参数从而高效表达目的蛋白[20]。马红丽等[21]利用10 L发酵罐扩大培养可将脂肪酶胞外酶活力从摇瓶水平的105.62 U/mL提升至680 U/mL。在工业生产上可利用发酵罐进一步优化Pro-PG在毕赤酵母中的表达。
本实验成功构建质粒pPIC9K-Pro-PG,通过电转法获得pPIC9K-Pro-PG/GS115工程菌,经PCR和基因测序验证无误。经SDS-PAGE分析确定Pro-PG的表达,经胰蛋白酶加工后确认获得0.878 U/mL的PG酶活力。
[1] LIUX, WANG C, ZHANG X W, et al.Application prospect of protein-glutaminase in the development of plant-based protein foods[J].Foods (Basel Switzerland),2022, 11(3):440.
[2] NIKBAKHTNASRABADI M, SEDAGHAT DOOST A, MEZZENGA R.Modification approaches of plant-based proteins to improve their techno-functionality and use in food products[J].Food Hydrocolloids, 2021, 118:106789.
[3] YONGY H, YAMAGUCHI S, MATSUMURA Y.Effects of enzymatic deamidation by protein-glutaminase on structure and functional properties of wheat gluten[J].Journal of Agricultural and Food Chemistry, 2006, 54(16):6 034-6 040.
[4] HAMADAJ S, SWANSON B.Deamidation of food proteins to improve functionality[J].Critical Reviews in Food Science and Nutrition, 1994, 34(3):283-292.
[5] YAMAGUCHIS, JEENES D J, ARCHER D B.Protein-glutaminase from Chryseobacterium proteolyticum, an enzyme that deamidates glutaminyl residues in proteins:Purification, characterization and gene cloning[J].European Journal of Biochemistry, 2001, 268(5):1 410-1 421.
[6] SUPPAVORASATITI, DE MEJIA E G, CADWALLADER K R.Optimization of the enzymatic deamidation of soy protein by protein-glutaminase and its effect on the functional properties of the protein[J].Journal of Agricultural and Food Chemistry, 2011, 59(21):11 621-11 628.
[7] YONGY H, YAMAGUCHI S, GU Y S, et al.Effects of enzymatic deamidation by protein-glutaminase on structure and functional properties of α-zein[J].Journal of Agricultural and Food Chemistry, 2004, 52(23):7 094-7 100.
[8] YINX X, ZHANG G Q, ZHOU J W, et al.Combinatorial engineering for efficient production of protein-glutaminase in Bacillus subtilis[J].Enzyme and Microbial Technology, 2021, 150:109863.
[9] DALYR, HEARN M T W.Expression of heterologous proteins in Pichia pastoris:A useful experimental tool in protein engineering and production[J].Journal of Molecular Recognition, 2005, 18(2):119-138.
[10] KARAO LAN M, ERDEN-KARAO
LAN M, ERDEN-KARAO LAN F.Effect of codon optimization and promoter choice on recombinant endo-polygalacturonase production in Pichia pastoris[J].Enzyme and Microbial Technology, 2020, 139:109589.
LAN F.Effect of codon optimization and promoter choice on recombinant endo-polygalacturonase production in Pichia pastoris[J].Enzyme and Microbial Technology, 2020, 139:109589.
[11] KARBALAEIM, REZAEE S A, FARSIANI H.Pichia pastoris:A highly successful expression system for optimal synthesis of heterologous proteins[J].Journal of Cellular Physiology, 2020, 235(9):5 867-5 881.
[12] 廖兆民,蔡俊, 林建国, 等.黑曲霉葡萄糖氧化酶基因在毕赤酵母中的表达及产酶条件的优化[J].生物技术通报, 2021, 37(6):97-107.
LIAO Z M, CAI J, LIN J G, et al.Expression of glucose oxidase gene from Aspergillus nigerin Pichia pastorisand optimization of enzyme production conditions[J].Biotechnology Bulletin, 2021, 37(6):97-107.
[13] YAMAGUCHIS, YOKOE M.A novel protein-deamidating enzyme from Chryseobacterium proteolyticumsp.nov., a newly isolated bacterium from soil[J].Applied and Environmental Microbiology, 2000, 66(8):3 337-3 343.
[14] 梁启星,石竟成, 金学荣, 等.肠激酶在毕赤酵母中的分泌表达优化[J].生物工程学报, 2020, 36(8):1 689-1 698.
LIANG Q X, SHI J C, JIN X R, et al.Optimization of enterokinase secretion in Pichia pastoris[J].Chinese Journal of Biotechnology, 2020, 36(8):1 689-1 698.
[15] 魏东升,段广东, 钱江潮.葡萄糖氧化酶在毕赤酵母中的高效分泌表达[J].华东理工大学学报(自然科学版), 2021, 47(3):300-307.
WEI D S, DUAN G D, QIAN J C.Efficient secretory expression of glucose oxidase in Pichia pastoris[J].Journal of East China University of Science and Technology, 2021, 47(3):300-307.
[16] XUY C, LIU K S, HAN Y, et al.Codonusage bias regulates gene expression and protein conformation in yeast expression system P.pastoris[J].Microbial Cell Factories, 2021, 20(1):91.
[17] LIJ D, XIE X, CAI J, et al.Enhanced secretory expression and surface display level of Bombyx moriacetylcholinesterase 2 by Pichia pastorisbased on codon optimization strategy for pesticides setection[J].Applied Biochemistry and Biotechnology, 2021, 193(10):3 321-3 335.
[18] 苗杨利,李站胜, 韩双艳.促进米黑根毛霉脂肪酶分泌的毕赤酵母内源信号肽的筛选[J].现代食品科技, 2019, 35(10):155-163.
MIAO Y L, LI Z S, HAN S Y.Screening endogenous signal peptides in Pichia pastorisfor promoting the secretion of lipase from Rhizomucor miehei[J].Modern Food Science and Technology, 2019, 35(10):155-163.
[19] 闫达中,许芳, 杨艳燕.毕赤酵母基因工程菌高密度发酵产纳豆激酶条件研究[J].中国酿造, 2009, 28(9):59-61.
YAN D Z, XU F, YANG Y Y.High cell density fermentation conditions of nattokinase by genetic engineering strain Pichia pastorisGS115/pPRONK1[J].China Brewing, 2009, 28(9):59-61.
[20] 武婕,张晓雪, 余河水, 等.毕赤酵母工程菌高密度发酵研究与进展[J].中国生物工程杂志, 2016, 36(1):108-114.
WU J, ZHANG X X, YU H S, et al.Research progress of high density fermentation process of Pichia pastoris[J].China Biotechnology, 2016, 36(1):108-114
[21] 马红丽,付晓平, 郑雯, 等.费希尔曲霉脂肪酶在毕赤酵母中的优化表达及高密度发酵[J].微生物学通报, 2020, 47(7):2 140-2 150.
MA H L, FU X P, ZHENG W, et al.Optimized expression and high-density fermentation of Aspergillus fischerilipase in Pichia pastoris[J].Microbiology, 2020, 47(7):2 140-2 150.