植酸酶(phytase,EC 3.1.3.8)又称肌醇六磷酸水解酶(myo-inositol hexakisphosphate phosphohydrolas)[1],能将谷物中的植酸水解成肌醇衍生物,并逐步释放出无机磷酸[2]。植酸酶已被广泛用作饲料添加剂[3],以减少无机磷的添加量及消除植酸的抗营养效应。同时,它也被广泛用于面团发酵前的处理,去除面粉中的植酸,改善面包的口感[4]。来源于微生物的植酸酶因活性高、生产成本低等优点,已被广泛用于饲料加工产业[3]。目前,畜禽饲料市场上的植酸酶主要源自真菌和细菌,以及一些经过酶学特性改造过的工程酶[5-7]。植酸酶广泛存在于多种微生物中,但原始菌株的产酶效率低下、培养条件苛刻,不适合规模化的生产。利用基因工程技术将植酸酶基因导入合适的宿主,进行高效异源表达仍是高品质植酸酶开发的有效途径。目前已有许多植酸酶基因成功地在多种表达系统中实现了异源表达,如大肠杆菌[8]、芽胞杆菌[9]、毕赤酵母[10-11]和丝状真菌[11]等。尽管如此,目前在用的植酸酶仍然还存在催化活性较低或稳定性较差的缺陷,尤其是热稳定性差的缺陷严重制约了它在饲料加工中的应用。
植物乳杆菌(Lactobacillus plantarum)已被广泛用于食品和饲料加工,能够产生大量的抗氧化物质,以及提高黄贮饲料的发酵品质[12]。此外,最近也有学者从植物乳杆菌中分离出了植酸酶,并对它们进行了部分酶学特性的分析,它表现出了良好的热稳定性和较高的最适反应温度,在饲料加工中具有更大的优势[13-14]。受限于植物乳杆菌生长速度缓慢,难以实现高密度发酵,从植物乳杆菌中提取植酸酶难以满足规模化的生产需求[15]。利用现代基因工程手段将高产目标酶的原始菌株中的目标酶基因进行克隆和异源表达,有利于对目标酶进行深入研究和应用推广[16-17]。
本研究拟借助基因组挖掘技术,从植物乳杆菌基因组中挖掘新型植酸酶基因,对获得的基因进行相关的生物信息学分析,借助计算机辅助的手段对其进行理性设计,再利用大肠杆菌作为宿主,构建能高效表达重组植酸酶的基因工程菌,并进行酶学特性的分析,旨在为进一步实现植物乳杆菌植酸酶的分子改造和高效表达奠定理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 主要试剂
常用的分子生物学试剂和试剂盒,大连TaKaRa公司;一步法His标记蛋白微型制备试剂盒,北京索莱宝生物技术有限公司;PCR扩增和定点突变的引物委托苏州鸿讯生物技术有限公司合成。
1.1.2 菌株、质粒和培养基
携带原核表达质粒的Escherichia coliBL21/pET28a菌株由本课题组保藏,用LB培养基培养。Lactobacillus plantarumCICC 20038购自中国工业微生物菌种保藏中心,用MRS培养基进行培养,主要用于植酸酶基因的PCR扩增。
1.1.3 常用网站及软件
NCBI用于搜索植酸酶的序列。采用Modeler 9.9和EasyModeler 2.0对植酸酶及其突变体的三维结构进行多模板同源性建模。采用PyMOL方法进行三维结构观察和分析。采用DNAMAN进行基因序列和限制性内切位点分析。采用二硫键设计程序(DbDv2.0)预测了植酸酶中二硫键的可能形成。采用Genious 4.7进行序列存储和管理。Gromacs 4.5.4用于分子动力学模拟。采用Origin 9.0进行统计和分析。
1.2 主要仪器与设备
VCX -130型超声波破碎仪,Sonics &Materials, Inc.;PowerPacTMHC/Mini-PROTEAN®蛋白电泳系统、UNIVERSAL Hood凝胶成像系统,Bio-rad公司;UV-1000紫外可见分光光度计,上海天美科学仪器有限公司。
1.3 实验方法
1.3.1 植物乳杆菌植酸酶的基因克隆及生物信息学分析
以Lactobacillus plantarum和phytase为关键词,在NCBI中搜索到1个潜在的植物乳杆菌植酸酶。根据报道的基因序列,设计了用于扩增植物乳杆菌植酸酶(LpPHY)基因的引物LpPHY-F和LpPHY-R,如表1所示。PCR产物克隆至pMD19-T载体后经测序获得实际的LpPHY基因序列,并通过DNAMAN推导出其氨基酸序列。再采用Modeler 9.9和EasyModeler 2.0对植酸酶进行多模板同源建模,获得较为准确的三维结构。然后,通过DbDv2.0预测了该植酸酶中可能形成二硫键的位置,将拟突变体进行多模板的同源建模和分子动力学模拟后,根据理性设计的结果选择理想的突变位点,并基于全质粒扩增的原理设计定点突变引物,引物SDM1和pET28-2254-F如表1所示。
表1 植物乳杆菌植酸酶基因的PCR扩增和定点突变的引物
Table 1 The primers for PCR amplification and site-directed mutagenesis of L.plantarumphytase
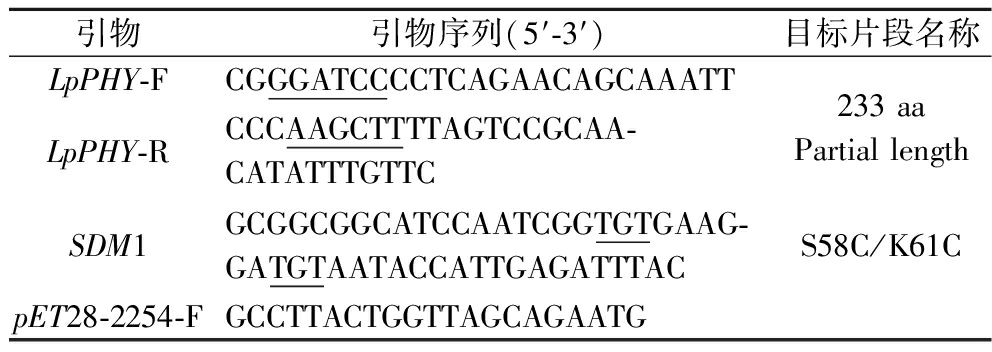
引物引物序列(5′-3′)目标片段名称LpPHY-FCGGGATCCCCTCAGAACAGCAAATTLpPHY-RCCCAAGCTTTTAGTCCGCAA-CATATTTGTTC233 aa Partial lengthSDM1GCGGCGGCATCCAATCGGTGTGAAG-GATGTAATACCATTGAGATTTACS58C/K61CpET28-2254-FGCCTTACTGGTTAGCAGAATG
1.3.2 重组表达质粒的构建
将上述获得的pMD19-T-LpPHY233用BamH I和Hind Ⅲ进行双酶切,然后连接至经同样双酶切的pET28a质粒,转化E.coliBL21感受态细胞,经卡那霉素抗性筛选、菌落PCR检测和测序鉴定后获得E.coliBL21/pET28a-LpPHY233重组子。然后,以pET28a-LpPHY233为模板,采用全质粒扩增的技术[18]进行定点突变,获得E.coliBL21/pET28a-LpPHY233S58C/K61C。
1.3.3 重组子的表达和纯化
为了对植酸酶及其突变体进行酶学特性分析及比较,将上述重组子进行低温低诱导剂的表达[19]。离心收集菌体,重悬后进行超声波破碎。离心收集上清液,再通过平衡好的镍琼脂糖柱上进行亲和层析。将纯化前和纯化后的产物进行SDS-PAGE和蛋白浓度测定。
1.3.4 植酸酶的酶活性分析
植酸酶活性测定采用改良的硫酸亚铁-钼酸盐蓝法。用100 mmol/L、pH 7.0的Tris-HCl缓冲液配制2 mmol/L的植酸钠溶液,取0.3 mL上述植酸钠溶液,加入0.5 mL稀释适当倍数的植酸酶溶液,在37 ℃、300 r/min反应30 min后,加入360 μL终止液终止反应,再加入600 μL显色液,混匀后静置5 min,用分光光度计在700 nm检测吸光值,计算酶活性,所有实验均重复3次。酶活性单位定义为:在上述测定条件下,以每分钟释放1 μmol无机磷所需的酶量定义为1个酶活性单位(IU)。
1.3.5 重组酶的温度特性
参照1.3.4中的方法,分别在35~60 ℃下测定重组植酸酶的催化活性,以获得它的最适反应温度。此外,将重组酶在25~65 ℃下孵育1 h后,分别在最适反应温度下测定其残留活性,以此来考察重组酶的温度稳定性,所有实验均重复3次。
1.3.6 重组酶的pH特性
参照1.3.4中的方法,分别测定重组酶在pH 1.0~4.5下的活性,考察它们最适反应pH值。为了考察重组酶的pH稳定性,将重组植酸酶分别在pH值1.0~4.0缓冲液中,冰浴条件下孵育1.0 h,然后在最适反应温度和pH值下测定残留活性。
1.3.7 金属离子特性
将重组植酸酶与终浓度为1 mmol/L的不同金属离子于0 ℃孵育0.5 h,采用1.3.4中的方法测定它们的残留酶活性,以仅加缓冲液的酶液为100%,考察金属离子对酶活性的影响。
1.3.8 分子对接分析
为进一步考察突变体酶学特性改变的分子机制,分别将野生型和突变型植酸酶与植酸钠进行分子对接分析。利用Chem Draw Ultra 8.0绘制植酸钠的一级结构,并去掉其中的Na+;利用Chem 3D Ultra 8.0软件进行三维结构的处理及能量最小化,再利用Auto Dock4.2软件分别将它们进行分子对接模拟。最后,利用PyMOL和Discovery Studio软件进行可视化分析,并分析酶学特性改变的分子机制[20]。
2 结果与分析
2.1 植酸酶的基因克隆
以Lactobacillus plantarum和phytase为关键词,在NCBI中搜索到了1个植物乳杆菌植酸酶(GenBank:AVV65759.1),其对应基因登录号为KY987115.1。根据报道的基因序列,设计并合成了用于PCR扩增LpPHY基因的引物LpPHY-F和LpPHY-R,PCR扩增产物经琼脂糖凝胶电泳检测,在约700 bp处获得了特异性的目的条带,与预期的长度一致,连接至pMD19-T载体后进行测序鉴定,结果与报道的序列完全一致。将测序后的pMD19-T-LpPHY233用BamH I和Hind Ⅲ进行双酶切,然后连接至经同样双酶切的pET-28a质粒,转化E.coliBL21感受态细胞,经卡那霉素抗性筛选、菌落PCR检测和测序鉴定后获得E.coliBL21/pET28a-LpPHY233重组子。
2.2 二硫键的理性引入
二硫键对于维持蛋白质的正确构象起着重要作用,在适当的位置引入二硫键可以显著提高蛋白质的热稳定性,如α-淀粉酶、纤维素酶、植酸酶和木聚糖酶[21]。本研究拟采用计算机辅助的二硫键设计方法,进一步提高LpPHY233的热稳定性。采用DbDv2.0分析了LpPHY233可能形成二硫键的位置,设计出了一系列的拟突变体,经过多模板同源建模和分子动力学模拟分析,获得了一个均方根偏差(root-mean-square deviation,RMSD)显著低于亲本的拟突变体LpPHY233S58C/K61C,结果如图1-a所示。此外,LpPHY233S58C/K61C的高RMSD值的频率也显著低于LpPHY233(图1-b)。据报道,RMSD作为评价热波动的指标[22-23],与蛋白质的热稳定性呈负相关。因此,我们选择了LpPHY233S58C/K61C突变体来考察二硫键引入对植酸酶热稳定性的影响。然后,根据1.3.2中的方法,采用全质粒扩增的技术进行了定点突变,获得了E.coliBL21/pET28a-LpPHY233S58C/K61C。
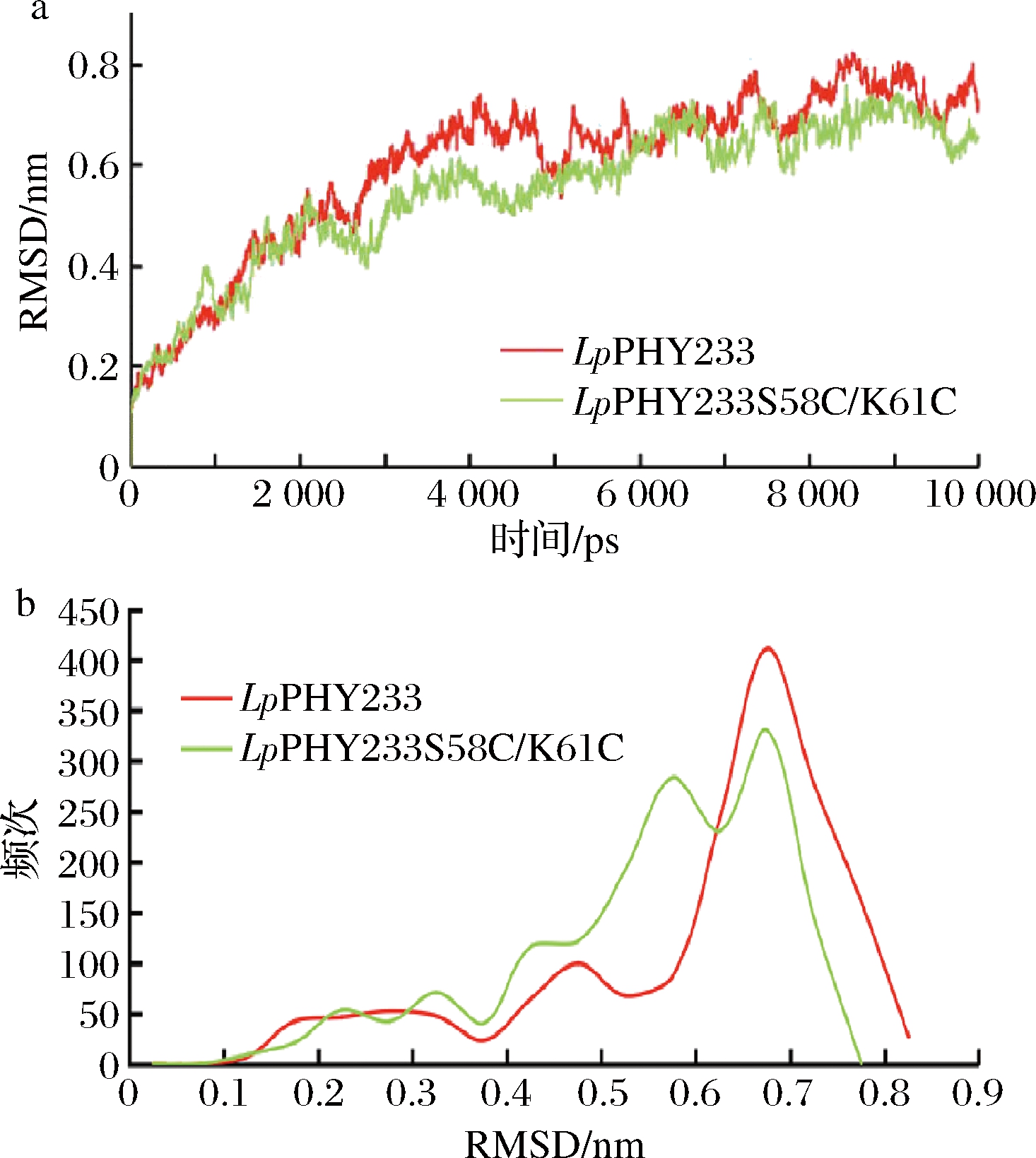
a-RMSD值的计算;b-分布
图1 LpPHY233及其突变体的RMSD值的计算和分布
Fig.1 Calculation and distribution for the RMSD values ofLpPHY233 and its mutants
2.3 重组植酸酶的表达纯化
为了研究LpPHY233及其突变体的表达水平和酶学特性,对重组子进行了低温低诱导剂浓度的诱导表达。经镍柱亲和层析,获得了电泳纯的重组LpPHY233和LpPHY233S58C/K61C,结果如图2所示。其表观相对分子质量约为30 kDa,与预期理论分子质量一致,其表观分子质量约为野生型的植物乳杆菌植酸酶的50%[14]。此外,经测定和换算后,E.coliBL21/pET28a-LpPHY233发酵液中的植酸酶活性可达560 IU/mL,显著高于植物乳杆菌的0.71 IU/mL[14]。结果表明,异源表达可显著提高目标植酸酶的表达水平。E.coliBL21/pET28a-LpPHY233S58C/K61C的表达水平有所降低,约为326.9 IU/mL,这可能是由于点突变后三维结构有所改变,蛋白质的刚性增强,柔性降低,导致酶活性稍有降低。
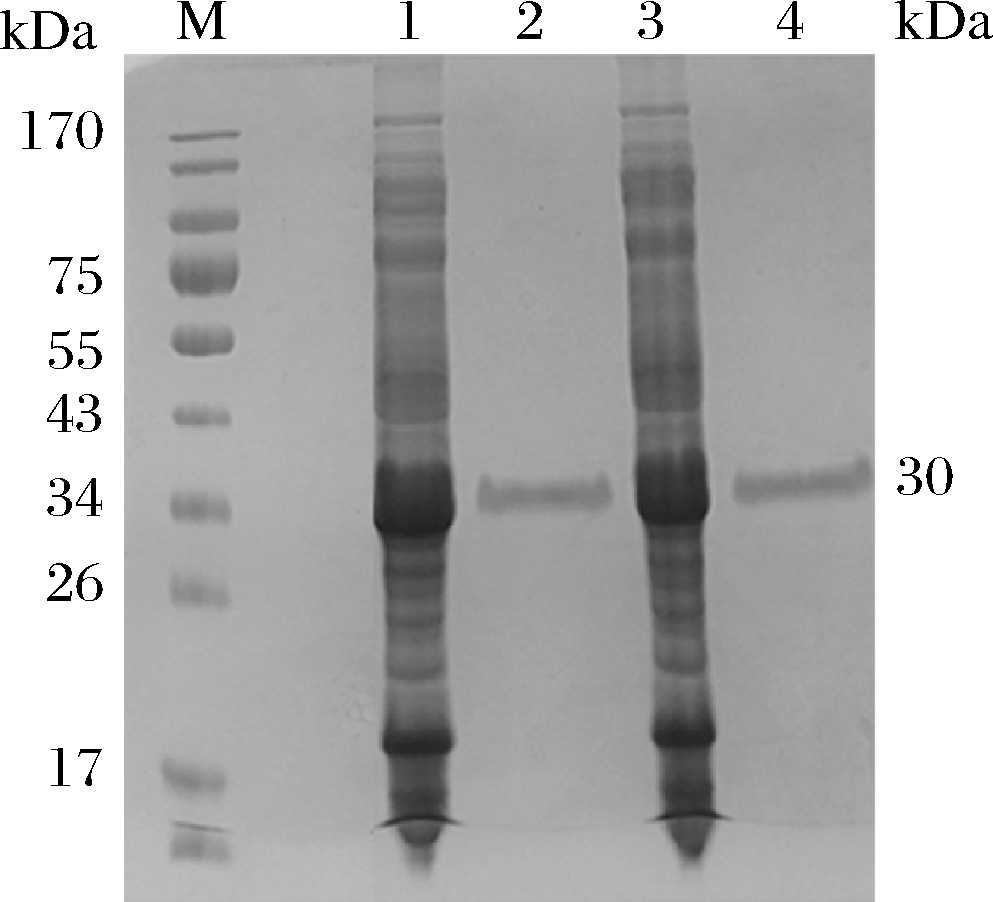
M-蛋白marker;1-LpPHY233粗酶液; 2-纯化LpPHY233酶液;3-LpPHY233S58C/K61C粗酶液; 4-纯化LpPHY233S58C/K61C酶液
图2 LpPHY233及其突变体的SDS-PAGE分析
Fig.2 SDS-PAGE analysis for the LpPHY233 and its mutants
2.4 重组酶的酶学特性分析
2.4.1 重组酶的温度特性
为了进一步考察植酸酶及其突变体的酶学性质,本研究以植酸钠为模型底物,测定了重组LpPHY233和LpPHY233S58C/K61C的酶学性质。重组植酸酶的温度特性如图3和图4所示,LpPHY233的最适反应温度为50 ℃,而突变体LpPHY233S58C/K61C的最适反应温度为65 ℃,较亲本提高了15 ℃。LpPHY233S58C/K61C在55 ℃和60 ℃下孵育1 h后的残余酶活性也略高于LpPHY233。结果表明,引入二硫键可显著提高了植酸酶的最适反应温度,与上述分子动力学模拟的结果吻合度较高,进一步提高了分子动力学模拟在指导热稳定性改造方面的可靠性。
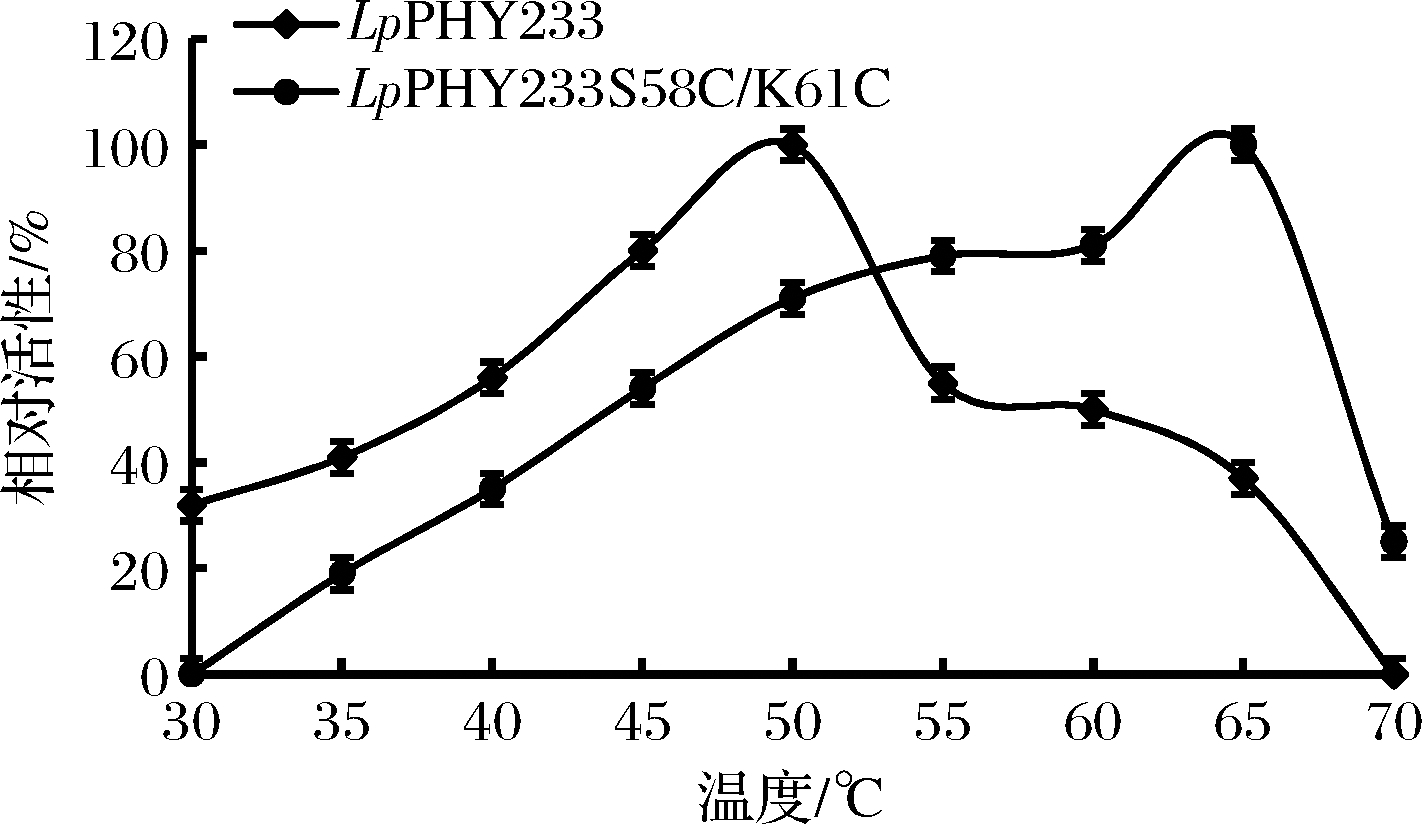
图3 重组植酸酶的最适反应温度
Fig.3 Temperature optima of recombinant phytases
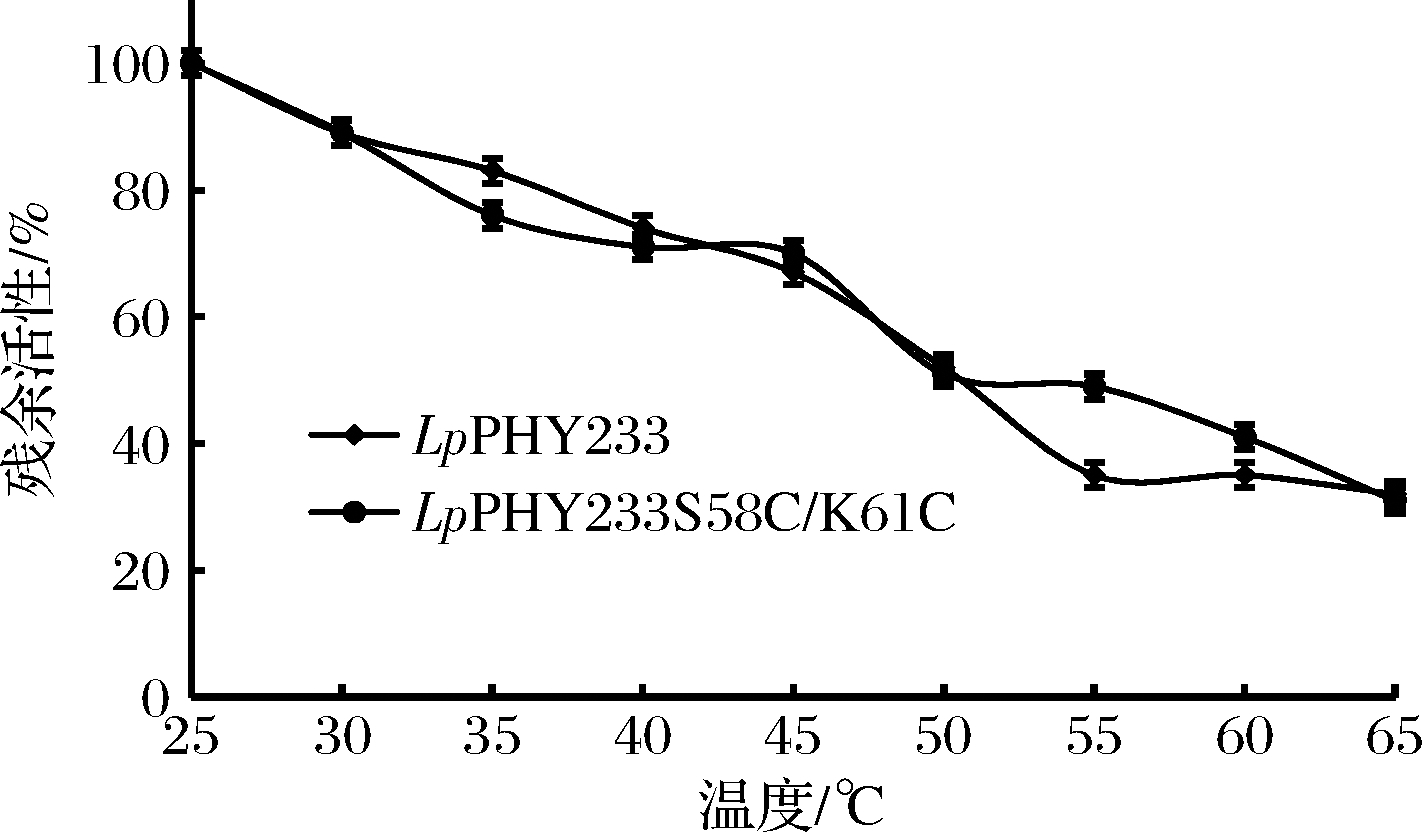
图4 重组植酸酶的温度稳定性
Fig.4 Temperature stability of recombinant phytases
2.4.2 重组酶的pH特性
按照1.3.6中的方法,测定了重组LpPHY233和LpPHY233S58C/K61C的最适反应pH值和pH稳定性,由图5可知,LpPHY233和LpPHY233S58C/K61C的最适反应pH值分别为3.5和2.5,属于嗜酸性的植酸酶,较植物乳杆菌来源植酸酶全酶的最适反应pH值(pH 4.5)有显著下降[14]。由图6可知,缓冲液的pH值可以显著影响这些植酸酶的活性。这2种植酸酶在pH 2.0下具有较高的催化活性和稳定性,这一特性有利于其耐受胃液极酸的pH环境,在饲料添加剂领域具有广阔的应用前景。
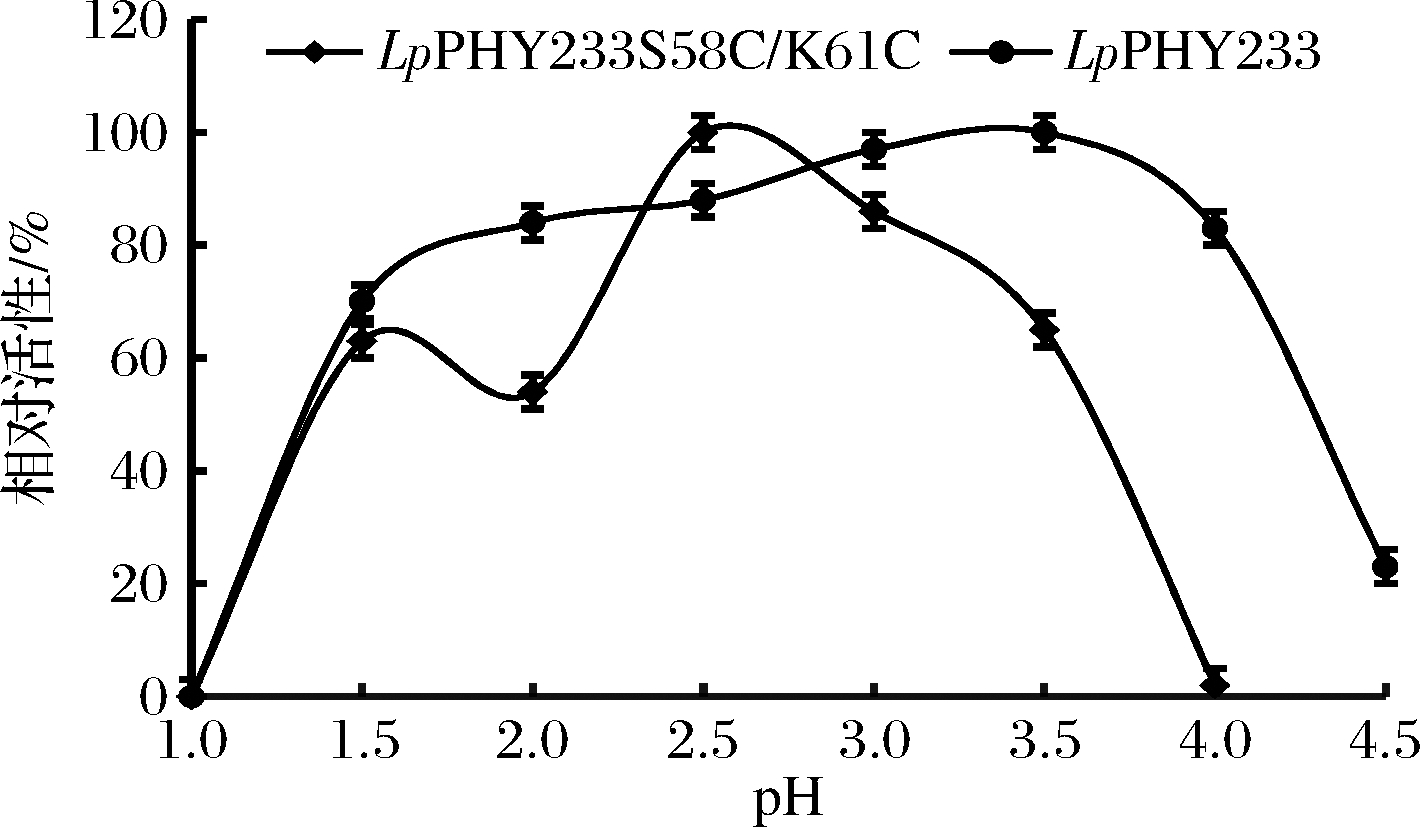
图5 重组植酸酶的最适pH值
Fig.5 Optimal pH value for these recombinant phytases
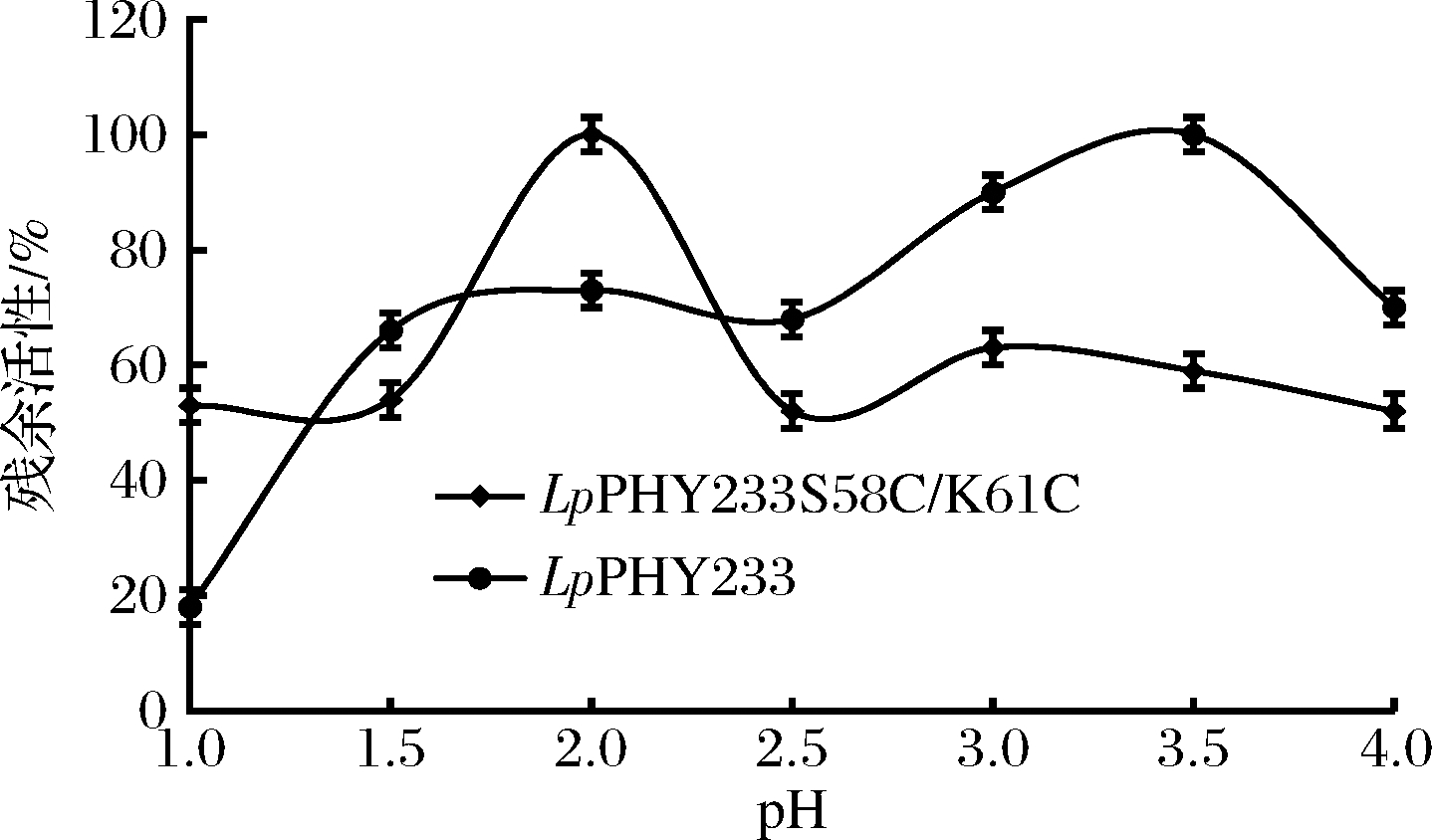
图6 重组植酸酶的pH稳定性
Fig.6 pH stability for these recombinant phytases
2.4.3 金属离子对重组酶的影响
不同金属离子对酶活性的影响如图7所示。
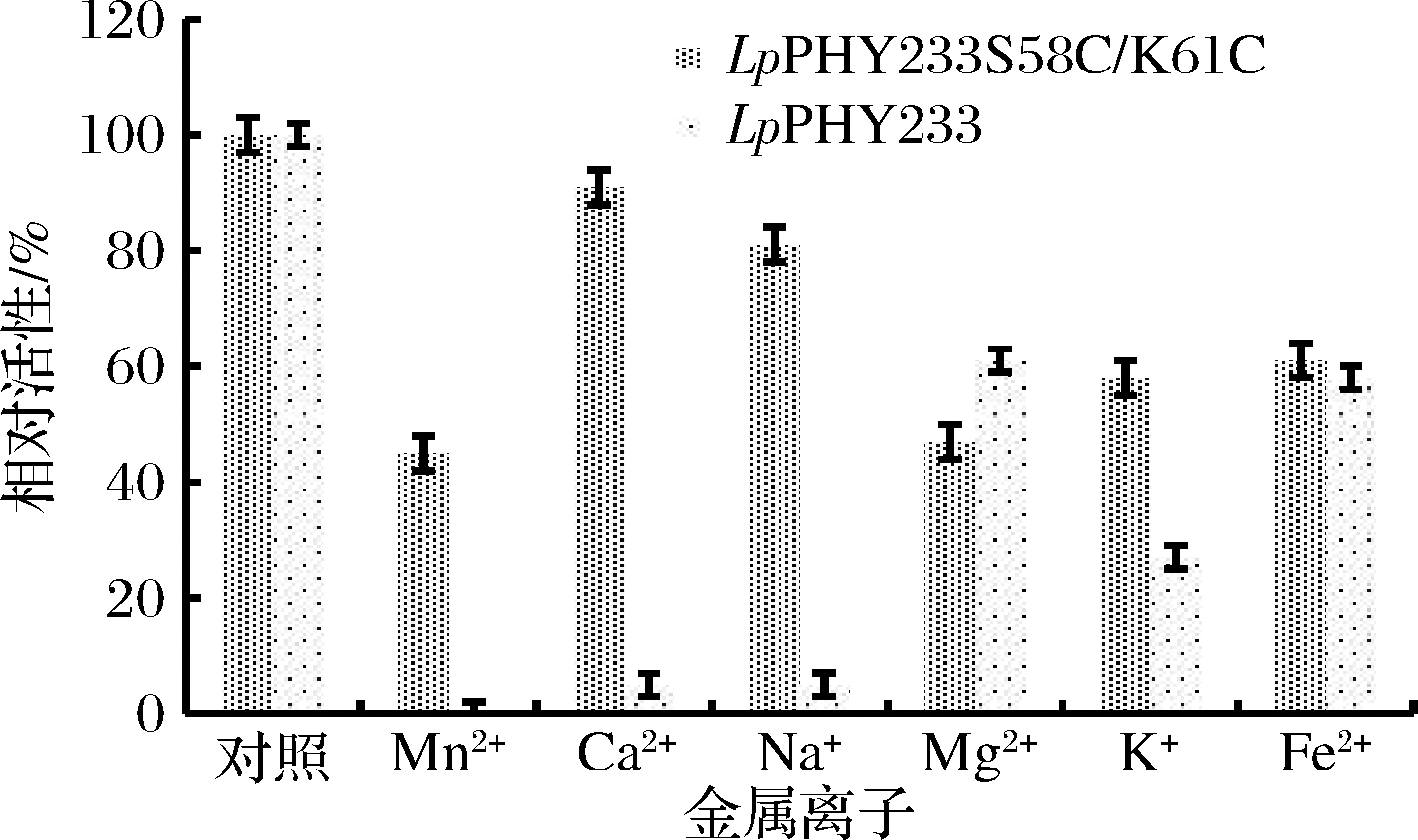
图7 重组植酸酶的金属离子特性
Fig.7 Metal ion properties of recombinant phytases
结果表明,金属离子Mn2+、Ca2+和Na+对LpPHY233的活性呈显著的抑制作用。金属离子对植酸酶的作用主要来自金属离子可以螯合底物形成稳定的络合物,从而影响植酸酶与底物的接触,而以往的研究表明酸性植酸酶很难分解金属-植酸络合物[24]。
2.5 分子对接分析
为了深入洞察这些植酸酶对植酸钠催化性能差异的分子基础,在相同对接参数下进行了分子对接分析,它们的首选构象如图8和图9所示。
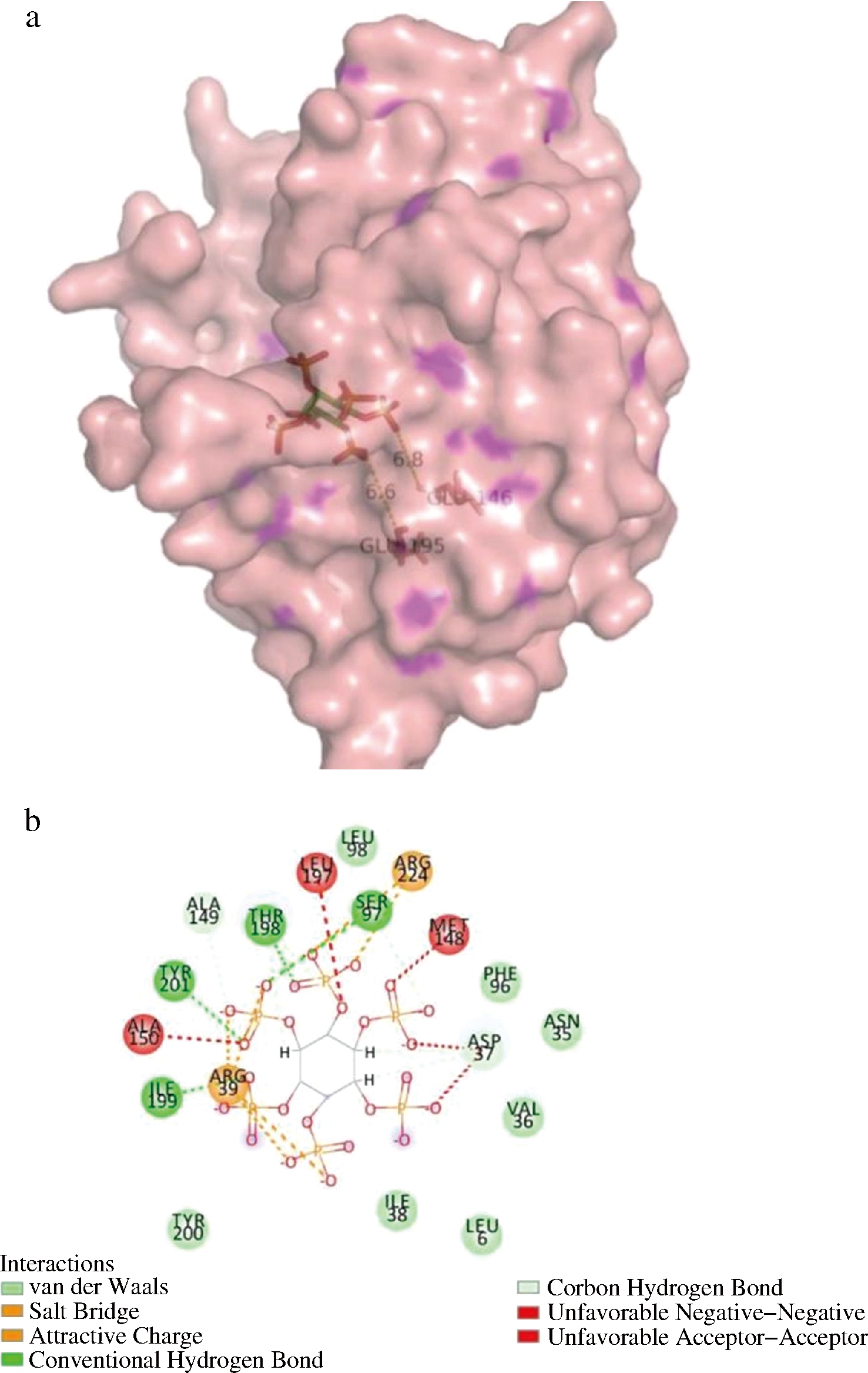
a-分子对接的首选构象;b-分子间的相互作用 `图8 植酸钠与LpPHY233的分子对接的首选 构象及分子间的相互作用
Fig.8 Preferred conformation of molecular docking and intermolecular interaction for sodium phytate with LpPHY233
如图8-a和图9-a所示,LpPHY233S58C/K61C的催化残基与底物的距离较LpPHY233的更近,理论上应该更有利于对底物的催化,但可能还受其他因素影响,LpPHY233S58C/K61C的催化活性并没有LpPHY233的高。此外,如图8-b和图9-b所示,LpPHY233S58C/K61C与底物之间的范德华力增强,表明二硫键的引入可能会改变酶与底物之间的范德华力,进而影响其最适反应温度和热稳定性。此外。尽管LpPHY233中的盐桥和常规氢键的数目明显多于LpPHY233S58C/K61C,但前者的最适反应温度显著低于后者,这也表明范德华力在维系植酸酶稳定性的作用上明显强于盐桥和氢键。
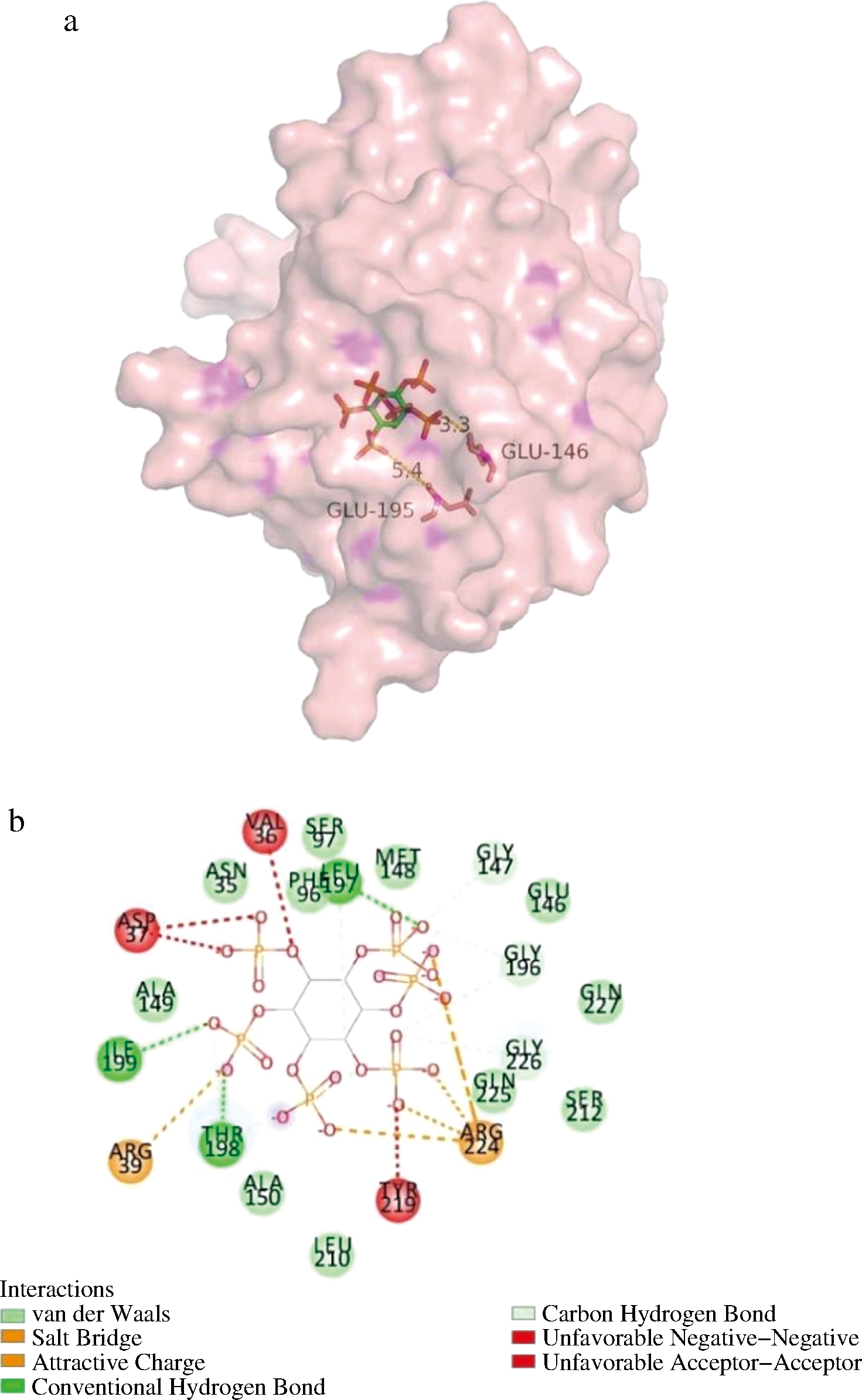
a-分子对接的首选构象;b-分子间的相互作用
图9 植酸钠与LpPHY233S58C/K61C分子对接的 首选构象及分子间的相互作用
Fig.9 Preferred conformation of molecular docking and intermolecular interaction for sodium phytate with LpPHY233S58C/K61C
3 结论与讨论
植酸酶可以高效去除谷物中的抗营养因子——植酸,因此在食品加工和饲料领域具有重要的应用潜力。此外,高活性、高稳定性的植酸酶在应用过程中更具前景[1]。尽管国内外的广大科研工作者针对植酸酶做了大量研究工作,取得了许多突破性进展,但是目前植酸酶依然存在热稳定差、比酶活性不够高的缺陷,这些缺陷严重制约了它的应用。植物乳杆菌是一种经FDA公认的安全乳杆菌,能够产生大量的抗氧化物质,它在食品加工领域有着许多天然的优势[12, 25]。本研究采用现代基因工程技术从植物乳杆菌中克隆出了1个新型植酸酶基因,并在大肠杆菌中实现了可溶性表达,其表达水平比植物乳杆菌原始菌株高出约800倍。然后,基于理性设计的结果,通过二硫键工程技术,进一步改善了LpPHY233的温度特性。同时,与LpPHY233相比,植酸酶突变体在饲料添加剂领域具有广阔的应用前景。此外,还借助分子对接的手段,对酶学特性改变的分子基础进行了初步探索,这些结果为植酸酶等工业酶的分子改造奠定了坚实的基础。
[1] HAN C, OH B C, SHIN S, et al.Crystal structures of a novel, thermostable phytase in partially and fully calcium-loaded states[J].Nature Structural Biology, 2000, 7(2):147-153.
[2] NAVONEL, VOGL T, LUANGTHONGKAM P, et al.Disulfide bond engineering of AppA phytase for increased thermostability requires co-expression of protein disulfide isomerase in Pichia pastoris[J].Biotechnology for Biofuels, 2021, 14(1):80.
[3] COWIESONA J, ACAMOVIC T, BEDFORD M R.Phytic acid and phytase:Implications for protein utilization by poultry[J].Poultry Science, 2006, 85(5):878-885.
[4] MUSZY SKIS, TOMASZEWSKA E, KWIECIE
SKIS, TOMASZEWSKA E, KWIECIE M, et al.Effect of dietary phytase supplementation on bone and hyaline cartilage development of broilers fed with organically complexed copper in a Cu-deficient diet[J].Biological Trace Element Research, 2018, 182(2):339-353.
M, et al.Effect of dietary phytase supplementation on bone and hyaline cartilage development of broilers fed with organically complexed copper in a Cu-deficient diet[J].Biological Trace Element Research, 2018, 182(2):339-353.
[5] FEIB J, XU H, CAO Y, et al.A multi-factors rational design strategy for enhancing the thermostability of Escherichia coliAppA phytase[J].Journal of Industrial Microbiology &Biotechnology, 2013, 40(5):457-464.
[6] NIUC F, YANG P L, LUO H Y, et al.Engineering the residual side chains of HAP phytases to improve their pepsin resistance and catalytic efficiency[J].Scientific Reports, 2017, 7(1):1-15.
[7] YAOM Z, WANG X, WANG W, et al.Improving the thermostability of Escherichia coliphytase, appA, by enhancement of glycosylation[J].Biotechnology Letters, 2013, 35(10):1 669-1 676.
[8] MIRZAEIM, SAFFAR B, SHAREGHI B.Cloning, codon optimization, and expression of Yersinia intermedia phytase gene in E.coli[J].Iranian Journal of Biotechnology, 2016, 14(2):63-69.
[9] TSUJIS, TANAKA K, TAKENAKA S, et al.Enhanced secretion of natto phytase by Bacillus subtilis[J].Bioscience, Biotechnology, and Biochemistry, 2015, 79(11):1 906-1 914.
[10] XIEZ M, FONG W P, TSANG P W K.Engineering and optimization of phosphate-responsive phytase expression in Pichia pastorisyeast for phytate hydrolysis[J].Enzyme and Microbial Technology, 2020, 137:109533.
[11] AJITHS, GHOSH J, SHET D, et al.Partial purification and characterization of phytase fromAspergillus foetidus MTCC 11682[J].AMB Express, 2019, 9(1):3.
[12] LIH C, HUANG J T, WANG Y Q, et al.Study on the nutritional characteristics and antioxidant activity of dealcoholized sequentially fermented apple juice with Saccharomyces cerevisiaeand Lactobacillus plantarumfermentation[J].Food Chemistry, 2021, 363:130351.
[13] KARAGÖZF P, DEMIR Y, KOTAN M  , et al.Purification of the phytase enzyme from Lactobacillus plantarum:The effect on pansy growth and macro-micro element content[J].Biotechnology and Applied Biochemistry, 2021, 68(5):1 067-1 075.
, et al.Purification of the phytase enzyme from Lactobacillus plantarum:The effect on pansy growth and macro-micro element content[J].Biotechnology and Applied Biochemistry, 2021, 68(5):1 067-1 075.
[14] SANDEZPENIDEZ S H, VELASCO MANINI M A, GEREZ C L, et al.Partial characterization and purification of phytase from Lactobacillus plantarumCRL1964 isolated from pseudocereals[J].Journal of Basic Microbiology, 2020, 60(9):787-798.
[15] LINQ, LI D N, QIN H Z.Molecular cloning, expression, and immobilization of glutamate decarboxylase from Lactobacillus fermentumYS2[J].Electronic Journal of Biotechnology, 2017, 27:8-13.
[16] GONGY Y, YIN X, ZHANG H M, et al.Cloning, expression of a feruloyl esterase from Aspergillus usamiiE001 and its applicability in generating ferulic acid from wheat bran[J].Journal of Industrial Microbiology &Biotechnology, 2013, 40(12):1 433-1 441.
[17] TANGC D, LI X, SHI H L, et al.Efficient expression of novel glutamate decarboxylases and high level production of γ-aminobutyric acid catalyzed by engineered Escherichia coli[J].International Journal of Biological Macromolecules, 2020, 160:372-379.
[18] HUD, ZONG X C, XUE F, et al.Manipulating regioselectivity of an epoxide hydrolase for single enzymatic synthesis of (R)-1, 2-diols from racemic epoxides[J].Chemical Communications (Cambridge, England), 2020, 56(18):2 799-2 802.
[19] TANGC D, DING P J, SHI H L, et al.One-pot synthesis of phenylglyoxylic acid from racemic mandelic acids via cascade biocatalysis[J].Journal of Agricultural and Food Chemistry, 2019, 67(10):2 946-2 953.
[20] TANGC D, LI J F, WEI X H, et al.Fusing a carbohydrate-binding module into the Aspergillus usamii β-mannanase to improve its thermostability and cellulose-binding capacity byin silicodesign[J].PLoS One, 2013, 8(5):e64766.
[21] WANGS, MENG K, SU X Y, et al.Cysteine engineering of an endo-polygalacturonase from Talaromyces leycettanusJCM 12802 to improve its thermostability[J].Journal of Agricultural and Food Chemistry, 2021, 69(22):6 351-6 359.
[22] WANGH Y, XIE Y L, SHI X, et al.Directed evolution of a D-mandelate dehydrogenase toward D-o-chloromandelic acid and insight into the molecular basis for its catalytic performance[J].Biochemical Engineering Journal, 2021, 166:107863.
[23] WANGJ Q, TAN Z B, WU M C, et al.Improving the thermostability of a mesophilic family 10 xylanase, AuXyn10A, from Aspergillus usamiiby in silicodesign[J].Journal of Industrial Microbiology &Biotechnology, 2014, 41(8):1 217-1 225.
[24] ANGELR, TAMIM N M, APPLEGATE T J, et al.Phytic acid chemistry:Influence on phytin-phosphorus availability and phytase efficacy1[J].Journal of Applied Poultry Research, 2002, 11(4):471-480.
[25] VANDEN NIEUWBOER M, VAN HEMERT S, CLAASSEN E, et al.Lactobacillus plantarumWCFS1 and its host interaction:A dozen years after the genome[J].Microbial Biotechnology, 2016, 9(4):452-465.