芝麻香型白酒具有“芳香馥郁、绵软醇厚、香味协调、芝麻香幽雅”的独特风格,深受消费者青睐。芝麻香型白酒的酿造工艺包括高温制曲、高氮配料、高温堆积、高温发酵、多微共酵、分层蒸馏等多个阶段[1],其中高温制曲阶段为白酒酿造提供了丰富的微生物、各种生物酶类以及多种香味前驱物质,在传统白酒酿造过程中发挥重要作用[2]。
扣囊复膜孢酵母(Saccharomycopsis fibuligera)是一类具有淀粉酶、酸性蛋白酶等多种水解酶系的二形态酵母[3-7],能够通过影响大曲前期“上霉”(呈现致密的白色小点)而直接影响成品曲品质和白酒酿造质量,是白酒酿造过程中重要的功能性微生物。王晓丹等[8]的研究表明该菌株可增加固态发酵物中乙酸乙酯、乙酸异戊酯、苯乙醇、乙酸苯乙酯、棕榈酸乙酯的含量;郝文军等[9]分析了分离自牛栏山酒醅的扣囊复膜酵母的代谢产物,发现该菌株在高粱培养基中可生成多种酯类、内酯类、醇类、萘酚类、酸类和醛酮类化合物,其中6种是牛栏山二锅头白酒中重要的香气成分;马美荣等[10]将2株扣囊复膜酵母制备成麸曲应用于清香型白酒发酵,显著提升了白酒的出酒率和品质;王浩等[11]通过模拟白酒固态混菌发酵体系来研究扣囊复膜孢酵母对微生物菌群结构的影响,结果显示该菌株会降低毕赤酵母属(Pichia)、横梗霉属(Lichtheimia)、肠杆菌属(Enterobacter)的丰度,增加芽孢杆菌属(Bacillus)和复膜酵母属(Saccharomycopsis)的丰度。
本课题组前期对扣囊复膜孢酵母的研究主要集中在菌株的生化性能[12-13]及菌株对芝麻香型高温大曲的改善作用[14],对该菌株在大曲制备过程中的具体功能机制尚未进一步解析。因此,本研究拟采用宏基因组测序和生物信息学分析技术,分析比较扣囊复膜孢酵母强化大曲和对照大曲的微生物群落结构的变化规律,探究2种大曲在物质代谢方面的异同,从而全面揭示扣囊复膜孢酵母CICC 33077对芝麻香型高温大曲群落结构和功能特性的影响,为该菌株在白酒酿造中的应用奠定基础。
1 材料与方法
1.1 实验菌株
扣囊复膜孢酵母(S.fibuligera) CICC 33077,中国工业微生物菌种保藏管理中心。
1.2 实验试剂及仪器设备
麦芽浸粉琼脂培养基、平板计数琼脂培养基,北京陆桥技术有限公司;麦芽浸粉、蛋白胨、大豆蛋白胨,北京奥博星生物技术有限责任公司;Tris、EDTA-2Na、溴化十六烷基三甲铵(cetyltrimethyl ammonium bromide,CTAB),以上均为分析纯,美国 Sigma公司;Tris饱和酚,北京博奥拓达有限公司;无水乙醇、异丙醇、三氯甲烷、K2HPO4、KH2PO4、NaCl,以上均为分析纯,北京化工厂;十二烷基硫酸钠(sodium dodecyl sulfate,SDS)(分析纯),天津市福晨化学试剂厂;Cycle-Pure Kit(100)纯化试剂盒,美国OMEGA生物技术公司;溶菌酶(50 mg/mL)、蛋白酶K(10 mg/mL),天根生化科技有限公司;MGIEasy DNA文库制备试剂盒V1.1、BGISEQ-500RS高通量测序试剂盒(PE100)V3.0,深圳华大智造科技有限公司。
BHG-8082型恒温振荡器,上海一恒科学仪器有限公司;低温离心机、各量程微量移液器,德国Eppendorf公司;VORTEX-5型旋转混合器,海门市其林贝尔仪器制造公司;SCD005喷金-离子溅射仪,瑞士BAL-TEC公司;EC250-90型电泳仪,赛默飞世尔科技公司;Explored-TWL-20n型铂金级凝胶成像系统,BIO-RAD有限公司;SU8010扫描电镜,日本日立公司。
1.3 实验方法
1.3.1 大曲的强化与采样
采用微生物强化技术,将浓度为6×105CFU/mL的扣囊复膜孢酵母菌悬液喷洒至高温大曲表面,强化大曲标记为“KS”,未强化扣囊复膜孢酵母CICC 33077的大曲为对照组,标记为“KD”。采用五点采样法,在曲房的4个边角和中心各取1块长势良好的高温大曲,充分破碎混匀,使用四分采样法,取其中的一份为本实验的研究样品[15-16]。
1.3.2 大曲样品的扫描电镜观察
使用扫描电镜对接种扣囊复膜孢酵母CICC 33077的强化大曲和对照大曲的内部结构进行解析,初步揭示其在高温大曲生产中的作用[14]。
1.3.3 大曲样品宏基因组的提取与纯化
样品宏基因组DNA提取采用ZHOU等[17]的CTAB提取方法,稍作修改。取约5 g样品,加入13.5 mL DNA裂解液和50 μL溶菌酶(50 mg/mL),220 r/min37 ℃振荡30 min;加入1.5 mL 20%(质量分数)SDS溶液和100 μL蛋白酶K(10 mg/mL),65 ℃水浴2 h,6 000 r/min离心10 min;取上清液,加入等体积V(酚)∶V(氯仿)∶V(异丙醇)=25∶24∶1抽提液,充分混匀,10 000 r/min离心20 min;取上层水相,加入0.6倍体积异丙醇,室温沉淀1 h,13 000 r/min 离心5 min;收集沉淀,70%乙醇冲洗并重复一次,室温放置使DNA干燥;使用60 μL ddH2O溶解DNA,利用纯化试剂盒纯化,使用Nanodrop测定宏基因组DNA浓度。
1.3.4 宏基因组测序、质控、组装、微生物物种和功能预测
采用MGIEasy DNA文库制备试剂盒制备测序文库,利用基因测序仪进行双端测序。通过SOAPnuke (v1.5.6)[18]软件对原始下机数据进行以下质控过滤:去除含10%不确定碱基(N碱基)的Reads、去除含测序接头的Reads(有15个碱基或更长的区域比对到接头序列)、去除含50%以上低质量碱基(Q<20的碱基)的Reads。以小麦基因组序列(GCA_002220415.2_Triticum _3.1_genomic.fna)为参考,利用snap-aligner (v1.0beta.23)[19]和samtools (v1.8)[20]过滤宿主序列,用于后续生物信息学分析。
使用IDBA_UD(v1.1.1)[21]对上述有效数据进行组装,通过MetaGeneMark(v2.1.0)[22]对组装序列进行开放阅读框(open reading frames,ORFs)预测。将预测出的ORFs合并,利用CD-Hit(v4.7)[23]开展聚类分析,相似度>95%且比对区域>90%的序列聚类为一个单元,去除重复的冗余序列,构建样品基因集。使用Bowite2 (v1.2.2)[24]将样品的高质量Reads与基因集比对,采用Pathoscope (v1.0)[25]将reads重新分配至“最可能来源”的基因。
利用DIAMOND (v0.8.36)[26]将编码基因映射到nr数据库(Non-redundant protein sequences from GenPept, Swissprot, PIR, PDF, PDB, and NCBI RefSeq) 和KEGG 数据库(Kyoto Encyclopedia of Genes and Genomes),获得相应基因的注释信息。基于nr数据库比对结果,使用MEGAN (v6)[27]进行微生物物种信息注释,并计算该物种的丰度。
2 实验结果
2.1 大曲样品内部结构解析
扫描电镜观察结果显示,对照大曲结构致密,淀粉颗粒排列整齐,微生物和菌丝体数量较少(图1-a和图1-c),而强化大曲结构疏松,淀粉颗粒排列松散,微生物数量较多,有大量菌丝体生长(图1-b和图1-d)。大曲内部结构的变化可能与添加的强化菌株有关。扣囊复膜孢酵母能够分泌多种胞外水解酶,且在营养充足时产生大量菌丝体,该生理特征有利于降解淀粉等大分子物质,同时进一步疏松大曲质地,为其他功能微生物的生长繁殖提供足够的营养和空间,为强化大曲中高效稳定的微生物群落结构形成奠定基础。

a-对照大曲400×;b-强化大曲400×; c-对照大曲1 000×;d-强化大曲1 000×
图1 高温大曲内部结构扫描电镜图
Fig.1 SEM of internal structure in high-temperature Daqu
2.2 微生物群落结构分析
对照大曲(KD)和强化大曲(KS)的宏基因组分别得到了包含5.76 Gbp和5.02 Gbp的有效数据,预测到124 174和112 840个编码基因,与nr数据库和KEGG数据库比对注释到的基因分别为92 617和51 068个。
在属水平上对样品的微生物群落结构进行统计分析,建立差异度指数,即强化大曲(KS)中微生物丰度与对照大曲(KD)中微生物丰度的比值。通过比较2个样品的差异度指数来分析其微生物群落结构差异。大曲微生物群落结构分析结果呈现三大特点(图2):强化大曲中酵母菌丰度高于对照大曲,其中复膜孢酵母属(Saccharomycopsis) 丰度变化最大,强化组丰度是对照组的5.374倍,与实验预期一致,表明强化菌株可在高温大曲中繁殖并发挥作用;且强化大曲中酵母菌属(Saccharomyces) 含量显著增多,差异度指数达2.278,这可能有利于提升后续白酒酿造过程的出酒率;强化大曲中乳酸菌的含量增加,乳酸菌中的乳球菌属(Lactococcus)、片球菌属(Pediococcus)、乳杆菌属(Lactobacillus)、明串珠菌属(Leuconostoc)和魏斯氏菌属(Weissella)在2个样品中的差异度指数分别为3.638、2.586、2.471、2.212和2.144,表明扣囊复膜孢酵母CICC 3307的强化改变了大曲中原有的微生物群落结构,显著提升了大曲中乳酸菌丰度,这可能利于丰富白酒的风味;食源性致病菌含量恒定且有下降趋势,强化大曲中肠杆菌属(Enterobacter)、欧文氏菌属(Erwinia)、葡萄球菌属(Staphylococcus)的菌种丰度降低,其他食源性致病菌包括肠球菌属(Enterococcus)、克洛诺斯杆菌属(Cronobacter)、沙雷菌属(Serratia)、克雷伯菌属(Klebsiella)、埃希氏菌属(Escherichia) 和沙门氏菌属(Salmonella) 的丰度在2个样品中基本保持恒定,表明扣囊复膜孢酵母CICC 3307的强化在一定程度上抑制了大曲中的食源性致病微生物。
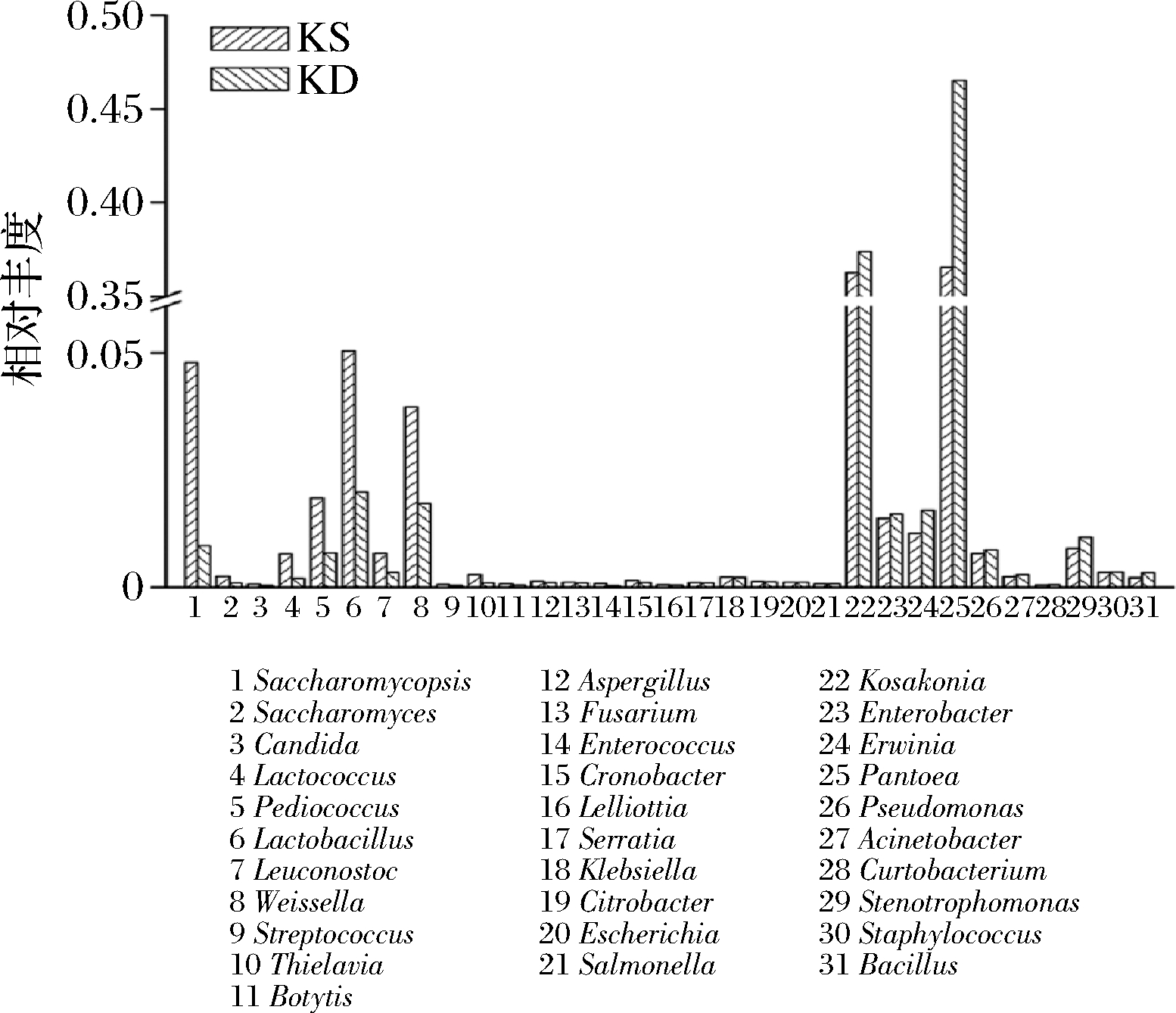
图2 强化大曲(KS)和对照大曲(KD)中微生物物种丰度
Fig.2 Abundance of microbial species in intensified Daqu(KS) and control Daqu(KD)
2.3 功能特性分析
使用KEGG对2个样品的宏基因组信息进行功能注释和代谢分析,比较强化大曲(KS)和对照大曲(KD)在代谢途径上的异同,探究扣囊复膜孢酵母CICC 33077对高温大曲功能特性的影响。
从全局代谢上看,强化大曲主要参与九大代谢途径(图3),涉及功能基因33 690个,比对照大曲提高了3%;强化大曲中参与碳水化合物、氨基酸、核苷酸、脂类物质合成与代谢过程以及膜转运系统方面的编码基因达14 058个,比对照大曲提高了3.5%(表1),这均表明扣囊复膜孢酵母的强化在整体上提升了高温大曲的代谢活力,为大曲原料的充分利用和大曲品质的提高奠定了基础。考虑到该强化菌株分泌高活力淀粉酶的特性,后续研究重点围绕高温大曲的淀粉代谢途径展开。
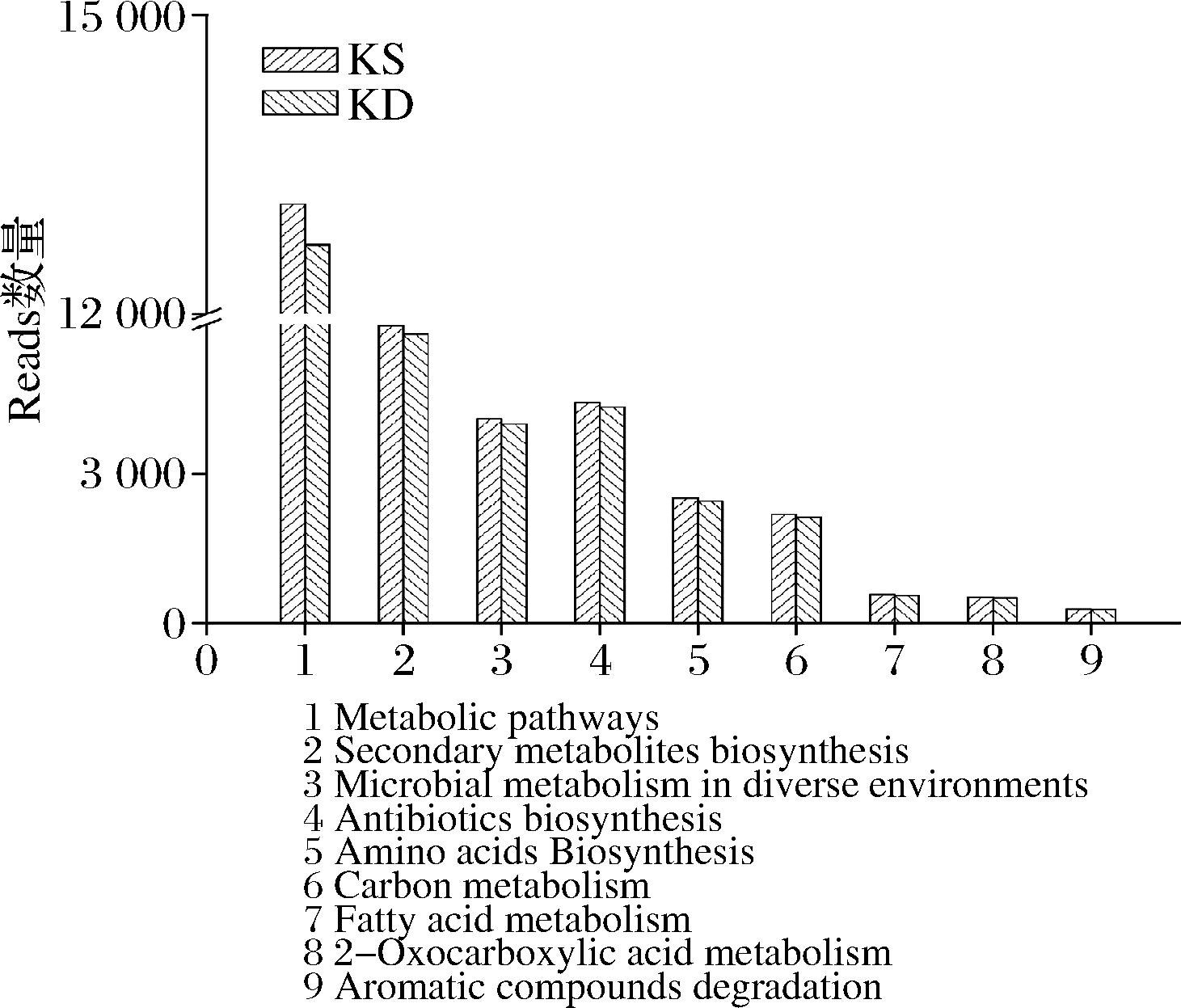
图3 强化大曲(KS)和对照大曲(KD)的全局代谢途径分析
Fig.3 Global metabolic pathway analysis of intensified Daqu(KS) and control Daqu(KD)
2.4 优势微生物与淀粉代谢途径的相关性分析
探究优势微生物,即复膜孢酵母属(Saccharomycopsis)、乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)、明串珠菌属(Leuconostoc)、片球菌属(Pediococcus) 和魏斯氏菌属(Weissella) 与淀粉代谢途径中各代谢酶的相关性(图4)。结果显示,强化大曲(KS)和对照大曲(KD)中参与淀粉代谢途径的代谢酶的种类基本恒定,但强化大曲中糖化酶(EC 3.2.1.133)、α-葡萄糖糖苷酶(EC 3.2.1.20) 等催化淀粉转变为小分子糖的水解酶数量明显高于对照大曲。该结果表明大曲自身代谢途径具有一定稳定性,扣囊复膜孢酵母的强化不会破坏大曲原有的淀粉代谢途径,而是能通过增加复膜孢酵母属、乳酸菌等功能微生物的丰度来增加淀粉代谢酶的数量,从而提高大曲对淀粉的降解能力,提高原料的利用率,为提升高温大曲的品质奠定基础。
表1 强化大曲(KS)和对照大曲(KD)参与的 KEGG代谢途径
Table 1 KEGG metabolic pathways of intensified Daqu(KS) and control Daqu(KD)

功能KEGG 代谢途径Reads数量KDKS碳水化合物代谢淀粉和蔗糖代谢932979糖酵解/糖异生1 0311 052戊糖磷酸途径811833果糖和甘露糖代谢594613半乳糖代谢462478丙酮酸代谢825848乙醛酸和二羧酸代谢484497氨基糖和核糖代谢951964氨基酸代谢半胱氨酸和甲硫氨酸代谢792828甘氨酸、丝氨酸和苏氨酸代谢726744精氨酸和脯氨酸代谢340355组氨酸代谢193206缬氨酸、亮氨酸和异亮氨酸代谢284296核苷酸代谢嘌呤代谢1 6111 664嘧啶代谢1 0901 125脂类代谢甘油磷脂代谢441472脂肪酸合成436453膜转运系统磷酸转移酶系统500532双组份系统755781ABC 转运蛋白320338

图4 强化大曲(KS)和对照大曲(KD)中优势微生物与淀粉代谢途径中代谢酶的相关性
Fig.4 Correlation between dominant microorganisms and metabolic enzymes of starch metabolic pathway in intensified Daqu(KS) and control Daqu(KD)
3 讨论
扣囊复膜孢酵母CICC 33077生长温度和pH值范围广,淀粉酶活力达到12 000 U/g,且酶活力最适反应温度为50 ℃,最适反应pH 4.0,适合芝麻香型白酒高温大曲的发酵环境[16]。本课题组先前的研究将扣囊复膜孢酵母CICC 33077应用于高温大曲生产过程,并从大曲内部结构、微生物指标、感官指标和理化指标等方面对高温大曲进行全面评价,直观反映了扣囊复膜孢酵母CICC 33077对大曲质地、感官、糖化力和液化力等理化特征的改善作用,而对其在高温大曲中的作用机制未进一步研究。本研究比较分析了扣囊复膜孢酵母强化大曲和对照大曲在内部结构、微生物群落结构、物质代谢方面的异同,解析扣囊复膜孢酵母CICC 33077对芝麻香型高温大曲品质改善作用的内在机制。
功能乳酸菌具有丰富的水解酶系,能够产生包括乳酸和乙酸在内的多种风味物质,且兼具益生功能,在提高传统发酵食品的原料利用率,提升产品口感和营养价值等方面发挥重要作用[28-29]。本研究发现扣囊复膜孢酵母CICC 33077的强化改变了大曲中原有的微生物群落结构,显著增加了高温大曲中功能乳酸菌的丰度;且全面提升了高温大曲的代谢活力,重构了高温大曲中功能乳酸菌的淀粉代谢途径(图5),显著增加了淀粉代谢酶的数量(图4),这可能为强化大曲品质的提升奠定了基础。

图5 高温大曲中功能乳酸菌的淀粉代谢途径
Fig.5 Starch metabolism pathway of functional lactic acid bacteria in high-temperature Daqu
4 结论
本研究结果显示,扣囊复膜孢酵母的强化能够疏松大曲内部空间;增加大曲中复膜孢酵母属(Saccharomycopsis)、酵母菌属(Saccharomyces)以及乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)、明串珠菌属(Leuconostoc)、片球菌属(Pediococcus)和魏斯氏菌属(Weissella)等功能乳酸菌的丰度,降低食源性致病菌丰度;全面提升高温大曲的代谢活力,显著增加淀粉代谢相关酶数量。表明扣囊复膜酵母菌的强化能够通过改变高温大曲的微生物群落结构进而影响体系代谢水平。
[1] 高传强.对芝麻香型白酒风格定位及相关技术的探讨[J].酿酒科技, 2014(4):60-64.
GAO C Q.Discussion on the production techniques of sesame-flavor liquor and its style positioning[J].Liquor-Making Science &Technology, 2014(4):60-64.
[2] 姚粟,葛媛媛, 李辉, 等.利用非培养技术研究芝麻香型白酒高温大曲的细菌群落多样性[J].食品与发酵工业, 2012, 38(6):1-6.
YAO S, GE Y Y, LI H, et al.Analysis on bacterial communities in high temperature Daquof sesame flavor liquor through culture-free approach[J].Food and Fermentation Industries, 2012, 38(6):1-6.
[3] KREGER-VANR I J, NELLY J W, et al.The Yeasts:A Taxonomic Study[M].Amsterdam:Elsevier, 2013.
[4] WICKERHAML J, LOCKWOOD L B, PETTIJOHN O G, et al.Starch hydrolysis and fermentation by the yeast Endomycopsis fibuliger[J].Journal of Bacteriology, 1944, 48(4):413-427.
[5] CHIZ M, CHI Z, LIU G L, et al.Saccharomycopsis fibuligeraand its applications in biotechnology[J].Biotechnology Advances, 2009, 27(4):423-431.
[6] 于欣君.扣囊复膜酵母菌酸性蛋白酶基因在解脂耶罗维亚酵母菌中表达和重组酸性蛋白酶的应用[D].青岛:中国海洋大学, 2011.
YU X J.Expression of acid protease gene from Saccharomycopsis fibuligerain Yarrowia lipolyticaand application of the recombinant acid protease[D].Qingdao:Ocean University of China, 2011.
[7] 周衍,张梁, 王正祥, 等.扣囊复膜孢酵母β-葡萄糖苷酶基因在工业酿酒酵母中的表达[J].中国生物工程杂志, 2007, 27(2):64-69.
ZHOU Y, ZHANG L, WANG Z X, et al.Expression of BGLgene from Saccharomycopsis fibuligerain industrial Saccharomyces cerevisiae[J].China Biotechnology, 2007, 27(2):64-69.
[8] 王晓丹,陈美竹, 班世栋, 等.茅台大曲中酵母的分离、鉴定及其功能初探[J].食品科学, 2017, 38(4):51-57.
WANG X D, CHEN M Z, BAN S D, et al.Separation, identification and functional characterization of yeast strains from Moutai-flavor Daqu, a traditional Chinese liquor fermentation starter[J].Food Science, 2017, 38(4):51-57.
[9] 郝文军,刘红霞, 于晓涛, 等.牛栏山白酒酿造过程中扣囊复膜酵母的分离与产物分析[J].酿酒科技, 2019(2):49-52.
HAO W J, LIU H X, YU X T, et al.Isolation of S.fibuligerafrom the production process of Niulanshan liquor and its metabolites[J].Liquor-Making Science &Technology, 2019(2):49-52.
[10] 马美荣,刘小改, 李洪媛, 等.两种扣囊复膜酵母在清香型麸曲白酒生产中的应用[J].食品工业科技, 2021, 42(21):133-139.
MA M R, LIU X G, LI H Y, et al.The application of two kinds of Saccharomycopsis fibuligerain mild-flavour Fuqu Baijiu production[J].Science and Technology of Food Industry, 2021, 42(21):133-139.
[11] 王浩,黄丹, 余东, 等.扣囊复膜酵母菌对固态混菌发酵体系微生物菌群结构及代谢的影响[J].食品与发酵工业, 2021, 47(11):45-52.
WANG H, HUANG D, YU D, et al.Effect of Saccharomycopsis fibuligeraon the microbial community structure and metabolism in mixed solid fermentation system[J].Food and Fermentation Industries, 2021, 47(11):45-52.
[12] 孙思佳,翟磊, 白飞荣, 等.菌株CICC 33077的鉴定及培养基组分的响应面优化[J].中国酿造, 2018, 37(4):44-50.
SUN S J, ZHAI L, BAI F R, et al.Identification of strain CICC 33077 and the optimization of medium composition by response surface methodology[J].China Brewing, 2018, 37(4):44-50.
[13] 孙思佳,翟磊, 白秀彬, 等.一株高产淀粉酶扣囊复膜孢酵母的产酶条件优化及酶学性质研究[J].食品与发酵工业, 2019, 45(21):31-37.
SUN S J, ZHAI L, BAI X B, et al.Optimization of enzyme production conditions and enzymatic properties of a high amylase-production Saccharomycopsis fibuligera[J].Food and Fermentation Industries, 2019, 45(21):31-37.
[14] 孙思佳,翟磊, 许玲, 等.扣囊复膜孢酵母CICC 33077在芝麻香型白酒高温大曲生产中的应用[J].酿酒科技, 2018(7):76-82.
SUN S J, ZHAI L, XU L, et al.Application of Saccharomycopsis fibuligeraCICC 33077 in the production of high-temperature Zhimaxiang Daqu[J].Liquor-Making Science &Technology, 2018(7):76-82.
[15] 戴奕杰,李宗军, 田志强.酱香型白酒大曲和糟醅的真菌多样性分析[J].现代食品科技, 2018, 34(7):97-104.
DAI Y J, LI Z J, TIAN Z Q.Analysis of Maotai-flavor Daquand fungal diversity of fermented grains[J].Modern Food Science and Technology, 2018, 34(7):97-104.
[16] 周健,郭志, 明红梅, 等.优质中高温浓香型大曲主要香味成分的初步研究[J].酿酒科技, 2014(4):11-14.
ZHOU J, GUO Z, MING H M, et al.Preliminary study of main flavoring components of quality high-temperature &medium-temperature Nong-Xiang Daqu[J].Liquor-Making Science &Technology, 2014(4):11-14.
[17] ZHOUJ, BRUNS M A, TIEDJE J M.DNA recovery from soils of diverse composition[J].Applied and Environmental Microbiology, 1996, 62(2):316-322.
[18] CHENY X, CHEN Y S, SHI C M, et al.SOAPnuke:A MapReduce acceleration-supported software for integrated quality control and preprocessing of high-throughput sequencing data[J].GigaScience, 2018, 7(1):gix120.
[19] ZAHARIAM, BOLOSKY W J, CURTIS K, et al.Faster and more accurate sequence alignment with SNAP[EB/OL].2011:arXiv:1111.5572.https://arxiv.org/abs/1111.5572.
[20] LIH, HANDSAKER B, WYSOKER A, et al.The sequence alignment/map format and SAMtools[J].Bioinformatics, 2009, 25(16):2 078-2 079.
[21] PENGY, LEUNG H C M, YIU S M, et al.IDBA-UD:A de novoassembler for single-cell and metagenomic sequencing data with highly uneven depth[J].Bioinformatics, 2012, 28(11):1 420-1 428.
[22] ZHUW H, LOMSADZE A, BORODOVSKY M.Ab initio gene identificationin metagenomic sequences[J].Nucleic Acids Research, 2010, 38(12):e132.
[23] LIW Z, GODZIK A.Cd-hit:A fast program for clustering and comparing large sets of protein or nucleotide sequences[J].Bioinformatics, 2006, 22(13):1 658-1 659.
[24] LANGMEADB, TRAPNELL C, POP M, et al.Ultrafast and memory-efficient alignment of short DNA sequences to the human genome[J].Genome Biology, 2009, 10(3):R25.
[25] FRANCISO E, BENDALL M, MANIMARAN S, et al.Pathoscope:Species identification and strain attribution with unassembled sequencing data[J].Genome Research, 2013, 23(10):1 721-1 729.
[26] BUCHFINKB, XIE C, HUSON D H.Fast and sensitive protein alignment using DIAMOND[J].Nature Methods, 2015, 12(1):59-60.
[27] HUSOND H, AUCH A F, QI J, et al.MEGAN analysis of metagenomic data[J].Genome Research, 2007, 17(3):377-386.
[28] WUL H, LU Z M, ZHANG X J, et al.Metagenomics reveals flavour metabolic network of cereal vinegar microbiota[J].Food Microbiology, 2017, 62:23-31.
[29] HOLZAPFELW H, HABERER P, SNEL J, et al.Overview of gut flora and probiotics[J].International Journal of Food Microbiology, 1998, 41(2):85-101.