大豆中含有多种功能性物质,如维生素、异黄酮、皂苷、低聚糖、磷脂、不饱和脂肪酸等,并且含有丰富的蛋白质以及种类齐全的氨基酸。传统豆浆是将大豆涨泡、磨浆和过滤豆渣后,将浆液煮沸后制成的,由于营养价值丰富,备受人们喜爱[1-2]。研究表明,制作传统豆浆过程中除去的豆渣里仍然含有丰富的营养物质,如膳食纤维、蛋白质、脂肪、碳水化合物、维生素、大豆异黄酮等功能性营养物质[3]。以全大豆为原料将大豆涨泡后使用胶体磨进行粗磨浆,再经过湿法超微粉碎机过200目筛网进行细磨浆,然后进行均质,最后煮沸可制得全营养豆浆。全豆豆浆与传统豆浆相比保留了更多的营养物质,尤其是膳食纤维,而大豆膳食纤维还有通过促进胃肠蠕动和排便,将肠道中大量的细菌、发酵物质带出体外,促进微生态平衡的作用[4]。
由于全豆豆浆中未被过滤的大分子物质难以被小肠消化吸收,所以在人体肠道中主要被利用的部位是结肠,结肠中的微生物可以将全豆豆浆中的膳食纤维等大分子物质转化为具有生物活性的物质,从而影响肠道的生态环境,促进有益菌的生长,抑制有害菌的生长[5-6]。体外消化实验可模拟体内胃和肠的生理条件,比如消化酶的种类、消化时间和温度以及盐离子浓度等条件,对物质进行体外消化[7]。体外结肠发酵是一种体外微生物培养体系,一般是利用人类粪菌进行发酵,促进未被小肠消化吸收的食物残渣在结肠中被肠道微生物利用,进而可以生成短链脂肪酸(short-chain fatty acids,SCFAs)。SCFAs是一些肠道微生物在生长过程中的代谢产物,能够促进结肠上皮细胞的生长和分化,影响结肠上皮细胞的转运;降低肠道pH值,抑制潜在的病原体;调节肠道免疫代谢;还具有减少炎症发生、抗肿瘤和抗肥胖等作用[8],SCFAs还可以有效促进有益菌的生长,提高营养物质的生物利用率。体外模拟结肠发酵模型可以模拟人体肠道菌群及代谢物SCFAs的生长和代谢,对于人体内的肠道微生态和食品微生物消化的研究有极其重要的作用。该模型对于探究食品中的营养素与肠道微生物之间的相互作用关系也有着极其重要的作用[9]。
本研究是以湿法超微粉碎加工制得的全豆豆浆为原料,通过体外消化和发酵实验,探究全豆豆浆对于人体肠道菌群及SCFAs的影响。为科学引导消费者摄入大豆制品、推广全营养大豆提供科学理论基础。
1 材料与方法
1.1 材料与试剂
安徽大豆,江苏无锡金桥批发市场。
唾液淀粉酶、胃脂肪酶、胃蛋白酶、胰脂肪酶、胰蛋白酶、玉米淀粉、蛋白胨、酵母膏、色氨酸、黏蛋白、L-半胱氨酸盐酸盐、胆盐、血红素、NaCl、吐温、KCl、KH2PO4、MgCl2·6H2O、CaCl2·6H2O、MgSO4·7H2O、FeSO4·7H2O、CaCl2·2H2O、MnCl2·4H2O、CoSO4·7H2O、CuSO4·7H2O、ZnSO4·7H2O、NiCl2·6H2O,上海麦克林生物科技有限公司。
1.2 仪器与设备
五连发酵罐(合作设计),上海百伦生物科技有限公司;WT10000X电子天平,常州万泰天平仪器有限公司;ZNCL-GS智能磁力搅拌器,上海越众仪器设备有限公司;QP2010Ultra气相色谱质谱联用仪,日本岛津公司。
1.3 实验方法
1.3.1 样品前处理
挑选并准确称量120 g优质安徽大豆,清洗除去杂质,在4 ℃环境下浸泡18 h。使用含0.5%(质量分数)NaHCO3的沸水对大豆进行热烫处理,将处理过的大豆与720 g纯净水一起进行破壁处理,将粗磨的豆浆再通过湿法超微粉碎机过200目筛网进行细磨浆,然后使用高压均质机在45 MPa下循环均质2遍,加热煮沸后放凉。
1.3.2 体外模拟胃肠消化
参照BRODKORB等[10]的方法,对实验室湿法粉碎制得的全豆豆浆进行口腔、胃和小肠3个阶段的体外模拟消化。
口腔阶段:称取840 mL全豆豆浆与336 mL的口腔微量元素离子液混合1 min, 随后加入4.2 mL 的CaCl2溶液(0.3 mol/L),7.88 g的α-淀粉酶(16 U/mL),加超纯水补充溶液至1 680 mL,在37 ℃下孵育5 min。
胃阶段:将1 680 mL 模拟胃液添加到前一阶段的消化液中,用HCl溶液将pH调节至3.0,再加入0.84 mL 的CaCl2溶液(0.3 mol/L),2.24 g的胃蛋白酶(3 000 U/mL)和2 g的胃脂肪酶(100 U/mL),之后用超纯水补充溶液至3 360 mL,将混合物在37 ℃下孵育2 h。
小肠阶段:将3 360 mL 模拟小肠液添加至前一阶段的消化液中,用NaOH溶液将pH调节至7.0,加入45.76 g的胆盐溶液(133.2 mmol/L),6.72 mL 的CaCl2溶液(0.3 mol/L),2.69 g的胰蛋白酶(250 U/mL)和6.72 g的胰脂肪酶(100 U/mL),用超纯水补充至6 720 mL,将混合物在37 ℃下孵育2 h。
反应结束后,将消化液转移至透析袋(1 000 Da)中,于5 000 mL NaCl透析液(10 mmol/L)中,在4 ℃透析24 h,透析袋内残渣冷冻干燥后作为样品用于后续结肠发酵实验。
1.3.3 体外模拟结肠发酵
发酵实验方案参考SHIME模型[11],结合本实验室之前的研究结果[12-13]综合分析,发现7 d可以稳定肠道微生物,因此确定了7 d稳定加7 d发酵的实验模型,后将全豆豆浆样品经过胃肠消化后未被吸收的物质进行体外发酵。
采集至少3个月以上没有胃肠道疾病或接受抗生素治疗的健康男性和女性(年龄范围为20~28岁)的新鲜粪便各4份,将采集的粪便样品等量混合,消除个体差异后,取10 g粪便于装有90 mL无菌PBS(pH = 6.8)的无菌容器中,充分搅拌混匀后,通过4层无菌纱布过滤掉固体颗粒,然后立即放置于厌氧环境中封闭保存。
3个发酵罐依次模拟人体升结肠、横结肠和降结肠3个肠腔,其中各个结肠肠段的条件如表1所示。
表1 各结肠肠段发酵参数
Table 1 Fermentation parameters of each colon segment
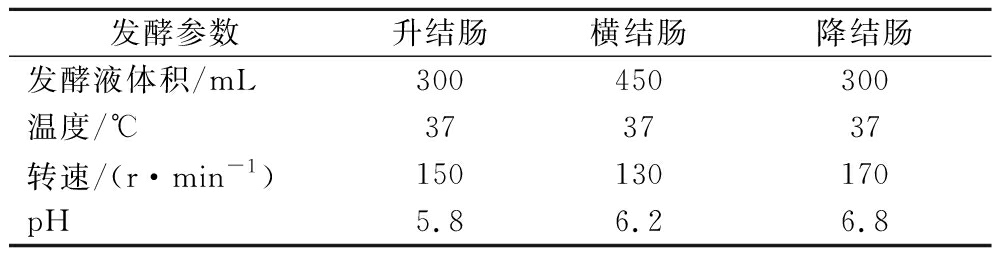
发酵参数升结肠横结肠降结肠发酵液体积/mL300450300温度/℃373737转速/(r·min-1)150130170pH5.86.26.8
通过pH控制系统自动添加HCl溶液(1.5 mol/L)或NaOH溶液(1.5 mol/L)来调节发酵罐中的pH。通过发酵罐自动加热冷凝系统来维持发酵罐中的温度恒定。为了维持发酵罐中严格的厌氧环境,每日3次通入N2以排尽发酵罐中的空气。为了使发酵罐中的微生物正常发酵,每日3次排出废料和补给养料各100 mL。发酵罐中的微生物在粪便菌群接种后连续发酵一周左右趋于稳定水平。之后每日额外在补料罐中加入样品与培养基,7 d后停止发酵。收集发酵前和发酵中第1、2、3、5和7天各个结肠部位的发酵液。
1.3.4 细菌16S rDNA测序分析
用 DNA提取试剂盒提取酵解液中的DNA,再用1%琼脂糖凝胶电泳检测DNA纯度和浓度。引物341F(5′-CCTAYGGGRBGCASCAG-3′)和806R (5′-GGACTACNNGGGTATCTAAT-3′)用于PCR 扩增 V3~V4 可变区。PCR扩增程序:98 ℃预变性1 min;30个循环(98 ℃变性10 s;50 ℃退火30 s;72 ℃延伸30 s),用2%琼脂糖凝胶电泳检测反应产物。
PCR产物经琼脂糖凝胶电泳纯化,使用Qiagen凝胶回收试剂盒回收目标条带。TruSeq DNA PCR-free Sample Preparation Kit 用于构建文库。经过 Qubit 量化和文库检测后,使用 PE250 进行机器测序。使用QIIME2 DADA2进行质量控制、剪枝、去噪、剪接和去除嵌合体,对相似度为97 %的序列进行OTU操作作为分类标准。使用QIIME2软件获取Green Gene数据库结果。
1.3.5 气质联用测定SCFAs
参照LIU等[12]的方法,称取体外发酵产物200 mg,加入碳酸二甲酯1 mL、KHSO4饱和溶液0.1 mL、2-羟基工业酸内标溶液(0.1 mg/mL)0.1 mL,摇匀10 min。然后将混合物4 000 r/min离心10 min以获得碳酸二甲酯相,其为SCFAs。SCFAs标准溶液的制备方法同上(甲酸、乙酸、丙酸、丁酸、异丁酸、戊酸)。质量浓度梯度为0.05、0.1、0.5、0.75、1.0、2.0、3.0、5.0 mg/mL。样品通过GC/MS检测,电离模式为EI,发射电流1 mA,电子能量70 eV;气相色谱柱DB-WAX UI(30 m×0.25 mm×0.25 mm);仪器初始温度设定为 60 ℃,保温 1 min,以 15 ℃/min 的速率升温至 240 ℃,保温 10 min。
1.4 数据分析
采用SPSS 26进行单因素方差分析,Tukey test方法对数据进行组间差异比较,P<0.05被认为具有统计学意义。采用Origin 2021和Graphpad prism 9进行绘图。
2 结果与讨论
2.1 α多样性
Chao 1常用来估计物种总数,是计算微生物群落丰度的指数,指数越大,OTU数目越多,说明样本物种数也越多。由图1-a可知第7天的Chao 1指数显著高于发酵前(P<0.01),随着发酵时间的延长,微生物总数呈现增高的趋势。
Shannon 通常用于评估菌群多样性,Shannon值越大,表示不确定性越大,说明群落中未知因素越多,即群落多样性越高。由图1-b可知,Shannon指数先降低后增高,且在发酵第7天显著高于发酵前(P<0.01),说明发酵过程中,微生物的物种多样性先略微降低后增高,第7天时物种多样性达到最高。
Simpson 是评价种类分布均匀性的常用指标,也是用来估算样品多样性的指数之一,其中稀有物种所起的作用较小。由图1-c可知,微生物的物种分布均匀性保持稳定,在第7天略微升高。
因此在全豆豆浆的干预下,微生物群落丰度显著增高,微生物物种多样性也显著增高,说明全豆豆浆具有提高肠道菌群生物多样性的作用。

a-Chao 1;b-Shannon;c-Simpson
图1 全豆豆浆对肠道菌群α多样性的影响
Fig.1 Effects of whole soybean milk on gut microbiota alpha diversity
注:*代表与D0相比,P<0.05,**代表与D0相比,P<0.01;D0、1、2、3、5、7表示第0、1、2、3、5、7天(下同)
2.2 β多样性
主坐标分析(principal coordinate analysis,PCoA)可以反映不同组之间微生物群落结构的差别,基于PCoA的结果,将多维数据的差异呈现在二维坐标图上,颜色相同的点表示同一分组,点间距离越近提示群落构成差异越小,样品群落组成越相似[14]。由图2可以看出,D0与其他组之间距离较远,D3、D5、D7的距离相对较近,表明D0的群落结构与其他组之间差异显著,发酵前3天菌群群落构成变化明显,后4天群落结构差异变小。进一步说明全豆豆浆可以影响人体肠道菌群的群落结构,且在第3天之后群落结构趋于稳定。

图2 全豆豆浆对肠道菌群β多样性的影响
Fig.2 Effects of whole soybean milk on the beta diversity of gut microbiota
2.3 在门水平上肠道菌群的变化
图3-a和3-b表示体外模拟人体升结肠、横结肠和降结肠在体外厌氧发酵7 d的时间里,在门水平上肠道菌群的相对丰度变化趋势,可以看出在经过体外模拟结肠发酵全豆豆浆过程中,肠道菌群在门水平上主要有Bacteroidetes、Proteobacteria、Firmicutes、Synergistetes、Fusobacteria和Actinobacteria。
随着发酵时间的延长,在发酵的第7天与发酵前相比,Bacteroidetes和Firmicutes的相对丰度相较于D0显著下降(P<0.05,P<0.01),Fusobacteria和Actinobacteria的相对丰度相较于D0均下降但不显著(P>0.05),Proteobacteria 的相对丰度相较于D0显著升高(P<0.01),Synergistetes的相对丰度相较于D0升高但不显著(P>0.05)。说明全豆豆浆在发酵过程中可以促进Proteobacteria和Synergistetes的增长,抑制Bacteroidetes、Firmicutes、Fusobacteria和Actinobacteria的生成,这与YAN等[15]的研究结果类似,这可能是由于豆浆中不可溶的大分子膳食纤维在门水平上改变了肠道微生物的组成。

a-门水平上肠道菌群的丰度;b-门水平上的关键微生物分析
图3 模拟结肠发酵全豆豆浆在门水平上对肠道菌群的影响
Fig.3 Influence of simulated colon fermented whole soybean milk on gut microbiota at phyla level
C0-发酵前;S-升结肠;H-横结肠;J-降结肠;0、1、2、3、5、7表示发酵天数(下同)
2.4 在属水平上肠道菌群的变化
对相对丰度之和排名前13名的菌属进行了分析,由图4-a和4-b可知,在发酵的第7天,Enterobacter、Megasphaera和Pyramidobacter的相对丰度相较于D0均显著升高(P<0.01),Acinetobacter和Phascolarctobacterium的相对丰度相较于D0均上升但不显著(P>0.05),Prevotella和Megamonas的相对丰度较D0显著下降(P<0.01,P<0.05),Sutterella和Bacteroides的相对丰度相较于D0均下降但不显著(P>0.05)。
有研究表明,Enterobacter通过产生的胆碱及其受体α7nAChR介导视黄酸和转化生长因子-β(transforming growth factor-β,TGF-β)上调,从而诱导树突状细胞的免疫耐受能力,促进调节性T细胞(Treg)分化,进而保护小鼠免受硫酸葡聚糖钠盐(dextran sulfate sodium,DSS)诱导的结肠炎[16]。Prevotella属于拟杆菌门,它可以在人体内水解乙酸、丁酸和异戊酸,并且对胆汁酸具有抗性[17]。Megamonas是一种亚洲人特有的菌属,其丰度变化与人的饮食习惯有极其紧密的关联[18]。有研究报道,肥胖人群中的Prevotella、Megamonas、Fusobacterium和Blautia的相对丰度显著增加[19]。此外,Sutterella与溃疡性结肠炎的缓解呈负相关[20]。因此,全豆豆浆可以抑制Prevotella和Megamonas等有害菌的增殖,调节脂代谢和缓解小鼠肠道疾病的菌属,说明其具有潜在的改善脂代谢及维护肠道健康作用。

a-属水平上肠道菌群的丰度;b-属水平上的关键微生物分析
图4 模拟结肠发酵全豆豆浆在属水平上对肠道菌群的影响
Fig.4 Influence of simulated colon fermentedwhole soybean milk on gut microbiota at the genus level
2.5 发酵液中微生物代谢功能的变化
京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)是一个整合基因组、化学和系统功能信息的集成数据库。在KEGG数据库中,可分为3个层次,将数据库与发酵微生物结果对比可以得出在不同层次上具有显著差异的代谢通路。图5-a为在KEGG通路上的主成分分析(principal component analysis,PCA)图,反映了在KEGG通路中不同组之间微生物群落结构的差别,结果显示,D3、D5和D7距离较近,但与D0距离较远,在发酵过程中群落结构与发酵前发生了明显变化,且在D3之后趋于稳定。Level 1结果如图5-b表示,全豆豆浆发酵后微生物代谢途径发生了变化,发酵后基因主要富集在环境信息处理、细胞过程和人类疾病方面,而发酵前基因富集主要集中在生物体系统、遗传信息处理和新陈代谢方面。如图5-c所示,在Level 2水平上对代谢途径进行LDA分析,得分越高说明相关性越强,发现全豆豆浆能够增强循环系统、脂质代谢、排泄系统、细胞运动性、外来生物的降解和代谢、环境适应以及萜类和多酮类化合物的代谢。由图5-d可以看出,Level 3筛选出的是代谢途径中最显著的代谢通路,结果进一步显示全豆豆浆可以促进ZnSO4的新陈代谢、磷酸酶转移、肽聚糖的生物合成以及氨基酸和生物合成。进一步说明全豆豆浆大分子物质可以影响肠道菌群,提高宿主肠道中营养物质的合成及其代谢能力。

a-KEGG通路上的PCA;b-Level 1上的微生物代谢水平;c-Level 2上的微生物代谢水平;d-Level 3上的微生物代谢水平
图5 全豆豆浆代谢功能预测
Fig.5 Prediction of metabolic function of whole soybean milk
2.6 全豆豆浆对SCFAs的影响
SCFAs,也称挥发性脂肪酸,是指碳原子数低于6的饱和脂肪酸,主要包括甲酸、乙酸、丙酸、异丁酸、丁酸、异戊酸和戊酸[21]。SCFAs可以作用于宿主局部和全身区域的多个靶点,在宿主的新陈代谢和免疫中发挥着重要作用[22]。如图6所示,在厌氧发酵7 d后,在升结肠、横结肠和降结肠肠腔内乙酸和丁酸的浓度都极显著升高(P<0.01),甲酸、丙酸、异丁酸和正戊酸的浓度也显著升高(P<0.05)。
如图6-b所示,在升结肠、横结肠和降结肠3个肠腔中乙酸的浓度随发酵时间的延长呈上升趋势。作为含量最丰富的SCFAs,乙酸的来源广泛,很多细菌均可产生,肠道菌群产生的乙酸主要是穿过肠上皮屏障,从而进入外周循环系统发挥作用,乙酸在肠道中主要参与促进胰岛素敏感性和增强饱腹感的作用[23-24]。而且,高乙酸盐饮食可降低病原体毒力基因的表达,增加肠道中调节性T细胞的数量,防止病原体感染肠道[25]。有研究表明,黄芪多糖促产乙酸菌生长,或能缓解脂肪肝的形成[26]。
如图6-c所示,肠道菌群产生的丙酸含量随着发酵时间的延长先增高,到第5天之后开始下降,但总体产量与发酵第1天相比是增多的,这可能与微生物的代谢活性有关。肠道菌群产生的丙酸通过门静脉循环到达肝脏,经过草酰乙酸的转化和蛋白激酶的活化,抑制胆固醇的合成,促进脂肪酸的氧化[27]。丙酸能够通过上调HECT结构域E3泛素蛋白连接酶2,促进常染色组蛋白赖氨酸N-甲基转移酶2蛋白酶体降解,抑制结直肠癌发生[28]。有研究表明,丙酸盐处理可减轻高脂饮食诱导ApoE-/-小鼠的动脉粥状硬化和高胆固醇血症;丙酸增加肠内Treg及白细胞介素-10(interleukin,IL-10)水平,抑制肠上皮细胞表达胆固醇转运蛋白Npc1L1,从而降低血液胆固醇水平[28]。
进一步发现,在升结肠、横结肠和降结肠3个肠腔中,丁酸的浓度随发酵时间的延长呈上升趋势(图6-d)。丁酸作为结肠上皮细胞的主要能量来源,对结肠上皮屏障功能的维持、免疫细胞的调节和上皮细胞的凋亡均起着重要的作用[29]。丁酸可以通过促进肠道M2巨噬细胞极化,经WNT-ERK1/2轴,恢复肠上皮杯状细胞的数量和功能,从而促进了结肠炎中的肠道黏液屏障修复[30]。有研究表明,丁酸盐可在体外及体内诱导Treg细胞的分化,并缓解小鼠的结肠炎[31]。
由此,全豆豆浆经消化后的物质可以在人体肠道菌群的发酵下代谢产生乙酸、丙酸和丁酸等SCFAs。如前所述,SCFAs不仅可以保护肠道屏障,还可以调节肠道免疫,以及促进肠道环境酸化,从而抑制有害微生物的生长。

a-甲酸;b-乙酸;c-丙酸;d-丁酸;e-异丁酸;f-正戊酸
图6 全豆豆浆在结肠发酵过程中SCFAs的变化
Fig.6 Changes of SCFAs during colonic fermentation of whole soybean milk
注:*代表在升结肠肠腔内与D0相比(P<0.05),**代表升结肠肠腔内与D0相比(P<0.01);#代表在横结肠肠腔内与D0相比(P<0.05),##代表横结肠肠腔内与D0相比(P<0.01);&代表在降结肠肠腔内与D0相比(P<0.05),&&代表在降结肠肠腔内与D0相比(P<0.01)
3 结论
综上所述,全豆豆浆经胃肠模拟消化后,部分大分子物质不能被吸收利用进而可能会进入结肠被肠道菌群利用。在全豆豆浆消化残渣经过模拟人体结肠发酵7 d后,由Chao 1指数与Shannon指数变化趋势可以得出,微生物群落丰度与物种多样性呈上升趋势,并在发酵的第7天显著高于发酵前;由β多样性分析也可以得出,全豆豆浆能够改变人体肠道菌群的结构;在门水平上,与发酵前相比,第7天时Proteobacteria和Synergistetes的相对丰度升高,Bacteroidetes、Firmicutes、Fusobacteria和Actinobacteria的相对丰度降低;在属水平上,与发酵前相比,第7天时Enterobacter、Megasphaera、Pyramidobacter、Acinetobacter和Phascolarctobacterium的相对丰度升高,Prevotella、Megamonas、Sutterella和Bacteroides的相对丰度降低;全豆豆浆大分子物质还可以增强代谢酶的活性,提高机体营养物质的合成与代谢能力。体外厌氧发酵7 d后,乙酸、丙酸和丁酸的浓度显著升高。全豆豆浆未被胃肠消化的部分在肠道中可以促使肠道菌群结构和组成发生改变,进而促进了SCFAs的产生。因此,全豆豆浆具有促进肠道健康的功效,为科学地指导消费者摄入大豆制品、推广全豆食品提供科学理论依据。
[1] 王艳萍, 蔚钏, 郗宏波, 等.发酵型酸豆乳工艺条件的研究[J].中国乳品工业, 2013, 41(3):61-64.
WANG Y P, YU C, XI H B, et al.Study on fermentation processing techonology of the soybean yogurt[J].China Dairy Industry, 2013, 41(3):61-64.
[2] HASSAN SOHOULI M, LARI A, FATAHI S, et al.Impact of soy milk consumption on cardiometabolic risk factors:A systematic review and meta-analysis of randomized controlled trials[J].Journal of Functional Foods, 2021, 83:104499.
[3] DU Y, ZHANG Q, ZHAO X Y, et al.Effect of reverse micelle on physicochemical properties of soybean 7S globulins[J].Journal of Food Engineering, 2020, 282:110026.
[4] SHUKLA A, BRANDSCH C, BETTZIECHE A, et al.Isoflavone-poor soy protein alters the lipid metabolism of rats by SREBP-mediated down-regulation of hepatic genes[J].The Journal of Nutritional Biochemistry, 2007, 18(5):313-321.
[5] BEZEK K, BARLI -MAGANJA D.Dietary polyphenols and their effect on the gut microbiota and human health[M]//Zdravje delovno aktivne populacije/Health of the Working-Age Population Koper Založba Univerze na Primorskem, 2020.
-MAGANJA D.Dietary polyphenols and their effect on the gut microbiota and human health[M]//Zdravje delovno aktivne populacije/Health of the Working-Age Population Koper Založba Univerze na Primorskem, 2020.
[6] WICI SKI M, GBALSKI J, MAZUREK E, et al.The influence of polyphenol compounds on human gastrointestinal tract microbiota[J].Nutrients, 2020, 12(2):350.
SKI M, GBALSKI J, MAZUREK E, et al.The influence of polyphenol compounds on human gastrointestinal tract microbiota[J].Nutrients, 2020, 12(2):350.
[7] CAMPOS-VEGA R, V ZQUEZ-S
ZQUEZ-S NCHEZ K, L
NCHEZ K, L PEZ-BARRERA D, et al.Simulated gastrointestinal digestion and in vitro colonic fermentation of spent coffee (Coffea arabica L.):Bioaccessibility and intestinal permeability[J].Food Research International, 2015, 77:156-161.
PEZ-BARRERA D, et al.Simulated gastrointestinal digestion and in vitro colonic fermentation of spent coffee (Coffea arabica L.):Bioaccessibility and intestinal permeability[J].Food Research International, 2015, 77:156-161.
[8] CHAMBERS E S, BYRNE C S, MORRISON D J, et al.Dietary supplementation with inulin-propionate ester or inulin improves insulin sensitivity in adults with overweight and obesity with distinct effects on the gut microbiota, plasma metabolome and systemic inflammatory responses:A randomised cross-over trial[J].Gut, 2019, 68(8):1 430-1 438.
[9] BOTHE M K, MAATHUIS A J H, BELLMANN S, et al.Dose-dependent prebiotic effect of lactulose in a computer-controlled in vitro model of the human large intestine[J].Nutrients, 2017, 9(7):767.
[10] BRODKORB A, EGGER L, ALMINGER M, et al.INFOGEST static in vitro simulation of gastrointestinal food digestion[J].Nature Protocols, 2019, 14(4):991-1 014.
[11] LIU L, WANG Q, WU X Y, et al.Vancomycin exposure caused opportunistic pathogens bloom in intestinal microbiome by simulator of the human intestinal microbial ecosystem (SHIME)[J].Environmental Pollution, 2020, 265:114399.
[12] LIU C, GUO Y H, CHENG Y L, et al.A colon-targeted delivery system of torularhodin encapsulated in electrospinning microspheres, and its co-metabolic regulation mechanism of gut microbiota[J].Food Hydrocolloids, 2023, 135:108189.
[13] XIANG S S, YE K, LI M, et al.Xylitol enhances synthesis of propionate in the colon via cross-feeding of gut microbiota[J].Microbiome, 2021, 9(1):62.
[14] 田小平, 夏军, 卫静雯, 等.基于16S rDNA测序研究当归芍药散调控肠道菌群改善SAMP8小鼠认知能力的作用机制[J].中国实验方剂学杂志, 2022, 28(24):26-34.
TIAN X P, XIA J, WEI J W, et al.Mechanisms of Danggui Shaoyaosan in improving cognitive ability in SAMP8 mice by regulating gut microbiota via 16S rDNA sequencing[J].Chinese Journal of Experimental Traditional Medical Formulae, 2022, 28(24):26-34.
[15] YAN H L, LU J M, WANG Y F, et al.Intake of total saponins and polysaccharides from Polygonatum kingianum affects the gut microbiota in diabetic rats[J].Phytomedicine, 2017, 26:45-54.
[16] LI Q Q, SUN X, YU K Y, et al.Enterobacter ludwigii protects experimental colitis through CD103+DC and Treg cells[J].2020.https://doi.org/10.21203/rs.3.rs-69321/v1.
[17] VANDEPUTTE D, KATHAGEN G, D’HOE K, et al.Quantitative microbiome profiling links gut community variation to microbial load[J].Nature, 2017, 551(7681):507-511.
[18] LIAO M, XIE Y L, MAO Y, et al.Comparative analyses of fecal microbiota in Chinese isolated Yao population, minority Zhuang and rural Han by 16 sRNA sequencing[J].Scientific Reports, 2018, 8(1):1-10.
[19] CHIU C M, HUANG W C, WENG S L, et al.Systematic analysis of the association between gut flora and obesity through high-throughput sequencing and bioinformatics approaches[J].BioMed Research International, 2014, 2014:906168.
[20] KAAKOUSH N O.Sutterella species, IgA-degrading bacteria in ulcerative colitis[J].Trends in Microbiology, 2020, 28(7):519-522.
[21] WU D T, NIE X R, GAN R Y, et al.In vitro digestion and fecal fermentation behaviors of a pectic polysaccharide from okra (Abelmoschus esculentus) and its impacts on human gut microbiota[J].Food Hydrocolloids, 2021, 114:106577.
[22] RINGSEIS R, GESSNER D K, EDER K.The gut-liver axis in the control of energy metabolism and food intake in animals[J].Annual Review of Animal Biosciences, 2020, 8:295-319.
[23] DELGADO-ANDRADE C, PASTORIZA DE LA CUEVA S, PEINADO M J, et al.Modifications in bacterial groups and short chain fatty acid production in the gut of healthy adult rats after long-term consumption of dietary Maillard reaction products[J].Food Research International, 2017, 100:134-142.
[24] FROST G, SLEETH M L, SAHURI-ARISOYLU M, et al.The short-chain fatty acid acetate reduces appetite via a central homeostatic mechanism[J].Nature Communications, 2014, 5(1):1-11.
[25] YAP Y A, MCLEOD K H, MCKENZIE C I, et al.An acetate-yielding diet imprints an immune and anti-microbial programme against enteric infection[J].Clinical &Translational Immunology, 2021, 10(1):e1233.
[26] HONG Y, SHENG L L, ZHONG J, et al.Desulfovibrio vulgaris, a potent acetic acid-producing bacterium, attenuates nonalcoholic fatty liver disease in mice[J].Gut Microbes, 2021, 13(1):1930874.
[27] PEREDO-LOVILLO A, ROMERO-LUNA H E, JIMÉNEZ-FERN NDEZ M.Health promoting microbial metabolites produced by gut microbiota after prebiotics metabolism[J].Food Research International, 2020, 136:109473.
NDEZ M.Health promoting microbial metabolites produced by gut microbiota after prebiotics metabolism[J].Food Research International, 2020, 136:109473.
[28] RYU T Y, KIM K, HAN T S, et al.Human gut-microbiome-derived propionate coordinates proteasomal degradation via HECTD2 upregulation to target EHMT2 in colorectal cancer[J].The ISME Journal, 2022, 16(5):1 205-1 221.
[29] DING Y, YAN Y M, PENG Y J, et al.In vitro digestion under simulated saliva, gastric and small intestinal conditions and fermentation by human gut microbiota of polysaccharides from the fruits of Lycium barbarum[J].International Journal of Biological Macromolecules, 2019, 125:751-760.
[30] LIANG L P, LIU L, ZHOU W Y, et al.Gut microbiota-derived butyrate regulates gut mucus barrier repair by activating the macrophage/WNT/ERK signaling pathway[J].Clinical Science (London, England:1979), 2022, 136(4):291-307.
[31] FURUSAWA Y, OBATA Y, FUKUDA S, et al.Commensal microbe-derived butyrate induces the differentiation of colonic regulatory T cells[J].Nature, 2013, 504(7480):446-450.