我国发酵乳历史悠久,但乳制品市场起步晚,且发酵剂大量依赖于进口,长此以往将阻碍我国发酵乳事业的步伐[1]。研发具有自主知识产权的优良发酵剂是促进我国乳业发展的基础工作[2],要从传统发酵乳制品入手,选择具有优良特性并可作为发酵剂的菌株是非常重要的。乳酸菌能够发酵碳水化合物产生大量乳酸[3],在自然界中分布广泛,物种多样性丰富。
嗜热链球菌(Streptococcus thermophilus)被称为第二大工业乳酸菌菌种,是乳品发酵剂的主要菌株之一[4]。它可以通过同型乳酸发酵的糖酵解途径将牛乳中的乳糖转化成乳酸,同样还能产生其他酸类、醛类、酮类等化学物质,影响发酵乳成品的风味和质地[5],并抑制有害微生物生长。长期生长在牛乳环境中使其糖类代谢能力退化[6],能够利用半乳糖和果糖的只占少数[7]。嗜热链球菌细胞中,负责转运碳水化合物的系统有四大类,分别是:同向转运体,指同时向同一方向转运2种或更多离子或分子的系统;磷酸转移酶转运系统(phosphotransferase system,PTS),一种仅存在于微生物细胞内的系统,主要依赖于磷酸级联反应将糖磷酸化后运输到细胞内[8];通透酶是一种膜蛋白,用于提高细胞膜对特定分子的通透性;ABC 转运体是一种膜转运蛋白,实现糖类的跨膜运输。PTS是嗜热链球菌摄取己糖、己糖醇和双糖等糖类物质时的主要转运系统[9]。
糖代谢的功能为细胞生长发育繁殖提供所需的能量,是嗜热链球菌代谢功能中的重要组成部分。学者发现所有嗜热链球菌都能够代谢乳糖、葡萄糖和果糖,但代谢其他碳水化合物的能力具有菌株特异性[10-11]。嗜热链球菌已在食品中广泛应用,一些制造商使用多种嗜热链球菌菌株来扩展产品风味和质地。高通量测序提高了全基因组的测序效率,全基因组测序在基因组水平上对细菌的功能提供了全新的认识。这些新兴技术释放了微生物全基因组关联研究的潜力,可以用于剖析已知表型性状(如碳水化合物代谢)的遗传基础。了解某些基因型-表型关联为更好地探索技术上有用的微生物带来了机会,并应用于食品技术中的发酵剂培养[12]。
本研究以27株分离自自然发酵乳的嗜热链球菌为试验菌株,经碳水化合物鉴定生化试剂条鉴定后,对比分离株对不同糖类的利用情况,并通过基因组重测序和相关性分析,定位与菌株代谢碳水化合物特性相关的关键基因。以期更好地了解嗜热链球菌的糖利用情况,为促进新产品的开发提供理论基础,并为嗜热链球菌发酵食品中糖种类的选择提供依据。
1 材料与方法
1.1 菌株信息
27株嗜热链球菌均来自于内蒙古农业大学乳酸菌菌种资源库(lactic acid bacteria collection center,LABCC),其中,11株分离自蒙古国酸牛奶中,1株分离自中国内蒙古自治区的酸马奶中,1株分离自中国西藏自治区的酸牦牛奶中,1株分离自青海省的酸山羊奶中,2株分离自青海省的酸牦牛奶中,其余11株分离自新疆自治区的酸牛奶中,具体信息见表1。
表1 27株嗜热链球菌菌株信息
Table 1 Information of 27 Streptococcus thermophilus strains

GenBank登录号LABCC编号分离地分离源分离时间GCA_021294235.1IMAU20587蒙古国后杭盖省臣赫尔苏木酸牛奶2009年GCA_021293975.1IMAU205623蒙古国后杭盖省塔日亚特苏木次老图河酸牛奶2009年GCA_021293985.1IMAU205622蒙古国后杭盖省塔日亚特苏木次老图河酸牛奶2009年GCA_021294135.1IMAU205607蒙古国后杭盖省塔日亚特苏木塔日赫湖酸牛奶2009年GCA_021296775.1IMAU20433蒙古国库苏古尔省库苏古尔湖酸牛奶2009年GCA_021296655.1IMAU20465蒙古国库苏古尔省扎尔格朗特苏木酸牛奶2009年GCA_021293535.1IMAU20764蒙古国乌兰巴托市汗搭盖图苏木酸牛奶2009年GCA_021294555.1IMAU20542蒙古国扎布汗省查干海尔汗苏木酸牛奶2009年GCA_021293695.1IMAU20741蒙古国中央省巴音杭盖苏木酸牛奶2009年GCA_021293805.1IMAU20730蒙古国中央省巴音杭盖苏木酸牛奶2009年GCA_021293715.1IMAU20742蒙古国中央省巴音杭盖苏木酸牛奶2009年GCA_021293275.1IMAU10630中国内蒙古呼伦贝尔盟海拉尔市酸马奶2009年GCA_021292975.1IMAU40071中国青海省海北州天峻县酸牦牛奶2005年GCA_021293005.1IMAU40024中国青海省海南州共和县江西沟乡酸牦牛奶2005年GCA_021293035.1IMAU40148中国青海省海西州德令哈市蓄集乡酸山羊奶2005年GCA_021292675.1IMAU62015中国西藏自治区拉萨市当雄县格达乡甲多村酸牦牛奶2012年GCA_021292685.1IMAU32090中国新疆伊犁哈萨克自治州巩留县七乡酸牛奶2012年GCA_021292635.1IMAU32092中国新疆伊犁哈萨克自治州巩留县七乡酸牛奶2012年GCA_021292615.1IMAU32109中国新疆伊犁哈萨克自治州巩留县七乡酸牛奶2012年GCA_021292295.1IMAU32186中国新疆伊犁哈萨克自治州巩留县七乡酸牛奶2012年GCA_021292435.1IMAU32148中国新疆伊犁哈萨克自治州特克斯二乡酸牛奶2012年GCA_021292535.1IMAU32126中国新疆伊犁哈萨克自治州特克斯镇酸牛奶2012年GCA_021292485.1IMAU32144中国新疆伊犁哈萨克自治州特克斯镇酸牛奶2012年GCA_021292475.1IMAU32139中国新疆伊犁哈萨克自治州特克斯镇酸牛奶2012年GCA_021292665.1IMAU32084中国新疆伊犁哈萨克自治州特克斯镇酸牛奶2012年GCA_021292595.1IMAU32140中国新疆伊犁哈萨克自治州特克斯镇酸牛奶2012年GCA_021292335.1IMAU32589中国新疆伊犁哈萨克自治州昭苏县军马场酸牛奶2012年
1.2 材料与试剂
主要材料及试剂:API 50CHL碳水化合物鉴定试剂条,BIOMERIEUX公司;M17固体、液体培养基,青海海博生物技术有限公司;全脂乳粉、PBS、DL2000 DNA Marker,TaKaRa公司;Wizard® Genomic DNA Purification Kit,PROMEGA公司;5×TBE电泳缓冲液、琼脂糖,维百奥(北京)生物科技有限公司;6×DNA Loading Buffer,天根生化科技(北京)有限公司;核酸染料GELVIEW,北京百泰克生物技术有限公司。
本试验的菌株全基因组测序由上海美吉生物医药科技有限公司完成。
1.3 仪器与设备
SJ-3F型pH计,上海精密科学仪器有限公司;AR2202CN电子天平,奥豪斯仪器上海有限公司;ZHJH-C1214C超净台,上海智城分析仪器制造有限公司;DYY-12电泳仪,北京六一仪器厂;HiSeq4000测序仪,美国Illumina公司;BX50光学显微镜,日本奥林巴斯;Vortex-genie2漩涡振荡器,美国Scientific Industries公司;GDS-8000凝胶成像仪,美国UVP公司;LRH-250电热恒温培养箱,上海一恒科技有限公司;5810R高速控温离心机,德国Eppendorf公司;HA-300M全自动高压蒸汽灭菌器,日本HIRAYAMA公司。
1.4 实验方法
1.4.1 菌种活化及基因组DNA的提取
将于安瓿管中保藏的嗜热链球菌分离株接种于脱脂乳中活化,然后将活化液体混匀后吸取2%于M17液体培养基中,传代2次,37 ℃培养24 h。且每一次传代要进行革兰氏染色后于显微镜下观察其形态是否单一呈紫色链球状。取出培养液于离心机中3 000 r/min离心10 min,用PBS清洗2次后收集菌泥,提取基因组DNA[13]。
1.4.2 DNA质控及基因组重测序
提取DNA后,测定DNA液的浓度,在1.8~2.0间判断合格,并用1%琼脂糖凝胶电泳测定基因组DNA的纯度和完整度。检测合格的基因组DNA经16S rRNA测序,确定为目标菌后用Illumina HiSeq 4000平台测序[14]。
1.4.3 基因组拼接组装
将27株嗜热链球菌的基因组重测序(已去除接头及阳性对照序列)过滤到100×;接着利用SOAPdenovo 程序包选择合适的kmer值对过滤后的数据进行拼接组装和单碱基校正。选取Scaffold数量较小、N50和N90较长、GC含量与参考基因组较接近的拼接结果;最后,选取组装结果较好的序列进行后续soap验证及校验[15]。
1.4.4 功能基因的预测与注释
用RAST(http://rast.nmpdr.org/)网上在线工具注释27株嗜热链球菌的全基因组[16],并记录其基因组大小,GC含量,CDS数量,RNA数量。
1.4.5 碳水化合物代谢特性研究方法
利用API 50CHL碳水化合物鉴定生化试剂条对27株嗜热链球菌糖利用情况进行表型研究。首先,将菌株活化传代至第三代,混匀将菌悬液加到鉴定试剂条上的50个微生化管中,最后用无菌矿物油封管,静置于30 ℃培养。24 h及48 h各观察一次结果。试剂条不变色为阴性反应,变为绿色为弱阳性反应,变为黄色(25号管变为黑色)为阳性反应,以48 h观察到的结果作为最终结果[17]。
1.5 生物信息学分析
1.5.1 碳水化合物活性酶基因注释
用CAZyme数据库(http://csbl.bmb.uga.edu/db CAN/)对样本基因组序列进行碳水化合物活性酶的注释;后根据结果在CAZy(http://www.cazy.org/)网页上查询详细的碳水化合物活性酶家族信息。
1.5.2 系统发育树的构建
整理RAST网上在线工具对嗜热链球菌基因组的注释,将27株菌的多糖代谢基因整理在文件中,利用网站MAFFT(MAFFT
1.5.3 相关性分析
27株嗜热链球菌的表型数据分别与基因组的注释结果和碳水化合物活性酶注释结果,利用斯皮尔曼的方法进行相关性分析,设置相关性阈值为±0.5,且当假设检验P<0.05时,其相关性显著。
2 结果与分析
2.1 嗜热链球菌碳水化合物利用能力评估
嗜热链球菌不同菌株之间的酶系统存在着差异,对不同碳源的利用能力也都不相同[20],基于这一现象,本研究利用碳水化合物鉴定试剂条对嗜热链球菌碳水化合物代谢进行研究,探究这27株嗜热链球菌利用碳源的种类,为之后的碳水化合物代谢中的功能基因分析提供表型数据,试验结果图1所示。

图1 27株嗜热链球菌对API 50CHL糖利用热图
Fig.1 Heat map of API 50CHL sugar utilization of 27 Streptococcus thermophilus strains
API 50CHL碳水化合物鉴定试剂条所涉及的49种碳水化合物中,葡萄糖、乳糖、蔗糖可以被27株嗜热链球菌利用,且其中有12株嗜热链球菌还可以利用核糖、半乳糖、果糖、甘露糖、N-乙酰葡萄糖胺、苦杏仁苷、熊果苷、水杨苷、纤维二糖、麦芽糖、海藻糖、龙胆二糖。根据表型结果,将此27株嗜热链球菌分为2组进行后续功能基因分析,第一组为只能利用葡萄糖、乳糖、蔗糖的15株菌,第二组为还可以利用其它12种糖的12株菌,如表2所示。结果表明有部分嗜热链球菌显示半乳糖阴性,与前人研究相符,并不是由于缺乏与半乳糖发酵的基因,这种表型是由于半乳糖激酶基因(galK)的启动子中存在突变,导致表达水平低[21]。
表2 实验用菌分组表
Table 2 Grouping table of experimental bacteria

分组菌名可利用的糖类组一IMAU20433,IMAU20465,IMAU20542,IMAU205607,IMAU205622,IMAU205623,IMAU20730,IMAU20742,IMAU32084,IMAU32139,IMAU32140,IMAU32144,IMAU32148,IMAU32589,IMAU32186葡萄糖、乳糖、蔗糖组二IMAU10630,IMAU20587,IMAU20741,IMAU20764,IMAU32090,IMAU32092, IMAU32109,IMAU32126,IMAU40024,IMAU40071,IMAU40148,IMAU62015葡萄糖、乳糖、蔗糖、核糖、半乳糖、果糖、甘露糖、N-乙酰葡萄糖胺、苦杏仁苷、熊果苷、水杨苷、纤维二糖、麦芽糖、海藻糖、龙胆糖
2.2 27株嗜热链球菌基因组的一般特点
27株嗜热链球菌基因组的基本信息如表3所示,嗜热链球菌的全基因组序列长度平均为(1.80±0.06)Mb。在嗜热链球菌基因组中共预测到的编码序列(coding sequence,CDS)为1 871~2 043个,平均为(1 959±51.10)个CDS;GC含量整体变化较稳定,平均值为(38.87±0.18)%。此外,在这27株嗜热链球菌基因组中均未检测到质粒。
表3 27株嗜热链球菌基因组一般特点表
Table 3 General characteristics of the genomes of 27 S. thermophilus strains

菌株保藏号基因组大小/MbGC含量/%CDSs/个RNA数量IMAU106301.7938.901 93239IMAU204331.6939.101 87142IMAU204651.7239.001 93136IMAU205421.7 39.101 88838IMAU205871.7639.101 90539IMAU207301.8338.901 99138IMAU207411.8638.901 99949IMAU207421.8338.901 99837IMAU207641.7939.001 93637IMAU320841.8238.601 97546IMAU320901.8538.602 01642IMAU320921.8438.602 00845IMAU321091.7339.101 92838IMAU321261.8638.702 02244IMAU321391.8438.702 02246IMAU321401.7139.101 89240IMAU321441.8438.701 93638IMAU321481.8538.702 02743IMAU321861.8438.601 99448IMAU325891.8438.701 92637IMAU400241.8538.901 98940IMAU400711.7239.001 88038IMAU401481.8638.902 04339IMAU620151.8338.901 99045IMAU2056071.8238.801 99041IMAU2056221.7239.101 90541IMAU2056231.7239.001 90637
2.3 嗜热链球菌糖代谢功能基因注释
两组间碳水化合物代谢基因统计图见图2,根据非配对T检验,对2组的碳水化合物代谢基因拷贝数进行差异分析,得到其在多糖代谢基因上具有显著差异(P<0.05)。在多糖代谢基因中包括2个子系统,一个是糖原代谢基因,与麦芽低聚糖利用的酶有关,另外一个是链球菌中的α-淀粉酶位点基因,此系统同样包含参与麦芽糖/麦芽糖糊精利用的基因[22],这一基因差异结果和2.1节中的表型特征相同,组一中的嗜热链球菌不能利用麦芽糖,而组二中的嗜热链球菌可利用麦芽糖。

图2 27株嗜热链球菌碳水化合物代谢基因统计
Fig.2 Carbohydrate metabolism gene statistics of 27 S. thermophilus strains
2.4 多糖代谢基因微进化分析
27株嗜热链球菌在多糖代谢基因上存在组间显著差异,因此基于:麦芽糖糊精磷酸化酶(EC 2.4.1.1)、4-α-葡聚糖转移酶(EC 2.4.1.25)、1,4-α-葡聚糖(糖原)分支酶,GH-13型(EC 2.4.1.18)、糖原脱支酶(EC 3.2.1.-)/普鲁兰酶(EC 3.2.1.41)、麦芽糖/麦芽糖糊精ABC转运体底物结合周质蛋白MalE编码基因序联,构建系统发育树,结果显示,在多糖代谢基因的微进化关系中,并未发现明显的组内聚类(图3-a)。通过27株嗜热链球菌的核心基因构建系统发育树(图3-b),也并没有在2组间发现明显的进化关系。

a-多糖代谢基因系统发育树;b-菌株系统发育树
图3 27株嗜热链球菌多糖代谢基因系统发育树及菌株系统发育树
Fig.3 Phylogenetic tree of polysaccharide metabolism gene and strain of 27 S. thermophilus
注:A:麦芽糖糊精磷酸化酶(EC 2.4.1.1);B:4-α-葡糖苷转移酶(淀粉酶)(EC 2.4.1.25);C:糖原分支酶GH-13型(EC 2.4.1.18),D:糖原脱支酶(EC 3.2.1.-)/普鲁兰酶(EC 3.2.1.41);E:麦芽糖/麦芽糊精ABC转运体,底物结合周质蛋白MalE。
黑色字体为组一,红色字体为组二
2.5 碳水化合物活性酶分析
运用CAZy数据库对嗜热链球菌的碳水化合物活性酶功能基因进行注释[23]。27株嗜热链球菌共注释到37个碳水化合物活性酶基因家族,包括16个GH家族、9个 GT家族、6个CE家族、5个CBM家族和1个AA家族(图4)。其中,GT2家族在嗜热链球菌基因组中的拷贝数最多(平均约8个),其次是GT4(平均约6个),这2类酶与合成蔗糖等二糖和壳聚糖,脂多糖和纤维素有关。组间差异分析显示,GT8碳水化合物活性酶编码基因差异显著(P<0.05)。碳水化合物活性酶GT8属于糖基转移酶家族,目前对GT8的研究主要集中于植物领域[24],对原核生物的研究较少,其中包括脂多糖α-1,3-半乳糖基转移酶,肌醇1-α-半乳糖基转移酶(半乳糖醇合酶),同型半乳糖醛酸α-1,4-半乳糖基转移酶,葡萄糖苷α-1,3-半乳糖基转移酶,均与半乳糖的代谢有关。这一结果同2.1节中的表型数据相符,第一组中的嗜热链球菌均不可利用半乳糖,第二组中的嗜热链球菌都可以利用半乳糖。
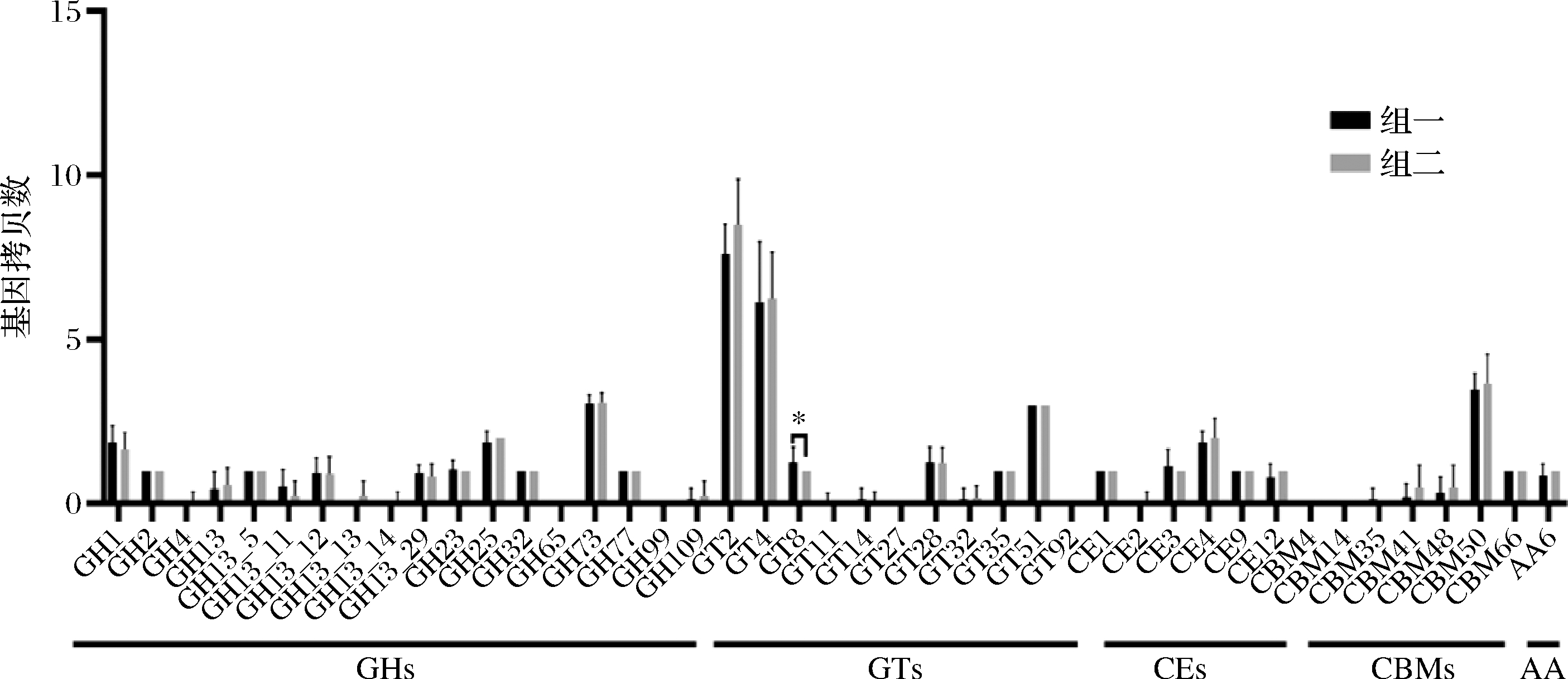
图4 27株嗜热链球菌碳水化合物活性酶功能基因统计
Fig.4 Carbohydrate active enzyme function gene statistics of 27 S. thermophilus strains
2.6 相关性分析
为了解27株嗜热链球菌表型与基因型的相关关系,最后进行表型与基因型的斯皮尔曼相关性分析。如图5-a所示,几丁质、N-乙酰葡萄糖氨基编码基因与菊粉、D-松三糖、木糖的利用呈正相关(P<0.05),木糖广泛存在于动物软骨素中,微生物对几丁质的有效降解通过几丁质酶(chitinase,EC 3.2.1.14)和几丁二糖酶,即β-N-乙酸氨基葡萄糖苷酶(β-N-acetylglucosaminidase,Nag,EC 3.2.1.52)等的协同作用得以实现[25]。研究表明,嗜热链球菌具有将N-乙酰葡萄糖胺转化为糖酵解,氨基酸代谢和肽聚糖生物合成底物,为细胞提供能量[26]。图5-b中显示淀粉的利用与基因家族GH13_14,CE2具有强相关性。研究显示GH表示糖苷水解酶,GH13家族亚家族14是作用于以有α-葡萄糖苷键为主要底物上的糖苷水解酶家族,其中α淀粉酶备受关注,α淀粉酶可以分解α-1,4-葡聚糖类型的多糖,如直链淀粉和支链淀粉[27]。

a-表型及基因相关性;b-碳水化合物活性酶及表型相关性
图5 基因与表型相关性分析
Fig.5 Correlation analysis between gene and phenotype
3 结论
27株嗜热链球菌均分离自自然发酵乳中,对其进行糖代谢表型实验,嗜热链球菌都表现出了对葡萄糖、乳糖、蔗糖的偏好性,但对于其他糖表现出了特异性,可明显将这27株嗜热链球菌归纳为2大组。接着对27株嗜热链球菌的全基因进行注释发现,组间在多糖代谢的功能基因上有显著差异(P<0.05),碳水化合物活性酶糖基转移酶中的GT8编码基因上差异显著(P<0.05),这2项差异分别与麦芽糖、半乳糖代谢在组间表型上的差异有关。最后用表型与RAST注释结果和CAZy注释结果分别进行相关性分析,得到几丁质、N-乙酰葡萄糖氨基编码基因与菊粉、D-松三糖、木糖的利用呈正相关,淀粉的利用与基因家族GH13_14、CE2具有强相关性。通过碳水化合物代谢表型及相关功能基因方面的分析,发现嗜热链球菌的麦芽糖代谢、半乳糖代谢、淀粉代谢与对应基因有密切关系,所以部分糖的代谢与其功能基因有一定的联系,其内部机制问题仍需探索。本实验可以为嗜热链球菌的基础生物研究和相关菌种的开发提供理论依据。
[1] 李鹏. 嗜热链球菌的功能性研究及其直投式发酵剂的开发应用[D].石家庄:河北科技大学, 2018.
LI P.Study on the functionality of Streptococcus thermophilus and the development and application of its DVS starter[D].Shijiazhuang:Hebei University of Science and Technology, 2018.
[2] 刘冬梅, 胡金双, 黄泳尧, 等.中国传统发酵乳中嗜热链球菌H2的培养及益生特性评价[J].华南理工大学学报(自然科学版), 2020, 48(9):133-140;148.
LIU D M, HU J S, HUANG Y Y, et al.Cultivation of Streptococcus thermophilus H2 and the evaluation on its prebiotic properties in Chinese traditional fermented milk[J].Journal of South China University of Technology (Natural Science Edition), 2020, 48(9):133-140;148.
[3] ZOU J, JIANG H, CHENG H, et al.Strategies for screening, purification and characterization of bacteriocins [J].International Journal of Biological Macromolecules, 2018, 117:781-789.
[4] 马立清, 张文羿, 孙天松.嗜热链球菌S4发酵乳代谢组学的研究[J].食品与发酵工业, 2021, 47(19):71-78.
MA L Q, ZHANG W Y, SUN T S.Metabolomics analysis of fermented milk with Streptococcus thermophilus S4[J].Food and Fermentation Industries, 2021, 47(19):71-78.
[5] 艾正文. 基于转录组学的Streptococcus thermophilus TF96中心碳代谢机制的研究[D].哈尔滨:东北农业大学, 2017.
AI Z W.Transcriptomics study on the mechanism of central carbon metabolism of Streptococcus thermophilus TF96[D].Harbin:Northeast Agricultural University, 2017.
[6] 张夙夙. 乳酸菌的乳糖/半乳糖代谢及其应用[D].济南:山东大学, 2020.
ZHANG S S.Lactose/galactose metabolism of lactic acid bacteria and its application[D].Jinan:Shandong University, 2020.
[7] 陆文伟. 全局转录调控因子CodY在嗜热链球菌中的调控机制研究[D].济南:山东大学, 2014.
LU W W.Study on the regulatory mechanism of global transcription regulator CodY in Streptococcus thermophilus[D].Jinan:Shandong University, 2014.
[8] 刘倩钰, 吴丽雯, 牛建军, 等.细菌磷酸转移酶系统(PTS)的组成与功能研究进展[J].微生物学通报, 2020, 47(7):2 266-2 277.
LIU Q Y, WU L W, NIU J J, et al.Research progress of the composition and function of bacterial phosphotransferase system[J].Microbiology China, 2020, 47(7):2 266-2 277.
[9] 季安营, 魏雪团.改造非磷酸转移酶葡萄糖转运途径强化解淀粉芽胞杆菌合成L-酪氨酸[J].食品与发酵工业, 2020, 46(15):27-31.
JI A Y, WEI X T.Engineering of the non-phosphotransferase glucose transport system for enhancing L-tyrosine production in Bacillus amyloliquefaciens[J].Food and Fermentation Industries, 2020, 46(15):27-31.
[10] ERKUS O, OKUKLU B, YENIDUNYA A F, et al.High genetic and phenotypic variability of Streptococcus thermophilus strains isolated from artisanal Yuruk yoghurts[J].LWT - Food Science and Technology, 2014, 58(2):348-354.
[11] MORANDI S, BRASCA M.Safety aspects, genetic diversity and technological characterisation of wild-type Streptococcus thermophilus strains isolated from north Italian traditional cheeses[J].Food Control, 2012, 23(1):203-209.
[12] WU C D, HUANG J, ZHOU R Q.Genomics of lactic acid bacteria:Current status and potential applications[J].Critical Reviews in Microbiology, 2017, 43(4):393-404.
[13] 赵洁.自然发酵乳中嗜热链球菌群体遗传学和功能基因组学研究[D].呼和浩特:内蒙古农业大学, 2018.
ZHAO J.Study on population genetics and functional genomics of Streptococcus thermophilus in naturally fermented milk[D].Hohhot:Inner Mongolia Agricultural University, 2018.
[14] 赵洁, 席晓霞, 张兴昌, 等.分子生物学法快速筛选具有优良产酸特性的嗜热链球菌[J].中国食品学报, 2018, 18(3):73-80.
ZHAO J, XI X X, ZHANG X C, et al.Rapid screening Streptococcus thermophilus strains with superior acid-producing character by molecular biology techniques[J].Journal of Chinese Institute of Food Science and Technology, 2018, 18(3):73-80.
[15] 安晓娜, 李伟程, 于洁, 等.比较基因组学分析不同来源罗伊氏乳杆菌基因多样性及生境适应性[J].微生物学报, 2020, 60(5):875-886.
AN X N, LI W C, YU J, et al.Comparative genomics analysis of genetic diversity and habitat adaptability of Lactobacillus reuteri from different sources[J].Acta Microbiologica Sinica, 2020, 60(5):875-886.
[16] AZIZ R K, BARTELS D, BEST A A, et al.The RAST Server:Rapid annotations using subsystems technology[J].BMC Genomics, 2008, 9:75.
[17] 董安利. 乳酸乳球菌乳酸亚种BL19的高密度培养研究[D].呼和浩特:内蒙古农业大学, 2019.
DONG A L.Study on high density culture of Lactococcus lactis subsp.lactis BL19[D].Hohhot:Inner Mongolia Agricultural University, 2019.
[18] SEEMANN T.Prokka:Rapid prokaryotic genome annotation[J].Bioinformatics, 2014, 30(14):2 068-2 069.
[19] PAGE A J, CUMMINS C A, HUNT M, et al.Roary:Rapid large-scale prokaryote pan genome analysis[J].Bioinformatics, 2015, 31(22):3 691-3 693.
[20] OVERBEEK R, OLSON R, PUSCH G D, et al.The SEED and the rapid annotation of microbial genomes using subsystems technology (RAST)[J].Nucleic Acids Research, 2014, 42(D1):D206-D214.
[21] GASSER C, GARAULT P, CHERVAUX C, et al.Co-utilization of saccharides in mixtures:Moving toward a new understanding of carbon metabolism in Streptococcus thermophilus[J].Food Microbiology, 2022, 107:104080.
[22] 姚纲, 张健鹏, 胡红焱, 等.用API50CH系统鉴定9株产高光学纯度D-乳酸野生菌株[J].中国实用医药, 2014, 9(8):263-265.
YAO G, ZHANG J P, HU H Y, et al.Identification of 9 wild strains producing high optical purity D-lactic acid by API50CH system[J].China Practical Medicine, 2014, 9(8):263-265.
[23] 韩晓龙. 赭曲霉全基因组测序和功能注释及其重要次级代谢产物生物合成基因簇的预测[D].广州:南方医科大学, 2017.
HAN X L.Genome sequencing and functional annotation of Aspergillus ochraceus and prediction of its important secondary metabolite biosynthesis gene cluster[D].Guangzhou:Southern Medical University, 2017.
[24] MIZUGUCHI K, DEANE C M, BLUNDELL T L, et al.HOMSTRAD:A database of protein structure alignments for homologous families[J].Protein Science:a Publication of the Protein Society, 1998, 7(11):2 469-2 471.
[25] 伍涵宇, 丁彦琴, 牛春, 等.微生物生产N-乙酰氨基葡萄糖的进展、应用及展望[J].轻工科技, 2021, 37(2):4-8.
WU H Y, DING Y Q, NIU C, et al.Progress, application and prospect of microbial production of N-acetylglucosamine[J].Light Industry Science and Technology, 2021, 37(2):4-8.
[26] GOH Y J, GOIN C, O’FLAHERTY S, et al.Specialized adaptation of a lactic acid bacterium to the milk environment:The comparative genomics of Streptococcus thermophilus LMD-9[J].Microbial Cell Factories, 2011, 10(Suppl 1):S22.
[27] 周亮亮. 柑橘溃疡病生防菌Bacillus velezensis QC-J全基因组测序及比较基因组学分析[D].赣州:江西理工大学, 2021.
ZHOU L L.Genome sequencing and comparative genomic analysis of Bacillus velezensis QC-J strain, a biocontrol bacterium against Citrus canker[D].Ganzhou:Jiangxi University of Science and Technology, 2021.