糖类、有机酸、油脂、醇类等是主要的发酵碳源,氨基酸主要作为氮源应用于工业发酵中。氨基酸由于其独特的结构,既可以进入中心代谢生成生物量,也可以通过脱氨反应为细胞提供氮源。氨基酸进入中心代谢通常需要经过多步催化,能量效率低且利用效率不高,是氨基酸成为微生物发酵碳源的主要限制因素。L-丝氨酸(L-serine)是组成蛋白质的基本氨基酸之一,可以通过L-丝氨酸脱水酶一步催化成丙酮酸进入中心代谢途径,因此,L-丝氨酸有望成为工业发酵中的备用碳源。L-丝氨酸参与细胞内多种代谢物的合成,也是合成L-半胱氨酸[1]、磷脂酰丝氨酸、L-丝氨酸等高附加值产物的重要前体物质,以丝氨酸为碳源进行微生物发酵或许可以提高L-半胱氨酸、磷脂酰丝氨酸的产量。目前,对丝氨酸合成代谢的研究较多[2-4],而对丝氨酸分解代谢的研究较少。优化丝氨酸代谢途径和效率,可以为丝氨酸的综合利用提供有益的支撑。并且丝氨酸的利用在某些特定的代谢途径,特别是本文中讨论的还原性甘氨酸途径,是限制性节点,具有重要的应用潜力。
巴斯德毕赤酵母(Pichia pastoris,即Komagataella phaffii)中丝氨酸的合成途径与酿酒酵母类似,由前体物质3-磷酸甘油酸经过三步反应合成L-丝氨酸(图1)。丝氨酸的分解途径有3条:1)经丝氨酸脱水酶(L-serine dehydratase,L-SerDH,EC 4.3.1.17)催化生成丙酮酸;2)在丙氨酸乙醛酸氨基转移酶(Alanine:glyoxylate aminotransferase,AGT,EC 2.1.6.45)催化下与乙醛酸反应生成羟基丙酮酸和甘氨酸;3)经丝氨酸羟甲基转移酶(serine hydroxymethyltransferase,SHMT,EC 2.1.2.1)催化生成甘氨酸和亚甲基四氢叶酸为巴斯德毕赤酵母的生长提供一碳单位。其中,丝氨酸脱水酶催化的分解途径最短,可以更快地连接中心代谢途径。除此之外,L-丝氨酸还参与L-半胱氨酸、L-色氨酸、磷脂、嘌呤和嘧啶等重要中间代谢物的生物合成[5-6]。
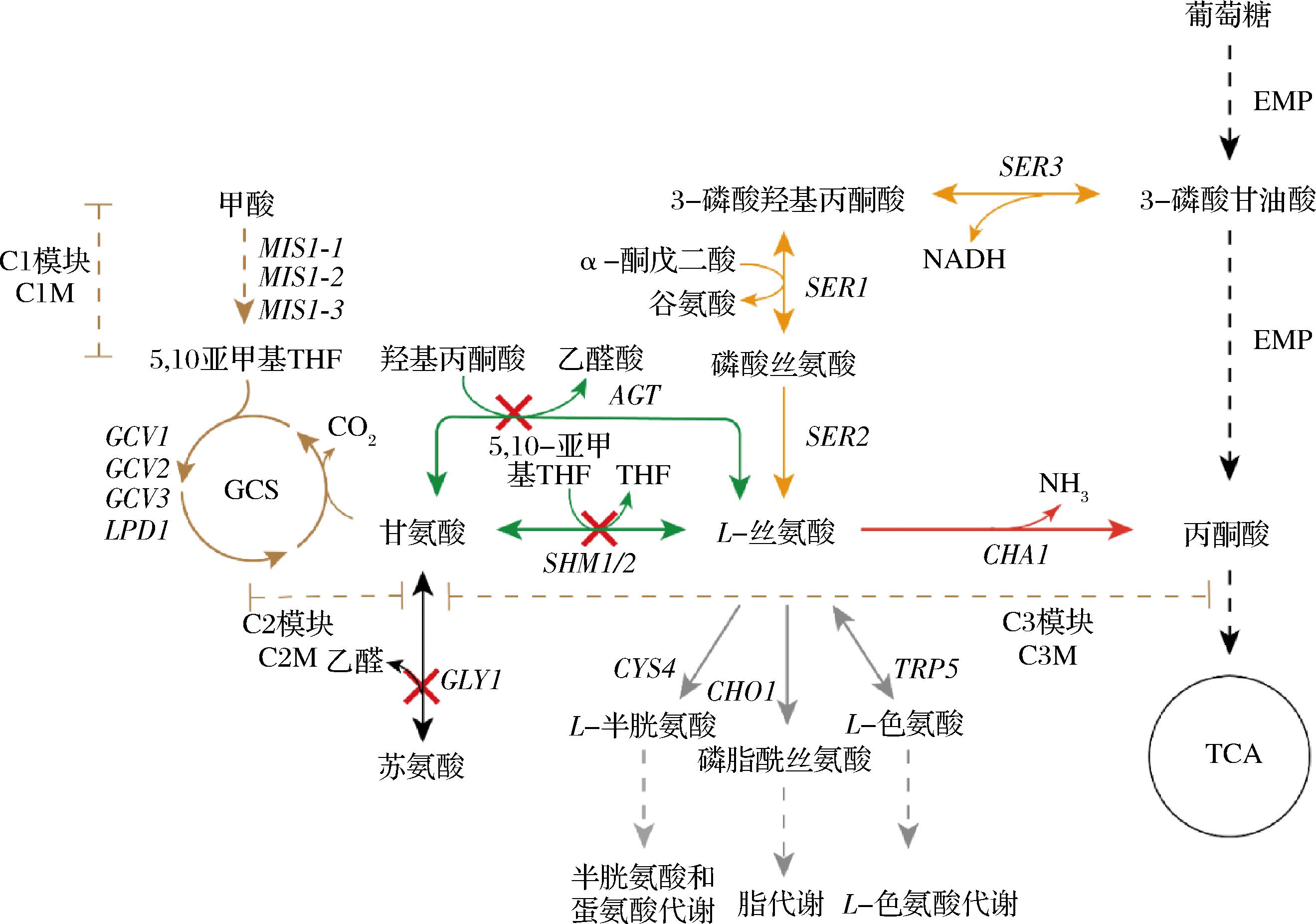
图1 巴斯德毕赤酵母丝氨酸代谢途径示意图
Fig.1 Diagram of the pathway of serine metabolism in Pichia pastoris
注:橙色箭头表示丝氨酸的合成途径;绿色和红色箭头表示3条丝氨酸分解代谢途径;棕色箭头和棕色虚线表示还原性甘氨酸途径;灰色箭头和化合物表示丝氨酸合成其他产物的代谢途径。
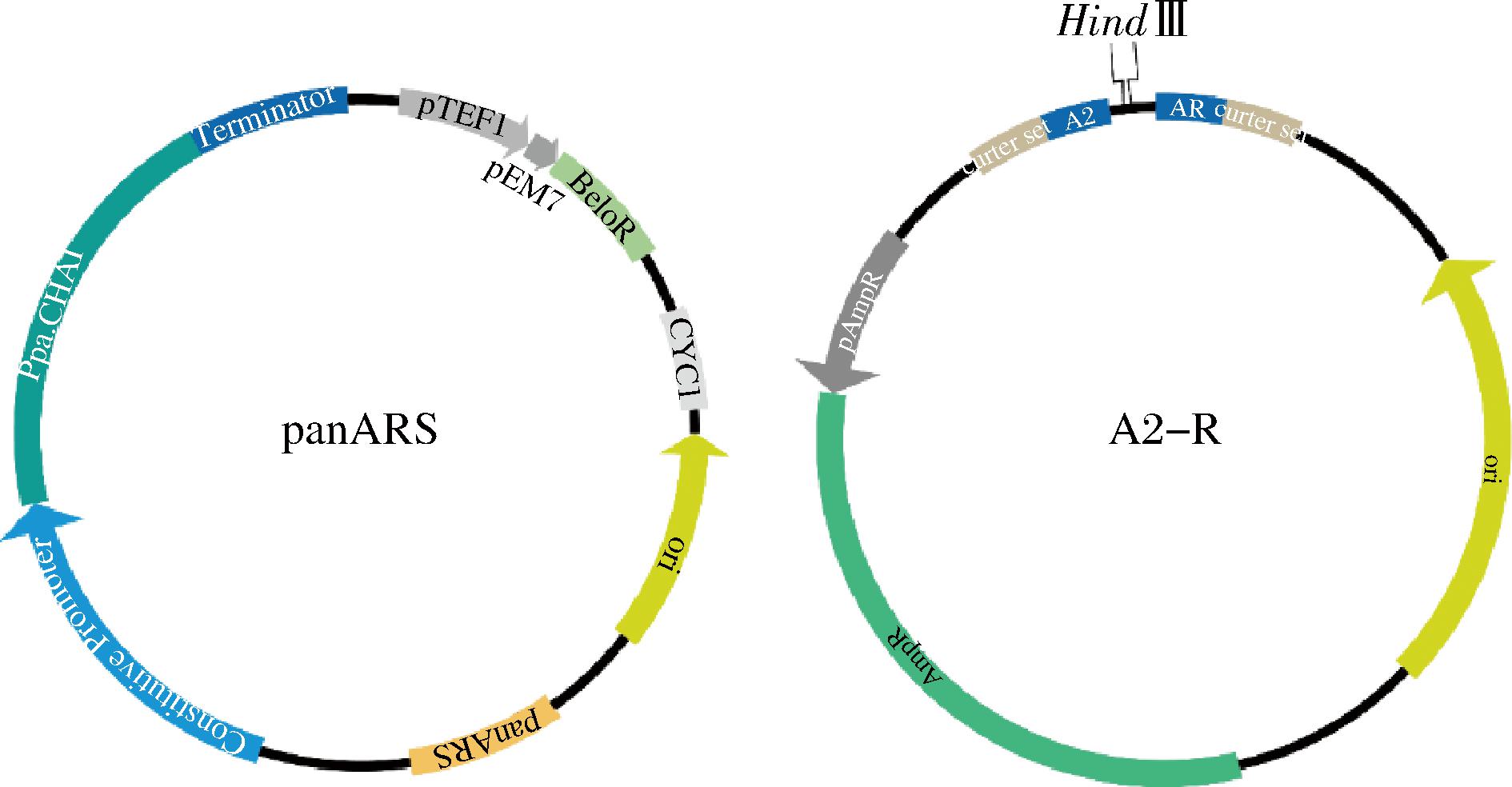
图2 panARS和A2-R质粒示意图
Fig.2 Diagram of panARS plasmid and A2-R plasmid
近年来,随着人们对可持续发展和环境问题的日益关注,CO2、甲醇和甲酸等一碳资源的第三代生物炼制可以在一定程度上减少对化石燃料和糖类的需求[7-8]。毕赤酵母可以利用甲醇为唯一碳源和能源生长,是一种很有潜力的一碳资源利用的底盘细胞[9],还被工程化用于CO2的固定[10]。因甲醇易燃易爆的缺点限制了其在大规模工业生产中的应用。还原性甘氨酸途径(reductive glycine pathway,rGlyP)是微生物利用甲酸进行有氧生长最有效的途径[2, 11],甲酸是有潜力替代甲醇的一碳原料。L-丝氨酸是三碳(C3)模块(C3M)的重要中间体(图1),是由甲酸生成的甘氨酸和连接中心碳代谢的丙酮酸之间的桥梁[12]。丝氨酸的利用效率已经成为该代谢途径的限制节点。本研究期望筛选出优良的L-丝氨酸脱水酶,促进丝氨酸高效生成丙酮酸,从而进入中心代谢途径,以维持甘氨酸营养缺陷型毕赤酵母的生长,为后续甲酸利用的代谢工程研究提供有价值的参考。
1 材料与方法
1.1 培养基及试剂
1.1.1 培养基
LLB(low-salt Luria-Bertani)培养基(g/L):酵母提取物 5,胰蛋白胨 10,NaCl 5,配制固体培养基时加入琼脂粉 20,博莱霉素工作质量浓度25 μg/mL,氨苄青霉素工作浓度100 μg/mL。酵母提取物胨葡萄糖(yeast extract peptone dextrose,YPD)培养基(g/L):酵母提取物 10,胰蛋白胨 20,葡萄糖 20,配制固体培养基时加入琼脂粉20。酵母提取物胨甲酵(yeast extract peptone methanol,YPM)培养基(g/L):酵母提取物 10,胰蛋白胨 20,甲醇 1%(体积分数)。
基础葡萄糖(minimal dextrose,MD)培养基(g/L):YNB 13.4,葡萄糖 20,培养甘氨酸营养缺陷型菌株时额外添加1 g/L的甘氨酸(以下称MDG培养基),培养野生型毕赤酵母及未回补组氨酸缺陷型的基因敲除菌时额外添加0.04 g/L组氨酸(以下称MDH培养基),配制固体培养基时加入琼脂粉 20。MDZ培养基(g/L):YNB(无硫酸铵) 3.4,硫酸铵 1,葡萄糖 20,50 μg/mL博莱霉素,配制固体培养基时加入琼脂粉 20。MMG(minimal methanol)培养基(g/L):YNB 13.4,甲醇 1%(体积分数),甘氨酸1。BMSGF(buffered minimal serine)培养基(g/L):磷酸钾缓冲液(K2HPO4 3.0, KH2PO4 11.8,pH 6.0),YNB 13.4,丝氨酸 10,甘氨酸 1,50 mmol/L甲酸钠。C1添加剂:0.3 mmol/L肌苷,0.3 mmol/L胸苷,0.3 mmol/L甲硫氨酸,6 μmol/L D-泛酸钙。
1.1.2 试剂
Hind Ⅲ核酸内切酶、PrimeSTAR Max DNA聚合酶,Takara;Rapid Taq Master Mix、Phanta Flash Master Mix、HiScript Ⅲ RT SuperMix for qPCR (+gDNA wiper)、ChamQ Universal SYBR qPCR Master Mix、质粒提取试剂盒、胶回收试剂盒,南京诺唯赞生物科技股份有限公司;MultiF Seamless Assembly Mix,ABclonal;超纯 RNA 提取试剂盒,北京康为世纪科技有限公司;甲酸钠、D-泛酸钙,上海麦克林生化科技有限公司;胸苷,北京百灵威科技有限公司;胰蛋白胨、酵母提取物、博莱霉素,Thermo Fisher Scientific;甘氨酸、YNB,生工生物工程股份有限公司;YNB(无硫酸铵),北京酷来搏科技有限公司;其他试剂均购自国药集团化学试剂有限公司。文中所需的引物均由苏州金唯智生物科技有限公司合成。
1.2 菌株及质粒
本研究使用大肠杆菌(Escherichia coli)JM109用于构建克隆,E.coli XL10-Gold用于sg RNA质粒的构建,上述两种菌均由实验室保存。
本研究所用质粒和菌株见表1。钩虫贪铜菌(Cupriavidus necator)和扭脱甲基杆菌(Methylobacterium extorquens)来源的丝氨酸脱水酶由江苏赛索飞生物科技有限公司合成。含有不同组成型启动子/终止子Ppa.CHA1的游离表达载体均以panARS-SfGFP质粒为模板构建,含不同物种来源的丝氨酸脱水酶表达载体均以A2-R质粒为模板构建,上述两种质粒均为实验室构建保存。
表1 菌株和质粒
Table 1 strains and plasmids

质粒/菌株特性来源AIO5-sgZeocin+实验室保存AIO5-sg PNS Ⅳ9Zeocin+,Ⅳ9位点sg质粒实验室保存AIO5-sg SHM1Zeocin+,SHM1基因 sg质粒实验室保存AIO5-sg SHM2Zeocin+,SHM2基因 sg质粒实验室保存AIO5-sg AGTZeocin+,AGT 基因sg质粒实验室保存A2-RAmpr实验室保存panARS-SfGFPZeocin+,pGCW14-SfGFP-AOX1tt实验室保存A2-R Ppa.CHA1Ampr,pADH2-Ppa.CHA1-ADH2tt本研究构建A2-R Ec.sdaAAmpr,pADH2-Ec.sdaA-ADH2tt本研究构建A2-R Ec.sdaBAmpr,pADH2-Ec.sdaB-ADH2tt本研究构建A2-R Ec.tdcGAmpr,pADH2- Ec.tdcG-ADH2tt本研究构建A2-R Sc.CHA1Ampr,pADH2-Sc.CHA1-ADH2tt本研究构建A2-R Ppu.tdcG1Ampr,pADH2-Ppu.tdcG1-ADH2tt本研究构建A2-R Ppu.tdcG3Ampr,pADH2-Ppu.tdcG3-ADH2tt本研究构建A2-R Cne.sdaAAmpr,pADH2-Cne.sdaA-ADH2tt本研究构建A2-R Me.sdaBAmpr,pADH2-Me.sdaB-ADH2tt本研究构建panARS-pGAP-1Zeocin+,pGAP-Ppa.CHA1-AOX1tt本研究构建panARS-pGCW14-1Zeocin+,pGCW14-Ppa.CHA1-AOX1tt本研究构建panARS-pTEF1-1Zeocin+,pTEF1-Ppa.CHA1-AOX1tt本研究构建panARS-pTEF1(Ppa)-1Zeocin+,pTEF1(Ppa)-Ppa.CHA1-AOX1tt本研究构建panARS-pGPM1-1Zeocin+,pGPM1-Ppa.CHA1-AOX1tt本研究构建panARS-pADH2-1Zeocin+,pADH2-Ppa.CHA1-AOX1tt本研究构建panARS-pTPI1-1Zeocin+,pTPI1-Ppa.CHA1-AOX1tt本研究构建panARS-pPGK1-1Zeocin+,pPGK1-Ppa.CHA1-AOX1tt本研究构建panARS-pSSA4-1Zeocin+,pSSA4-Ppa.CHA1-AOX1tt本研究构建panARS-pGAP-2Zeocin+,pGAP-Ppa.CHA1-ADH2tt本研究构建panARS-pGCW14-2Zeocin+,pGCW14-Ppa.CHA1-ADH2tt本研究构建panARS-pTEF1-2Zeocin+,pTEF1-Ppa.CHA1-ADH2tt本研究构建panARS-pTEF1(Ppa)-2Zeocin+,pTEF1(Ppa)-Ppa.CHA1-ADH2tt本研究构建panARS-pGPM1-2Zeocin+,pGPM1-Ppa.CHA1-ADH2tt本研究构建panARS-pADH2-2Zeocin+,pADH2-Ppa.CHA1-ADH2tt本研究构建panARS-pTPI1-2Zeocin+,pTPI1-Ppa.CHA1-ADH2tt本研究构建panARS-pPGK1-2Zeocin+,pPGK1-Ppa.CHA1-ADH2tt本研究构建panARS-pSSA4-2Zeocin+,pSSA4-Ppa.CHA1-ADH2tt本研究构建P.pastoris GS115his-实验室保存GS115P.pastoris GS115:PNS Ⅰ 6 pGAP-Ppa.Rad52-AOX1tt实验室保存GS115-△GLY1GS115, △GLY1实验室保存GS115-△SHM1GS115, △SHM1本研究构建GS115-△SHM2GS115, △SHM2本研究构建GS115-△AGTGS115, △AGT本研究构建
续表1

质粒/菌株特性来源GS115-△GLY1△AGTGS115, △AGT本研究构建GS115-4△GS115, his+,△GLY1△AGT△SHM1△AGT实验室保存GS115-Ppa.CHA1 1-1P.pastoris GS115,游离质粒表达pGAP-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-2P.pastoris GS115,游离质粒表达pGCW14-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-3P.pastoris GS115,游离质粒表达pTEF1-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-4P.pastoris GS115,游离质粒表达pTEF1(Ppa)-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-5P.pastoris GS115,游离质粒表达pGPM1-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-6P.pastoris GS115,游离质粒表达pADH2-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-7P.pastoris GS115,游离质粒表达pTPI1-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-8P.pastoris GS115,游离质粒表达pPGK1-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 1-9P.pastoris GS115,游离质粒表达pSSA4-Ppa.CHA1-AOX1tt本研究构建GS115-Ppa.CHA1 2-1P.pastoris GS115,游离质粒表达pGAP-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-2P.pastoris GS115,游离质粒表达pGCW14-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-3P.pastoris GS115,游离质粒表达pTEF1-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-4P.pastoris GS115,游离质粒表达pTEF1(Ppa)-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-5P.pastoris GS115,游离质粒表达pGPM1-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-6P.pastoris GS115,游离质粒表达pADH2-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-7P.pastoris GS115,游离质粒表达pTPI1-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-8P.pastoris GS115,游离质粒表达pPGK1-Ppa.CHA1-ADH2tt本研究构建GS115-Ppa.CHA1 2-9P.pastoris GS115,游离质粒表达pSSA4-Ppa.CHA1-ADH2tt本研究构建GS115-4△ Ppa.CHA1GS115-4△,PNS Ⅳ9 pADH2-Ppa.CHA1-ADH2tt本研究构建GS115-4△ Ec.sdaAGS115-4△,PNS Ⅳ9 pADH2-Ec.sdaA-ADH2tt本研究构建GS115-4△ Ec.sdaBGS115-4△,PNS Ⅳ9 pADH2-Ec.sdaB-ADH2tt本研究构建GS115-4△ Ec.tdcGGS115-4△,PNS Ⅳ9 pADH2- Ec.tdcG-ADH2tt本研究构建GS115-4△ Sc.CHA1GS115-4△,PNS Ⅳ9 pADH2-Sc.CHA1-ADH2tt本研究构建GS115-4△ Ppu.tdcG1GS115-4△,PNS Ⅳ9 pADH2-Ppu.tdcG1-ADH2tt本研究构建GS115-4△ Ppu.tdcG3GS115-4△,PNS Ⅳ9 pADH2-Ppu.tdcG3-ADH2tt本研究构建GS115-4△ Cne.sdaAGS115-4△,PNS Ⅳ9 pADH2-Cne.sdaA-ADH2tt本研究构建GS115-4△ Me.sdaBGS115-4△,PNS Ⅳ9 pADH2-Me.sdaB-ADH2tt本研究构建
1.3 方法
1.3.1 质粒构建
Ppa.CHA1游离表达质粒均通过Gibson assembly方法构建。以panARS-SfGFP质粒为模板进行反向PCR得到质粒骨架。通过PCR得到的Ppa.CHA1基因、9种组成型启动子和2种终止子经DNA纯化后,使用MultiF Seamless Assembly Mix与质粒骨架进行Gibson assembly。随后转化至E.coli JM109中,通过菌落PCR验证并挑取阳性克隆至液体培养基中培养。提取质粒并测序验证正确的质粒进行下一步实验。
不同物种来源的丝氨酸脱水酶表达质粒使用相同的方法构建,其质粒骨架是A2-R质粒通过Hind Ⅲ酶切获得的。大肠杆菌、酿酒酵母和恶臭假单胞菌来源的丝氨酸脱水酶是分别以E.coli JM109、酿酒酵母S288C、恶臭假单胞菌KT2440基因组为模板通过PCR扩增得到的,而C.necator和M.extorquens来源的丝氨酸脱水酶是以赛索飞合成得到的pUC57克隆质粒为模板扩增得到的。
1.3.2 菌株构建
不同强度组成型启动子游离表达Ppa.CHA1菌株的构建:直接将测序正确的500 ng游离表达质粒转化至P.pastoris GS115感受态细胞中,转化平板形成单克隆菌株后挑取无绿色荧光的单克隆菌株至YPD液体培养基中,随后提取基因组并进行PCR验证,选择验证正确的菌株进行后续的蛋白表达实验。
使用实验室构建的CRISPR-Cas9基因编辑系统(实验结果未发表)完成GS115-△SHM1、GS115-△SHM2、GS115-△AGT、GS115-△GLY1△AGT和9种不同来源的丝氨酸脱水酶过表达菌株的构建,使用CRISPR-Cas9基因编辑系统进行基因敲入的方法,具体如下:
首先,以GS115菌株的基因组为模板通过PCR扩增无义位点PNS Ⅳ9上下游各1 000 bp的同源臂片段,从9种不同来源的丝氨酸脱水酶表达质粒上进行PCR扩增得到完整的基因表达框,然后使用上游同源臂的正向引物和下游同源臂的反向引物进行Overlap PCR。得到的融合片段经DNA纯化后与1 μg AIO5-sg PNS Ⅳ9质粒混合后转化至GS115-4△感受态细胞中(图3)。提取基因组进行PCR验证正确的菌株通过在YPM/MMG培养基中诱导12 h消除sg RNA质粒。基因敲除菌株的构建只需将上下游同源臂进行一次双片段的Overlap PCR,与目标基因的sg RNA质粒混合转化至相应的酵母感受态细胞中,其余与上述基因敲入的构建方法一致。
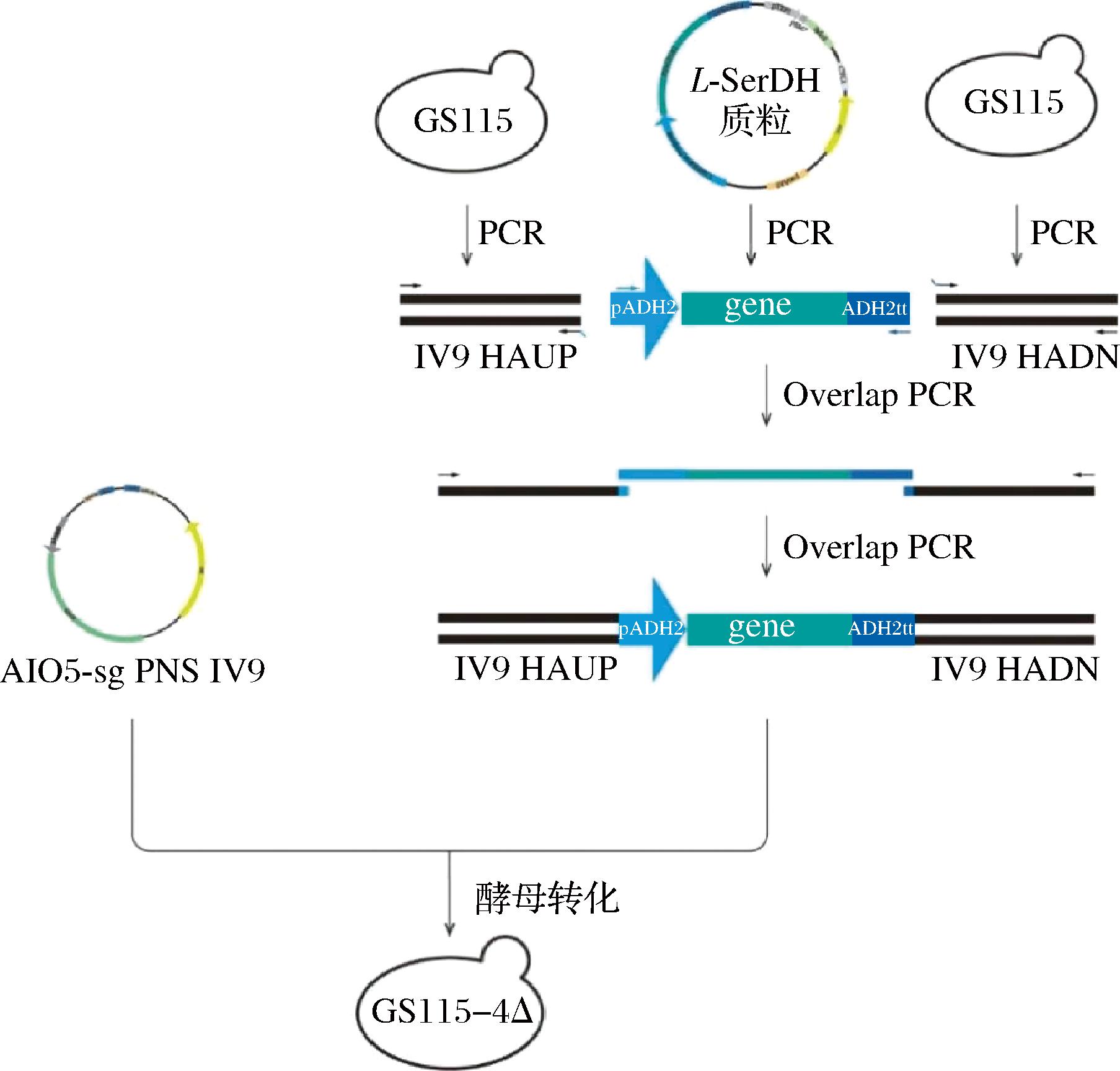
图3 不同来源L-SerDH敲入菌株构建示意图
Fig.3 Diagram of constructing strains knocking in different sources L-SerDH
1.3.3 丝氨酸耐受性实验
将20 μL P.pastoris GS115及4种单敲菌的甘油菌液接种至2 mL YPD培养基,30 ℃、200 r/min下培养36 h;而 GS115-4△和GS115-4△::Ec.tdcG需接种至MDG培养基中培养96 h。使用多功能酶标仪(Synergy H1,BioTek)测定菌液OD600,收集OD600=1菌液, 10 000 r/min离心5 min,弃上清液后用1 mL 无菌水重悬菌体,用无菌水依次稀释10倍、100倍和1 000倍。各取1 μL 稀释菌液点板至含不同浓度丝氨酸的MDH+C1(MDH培养基中添加C1添加剂),GS115-4△和GS115-4△::Ec.tdcG需要点板至含不同浓度丝氨酸的MDG或MDG+F(MDG培养基中添加50 mmol/L甲酸钠),平板正置于30 ℃恒温培养箱,培养3~6 d。
1.3.4 SDS-PAGE分析及Western-Blot分析
收集18种游离表达Ppa.L-SerDH的工程毕赤酵母发酵菌液于破碎管中,10 000 r/min离心5 min,加入1~2勺酸性玻璃珠,设置生物样品均质器(OMNI)速度为2 m/s,破碎30 s 停止30 s,循环6次,进行酵母细胞破碎。随后12 000 r/min离心10 min,上清液即破碎后的粗蛋白样。
使用BCA蛋白浓度测定试剂盒(YEASEN)测定粗蛋白样浓度,取2~5 mg/mL 蛋白与适量上样缓冲液混合,沸水浴10 min 变性。使用免封闭PAGE凝胶快速制备试剂盒(YEASEN)进行蛋白胶制备(浓缩胶和分离胶浓度均为12.5%)。取10 μL处理好的样品点入凝固的蛋白胶孔中,120 V 电泳约1.5 h。电泳结束后,用考马斯亮蓝R-250染色液进行染色,脱色后用凝胶成像系统拍照。将未染色的蛋白胶用胶板切下目标条带处的胶,将蛋白转印至聚偏二氟乙烯(polyvinylidene fluoride,PVDF)膜上。转膜完毕后,使用1% 脱脂牛奶封闭1 h。含His-HRP抗体(PROTEINTECH)的脱脂牛奶,摇动孵育1 h。抗体孵育完成后,使用TBST缓冲液洗膜3次,在转印膜表面加适量显影液(Tanon)后使用化学发光成像仪(Tanon)进行显影。
1.3.5 实时荧光定量(real-time quantitative PCR,RT-qPCR)
将GS115-4△和9种敲入不同来源丝氨酸脱水酶的工程菌以1%的比例接种于2 mL MDG培养基中,30 ℃、200 r/min下培养4 d。离心收集菌体,加入2 mL BMSGF培养基诱导48 h后离心取菌体。使用超纯RNA提取试剂盒提取GS115-4△和9种工程菌的总RNA。使用HiScript Ⅲ RT SuperMix for qPCR (+gDNA wiper)试剂盒逆转录1 μg RNA,获得cDNA片段。
使用NBCI网站设计各种丝氨酸脱水酶的RT-qPCR引物。qPCR 反应液的组成:5 μL 2×ChamQ Universal SYBR qPCR Master Mix、0.2 μL 10 μmol/L 的正向及反向引物、50 ng cDNA 和 ddH2O 组成,总体积为 10 μL,在 StepOnePlus RT-qPCR 系统上进行反应。
1.3.6 工程菌生长曲线测定
将GS115-4△和9种敲入不同来源丝氨酸脱水酶编码基因的工程菌株接至5 mL MDG培养基中进行活化,离心收集菌体后,加入5 mL BMSGF培养基短期实验室适应性进化24 h后,用多功能酶标仪测菌液OD600,取OD600=16的菌液离心去上清液,用2 mL 无菌水重悬,以2.5%接种量至20 mL BMSGF培养基中,每2 d取1次菌液,使用多功能酶标仪测量OD600。
2 结果与分析
2.1 巴斯德毕赤酵母丝氨酸耐受性
丝氨酸的积累会影响E.coli的生长和分裂[13]。MUNDHADA等[14]发现敲除3个编码L-丝氨酸脱水酶基因(sdaA、sdaB、tdcG)会导致生产丝氨酸的大肠杆菌生物量显著下降。此外,在基本培养基中添加244 μmol/L丝氨酸即可抑制枯草芽孢杆菌168(Bacillus subtilis 168)的生长, 敲除一个丝氨酸转运蛋白(YbeC)和两个苏氨酸转运蛋白(BcaP和YbxG)后,B. subtilis 168对丝氨酸耐受性达100 mmol/L[15]。目前,尚未发现有关丝氨酸抑制巴斯德毕赤酵母生长的报道。本研究对巴斯德毕赤酵母进行了丝氨酸耐受性实验,将不同稀释浓度的GS115菌株点板至含不同浓度丝氨酸的MDH+C1培养基(添加C1添加剂)考察其生长状况。图4结果表明GS115菌株可以耐受20 g/L的丝氨酸。

图4 各重组菌株的丝氨酸耐受性
Fig.4 Serine tolerance of various recombinant strains
GS115-4△(基因型为GS115-△GLY1△AGT△SHM1/2)是实验室构建的一株甘氨酸营养缺陷型毕赤酵母菌株。GS115-4△不能在基本培养基中生长,需额外添加0.1%的甘氨酸可以恢复生长。对4种基因的单敲菌和GLY1与AGT的双敲菌进行丝氨酸耐受性实验,发现GS115-△SHM2的生长受限,说明毕赤酵母胞内丝氨酸主要通过SHMT2催化生成甘氨酸进行分解代谢。推测SHMT2缺失后,甘氨酸合成效率下降,进一步引起5,10-亚甲基四氢叶酸和一碳单位的生成不足。GS115-4△阻断了其余两条丝氨酸分解途径,其在丝氨酸为唯一碳源下的生长完全依赖于L-丝氨酸脱水酶催化产生丙酮酸,进入中心代谢产生生物量。因此,加强GS115-4△中丝氨酸到丙酮酸的转化,可以增加丝氨酸的同化效率,将丝氨酸用于菌株生长。
2.2 内源丝氨酸脱水酶表达元件的选择
为得到能表达毕赤酵母内源L-SerDH的表达元件,在P.pastoris GS115中使用9种不同强度的组成型启动子和2种终止子组合(表2)对内源L-SerDH进行游离表达。内源L-SerDH蛋白大小约为36.2 kDa,受毕赤酵母胞内其他蛋白的影响,无法通过SDS-PAGE分辨L-SerDH的表达效果(图5-a);Western Blot结果显示,ADH2启动子和ADH2终止子的组合对内源L-SerDH表达效果最好(图5-b)。
表2 组成型启动子和终止子
Table 2 Constructive promoters and terminators
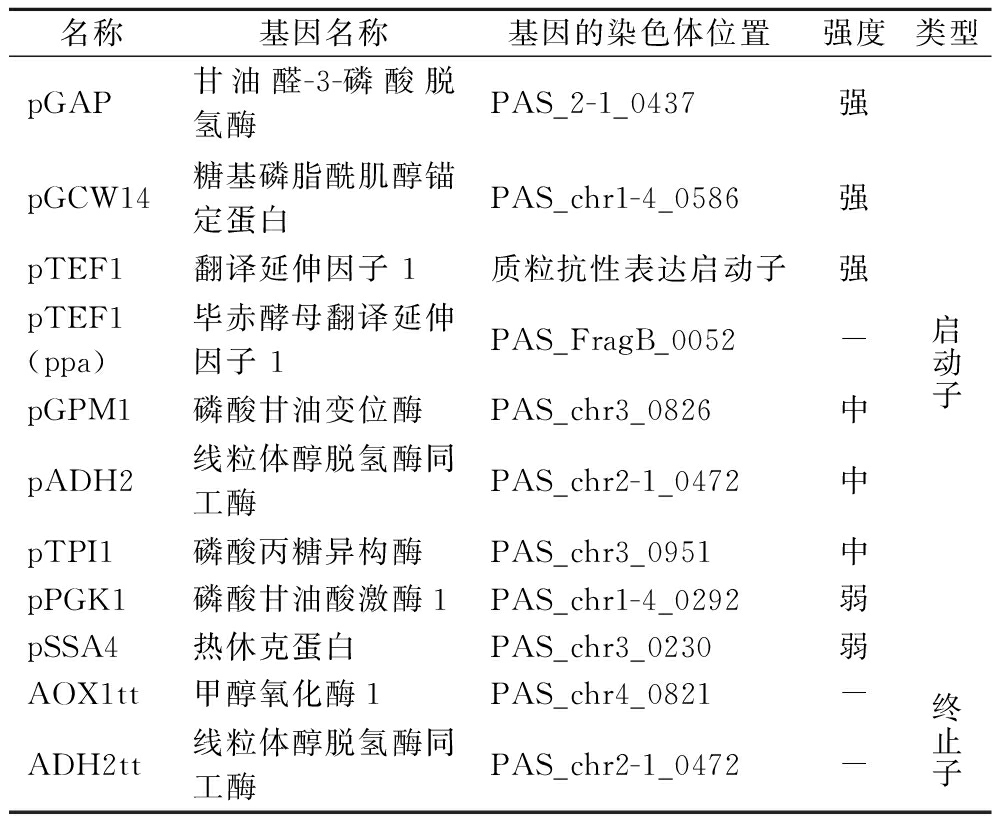
名称基因名称基因的染色体位置强度类型pGAP甘油醛-3-磷酸脱氢酶PAS_2-1_0437强pGCW14糖基磷脂酰肌醇锚定蛋白PAS_chr1-4_0586强pTEF1翻译延伸因子 1质粒抗性表达启动子强pTEF1(ppa)毕赤酵母翻译延伸因子 1PAS_FragB_0052-pGPM1磷酸甘油变位酶PAS_chr3_0826中pADH2线粒体醇脱氢酶同工酶PAS_chr2-1_0472中pTPI1磷酸丙糖异构酶PAS_chr3_0951中pPGK1磷酸甘油酸激酶1PAS_chr1-4_0292弱pSSA4热休克蛋白PAS_chr3_0230弱启动子AOX1tt甲醇氧化酶1PAS_chr4_0821-ADH2tt线粒体醇脱氢酶同工酶PAS_chr2-1_0472-终止子
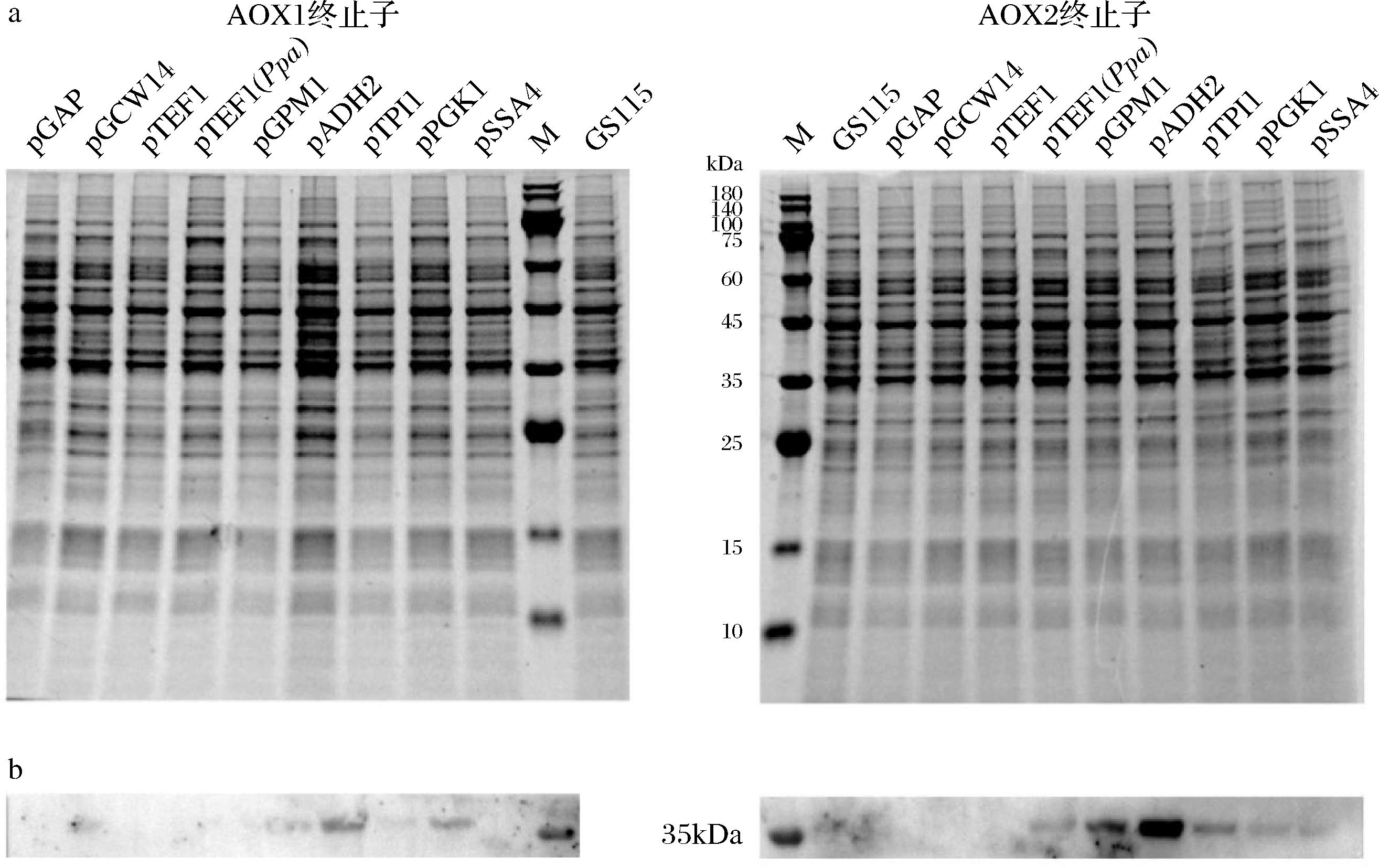
a-SDS-PAGE;b-Western印迹分析
图5 不同启动子和终止子对毕赤酵母L-SerDH表达影响
Fig.5 SDS-PAGE and Western-Blot analysis of the effect of different promoters and terminators on P.pastoris L-SerDH expression
2.3 不同物种来源丝氨酸脱水酶转录验证
利用CRISPR-Cas9技术在GS115-4△的Ⅳ9无义位点(PNS Ⅳ9)[16]处分别敲入9种L-SerDH(L-SerDH基因来源如表3所示)。在BMSGF培养基中诱导48 h,检测9种L-SerDH的CT值,计算每株菌L-SerDH的△CT值(△CT= CT(L-SerDH)-CT(ACT))。图6的结果显示, GS115-4△::Ppa.CHA1菌株内源CHA1比GS115-4△菌株内源CHA1的转录有所提高。除Me.sdaB未检测到CT值外,其余7种外源L-SerDH均检测到了CT值,说明7种外源L-SerDH都得到了转录表达,但转录水平并不一致。
表3 L-丝氨酸脱水酶来源
Table 3 L-Serine dehydratase source

基因名称物种来源获取方式参考文献Ppa.CHA1Ec.sdaAEc.sdaBEc.tdcGSc.CHA1Ppu.tdcG1Ppu.tdcG3Cne.sdaAMe.sdaB巴斯德毕赤酵母大肠杆菌酿酒酵母恶臭假单胞菌钩虫贪铜菌扭脱甲基杆菌基因组PCR生物公司合成-[17]-[18][19][12]
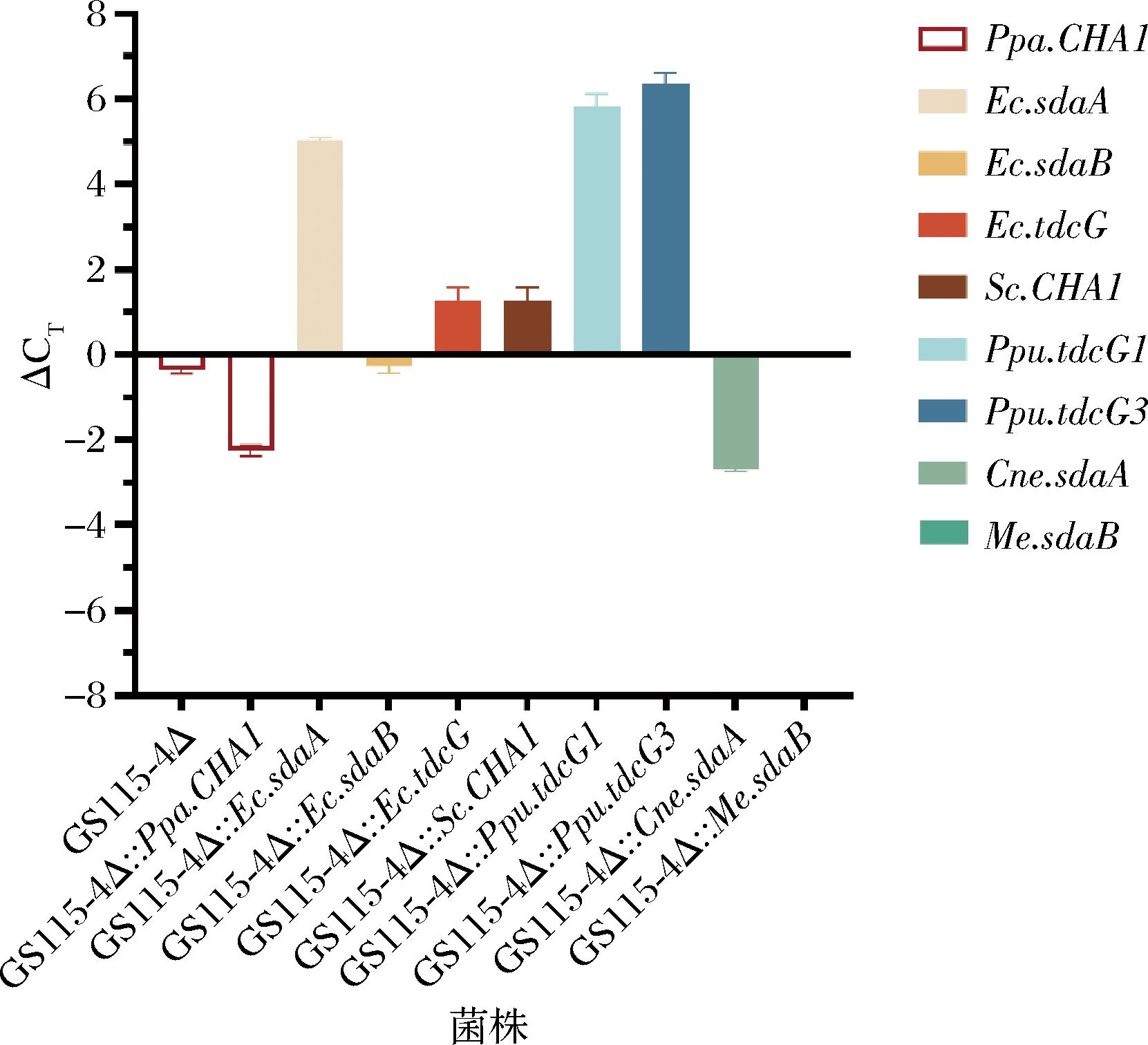
图6 不同来源L-丝氨酸脱水酶转录△CT值
Fig.6 Transcriptional ΔCT values of L-serine dehydratase from different sources
2.4 异源L-丝氨酸脱水酶的表达对毕赤酵母丝氨酸生长的影响
对GS115-4△在以丝氨酸作为唯一碳源的基本培养基(BMSG培养基)中的生长表型进行验证,不同浓度丝氨酸的BMSG培养基中都未观察到生长(图7-a)。推测是因为丝氨酸羟甲基转移酶(SHM1/2)的敲除阻断了丝氨酸和甘氨酸的相互转化,导致一碳单位供应不足而影响了生长,与图4结果一致。
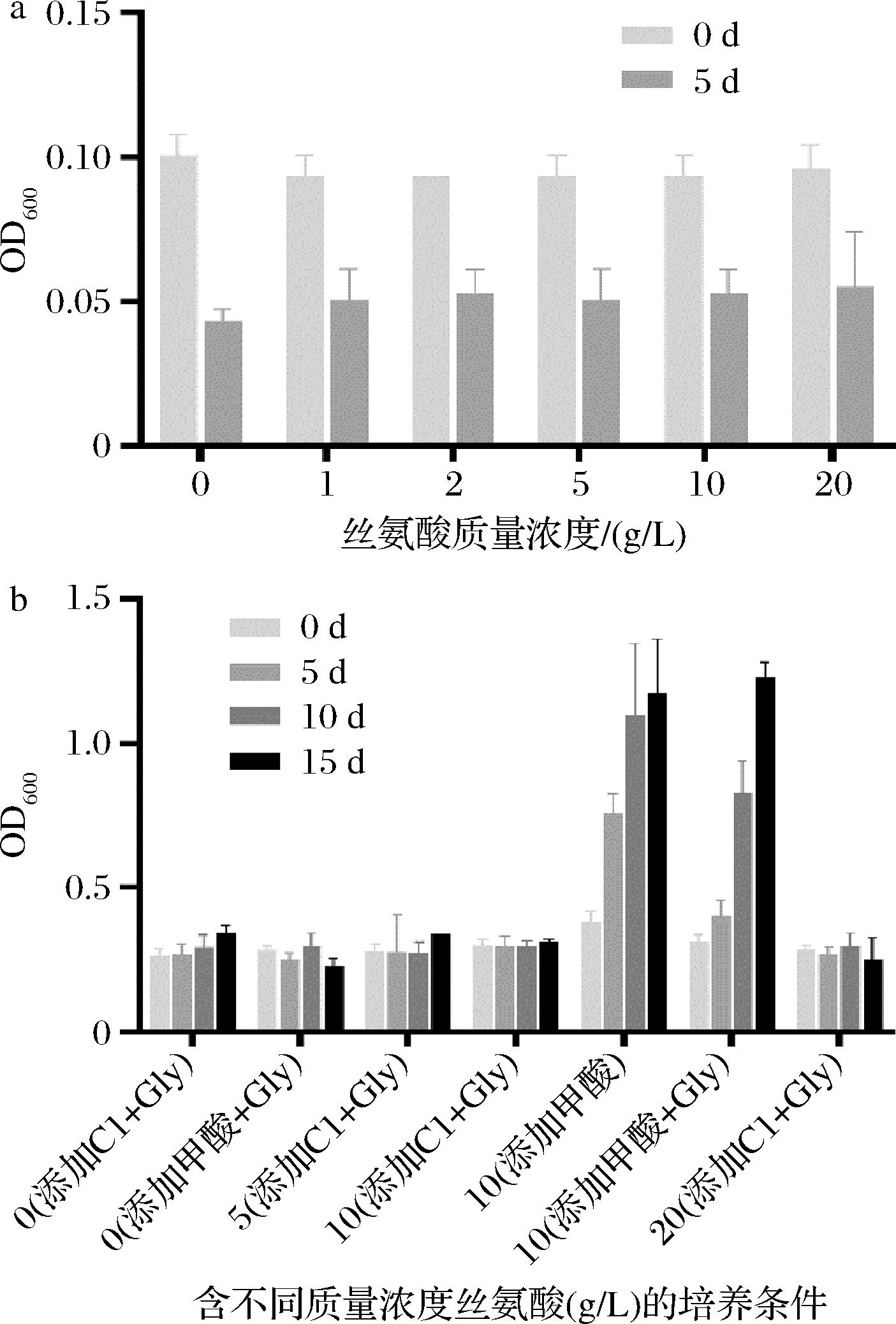
a-GS115-4△在不同浓度丝氨酸BMSG中的生长情况;b-GS115-4△在添加C1添加剂或甲酸钠的丝氨酸培养条件下的生长情况
图7 甘氨酸营养缺陷型菌株丝氨酸生长情况
Fig.7 Growth of glycine auxotroph strain on serine
图7-b显示培养基中添加甘氨酸不足以维持生长,说明甘氨酸的添加不能产生足够的一碳单位支撑丝氨酸的利用。所以,在培养基中额外补充C1添加剂(添加量见方法1.1.1节)或者甲酸钠(终浓度为50 mmol/L)来解除一碳单位对GS115-4△丝氨酸生长的限制。结果显示C1添加剂的补充无法恢复菌株的生长,而甲酸钠的补充恢复了GS115-4△的丝氨酸生长表型,且添加甲酸钠的效果最优。说明甲酸钠在体内代谢产生的一碳单位种类更丰富,能更好地支撑菌体的生长。
GS115-4△及9种L-SerDH敲入菌按1.3.6节所述的培养方法测定其在BMSGF培养基中的生长曲线,结果如图8所示,GS115-4△::Ec.tdcG展示出了优于出发菌株的丝氨酸生长特性,终OD600是GS115-4△的1.6倍。GS115-4△::Ec.sdaA终OD600是GS115-4△的1.3倍,但GS115-4△::Ec.sdaA的迟滞期有所延长。其余7种工程菌的生长情况弱于出发菌株GS115-4△。说明Ec.tdcG是编码最优的丝氨酸脱水酶。
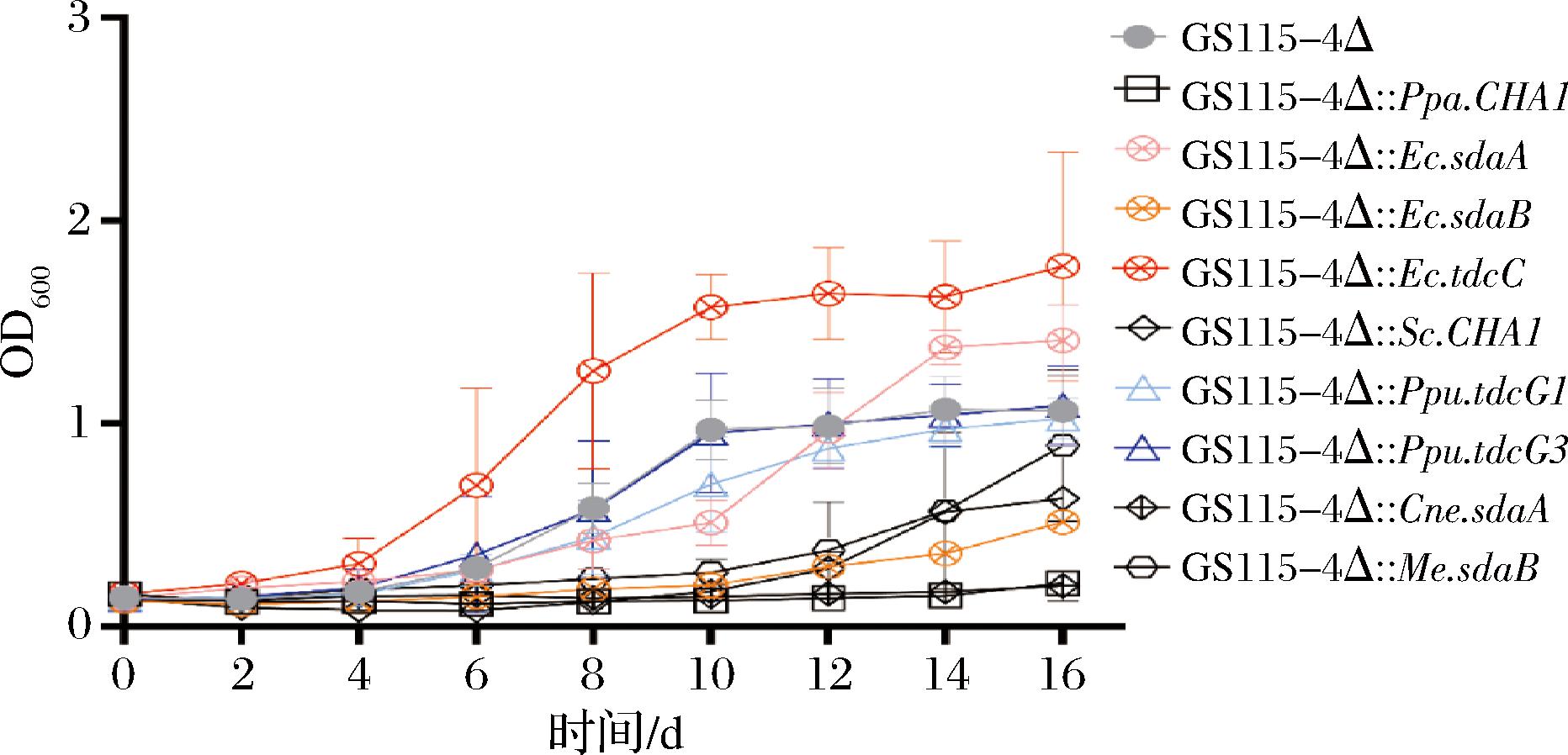
图8 L-丝氨酸脱水酶表达工程毕赤酵母在丝氨酸下的生长曲线
Fig.8 Growth curves of engineered P.astoris expressing L-serine dehydratase on serine
GS115-4△点板实验的结果显示(图9),其在含高浓度丝氨酸的MDG平板上无法生长,而GS115-4△::Ec.tdcG在含高浓度丝氨酸的MDG平板上生长良好,说明大肠杆菌来源的基因可以有效催化丝氨酸生成丙酮酸,快速利用丝氨酸,减少因丝氨酸积累而导致的菌株生长受限。在培养基中补充50 mmol/L的甲酸钠,GS115-4△恢复了对高浓度丝氨酸的耐受性。
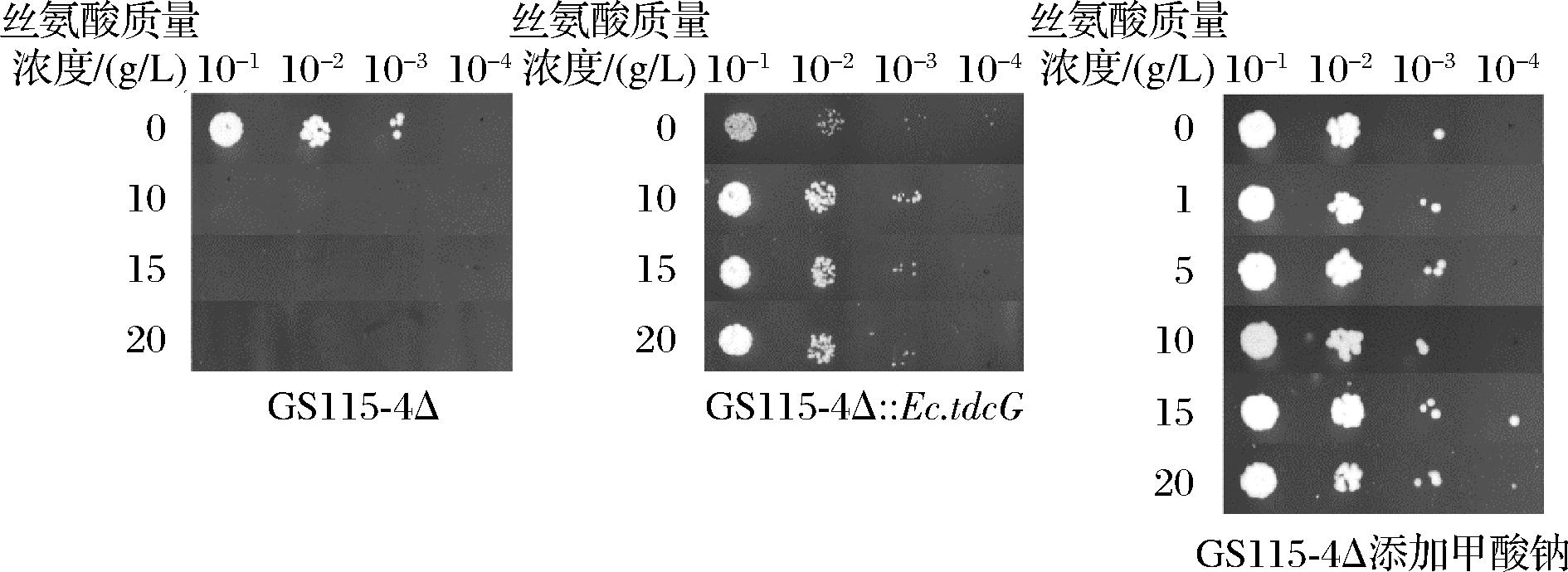
图9 GS115-4△和GS115-4△::Ec.tdcG的丝氨酸耐受性
Fig.9 Serine tolerance of GS115-4△ and GS115-4△::Ec.tdcG
3 结论与讨论
本研究通过丝氨酸耐受性点板实验发现巴斯德毕赤酵母可以耐受20 g/L的丝氨酸。GLY1、AGT、SHM1的单独敲除和GLY1与AGT双敲除对毕赤酵母的丝氨酸耐受性不产生影响。但SHM2的单独敲除会影响毕赤酵母在基本培养基平板的生长,且其生长不随丝氨酸浓度的变化而改变。推测SHM2主要催化丝氨酸合成甘氨酸和亚甲基四氢叶酸,其敲除会导致毕赤酵母因甘氨酸或C1单位合成不足而生长受限。图4结果也说明了毕赤酵母中SHMT1和SHMT2催化方向不同。HAMA等[20]和TAZUYA-MURAYAMA等[21]报道,丝氨酸可以抑制高丝氨酸脱氢酶Ⅰ 和预苯酸脱水酶从而影响高丝氨酸和L-苯丙氨酸的合成,影响E.coli的生长。高浓度丝氨酸抑制了GS115-4△的生长,添加甲酸后恢复生长,推测高浓度丝氨酸或许抑制了某种酶的活性而影响了GS115-4△的生长,通过添加甲酸缓解丝氨酸耐受性。
通过对毕赤酵母内源L-SerDH启动子和终止子的优化,筛选得到了有利于内源L-SerDH表达的组成型启动子(pADH2)和终止子(ADH2tt)。尚不清楚使用最优的表达元件表达不同来源的L-SerDH其转录水平却有很大差异(图6)的原因。GS115-4△在以丝氨酸为唯一碳源时不能生长,但是仅添加甲酸钠即可恢复其在丝氨酸中的生长(图7-b),推测甲酸对一碳单位和甘氨酸的补充效果更优。在GS115-4△中敲入表达9种不同来源的L-丝氨酸脱水酶,发现Ec.tdcG编码的L-丝氨酸脱水酶使GS115-4△以L-丝氨酸为碳源时生长的生物量是出发菌株生物量的1.6倍。
本研究聚焦于毕赤酵母经L-丝氨酸脱水酶催化的L-丝氨酸的碳源利用性,弥补了毕赤酵母在L-丝氨酸利用方面研究的空白,并有望为在毕赤酵母中丝氨酸的综合利用提供更优的L-丝氨酸脱水酶来源。
[1] XU G Q, ZHANG X M, XIAO W H, et al.Production of L-serine and its derivative L-cysteine from renewable feedstocks using Corynebacterium glutamicum:Advances and perspectives[J].Critical Reviews in Biotechnology, 2024, 44(3):448-461.
[2] DE LA CRUZ J G, MACHENS F, MESSERSCHMIDT K, et al.Core catalysis of the reductive Glycine pathway demonstrated in yeast[J].ACS Synthetic Biology, 2019, 8(5):911-917.
[3] RENNIG M, MUNDHADA H, WORDOFA G G, et al.Industrializing a bacterial strain for L-serine production through translation initiation optimization[J].ACS Synthetic Biology, 2019, 8(10):2347-2358.
[4] ZHU Q J, ZHANG X M, LUO Y C, et al.L-Serine overproduction with minimization of by-product synthesis by engineered Corynebacterium glutamicum[J].Applied Microbiology and Biotechnology, 2015, 99(4):1665-1673.
[5] 康培. 代谢工程改造大肠杆菌glyA敲除菌株提高L-丝氨酸产量[D].天津:天津大学, 2016.KANG P.Increased L-serine production by metabolic engineering of glyA knockout E.coli[D].Tianjin:Tianjin University, 2016.
[6] ZHANG X M, XU G Q, SHI J S, et al.Microbial production of L-serine from renewable feedstocks[J].Trends in Biotechnology, 2018, 36(7):700-712.
[7] 王凯, 刘子鹤, 陈必强, 等.微生物利用二氧化碳合成燃料及化学品:第三代生物炼制[J].合成生物学, 2020, 1(1):60-70.WANG K, LIU Z H, CHEN B Q, et al.Microbial utilization of carbon dioxide to synthesize fuels and chemicals:Third-generation biorefineries[J].Synthetic Biology, 2020, 1(1):60-70.
[8] 程真真, 张健, 高聪, 等.代谢工程改造微生物利用甲酸研究进展 [J].合成生物学, 2023, 4(4):756-778.CHENG Z Z, ZHANG J, GAO C, et al.Progress in metabolic engineering of microorganisms for the utilization of formate[J].Synthetic Biology, 2023, 4(4):756-778.
[9] GUO F, QIAO Y Y, XIN F X, et al.Bioconversion of C1 feedstocks for chemical production using Pichia pastoris[J].Trends in Biotechnology, 2023, 41(8):1066-1079.[10] GASSLER T, SAUER M, GASSER B, et al.The industrial yeast Pichia pastoris is converted from a heterotroph into an autotroph capable of growth on CO2[J].Nature Biotechnology, 2020, 38(2):210-216.
[11] BAR-EVEN A, NOOR E, FLAMHOLZ A, et al.Design and analysis of metabolic pathways supporting formatotrophic growth for electricity-dependent cultivation of microbes[J].Biochimica et Biophysica Acta, 2013, 1827(8-9):1039-1047.
[12] KIM S, LINDNER S N, ASLAN S, et al.Growth of E.coli on formate and methanol via the reductive glycine pathway[J].Nature Chemical Biology, 2020, 16(5):538-545.
[13] ZHANG X, NEWMAN E.Deficiency in L-serine deaminase results in abnormal growth and cell division of Escherichia coli K-12[J].Molecular Microbiology, 2008, 69(4):870-881.
[14] MUNDHADA H, SCHNEIDER K, CHRISTENSEN H B, et al.Engineering of high yield production of L-serine in Escherichia coli[J].Biotechnology and Bioengineering, 2016, 113(4):807-816.
[15] KLEWING A, KOO B M, KRÜGER L, et al.Resistance to serine in Bacillus subtilis:Identification of the serine transporter YbeC and of a metabolic network that links serine and threonine metabolism[J].Environmental Microbiology, 2020, 22(9):3937-3949.
[16] CAI P, DUAN X P, WU X Y, et al.Recombination machinery engineering facilitates metabolic engineering of the industrial yeast Pichia pastoris[J].Nucleic Acids Research, 2021, 49(13):7791-7805.
[17] BANG J, HWANG C H, AHN J H, et al.Escherichia coli is engineered to grow on CO2 and formic acid[J].Nature Microbiology, 2020, 5(12):1459-1463.
[18] TURLIN J, DRONSELLA B, DE MARIA A, et al.Integrated rational and evolutionary engineering of genome-reduced Pseudomonas putida strains promotes synthetic formate assimilation[J].Metabolic Engineering, 2022, 74:191-205.
[19] CLAASSENS N J, BORDANABA-FLORIT G, COTTON C A R, et al.Replacing the Calvin cycle with the reductive glycine pathway in Cupriavidus necator[J].Metabolic Engineering, 2020, 62:30-41.
[20] HAMA H, SUMITA Y, KAKUTANI Y, et al.Target of serine inhibition in Escherichia coli[J].Biochemical and Biophysical Research Communications, 1990, 168(3):1211-1216.
[21] TAZUYA-MURAYAMA K, ARAMAKI H, MISHIMA M, et al.Effect of L-serine on the biosynthesis of aromatic amino acids in Escherichia coli[J].Journal of Nutritional Science and Vitaminology, 2006, 52(4):256-260.